Method Article
Одномолекулярная флуоресцентная визуализация динамики ДНК-полимеразы в G-квадруплексах
В этой статье
Резюме
Этот протокол описывает анализ репликации одной молекулы ДНК на основе флуоресцентной микроскопии, позволяющий в режиме реального времени визуализировать взаимодействия между ДНК-полимеразами и препятствиями, такими как G-квадруплексные структуры.
Аннотация
Способность белков, участвующих в репликации эукариотической ДНК, преодолевать препятствия, такие как «препятствия» на белках и ДНК, имеет решающее значение для обеспечения достоверной дупликации генома. G-квадруплексы — это структуры нуклеиновых кислот более высокого порядка, которые образуются в богатых гуанином областях ДНК и, как было показано, действуют как препятствия, мешая путям поддержания генома. В этом исследовании представлен метод на основе флуоресцентной микроскопии в реальном времени для наблюдения за взаимодействиями ДНК-полимеразы со структурами G-квадруплекса. Короткие, праймированные олигонуклеотиды ДНК, содержащие G-квадруплекс, были иммобилизованы на функционализированных стеклянных покровных стеклах в микрофлюидной проточной ячейке. Были введены флуоресцентно меченые ДНК-полимеразы, что позволило контролировать их поведение и стехиометрию с течением времени. Этот подход позволил наблюдать поведение полимеразы при его остановке из-за G-квадруплекса. В частности, с помощью флуоресцентно меченых δ дрожжевой полимеразы было обнаружено, что при встрече с G-квадруплексом полимераза проходит непрерывный цикл связывания и рассвязывания. Этот анализ одной молекулы может быть адаптирован для изучения взаимодействий между различными белками, поддерживающими ДНК, и препятствиями на субстрате ДНК.
Введение
ДНК-полимеразы — это ферменты, которые катализируют включение нуклеозидтрифосфатов для дублирования ДНК 1,2,3,4,5. Таким образом, они играют ключевую роль в основных процессах поддержания ДНК, включая репликацию ДНК 6,7,8 и репарацию 9,10,11,12. ДНК-полимеразы должны точно и эффективно реплицировать геном, чтобы обеспечить целостность генома, предотвращая накопление мутаций в геноме. Во время синтеза полимеразы часто сталкиваются с «препятствиями», такими как связанные с ДНК белки или вторичные структуры ДНК13. Эти препятствия могут замедлить или даже заблокировать прогрессирование полимеразы14. Преодоление этих препятствий важно для обеспечения достоверной дупликации генома, поскольку невыполнение этого требования может привести к нестабильности генома15,16.
Одним из основных классов препятствий являются G-квадруплексы (G4), неканонические вторичные структуры ДНК, которые, как было показано, формируются в богатых гуанином последовательностях в геноме человека. В геноме человека существует более 700 000 различных последовательностей, способных образовывать G4, включая области внутри теломер ипромоторы онкогенов. Эти структуры ДНК принимают различные конформации в зависимости от последовательности нуклеотидов, длины и связанного катиона металла 19,20,21. Это разнообразие означает, что полимеразы должны преодолевать ряд различных топологий G4, потенциально с разной степенью эффективности. Было показано, что неспособность полимераз преодолеть или обойти структуру G4 препятствует прогрессированию репликационной вилки in vivo, что приводит к геномной нестабильности22. Исследования in vitro показали, что структуры G4 могут останавливать или полностью блокировать дрожжевые полимеразы 23,24,25,26,27. Способность структур G4 останавливать или блокировать ДНК-полимеразы полностью зависит от их кинетической и термодинамической стабильности, при этом некоторые полимеразы способны разворачивать определенные G428. В то время как эти исследования дают представление о способности полимераз преодолевать препятствия G-квадруплекса, им не хватает возможности непосредственно визуализировать поведение полимеразы при встрече с G4. Судьба полимераз — остаются ли они связанными, отпадают или динамически обмениваются — определяет, какие последующие процессы доступны для разрешения G4.
В этом исследовании был разработан флуоресцентный анализ одномолекулярной микроскопии для прямой визуализации и мониторинга связывания ДНК-полимеразы со структурами G4 в режиме реального времени. Этот анализ включает в себя привязку G4-образующих матриц ДНК к биотинилированному покровному стеклу в микрофлюидной проточной ячейке, куда могут быть введены флуоресцентно меченные ДНК-полимеразы для инициирования синтеза ДНК. Измеряя флуоресценцию полимераз с течением времени, можно непосредственно наблюдать их поведение при столкновении со структурой G4. Структура G4, обнаруженная в онкогене рака c-MYC , была выбрана для этого анализа из-за ее высокого уровня стабильности. Этот протокол теперь может быть адаптирован для каталогизации поведения различных полимераз во всех областях жизни, связанных с различными топологиями и стабильностью G4. Этот анализ предлагает инновационный и высокопроизводительный подход к выяснению механизмов, с помощью которых ДНК-полимеразы преодолевают препятствия ДНК, обеспечивая мощный инструмент для углубления понимания динамики полимеразы.
протокол
Подробная информация об используемых реагентах и оборудовании приведена в Таблице материалов.
1. Спектроскопия кругового дихроизма
ПРИМЕЧАНИЕ: Перед разработкой анализа необходимо было провести спектроскопию кругового дихроизма (CD) на выбранной последовательности G4, чтобы обеспечить правильное сворачивание. Последовательность 22 nt (5'-TGAGGGTGGGGGGAGGGGGGGAA-3') образует структуру G4, полученную из онкогена рака c-MYC . Спектроскопия БК также проводилась на контрольной последовательности (5'-TGAGTGTGAAGACGATGATAGAA -3), в которой изменены ключевые нуклеотиды, предотвращающие образование G4.
- Для подготовки ДНК-матриц для CD-спектроскопии добавьте 40 мкл 100 мкМ из 22 нт-матриц в 360 мкл буфера Tris-HCl (pH 8,0), этилендиаминтетрауксусной кислоты (ЭДТА) (TE) с добавлением 200 мМ хлорида калия (KCl). Для экспериментов с помощью CD-спектроскопии требуется конечная концентрация ДНК в 10 мкМ.
Примечание: Присутствие ионов K+ имеет важное значение для стабилизации структуры G4 в матрице. - Нагрейте растворы ДНК до 95 °C в цифровой сухой ванне в течение 15 минут. После завершения медленно охладите растворы в течение ночи, поместив нагревательный блок в полистирол. Этот медленный процесс охлаждения отвечает за складывание G4.
- Перенесите 400 мкл растворов для формирования G4 и контрольной последовательности в кварцевую кювету длиной 0,1 см с помощью газонепроницаемого шприца. Наполняйте кювету медленно, чтобы избежать образования пузырьков воздуха.
- Измерьте спектр CD в диапазоне от 200 нм до 400 нм с помощью спектрополяриметра при температуре 25 °C. Параметры и условия для этого измерения были описаны ранее29. Результаты можно найти на дополнительном рисунке 1.
ПРИМЕЧАНИЕ: Убедитесь, что вы записали спектр CD, чтобы буфер действовал как пустой.
2. Подготовка шаблонов ДНК
ПРИМЕЧАНИЕ: Матрицы, которые могут быть воспроизведены, представляют собой короткие, 100 nt загрунтованные линейные матрицы, изготовленные с использованием стандартных методов молекулярной биологии. G4-образующий шаблон (5'-GAATTACATTTAAATTTTACAGATACAGTCAATGAGAA
CTTCCAGGCGTAACGAGAGCACGGGGGGGGGGGGT
GGGACCTTAGCTTCGAGTTCCGAT-3') содержит последовательность, способную формировать в своем центре G-квадруплекс, полученный из MYC (из стадии 1). Шаблон управления (5'-GAATTACATTTAAATTTTACACAGATACAGTCAATGAGA
ACTTCCAGGCGTAACGAGAGCACGATGTAGCAGAACT
GTGACCTTAGCTTCGAGTTCCGAT-3′) имеет ту же область управления (из шага 1) также в своем центре.
- Для отжига праймера на G4-образующие и контрольные матрицы ДНК смешайте 20 мкМ праймера (5'-Biotin-AF647-TCTTAATGTAAAAATGTC-3') с 20 мкМ 100 нт матриц в TE-буфере с добавлением 200 мМ KCl.
- Нагрейте матричные растворы ДНК до 95 °C в цифровой сухой ванне в течение 15 минут. После завершения медленно охладите растворы в течение ночи, поместив нагревательный блок в полистирол, чтобы обеспечить складывание G4 и отжиг двух нитей.
3. Мечение ДНК-полимеразы с помощью AF647
- Маркировка полимеразы красителем
Примечание: Дрожжевой Pol δ экспрессировали и очищали, как описано ранее30. Затем полимеразу необходимо было пометить монореактивным эфирным красителем Alexa Fluor NHS (AF647), чтобы обеспечить визуализацию на уровне одной молекулы. Это было завершено в соответствии с предыдущими описаниями5.- Для мечения N-конца инкубируют 3-кратный избыток красителя AF647 с 320 мкл 58,5 мкМ Pol δ в буфере, содержащем 30 мМ Tris-HCl (pH 7,6), 2 мМ дитиотреитола (DTT), 300 мМ натрия хлорида (NaCl), 50 мМ глутамата калия и 10% (v/v) глицерина в течение 10 мин при 4 °C. Осторожно поворачивайте раствор, чтобы обеспечить тщательное перемешивание и повысить эффективность этикетирования.
Примечание: Белковая последовательность может содержать дополнительные первичные амины в аминокислотах лизина. Регулируя условия реакции, такие как pH, метка может быть преимущественно прикреплена к N-концу в отличие от остатков лизина. Для этого может потребоваться дальнейшая оптимизация для повышения эффективности маркировки. - Для приготовления спиновых обессоливающих колонн объемом 0,5 мл (отсечение молекулярной массы 7 К) сначала центрифугируйте колонны при давлении 1500 x g в течение 1 минуты, чтобы удалить накопительный раствор. Выбросьте растворы, нанесенные в сборную пробирку после центрифугирования.
ПРИМЕЧАНИЕ: Обязательно ослабляйте крышку спиновых колонн во время этапов центрифугирования. Это обеспечит надлежащую элюировку колонн. - Уравновесьте колонки в буфере, содержащем 30 мМ Tris-HCl (pH 7,6), 300 мМ NaCl, 3 мМ DTT, 50 мМ глутамата калия и 5% (v/v) глицерина, добавив 300 мкл буфера в верхнюю часть колонки. Центрифугируйте колонку при давлении 1500 x g в течение 1 минуты и выбросьте проточный поток. Повторите этот процесс еще два раза.
- Нанесите на колонку по 200 мкл каждого образца Pol δ. Центрифугируйте колонки при давлении 1500 x g в течение 2 мин и соберите очищенный Pol δ в микроцентрифужную пробирку объемом 1,5 мл.
- Заморозьте очищенные фракции в жидкости N2 и храните аликвоты при температуре -80 °C, чтобы предотвратить деградацию белка с течением времени.
- Для мечения N-конца инкубируют 3-кратный избыток красителя AF647 с 320 мкл 58,5 мкМ Pol δ в буфере, содержащем 30 мМ Tris-HCl (pH 7,6), 2 мМ дитиотреитола (DTT), 300 мМ натрия хлорида (NaCl), 50 мМ глутамата калия и 10% (v/v) глицерина в течение 10 мин при 4 °C. Осторожно поворачивайте раствор, чтобы обеспечить тщательное перемешивание и повысить эффективность этикетирования.
- Определение эффективности маркировки
- Загрузите 2 мкл меченого AF647 образца Pol δ на УФ-видимый спектрофотометр31.
ПРИМЕЧАНИЕ: Сначала убедитесь, что был измерен бланк буфера Pol δ. - Измерьте длины волн возбуждения 650 нм для красителя AF647 и 280 нм для δ Pol с помощью спектрофотометра.
- Используя закон Бера-Ламберта и молярные коэффициенты экстинкции 270 000 см-1 М-1 для красителя AF647 и 195 960 см-1 М-1 для полимеразы, рассчитать эффективность мечения.
ПРИМЕЧАНИЕ: Эффективность мечения, определенная для меченых AF647 дрожжей Pol δ, используемых в этом анализе, была рассчитана примерно на 67%.
- Загрузите 2 мкл меченого AF647 образца Pol δ на УФ-видимый спектрофотометр31.
4. Анализ удлинения ансамбля праймера
Примечание: Перед проведением экспериментов по репликации одной молекулы необходимо подтвердить, что ДНК-полимераза блокируется G-квадруплексом, с помощью анализов объемной репликации.
- Установите температуру нагревательного блока в цифровой сухой бане на 30 °C. Это обеспечит максимальную активность дрожжей Pol δ.
- Готовьте «мастер-миксы» для G4-образующих и контрольных матриц в репликационном буфере (25 мМ Tris-HCl (pH 7,6), 10 мМ ацетата магния, 50 мМ глутамата калия, 40 мкг/мл бычьего сывороточного альбумина (BSA), 0,1 мМ ЭДТА, 5 мМ DTT и 0,0025% (v/v) Tween-20). Дополните каждую смесь 1 мМ DTT, 250 мкМ dTTP, dCTP, dATP, dGTP (dNTP) и 10 нМ соответствующей матрицы ДНК (G4-образующей или контрольной). Поместите их в нагревательный блок, чтобы они достигли 30 °C.
- Закалите 12 μL каждой исходной смеси с 12 μL буфера загрузки формамида (80% (w/v) формамида, 10 мМ ЭДТА) в качестве регулятора T = 0 мин.
- Чтобы начать синтез ДНК, добавьте меченные AF647 дрожжи Pol δ в каждую мастер-смесь (конечная концентрация 20 нМ). Тщательно перемешайте, чтобы ДНК-полимеразы воспроизводили шаблоны с максимальной эффективностью.
ПРИМЕЧАНИЕ: Держите дрожжи Pol δ на льду во время использования, чтобы сохранить активность ферментов. - Через 30 с, 60 с и 180 с удалите по 12 мкл каждой мастер-смеси и охладите 12 мкл буфера для загрузки формамида. Соотношение 1:1 исходной смеси и буфера загрузки формамида гарантирует, что реакция репликации не может продолжаться после гашения.
- Извлеките теперь закаленные растворы из нагревательного блока при температуре 30 °C и поместите их в нагревательный блок с температурой 98 °C для денатурации дцДНК до одноцепочечной ДНК. По истечении 10 мин поместите весь нагревательный блок в резервуар для геля для загрузки растворов в денатурирующий 15% трис-борат-ЭДТА-ЭДТА (КЭ)-мочевинный полиакриламид (PAGE) гель32.
ПРИМЕЧАНИЕ: Гель PAGE необходимо нагреть перед загрузкой образцов, запустив гель-электрофорез при напряжении 180 В в течение 30 минут в буфере 1x TBE. По завершении промойте каждую лунку, чтобы удалить мочевину. - Загрузите 15 μL каждого образца в гель вместе с лестницей, содержащей 0,02 μM меченых олигонуклеотидов 20 nt, 60 nt и 100 nt в буфере для загрузки формамида.
- Запустите гель в течение 60 минут при напряжении 180 В в буфере 1x TBE, чтобы обеспечить полное разделение.
- Визуализируйте гель в канале Cy5 биомолекулярного имиджера. Это позволит измерить реплицированную цепь ДНК, содержащую метку AF-647.
5. Одномолекулярная флуоресцентная микроскопия
- Функционализация покровного стекла
ПРИМЕЧАНИЕ: Чтобы прикрепить матрицы ДНК к стеклянному покровному стеколу, его необходимо сначала функционализировать аминосиланом, а затем связать биотинилированные молекулы ПЭГ. Этот процесс сводит к минимуму неспецифические взаимодействия между ДНК и/или белками и поверхностью.- Очистите покровные стекла (24 x 24 мм), вставив их в банки для окрашивания и засыпав этанол. Протрите банки ультразвуком в течение 30 минут, прежде чем промыть покровные листы чистой водой. Повторите этот процесс с 1 М гидроксида калия (KOH) перед повторным полосканием. Повторите эти действия по очистке еще раз.
- Промойте и наполните новую банку ацетоном и поместите внутрь покровные листы. Распылите 3-аминопропилтриэтоксисилан в банку с образованием 2% v/v раствора. Помешивайте банку в течение 3 минут до того как погасить реакцию большим избытком воды.
- Высушите покровные стекла с помощью N2 и поместите их по отдельности на наполненный водой влажный ящик. Это предотвратит высыхание покровных стекол.
- Приготовьте эфир Biotin-PEG-SVA:mPEG-SVA в соотношении 1:25 в 100 мМ NaHCO3 (pH 8,2). Ввергните эту смесь в течение 20 с, чтобы обеспечить тщательное перемешивание.
- Нанесите 40 μл раствора PEG на сухую защитную пленку. Поместите сверху еще одну защитную крышку, чтобы «зажать» раствор между двумя защитными крышками. Это позволит функционализировать грани покровного стекла с внутренней стороны. Повторите для всех покровных накладок.
- Инкубируйте покровные стекла в темноте в течение 3 часов. После инкубации разделите покровные пары, промойте их лишней водой и высушите сжатым газом N2 .
- Повторите шаги 4.1.4-4.1.5, чтобы нанести второй слой PEG на покровные стекла. Убедитесь, что ранее функциональные стороны сложены вместе, поэтому будьте осторожны, чтобы случайно не перевернуть их на этапах стирки. Инкубируйте растворы в темноте в течение ночи, прежде чем промыть и высушить их.
- Храните покровные стекла в вакууме, чтобы сохранить их функциональность. Функциональные покровные стекла стабильны в течение месяца при правильном хранении.
- Подготовка микрофлюидных проточных ячеек
Примечание: Микрофлюидная проточная ячейка33 сконструирована для экспериментов с одной молекулой (см. дополнительный рисунок 2). Это позволит буферам, матрицам ДНК и белкам вступать в контакт с функционализированным покровным стеклом (см. шаг 5.1).- Извлеките функционализированный покровный колпак из вакуума и поместите его на штатив для микропробирок, частично заполненный водой (влажный бокс). Смешайте 100 мкл блокирующего буфера (50 мМ Tris-HCl (pH 7,6), 50 мМ KCl, 2% (v/v) Tween-20) с 25 μл 1 мг/мл раствора NeutrAvidin (10% PBS) и распределите его по поверхности покровного стекла. Дайте ему понервироваться в течение 15 минут при комнатной температуре во влажной камере.
- Постирайте покровное стекло в воде и высушите в N2. Помните, что функционализирована только одна сторона покровного стекла, поэтому помните, какая она сторона.
- Поместите изготовленный на заказ блок34 из полидиметилсилоксана (PDMS) на покровное стекло внутри держателя проточной ячейки. В результате образуется проточный канал высотой 100 мкм и шириной 1 мм. Полиэтиленовые трубки (PE-60: входной диаметр 0,76 мм и внешний диаметр 1,22 мм) могут быть вставлены в отверстия проточной ячейки для обеспечения доступа к буферам и подложкам.
- Флуоресцентная визуализация с использованием одной молекулы
ПРИМЕЧАНИЕ: Эти эксперименты должны проводиться как с G4-формирующими, так и с контрольными шаблонами. Это позволит визуализировать оба условия (блокирование Pol δ структурой G4 и отсутствие блокировки) на уровне отдельных молекул с помощью микроскопии полного внутреннего отражения (TIRF).- Нагрейте 1 мл аликвот буфера, блокирующего Tween (50 мМ Tris-HCl (pH 7,6), 50 мМ KCl, 2% (v/v) Tween-20) и 500 мкл аликвот промывочного буфера (25 мМ Tris-HCl (pH 7,6), 10 мМ ацетата магния, 250 мМ глутамата калия, 40 мкг/мл BSA, 0,1 мМ ЭДТА, 5 мМ DTT, 0,0025% (v/v) Tween-20) и буфер репликации до 40 °C в течение 15 мин. Это позволит высвободить газы из растворов. Дегазируйте растворы в вакуумной камере в течение еще 15 минут при давлении на 800 мбар ниже атмосферного.
- Возьмите сконструированную проточную ячейку (см. шаг 4.2) и поместите ее на предметное столико микроскопа. Нанеся каплю масла на объектив, поднимите его так, чтобы он соответствовал покровному покрытию.
ПРИМЕЧАНИЕ: Убедитесь, что объектив и проточная ячейка успевают достичь 31 °C. Это повысит активность дрожжей Pol δ и поможет обеспечить воспроизводимость данных. - Вставьте входную трубку в буфер блокировки дегазированного подростка и подсоедините выпускное отверстие к шприцевому насосу. Потяните шприц назад, чтобы втянуть блокирующий буфер Tween через трубку в канал. Дайте этому буферу инкубироваться не менее 30 минут, чтобы свести к минимуму неспецифические взаимодействия.
- Подайте 200 мкл дегазированного промывочного буфера в канал со скоростью 100 мкл/мин. Это очистит буфер блокировки Tween.
- Растворы матриц ДНК разбавить до 0,5 пМ в 500 мкл репликационного буфера. Подайте в канал 150 μL со скоростью 10 μL/мин. Осветите образец с помощью лазера с длиной волны 647 нм при давлении приблизительно 900 мВтсм-2 в плоскости образца для визуализации отдельных матриц ДНК.
ПРИМЕЧАНИЕ: Если поле зрения (FOV) с образцом не полностью сфокусировано, защитное стекло может не прилегать заподлицо к нижней части сконструированной проточной ячейки. В этом случае постройте новую проточную ячейку с использованием свежего покровного стекла. - Как только станет видна достаточная плотность пятен (около 1 пятна на 10мкм2), влейте свежий раствор репликационного буфера (с добавлением 1 мМ DTT) в канал для вымывания избытка ДНК. Объем в 250 мкл достаточен для полного удаления всей несвязанной ДНК.
- Перейдите к новому полю обзора и сделайте снимок ДНК, чтобы определить степень колокализации между меченой полимеразой и субстратом ДНК. После завершения увеличьте мощность лазера, чтобы фотоотбелить оставшиеся пятна.
- Готовят раствор полимеразы, содержащий 1 мМ DTT, 250 мкМ dNTP и 20 нМ δ Pol, меченного AF647, в 200 мкл репликационного буфера. Введите в канал 100 мкл раствора полимеразы со скоростью 5 мкл/мин.
- После того, как образец находится в фокусе и угол TIRF отрегулирован, установите мощность лазера с длиной волны волны 647 нм примерно на 900 мВтсм-2 в плоскости образца. Затем начните визуализацию поля зрения в течение желаемого периода времени. Собирайте данные со скоростью от 1 до 5 кадров в секунду в течение 10–20 минут, чтобы записать все события репликации.
- Анализ данных
ПРИМЕЧАНИЕ: Все анализы проводились с использованием Python (v. 3.11.7). Пользовательский код, используемый для анализа данных, доступен здесь: https://github.com/Single-molecule-Biophysics-UOW/G-quadruplex_binding_analysis.- Во-первых, колокализуйте пятна матрицы ДНК с флуоресцентными пятнами меченых дрожжей Pol δ. Два пика были определены как колокализованные, если их расстояние после коррекции дрейфа составляло ≤ 3 пикселя. Только пятна Pol δ, которые колокализуют матрицы ДНК, анализируются на предмет их кинетики связывания и общих поведенческих тенденций.
- Чтобы определить данные о событиях привязки, измерьте интенсивность каждого ролика с течением времени, поскольку они демонстрируют поведение «вкл/выкл» на протяжении всего фильма. Это позволяет определить, сколько раз связывание Pol δ происходит на одном матрице ДНК.
ПРИМЕЧАНИЕ: Этот анализ проводится как для экспериментов с формированием G4, так и для экспериментов с контрольным шаблоном для сравнения динамики связывания.
Примечание: Были проведены контрольные эксперименты, которые гарантировали, что неспецифические события связывания с G-квадруплексом или самой матрицей (23,2 с ± 4,9 с) не включаются в анализ событий связывания.
Результаты
Для этого анализа были разработаны два субстрата ДНК с флуоресцентно меченным 20-нт праймером: один, содержащий G4-образующую последовательность (рис. 1А, слева), и другой, в котором эта последовательность отсутствует (рис. 1А, справа). Чтобы подтвердить, что G-квадруплекс является эффективным препятствием для полимеразной активности, синтез ДНК с помощью Pol δ контролировали на денатурирующем геле PAGE. Активность очищенного меченого Pol δ на субстраты ДНК исследовали методом гель-электрофореза. На рисунке 1B (слева) показано, что флуоресцентно меченый Pol δ не способен синтезироваться за пределами G-квадруплекса. Перед началом синтеза (t = 0 мин) присутствует полоса, соответствующая 20 nt, представляющая собой меченый праймер, который был денатурирован из матричной цепи. Через 3 минуты эта 20-нтная полоса была преобразована в 60-нтн, что указывает на синтез всей ДНК и подтверждает, что полимераза полностью блокируется структурой G-квадруплекса. Эта блокировка подразумевает, что полимераза не может ни развернуть, ни обойти структуру. В противоположность этому, синтез контрольного шаблона, который не содержит последовательности, образующей G-квадруплекс (рис. 1A, справа), дал полосу в 100 нл через 3 минуты (рис. 1B, справа).
После подтверждения синтеза на матрицах и эффективной блокировки G-квадруплексом, эти измерения были повторены на одномолекулярном флуоресцентном микроскопе для мониторинга поведения полимеразы. Матрицы ДНК были привязаны к функционализированным покровным стеклам (рис. 2A) в микрофлюидной проточной ячейке, и положение каждого субстрата определяли путем визуализации флуоресцентно меченного праймера. Затем меченый Pol δ загружали в присутствии dNTP для инициирования синтеза. В рамках типичного поля зрения отслеживаются отдельные пятна, чтобы количественно определить, как часто Pol δ связывается и диссоциирует с ДНК (рис. 2B, C). Измеряя интенсивность в зависимости от времени на каждом субстрате ДНК, можно создать траектории отдельных молекул (рис. 2C, дополнительный рис. 3). Можно измерить характерное «время задержки», или среднюю продолжительность, в течение которой Pol δ остается привязанным к шаблону. Для подложки G4 время выдержки было определено равным 6 с ± 2 с, в то время как для контрольной подложки оно составляло 10 с ± 3 с (рис. 2D, дополнительный рис. 4). Кроме того, для каждой траектории можно количественно определить, сколько раз Pol δ связывается с шаблоном. Количество событий связывания с подложкой G4 значительно выше по сравнению с контрольным шаблоном (рисунок 2E). Несмотря на то, что из-за схоластической привязки и отмены привязки в контрольном шаблоне имеется более одного события привязки, в среднем наблюдается явное увеличение числа событий привязки для шаблона, формирующего G4. Это говорит о том, что после того, как синтез останавливается G-квадруплексом, связанная полимераза диссоциирует от ДНК до того, как новые полимеразы из раствора вступают в непрерывный цикл связывания и разъединения. Таким образом, этот анализ одной молекулы дает беспрецедентное понимание того, как ДНК-полимеразы реагируют на препятствия ДНК.

Рисунок 1: Анализ удлинения ансамблевого праймера. (А) Схематическое изображение субстратов ДНК. Субстрат G4 (слева) содержит последовательность, образующую G-квадруплекс, в то время как контрольный субстрат (справа) его не содержит. (B) Анализ расширения праймированной ДНК праймированной G4 и контрольных субстратов ДНК показывает, что флуоресцентно меченный дрожжевой Pol δ полностью блокируется G-квадруплексом. Гель PAGE показывает репликацию матриц ДНК с течением времени, что приводит к сдвигу меченого 20-нт праймера в 60-нт продукта для субстрата G4 (слева) и 100-нт продукта для контрольной матрицы (справа). M представляет собой лестницу, содержащую меченые 20 nt, 60 nt и 100 nt олигону. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этой цифры.
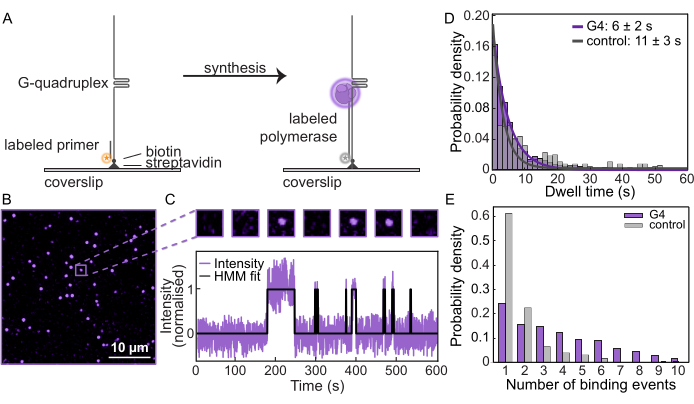
Рисунок 2: Флуоресцентная микроскопия с использованием одномолекулярной флуоресцентной микроскопии. (A) Схематическое изображение анализа удлинения одномолекулярного праймера с использованием меченых AF647 дрожжей Pol δ. (B) Типичное поле зрения, полученное в результате анализа одной молекулы. Каждое пятно представляет собой флуоресцентно меченный Pol δ связывание с матрицей ДНК. Масштабная линейка: 10 мкм. (C) (Вверху) Пример молекулы из анализа одной молекулы, показывающей включение/выключение связывания при обмене Pol δ. (внизу) Траектория движения одной и той же молекулы по одной молекуле, показывающая интенсивность во времени эксперимента. Черная линия представляет подгонку скрытой модели Маркова (HMM) к данным. (D) Время выдержки Pol δ на подложке G4 (фиолетовый) и контрольной подложке (серый). Линии представляют собой экспоненциальную аппроксимацию, что дает время жизни 6 с ± 2 с на подложке G4 и 10 с ± 3 с на управляющей подложке. (E) Количество событий связывания Pol δ с одиночными субстратами ДНК. Медианное число событий связывания на субстрате G4 (фиолетовый) составляет 3,5 по сравнению с контрольным субстратом 1 (серый). Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этой цифры.
Дополнительный рисунок 1:CD-спектроскопия. CD-спектры G4-образующей (фиолетовый) и контрольной последовательности (серый) в TE-буфере, содержащем 200 мМ KCl при 25 °C. Характерный пик на длине волны 260 нм, за которым следует отрицательный пик на длине волны 240 нм, характерен для параллельной внутримолекулярной GQ. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Дополнительный рисунок 2: Конструкция проточной ячейки. (А) Схема отдельных частей проточной ячейки. Квадратный блок PDMS расположен в верхней части покровного стекла микроскопа. Затем эти части соединяются вместе внутри крышки и дна держателя проточной ячейки. (В) Проточная ячейка в сборе. Трубки могут быть вставлены в каждую сторону блока PDMS для формирования каналов, через которые буферы могут проходить через устройство с помощью микрофлюидики. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Дополнительный рисунок 3: Траектории одиночных молекул. (А) Пример траектории молекулы, претерпевающей однократное связывание в течение 10-минутного временного интервала эксперимента. (B) Пример траектории молекулы, претерпевающей нулевое связывание в течение 10-минутного временного интервала эксперимента. Черная линия в точках (A) и (B) представляет подгонку скрытой марковской модели (HMM) к данным. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Дополнительный рисунок 4: Время задержки. (A) Время пребывания Pol δ на подложке G4. Линия представляет собой экспоненциальную аппроксимацию, что дает время жизни 6 с ± 2 с. (B) Время задержки Pol δ на управляющей подложке. Экспоненциальная аппроксимация дает время жизни от 10 с ± 3 с. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Дополнительный рисунок 5: Контроль фотоотбеливания. На графике показано среднее время, за которое отдельный фермент Pol δ дрожжей, меченный AF647, подвергается фотообесцвечиванию лазером с длиной волны 647 нм. Линия представляет собой экспоненциальную аппроксимацию, что дает срок службы 39 ± 6 с. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Обсуждение
В данной работе был описан анализ на основе флуоресценции одной молекулы, который дает представление о поведении ДНК-полимеразы при столкновении с G-квадруплексом. В то время как протоколы генерации матриц ДНК, мечения Pol δ и анализа объемной репликации ДНК просты, выполнение анализов микроскопии с использованием одной молекулы является более сложным с технической точки зрения. Из-за особенностей одномолекулярных методов необходимо проявлять большую осторожность, чтобы избежать попадания пыли, загрязнений или пузырьков воздуха, так как они заслоняют поле зрения и затрудняют сбор данных.
Ограничением экспериментов с использованием одномолекулярной микроскопии TIRF является фотообесцвечивание флуорофоров, которые ковалентно связаны с представляющими интерес биомолекулами. Фотообесцвечивание является необратимым процессом, приводящим к необратимой потере флуоресценции35. Чтобы смягчить эту проблему во время экспериментов, важно ограничить продолжительность лазерного воздействия, отрегулировать интенсивность лазера и оптимизировать время получения изображения. Эти стратегии помогают сохранять флуоресцентные сигналы, обеспечивая более надежные и продолжительные периоды наблюдений. При тонкой настройке этих параметров сигнал Pol δ сохраняется на протяжении всего измерения. Для оптимизации мощности лазера для анализа синтеза одномолекулярной флуоресцентной микроскопии рекомендуется измерять скорость фотообесцвечивания путем визуализации меченой полимеразы на чистом стеклянном покровном стекле. Систематически изменяя мощность лазера и оценивая скорость фотообесцвечивания в поле зрения, можно определить оптимальную интенсивность лазера, которая уравновешивает силу сигнала флуорофора с устойчивостью к фотообесцвечиванию (см. дополнительный рисунок 5).
Ключевым преимуществом этого одномолекулярного подхода по сравнению с традиционными методами, основанными на ансамблях, является его способность непосредственно визуализировать, когда отдельная ДНК-полимераза сталкивается со структурой G4 и взаимодействует с ней. Традиционные методы, основанные на ансамбле (такие как гель-электрофорез), продемонстрировали способность структур G4 блокировать ДНК-полимеразы 23,36,37. Эти методы, однако, не в состоянии предоставить кинетическую и механистическую информацию об этом взаимодействии в реальном времени, которая необходима для распутывания различных молекулярных кинетических этапов и результатов. Методы работы с одиночными молекулами предлагают беспрецедентное понимание кинетики, механизмов и поведения биомолекул, часто скрытых усреднением ансамбля38. Теперь можно видеть, как действуют ДНК-полимеразы в режиме реального времени – обмениваются, останавливаются, диссоциируют или обходят препятствия ДНК39. С помощью этого протокола идентификатор G4 может быть легко изменен с выбранной параллельной структуры c-MYC на любую параллельную, антипараллельную или гибридную топологию. Применение этого анализа на одной молекуле покажет, ведут ли себя одни и те же ДНК-полимеразы по-разному при столкновении с альтернативными топологиями G4. Таким образом, одномолекулярные методы жизненно важны для ответа на конкретные вопросы о том, как взаимодействуют белки и ДНК организма.
Посредством прямой визуализации взаимодействий ДНК-полимеразы с G-квадруплексами был идентифицирован ранее не охарактеризованный путь обмена дрожжевым Pol δ. Это открытие предполагает, что полимераза разъединяется при встрече с G-квадруплексом, ожидая вмешательства другого белка для разрешения структуры, прежде чем возобновить синтез ДНК. Этот протокол может быть адаптирован для исследования взаимодействий между различными белками поддержания генома и препятствиями ДНК, что дает беспрецедентное понимание того, как клеточные ферменты преодолевают геномные препятствия. Например, препятствие в этом анализе может быть изменено со структуры G4 на белково-ДНК-сшивку, тип повреждения ДНК, при котором белок необратимо ковалентно связывается с ДНК, выступая в качестве препятствия для репликации ДНК40. Такие исследования имеют решающее значение для понимания фундаментальных процессов репликации, восстановления и рекомбинации ДНК. Позволяя изучать динамику ДНК-белка на молекулярном уровне, этот анализ предоставляет мощный инструмент для выяснения механизмов, лежащих в основе целостности генома.
Раскрытие информации
Авторы заявляют, что у них нет конкурирующих финансовых интересов.
Благодарности
Н.К.-А. выражает признательность за финансирование, предоставленное в рамках стипендии Программы подготовки научных кадров правительства Австралии. Л.М.С. благодарна за финансирование, которое она получила от Национального совета по здравоохранению и медицинским исследованиям (Грант исследователя 2007778). Компания J.S.L благодарна за то, что получила награду Discovery Early Career Award (DE240100780) и награду NHMRC Investigator EL1 (2025412), финансируемую правительством Австралии. Компания S.H.M благодарна за то, что стала получателем карьерной стипендии Брюса Уоррена Molecular Horizons Ealy.
Материалы
| Name | Company | Catalog Number | Comments |
| 100 nt control DNA template | Integrated DNA Technologies | Primary control template in primer extension assays | |
| 100 nt G4-forming DNA template | Integrated DNA Technologies | Primary G4-forming template in primer extension assays | |
| 15% Mini-PROTEAN TBE-Urea Gel, 12 well, 20 µL | Bio-Rad | 4566055 | Gel electrophoresis |
| 20 nt labelled/biotinylated primer oligonucleotide | Integrated DNA Technologies | Required for primer extension of the 100 nt templates | |
| 22 nt control DNA template | Integrated DNA Technologies | Primary control template in circular dichroism spectroscopy experiments | |
| 22 nt G4-forming DNA template | Integrated DNA Technologies | Primary G4-forming template in circular dichroism spectroscopy experiments | |
| 24 x 24 mm coverslip | Marienfeld Superior | 100062 | Required for TIRF microscopy |
| 3-aminopropyltriethoxysilane | Thermo Fisher Scientific | A10668.22 | Coverslip functionalization |
| 647 nm laser | Coherent | 1196627 | Select wavelength to correspond to stain/dye of choice |
| 670 nm emission filter | Chroma Technology Corp | ET655lp | Ensures only the relevant wavelengths are let through to the detector |
| Amersham Typhoon 5 biomolecular imager | Cytiva | 29187191 | Gel analysis |
| Biotin-PEG-SVA (MW 5000) | Laysan Bio | BIO-PEG-SVA-5K-100MG | Coverslip functionalization |
| Deoxynucleotides triphosphate solution mix | Jena Bioscience | NU-1005L | Replication building blocks |
| Digital dry bath (115V) | Bio-Rad | 1660571 | Heating solutions |
| Dithiothreitol | Sigma-Aldrich | 646563 | Disulfide bond reduction |
| EM-CCD camera | Andor | iXon 897 | Capturing single-molecule movies |
| Formamide | Sigma-Aldrich | F9037 | DNA denaturing during ensemble DNA replication assays |
| Gas tight syringe, 1 mL | SGE | 21964 | Filling cuvette for circular dichroism spectroscopy |
| Inverted microscope with CFI Apo TIRF 100x oil-immersion objective | Nikon | Eclipse Ti-E | Facilitates single-molecule TIRF microscopy |
| J-810 Spectrophotometer | Jasco | J-810 | Measuring circular dichroism spectroscopy |
| mPEG-SVA (MW 5000) | Laysan Bio | MPEG-SVA-5K-100MG | Coverslip functionalization |
| NanoDrop One Spectrophotometer | Thermofisher Scientific | ND-ONE | Measuring protein and label concentrations |
| NeutrAvidin | Thermo Fisher Scientific | 31000 | Tethering DNA templates to the functionalized coverslips during single-molecule experiments |
| Polyethylene tubing | Instech | BTPE-60 | Needed for flow cell construction |
| Quartz cuvette, 10 mm | Starna Scientific | 1/Q/10/CD | Holds sample for circular dichroism spectroscopy |
| Syringe pump | Adelab Scientific | NE-1002X | Used to flow buffers and substrates through a microfluidic flow cell |
| Tris-EDTA buffer | Thermofisher Scientific | 93283 | DNA solution buffer |
| Yeast polymerase δ | Michael O'Donnell Lab | Replicating the DNA templates | |
| Zeba spin desalting column, 7 K MWCO, 0.5 mL | Thermo Fisher Scientific | 89882 | Polymerase purification |
Ссылки
- Sutton, M. D. Walker, G. C. Managing DNA polymerases: Coordinating DNA replication, DNA repair, and DNA recombination. Proc Natl Acad Sci U. S. A. 98 (15), 8342-8349 (2001).
- Garg, P. Burgers, P. M. J. DNA polymerases that propagate the eukaryotic DNA replication fork. Crit Rev Biochem Mol Biol. 40 (2), 115-128 (2005).
- Burgers, P. M. J. Eukaryotic DNA polymerases in DNA replication and DNA repair. Chromosoma. 107 (4), 218-227 (1998).
- Tsai, M.-D. How DNA polymerases catalyze DNA replication, repair, and mutation. Biochem. 53 (17), 2749-2751 (2014).
- Lewis, J. S. et al. Tunability of DNA polymerase stability during eukaryotic DNA replication. Mol Cell. 77 (1), 17-25 (2020).
- Chilkova, O. et al. The eukaryotic leading and lagging strand DNA polymerases are loaded onto primer-ends via separate mechanisms but have comparable processivity in the presence of PCNA. Nucleic Acids Res. 35 (19), 6588-6597 (2007).
- Kawasaki, Y. Sugino, A. Yeast replicative DNA polymerases and their role at the replication fork. Mol Cells. 12 (3), 277-285 (2001).
- Stillman, B. DNA polymerases at the replication fork in eukaryotes. Mol Cell. 30 (3), 259-260 (2008).
- Wood, R. D. Shivji, M. K. Which DNA polymerases are used for DNA repair in eukaryotes? Carcinogenesis. 18 (4), 605-610 (1997).
- Budd, M. E. Campbell, J. L. The roles of the eukaryotic DNA polymerases in DNA repair synthesis. Mutat Res DNA Repair. 384 (3), 157-167 (1997).
- Yang, W. Gao, Y. Translesion and repair DNA polymerases: Diverse structure and mechanism. Annu Rev Biochem. 87, 239-261 (2018).
- Bridges, B. A. DNA repair: Polymerases for passing lesions. Curr Biol. 9 (13), 475-477 (1999).
- Wilkinson, E. M., Spenkelink, L. M., Van Oijen, A. M. Observing protein dynamics during DNA-lesion bypass by the replisome. Front Mol Biosci. 9, 968424 (2022).
- Mueller, S. H., Spenkelink, L. M., Van Oijen, A. M. When proteins play tag: The dynamic nature of the replisome. Biophys Rev. 11 (4), 641-651 (2019).
- Zybailov, B. L., Sherpa, M. D., Glazko, G. V., Raney, K. D., Glazko, V. I. G4-quadruplexes, and genome instability. Mol Biol. 47 (2), 197-204 (2013).
- Loeb, L. A. Monnat, R. J. DNA polymerases and human disease. Nat Rev Genet. 9 (8), 594-604 (2008).
- Oganesian, L. Bryan, T. M. Physiological relevance of telomeric G-quadruplex formation: A potential drug target. Bioessays. 29 (2), 155-165 (2007).
- Chambers, V. S. et al. High-throughput sequencing of DNA G-quadruplex structures in the human genome. Nat Biotechnol. 33 (8), 877-881 (2015).
- Cheng, Y., Zhang, Y., You, H. Characterization of G-quadruplexes folding/unfolding dynamics and interactions with proteins from single-molecule force spectroscopy. Biomol. 11 (11), 1579 (2021).
- Tippana, R., Xiao, W., Myong, S. G-quadruplex conformation and dynamics are determined by loop length and sequence. Nucleic Acids Res. 42 (12), 8106-8114 (2014).
- Kusi-Appauh, N., Ralph, S. F., Van Oijen, A. M., Spenkelink, L. M. Understanding G-quadruplex biology and stability using single-molecule techniques. J Phys Chem B. 127 (25), 5521-5540 (2023).
- Obi, I. et al. Stabilization of G-quadruplex DNA structures in Schizosaccharomyces pombe causes single-strand DNA lesions and impedes DNA replication. Nucleic Acids Res. 48 (19), 10998-11015 (2020).
- Kamath-Loeb, A. S., Loeb, L. A., Johansson, E., Burgers, P. M., Fry, M. Interactions between the Werner syndrome helicase and DNA polymerase delta specifically facilitate copying of tetraplex and hairpin structures of the d(CGG)n trinucleotide repeat sequence. J Biol Chem. 276 (19), 16439-16446 (2001).
- Kaguni, L. S. Clayton, D. A. Template-directed pausing in in vitro DNA synthesis by DNA polymerase a from Drosophila melanogaster embryos. Proc Natl Acad Sci. 79 (4), 983-987 (1982).
- Sato, K., Martin-Pintado, N., Post, H., Altelaar, M., Knipscheer, P. Multistep mechanism of G-quadruplex resolution during DNA replication. Sci Adv. 7 (39), eabf8653 (2021).
- Castillo Bosch, P. et al. FANCJ promotes DNA synthesis through G-quadruplex structures. EMBO J. 33 (21), 2521-2533 (2014).
- Paeschke, K., Capra, John A., Zakian, Virginia A. DNA replication through G-quadruplex motifs is promoted by the Saccharomyces cerevisiae Pif1 DNA helicase. Cell. 145 (5), 678-691 (2011).
- Guo, L. et al. Joint efforts of replicative helicase and SSB ensure inherent replicative tolerance of G-quadruplex. Adv Sci. (Weinh.). 11 (9), e2307696 (2024).
- Pham, S. Q. T. et al. A new class of quadruplex DNA-binding nickel Schiff base complexes. Dalton Trans. 49 (15), 4843-4860 (2020).
- Georgescu, R. E. et al. Mechanism of asymmetric polymerase assembly at the eukaryotic replication fork. Nat Struct Mol Biol. 21 (8), 664-670 (2014).
- Au - Desjardins, P., Au - Hansen, J. B., Au - Allen, M. Microvolume protein concentration determination using the NanoDrop 2000c spectrophotometer. J Vis Exp. 33, e1610 (2009).
- Summer, H., Grämer, R., Dröge, P. Denaturing urea polyacrylamide gel electrophoresis (Urea PAGE). J Vis Exp. 32, e1485 (2009).
- Geertsema, H. J., Duderstadt, K. E., Van Oijen, A. M. in DNA Replication: Methods and Protocols., eds Vengrova, S., Dalgaard, J.) 219-238, Springer New York, New York, NY (2015).
- Lewis, J. S. et al. Single-molecule visualization of fast polymerase turnover in the bacterial replisome. eLife. 6, e23932 (2017).
- Fish, K. N. Total internal reflection fluorescence (TIRF) microscopy. Curr Protoc Cytom. 12, 12-18 (2009).
- Sommers, J. A., Estep, K. N., Maul, R. W., Brosh, R. M. Biochemical analysis of DNA synthesis blockage by G-quadruplex structure and bypass facilitated by a G4-resolving helicase. Methods. 204, 207-214 (2022).
- Edwards, D. N., Machwe, A., Wang, Z., Orren, D. K. Intramolecular telomeric G-quadruplexes dramatically inhibit DNA synthesis by replicative and translesion polymerases, revealing their potential to lead to genetic change. PLoS One. 9 (1), 80664 (2014).
- Mueller, S. H. et al. Rapid single-molecule characterization of enzymes involved in nucleic-acid metabolism. Nucleic Acids Res. 51 (1), 5 (2022).
- Lewis, J. S., Van Oijen, A. M., Spenkelink, L. M. Embracing heterogeneity: Challenging the paradigm of replisomes as deterministic machines. Chem Rev. 123 (23), 13419-13440 (2023).
- Stingele, J., Bellelli, R., Boulton, S. J. Mechanisms of DNA-protein crosslink repair. Nat Rev Mol Cell Biol. 18 (9), 563-573 (2017).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеThis article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены