Method Article
Visualização de fluorescência de molécula única da dinâmica da DNA polimerase em G-quadruplexes
Neste Artigo
Resumo
Este protocolo descreve um ensaio de replicação de DNA de molécula única baseado em microscopia de fluorescência, permitindo a visualização em tempo real das interações entre DNA polimerases e obstáculos como estruturas G-quadruplex.
Resumo
A capacidade das proteínas envolvidas na replicação do DNA eucariótico de superar obstáculos - como "obstáculos" de proteínas e DNA - é fundamental para garantir a duplicação fiel do genoma. G-quadruplexes são estruturas de ácidos nucleicos de ordem superior que se formam em regiões ricas em guanina do DNA e demonstraram atuar como obstáculos, interferindo nas vias de manutenção genômica. Este estudo apresenta um método baseado em microscopia de fluorescência em tempo real para observar as interações da DNA polimerase com estruturas G-quadruplex. Oligonucleotídeos de DNA curtos e preparados contendo um G-quadruplex foram imobilizados em lamínulas de vidro funcionalizadas dentro de uma célula de fluxo microfluídico. As DNA polimerases marcadas com fluorescência foram introduzidas, permitindo que seu comportamento e estequiometria fossem monitorados ao longo do tempo. Essa abordagem permitiu a observação do comportamento da polimerase à medida que ela era paralisada por um G-quadruplex. Especificamente, usando δ de polimerase de levedura marcada com fluorescência, descobriu-se que, ao encontrar um G-quadruplex, a polimerase passa por um ciclo contínuo de ligação e desligação. Este ensaio de molécula única pode ser adaptado para estudar interações entre várias proteínas de manutenção de DNA e obstáculos no substrato de DNA.
Introdução
As DNA polimerases são enzimas que catalisam a incorporação de trifosfatos de nucleosídeos para duplicar o DNA 1,2,3,4,5. Como tal, eles desempenham um papel fundamental nos processos essenciais de manutenção do DNA, incluindo replicação do DNA 6,7,8 e reparo 9,10,11,12. As DNA polimerases devem replicar o genoma com precisão e eficiência para garantir a integridade genômica, evitando o acúmulo de mutações no genoma. Durante a síntese, as polimerases freqüentemente encontram "obstáculos", como proteínas ligadas ao DNA ou estruturas secundárias do DNA13. Esses obstáculos podem retardar ou até mesmo bloquear a progressão da polimerase14. Superar esses obstáculos é importante para garantir a duplicação fiel do genoma, pois a falha em fazê-lo pode levar à instabilidade genômica 15,16.
Uma classe importante de obstáculos são os G-quadruplexes (G4), estruturas secundárias de DNA não canônicas que demonstraram se formar em sequências ricas em guanina dentro do genoma humano17. Existem mais de 700.000 sequências diferentes no genoma humano capazes de formar um G4, incluindo regiões dentro dos telômeros e promotores de oncogenes18. Essas estruturas de DNA adotam várias conformações, dependendo da sequência de nucleotídeos, comprimento e cátion metálico ligado 19,20,21. Essa diversidade significa que as polimerases devem superar uma variedade de diferentes topologias G4, potencialmente com vários graus de eficiência. A falha das polimerases em superar ou contornar uma estrutura G4 demonstrou impedir a progressão do garfo de replicação in vivo, levando à instabilidade genômica22. Estudos in vitro mostraram que as estruturas G4 podem paralisar ou bloquear completamente as polimerases de levedura 23,24,25,26,27. A capacidade das estruturas G4 de paralisar ou bloquear as DNA polimerases é inteiramente dependente de sua estabilidade cinética e termodinâmica, com algumas polimerases sendo capazes de desdobrar certas G428. Embora esses estudos forneçam informações sobre a capacidade das polimerases de superar os obstáculos do G-quadruplex, eles não têm a capacidade de visualizar diretamente o comportamento da polimerase ao encontrar um G4. O destino das polimerases - se elas permanecem ligadas, caem ou trocam dinamicamente - determina quais processos a jusante são acessíveis para resolver o G4.
Neste estudo, um ensaio de microscopia de molécula única baseado em fluorescência foi desenvolvido para visualizar e monitorar diretamente a ligação da DNA polimerase com estruturas G4 em tempo real. Este ensaio envolve amarrar modelos de DNA formadores de G4 a uma lamínula biotinilada em uma célula de fluxo microfluídico, onde DNA polimerases marcadas com fluorescência podem ser introduzidas para iniciar a síntese de DNA. Ao medir a fluorescência das polimerases ao longo do tempo, seu comportamento ao encontrar uma estrutura G4 pode ser observado diretamente. A estrutura G4 encontrada no oncogene do câncer c-MYC foi escolhida para este ensaio devido ao seu alto nível de estabilidade. Este protocolo agora pode ser adaptado para catalogar o comportamento de uma variedade de polimerases em todos os domínios da vida associados a diferentes topologias e estabilidades G4. Este ensaio oferece uma abordagem inovadora e de alto rendimento para elucidar os mecanismos pelos quais as DNA polimerases navegam pelos obstáculos do DNA, fornecendo uma ferramenta poderosa para avançar na compreensão da dinâmica da polimerase.
Protocolo
Os detalhes dos reagentes e do equipamento utilizado estão listados na Tabela de Materiais.
1. Espectroscopia de dicroísmo circular
NOTA: Antes de desenvolver o ensaio, foi necessário realizar espectroscopia de dicroísmo circular (CD) na sequência G4 selecionada para garantir o dobramento correto. A sequência de 22 nt (5′-TGAGGGTGGAGGGGGGGGAA-3′) forma a estrutura G4 derivada do oncogene do câncer c-MYC . A espectroscopia de CD também foi realizada em uma sequência de controle (5′- TGAGTGTGAAGACGATGTAGAA -3) que possui nucleotídeos-chave alterados que impedem a formação de G4.
- Para preparar os moldes de DNA para espectroscopia de CD, adicione 40 μL de 100 μM dos moldes de 22 nt a 360 μL de Tris-HCl (pH 8,0), tampão de ácido etilenodiaminotetracético (EDTA) (TE) suplementado com 200 mM de cloreto de potássio (KCl). Um 10 μM de concentração final de DNA é desejado para experimentos de espectroscopia de CD.
NOTA: A presença de íons K+ é essencial para estabilizar a estrutura G4 no modelo. - Aquecer as soluções de ADN a 95 °C num banho seco digital durante 15 min. Depois de concluído, resfrie as soluções lentamente durante a noite, colocando o bloco de aquecimento em poliestireno. Este processo de resfriamento lento é responsável pela dobra do G4.
- Transfira 400 μL das soluções de sequência de formação e controle de G4 para uma cubeta de quartzo de 0,1 cm de comprimento de caminho usando uma seringa à prova de gás. Encha a cubeta lentamente para evitar a formação de bolhas de ar.
- Medir o espectro de CD entre 200 nm e 400 nm utilizando um espectropolarímetro a 25 °C. Os parâmetros e condições para essa medição foram descritos anteriormente29. Os resultados podem ser encontrados na Figura Suplementar 1.
NOTA: Certifique-se de gravar o espectro de CD para que o buffer sozinho atue como um espaço em branco.
2. Preparação de modelos de DNA
NOTA: Os modelos capazes de serem replicados são modelos lineares curtos de 100 nt preparados usando técnicas padrão de biologia molecular. O molde formador de G4 (5′-GAATTACATTTAAATTTTACACAGATACAGTCAATGAGAA
CTTCCAGGCGTAACGAGAGCACGGGGGGGGAGGG
GGGACCTTAGCTTCGAGTTCCGAT-3 ′) contém a sequência capaz de formar um G-quadruplex derivado de MYC (da etapa 1) em seu centro. O modelo de controle (5′-GAATTACATTTAAATTTTACACAGATACAGTCAATGAGA
ACTTCCAGGCGTAACGAGAGCACGATGTAGCAGAACT
GTGACCTTAGCTTCGAGTTCCGAT-3′) tem a mesma região de controle (da etapa 1) também em seu centro.
- Para recozer o primer para os moldes de DNA formadores e de controle de G4, misture 20 μM do primer (5′-Biotina-AF647-TCTTAATGTAAATTTAAAATGTGTC-3′) a 20 μM dos moldes de 100 nt em tampão TE suplementado com 200 mM KCl.
- Aquecer as soluções molde de ADN a 95 °C num banho seco digital durante 15 min. Depois de concluído, resfrie as soluções lentamente durante a noite, colocando o bloco de aquecimento em poliestireno para permitir o dobramento do G4 e o recozimento dos dois fios.
3. Marcação da DNA polimerase com AF647
- Rotulando a polimerase com o corante
NOTA: A levedura Pol δ foi expressa e purificada conforme descrito anteriormente30. A polimerase então precisava ser marcada com o corante monorreativo éster Alexa Fluor NHS (AF647) para permitir a visualização no nível de molécula única. Isso foi concluído conforme descrito anteriormente5.- Para rotular o terminal N, incube um excesso de 3 vezes do corante AF647 com 320 μL de 58,5 μM Pol δ em um tampão contendo 30 mM de Tris-HCl (pH 7,6), 2 mM de ditiotreitol (DTT), 300 mM de cloreto de sódio (NaCl), 50 mM de glutamato de potássio e 10% (v / v) de glicerol por 10 min a 4 ° C. Gire suavemente a solução para garantir uma mistura completa e melhorar a eficiência da rotulagem.
NOTA: A sequência de proteínas pode conter aminas primárias adicionais em aminoácidos lisina. Ajustando as condições de reação, como o pH, o rótulo pode ser preferencialmente anexado ao terminal N em oposição aos resíduos de lisina. Isso pode exigir mais otimização para aumentar a eficiência da rotulagem. - Para preparar as colunas de dessalinização por centrifugação de 0,5 mL (7 K de peso molecular cortado), primeiro centrifugue as colunas a 1500 x g por 1 min para remover a solução de armazenamento. Rejeitar as soluções depositadas no tubo de recolha após centrifugação.
NOTA: Certifique-se de afrouxar a tampa das colunas de centrifugação durante as etapas de centrifugação. Isso garantirá que as colunas sejam eluídas corretamente. - Equilibre as colunas em um tampão contendo 30 mM de Tris-HCl (pH 7,6), 300 mM de NaCl, 3 mM de DTT, 50 mM de glutamato de potássio e 5% (v / v) de glicerol adicionando 300 μL de tampão ao topo da coluna. Centrifugar a coluna a 1500 x g durante 1 min e rejeitar o escoamento. Repita esse processo mais duas vezes.
- Aplique 200 μL de cada amostra de δ Pol na coluna. Centrifugue as colunas a 1500 x g por 2 min e colete o Pol δ purificado em um tubo de microcentrífuga de 1,5 mL.
- Congelar as fracções purificadas em N2 líquido e armazenar as alíquotas a -80 °C para evitar a degradação das proteínas ao longo do tempo.
- Para rotular o terminal N, incube um excesso de 3 vezes do corante AF647 com 320 μL de 58,5 μM Pol δ em um tampão contendo 30 mM de Tris-HCl (pH 7,6), 2 mM de ditiotreitol (DTT), 300 mM de cloreto de sódio (NaCl), 50 mM de glutamato de potássio e 10% (v / v) de glicerol por 10 min a 4 ° C. Gire suavemente a solução para garantir uma mistura completa e melhorar a eficiência da rotulagem.
- Determinando a eficiência da rotulagem
- Carregue 2 μL da amostra de δ Pol agora marcada com AF647 em um espectrofotômetro UV-vis31.
NOTA: Certifique-se de que um espaço em branco do buffer Pol δ seja medido primeiro. - Meça os comprimentos de onda de excitação de 650 nm para o corante AF647 e 280 nm para o Pol δ usando o espectrofotômetro.
- Usando a lei de Beer-Lambert e os coeficientes de extinção molar de 270.000 cm-1 M-1 para o corante AF647 e 195.960 cm-1 M-1 para a polimerase, calcule a eficiência de marcação.
NOTA: A eficiência de rotulagem determinada para a levedura marcada com AF647 Pol δ usada neste ensaio foi calculada em aproximadamente 67%.
- Carregue 2 μL da amostra de δ Pol agora marcada com AF647 em um espectrofotômetro UV-vis31.
4. Ensaio de extensão de primer de conjunto
NOTA: Antes de realizar experimentos de replicação de molécula única, é necessário confirmar se a DNA polimerase está bloqueada pelo G-quadruplex por meio de ensaios de replicação em massa.
- Defina a temperatura de um bloco de aquecimento dentro de um banho seco digital para 30 °C. Isso garantirá a atividade máxima da levedura Pol δ.
- Prepare "master mixes" para os modelos de formação e controle de G4 em tampão de replicação (25 mM de Tris-HCl (pH 7,6), 10 mM de acetato de magnésio, 50 mM de glutamato de potássio, 40 μg / mL de albumina de soro bovino (BSA), 0,1 mM de EDTA, 5 mM de DTT e 0,0025% (v / v) Tween-20). Suplementar cada mistura com 1 mM de DTT, 250 μM de dTTP, dCTP, dATP, dGTP (dNTPs) e 10 nM do respectivo molde de DNA (formador de G4 ou controle). Coloque-os em um bloco de aquecimento para garantir que atinjam 30 °C.
- Temperar 12 μL de cada master mix com 12 μL de tampão de carga de formamida (80% (p/v) de formamida, 10 mM de EDTA) para atuar como controle T = 0 min.
- Para iniciar a síntese de DNA, adicione a levedura Pol δ marcada com AF647 a cada mistura principal (concentração final de 20 nM). Misture bem para garantir que as DNA polimerases repliquem os moldes com a máxima eficiência.
NOTA: Mantenha o fermento Pol δ no gelo durante o uso para preservar a atividade enzimática. - Após 30 s, 60 s e 180 s, remova 12 μL de cada master mix e resfrie com 12 μL de tampão de carga de formamida. A proporção de 1:1 da mistura principal para o tampão de carregamento de formamida garante que a reação de replicação não possa continuar depois de extinta.
- Retirar as soluções agora temperadas do bloco de aquecimento a 30 °C e colocá-las num bloco de aquecimento a 98 °C para desnaturar o dsDNA em ssDNA. Depois de decorridos 10 minutos, leve todo o bloco de aquecimento ao tanque de gel para carregar as soluções em um gel desnaturante de tris-borato-EDTA (TBE)-uréia poliacrilamida (PAGE)a 15% 32.
NOTA: O gel PAGE precisa ser aquecido antes do carregamento das amostras, executando a eletroforese em gel a 180 V por 30 min em 1x tampão TBE. Depois de concluído, enxágue cada poço para remover a uréia. - Carregue 15 μL de cada amostra no gel ao lado de uma escada contendo 0,02 μM de oligonucleotídeos marcados com 20 nt, 60 nt e 100 nt em tampão de carga de formamida.
- Execute o gel por 60 min a 180 V em 1x tampão TBE para garantir a separação completa.
- Imagem do gel no canal Cy5 de um gerador de imagens biomolecular. Isso permitirá que a fita de DNA replicada contendo o rótulo AF-647 seja medida.
5. Microscopia de fluorescência de molécula única
- Funcionalidade da lamínula
NOTA: Para anexar os moldes de DNA à lamínula de vidro, ela deve primeiro ser funcionalizada com aminosilano, seguida pela amarração de moléculas de PEG biotiniladas. Esse processo minimiza as interações inespecíficas entre o DNA e/ou proteínas e a superfície.- Limpe as lamínulas (24 x 24 mm) inserindo-as em frascos de coloração e despejando etanol. Sonicar os frascos por 30 min antes de enxaguar as lamínulas com água pura. Repita este processo com hidróxido de potássio 1 M (KOH) antes de enxaguar novamente. Repita essas etapas de limpeza mais uma vez.
- Enxágue e encha um novo frasco com acetona e coloque as lamínulas dentro. Espalhe o 3-aminopropiltrietoxissilano no frasco para formar uma solução a 2% v / v. Agite o frasco por 3 min antes de extinguir a reação com um grande excesso de água.
- Seque as lamínulas com N2 e coloque-as individualmente em uma caixa úmida cheia de água. Isso evitará que as lamínulas sequem.
- Preparar um éster de biotina-PEG-SVA:mPEG-SVA 1:25 em 100 mM de NaHCO3 (pH 8,2). Vortex esta mistura por 20 s para garantir uma mistura completa.
- Pipetar 40 μL da solução de PEG para uma lamínula seca. Coloque outra lamínula por cima para "ensanduchar" a solução entre as duas lamínulas. Isso irá funcionalizar as faces da lamínula no interior. Repita para todas as lamínulas.
- Incube as lamínulas no escuro por 3 h. Após a incubação, separe os pares de lamínulas, enxágue-os com excesso de água e seque-os com gás N2 comprimido.
- Repita as etapas 4.1.4-4.1.5 para aplicar uma segunda camada de PEG nas lamínulas. Certifique-se de colocar os lados previamente funcionalizados juntos, portanto, tome cuidado para não virá-los acidentalmente durante as etapas de lavagem. Incube as soluções no escuro durante a noite antes de enxaguá-las e secá-las.
- Armazene as lamínulas no vácuo para preservar sua funcionalidade. As lamínulas funcionalizadas são estáveis por um mês quando armazenadas corretamente.
- Preparação de células de fluxo microfluídico
NOTA: Uma célula de fluxo microfluídico33 é construída para experimentos de molécula única (ver Figura Suplementar 2). Isso permitirá que tampões, modelos de DNA e proteínas entrem em contato com uma lamínula funcionalizada (consulte a etapa 5.1).- Remova uma lamínula funcionalizada do aspirador e coloque-a em um suporte de microtubos parcialmente cheio de água (caixa úmida). Misture 100 μL de um tampão de bloqueio (50 mM de Tris-HCl (pH 7,6), 50 mM de KCl, 2% (v / v) Tween-20) com 25 μL de solução de NeutrAvidina 1 mg / mL (10% PBS) e espalhe na superfície da lamínula. Deixe incubar por 15 min em temperatura ambiente na caixa úmida.
- Lavar a lamínula com água e secar em N2. Lembre-se de que apenas um lado da lamínula é funcionalizado, portanto, lembre-se de qual lado está.
- Coloque o bloco34 de polidimetilsiloxano (PDMS) feito sob medida na lamínula dentro do suporte da célula de fluxo. Isso gerará um canal de fluxo de 100 μm de altura e 1 mm de largura. Tubos de polietileno (PE-60: diâmetro de entrada de 0,76 mm e diâmetro externo de 1,22 mm) podem então ser inseridos nos orifícios da célula de fluxo para fornecer acesso a tampões e substratos.
- Imagem de fluorescência de molécula única
NOTA: Esses experimentos devem ser realizados com os modelos de formação e controle de G4. Isso permitirá que ambas as condições (bloqueio do Pol δ por uma estrutura G4 e nenhum bloqueio) sejam visualizadas no nível de molécula única usando microscopia de reflexão interna total (TIRF).- Aquecer alíquotas de 1 ml de um tampão de bloqueio de Tween (50 mM de Tris-HCl (pH 7,6), 50 mM de KCl, 2% (v/v) de Tween-20) e alíquotas de 500 μL de tampão de lavagem [25 mM de Tris-HCl (pH 7,6), 10 mM de acetato de magnésio, 250 mM de glutamato de potássio, 40 μg/ml de BSA, 0,1 mM de EDTA, 5 mM de TDT, 0,0025% (v/v) Tween-20) e tampão de replicação a 40 °C por 15 min. Isso liberará os gases das soluções. Desgaseifique as soluções em uma câmara de vácuo por mais 15 minutos a 800 mbar abaixo da pressão atmosférica.
- Pegue a célula de fluxo construída (consulte a etapa 4.2) e coloque-a no stage do microscópio. Depois de colocar uma gota de óleo na objetiva, levante a objetiva para atender à lamínula.
NOTA: Certifique-se de que a objetiva e a célula de fluxo tenham tempo para atingir 31 °C. Isso aumentará a atividade da levedura Pol δ e ajudará a garantir dados reprodutíveis. - Insira o tubo de entrada no tampão de bloqueio Tween desgaseificado e conecte a saída à bomba de seringa. Puxe a seringa para trás para puxar o tampão de bloqueio de Tween através do tubo para o canal. Permita que esse buffer incube por pelo menos 30 minutos para minimizar interações não específicas.
- Fluxo em tampão de lavagem desgaseificado de 200 μL no canal a uma taxa de 100 μL/min. Isso eliminará o buffer de bloqueio de interpolação.
- Dilua as soluções de molde de DNA a 0,5 pM em tampão de replicação de 500 μL. Fluxo de 150 μL para o canal a uma taxa de 10 μL/min. Ilumine a amostra usando um laser de 647 nm a aproximadamente 900 mWcm-2 no plano da amostra para visualizar modelos de DNA individuais.
NOTA: Se o campo de view (FOV) com a amostra não estiver completamente em foco, a lamínula pode não estar nivelada com a parte inferior da célula de fluxo construída. Construa uma nova célula de fluxo usando uma nova lamínula, se isso ocorrer. - Uma vez visível uma densidade suficiente de manchas (cerca de 1 mancha por 10 μm2), flua em uma nova solução de tampão de replicação (suplementada com 1 mM de DTT) para o canal para lavar o excesso de DNA. Um volume de 250 μL é suficiente para remover completamente todo o DNA não ligado.
- Mova para um novo FOV e capture uma imagem do DNA para determinar o grau de colocalização entre a polimerase marcada e o substrato de DNA. Depois de concluído, aumente a potência do laser para fotobranquear os pontos restantes.
- Prepare uma solução de polimerase contendo 1 mM de DTT, 250 μM de dNTPs e 20 nM de Pol δ marcado com AF647 em 200 μL de tampão de replicação. Fluxo em 100 μL da solução de polimerase a uma taxa de 5 μL / min no canal.
- Quando a amostra estiver em foco e o ângulo TIRF tiver sido ajustado, defina a potência do laser de um laser de 647 nm para aproximadamente 900 mWcm-2 no plano da amostra. Em seguida, comece a obter imagens do FOV pelo período de tempo desejado. Adquira entre 1 e 5 quadros/s por 10 a 20 minutos para capturar todos os eventos de replicação.
- Análise de dados
NOTA: Todas as análises foram realizadas usando Python (v. 3.11.7). O código personalizado usado para a análise de dados está disponível aqui: https://github.com/Single-molecule-Biophysics-UOW/G-quadruplex_binding_analysis.- Primeiro, colocalize os pontos de molde de DNA com os pontos fluorescentes da levedura marcada Pol δ. Dois picos foram determinados como colocalizados se sua distância após a correção do desvio fosse ≤ 3 pixels. Apenas os pontos Pol δ que colocalizam modelos de DNA são analisados quanto à sua cinética de ligação e tendências comportamentais gerais.
- Para determinar os dados de eventos de ligação, meça a intensidade ao longo do tempo de cada ponto à medida que eles exibem o comportamento "ligado/desligado" ao longo do filme. Isso permite determinar quantas vezes a ligação do δ Pol ocorre em um único molde de DNA.
NOTA: Esta análise é feita para os experimentos de modelo de formação e controle de G4 para comparar a dinâmica de ligação.
NOTA: Experimentos de controle foram realizados, o que garante que eventos de ligação inespecíficos ao G-quadruplex ou ao próprio modelo (23,2 s ± 4,9 s) não sejam incluídos na análise dos eventos de ligação.
Resultados
Para este ensaio, dois substratos de DNA com um primer de 20 nt marcado com fluorescência foram projetados: um contendo uma sequência formadora de G4 (Figura 1A, à esquerda) e outro sem essa sequência (Figura 1A, à direita). Para confirmar que o G-quadruplex é um obstáculo eficaz à atividade da polimerase, a síntese de DNA por Pol δ foi monitorada em um gel PAGE desnaturante. A atividade do Pol δ purificado marcado nos substratos de DNA foi examinada por eletroforese em gel. A Figura 1B (à esquerda) mostra que o Pol marcado com fluorescência δ é incapaz de sintetizar além do G-quadruplex. Antes do início da síntese (t = 0 min), uma banda correspondente a 20 nt está presente, representando o primer marcado que foi desnaturado da fita molde. Após 3 min, essa banda de 20 nt foi convertida em uma banda de 60 nt, indicando que a síntese ocorreu em todo o DNA e confirmando que a polimerase foi completamente bloqueada pela estrutura G-quadruplex. Esse bloqueio implica que a polimerase não pode se desdobrar nem contornar a estrutura. Em contraste, a síntese de um modelo de controle que não contém a sequência de formação de G-quadruplex (Figura 1A, direita) produziu uma banda de 100 nt após 3 min (Figura 1B, direita).
Após a confirmação da síntese nos moldes e o bloqueio eficiente pelo G-quadruplex, essas medições foram repetidas em um microscópio de fluorescência de molécula única para monitorar o comportamento da polimerase. Os moldes de DNA foram amarrados a lamínulas funcionalizadas (Figura 2A) em uma célula de fluxo microfluídico, e a posição de cada substrato foi determinada pela visualização do primer marcado com fluorescência. Em seguida, Pol δ rotulado foi carregado na presença de dNTPs para iniciar a síntese. Dentro de um FOV típico, manchas individuais são rastreadas para quantificar a frequência com que Pol δ se liga e se dissocia do DNA (Figura 2B, C). Medindo a intensidade em função do tempo em cada substrato de DNA, trajetórias de molécula única podem ser geradas (Figura 2C, Figura Suplementar 3). A característica "tempo de permanência", ou a duração média em que Pol δ permanece vinculado ao modelo, pode ser medida. Para o substrato G4, o tempo de permanência foi determinado em 6 s ± 2 s, enquanto para o substrato controle, foi de 10 s ± 3 s (Figura 2D, Figura Suplementar 4). Além disso, para cada trajetória, o número de vezes que Pol δ se liga ao modelo pode ser quantificado. O número de eventos de ligação ao substrato G4 é muito maior em comparação com o modelo de controle (Figura 2E). Embora haja instâncias de mais de um evento de associação no modelo de controle devido à associação e desvinculação escolar, há um claro aumento no número de eventos de associação em média para o modelo de formação de G4. Isso sugere que, após a síntese ser interrompida pelo G-quadruplex, a polimerase ligada se dissocia do DNA antes que novas polimerases da solução envolvam um ciclo contínuo de ligação e desligação. Portanto, este ensaio de molécula única fornece uma visão incomparável de como as DNA polimerases respondem aos bloqueios de DNA.

Figura 1: Ensaio de extensão do primer do conjunto. (A) Representação esquemática dos substratos de DNA. O substrato G4 (à esquerda) contém uma sequência de formação de G-quadruplex, enquanto o substrato de controle (à direita) não. (B) Ensaio de extensão de primase de DNA de conjunto do G4 preparado e substratos de DNA de controle mostrando que a levedura marcada com fluorescência Pol δ é totalmente bloqueada pelo G-quadruplex. O gel PAGE mostra a replicação dos moldes de DNA ao longo do tempo, resultando em uma mudança do primer de 20 nt marcado para o produto de 60 nt para o substrato G4 (à esquerda) e o produto de 100 nt para o modelo de controle (à direita). M representa uma escada contendo oligos marcados de 20 nt, 60 nt e 100 nt. Clique aqui para ver uma versão maior desta figura.
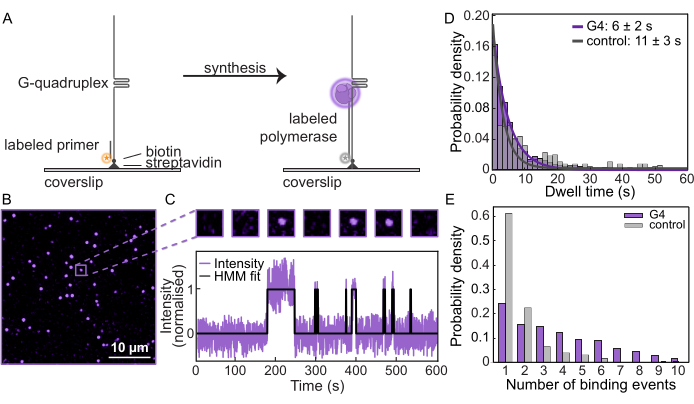
Figura 2: Microscopia de fluorescência de molécula única. (A) Representação esquemática do ensaio de extensão de primer de molécula única usando levedura marcada com AF647 Pol δ. (B) FOV típico obtido de um ensaio de molécula única. Cada mancha representa um Pol marcado com fluorescência δ ligação a um molde de DNA. Barra de escala: 10 μm. (C) (Topo) Exemplo de molécula do ensaio de molécula única mostrando a ligação liga/desliga à medida que o Pol δ troca. (Abaixo) Trajetória de molécula única da mesma molécula mostrando a intensidade ao longo do curso de tempo do experimento. A linha preta representa o ajuste do Modelo de Markov Oculto (HMM) aos dados. (D) Tempo de permanência do Pol δ no substrato G4 (roxo) e no substrato controle (cinza). As linhas representam ajustes exponenciais, dando uma vida útil de 6 s ± 2 s no substrato G4 e 10 s ± 3 s no substrato de controle. (E) Número de eventos de ligação de Pol δ a substratos de DNA únicos. O número médio de eventos de ligação no substrato G4 (roxo) é 3,5, em comparação com o substrato de controle 1 (cinza). Clique aqui para ver uma versão maior desta figura.
Figura Suplementar 1: Espectroscopia CD. Espectros CD da sequência de formação de G4 (roxo) e controle (cinza) em tampão TE contendo 200 mM de KCl a 25 ° C. O pico característico em 260 nm, seguido pelo pico negativo em 240 nm, é característico de um GQ intramolecular paralelo. Clique aqui para baixar este arquivo.
Figura suplementar 2: Construção da célula de fluxo. (A) Esquema das peças individuais da célula de fluxo. O bloco quadrado do PDMS fica no topo da lamínula do microscópio. Essas peças são então encaixadas dentro da tampa e da parte inferior do suporte da célula de fluxo. (B) A célula de fluxo completa. Os tubos podem ser inseridos em cada lado do bloco PDMS para formar os canais através dos quais os buffers podem fluir através do dispositivo por meio de microfluídica. Clique aqui para baixar este arquivo.
Figura suplementar 3: Trajetórias de molécula única. (A) Exemplo de trajetória de uma molécula passando por um único evento de ligação durante o período de 10 minutos do experimento. (B) Exemplo de trajetória de uma molécula submetida a eventos de ligação zero durante o período de 10 minutos do experimento. A linha preta em (A) e (B) representa o ajuste do Modelo de Markov Oculto (HMM) aos dados. Clique aqui para baixar este arquivo.
Figura Suplementar 4: Tempos de permanência. (A) Tempo de permanência do Pol δ no substrato G4. A linha representa o ajuste exponencial, dando uma vida útil de 6 s ± 2 s. (B) Tempo de permanência de Pol δ no substrato de controle. O ajuste exponencial dá uma vida útil de 10 s ± 3 s. Clique aqui para baixar este arquivo.
Figura 5 suplementar: Controle de fotobranqueamento. O gráfico mostra o tempo médio para uma enzima Pol δ de levedura marcada com AF647 individual ser fotobranqueada pelo laser de 647 nm. A linha representa o ajuste exponencial, dando uma vida útil de 39 ± 6 s. Clique aqui para baixar este arquivo.
Discussão
Aqui, foi descrito um ensaio baseado em fluorescência de molécula única que fornece informações sobre o comportamento de uma DNA polimerase ao encontrar um G-quadruplex. Embora os protocolos de geração de modelos de DNA, marcação de δ Pol e ensaios de replicação de DNA em massa sejam todos diretos, a execução de ensaios de microscopia de molécula única é tecnicamente mais desafiadora. Devido à natureza das técnicas de molécula única, deve-se tomar muito cuidado para evitar a introdução de poeira, contaminação ou bolhas de ar, pois isso obscurecerá o FOV e dificultará a coleta de dados.
Uma limitação dos experimentos de microscopia TIRF de molécula única é o fotobranqueamento dos fluoróforos que são acoplados covalentemente às biomoléculas de interesse. O fotobranqueamento é um processo irreversível que leva à perda permanente de fluorescência35. Para mitigar isso durante os experimentos, é essencial limitar a duração da exposição ao laser, ajustar a intensidade do laser e otimizar o tempo de imagem. Essas estratégias ajudam a preservar os sinais de fluorescência, garantindo períodos de observação mais confiáveis e prolongados. Ao ajustar esses parâmetros, o sinal Pol δ permanece durante a medição. Para otimizar a potência do laser para os ensaios de síntese de microscopia de fluorescência de molécula única, é aconselhável medir a taxa de fotobranqueamento por meio de imagens da polimerase marcada em uma lamínula de vidro limpa. Variando sistematicamente a potência do laser e avaliando a taxa de fotobranqueamento em todo o campo de visão, pode-se identificar a intensidade ideal do laser que equilibra a intensidade do sinal do fluoróforo com a resistência ao fotobranqueamento (consulte a Figura 5 suplementar).
A principal vantagem dessa abordagem de molécula única em relação aos métodos tradicionais baseados em conjunto é sua capacidade de visualizar diretamente quando uma DNA polimerase individual encontra e interage com uma estrutura G4. Métodos tradicionais baseados em conjuntos (como eletroforese em gel) demonstraram a capacidade das estruturas G4 de bloquear as DNA polimerases23 , 36 , 37 . Essas técnicas, no entanto, não fornecem as informações cinéticas e mecanicistas em tempo real dessa interação, necessárias para desvendar diferentes etapas e resultados cinéticos moleculares. As técnicas de molécula única oferecem uma visão incomparável da cinética, mecanismos e comportamentos das biomoléculas, muitas vezes ocultas pela média do conjunto38. Agora é possível ver como as DNA polimerases estão agindo em tempo real - se elas trocam, param, dissociam ou contornam os obstáculos do DNA39. Com este protocolo estabelecido, a identidade do G4 pode ser facilmente alterada da estrutura c-MYC paralela escolhida para qualquer topologia paralela, antiparalela ou híbrida. A aplicação deste ensaio de molécula única revelará se as mesmas DNA polimerases se comportam de maneira diferente ao encontrar topologias G4 alternativas. Como tal, os métodos de molécula única são vitais para responder a perguntas específicas sobre como as proteínas e o DNA do corpo interagem.
Por meio da visualização direta das interações da DNA polimerase com G-quadruplexes, foi identificada uma via de troca anteriormente não caracterizada para a levedura Pol δ. Esta descoberta sugere que a polimerase se desprende ao encontrar um G-quadruplex, aguardando a intervenção de outra proteína para resolver a estrutura antes de reiniciar a síntese de DNA. Este protocolo pode ser adaptado para investigar interações entre várias proteínas de manutenção do genoma e obstáculos de DNA, oferecendo insights incomparáveis sobre como as enzimas celulares navegam pelos impedimentos genômicos. Por exemplo, o bloqueio deste ensaio pode ser alterado de uma estrutura G4 para uma reticulação proteína-DNA, um tipo de lesão de DNA na qual uma proteína é irreversivelmente ligada covalentemente ao DNA, atuando como um obstáculo para a replicação do DNA40. Esses exames são cruciais para a compreensão dos processos fundamentais de replicação, reparo e recombinação do DNA. Ao permitir o estudo da dinâmica DNA-proteína no nível molecular, este ensaio fornece uma ferramenta poderosa para elucidar os mecanismos subjacentes à integridade genômica.
Divulgações
Os autores declaram não ter interesses financeiros concorrentes.
Agradecimentos
N.K.-A. reconhece o financiamento concedido pela bolsa de estudos do Programa de Treinamento em Pesquisa do Governo Australiano. LMS é grata pelo financiamento que recebeu do Conselho Nacional de Saúde e Pesquisa Médica (Investigator Grant 2007778). JSL é grato por receber um Prêmio Discovery Early Career (DE240100780) e NHMRC Investigator EL1 (2025412) financiado pelo governo australiano. A S.H.M é grata por receber a bolsa de carreira Bruce Warren Molecular Horizons Ealy.
Materiais
| Name | Company | Catalog Number | Comments |
| 100 nt control DNA template | Integrated DNA Technologies | Primary control template in primer extension assays | |
| 100 nt G4-forming DNA template | Integrated DNA Technologies | Primary G4-forming template in primer extension assays | |
| 15% Mini-PROTEAN TBE-Urea Gel, 12 well, 20 µL | Bio-Rad | 4566055 | Gel electrophoresis |
| 20 nt labelled/biotinylated primer oligonucleotide | Integrated DNA Technologies | Required for primer extension of the 100 nt templates | |
| 22 nt control DNA template | Integrated DNA Technologies | Primary control template in circular dichroism spectroscopy experiments | |
| 22 nt G4-forming DNA template | Integrated DNA Technologies | Primary G4-forming template in circular dichroism spectroscopy experiments | |
| 24 x 24 mm coverslip | Marienfeld Superior | 100062 | Required for TIRF microscopy |
| 3-aminopropyltriethoxysilane | Thermo Fisher Scientific | A10668.22 | Coverslip functionalization |
| 647 nm laser | Coherent | 1196627 | Select wavelength to correspond to stain/dye of choice |
| 670 nm emission filter | Chroma Technology Corp | ET655lp | Ensures only the relevant wavelengths are let through to the detector |
| Amersham Typhoon 5 biomolecular imager | Cytiva | 29187191 | Gel analysis |
| Biotin-PEG-SVA (MW 5000) | Laysan Bio | BIO-PEG-SVA-5K-100MG | Coverslip functionalization |
| Deoxynucleotides triphosphate solution mix | Jena Bioscience | NU-1005L | Replication building blocks |
| Digital dry bath (115V) | Bio-Rad | 1660571 | Heating solutions |
| Dithiothreitol | Sigma-Aldrich | 646563 | Disulfide bond reduction |
| EM-CCD camera | Andor | iXon 897 | Capturing single-molecule movies |
| Formamide | Sigma-Aldrich | F9037 | DNA denaturing during ensemble DNA replication assays |
| Gas tight syringe, 1 mL | SGE | 21964 | Filling cuvette for circular dichroism spectroscopy |
| Inverted microscope with CFI Apo TIRF 100x oil-immersion objective | Nikon | Eclipse Ti-E | Facilitates single-molecule TIRF microscopy |
| J-810 Spectrophotometer | Jasco | J-810 | Measuring circular dichroism spectroscopy |
| mPEG-SVA (MW 5000) | Laysan Bio | MPEG-SVA-5K-100MG | Coverslip functionalization |
| NanoDrop One Spectrophotometer | Thermofisher Scientific | ND-ONE | Measuring protein and label concentrations |
| NeutrAvidin | Thermo Fisher Scientific | 31000 | Tethering DNA templates to the functionalized coverslips during single-molecule experiments |
| Polyethylene tubing | Instech | BTPE-60 | Needed for flow cell construction |
| Quartz cuvette, 10 mm | Starna Scientific | 1/Q/10/CD | Holds sample for circular dichroism spectroscopy |
| Syringe pump | Adelab Scientific | NE-1002X | Used to flow buffers and substrates through a microfluidic flow cell |
| Tris-EDTA buffer | Thermofisher Scientific | 93283 | DNA solution buffer |
| Yeast polymerase δ | Michael O'Donnell Lab | Replicating the DNA templates | |
| Zeba spin desalting column, 7 K MWCO, 0.5 mL | Thermo Fisher Scientific | 89882 | Polymerase purification |
Referências
- Sutton, M. D., Walker, G. C. Managing DNA polymerases: Coordinating DNA replication, DNA repair, and DNA recombination. Proc Natl Acad Sci. 98 (15), 8342-8349 (2001).
- Burgers Garg, P., J, P. M. DNA polymerases that propagate the eukaryotic DNA replication fork. Crit Rev Biochem Mol Biol. 40 (2), 115-128 (2005).
- Burgers, P. M. J. Eukaryotic DNA polymerases in DNA replication and DNA repair. Chromosoma. 107 (4), 218-227 (1998).
- Tsai, M. -D. How DNA polymerases catalyze DNA replication, repair, and mutation. Biochem. 53 (17), 2749-2751 (2014).
- Lewis, J. S., et al. Tunability of DNA polymerase stability during eukaryotic DNA replication. Mol Cell. 77 (1), 17-25 (2020).
- Chilkova, O., et al. The eukaryotic leading and lagging strand DNA polymerases are loaded onto primer-ends via separate mechanisms but have comparable processivity in the presence of PCNA. Nucleic Acids Res. 35 (19), 6588-6597 (2007).
- Sugino Kawasaki, Y., A, Yeast replicative DNA polymerases and their role at the replication fork. Mol Cells. 12 (3), 277-285 (2001).
- Stillman, B. DNA polymerases at the replication fork in eukaryotes. Mol Cell. 30 (3), 259-260 (2008).
- Shivji Wood, R. D., K, M. Which DNA polymerases are used for DNA repair in eukaryotes. Carcinogenesis. 18 (4), 605-610 (1997).
- Campbell Budd, M. E., L, J. The roles of the eukaryotic DNA polymerases in DNA repair synthesis. Mutat Res DNA Repair. 384 (3), 157-167 (1997).
- Gao Yang, W., Y, Translesion and repair DNA polymerases: Diverse structure and mechanism. Annu Rev Biochem. 87, 239-261 (2018).
- Bridges, B. A. DNA repair: Polymerases for passing lesions. Curr Biol. 9 (13), 475-477 (1999).
- Wilkinson, E. M., Spenkelink, L. M., Van Oijen, A. M. Observing protein dynamics during DNA-lesion bypass by the replisome. Front Mol Biosci. 9. 968424, (2022).
- Mueller, S. H., Spenkelink, L. M., Van Oijen, A. M. When proteins play tag: The dynamic nature of the replisome. Biophys Rev. 11 (4), 641-651 (2019).
- Zybailov, B. L., Sherpa, M. D., Glazko, G. V., Raney, K. D., Glazko, V. I. G4-quadruplexes, and genome instability. Mol Biol. 47 (2), 197-204 (2013).
- Monnat Loeb, L. A., J, R. DNA polymerases and human disease. Nat Rev Genet. 9 (8), 594-604 (2008).
- Bryan Oganesian, L., M, T. Physiological relevance of telomeric G-quadruplex formation: A potential drug target. Bioessays. 29 (2), 155-165 (2007).
- Chambers, V. S., et al. High-throughput sequencing of DNA G-quadruplex structures in the human genome. Nat Biotechnol. 33 (8), 877-881 (2015).
- Cheng, Y., Zhang, Y., You, H. Characterization of G-quadruplexes folding/unfolding dynamics and interactions with proteins from single-molecule force spectroscopy. Biomol. 11 (11), (2021).
- Tippana, R., Xiao, W., Myong, S. G-quadruplex conformation and dynamics are determined by loop length and sequence. Nucleic Acids Res. 42 (12), 8106-8114 (2014).
- Kusi-Appauh, N., Ralph, S. F., Van Oijen, A. M., Spenkelink, L. M. Understanding G-quadruplex biology and stability using single-molecule techniques. J Phys Chem B. 127 (25), 5521-5540 (2023).
- Obi, I., et al. Stabilization of G-quadruplex DNA structures in Schizosaccharomyces pombe causes single-strand DNA lesions and impedes DNA replication. Nucleic Acids Res. 48 (19), 10998-11015 (2020).
- Kamath-Loeb, A. S., Loeb, L. A., Johansson, E., Burgers, P. M., Fry, M. Interactions between the Werner syndrome helicase and DNA polymerase delta specifically facilitate copying of tetraplex and hairpin structures of the d(CGG)n trinucleotide repeat sequence. J Biol Chem. 276 (19), 16439-16446 (2001).
- Clayton Kaguni, L. S., A, D. Template-directed pausing in in vitro DNA synthesis by DNA polymerase a from Drosophila melanogaster embryos. Proc Natl Acad Sci. 79 (4), 983-987 (1982).
- Sato, K., Martin-Pintado, N., Post, H., Altelaar, M., Knipscheer, P. Multistep mechanism of G-quadruplex resolution during DNA replication. Sci Adv. 7 (39), (2021).
- Castillo Bosch,, P,, et al. FANCJ promotes DNA synthesis through G-quadruplex structures. EMBO J. 33 (21), 2521-2533 (2014).
- Paeschke, K., Capra, J. ohnA., Zakian, V. irginiaA. DNA replication through G-quadruplex motifs is promoted by the Saccharomyces cerevisiae Pif1 DNA helicase. Cell. 145 (5), 678-691 (2011).
- Guo, L., et al. Joint efforts of replicative helicase and SSB ensure inherent replicative tolerance of G-quadruplex. Adv Sci. (Weinh. 11 (9), (2024).
- Pham, S. Q. T., et al. A new class of quadruplex DNA-binding nickel Schiff base complexes). Dalton Trans. 49 (15), 4843-4860 (2020).
- Georgescu, R. E., et al. Mechanism of asymmetric polymerase assembly at the eukaryotic replication fork. Nat Struct Mol Biol. 21 (8), 664-670 (2014).
- Hansen, J. B., Au, Microvolume protein concentration determination using the NanoDrop 2000c spectrophotometer. J Vis Exp. 33, (2009).
- Summer, H., Grämer, R., Dröge, P. Denaturing urea polyacrylamide gel electrophoresis (Urea PAGE). J Vis Exp. 32, (2009).
- Geertsema, H. J., Duderstadt, K. E., Van Oijen, A. M. in DNA Replication: Methods and Protocols., eds Vengrova, S., Dalgaard, J. , Springer. New York, New York, NY. 219-238 (2015).
- Lewis, J. S., et al. Single-molecule visualization of fast polymerase turnover in the bacterial replisome. eLife. 6, 23932(2017).
- Fish, K. N. Total internal reflection fluorescence (TIRF) microscopy. Curr Protoc Cytom. 12, 12-18 (2009).
- Sommers, J. A., Estep, K. N., Maul, R. W., Brosh, R. M. Biochemical analysis of DNA synthesis blockage by G-quadruplex structure and bypass facilitated by a G4-resolving helicase. Methods. , 207-214 (2022).
- Edwards, D. N., Machwe, A., Wang, Z., Orren, D. K. Intramolecular telomeric G-quadruplexes dramatically inhibit DNA synthesis by replicative and translesion polymerases, revealing their potential to lead to genetic change. PLoS One. 9 (1), 80664(2014).
- Mueller, S. H., et al. Rapid single-molecule characterization of enzymes involved in nucleic-acid metabolism. Nucleic Acids Res. 51 (1), 5(2022).
- Lewis, J. S., Van Oijen, A. M., Spenkelink, L. M. Embracing heterogeneity: Challenging the paradigm of replisomes as deterministic machines. Chem Rev. 123 (23), 13419-13440 (2023).
- Stingele, J., Bellelli, R., Boulton, S. J. Mechanisms of DNA-protein crosslink repair. Nat Rev Mol Cell Biol. 18 (9), 563-573 (2017).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados