Method Article
الكشف عن نسخة عدد التعديلات عن طريق التسلسل خلية واحدة
In This Article
Summary
تسلسل وحيد الخلية هو أداة شعبية متزايدة ويمكن الوصول إليها لمعالجة التغيرات الجينية بدقة عالية. ونحن نقدم البروتوكول الذي يستخدم التسلسل خلية واحدة لتحديد عدد النسخ تغييرات في الخلايا وحيدة.
Abstract
الكشف عن التغيرات الجينية في قرار وحيد الخلية مهم لوصف التباين الوراثي والتطور في الأنسجة الطبيعية، والسرطانات، والسكان الميكروبية. وقد الطرق التقليدية لتقييم التباين الوراثي محدودة بسبب دقة منخفضة، انخفاض الحساسية، و / أو منخفضة النوعية. وقد برزت تسلسل خلية واحدة باعتبارها أداة قوية للكشف عن تباين وراثي مع ارتفاع القرار، وحساسية عالية، وعندما حللت بشكل مناسب، خصوصية عالية. نحن هنا نقدم بروتوكول لعزل، كامل الجينوم التضخيم، والتسلسل، وتحليل الخلايا واحد. يسمح نهجنا لتحديد موثوق للmegabase نطاق المتغيرات في عدد النسخ في زنازين انفرادية. ومع ذلك، يمكن أن تطبق أيضا جوانب من هذا البروتوكول للتحقيق في أنواع أخرى من تغيرات جينية في خلايا مفردة.
Introduction
تغييرات في عدد النسخ الحمض النووي يمكن أن تتراوح في حجمها من عدة أزواج قاعدة (عدد النسخ المتغيرات) (التنوعات في عدد النسخ) على الكروموسومات بأكملها (عدم توازن الصبغيات). تغييرات في عدد النسخ التي تؤثر على مناطق واسعة من الجينوم يمكن أن يكون لها عواقب كبيرة المظهرية عن طريق تغيير التعبير تصل إلى الآلاف من الجينات 1، 2. التنوعات في عدد النسخ التي تكون موجودة في جميع الخلايا من السكان يمكن الكشف عنها بواسطة التسلسل السائبة أو أساليب تعتمد على ميكروأري 3 و 4. ومع ذلك، يمكن السكان أيضا أن تكون متباينة وراثيا، مع التنوعات الموجودة في مجموعة فرعية من السكان أو حتى الخلايا وحيدة. تباين وراثي شائع في السرطان، والقيادة تطور الورم، وأيضا في الأنسجة الطبيعية، مع مجهولة العواقب 5، 6، 7، 8، 9 ، 10.
تقليديا، تم تقييم التباين الوراثي إما عن طريق نهج سيتولوجي أو التسلسل بالجملة. النهج سيتولوجي، مثل مضان في الموقع التهجين (FISH)، وينتشر كروموسوم، وتنميط نووي الطيفي (سكاى)، لديها مصلحة تحديد التعديلات موجودة في الخلايا الفردية ولكن لديها معدلات الخطأ عالية بسبب القطع الأثرية من التهجين ونشر 11. تقتصر هذه الأساليب أيضا في عدد نسخ قرار فقط كاشفا على التغييرات تغطي العديد من megabases. التسلسل أو المجهرية من الحمض النووي بالجملة، على الرغم العالي في الدقة والقرار، هو أقل حساسية. من أجل الكشف عن التباين الوراثي من خلال النهج القائمة على السكان، يجب أن تكون المتغيرات الحالية في جزء كبير من الخلايا في عدد السكان. جعلت ظهور أساليب لتضخيم الحمض النووي الجيني من الخلايا واحد من الممكن أن تسلسل جينوم الخلايا وحيدة. seque خلية واحدةncing لديها مزايا عالية الدقة، حساسية عالية، وعندما يتم تطبيق الأساليب المناسبة لمراقبة الجودة، وارتفاع دقة 12.
هنا، نحن تصف طريقة للكشف عن megabase النطاق في عدد النسخ تغييرات في الخلايا وحيدة. نحن عزل الخلايا واحد من قبل microaspiration، تضخيم الحمض النووي الجيني باستخدام رابط محول PCR، وإعداد مكتبات للجيل المقبل من التسلسل، وكشف عدد النسخ المتغيرات من قبل كل من نموذج ماركوف الخفي وتجزئة الثنائية دائرية.
Protocol
1. عزل الخلايا واحدة
- إعداد microaspirator
- إزالة نهاية بلاستيكية واضحة من أنبوب التجميع الشافطة وإدراج نهاية الضيقة في واحدة من نهاية الأنابيب البلاستيكية 1 أقدام طويلة مع 3/16 "قطرها الداخلي.
- إدراج مخرج مرشح حقنة 0.2 ميكرون في الطرف الآخر من الأنابيب البلاستيكية 1 أقدام طويلة مع 3/6 "القطر الداخلي.
- إدراج مدخل للمرشح حقنة 0.2 ميكرون في واحدة من نهاية الأنابيب البلاستيكية 6 بوصة طويلة مع 5/16 "قطرها الداخلي.
- اختياري: قطع الأنابيب البلاستيكية 6 بوصة طويلة مع 5/16 "قطرها الداخلي في نصف وإدراج فخ المياه في الخط الفاصل بين شطري.
- كسر ماصة المصلية 5 مل من البلاستيك في مل التخرج (1) وأدخل الطرف كسر في نهاية مفتوحة للأنابيب البلاستيكية مع 5/16 "قطرها الداخلي.
- إدراج مخرج 5 مل من البلاستيك ماصة المصلية في نهاية مرنة من تجميع أنبوب الشافطة (حيث نهاية بلاستيكية واضحةقد أزيلت).
- للتخزين، وتغطي لسان حال الأحمر من أنبوب التجميع الشافطة مع أنبوب بلاستيكية معقمة.
- استخلاص أنبوب زجاجي الشعرية إلى القطر الداخلي من 10-30 ميكرون من خلال جلب منتصف أنبوب بالقرب من لهب الموقد بنسن وتطبيق التوتر على طرفي الأنبوب. كسر أنبوب تعادل في وسط لإنشاء اثنين من الإبر الشافطة المحتملة. كرر هذه الخطوة مع عدة أنابيب وكما فقط بعض الأنابيب في نهاية المطاف مع قطرها الداخلي الذي يتناسب مع التقاط الخلايا وحيدة.
- للتخزين، ووضع قطعتين من الصلصال في 15 سم طبق بتري وتأمين الإبر الشافطة عبر شرائط.
- اختيار الخلايا
- إعداد تعليق خلية واحدة بما يتناسب مع نوع من الخلايا والتجربة. على سبيل المثال، لإعداد الخلايا الملتصقة مثل خطوط الإنسان الليفية الخلايا، والخلايا حصاد الخلايا trypsinization ونقل إلى أنبوب مخروطي يحتوي على وسائل الإعلام المناسبة.
- قبل وأثناء، أوfter إعداد تعليق واحد خلية (اعتمادا على كم من الوقت يستغرق لإعداد تعليق خلية واحدة)، والإعداد غطاء محرك السيارة لكامل الجينوم التضخيم.
- رش أسفل سطح غطاء محرك السيارة، وصناديق ماصة، ونصائح ماصة مع التبييض 10٪ ويمسح بقطعة منشفة ورقية. كرر هذه الخطوة مع 70٪ من الإيثانول.
- إضافة 8 ميكرولتر من المياه من عدة كله الجينوم التضخيم (WGA) إلى الآبار الفردية من 96-جيدا لوحة PCR، وأيضا واحدة لكل خلية لتكون متسلسلة. تغطية جيدا 96 لوحة PCR مع غطاء من 96-جيدا لوحة زراعة الأنسجة ومكان على الجليد.
- إضافة 1000 الخلايا إلى 10 مل سائل الإعلام أو الفوسفات مخزنة المالحة (PBS) في 15 سم طبق بتري ووضع الطبق على الجليد لمنع الخلايا من الانضمام إلى الطبق.
- جعل الخلايا في طبق بتري وجيد 96 لوحة PCR مع غطاء على الجليد لمجهر الضوء مع الهدف 10X.
- زيادة افتتاح إبرة الشافطة من خلال استغلال بلطف نهاية تعادل خارج من الالبريد الشافطة إبرة على سطح صلب مثل هذا غيض من الراحة. إدراج نهاية واسعة من الإبرة الشافطة في نهاية واضحة للmicroaspirator.
- وضع طبق بتري تحتوي الخلايا على المسرح المجهر. وضع لسان حال الأحمر من الشافطة في الفم. استخدم يدا واحدة لتحريك الإبرة الشافطة واليد الأخرى لنقل طبق بتري تحتوي على خلايا. تحديد الخلايا واحدة لتكون متسلسلة.
- عن طريق شفط الفم، واستخلاص خلية واحدة في إبرة الشافطة جنبا إلى جنب مع ~ 1-2 ميكرولتر من وسائل الإعلام أو برنامج تلفزيوني. نقل الخلية إلى 8 ميكرولتر من المياه داخل بئر واحدة من لوحة 96-جيدا PCR. تجنب إدخال فقاعات عند نقل الخلية.
- كرر الخطوة 1.2.7 حتى تم عزل العدد المطلوب من الخلايا. الحفاظ على لوحة PCR على الجليد ومغطاة غطاء في بين الخلايا اختيار. بمناسبة الآبار التي تلقت الخلايا.
- في حين التقاط الخلايا وحيدة، ذوبان الجليد في 10X تحلل خلية واحدة وعازلة تجزئة من ز كلهعدة التضخيم enome. بعد حصوله على العدد المطلوب من الخلايا، الشروع فورا في كامل الجينوم التضخيم.
2. الجامع الجينوم التضخيم
- لمنع التلوث خلال كامل الجينوم التضخيم، إضافة جميع الكواشف داخل نسيج الثقافة هود، استخدم نصائح ماصة مع المرشحات، وتغيير غيض ماصة بين الآبار.
- إعداد محلول تحلل وتفتت عازلة عمل من عدة كله الجينوم التضخيم (WGA). لكل مجموعة تصل إلى 32 خلية، والجمع بين 32 ميكرولتر من 10X تحلل خلية واحدة وعازلة تجزئة و 2 ميكرولتر من بروتين حل K في أنبوب microcentrifuge. دوامة أنبوب لخلط الحلول.
- إضافة 1 ميكرولتر من تحلل وحل العازلة تجزئة العمل إلى كل بئر وماصة صعودا وهبوطا إلى المزيج.
- تغطية لوحة وختم جميع الآبار مع فيلم من البلاستيك. أجهزة الطرد المركزي لفترة وجيزة لوحة في لوحة الدوار صغير.
- دورة الحرارية كما الفلالدودة الحلزونية: 50 ° C، 1 ساعة و 99 درجة مئوية، 4 دقيقة.
- تبريد لوحة على الجليد وأجهزة الطرد المركزي لفترة وجيزة لوحة في لوحة الدوار صغير.
- إعداد مكتبة حل العازلة إعداد عمل من كامل مجموعة الجينوم التضخيم. لكل خلية، والجمع بين 2 ميكرولتر من العازلة إعداد مكتبة خلية واحدة 1X و 1 ميكرولتر من محلول الاستقرار مكتبة في أنبوب microcentrifuge. تحضير محلول العمل لعدة خلايا في نفس أنبوب microcentrifuge.
- إزالة فيلم من البلاستيك وإضافة 3 ميكرولتر من العمل المكتبة حل العازلة تمهيدا لكل بئر. ماصة محتويات البئر صعودا وهبوطا إلى المزيج. استبدال فيلم من البلاستيك.
ملاحظة: فيلم من البلاستيك يمكن إعادة استخدامها في جميع مراحل عملية الجينوم التضخيم كلها حتى تبدأ الآبار لحفر الآبار في الفيلم. عندما يحدث هذا، والتحول إلى فيلم من البلاستيك الجديد. - لفترة وجيزة أجهزة الطرد المركزي لوحة في لوحة الدوار صغير واحتضان عند 95 درجة مئوية لمدة 2 دقيقة.
- تبريد لوحة على الجليد لفترة وجيزةأجهزة الطرد المركزي لوحة في لوحة الدوار صغير.
- إزالة فيلم من البلاستيك، إضافة 1 ميكرولتر انزيم إعداد مكتبة على كل جانب، وماصة محتويات البئر صعودا وهبوطا إلى المزيج. الحفاظ على انزيم إعداد مكتبة على الجليد أو في كتلة باردة طوال هذه الخطوة. استبدال فيلم من البلاستيك.
- لفترة وجيزة أجهزة الطرد المركزي لوحة في الدوار لوحة صغير ودورة الحرارية على النحو التالي: 16 درجة مئوية، 20 دقيقة، 24 ° C، 20 دقيقة، 37 ° C، 20 دقيقة، 75 ° C، 5 دقائق، وعقد في 4 درجات مئوية.
- تبريد لوحة على الجليد وأجهزة الطرد المركزي لفترة وجيزة لوحة في لوحة الدوار صغير.
- إعداد مزيج التضخيم عمل من كامل مجموعة الجينوم التضخيم. لكل خلية، والجمع بين 48.5 المياه ميكرولتر و 7.5 ميكرولتر مزيج الرئيسي 10X التضخيم، و 5 البلمرة ميكرولتر WGA الحمض النووي في أنبوب microcentrifuge. تحضير مزيج العمل لعدة خلايا في نفس أنبوب microcentrifuge. الحفاظ على مزيج العمل على الجليد.
- إزالة فيلم من البلاستيك، إضافة 61 ميكرولتر العمل مزيج التضخيمإلى كل بئر، ومحتويات ماصة من جانب صعودا وهبوطا إلى المزيج. الحفاظ على مزيج التضخيم تعمل على الجليد أو في كتلة باردة طوال هذه الخطوة. استبدال فيلم من البلاستيك.
- لفترة وجيزة أجهزة الطرد المركزي لوحة في لوحة الدوار صغير ودورة الحرارية على النحو التالي: 95 درجة مئوية، 3 دقائق، 25 دورات من 94 درجة مئوية، و 30 ثانية و 65 درجة مئوية، 5 دقائق، وعقد في 4 درجات مئوية.
- نقل 60 ميكرولتر من كل عينة إلى أنبوب microcentrifuge منفصلة وتخزينها في - 20 درجة مئوية لاستخدامها في الخطوة 3.
- إضافة صبغة الحمض النووي تحميل لحجم ما تبقى من كل عينة (أي 3 ميكرولتر من 6X الحمض النووي تحميل صبغ إلى 15 ميكرولتر من العينة) وتشغيل 5 ميكرولتر من كل رد فعل على هلام الاغاروز 1٪.
ملاحظة: الخلايا التي تضخمت بنجاح سوف تظهر مسحة من 250 إلى 1000 سنة مضت. العينات التي لا تظهر مسحة أو تظهر فقط مسحة باهتة من غير المرجح أن تسفر عن تسلسل البيانات المفيدة.
3. التسلسل
- تنقية العينات مع الخرز ممغطس.
- ثاعينات من الحمض النووي ث على الجليد. الخرز دوامة ممغطس حتى الحل هو متجانس واحتضان حبات في درجة حرارة الغرفة لمدة 30 دقيقة على الأقل.
- نقل 20 ميكرولتر من كل عينة الحمض النووي في أنبوب microcentrifuge نظيفة. ما تبقى من العينة يمكن تخزينها في -20 درجة مئوية.
- إضافة 30 ميكرولتر (1.5X) من الخرز ممغطس لكل عينة الحمض النووي ودوامة لخلط. احتضان العينات في درجة حرارة الغرفة لمدة 10 دقيقة. ترك محلول المخزون من الخرز في درجة حرارة الغرفة لاستخدامها في الخطوة 3.4.
- وضع أنابيب على شريط أنبوب المغناطيسي لمدة 2 دقيقة، أو حتى شكل حبات بيليه وطاف هو واضح.
- باستخدام ماصة P200، إزالة أكبر قدر من طاف ممكن من دون إزعاج الخرز.
- إضافة 180 ميكرولتر من 80٪ من الإيثانول إلى خليط الحمض النووي حبة. تدوير أنابيب عدة مرات النسبية إلى المغناطيس ل"شطف" حبات من خلال حل الإيثانول.
- باستخدام ماصة P200، إزالة أكبر قدر من غسل الايثانول كما possiبليه دون إزعاج الخرز.
- كرر 3.1.6 و3.1.7.
- الهواء الجاف الخرز لحوالي 10 دقيقة أو حتى لا مزيد من الإيثانول مرئيا. انتقل إلى الخطوة التالية عندما تظهر حبات متصدع أو تسقط جدار الأنبوب.
- إزالة عينات من الشريط المغناطيسي. إضافة 40 ميكرولتر من 10 ملي تريس درجة الحموضة 8.0 إلى أزل الحمض النووي من الخرز ودوامة العينات ل resuspend الخرز. احتضان لمدة 2 دقيقة في درجة حرارة الغرفة.
- أجهزة الطرد المركزي لفترة وجيزة على عينات لجمع السائل في الجزء السفلي من الأنبوب ووضع أنابيب على شريط أنبوب المغناطيسي لمدة دقيقة على الأقل. نقل شاطف في أنابيب microcentrifuge نظيفة من دون إزعاج الخرز.
- تطبيع عينة
- قياس تركيز كل عينة باستخدام مطياف. ينبغي أن تركيزات تتراوح 10-30 نانوغرام / ميكرولتر.
- تمييع كل عينة إلى 0.2 نانوغرام / ميكرولتر باستخدام 10 ملي تريس درجة الحموضة 8.0. وسوف تتطلب الخطوات التالية الحد الأدنى للطnput من 5 ميكرولتر من هذا التخفيف.
- إعداد مكتبة
- إعداد مكتبات التسلسل عن طريق اتباع تعليمات إعداد عدة مكتبة 13.
- تنظيف النهائي
- تنظيف العينات وفقا لخطوة 3.1. لفترات القراءة من 50 نقطة، 75 نقطة أساس، أو 150 سنة مضت، واستخدام نسبة من حبة لعينة حجم 1.5X، 1X، أو 0.6x، على التوالي. أزل مع 15 ميكرولتر من 10 ملي تريس درجة الحموضة 8.0.
- تشغيل العينات على محلل جزء للتحقق من حجم التوزيع من المكتبة 14. حجم التوزيع يجب أن تنتشر بالتساوي 150-900 نقطة أساس.
- تحديد العينات باستخدام QPCR
- استخدام عدة الكمي المكتبة، إعداد رد فعل QPCR وفقا للتعليمات مجموعة 15. ترك فارغا عمود لإضافة معايير إيجابية (معايير الحمض النووي من عدة) والمعايير السلبية (الماء أو محلول فارغ آخر).
- دورة الحرارية كما فلأدنى: 95 درجة مئوية، و 5 دقيقة (معدل زيادة قدرها 4.8 ° C / ثانية) و 35 دورات من 95 درجة مئوية، 30 ثانية (معدل زيادة قدرها 4.8 ° C / ثانية) و 60 درجة مئوية، و 45 ثانية (معدل المنحدر من 2.5 ° C / ثانية).
- تجميع
- تحديد كيفية هناك حاجة إلى العديد من الممرات على flowcell وتقسيم العينات إلى مجموعات، مع مجموعة واحدة لكل حارة.
- لكل مجموعة من عينات، واختيار عينة بأقل تركيز استنادا إلى البيانات QPCR من الخطوة 3.5.2 وتطبيع جميع العينات في تلك المجموعة إلى أن تركيز باستخدام 10 ملي تريس درجة الحموضة 8.0.
- تجميع العينات في كل مجموعة معا.
- التسلسل
- حمامات الحمل على الجهاز تسلسل الجيل القادم فقا للإجراءات العادية 16.
تحليل 4. البيانات
ملاحظة: مطلوب بيئة يونيكس لتشغيل البرامج والكتابات في هذا القسم. تثبيت البرنامج المذكور في بروتوكول التالية في حياتهم أدلة stallation. ويمكن الاطلاع على جميع النصوص في https://sourceforge.net/projects/singlecellseqcnv/.
- تقليم يقرأ إلى 40 الإقليم الشمالي باستخدام fastx_trimmer من FASTX-أدوات الإصدار 0.0.13 17.
fastx_trimmer -Q33 -i example.fastq -l 40 -o example.trim.fastq - محاذاة يقرأ على الجينوم المناسب الإشارة (MM9 فأرة الحاسوب، hg19 للإنسان) باستخدام التحالف النسخة 0.6.1 مع الخيارات الافتراضية 18.
التحالف ن. mm9.fa example.trim.fastq> example.sai
التحالف samse mm9.fa example.sai example.trim.fastq> example.sam - إزالة التحالفات لchrM والكروموسومات عشوائية، ثم فرز وفهرسة الملفات BAM الناتجة باستخدام SAMTools النسخة 0.1.19 19.
البقرى -v -w chrM example.sam | البقرى -v عشوائي> example.filtered.sam
samtools عرض example.filtered.sam -uSh | samtools نوع - example.filtered
samtools مؤشر example.filtered.bam - تشغيل HMMcopy للكشف عن التنوعات= "XREF"> 20
- استخدام gcCounter في HMMcopy لإنشاء ملف إشارة نسبة GC للجينوم. استخدام الخيار "-w 500000" لتحديد حجم النافذة. استخدام نفس إصدار الملف FASTA المرجعية المستخدمة في الخطوة 4.2، ولكن تأكد من chrM وتتم إزالة الصبغيات عشوائية من ملف FASTA.
gcCounter -w 500000 mm9.fa> mm9_gc.wig - استخدام generateMap.pl في HMMcopy لإنشاء ملف مناورة لmappability. استخدام الخيار "-w 40" لتحديد قراءة طول. استخدام نفس الملف FASTA المرجعية المستخدمة في الخطوة 4.4.1.
generateMap.pl -b mm9.fa
generateMap.pl -w 40 -i mm9.fa mm9.fa -o mm9.bigwig - استخدام mapCounter في HMMcopy لإنشاء ملف إشارة mappability للجينوم. استخدام الخيار "-w 500000" لتحديد حجم النافذة.
mapCounter -w 500000 mm9.bigwig> mm9_map.wig - استخدام readCounter في HMMcopy لإنشاء ملف تذبذب لكل ملف BAM.
readCounter -w 500000 example.filtered.bam> input.wig - تعديل المسارات إلى الملفات المرجعية في النصوص R (run_hmmcopy.mm9.r أو run_hmmcopy.hg19.r)، استخدم الملفات التي تم إنشاؤها أعلاه في 4.4.1 (على سبيل المثال، mm9_gc.wig) و4.4.3 (على سبيل المثال، mm9_map. شعر مستعار) لgfile المتغيرات وmfile، والتي تشير إلى ملف مئوية GC ملف إشارة وإشارة mappability، على التوالي. قم بتشغيل R النصي تقدم "run_hmmcopy.mm9.r" أو "run_hmmcopy.hg19.r".
R CMD دفعة run_hmmcopy.mm9.r
أو
R CMD دفعة run_hmmcopy.hg19.r - لتجهيز دفعة من مجموعة من الملفات BAM، وضع الملفات BAM في مجلد واحد وتشغيل المقدمة النصي "HMMpipe.pl" في حزمة لدعوة قطاعات وحساب عشرات تقلب (VS). اتبع الشكل:
بيرل HMMpipe.pl Folder_to_BAMfiles MM9
- استخدام gcCounter في HMMcopy لإنشاء ملف إشارة نسبة GC للجينوم. استخدام الخيار "-w 500000" لتحديد حجم النافذة. استخدام نفس إصدار الملف FASTA المرجعية المستخدمة في الخطوة 4.2، ولكن تأكد من chrM وتتم إزالة الصبغيات عشوائية من ملف FASTA.
- تشغيل DNAcopy للكشف عن التنوعات 21.
- استخدام SAMtools النسخة 0.1.19 لاستخراج تعيين فريد يقرأ من كل ملف BAM.
عرض samtools -h -F 0x0004 example.filtered.bam | egrep -i "^ @ | XT: A: U" | samtools عرض -Shu -> example.mapped.bam - استخدام MarkDuplicates في بيكار النسخة 1.94 للاحتفال مكررة PCR في BAM ملف 22.
جافا جرة MarkDuplicates.jar INPUT = example.mapped.bam الناتج = example.nondup.bam METRICS_FILE = example.dup REMOVE_DUPLICATES = صحيح - استخدام coveragebed في bedtools نسخة 2.17.0 لحساب تعيين يقرأ في كل من النوافذ الديناميكي 500KB mappable محددة مسبقا في ملف BED تقدم "mm9.500k.dynamic.win.bed" أو "hg19.500k.dynamic.win.bed" 23 .
coverageBed -abam example.nondup.bam -b mm9.500k.dynamic.win.bed -counts | نوع -k1،1 -k2،2n> example.nondup.counts - استخدام المقدمة برل "normalizeGC.pl" لتطبيع التهم قراءة تقوم على توفير محتوى GC ملف مرجعي "mm9.500k.dynamic.win.fa.gc.txt" أو "hg19.500k.dynamic.win.fa. gc.txt ".
بيرل normalizeGC.رر mm9.500k.dynamic.win.fa.gc.txt example.nondup.counts> example.bam.norm.counts - استخدام قدمت R النصي "dnacopy.r" لاستدعاء شرائح.
R CMD دفعة dnacopy.r
- استخدام SAMtools النسخة 0.1.19 لاستخراج تعيين فريد يقرأ من كل ملف BAM.
- تحديد التنوعات
- استبعاد الخلايا التي لVS المحسوبة في 4.4.6 يتجاوز 0.26.
- تصفية الشرائح التي دعا اليها HMMcopy في 4.4.6 لتشمل فقط تلك التي في متوسط السجل 2 نسبة أكبر من 0.4 (الربح المفترض) أو أقل من -0.35 (الخسارة المفترضة).
- تصفية الشرائح التي دعا اليها DNAcopy في 4.5.5 لتشمل فقط تلك التي الشريحة يعني أكبر من 1.32 أو أقل من 0.6.
- تتداخل شرائح من 4.6.2 و4.6.3، داعيا التنوعات فقط في المناطق التي على حد سواء HMMcopy وDNAcopy تحديد الربح أو الخسارة المفترضة المفترضة.
النتائج
يجب أن تظهر الشافطة تجميعها مماثلة لتلك التي في الشكل 1A. ينبغي وضع الإبرة بحيث يكون عريضا بما فيه الكفاية لاستيعاب خلية واحدة ولكن ليس ذلك على نطاق واسع أن كمية كبيرة وضعت مع خلية واحدة. الشفط الخلايا واحد هو أسهل عندما يكون هناك ما بين واحد وخمسة خلايا في حقل 10X (الشكل 1B).
إذا كله الجينوم التضخيم ناجحا، سوف تظهر العينة كما مسحة على هلام الاغاروز (الشكل 2، والممرات 1 و 2 و 4 و 5 و 6 و 7). تشير مسحة باهتة أو غائبة رد فعل التضخيم الفاشلة وعينة لا ينبغي أن تكون متسلسلة (الشكل 2، والممرات 3 و 8).
وبعد إعداد مكتبة، ينبغي تقييم حجم التوزيع جزء من العينات من قبل الكهربائي الشعري على تحليل شظية.بنجاح المكتبات مستعدة سوف يكون بدلا حتى توزيع أحجام جزء 150-900 سنة مضت (الشكل 3، A، B). وإعداد مكتبة فشلت يؤدي إلى توزيع يميل حجم جزء، وينبغي أن مثل هذه المكتبات لا تكون متسلسلة (الشكل 3C).
ومعالجة البيانات التسلسل من خلال نموذج ماركوف الخفي (HMMcopy) وتجزئة الثنائية دائرية (DNAcopy) تحليل جينوم كل خلية في أجزاء من عدد نسخ المقدرة. ويمكن بعد ذلك تتم تصفيته هذه الشرائح لتحديد أولئك الذين لديهم عدد النسخ المقدرة متوافقة مع الربح أو الخسارة في خلية واحدة (الجدول 1). ثم ينبغي أن تتداخل هذه القطاعات التي تمت تصفيتها من HMMcopy وDNAcopy لتحديد ارتفاع الثقة التنوعات في عدد النسخ.
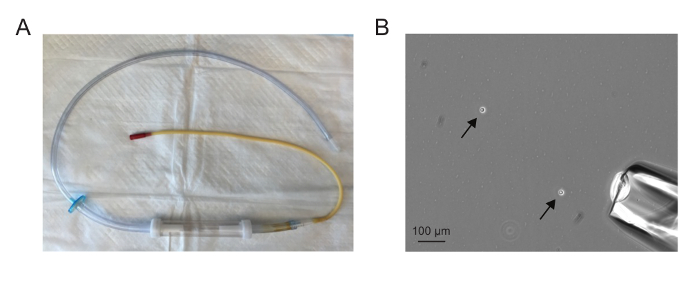
الشكل 1. عزل خلية واحدة. ( أ) تجميعها هناك microaspirator. تبين (ب) المجال و10X فصل الخلايا (السهام) والإبرة microaspirator (الزاوية اليمنى السفلى). الشفط الخلايا واحد هو أسهل عندما يكون هناك ما بين واحد وخمسة خلايا في حقل 10X. الرجاء انقر هنا لعرض نسخة أكبر من هذا الرقم.
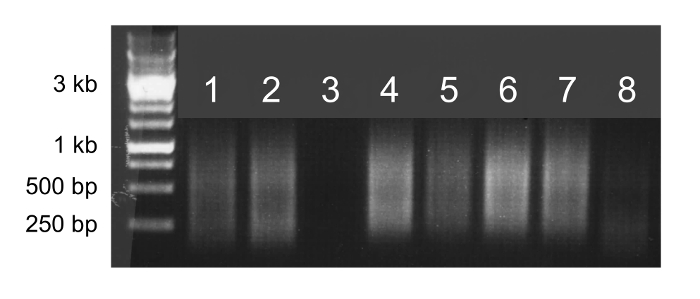
الشكل 2. كله تضخيم الجينوم. الكهربائي للهلام الاغاروز من 5 ميكرولتر من المنتجات الجينوم التضخيم بأكملها. العينات التي تضخيم بنجاح سوف تظهر مسحات كما مشرقة من 100 نقطة أساس إلى 1 كيلو بايت (حارات 1 و 2 و 4 و 5 و 6 و 7)، ويمكن أن تكون متسلسلة. العينات التي لا تضخيم بنجاح سوف ينتج مسحة باهتة أو أي تشويه (الحارات 3 و 8)، وينبغي ألا تكون متسلسلة.large.jpg "الهدف =" _ فارغة "> الرجاء انقر هنا لعرض نسخة أكبر من هذا الرقم.
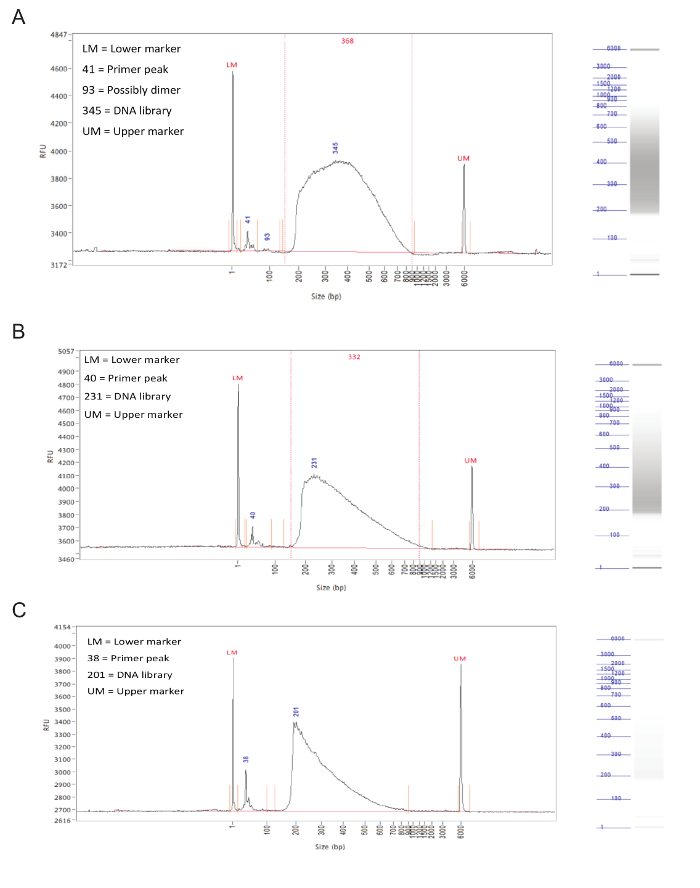
الشكل 3. إعداد مكتبة. نتائج ممثلة من محلل شظية. وتظهر الرسوم البيانية حجم جزء (في بي بي) على محور X وحدات مضان النسبي (RFU) على محور Y. إلى اليمين من كل رسم بياني هو هلام ممر محاكاة. (A) النتائج لعينة مثالية، حتى مع توزيع ما بين 150 و 900 نقطة أساس ودون القمم الحادة أو انحياز جانب واحد. هذه عينة مقبولة للتسلسل. تفاوت (ب) نتائج من عينة بخير، مع حجم التوزيع نحو الأحجام شظية أقل. في حين أن هذا ليس الأمثل، لا يزال العينة تكون متسلسلة. (C) النتائج من عينة فشلت، مع أحجام جزء في الغالب صغيرة. من المرجح ان تسبب ذلك عن طريق حضانة طويلة خلال الخطوة tagmentationمن إعداد مكتبة. لا ينبغي أن تكون متسلسلة هذه العينة. الرجاء انقر هنا لعرض نسخة أكبر من هذا الرقم.
| HMMcopy | |||||||
| عينة | قطعة | مركز حقوق الانسان | بداية | النهاية | حالة | الوسيط | |
| D15-4998 | 23 | chr8 | 144500001 | 146500000 | 6 | 0.5008794 | |
| D15-4998 | 29 | chr10 | 67000001 | 134500000 | 6 | 0.4031945 | |
| D15-4998 | 52 | جhr19 | 1 | 20،000،000 | 6 | 0.4616884 | |
| D15-4998 | 57 | chrY | 1 | 59500000 | 2 | -1،506532 | |
| DNAcopy | |||||||
| عينة | معدن الكروم | بداية بن | نهاية بن ل | بداية | النهاية | SEG متوسط | ميد SEG |
| D15-4998 | chr10 | 88 | 197 | 62612945 | 129971511 | 1.4688 | 0.157 |
| D15-4998 | chr19 | 0 | 31 | 0 | 28416392 | 1.4141 | 0.1674 |
| D15-4998 | chrX | 77 | 126 | 51659160 | 95343369 | 1.3548 | 0.1874 |
| D15-4998 | chrY | 0 | 14 | 0 | 23805358 | -2.7004 | 0.3591 |
الجدول 1. تحليل البيانات. شرائح المصفاة التي تولدها HMMcopy وDNAcopy من خلية واحدة. تداخل هاتين النتيجتين يكشف عن الربح من كروموسوم 10 67-130 ميجا بايت فضلا عن تحقيق مكاسب على الصبغي 19 0-20 ميجا بايت. لقد وجدنا أن المكاسب على الجزء القريب من كروموسوم 19 هي قطعة أثرية من خلية واحدة تسلسل 12.
Discussion
تقليديا، وتحديد التنوعات وعدم توازن الصبغيات على مستوى الخلايا وحيدة المطلوبة طرق سيتولوجي مثل الأسماك والسماء. الآن، وقد برزت تسلسل خلية واحدة كنهج بديل لمثل هذه الأسئلة. التسلسل خلية واحدة لديها مزايا أكثر من الأسماك وSKY حيث أنها الجينوم على نطاق وعالية الدقة. وعلاوة على ذلك، عندما يتم تطبيق الأساليب المناسبة لمراقبة الجودة، والتسلسل خلية واحدة يمكن أن توفر تقييما أكثر موثوقية من التنوعات وعدم توازن الصبغيات كما أنها ليست عرضة للتهجين ونشر الأعمال الفنية الأصيلة للأسماك وSKY. ومع ذلك، فإن العديد من التطبيقات الحديثة التسلسل خلية واحدة لم يتم إثباتها عن طريق تقييم شامل للحساسية وخصوصية الطرق والتحليلات. في الواقع، وترتبط بعض المناهج التحليلية المستخدمة من قبل دراسات أخرى مع ترددات عالية (> 50٪) من CNV ايجابية كاذبة يدعو 12. النهج وصفنا وقد تم اختبار صارم باستخدام خلايا KNعبء CNV الخاصة من أجل تحديد معدلات اكتشاف الصواب والخطأ وتحسين حساسية وخصوصية كشف CNV 12. عن طريق مراقبة الجودة والأساليب التحليلية الموصوفة في هذا البروتوكول، ما يقرب من 20٪ من 5 المكاسب ميجا بايت، 75٪ من 5 خسائر ميغابايت، وجميع التنوعات تزيد عن 10 ميجا بايت يمكن الكشف عنها. على الرغم من تحديد معدل اكتشاف كاذبة التسلسل خلية واحدة أمر صعب، ونحن قد يقدر لها أن تكون أقل من 25٪. ويمكن تطبيق هذا البروتوكول إلى الخلايا من مجموعة متنوعة من المصادر، والنصوص يمكن تعديلها لضبط دقة الكشف CNV، والبروتوكول يمكن تكييفها لتحديد أنواع أخرى من التعديلات الجينوم.
وهناك مجموعة متنوعة من وسائل النأي أنسجة جديدة في زنازين انفرادية، والعديد من المنشورات يصف الإجراءات الأمثل لأنسجة معينة، مثل الجلد 24 والدماغ 25. ونحن نفضل أن عزل الخلايا واحد من قبل microaspiration لأنها تسمح FO ص التقييم البصري للكل خلية لتكون متسلسلة. ومع ذلك، فمن الممكن أيضا أن عزل الخلايا واحد من قبل مضان تنشيط الخلايا الفرز (FACS) 26 وأجهزة ميكروفلويديك 27. إذا تم تنفيذ العزلة خلية واحدة وكله الجينوم التضخيم يدويا، فمن المعقول أن عزل وتضخيم ما يصل إلى أربعين الخلايا في جلسة واحدة. من أجل الحصول على بيانات خلية واحدة تسلسل جودة عالية، فمن الأهمية بمكان أن التضخيم من الجينوم خلية واحدة غير موحدة وكاملة. نجد أن نوعية الخلايا وحيدة معزولة فضلا عن كفاءة تحلل والتضخيم لديها تأثير كبير على نوعية البيانات التسلسل. على هذا النحو، ينبغي أن تحصد الخلايا من بيئتها المحلية قبيل عزل وكامل الجينوم التضخيم ينبغي أن تبدأ على الفور بعد أن يتم عزل الخلايا. وعلاوة على ذلك، ينبغي اتباع خطوة تحلل وتفتت بالضبط كما هو موضح في 2،3-2،6 الخطوات.
"> والخوارزميات يمكن تعديلها لتغيير دقة الكشف CNV، مع معارضة تأثيرات على حساسية وخصوصية (12). ومن الممكن أيضا لضبط عتبات للكشف عن كامل الكروموسوم عدم توازن الصبغيات في تحديد الرباعي (11). ومع ذلك، نجد أن يقتصر مقاربتنا للكشف عن التنوعات أكبر من 5 ميغابايت، كما الضجيج قدم خلال كامل الجينوم التضخيم يعقد الكشف عن المتغيرات أصغر 12. يجب أن التحسينات المستقبلية في النهج الجينوم التضخيم كلها تعزز في نهاية المطاف قرار الكشف CNV باستخدام التسلسل خلية واحدة.التسلسل خلية واحدة يسمح للتحقيق ليس فقط للتغييرات في عدد النسخ ولكن أيضا اختلافات النووية المنفردة 28 و 29 و التباين الهيكلي 30. بروتوكول لدينا لعزل خلية واحدة يمكن تطبيقها على الإجابة على هذه كيو الآخرينstions. ومع ذلك، واختيار كامل طريقة الجينوم التضخيم يعتمد على تطبيق معين. الطريقة الموصوفة في هذا البروتوكول، الذي يقوم على تفاعل البلمرة المتسلسل، هو الانسب للكشف عن تغييرات في عدد النسخ لأنه يرتبط مع انخفاض مستويات التضخيم التحيز 32. للتحقيق في أنواع أخرى من التعديلات الجينومية، مثل النوكليوتيدات المفردة، ويعتقد أن أساليب أخرى لكامل الجينوم التضخيم لتكون أكثر ملاءمة 31، 32.
Disclosures
الكتاب ليس لديهم ما يكشف.
Acknowledgements
نشكر ستيوارت ليفين للتعليقات على هذه المخطوطة. وأيد هذا العمل من قبل المعاهد الوطنية للصحة منح GM056800 وكاثي وصندوق أبحاث السرطان كيرت الرخام لانجيليكا آمون والجزء الآخر من معهد كوخ منحة دعم P30-CA14051. أنجيليكا آمون هو أيضا محقق من معهد هوارد هيوز الطبي ومؤسسة جلين للبحوث الطبية الحيوية. ويدعم كاك من المنحة T32GM007753 NIGMS التدريب.
Materials
| Name | Company | Catalog Number | Comments |
| Aspirator tube assemblies for calibrated microcapillary pipettes | Sigma | A5177 | |
| PVC tubing (ID 3/16", OD 5/16", ~1 foot) | VWR | 89068-500 | |
| PVC tubing (ID 5/16", OD 7/16", ~6 inches) | VWR | 89068-508 | |
| Acrodisc Syringe Filter with HT Tuffryn Membrane (diameter 25 mm, pore size 0.2 µm) | VWR | 28144-040 | |
| Serological pipette (5 mL) | BioExpress | P-2837-5 | Any plastic 5 mL pipette should suffice |
| In-Line Water Trap for Oxygen Use | Amazon.com | 700220210813 | |
| Capillary Melting Point Tubes (ID 0.8-1.1 mm, 100 mm length) | VWR | 34502-99 | |
| Modeling clay | VWR | 470156-850 | |
| Petri dish (150 mm diameter) | VWR | 25384-326 | Surface should be non-adherent to facilitate aspiration of single cells |
| Hard-Shell Full-Height 96-Well Semi-Skirted PCR Plates | Bio-Rad | HSS9601 | Any 96-well PCR plate should suffice |
| 96-well tissue culture plate | VWR | 62406-081 | Any 96-well tissue culture plate should suffice |
| GenomePlex Single Cell Whole Genome Amplification Kit | Sigma | WGA4 | |
| Microseal 'A' Film | Bio-Rad | MSA5001 | |
| Mini plate spinner | Thomas Scientific | 1225Z37 | |
| Thermal cycler | Bio-Rad | 1861096 | Any thermal cycler should suffice so long as it can accommodate a 96-well PCR plate |
| Agencourt Ampure Beads | Beckman Coulter | A63880 | |
| Dynamag-2 Magnet | Thermo Fisher Scientific | 12321D | Any similar magnetic tube strip should suffice |
| Nextera XT DNA Library Preparation Kit | Illumina | FC-131-1096 | |
| Nextera XT Index Kit | Illumina | FC-121-1012 | |
| Complete kit (optimized for Roche LightCycler 480) | Kapa Biosystems | KK4845 | This kit is optimized for the Roche LightCycler 480 real-time PCR machine. If using a different machine, substitute a kit optimized for your machine. |
References
- Kahlem, P. Transcript Level Alterations Reflect Gene Dosage Effects Across Multiple Tissues in a Mouse Model of Down Syndrome. Genome Res. 14 (7), 1258-1267 (2004).
- Torres, E. M. Effects of Aneuploidy on Cellular Physiology and Cell Division in Haploid Yeast. Science. 317 (5840), 916-924 (2007).
- Itsara, A. Population Analysis of Large Copy Number Variants and Hotspots of Human Genetic Disease. Am. J. Human Gen. 84 (2), 148-161 (2009).
- Sudmant, P. H. Global diversity, population stratification, and selection of human copy-number variation. Science. 349 (6253), aab3761 (2015).
- Navin, N. Inferring tumor progression from genomic heterogeneity. Genome Res. 20 (1), 68-80 (2010).
- Torres, L., Ribeiro, F. R., Pandis, N., Andersen, J. A., Heim, S., Teixeira, M. R. Intratumor genomic heterogeneity in breast cancer with clonal divergence between primary carcinomas and lymph node metastases. Breast cancer res and treat. 102 (2), 143-155 (2007).
- Yates, L. R. Subclonal diversification of primary breast cancer revealed by multiregion sequencing. Nat. Med. 21 (7), 751-759 (2015).
- Forsberg, L. A. Age-related somatic structural changes in the nuclear genome of human blood cells. Am. J. Human Gen. 90 (2), 217-228 (2012).
- Laurie, C. C. Detectable clonal mosaicism from birth to old age and its relationship to cancer. Nat. Gen. 44 (6), 642-650 (2012).
- Jacobs, K. B. Detectable clonal mosaicism and its relationship to aging and cancer. Nat. Gen. 44 (6), 651-658 (2012).
- Knouse, K. A., Wu, J., Whittaker, C. A., Amon, A. Single cell sequencing reveals low levels of aneuploidy across mammalian tissues. Proc. Natl. Acad. Sci. 111 (37), 13409-13414 (2014).
- Knouse, K. A., Wu, J., Amon, A. Assessment of megabase-scale somatic copy number variation using single-cell sequencing. Genome Res. 26 (3), 376-384 (2016).
- . KAPA Library Quantification Kit Available from: https://www.kapabiosystems.com/product-applications/products/next-generation-sequencing-2/library-quantification/ (2016)
- . DNAcopy Available from: https://bioconductor.org/packages/release/bioc/html/DNAcopy.html (2016)
- Lichti, U., Anders, J., Yuspa, S. H. Isolation and short-term culture of primary keratinocytes, hair follicle populations and dermal cells from newborn mice and keratinocytes from adult mice for in vitro analysis and for grafting to immunodeficient mice. Nat. Protoc. 3 (5), 799-810 (2008).
- Brewer, G. J., Torricelli, J. R. Isolation and culture of adult neurons and neurospheres. Nat. Protoc. 2 (6), 1490-1498 (2007).
- Navin, N. Tumour evolution inferred by single-cell sequencing. Nature. 472 (7341), 90-94 (2011).
- Szulwach, K. E., Chen, P. Single-Cell Genetic Analysis Using Automated Microfluidics to Resolve Somatic Mosaicism. PLoS ONE. 10 (8), e0135007 (2015).
- Zong, C., Lu, S., Chapman, A. R., Xie, X. S. Genome-Wide Detection of Single-Nucleotide and Copy-Number Variations of a Single Human Cell. Science. 338 (6114), 1622-1626 (2012).
- Wang, Y. Clonal evolution in breast cancer revealed by single nucleus genome sequencing. Nature. 512 (7513), 155-160 (2014).
- Zhang, C. Z. Chromothripsis from DNA damage in micronuclei. Nature. 522 (7555), 179-184 (2015).
- Macaulay, I. C., Voet, T. Single Cell Genomics: Advances and Future Perspectives. PLoS Genetics. 10 (1), e1004126 (2014).
- Gawad, C., Koh, W., Quake, S. R. Single-cell genome sequencing: current state of the science. Nat. Rev. Gen. , 1-14 (2016).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved