Method Article
単一細胞のシーケンシングを使用したコピー数変化の検出
要約
単一細胞配列決定は、高解像度でゲノムの変化に対処するため、ますます人気とアクセス可能なツールです。我々は、単一の細胞内のコピー数の変化を識別するために、単一細胞の配列決定を使用するプロトコルを提供します。
要約
単細胞解像度におけるゲノム変化の検出は、正常組織、癌、微生物集団における遺伝的多様性と進化を特徴づけるために重要です。遺伝的異質性を評価するための従来の方法は、低解像度、低感度、および/または低特異性によって制限されています。単一細胞配列決定は適切に分析し、高解像度、高感度かつ、高い特異性で遺伝的多様性を検出するための強力なツールとして浮上しています。ここでは、単一細胞の単離、全ゲノム増幅、シーケンシング、および分析のためのプロトコルを提供します。我々のアプローチは、単一細胞におけるメガベーススケールコピー数変異体の信頼性の同定を可能にします。しかしながら、このプロトコルの態様は、単一細胞における遺伝子変化の他のタイプを調査するために適用することができます。
概要
DNAコピー数の変化は、いくつかの塩基対(コピー数変異体()のCNV)から全染色体(異数性)のサイズの範囲であり得ます。ゲノムの大部分に影響を与えるコピー数変化は遺伝子1、2、数千までの発現を変化させることにより、大幅な表現型の結果を持つことができます。集団の全ての細胞に存在しているのCNVは、バルク配列決定またはマイクロアレイに基づく方法3,4により検出することができます。しかし、集団はまた、人口、あるいは単一細胞のサブセットで既存のCNVと、遺伝的に不均一であることができます。遺伝的異質性は、未知の結果5、6、7、8、9で、また、正常組織に存在する腫瘍の進化を駆動し、癌において一般的であり、かつP>、10。
伝統的に、遺伝的異質性は、細胞学的アプローチまたはバルク配列決定によっていずれかを評価しました。このような蛍光in situハイブリダイゼーション(FISH)、染色体スプレッド、スペクトル核型分析(SKY)のような細胞学的手法は、個々の細胞内に存在する変化を同定する利点を有するが、ハイブリダイゼーションおよび11の拡散アーチファクトによる高いエラー率を有します。これらのアプローチはまた、いくつかのメガベースに及ぶ彼らの解像度のみ明らかコピー数変化に限定されています。バルクのDNAの配列決定またはマイクロアレイは、精度と分解能が高いものの、あまり敏感です。集団ベースのアプローチによって遺伝的多様性を検出するために、変異体は、集団中の細胞のかなりの割合で存在しなければなりません。単一細胞からのゲノムDNAを増幅する方法の出現は、それが可能な単一細胞のゲノムの配列を決定することが可能となりました。単一細胞sequencingは、高解像度、高感度、かつ、適切な品質管理方法が適用され、高精度12の利点を有します。
ここでは、単一細胞におけるメガベーススケールコピー数変化を検出するための方法を記載します。私たちは、微小誤嚥により単一細胞を単離し、リンカー - アダプターPCRを用いてゲノムDNAを増幅し、次世代シーケンシングのためのライブラリを準備し、隠れマルコフモデルと円形バイナリセグメンテーションの両方でコピー数の変異体を検出します。
プロトコル
1.単一細胞を単離します
- microaspiratorを準備
- 吸引管アセンブリから透明なプラスチックの端を削除し、3月16日「内径1フィート長PVCチューブの一方の端部に狭い方の端を挿入します。
- 3/6 "内径1フィート長PVCチューブのもう一方の端に0.2μmシリンジフィルターの出口を挿入します。
- 5/16 "内径6インチの長さのPVC管の一端に0.2μmシリンジフィルタの入口に挿入します。
- オプション:半分に5/16 "内径6インチの長さのPVCチューブをカットし、二つの半分の間のインライン水トラップを挿入します。
- 1 mLの目盛りで5 mLのプラスチック血清学的ピペットを破ると5/16 "内径PVC管の開放端に切断端を挿入します。
- 吸引管アセンブリの柔軟なエンド(透明なプラスチックの端に5 mLのプラスチック血清学的ピペットの出口を挿入します)は削除されていました。
- ストレージについては、滅菌プラスチックチューブで吸引管アセンブリの赤いマウスピースをカバーしています。
- ブンゼンバーナーの炎の近くに管の中央をもたらし、チューブの両端に張力を印加することにより、10〜30ミクロンの内径にガラス毛細管を引き出します。 2潜在的な吸引針を作成するために、中央に描かれたチューブを破ります。唯一のいくつかのチューブが単一細胞をピッキングに適した内径で終わるように、いくつかのチューブと、この手順を繰り返します。
- ストレージについては、15センチメートルペトリ皿にモデリング粘土の2つのストリップを配置し、ストリップを横切って吸引針を固定します。
- セルを選択してください
- 細胞の種類や実験のための適切な単一細胞懸濁液を準備します。例えば、適切な培地を含むコニカルチューブにトリプシン処理および転送細胞によるヒト線維芽細胞株、収穫細胞と接着細胞を調製しました。
- 前、中、またはA単一細胞懸濁液(これは、単一細胞懸濁液を調製するのにかかる時間の長さに応じて)、セットアップ全ゲノム増幅のためのフードを準備FTER。
- 10%の漂白剤でフード、ピペットチップボックス、およびピペットチップの表面を下にスプレーし、紙タオルで拭いてください。 70%エタノールで、この手順を繰り返します。
- 配列決定される各セルに対して1つのウェル、全ゲノム増幅(WGA)キットの96ウェルPCRプレートの個々のウェルに水の8μLを追加します。氷上で96ウェル組織培養プレートと場所の蓋付き96ウェルPCRプレートを覆います。
- 15 cmのペトリ皿に10mlの培地またはリン酸緩衝生理食塩水(PBS)に1,000細胞を追加し、皿に付着した細胞を防ぐために、氷の上皿を置きます。
- 10X対物レンズの光顕微鏡に氷の上で蓋をペトリ皿内の細胞と96ウェルPCRプレートを持参してください。
- そっと目の引き出された端をタップしてアスピレータ針の開口部を増やします硬い表面上の電子吸引器の針は、先端が途切れるようになっています。 microaspiratorの明確な端部に吸引針の広い方の端を差し込みます。
- 顕微鏡ステージ上の細胞を含むペトリ皿を置きます。口の中に吸引器の赤いマウスピースを置きます。細胞を含有するペトリ皿を移動するために吸引針ともう一方の手を移動するために片手を使用してください。配列決定される単一細胞を識別します。
- 口の吸引を使用して、メディアまたはPBSの〜1-2μLと一緒に吸引針に1つのセルを描画します。 96ウェルPCRプレートの1ウェル内の水の8μLに細胞を転送します。セルを転送する際に気泡を導入することは避けてください。
- 細胞の所望の数が単離されているまで、ステップ1.2.7を繰り返します。氷上でPCRプレートを保持し、ピッキング細胞との間で蓋をし。細胞を受けた井戸をマークします。
- 単一細胞を採取しながら、全体のGから10X単一細胞溶解および断片化緩衝液を解凍enome増幅キット。細胞の所望の数をピッキングした後、全ゲノム増幅にすぐに進んでください。
2.全ゲノム増幅
- 、全ゲノム増幅中の汚染を防止するための組織培養フード内のすべての試薬を追加し、フィルターをピペットチップを使用し、ウェル間にピペットチップを変更します。
- 全ゲノム増幅(WGA)キットから作業を溶解および断片化緩衝液を準備します。最大32セルの各セットについて、マイクロチューブにプロテイナーゼK溶液の10倍単一細胞溶解および断片化緩衝液および2μLの32μLを兼ね備えています。溶液を混合し、チューブをボルテックスし。
- 作業を各ウェルに溶解および断片化緩衝液およびピペットアップの1μLを加え、ミックスダウンします。
- プレートをカバーし、プラスチックフィルムで全てのウェルを密閉します。簡単に言えば、ミニプレートスピナーでプレートを遠心します。
- follとして熱サイクルOWS:50°C、1時間および99℃、4分。
- 氷の上でプレートを冷却し、簡潔にミニプレートスピナーでプレートを遠心分離します。
- 全ゲノム増幅キットから作業ライブラリー調製緩衝液を準備します。各セルについて、1xシングルセルライブラリ調製緩衝液とマイクロチューブ内のライブラリ安定化溶液の1μLの2μLを兼ね備えています。同じマイクロ遠心チューブ内の複数のセルのための作業溶液を準備します。
- プラスチックフィルムを取り外し、各ウェルに、ライブラリの準備緩衝液を作業の3μLを追加します。混合するだけでなく、上下の内容をピペット。プラスチックフィルムを交換してください。
注意:ウェル膜のボア孔を開始するまで、プラスチックフィルムは、全ゲノム増幅プロセスを通じて再利用することができます。これが発生すると、新しいプラスチックフィルムに切り替えます。 - 簡単に言えば、ミニプレートスピナーでプレートを遠心分離し、95℃で2分間インキュベートします。
- 氷と簡単にプレートを冷却ミニプレートスピナーでプレートを遠心します。
- プラスチックフィルムを取り外し、各ウェルにライブラリ準備酵素1μLを追加し、混合するだけでなく、上下の内容をピペット。このステップ全体で氷の上またはコールドブロック内のライブラリの準備酵素を保管してください。プラスチックフィルムを交換してください。
- 16°C、20分間、24°C、20分間、37°C、20分間、75℃、5分間、4℃で保持する。簡単に説明すると次のようにミニプレートスピナー及び熱サイクルでプレートを遠心分離します。
- 氷の上でプレートを冷却し、簡潔にミニプレートスピナーでプレートを遠心分離します。
- 全ゲノム増幅キットからの作動増幅ミックスを調製します。各セルについて、48.5μLの水、7.5μL10倍増幅マスターミックス、およびマイクロ遠心チューブ中5μLWGA DNAポリメラーゼを兼ね備えています。同じマイクロ遠心チューブ内のいくつかのセルのために働いてミックスを調製します。氷の上で作業のミックスをしてください。
- プラスチックフィルムを外し、増幅混合物を作業61μLを追加よく上下との各ウェル、およびピペットの内容に混合します。氷の上またはこのステップを通して冷たいブロックで働く増幅混合物を保管してください。プラスチックフィルムを交換してください。
- 95℃、3分、94°Cの25サイクル、30秒、65℃、5分間、4℃で保持する:簡単に言うと次のようにミニプレートスピナー内プレートと熱サイクルを遠心分離します。
- 別々のマイクロ遠心チューブや店舗で、各サンプルの60μLを移し - 20°Cステップ3で使用するために。
- 各サンプル(サンプルの15μLにすなわち 3μL6倍のDNAローディング色素)の残り容量にDNAローディング色素を追加し、1%アガロースゲル上の各反応から5μLを実行します。
注:正常に増幅された細胞は、250〜1,000塩基対からスメアとして表示されます。スミアを表示したり、かすかなスメアを示していないサンプルは、有用な配列決定データを得にくいです。
3.シーケンシング
- 常磁性ビーズでサンプルを精製します。
- ター氷上のワットDNAサンプル。渦常磁性ビーズ溶液が均質であり、少なくとも30分間室温でビーズをインキュベートするまで。
- きれいな微量遠心チューブに各DNAサンプルの20μLを転送します。サンプルの残りは-20℃で保存することができます。
- 各DNAサンプルと混合する渦に常磁性ビーズの30μL(1.5倍)を追加します。 10分間、室温でサンプルをインキュベートします。ステップ3.4で使用するために、室温でビーズの原液のままにしておきます。
- 2分間の磁性体管ストリップ上にチューブを置き、またはビーズは、ペレットを形成し、上澄み液が透明になるまで。
- P200のピペットを用いて、ビーズを乱すことなく、可能な限り上清をできるだけ多く除去します。
- DNA-ビーズ混合物に80%エタノールを180μLを追加します。エタノール溶液を介してビーズを「すすぎ」にチューブを磁石に比べて数回回転させます。
- P200のピペットを用いて、possiとしてエタノール洗浄の限りを削除ビーズを乱すことなくBLE。
- 3.1.6と3.1.7を繰り返します。
- 空気は約10分間、またはそれ以上のエタノールが見えなくなるまでビーズを乾燥させます。ビーズが割れ現れたり、チューブの壁から落ち、次のステップに進みます。
- 磁気ストライプからサンプルを削除します。ビーズからDNAを溶出し、ビーズを再懸濁したサンプルをボルテックスする10 mMトリスpHが8.0の40μLを追加します。室温で2分間インキュベートします。
- 簡潔には、チューブの底に液体を収集し、少なくとも二つ分間磁気チューブストリップにチューブを配置するために、サンプルを遠心します。ビーズを乱すことなく、きれいな微量遠心チューブに溶出液を移します。
- サンプルの正規化
- 分光光度計を用いて、各試料の濃度を定量します。濃度は10から30 ngの/μLの範囲であるべきです。
- 10 mMトリスpHは8.0を使用して、0.2 ngの/μLに、各試料を希釈します。次の手順は、最小私を必要としますこの希釈液から5μLのNPUT。
- ライブラリの準備
- ライブラリー作製キット13の指示に従ってシーケンシングライブラリを準備します。
- 最終的なクリーンアップ
- ステップ3.1に従ってサンプルを清掃してください。 50塩基対、75塩基対、または150塩基対の読み取り長さについて、それぞれ、1.5倍、1倍、または0.6xのボリュームをサンプリングするために、ビーズの比率を使用しています。 10 mMトリスpHが8.0の15μLで溶出。
- ライブラリー14のサイズ分布を確認するためにフラグメント分析のサンプルを実行します。サイズ分布は均等に150から900塩基対に分散させる必要があります。
- 定量PCRを使用してサンプルを定量化
- ライブラリー定量キットを使用して、キットの説明書15に従って定量PCR反応を設定します。正の標準(キットからのDNA標準)と負の標準(水または他のブランク溶液)を追加する列を空白のままにします。
- FOLとして熱サイクル安値:95°C、5分(4.8℃/秒のランプ速度)と95℃、30秒(4.8℃/秒のランプ速度)と2.5の60°C、45秒(ランプ速度の35サイクル°C /秒)。
- プーリング
- フローセル上の多くのレーンが必要とされる方法を決定し、レーンあたり1グループと、グループにサンプルを分けます。
- サンプルのグループごとに、ステップ3.5.2からのqPCRデータに基づいて、最も低い濃度のサンプルを選択し、10 mMトリスpHは8.0を使用してその濃度に、そのグループ内のすべてのサンプルを正規化します。
- 一緒に、各グループ内のサンプルをプール。
- シークエンシング
- 標準的な手順16以下の次世代シーケンシングマシンの負荷プール。
4.データ解析
注:UNIXベースの環境は、このセクションではプログラムやスクリプトを実行するために必要です。その中で、以下のプロトコールに記載されたソフトウェアをインストールします。ンストールガイド。すべてのスクリプトはhttps://sourceforge.net/projects/singlecellseqcnv/で見つけることができます。
- FASTX-Toolkitのバージョン0.0.13 17からfastx_trimmerを使用して、40-ntのに読み込みトリム。
fastx_trimmer -Q33 -i example.fastq -l 40 -o example.trim.fastq - デフォルトのオプション18でBWAのバージョン0.6.1を使用して、適切な参照ゲノム(マウス用MM9、人間のためのhg19)に読み込む合わせます。
BWAのAlN mm9.fa example.trim.fastq> example.sai
BWA samse mm9.fa example.sai example.trim.fastq> example.sam - SAMToolsバージョン0.1.19 19を使用して、得られた BAMファイルにcHRMとランダム染色体への整列を削除し、その後、ソートとインデックス。
grepの-v -wにcHRM example.sam | grepの-vランダム> example.filtered.sam
samtoolsは-uSh example.filtered.samを見ます|ソートsamtools - example.filtered
samtoolsインデックスexample.filtered.bam - CNVを検出するためにHMMcopyを実行します。= "外部参照"> 20
- ゲノムのためのGCの割合参照ファイルを生成するHMMcopyにgcCounterを使用してください。ウィンドウサイズを指定するには、「500000 -w」オプションを使用します。ステップ4.2で使用されるFASTA参照ファイルの同じバージョンを使用しますが、にcHRMとランダム染色体はFASTAファイルから削除されていることを確認してください。
500000 mm9.fa> mm9_gc.wig -w gcCounter - mappabilityためのウィグルファイルを生成するHMMcopyにgenerateMap.plを使用してください。長さを読んで指定するには、「40 -w」オプションを使用します。ステップ4.4.1で使用したのと同じFASTA参照ファイルを使用してください。
generateMap.pl -b mm9.fa
generateMap.pl -w 40 -i mm9.fa mm9.fa -o mm9.bigwig - ゲノムのためのmappability参照ファイルを生成するHMMcopyにmapCounterを使用してください。ウィンドウサイズを指定するには、「500000 -w」オプションを使用します。
500000 -w mapCounter mm9.bigwig> mm9_map.wig - 各BAMファイルのウィグルファイルを生成するHMMcopyにreadCounterを使用してください。
readCounter -w 500000 example.filtered.bam> input.wig - 4.4.1で上記生成されたファイル( 例えば 、mm9_gc.wig)および4.4.3( 例えば 、mm9_mapを使用し、(run_hmmcopy.mm9.rまたはrun_hmmcopy.hg19.r)Rスクリプトで参照ファイルへのパスを変更します。それぞれ、GC百分率参照ファイルとmappability参照ファイルを参照する変数GFILEとMFILE、用かつら)。そして、提供さRスクリプト」run_hmmcopy.mm9.r」または「run_hmmcopy.hg19.r」を実行します。
R CMD BATCHにrun_hmmcopy.mm9.r
または
R CMD BATCHにrun_hmmcopy.hg19.r - BAMファイルのセットのバッチ処理のために、単一のフォルダにBAMファイルを置くと、セグメントを呼び出すと変動スコア(VS)を計算するためのパッケージで提供されるスクリプト「HMMpipe.pl」を実行します。フォーマットに従ってください:
perlのHMMpipe.pl Folder_to_BAMfiles MM9
- ゲノムのためのGCの割合参照ファイルを生成するHMMcopyにgcCounterを使用してください。ウィンドウサイズを指定するには、「500000 -w」オプションを使用します。ステップ4.2で使用されるFASTA参照ファイルの同じバージョンを使用しますが、にcHRMとランダム染色体はFASTAファイルから削除されていることを確認してください。
- CNV 21を検出するためにDNAcopyを実行します。
- 一意にBAMファイルから読み込み、マップされた抽出するためにSAMtoolsバージョン0.1.19を使用してください。
samtools表示-h -F 0x0004のexample.filtered.bam | egrepの-i "^ @ | XT:U" | samtoolsは-Shuを表示 - > example.mapped.bam - 22ファイル BAMにPCR重複をマークするためにピカールバージョン1.94でMarkDuplicatesを使用してください。
MarkDuplicates.jar入力-jar javaの= example.mapped.bamのOUTPUT = example.nondup.bam METRICS_FILE = example.dup REMOVE_DUPLICATES =真 - 提供BEDファイル」mm9.500k.dynamic.win.bed」または「hg19.500k.dynamic.win.bed」23で定義済みの動的500キロバイトのマッピング可能なウィンドウのそれぞれに読み込むマッピングされたカウントするbedtoolsバージョン2.17.0でcoveragebedを使用します。
coverageBed -abam example.nondup.bam -b mm9.500k.dynamic.win.bed -counts |ソート-k1,1 -k2,2n> example.nondup.counts - 提供GC含量参照ファイル」mm9.500k.dynamic.win.fa.gc.txt」または「hg19.500k.dynamic.win.faに基づいて、リードカウントを正規化するために提供されるPerlスクリプト「normalizeGC.pl "を使用してください。 gc.txt」。
perlのnormalizeGC。PL mm9.500k.dynamic.win.fa.gc.txt example.nondup.counts> example.bam.norm.counts - セグメントを呼び出すために設けられたRスクリプト」dnacopy.r "を使用してください。
R CMDバッチdnacopy.r
- 一意にBAMファイルから読み込み、マップされた抽出するためにSAMtoolsバージョン0.1.19を使用してください。
- CNVを特定
- 4.4.6で計算されたVSが0.26を超えている細胞を除外します。
- 中央値は log 2比は0.4(推定ゲイン)未満または-0.35(推定損失)よりも大きいためにのみが含まれるように4.4.6にHMMcopyによって呼び出されたセグメントをフィルタリングします。
- セグメントは、平均0.6より大きく1.32以下であるもののみのために含まれるように4.5.5にDNAcopyによって呼び出されたセグメントをフィルタリングします。
- のみHMMcopyとDNAcopy両方が推定されるゲインまたは推定損失を特定の地域でのCNVを呼び出し、4.6.2および4.6.3からのセグメントをオーバーラップします。
結果
組み立てられた吸引器は、 図1Aの1のようになります。針はなく、とても広い大ボリュームは、単一のセルに引き上げられることを1つのセルを収容するのに十分な幅であるように描かれるべきです。 10Xフィールドに1と5細胞( 図1B)との間に存在する場合、単一の細胞を吸引することが最も簡単です。
全ゲノム増幅が成功した場合、試料は、アガロースゲル上のスメアとして現れる( 図2、レーン1、2、4、5、6、および7)であろう。かすかまたは不在スミアが失敗した増幅反応を示し、サンプルが(レーン3および8、 図2)配列決定されるべきではありません。
ライブラリーの調製後、試料の断片サイズ分布は、フラグメント分析のキャピラリー電気泳動によって評価されるべきです。正常に準備ライブラリは、150から900塩基対( 図3、A、B)にフラグメントサイズのかなり均一な分布を持つことになります。 ( 図3C)配列決定されるべきでないライブラリの準備がスキュー断片サイズ分布およびこのようなライブラリーになりませんでした。
隠れマルコフモデル(HMMcopy)と円形バイナリセグメンテーション(DNAcopy)を介して配列決定データを処理すると、推定コピー数のセグメントに各細胞のゲノムを解析します。これらのセグメントは、単一のセル( 表1)の利得または損失と一致推定コピー数を有するものを同定するために濾過することができます。 HMMcopyとDNAcopyからこれらのフィルタ処理のセグメントは、その後、高信頼のCNVを識別するためにオーバーラップする必要があります。
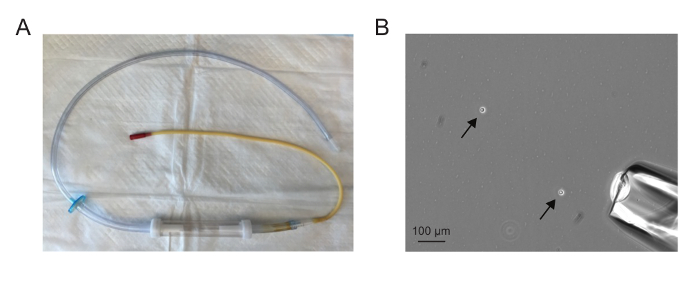
1.単一細胞の単離図。 ( A)Anがmicroaspiratorを組み立てました。 (B)10Xフィールドかを示すには、細胞(矢印)とmicroaspirator針(右下)を解離しました。 10Xフィールドに1〜5つのセルがある場合、単一の細胞を吸引することが最も簡単です。 この図の拡大版をご覧になるにはこちらをクリックしてください。
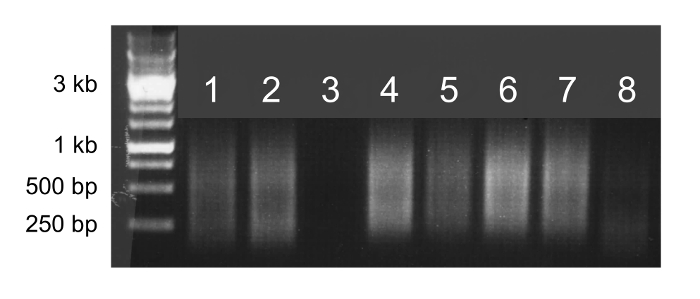
2.全ゲノム増幅図。全ゲノム増幅産物の5μlをアガロースゲル電気泳動。正常に1 kbの100塩基対から明るいスミアが現れる増幅サンプル(レーン1、2、4、5、6、および7)、配列決定することができます。正常に増幅しないサンプルは、かすかなスメアまたは全く汚れ(レーン3および8)を生成し、配列決定されるべきではありません。large.jpg "ターゲット=" _空白 ">この図の拡大版をご覧になるにはこちらをクリックしてください。
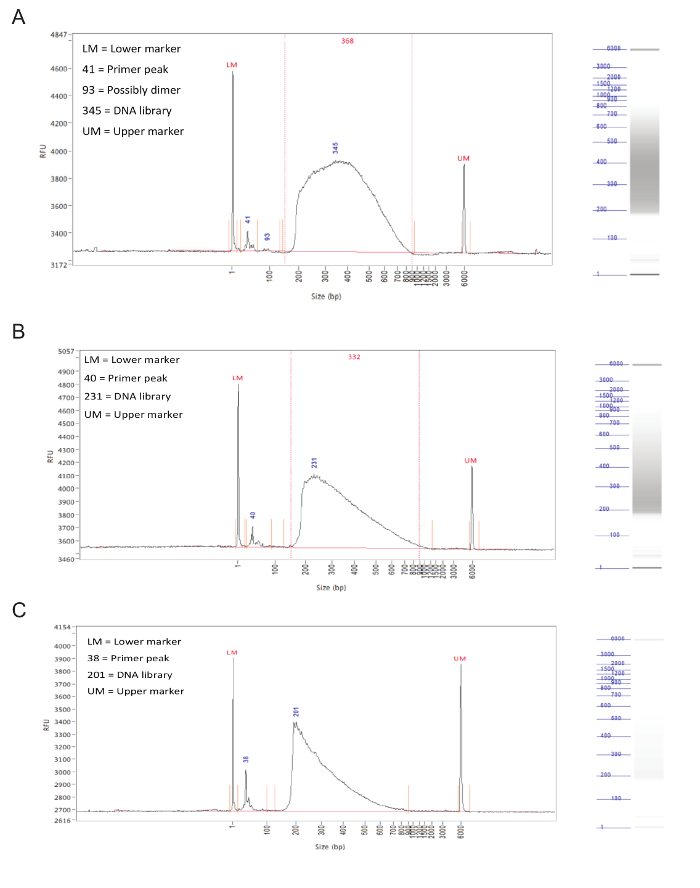
図3.ライブラリの準備。フラグメント分析から代表的な結果。グラフは、Y軸上にX軸、相対蛍光単位(RFU)で(BP)にフラグメントサイズを示します。各グラフの右側にシミュレートされたゲルのレーンです。 (A)は、150〜900 bpおよび鋭いピークまたは1側に偏りのない均一な分布で、理想的なサンプルについての結果。このサンプルでは、配列決定のために許容可能です。サイズ分布を有する大丈夫サンプルから(B)の結果は、下のフラグメントサイズに偏っ。これは最適ではないが、サンプルがまだ配列決定され得ます。 (C)は 、主に小断片サイズで、失敗したサンプルから得られた結果。これはおそらくtagmentationステップ中に長時間のインキュベーションによって引き起こされますライブラリの準備の。このサンプルでは、配列決定されるべきではありません。 この図の拡大版をご覧になるにはこちらをクリックしてください。
| HMMcopy | |||||||
| サンプル | セグメント | chrです | 開始 | 終わり | 状態 | 中央値 | |
| D15-4998 | 23 | chr8 | 144500001 | 1.465億 | 6 | 0.5008794 | |
| D15-4998 | 29 | chr10 | 67000001 | 1.345億 | 6 | 0.4031945 | |
| D15-4998 | 52 | C言語hr19 | 1 | 20,000,000 | 6 | 0.4616884 | |
| D15-4998 | 57 | chrY | 1 | 5950万 | 2 | -1.506532 | |
| DNAcopy | |||||||
| サンプル | CHROM | スタートビン | エンドビン | 開始 | 終わり | ワンセグ平均 | ワンセグMED |
| D15-4998 | chr10 | 88 | 197 | 62612945 | 129971511 | 1.4688 | 0.157 |
| D15-4998 | chr19 | 0 | 31 | 0 | 28416392 | 1.4141 | 0.1674 |
| D15-4998 | chrX | 77 | 126 | 51659160 | 95343369 | 1.3548 | 0.1874 |
| D15-4998 | chrY | 0 | 14 | 0 | 23805358 | -2.7004 | 0.3591 |
表1.データ分析。単一の細胞からHMMcopyとDNAcopyによって生成されたフィルタ処理セグメント。これら二つの結果が重複すると、67〜130メガビットから10番染色体上の利益だけでなく、20 MBに0から19番染色体上の利益を明らかにする。私たちは、19番染色体の近位部益は、単一細胞シーケンシング12の人工物であることを見出しました。
ディスカッション
伝統的に、このようなFISHおよびSKYなどの細胞学的方法を必要とした単一細胞のレベルでのCNVおよび異数性を識別する。今、単一のセル配列決定は、そのような質問のための代替的なアプローチとして浮上しています。それはゲノムワイドと高解像度の両方とおり、単一のセル配列決定は、FISHおよびSKYを上回る利点があります。適切な品質管理方法が適用される場合、それはFISHおよびSKYに固有のハイブリダイゼーションおよび拡散アーティファクトの影響を受けないようにまた、単一のセル配列決定は、より信頼性のCNVの評価と異数性を提供することができます。しかし、単一の細胞配列の最近のアプリケーションの多くは、方法および分析の感度及び特異性の徹底的な評価によって実証されていません。実際、他の研究で使用される分析的アプローチのいくつかは、偽陽性のCNV 12を呼び出すの高周波数(> 50%)に関連しています。私たちが説明する手法は、厳密にKNの細胞を使用してテストされています真と偽発見率を決定し、CNV検出12の感度と特異性を最適化するために独自のCNV負担。このプロトコルに記載の品質管理および分析手法を用いて、5 MBの利得の約20%、5 MBの損失の75%、及び10メガビットを超えるすべてのCNVを検出することができます。単一セル配列の偽発見率を決定することは困難であるが、我々はそれが25%未満であると推定しています。このプロトコルは、さまざまなソースからの細胞に適用することができ、スクリプトは、CNVの検出の分解能を調整するために変更することができ、プロトコルは、ゲノム変化の他のタイプを識別するように構成することができます。
そこに単一細胞に新鮮な組織解離の様々な手段があり、多くの刊行物は、皮膚24および脳25のような特定の組織のために最適化された手順を説明します。それはfoを可能にするように私たちは、微小誤嚥により単一細胞を単離することを好みます各セルのR視覚的評価を配列決定します。しかし、蛍光活性化細胞選別(FACS)26と、マイクロ流体デバイス27によって単一細胞を単離することも可能です。単一細胞の単離および全ゲノム増幅が手動で実行された場合、単離および単一座っに40セルまで増幅することが合理的です。高品質の単セルの配列決定データを得るためには、単一細胞のゲノムの増幅が均一かつ完全であることが重要です。我々は、溶解および増幅の効率、ならびに単離された単一細胞の品質は、配列決定データの品質に大きな影響を与えることを見出しました。このように、直前に分離し、全ゲノム増幅にその天然環境から採取されるべき細胞は、細胞が単離されている直後に開始すべきです。また、溶解および断片化のステップは、ステップ2.3から2.6に記載とおりに従うべきです。
">アルゴリズムは、感度および特異性12に対向する効果と、CNVの検出の解像度を変更するように調整することができる。四倍11の設定で全染色体の異数性を検出するための閾値を調整することも可能であるが、我々は発見します全ゲノム増幅の間に導入されたノイズが小さく変種12の検出を複雑として我々のアプローチは、より大きい5メガビットのCNVを検出することに限定されています。全ゲノム増幅アプローチにおける今後の改善が、最終的に単一細胞の配列決定を使用して、CNV検出の分解能を向上させる必要があります。単一細胞シーケンシングだけでなく、コピー数変化の調査だけでなく、一塩基変異28、29および構造的変化30を可能にします。単一細胞単離のための私たちのプロトコルは、これらの他のQUEに答えるためにも適用することができますstions。しかし、全ゲノム増幅方法の選択は、特定の用途に依存します。ポリメラーゼ連鎖反応に基づくもので、このプロトコルに記載された方法は、それが増幅偏り32のより低いレベルに関連付けられているため、コピー数の変化を検出するのに最適です。そのような一塩基多型などのゲノム変化の他のタイプを調べるために、全ゲノム増幅の他の方法は、31、32、より適していると考えられます。
開示事項
著者らは、開示することは何もありません。
謝辞
我々は、この原稿上のコメントのスチュアート・レヴァインに感謝します。この作品は、コッホ研究所のサポート・グラントP30-CA14051によりアンゲリカアモンに部分的には健康グラントGM056800とキャシーとカートマーブルがん研究基金の国立研究所によってサポートされていました。アンジェリカアモンはまた、ハワード・ヒューズ医学研究所と生物医学研究のためのグレン財団の研究者です。 KAKはNIGMSトレーニンググラントT32GM007753でサポートされています。
資料
| Name | Company | Catalog Number | Comments |
| Aspirator tube assemblies for calibrated microcapillary pipettes | Sigma | A5177 | |
| PVC tubing (ID 3/16", OD 5/16", ~1 foot) | VWR | 89068-500 | |
| PVC tubing (ID 5/16", OD 7/16", ~6 inches) | VWR | 89068-508 | |
| Acrodisc Syringe Filter with HT Tuffryn Membrane (diameter 25 mm, pore size 0.2 µm) | VWR | 28144-040 | |
| Serological pipette (5 mL) | BioExpress | P-2837-5 | Any plastic 5 mL pipette should suffice |
| In-Line Water Trap for Oxygen Use | Amazon.com | 700220210813 | |
| Capillary Melting Point Tubes (ID 0.8-1.1 mm, 100 mm length) | VWR | 34502-99 | |
| Modeling clay | VWR | 470156-850 | |
| Petri dish (150 mm diameter) | VWR | 25384-326 | Surface should be non-adherent to facilitate aspiration of single cells |
| Hard-Shell Full-Height 96-Well Semi-Skirted PCR Plates | Bio-Rad | HSS9601 | Any 96-well PCR plate should suffice |
| 96-well tissue culture plate | VWR | 62406-081 | Any 96-well tissue culture plate should suffice |
| GenomePlex Single Cell Whole Genome Amplification Kit | Sigma | WGA4 | |
| Microseal 'A' Film | Bio-Rad | MSA5001 | |
| Mini plate spinner | Thomas Scientific | 1225Z37 | |
| Thermal cycler | Bio-Rad | 1861096 | Any thermal cycler should suffice so long as it can accommodate a 96-well PCR plate |
| Agencourt Ampure Beads | Beckman Coulter | A63880 | |
| Dynamag-2 Magnet | Thermo Fisher Scientific | 12321D | Any similar magnetic tube strip should suffice |
| Nextera XT DNA Library Preparation Kit | Illumina | FC-131-1096 | |
| Nextera XT Index Kit | Illumina | FC-121-1012 | |
| Complete kit (optimized for Roche LightCycler 480) | Kapa Biosystems | KK4845 | This kit is optimized for the Roche LightCycler 480 real-time PCR machine. If using a different machine, substitute a kit optimized for your machine. |
参考文献
- Kahlem, P. Transcript Level Alterations Reflect Gene Dosage Effects Across Multiple Tissues in a Mouse Model of Down Syndrome. Genome Res. 14 (7), 1258-1267 (2004).
- Torres, E. M. Effects of Aneuploidy on Cellular Physiology and Cell Division in Haploid Yeast. Science. 317 (5840), 916-924 (2007).
- Itsara, A. Population Analysis of Large Copy Number Variants and Hotspots of Human Genetic Disease. Am. J. Human Gen. 84 (2), 148-161 (2009).
- Sudmant, P. H. Global diversity, population stratification, and selection of human copy-number variation. Science. 349 (6253), aab3761 (2015).
- Navin, N. Inferring tumor progression from genomic heterogeneity. Genome Res. 20 (1), 68-80 (2010).
- Torres, L., Ribeiro, F. R., Pandis, N., Andersen, J. A., Heim, S., Teixeira, M. R. Intratumor genomic heterogeneity in breast cancer with clonal divergence between primary carcinomas and lymph node metastases. Breast cancer res and treat. 102 (2), 143-155 (2007).
- Yates, L. R. Subclonal diversification of primary breast cancer revealed by multiregion sequencing. Nat. Med. 21 (7), 751-759 (2015).
- Forsberg, L. A. Age-related somatic structural changes in the nuclear genome of human blood cells. Am. J. Human Gen. 90 (2), 217-228 (2012).
- Laurie, C. C. Detectable clonal mosaicism from birth to old age and its relationship to cancer. Nat. Gen. 44 (6), 642-650 (2012).
- Jacobs, K. B. Detectable clonal mosaicism and its relationship to aging and cancer. Nat. Gen. 44 (6), 651-658 (2012).
- Knouse, K. A., Wu, J., Whittaker, C. A., Amon, A. Single cell sequencing reveals low levels of aneuploidy across mammalian tissues. Proc. Natl. Acad. Sci. 111 (37), 13409-13414 (2014).
- Knouse, K. A., Wu, J., Amon, A. Assessment of megabase-scale somatic copy number variation using single-cell sequencing. Genome Res. 26 (3), 376-384 (2016).
- . KAPA Library Quantification Kit Available from: https://www.kapabiosystems.com/product-applications/products/next-generation-sequencing-2/library-quantification/ (2016)
- . DNAcopy Available from: https://bioconductor.org/packages/release/bioc/html/DNAcopy.html (2016)
- Lichti, U., Anders, J., Yuspa, S. H. Isolation and short-term culture of primary keratinocytes, hair follicle populations and dermal cells from newborn mice and keratinocytes from adult mice for in vitro analysis and for grafting to immunodeficient mice. Nat. Protoc. 3 (5), 799-810 (2008).
- Brewer, G. J., Torricelli, J. R. Isolation and culture of adult neurons and neurospheres. Nat. Protoc. 2 (6), 1490-1498 (2007).
- Navin, N. Tumour evolution inferred by single-cell sequencing. Nature. 472 (7341), 90-94 (2011).
- Szulwach, K. E., Chen, P. Single-Cell Genetic Analysis Using Automated Microfluidics to Resolve Somatic Mosaicism. PLoS ONE. 10 (8), e0135007 (2015).
- Zong, C., Lu, S., Chapman, A. R., Xie, X. S. Genome-Wide Detection of Single-Nucleotide and Copy-Number Variations of a Single Human Cell. Science. 338 (6114), 1622-1626 (2012).
- Wang, Y. Clonal evolution in breast cancer revealed by single nucleus genome sequencing. Nature. 512 (7513), 155-160 (2014).
- Zhang, C. Z. Chromothripsis from DNA damage in micronuclei. Nature. 522 (7555), 179-184 (2015).
- Macaulay, I. C., Voet, T. Single Cell Genomics: Advances and Future Perspectives. PLoS Genetics. 10 (1), e1004126 (2014).
- Gawad, C., Koh, W., Quake, S. R. Single-cell genome sequencing: current state of the science. Nat. Rev. Gen. , 1-14 (2016).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved