Method Article
Tek Hücre sıralama kullanarak Kopya sayısı Değişmeler Algılama
Bu Makalede
Özet
Tek hücre sıralama yüksek çözünürlükte genomik değişikliklerin ele almak için giderek daha popüler ve erişilebilir bir araçtır. Biz tek hücrelerde kopya sayısı değişiklikleri tanımlamak için tek bir hücre sıralama kullanan bir protokol sağlar.
Özet
Tek hücre çözünürlükte genomik değişim tespiti, normal dokularda, kanser ve mikrobik popülasyonların genetik heterojenliği ve evrim karakterize edilmesi için önemlidir. Genetik heterojenite değerlendirmek için geleneksel yöntemler düşük çözünürlük, düşük hassasiyet, ve / veya düşük özgüllük ile sınırlı kalmıştır. Tek hücre sıralama uygun analiz yüksek çözünürlük, yüksek hassasiyet ve yüksek özgüllük ile genetik heterojenite tespit etmek için güçlü bir araç olarak ortaya çıkmıştır. Burada tek hücrelerin izolasyonu, tüm genom amplifikasyonu, sekanslama ve analiz için bir protokol sağlar. Bizim yaklaşım tek hücrelerde megabase ölçekli kopya sayısı varyantlarının güvenilir tanımlanması için izin verir. Bununla birlikte, bu protokol yönleri de tek hücre genetik değişikliklerin diğer tip araştırmak için uygulanabilir.
Giriş
DNA kopya sayısı değişiklikler birkaç baz çifti büyüklükleri değişebilir tüm kromozomlar (anöploidi) ila (CNVs) (kopya sayısı varyantları). Genomun büyük bölgeleri etkileyen kopya sayısı değişiklikleri kadar genler 1, 2 binlerce ifadesini değiştirerek önemli fenotipik sonuçlar doğurabilir. Toplu dizileme veya mikroarray bazlı yöntemler, 3, 4 ile tespit edilebilen bir nüfusun bütün hücrelerde bulunur CNVs. Ancak, popülasyonlar da nüfus ya da hatta tek hücrelerinin bir alt kümesi mevcut CNVs ile, genetik olarak heterojen olabilir. Genetik heterojenite bilinmiyor sonucu 5, 6, 7, 8, 9, aynı zamanda normal dokularda da tümör gelişimini sürücü, kanser yaygındır ve 10.
Geleneksel olarak, genetik heterojenite sitolojik yaklaşımlar veya dökme sıralama yoluyla değerlendirildi. Böyle situ hibridizasyon (FISH), kromozom yayılır ve spektral karyotipleme (SKY) floresan olarak sitoloji yaklaşımlar, tek tek hücrelerin içinde bulunan tanımlama değişikliklerin yarar var ama melezleme ve 11 yayılan eserler nedeniyle yüksek hata oranlarına sahiptir. Bu yaklaşımlar da kendi çözünürlük sadece açığa kopya sayısı sınırlıdır birkaç megabases kapsayan değişmektedir. Ardışık veya toplu DNA mikroarray'ler, doğruluk ve çözünürlük olsa daha yüksek, daha az duyarlıdır. popülasyon bazlı yaklaşımlar ile genetik heterojenite tespit etmek için, varyantlar popülasyonunda hücre önemli bir fraksiyonunda mevcut olmalıdır. tek hücre genomik DNA'yı büyütmek için yöntemler ortaya mümkün tek hücre genomunu dizilemek için yaptı. Tek hücre sequencing uygun kalite kontrol yöntemleri uygulanmaktadır yüksek çözünürlük, yüksek hassasiyet ve, yüksek doğruluk 12 avantajlara sahiptir.
Burada, tek bir hücre içinde megabaz ölçekli kopya sayısı değişimleri tespit etmek için bir yöntem açıklanmaktadır. Biz, bağlayıcı adaptörü PCR kullanılarak genomik DNA yükseltmek nesil dizileme için kütüphaneler hazırlamak ve kopya numarası gizli Markov modeli ve dairesel ikili segmentasyon hem varyantları tespit, Mikroaspirasyon tarafından tek hücreleri izole.
Protokol
1. Tek Hücreleri İzolasyon
- microaspirator hazırlayın
- aspiratör tüp montajı net plastik ucunu çıkarın ve 3/16 "iç çaplı bir 1 metrelik PVC boru bir ucunu dar ucunu.
- 3/6 "iç çapı 1 metrelik PVC boru diğer ucunu bir 0.2 mikron şırınga filtresi çıkışını takın.
- 5/16 "iç çapa sahip bir 6 inç uzunluğunda PVC boru donanımının bir ucuna 0.2 mikron bir şırınga filtreden girişini yerleştirin.
- İsteğe bağlı: yarısında 5/16 "iç çapı 6 inç uzunluğunda PVC boru kesin ve iki yarısı arasında bir in-line su tuzağı yerleştirin.
- 1 ml mezuniyet 5 ml plastik serolojik pipet kırmak ve 5/16 "iç çapı ile PVC boru açık ucunu kırık ucunu.
- Bir aspiratör borusu düzeneğinin esnek ucunda (şeffaf plastik uç içine 5 ml plastik serolojik pipet çıkışını takın) Çıkarmıştı.
- depolama için, steril bir plastik tüp ile aspiratör boru montaj kırmızı ağızlık kapsamaktadır.
- Bir Bunsen beki alev yakınında tüp orta getiren ve tüpün iki ucundaki gerilim uygulayarak 10-30 mikron iç çapı bir cam kılcal tüp dışarı çizin. iki potansiyel aspiratör iğneler oluşturmak için ortada çizilen tüp kırın. sadece bazı tüpler tek hücre toplamak için uygun bir iç çapı ile sona erecek birkaç tüpleri ile bu adımı yineleyin.
- depolama için, bir 15 cm'lik Petri kabındaki modelleme kil iki şeritler yerleştirin ve şeritler arasında aspiratör iğne sabitleyin.
- hücreleri seç
- Hücre tipine ve deney için uygun tek bir hücre süspansiyonu hazırlanır. Örneğin, bu gibi uygun bir ortam ihtiva eden bir konik tüp tripsinizasyon ve transfer hücreler tarafından insan fibroblast hücre hatları hasat hücreler olarak yapışmış olan hücrelerin hazırlanması için.
- Önce, sırasında ya dafter tek bir hücre süspansiyonu (o tek bir hücre süspansiyonu hazırlamak için ne kadar sürer bağlı olarak), kurulum tüm genom amplifikasyonu için kaput hazırlanıyor.
- % 10 çamaşır suyu ile kaput, pipet kutuları ve pipet uçları yüzeyini aşağı Sprey ve bir kağıt havlu ile silin. % 70 etanol ile bu adımı yineleyin.
- 96 oyuklu bir PCR plaka, tek tek oyuklara tüm genom amplifikasyonu (WGA) kit su 8 uL ekleyin, her bir hücre için bir de sekanslanacak olan. Buz üzerinde 96 oyuklu bir doku kültür plakasının ve bir yerden bir kapakla 96 gözlü PCR plakası kapatılır.
- 15 cm'lik bir Petri kabı içinde 10 ml ortamı veya fosfat tamponlu tuz (PBS) 1000 hücreleri ekleyin ve kabına yapışan hücreler önlemek için buz üzerinde çanak yerleştirin.
- 10X objektif bir ışık mikroskobu buz kapak ile Petri kabındaki hücrelere ve 96 PCR plaka getirin.
- yavaşça th çizilmiş ucunu hafifçe vurarak aspiratör iğne açılmasını artırmaksert bir yüzeye e aspiratör iğne ucu kırılır şekilde. microaspirator açık ucunu aspiratör iğne geniş ucunu takın.
- mikroskop sahnede hücreleri içeren Petri kabı yerleştirin. Ağızda aspiratör kırmızı ağızlık yerleştirin. hücreleri içeren Petri kabı taşımak için aspiratör iğne ve diğer yandan taşımak için tek bir elinizi kullanın. Tek hücreleri tanımlamak sekanslanacak olan.
- ağız emme kullanarak, ortam veya PBS ~ 1-2 mcL ile birlikte aspiratör iğne içine tek bir hücreyi çizin. 96 gözlü PCR plaka tek bir kuyu içine su 8 uL hücreyi aktarın. hücreyi aktarırken kabarcıkları tanıtan kaçının.
- Hücrelerin istediğiniz sayı izole edilene kadar adımı tekrarlayın 1.2.7. buz üzerinde PCR plaka tutun ve toplama hücreleri arasındaki kapak ile örtülü. Hücreleri almış kuyu işaretleyin.
- tek hücre toplama ederken, bütün g 10X tek hücre parçalama ve parçalanma tampon çözülmeenome amplifikasyon kiti. hücrelerin istenilen sayıda toplama sonra, tüm genom amplifikasyonu hemen geçin.
2. Tüm Genom Büyütme
- , Tüm genom amplifikasyonu sırasında kirlenmesini önlemek bir doku kültürü kaput içinde tüm reaktifler ekleyin filtreleri ile pipet uçları kullanın ve kuyularda arasında pipet değiştirmek için.
- tüm genom amplifikasyonu (WGA) kiti bir çalışma parçalama ve parçalanma tampon çözeltisi hazırlayın. En fazla 32 hücrelerinin her biri için, bir mikrosantrifüj tüpü içinde, proteinaz K çözeltisi 10x tek hücre lisis ve parçalanma tamponu, 2 uL 32 uL birleştirir. çözümler karıştırmak için tüp Vortex.
- her oyuğa lizis ve parçalanma tampon çözeltisi çalışma 1 mcL ekleyin ve yukarı pipet ve aşağı karıştırın.
- plaka Kapak ve bir plastik filmle tüm kuyuları mühür. Kısaca mini plaka spinner tabak santrifüj.
- foll olarak Termal döngüakımları: 50 ° C, 1 saat ve 99 ° C, 4 dakika.
- buz üzerinde plaka soğutun ve kısaca mini plaka spinner tabak santrifüj.
- tüm genom amplifikasyon kiti çalışan bir kütüphane hazırlık tampon çözeltisi hazırlayın. her bir hücre için, 1 x tek hücre kütüphanesi hazırlanması tamponu, 2 ul ve mikrosantrifüj tüpü içinde bir kütüphane sabitleme çözeltisi 1 uL birleştirir. Aynı mikrosantrifüj tüpü içinde birçok hücreler için çalışma çözeltisi hazırlayın.
- plastik filmi çıkarın ve her bir kütüphane hazırlanması tampon çözeltisi çalışma 3 uL ekleyin. karıştırmak için iyi yukarı ve aşağı içeriğini pipetle. plastik film değiştirin.
Not: Wells filmde delikler başlayana dek plastik film tüm genom amplifikasyonu süreci boyunca yeniden kullanılabilir. Bu durumda, yeni bir plastik film geçin. - Kısaca, bir mini plaka spinner plaka santrifüj 2 dakika boyunca 95 ° C'de inkübe edin.
- buz ve kısaca plaka soğutunmini plaka spinner plaka santrifüj.
- Plastik filmi çıkarın her kuyuya 1 ul kütüphane hazırlık enzim ekleyin ve yukarı iyi içeriğini pipet ve aşağı karıştırın. buz üzerinde veya bu adımı boyunca soğuk bir blokta kütüphane hazırlık enzim tutun. plastik film değiştirin.
- 16 ° C, 20 dakika, 24 ° C, 20 dakika, 37 ° C, 20 dakika, 75 ° C, 5 dakika, 4 ° C 'de sabit: Kısaca aşağıdaki mini plakası döndürücü ve termal döngüsü içinde plaka santrifüj.
- buz üzerinde plaka soğutun ve kısaca mini plaka spinner tabak santrifüj.
- tüm genom amplifikasyon kiti bir çalışma amplifikasyon karışımı hazırlayın. Her hücre için, 48.5 ul su, 7.5 ul 10x büyütme ana karışımı ve bir mikrosantrifüj tüp içinde 5 uL WGA DNA polimeraz birleştirir. Aynı mikrosantrifüj tüp içinde birkaç hücreler için çalışma karışımı hazırlayın. buz üzerinde çalışan karışımı tutun.
- Plastik filmi çıkarın amplifikasyon karışımı çalışan 61 mcLde yukarı ve aşağı her sıra ve pipet içeriğine karıştırın. buz üzerinde veya bu adımı boyunca soğuk bir blokta çalışan amplifikasyon karışımı tutun. plastik film değiştirin.
- 95 ° C, 3 dakika, 94 ° C'de 25 döngü, 30 saniye 65 ° C, 5 dak, 4 ° C 'de sabit: Kısaca şu şekilde mini plaka spinner plaka ve termal döngüsü santrifüj.
- 3. adımda kullanılmak üzere, 20 ° C - ayrı bir mikrosantrifüj tüpü ve saklamak için, her numune devri 60 uL.
- Her numune (örnek 15 uL, yani 3 uL 6x DNA yükleme boyası) geri kalan hacim DNA yükleme boyası ekleyin ve bir% 1 agaroz jeli üzerinde Her reaksiyondan 5 uL çalıştırın.
NOT: başarıyla 250 ila 1,000 bp bir smear olarak görünür güçlendirilmiş hücreler. bir karalama göstermek veya sadece bir soluk smear görünmüyor numuneler yararlı sıralama verilerini elde etmek olası değildir.
3. Sıralama
- paramanyetik boncuklar ile örnekleri arındırın.
- ThaBuz üzerinde ağırlık DNA örnekleri. Çözelti homojen olması ve en az 30 dakika oda sıcaklığında inkübe boncuk Vortex para-manyetik boncuk kadar.
- Temiz bir mikrosantrifüj tüpü içine, her bir DNA numunesi 20 uL aktarın. Numunenin geri kalan kısmı, -20 ° C'de saklanabilir.
- Her DNA örneği ve karıştırmak için vorteks paramanyetik boncuk 30 mcL (1.5x) ekleyin. 10 dakika oda sıcaklığında inkübe edin. Aşama 3.4 kullanılmak için oda sıcaklığında boncuk stok solüsyonu bırakın.
- 2 dakika süreyle manyetik tüp şerit üzerinde tüpler yerleştirin veya boncuk bir pelet oluşturur ve kadar süpernatant açıktır.
- P200 pipet kullanarak, boncuk bozmadan mümkün olduğunca süpernatant kadar kaldırın.
- DNA boncuk karışımı% 80 etanol içinde 180 uL ekleyin. boruları etanol çözeltisi ile boncuk "durulama" mıknatıs göre birkaç kez çevirin.
- P200 pipet kullanarak, çekmenin, aslında etanol yıkama kadar kaldırınboncuk bozmadan ble.
- 3.1.6 ve 3.1.7 tekrarlayın.
- Hava yaklaşık olarak 10 dakika ya da daha fazla etanol görünene kadar boncuk kurutun. boncuklar kırık görünür bir sonraki adıma geçin ya da tüpün duvarı düşmek.
- manyetik şerit örnekleri çıkarın. boncuklardan DNA elüte boncuk tekrar süspansiyon örnekleri girdaba 10 mM Tris pH 8.0, 40 uL ekleyin. Oda sıcaklığında 2 dakika süreyle inkübe edilir.
- Kısaca tüpün altındaki sıvı toplama ve en az iki dakika için bir manyetik şerit üzerinde boru tüpleri örnekleri santrifüj. boncuk bozmadan temiz mikrosantrifüj tüpler içine eluent aktarın.
- örnek normalleştirme
- Bir spektrofotometre kullanılarak, her bir numune konsantrasyonunu belirlemek. konsantrasyonları 10 ile 30 ng / ml aralığında olmalıdır.
- 10 mM Tris pH 8.0 kullanılarak 0.2 ng / ml, her örnek seyreltilir. Aşağıdaki adımlar asgari i gerektirirBu seyreltme 5 uL Temel giriş.
- Kütüphane hazırlanması
- Kütüphane hazırlık seti 13 yönergeleri izleyerek sıralama kütüphaneleri hazırlayın.
- nihai temizleme
- Adım 3.1 göre örnekleri temizleyin. 50 bp, 75 bp, ya da 150 bp okuma uzunlukları sırasıyla 1.5x, 1x veya 0.6x hacmini örnek boncuk oranını kullanın. 10 mM Tris pH 8.0 15 ul ile elüte.
- Kütüphanede 14 boyutu dağılımını kontrol etmek için bir parçası analizörü örnekleri çalıştırın. boyut dağılımı eşit 150 900 bp yayılmalıdır.
- qPCR kullanarak örnekleri sayısal olarak
- Kütüphane Niceleme Kiti kullanılarak, kit talimatlarına 15'e göre bir qPCR reaksiyon ayarlayın. Pozitif standartları (kit DNA standartları) ve negatif standartları (su veya başka boş çözelti) eklemek için bir sütun boş bırakın.
- fol olarak Termal döngüdüşükler: 95 ° C, 5 dakika (4.8 ° C / saniyenin altındaki bir rampa) ile 95 ° C, 30 saniye (4.8 ° C / saniyenin altındaki bir rampa) ve 2.5, 60 ° C, 45 saniye (rampa 35 döngü ° C / sn).
- havuzu
- akış hücresi üzerinde birçok şerit ihtiyaç vardır nasıl belirleyin ve hat başına bir grup ile, gruplar halinde örnekleri bölün.
- Numunelerin her bir grup için, aşama 3.5.2 arasında qPCR verilere dayanarak düşük konsantrasyon ile örnek seçmek ve 10 mM Tris pH 8.0 kullanılarak bu konsantrasyona kadar bu gruptaki tüm örnekleri normalleştirmek.
- birlikte, her grup örnekleri havuz.
- Ardışık
- Standart prosedürler 16 aşağıdaki nesil dizileme makinede yük havuzları.
4. Veri Analizi
NOT: Unix tabanlı ortamda bu bölümde programları ve komut dosyalarını çalıştırmak için gereklidir. onların in aşağıdaki protokolde belirtilen yazılımı yükleyin enstalasyonda kılavuzları. Tüm komut https://sourceforge.net/projects/singlecellseqcnv/ bulunabilir.
- FASTX-Toolkit sürüm 0.0.13 17'den fastx_trimmer kullanarak 40-nt okur Trim.
fastx_trimmer -Q33 -i example.fastq -l 40 -o example.trim.fastq - Varsayılan seçenekleri 18 ile BWA sürüm 0.6.1 kullanarak uygun referans genomu (fare için MM9, insana hg19) okur hizalayın.
bwa aln mm9.fa example.trim.fastq> example.sai
bwa Samse mm9.fa example.sai example.trim.fastq> example.sam - Daha sonra sıralamak ve endeks elde edilen BAM dosya SAMTools sürümü 0.1.19 19 kullanılarak, chrM ve rastgele kromozomlarına hizalamalarını çıkarın.
grep -v -w chrM example.sam | grep -v rasgele> example.filtered.sam
| Samtools -uSh example.filtered.sam görmek samtools sıralama - example.filtered
samtools endeksi example.filtered.bam - CNVs tespit HMMcopy başlat= "Xref"> 20
- genomu için GC yüzdesi referans dosyası oluşturmak için HMMcopy içinde gcCounter kullanın. pencere boyutunu belirlemek için "500000 -w" seçeneğini kullanın. Adım 4.2 kullanılan Fasta referans dosyası aynı sürümünü kullanın, ancak emin chrM yapmak ve rastgele kromozomlar Fasta dosyadan kaldırılır.
gcCounter -w 500000 mm9.fa> mm9_gc.wig - mappability için kıpırdatmak dosyası oluşturmak için HMMcopy içinde generateMap.pl kullanın. uzunluğu okumak belirtmek için "40 -w" seçeneğini kullanın. Adım 4.4.1 kullanılan aynı Fasta referans dosyasını kullanın.
generateMap.pl -b mm9.fa
generateMap.pl -w 40 -i mm9.fa mm9.fa -o mm9.bigwig - genomu için bir mappability referans dosyası oluşturmak için HMMcopy içinde mapCounter kullanın. pencere boyutunu belirlemek için "500000 -w" seçeneğini kullanın.
mapCounter -w 500000 mm9.bigwig> mm9_map.wig - Her BAM dosyası için bir kıpırdatmak dosyası oluşturmak için HMMcopy içinde readCounter kullanın.
readCounter 500000 example.filtered.bam> input.wig -w - R komut dosyalarında referans dosyalarına yollarını değiştirmek (run_hmmcopy.mm9.r veya run_hmmcopy.hg19.r), 4.4.1 (örneğin, mm9_gc.wig) ve 4.4.3 (örneğin, mm9_map yukarıda oluşturulan dosyaları kullanabilirsiniz. sırasıyla, GC yüzdesi referans dosyası ve mappability referans dosyasına bakın değişkenler gfile ve MFILE için peruk). Sonra sağlanan R komut "run_hmmcopy.mm9.r" ya da "run_hmmcopy.hg19.r" çalıştırın.
R CMD TOPLU run_hmmcopy.mm9.r
veya
R CMD TOPLU run_hmmcopy.hg19.r - BAM dosyaları bir dizi toplu işlem için, (VS) segmentleri arama ve değişkenlik puanları hesaplamak için paketinde sağlanan komut "HMMpipe.pl" tek bir klasörde BAM dosyaları koymak ve çalıştırın. biçimi uygulayın:
Perl HMMpipe.pl Folder_to_BAMfiles mm9
- genomu için GC yüzdesi referans dosyası oluşturmak için HMMcopy içinde gcCounter kullanın. pencere boyutunu belirlemek için "500000 -w" seçeneğini kullanın. Adım 4.2 kullanılan Fasta referans dosyası aynı sürümünü kullanın, ancak emin chrM yapmak ve rastgele kromozomlar Fasta dosyadan kaldırılır.
- CNVs 21 tespit etmek için DNAcopy çalıştırın.
- benzersiz her BAM dosyasından okur eşlenen ayıklamak için SAMtools sürüm 0.1.19 kullanın.
samtools görüntülemek -h -F 0x0004 example.filtered.bam | egrep -i "^ @ | XT A: U" | samtools -Shu görünümü -> example.mapped.bam - BAM PCR çiftleri 22 dosya işaretlemek için Picard sürüm 1.94 yılında MarkDuplicates kullanın.
MarkDuplicates.jar GİRİŞİ jar java = example.mapped.bam ÇIKIŞ = example.nondup.bam METRICS_FILE = example.dup REMOVE_DUPLICATES = true - Sürüm 2.17.0 eşlenen sayısı için bedtools olarak coveragebed kullanımı sağlanmıştır YATAK dosyası "mm9.500k.dynamic.win.bed" ya da "hg19.500k.dynamic.win.bed" 23 önceden tanımlanmış dinamik 500kb eşlenebilir pencerelerinin her okur .
coverageBed -abam example.nondup.bam -b mm9.500k.dynamic.win.bed -counts | sıralama -k1,1 -k2,2n> example.nondup.counts - sağlanan GC içeriği referans dosyası "mm9.500k.dynamic.win.fa.gc.txt" ya da "hg19.500k.dynamic.win.fa dayalı okuma sayar normalleştirmek için sağlanan Perl betiği" normalizeGC.pl "kullanın. gc.txt ".
perl normalizeGC.pl mm9.500k.dynamic.win.fa.gc.txt example.nondup.counts> example.bam.norm.counts - segmentleri aramak için sağlanan R komut "dnacopy.r" kullanın.
R CMD TOPLU dnacopy.r
- benzersiz her BAM dosyasından okur eşlenen ayıklamak için SAMtools sürüm 0.1.19 kullanın.
- CNVs tespit
- 4.4.6 hesaplanan VS 0.26 aştığı için hücreleri hariç.
- Hangi medyan günlük 2 oranı fazla 0.4 (farazi kazanç) ya da daha az -0.35 (varsayılan kayıp) sadece bu içerecek şekilde 4.4.6 HMMcopy tarafından çağrılan segmentleri Filtre.
- segmenti ortalama 0.6 daha fazla 1.32 veya daha az sadece bu hangi eklemek 4.5.5 yılında DNAcopy tarafından çağrılan segmentleri Filtre.
- Sadece HMMcopy ve DNAcopy hem farazi kazanç veya varsayılan kaybı tespit bölgelerde CNVs çağırarak, 4.6.2 ve 4.6.3 segmentleri örtüşüyor.
Sonuçlar
Monte aspiratör Şekil 1A benzer görünmelidir. İğne o ama çok geniş bir büyük hacimli tek bir hücre ile çizilmiş bir tek hücre karşılamak için yeterince geniş olacak şekilde çizilmelidir. 10X alan (Şekil 1B) bir ila beş hücre olduğunda tek hücre aspire en kolay yoldur.
Tüm genom amplifikasyonu başarılı ise, örnek bir agaroz jeli üzerinde leke olarak gösterilir (Şekil 2, bölmeler, 1, 2, 4, 5, 6, ve 7) olacak. Bir soluk veya eksik smear başarısız amplifikasyon reaksiyonu gösterir ve örnek sıralı olmamalıdır (Şekil 2, 3 şeritleri ve 8).
Kütüphane hazırlandıktan sonra, numune parçası boyutu dağılımı, bir fragmanı analizöründe kapiler elektroforez ile değerlendirilmelidir.Hazırlanabilmişse kütüphaneleri 150 900 bp fragmanı, boyutları çok daha dağılımına sahip olacaktır (Şekil 3, A, B). Başarısız bir kütüphane hazırlanması yamuk parçası boyutu dağılımına neden olur ve bu kütüphaneler (Şekil 3C) sıralanmamalıdır.
gizli Markov modelinin (HMMcopy) ve dairesel ikili bölümleme (DNAcopy) üzerinden dizi verilerinin işlenmesi tahmini kopya sayısının parça halinde her bir hücrenin genomu ayrıştırma olacaktır. Bu segmentler daha sonra tek bir hücre (Tablo 1) kazanç ya da kaybı ile tutarlıdır tahmini kopyalama sayısı ile bu tespit etmek filtrelenebilir. HMMcopy ve DNAcopy Bunlar süzülmüş segmentler daha sonra yüksek güven CNVs tanımlamak için üst üste edilmelidir.
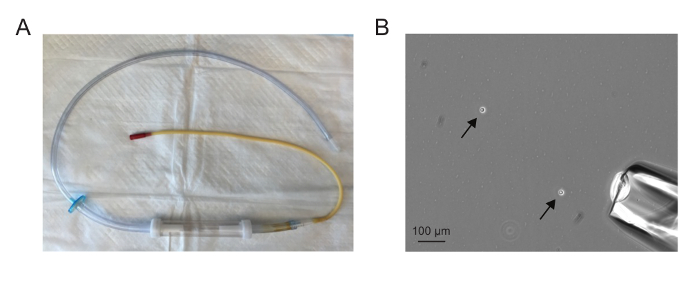
1. Tek hücre izolasyonu rakam. ( A) Bir microaspirator toplandı. (B) 10X alan gösteren hücreleri (oklar) ve microaspirator iğne (sağ alt köşe) ayrışmış. 10X alanında bir ila beş hücre olduğunda tek hücre aspire en kolay yoldur. Bu rakamın büyük halini görmek için lütfen buraya tıklayınız.
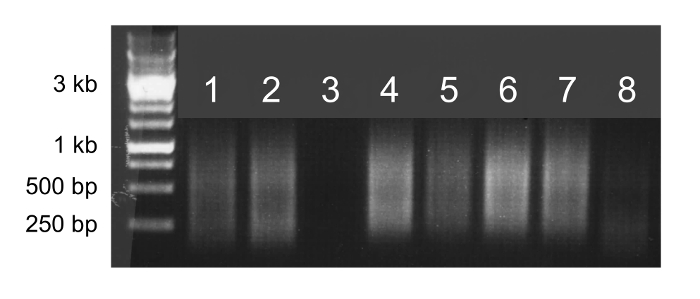
2. Tüm genom amplifikasyon rakam. tüm genom amplifikasyonu ürünleri 5 ul agaroz jel elektroforezi. başarılı bir şekilde 1 kb 100 bp'den parlak smear görünür amplifiye Numuneler (kulvarlar 1, 2, 4, 5, 6 ve 7) ve dizilebilir. başarıyla soluk lekeleri ya da hiç smear (kulvarlar 3 ve 8) üretecek yükseltmek yoktur ve sıralı olmamalıdır örnekler.large.jpg "target =" _ blank "> bu rakamın daha büyük bir versiyonunu görmek için lütfen buraya tıklayınız.
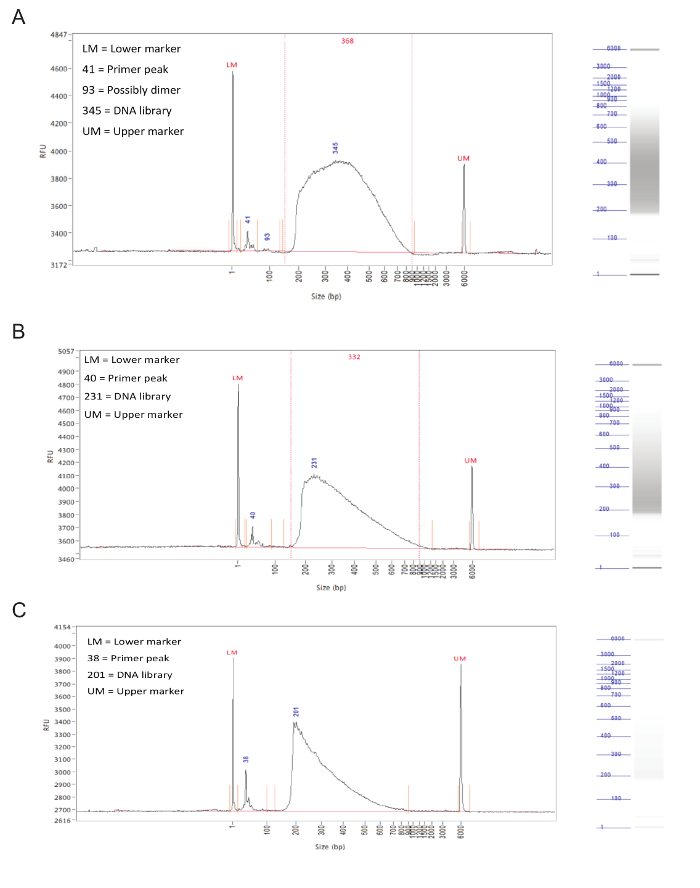
Şekil 3. kütüphane hazırlanması. Bir fragman analizörü Temsilcisi sonuçlar. grafikleri Y ekseni üzerinde X ekseninde ve bağıl floresan birimi (RFU) üzerine (bp olarak) fragmanı boyutunu gösterir. Her grafiğin sağında bir simüle jel şerit olduğunu. (A) 150 ve 900 bp arasında ve keskin zirveleri ya da bir tarafa doğru önyargısız eşit dağıtımı ile, ideal bir örnek için bulundu. Bu örnek, dizileme için kabul edilebilir. Bir boyut dağılımı ile iyi bir örnek (B) Sonuçlar, düşük değerli parçası boyutları eğilmiş. Bu en uygun olmasa da, numune hala dizilenebilmektedir. (C) ağırlıklı olarak küçük fragman boyutları ile, başarısız bir örnek bulundu. Bu büyük olasılıkla tagmentation aşaması esnasında uzun inkübasyon kaynaklanırKütüphane hazırlama. Bu örnek sıralanmamalıdır. Bu rakamın büyük halini görmek için lütfen buraya tıklayınız.
| HMMcopy | |||||||
| Numune | bölüm | Chr | Başlama | Son | Belirtmek, bildirmek | Medyan | |
| D15-4998 | 23 | chr8 | 144.500.001 | 146.500.000 | 6 | 0.5008794 | |
| D15-4998 | 29 | chr10 | 67.000.001 | 134.500.000 | 6 | 0.4031945 | |
| D15-4998 | 52 | chr19 | 1 | 20000000 | 6 | 0.4616884 | |
| D15-4998 | 57 | chrY | 1 | 59.500.000 | 2 | -1,506532 | |
| DNAcopy | |||||||
| Numune | krom | Başlangıç bin | bitiş bin | Başlama | Son | Seg ortalama | Seg med |
| D15-4998 | chr10 | 88 | 197 | 62.612.945 | 129.971.511 | 1,4688 | 0.157 |
| D15-4998 | chr19 | 0 | 31 | 0 | 28.416.392 | 1,4141 | 0,1674 |
| D15-4998 | chrX | 77 | 126 | 51.659.160 | 95.343.369 | 1,3548 | 0,1874 |
| D15-4998 | chrY | 0 | 14 | 0 | 23.805.358 | -2,7004 | 0,3591 |
Tablo 1. Veri analizi. Tek bir hücreden HMMcopy ve DNAcopy tarafından oluşturulan Filtreli segmentler. Bu iki sonuç örtüşen 67 130 Mb gelen kromozom 10 üzerindeki bir kazanç yanı sıra 0 ila 20 Mb gelen kromozomda 19 bir kazanç ortaya koymaktadır. Biz kromozomun 19 proksimal kısmında kazançlar tek hücre sıralama 12 bir dışlayıcı olduğunu bulduk.
Tartışmalar
Geleneksel olarak, bu tür FISH ve SKY olarak sitolojik yöntemler gerekli tek hücre düzeyinde CNVs ve anöploidi belirlenmesi. Şimdi, tek bir hücre sıralama bu tür sorulara için alternatif bir yaklaşım olarak ortaya çıkmıştır. bu genom ve yüksek çözünürlüklü hem olduğu gibi tek hücreli sıralama FISH ve SKY üzerinde avantajlara sahiptir. Uygun kalite kontrol yöntemleri uygulandığında bu FISH ve SKY doğasında melezleme ve yayma eserler duyarlı değildir Dahası, tek hücre sıralama daha güvenilir bir CNVs değerlendirilmesini ve anöploidi sağlayabilir. Ancak, tek bir hücre sıralama son uygulamaların birçok yöntem kapsamlı duyarlılık değerlendirilmesi ve özgünlüğü ile doğrulanmış ve analizler edilmemiştir. Gerçekten de, diğer çalışmalarda kullanılan analitik yaklaşımlar bazı yanlış pozitif KNV 12 çağrıları yüksek frekanslarda (>% 50) ile ilişkilidir. biz tarif yaklaşım titizlikle kn hücreleri kullanılarak test edilmiştirKNV tespiti 12 duyarlılık ve özgüllüğünü doğru ve yanlış keşif oranlarını belirlemek ve optimize etmek için kendi CNV yükü. 10 Mb aşan kalite kontrol ve bu protokolü açıklanan analitik yaklaşımlar, 5 Mb kazançlarının yaklaşık% 20, 5 Mb kayıplarının% 75'i ve tüm CNVs kullanılarak tespit edilebilir. Tek hücre sıralama yanlış keşif oranının belirlenmesinde zor olsa da, biz% 25 daha az olduğu tahmin var. Bu protokol, çeşitli kaynaklardan gelen hücrelere uygulanabilir, komut KNV algılama çözünürlüğünü ayarlamak için değiştirilebilir, ve protokol genomik değişiklikler diğer türleri tanımlamak için adapte edilebilir.
Orada tek hücre içine taze dokuları ayrışan araçlarının çeşitli ve birçok yayın cilt 24 ve beyin 25 gibi belirli dokular için optimize prosedürleri açıklanmaktadır. o fo verdiğinden Biz Mikroaspirasyon tarafından tek hücrelerin izole tercih Her bir hücrenin R görsel değerlendirme sekanslanacak olan. Bununla birlikte, floresans ile aktive edilen hücre sıralama (FACS) 26 ve mikroakışkan cihazlar 27 tarafından tek hücrelerin izole edilmesi de mümkündür. Tek hücre izolasyonu ve tüm genom amplifikasyonu elle gerçekleştirilir, izole etmek ve tek bir seferde kırk hücrelere kadar yükseltmek için uygun olur. Yüksek kaliteli tek hücre sıralama verileri elde etmek için, tek bir hücre genomlarının amplifikasyonu düzgün ve tam olması çok önemlidir. Bu liziz ve amplifikasyon verimi hem de izole edilmiş tek hücrelerin kalitesi dizi verinin kalitesi üzerinde önemli bir etkisi olduğunu belirledik. Bu nedenle, hücreleri hücreler izole hemen sonra başlamalı hemen önce izolasyonu ve tüm genom amplifikasyonu için kendi doğal ortamından hasat edilmelidir. adım 2.3-2.6 de tarif edildiği gibi, ayrıca, parçalama ve parçalanma adımı tam olarak izlenmelidir.
"> Algoritmalar duyarlılık ve özgüllük 12 üzerindeki etkilerinin karşıt, KNV algılama çözünürlüğünü değiştirmek için ayarlanabilir. Tetraploidi 11 ayar tam kromozom anöploidi tespit eşikleri ayarlamak da mümkündür. Ancak, biz bulmak tüm genom amplifikasyonu esnasında katılan gürültü küçük varyantları 12 tespitini zorlaştırmaktadır olarak yaklaşım CNVs 5'inden Mb tespit edebilir. tüm genom amplifikasyonu yaklaşımlar ileri geliştirmeler, sonuçta tek bir hücre sıralama kullanılarak KNV algılama çözünürlüğünü arttırmak gerekir.Tek hücre sıralaması sadece kopya sayısı değişikliklerinin araştırılması hem de tek nükleotid değişimleri 28, 29 ve bir yapısal versiyonunun 30 sağlar. tek bir hücre izolasyonu için protokol bu que cevap uygulanabilirparagrafındaki. Bununla birlikte, tüm genom amplifikasyonu yönteminin seçimi, belirli uygulamaya bağlıdır. Polimeraz zincir reaksiyonu dayanan bu protokol, tarif edilen yöntem, bu amplifikasyon önyargı 32 daha düşük seviyeleri ile ilişkili olduğu için kopya sayısı değişiklikleri tespit etmek için uygundur. Bu tür tek nükleotid polimorfizm genomik değişiklikler, diğer türde araştırmak için, tüm genom amplifikasyonu için diğer yöntemler 31, 32 daha uygun olduğuna inanılmaktadır.
Açıklamalar
Yazarlar ifşa hiçbir şey yok.
Teşekkürler
Biz bu yazının yorumları için Stuart Levine teşekkür ederiz. Bu çalışma Sağlık Hibe GM056800 National Institutes ve Angelika Amon için Kathy ve Curt Mermer Kanser Araştırma Fonu tarafından ve Koch Enstitüsü Destek Hibe P30-CA14051 tarafından kısmen desteklenmiştir. Angelika Amon da Howard Hughes Tıp Enstitüsü ve Biyomedikal Araştırma Glenn Vakfı'nın bir araştırmacı. KAK NIGMS Eğitim Hibe T32GM007753 tarafından desteklenmektedir.
Malzemeler
| Name | Company | Catalog Number | Comments |
| Aspirator tube assemblies for calibrated microcapillary pipettes | Sigma | A5177 | |
| PVC tubing (ID 3/16", OD 5/16", ~1 foot) | VWR | 89068-500 | |
| PVC tubing (ID 5/16", OD 7/16", ~6 inches) | VWR | 89068-508 | |
| Acrodisc Syringe Filter with HT Tuffryn Membrane (diameter 25 mm, pore size 0.2 µm) | VWR | 28144-040 | |
| Serological pipette (5 mL) | BioExpress | P-2837-5 | Any plastic 5 mL pipette should suffice |
| In-Line Water Trap for Oxygen Use | Amazon.com | 700220210813 | |
| Capillary Melting Point Tubes (ID 0.8-1.1 mm, 100 mm length) | VWR | 34502-99 | |
| Modeling clay | VWR | 470156-850 | |
| Petri dish (150 mm diameter) | VWR | 25384-326 | Surface should be non-adherent to facilitate aspiration of single cells |
| Hard-Shell Full-Height 96-Well Semi-Skirted PCR Plates | Bio-Rad | HSS9601 | Any 96-well PCR plate should suffice |
| 96-well tissue culture plate | VWR | 62406-081 | Any 96-well tissue culture plate should suffice |
| GenomePlex Single Cell Whole Genome Amplification Kit | Sigma | WGA4 | |
| Microseal 'A' Film | Bio-Rad | MSA5001 | |
| Mini plate spinner | Thomas Scientific | 1225Z37 | |
| Thermal cycler | Bio-Rad | 1861096 | Any thermal cycler should suffice so long as it can accommodate a 96-well PCR plate |
| Agencourt Ampure Beads | Beckman Coulter | A63880 | |
| Dynamag-2 Magnet | Thermo Fisher Scientific | 12321D | Any similar magnetic tube strip should suffice |
| Nextera XT DNA Library Preparation Kit | Illumina | FC-131-1096 | |
| Nextera XT Index Kit | Illumina | FC-121-1012 | |
| Complete kit (optimized for Roche LightCycler 480) | Kapa Biosystems | KK4845 | This kit is optimized for the Roche LightCycler 480 real-time PCR machine. If using a different machine, substitute a kit optimized for your machine. |
Referanslar
- Kahlem, P. Transcript Level Alterations Reflect Gene Dosage Effects Across Multiple Tissues in a Mouse Model of Down Syndrome. Genome Res. 14 (7), 1258-1267 (2004).
- Torres, E. M. Effects of Aneuploidy on Cellular Physiology and Cell Division in Haploid Yeast. Science. 317 (5840), 916-924 (2007).
- Itsara, A. Population Analysis of Large Copy Number Variants and Hotspots of Human Genetic Disease. Am. J. Human Gen. 84 (2), 148-161 (2009).
- Sudmant, P. H. Global diversity, population stratification, and selection of human copy-number variation. Science. 349 (6253), aab3761 (2015).
- Navin, N. Inferring tumor progression from genomic heterogeneity. Genome Res. 20 (1), 68-80 (2010).
- Torres, L., Ribeiro, F. R., Pandis, N., Andersen, J. A., Heim, S., Teixeira, M. R. Intratumor genomic heterogeneity in breast cancer with clonal divergence between primary carcinomas and lymph node metastases. Breast cancer res and treat. 102 (2), 143-155 (2007).
- Yates, L. R. Subclonal diversification of primary breast cancer revealed by multiregion sequencing. Nat. Med. 21 (7), 751-759 (2015).
- Forsberg, L. A. Age-related somatic structural changes in the nuclear genome of human blood cells. Am. J. Human Gen. 90 (2), 217-228 (2012).
- Laurie, C. C. Detectable clonal mosaicism from birth to old age and its relationship to cancer. Nat. Gen. 44 (6), 642-650 (2012).
- Jacobs, K. B. Detectable clonal mosaicism and its relationship to aging and cancer. Nat. Gen. 44 (6), 651-658 (2012).
- Knouse, K. A., Wu, J., Whittaker, C. A., Amon, A. Single cell sequencing reveals low levels of aneuploidy across mammalian tissues. Proc. Natl. Acad. Sci. 111 (37), 13409-13414 (2014).
- Knouse, K. A., Wu, J., Amon, A. Assessment of megabase-scale somatic copy number variation using single-cell sequencing. Genome Res. 26 (3), 376-384 (2016).
- . KAPA Library Quantification Kit Available from: https://www.kapabiosystems.com/product-applications/products/next-generation-sequencing-2/library-quantification/ (2016)
- . DNAcopy Available from: https://bioconductor.org/packages/release/bioc/html/DNAcopy.html (2016)
- Lichti, U., Anders, J., Yuspa, S. H. Isolation and short-term culture of primary keratinocytes, hair follicle populations and dermal cells from newborn mice and keratinocytes from adult mice for in vitro analysis and for grafting to immunodeficient mice. Nat. Protoc. 3 (5), 799-810 (2008).
- Brewer, G. J., Torricelli, J. R. Isolation and culture of adult neurons and neurospheres. Nat. Protoc. 2 (6), 1490-1498 (2007).
- Navin, N. Tumour evolution inferred by single-cell sequencing. Nature. 472 (7341), 90-94 (2011).
- Szulwach, K. E., Chen, P. Single-Cell Genetic Analysis Using Automated Microfluidics to Resolve Somatic Mosaicism. PLoS ONE. 10 (8), e0135007 (2015).
- Zong, C., Lu, S., Chapman, A. R., Xie, X. S. Genome-Wide Detection of Single-Nucleotide and Copy-Number Variations of a Single Human Cell. Science. 338 (6114), 1622-1626 (2012).
- Wang, Y. Clonal evolution in breast cancer revealed by single nucleus genome sequencing. Nature. 512 (7513), 155-160 (2014).
- Zhang, C. Z. Chromothripsis from DNA damage in micronuclei. Nature. 522 (7555), 179-184 (2015).
- Macaulay, I. C., Voet, T. Single Cell Genomics: Advances and Future Perspectives. PLoS Genetics. 10 (1), e1004126 (2014).
- Gawad, C., Koh, W., Quake, S. R. Single-cell genome sequencing: current state of the science. Nat. Rev. Gen. , 1-14 (2016).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır