A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
الفيديو خالية من عدسة مجهرية للدينامية والتحليل الكمي للثقافة الخلية ملتصقة
In This Article
Summary
مجهرية خالية من عدسة الفيديو تمكننا من رصد الثقافات الخلية مباشرة داخل الحاضنة. هنا يمكننا وصف كامل البروتوكول المستخدمة لاكتساب وتحليل عملية شراء 2.7 يوم طويل من خلايا هيلا مثقف، مما يؤدي إلى dataset من 2.2 x 106 قياسات مورفولوجيا الخلايا الفردية ودورة الخلية 10584 المسارات.
Abstract
هنا، علينا أن نبرهن أن الفيديو خالية من عدسة مجهرية تمكننا من التقاط حركية الآلاف من الخلايا مباشرة داخل الحاضنة في وقت واحد، وأنه من الممكن لرصد وتحديد خلايا مفردة على طول عدة دورات الخلية. يصف لنا كامل البروتوكول المستخدمة لرصد وقياس ثقافة خلية هيلا لأيام 2.7. أولاً، يتم تنفيذ اكتساب ثقافة الخلية مجهر خالية من عدسة فيديو، وثم يتم تحليل البيانات بعد عملية من أربع خطوات: التعمير المجسم متعدد الطول الموجي، تتبع الخلية، الخلية تجزئة وانقسام الخلية الكشف خوارزميات. نتيجة لذلك نحن تظهر أنه من الممكن جمع من dataset يضم أكثر من المسارات دورة الخلية 10,000 وأكثر من 2 × 106 خلية القياسات المورفولوجية.
Introduction
رصد خلايا الثدييات مثقف طوال عدة دورات الخلية وقياس دقة الخلية خلية وحجم الكتلة الجافة مهمة صعبة. العديد من التقنيات البصرية خالية من التسمية قادرة على أداء هذه المهمة1،2: التحول من مرحلة قياس التداخل3، الميكروسكوب المجسم الرقمي (DHM)4،،من56، 7، كوادريوافي الجانبي القص التداخلي8،9 والكمية المرحلة التصوير المقطعي10،11. وأدت هذه الأساليب إلى العديد من الأفكار الجديدة في فهم دورة خلية من خلايا الثدييات. لكن نادراً ما كانت تقترن بالتلقائية خلية تتبع خوارزميات والإنتاجية بهم لا يزال محدودا عند قياس الخلية مسارات الشامل1 (N < 20 على التوالي3،،من45 , 6). وبالتالي مطلوب طريقة بصرية جديدة لقياس مسارات الخلية الجماعية مع إحصاءات كبيرة (N > 1000).
في هذه الورقة، ونظهر قدرة مجهرية خالية من عدسة الفيديو الصورة في الوقت نفسه آلاف خلايا مباشرة داخل الحاضنة، وثم تحديد المقاييس خلية واحدة على طول آلاف مسارات الفردية دورة الخلية. خالية من عدسة مجهرية هو مرحلة كمية التصوير الأسلوب الذي يتيح الحصول على صورة المرحلة من كثافة الخلايا عبر مجال رؤية كبير جداً (عادة عدة عشرات من مم2، هنا 29.4 مم2)12،13 ،،من1415. يتم تحديد المقاييس عدة على مستوى الخلية الواحدة، مثلاً، خلية المنطقة وخلية الكتلة الجافة، وسمك الخلية، طول المحور الرئيسية الخلية والخلية نسبة العرض إلى الارتفاع12،15عاماً من كل صورة. ثم قم بتطبيق خوارزمية تتبع الخلية، يمكن رسم هذه الميزات لكل خلية واحدة كدالة لل14،وقت التجربة15. وعلاوة على ذلك، عن طريق الكشف عن حدوث انقسامات الخلية في مسارات الخلية، فمن الممكن لاستخراج معلومات هامة أخرى مثل الخلية الأولى الجماهيري (فقط بعد انقسام الخلية)، والكتلة الجافة الخلية الأخيرة (قبل انقسام الخلية) والخلية الجافة دورة مدتها، أي، والفترة الزمنية بين شعبتين على التوالي15. يمكن أن تحسب جميع هذه القياسات مع إحصاءات جيدة جداً (N > 1000) نظراً لمجال الرؤية الكبيرة عادة ما يسمح تحليل الخلايا 200 إلى 10,000 في عملية شراء واحدة خالية من العدسة.
من أجل شرح هذه المنهجية استناداً إلى الفحص المجهري خالية من عدسة الفيديو، يصف لنا بروتوكول رصد وقياس ثقافة خلية هيلا لأيام 2.7. تحليل البيانات من أربع خطوات عملية تقوم على التعمير المجسم متعدد الطول الموجي وتقسيم الخلية وخلية تتبع خوارزميات انقسام الخلية. ويتضح هنا أن القرار المكانية ومعدل الإطار سريع نسبيا (شراء واحدة كل 10 دقائق) التي تم الحصول عليها مع هذا الإعداد مجهرية خالية من عدسة الفيديو متوافق مع معيار خلية تتبع خوارزميات. تحليل كامل لمجموعة البيانات هذه النتائج في قياس 10,584 خلية المسارات عبر دورات خلية كاملة.
وباختصار، مجهرية خالية من عدسة الفيديو أداة قوية لرصد تلقائياً الآلاف من غير مسمى، وغير المتزامنة، وخلايا غير معدلة كل تجربة؛ كل خلية يتم تعقب على مدى عدة دورات الخلية. وهكذا توفر لدينا قياسات متوسط قيمة العديد من المعلمات في الخلية، ولكن الأهم من ذلك، تباين بين الخلية على عدد كبير من الخلايا.
Protocol
1-خلية ثقافة رصد اكتساب
- تنمو خلايا هيلا في دميم + الجلوتامين (مثلاً، جلوتاماكس) المتوسطة تستكمل مع 10% (v/v) إبطال الحرارة العجل الجنين المصل و 1% البنسلين ووالستربتوميسين.
- معطف لوحات الثقافة أسفل الزجاج 6-جيدا مع فيبرونيكتين (25 ميكروغرام/مل) ح 1. ثم البذور 2 × 104 خلايا كل بئر.
- خلال اقتناء، تغيير في المتوسط كل 3 أيام.
- للحصول على مرور الزمن، استخدام المجهر خالية من عدسة الفيديو (المتاحة تجارياً).
ملاحظة: يستند هذا تقنية التصوير عدسة خالية الحسابية كما وصفها أوزكان et al. 16 الذي عدل لإجراء الرصد المستمر في حاضنة عند درجة حرارة 37 درجة مئوية12،15التي تسيطر عليها. ويتميز جهاز استشعار صورة متمم (CMOS) أكسيد معدن المتمم مع الملعب بكسل من 1.67 ميكرومتر ومنطقة تصوير من 6.4 x 4.6 مم2. فتوفرها متعددة الطول الموجي الإضاءة multichip خفيفة التي ينبعث منها قدر صمام ثنائي (المصابيح) جهاز، التي توفر إضاءة الأحمر والأخضر والأزرق. يتم توسيط في أطوال موجية، في 636 و 521 و 452 نانومتر على التوالي، مع عرض النطاق الترددي طيفية من 25 و 45 و 25 نانومتر على التوالي. أحمر-أخضر-أزرق (RGB) المصابيح تقع فوق ثقب ميكرومتر 150 على مسافة 5 سم تقريبا من الخلايا. قوة الضوء المقاسة قريبة من الصمام منخفضة قدر µW 10 في كل طول موجي والوقت الإضاءة ثانية واحدة فقط للحصول على. ومن ثم فمن المتوقع أي مشاكل صور-سمية عند ملاحظة الثقافات الخلية تحت المجهر خالية من عدسة الفيديو. - وضع الحاوية ثقافة الخلية وضعت في اتصال مع جهاز استشعار CMOS (الشكل 1). استخدام حاوية مع ساترة زجاجية في الجزء السفلي، ومهم للنوعية لاقتناء عدسة خالية. يمكن أيضا استخدام الحاويات البلاستيكية ولكنها ستنخفض الحصول على عدسة مجاناً نظراً لرداءة نوعية هذه الحاويات الضوئية.
- التحكم بالمجهر خالية من عدسة الفيديو مع اقتناء البرمجيات (متاح تجارياً)، الذي يقوم بالحصول على مرور الزمن وإعادة بناء المجسم. المعلمات التي يتم إدخالها بواسطة المستخدم في واجهة البرنامج هي معدل الإطار، مدة التجربة، ونوع الثقافة الخلية، أي ملتصقة الخلايا أو الخلايا العائمة.
- تعيين معلمات الإدخال من اقتناء البرمجيات. تعيين معدل الإطار تعيين إلى اقتناء واحد كل 10 دقائق، وتعيين نوع الثقافة الخلية إلى 'ملتصقة'.
ملاحظة: معدل إطار لاقتناء واحد كل 10 دقائق حلاً وسطا جيدا كما أنه يضمن موثوق خلية تتبع بينما لا تزال معقولة حجم مجموعة البيانات الناتجة إلى تحليل. أسرع معدل الإطار يمكن تحقيقه مع هذا الإعداد خالية من عدسة مجهرية من اقتناء واحدة كل 5 دقائق. زيادة في معدل الإطارات يؤدي التسخين المفرط للاستشعار CMOS التي يمكن أن تؤثر على بقاء الخلايا في الثقافة. - بدء اكتساب الوقت الفاصل بين، وخوارزميه إعادة البناء الثلاثية الأبعاد (متاح تجارياً) سوف تلقائياً عملية اقتناء خالية من العدسة للحصول على صورة المرحلة ثقافة الخلية. خوارزمية إعادة البناء الثلاثية الأبعاد استناداً إلى خوارزمية استرداد متعددة الطول موجي مرحلة17 ويقدم صورة RGB مرحلة أعيد بناؤها في النطاق [-π، + π] لكل خلية واحدة عبر مجال رؤية كبير من 29.4 مم2. مبدأ التجسيدي خالية من العدسة في خط هو تسليط الضوء على عينة رقيقة (في z = 0) مع موجه طائرة (من السعة 1 بعد تطبيع) والكشف عن الصورة كثافة حيود اللاحقة في z مسافة قصيرة على جهاز استشعار CMOS = Z، عادة 1 ملم بعد عينة. في الإعداد لدينا، يتم تبديل الإضاءة تسلسلياً إلى أطوال موجية 3 (λ1= 0.450 ميكرومتر، λ2= 0.540 ميكرومتر، λ3= 0.647 ميكرومتر) لقياس أنماط الحيود ثلاثة من نفس العينة.
2-خلية ثقافة تحليل البيانات
- لتتبع خلية، استخدام خوارزمية تراكماتي، المساعد فيجي مفتوحة مصدر التتبع الآلي من جزيئات مفردة18. أولاً، تحميل في الحصول على كامل الوقت الفاصل بين في فيجي (تحميل صورة تسلسل الأوامر). ونظرا للحجم الكبير لمجموعات البيانات، الذي يضم عادة 400 إطارات RGB 29.7 ميغا بايت، أنها أسرع تحميلها في الشكل 8-بت. التالية المساعد فيجي تراكماتي إرشاد المستخدم من خلال عدة مراحل من خوارزمية تتبع الخلية، إلا وهي مرحلة الكشف عن خلية ومرحلة تعقب خلية عدة عوامل التصفية المطبقة إلى اكتشافات الخلية والمسارات المحسوبة. في كل مرحلة، تكوين الخوارزميات وعرض النتائج فورا وذلك إذا لزم الأمر، واحد يمكن بسهولة التنقل ذهابا وإيابا إعادة ضبط الإعدادات. القوائم المختلفة لواجهة تراكماتي مبينة في الشكل 2 مع الإعدادات الفعلية المستخدمة في التجربة خلايا هيلا.
- تعيين معلمات الإدخال للبرنامج الحصول على النحو التالي (انظر الشكل 2). تعيين قطر النقطة المقدرة إلى 15 بكسل، عتبة الكاشف إلى 0.25، المسافة القصوى الربط إلى 15 بكسل، وسد فجوة المسافة كحد أقصى إلى 15 بكسل، والعدد من المواقع في المسارات إلى 3.5.
ملاحظة: تختلف هذه الإعدادات من الواضح من تجربة واحدة إلى آخر، ولا سيما 'النقطة المقدرة القطر' الذي يتوافق مع حجم الخلية (نظراً لهنا في بكسل 1.67 ميكرومتر). بهذا التصريح نفسه ينطبق على 'ربط ماكس المسافة' التي تمثل المسافة القصوى لخلية ضمن إطارين متتاليين (نظراً لهنا في بكسل 1.67 ميكرومتر). وسيتوقف قيمته المثلى على حركية الخلية ولكن أيضا على كثافة الخلية. - في نهاية عملية تتبع الخلية، تولد النتائج في شكل ثلاثة ملفات نصية ('تحليل' زر، انظر الشكل 2). الملف الأكثر فائدة، المسمى 'البقع في إحصاءات المسارات'، يتكون من قائمة بالكشف عن جميع الخلايا مع مواقف كل منهما (x, y) في اقتناء وعددهم الإطار وعددهم المسار. واستنادا إلى هذه البيانات، إجراء تقسيم الخلية والكشف عن انقسامات الخلية استخدام خوارزميات مخصصة (انظر ملفات التعليمات البرمجية التكميلية). يتم تسمية الملفات الأخرى 'الروابط في إحصاءات المسارات' و 'تعقب إحصائيات' وتشمل جميع النتائج التي تتعامل مع المسارات، مثل تعقب المدد، وعدد من الثغرات المكتشفة، وتتبع الإطار الأولى، إلخ.
- استخدام خوارزمية تجزئة الخلية (انظر ملف التعليمات البرمجية التكميلية) استخراج عدة مقاييس تصف مورفولوجيا الخلايا من مرحلة إعادة بناء الصورة تلقائياً. هذه المقاييس هي مساحة الخلية (S) وطول المحور الرئيسية الخلية (L)، وطول المحور طفيفة الخلية (l) وخلية نسبة العرض إلى الارتفاع (L/l). مرحلة تعافي من مجهرية خالية من عدسة تتناسب مع كثافة وسمك طبقة العينة كما نوقش سابقا من15.
- بالإضافة إلى السمات المورفولوجية للخلية، استخراج الكمية الخلية الجافة الشامل (إليه التنمية النظيفة) قياسات من19،،من13مرحلة أعيد بناؤها20
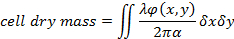 (1) مكافئ.
(1) مكافئ.
حيث φ(x,y) هو المرحلة أعيد بناؤها تحول19، λ الطول الموجي و α = 1.8 × 10-4 م3/كغ الانكسار المحددة التي تتعلق بتفاوت الانكسار الجافة الشامل1، 19. - تقييم سمك الخلية باستخدام تقدير الفرق بين الخلية الانكسار، وأن وسائل الإعلام الثقافة. ونظرا للعلاقة بين سمك الخلية h(x,y) ومرحلة قياس التحول هو قبل20:
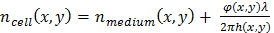 (2.1) مكافئ.
(2.1) مكافئ. (2.2) مكافئ.
(2.2) مكافئ.
مع Δn(x,y) = nخلية (x,y)- نالمتوسطة (x,y)، الانكسار الفرق بين الخلية وثقافة الإعلام. في ما يلي، تحمل قيمة ثابتة من Δn = 0.025، الذي يقدر من الانكسار المقاسة في نواة الخلية هيلا (n = 1.35511) ووسائط الثقافة مخزنة الفوسفات المالحة (PBS) (n = 1.33). على الرغم من أن قيمة سمك الخلية فقط الإرشادية كما أنه يقوم على افتراض، الاختلافات النسبية هي ذات مغزى ويمكن استغلالها للكشف عن تقسيم الخلايا. - استخدام خوارزمية مخصصة الخلايا (انظر ملف التعليمات البرمجية التكميلية) الكشف عن حدوث انقسامات الخلية ومن ثم استخراج المسارات الخلية المرتبطة إلى انقسامات الخلية المكتشفة، بغية الكشف عن انقسام الخلية، مع قياس سمك أكبر من 8 ميكرومتر يتم أولاً تحديد، ثم تحقق ما إذا كانت خلية جديدة تظهر عن كثب في الفضاء والوقت. وبهذه الطريقة، الكشف عن انقسامات الخلية يعتمد على معيارين قوية. يمكن تطبيق البديل وطرق أكثر تفصيلاً للكشف عن انقسام الخلية لاكتساب الوقت الفاصل بين لينسفري21،،من2223.
النتائج
لعملية إعادة البناء الثلاثية الأبعاد، يتم وصف الحقل الخفيفة بحقل القيم مفردة (حيث 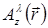 هي قيمة A معقدة على متن الطائرة على مسافة ض من الموقف عينة، والأفقي
هي قيمة A معقدة على متن الطائرة على مسافة ض من الموقف عينة، والأفقي 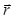 وفي الطول الموجي λ). وعلى غرار نش...
وفي الطول الموجي λ). وعلى غرار نش...
Discussion
في هذه الورقة، ونظهر أن الفحص المجهري خالية من عدسة الفيديو يمكن استخدامها داخل حاضنة لالتقاط حركية الآلاف من الخلايا. من أجل وصف المنهجية الشاملة التي شرحنا كيف يمكن تحليلها يوم 2.7 اكتساب الوقت الفاصل بين خلايا هيلا في الثقافة مع معيار خلية تتبع خوارزميات. والنتيجة هي إحدى وحدات dataset يضم 2...
Disclosures
الكتاب ليس لها علاقة بالكشف عن.
Acknowledgements
الكتاب ليس الاعتراف لها.
Materials
| Name | Company | Catalog Number | Comments |
| Cytonote lens-free video microscope | Iprasense | ||
| Horus acquisition software | Iprasense | ||
| 6-well glass bottom culture plates | MatTek corporation | Part No: P06G-0-14-F | |
| DMEM + GlutaMAX medium | Gibco | ||
| heat-inactivated fetal calf serum | Eurobio | ||
| penicillin and streptomycin | Gibco | ||
| Fibronectin | Sigma Aldrich | ||
| Matlab, image processing toolbox | Mathworks |
References
- Zangle, T. A., Teitell, M. A. Live-cell mass profiling: an emerging approach in quantitative biophysics. Nat Methods. 11 (12), 1221-1228 (2014).
- Popescu, G., Park, K., Mir, M., Bashir, R. New technologies for measuring single cell mass. Lab Chip. 14 (4), 646-652 (2014).
- Reed, J., et al. Rapid, massively parallel single-cell drug response measurements via live cell interferometry. Biophys J. 101 (5), 1025-1031 (2011).
- Mir, M., et al. Optical measurement of cycle-dependent cell growth. Proc Natl Acad Sci. USA. 108 (32), 13124-13129 (2011).
- Girshovitz, P., Shaked, N. T. Generalized cell morphological parameters based on interferometric phase microscopy and their application to cell life cycle characterization. Biomed Opt Express. 3 (8), 1757-1773 (2012).
- Kemper, B., Bauwens, A., Vollmer, A., Ketelhut, S., Langehanenberg, P. Label-free quantitative cell division monitoring of endothelial cells by digital holographic microscopy. J Biomed Opt. 15 (3), (2010).
- Mir, M., Bergamaschi, A., Katzenellenbogen, B. S., Popescu, G. Highly sensitive quantitative imaging for monitoring single cancer cell growth kinetics and drug response. PLoS One. 9 (2), 1-8 (2014).
- Bon, P., Savatier, J., Merlin, M., Wattellier, B., Monneret, S. Optical detection and measurement of living cell morphometric features with single-shot quantitative phase microscopy. J Biomed Opt. 17 (7), (2012).
- Aknoun, S., et al. Living cell dry mass measurement usinq quantitative phase imaging with quadriwave lateral shearing interferometry: an accuracy and sensitivity discussion. J Biomed Opt. 20 (1), 1-4 (2015).
- Cotte, Y., et al. Marker-free phase nanoscopy. Nature Photonics. 7 (2), 113-117 (2013).
- Choi, W., et al. Tomographic phase microscopy. Nat Methods. 4 (9), 717-719 (2007).
- Kesavan, S. V., et al. High-throughput monitoring of major cell functions by means of lensfree video microscopy. Sci Rep. 4, 1-11 (2014).
- Zheng, G., Lee, S. A., Antebi, Y., Elowitz, M. B., Yang, C. The ePetri dish, an on-chip cell imaging platform based on subpixel perspective sweeping microscopy (SPSM). Proc Natl Acad Sci USA. 108 (41), 16889-16894 (2011).
- Pushkarsky, I., et al. Automated single-cell motility analysis on a chip using lensfree microscopy. Sci Rep. 4, 4717 (2014).
- Allier, C., et al. Imaging of dense cell cultures by multiwavelength lens-free video microscopy. Cytom Part A. 91 (5), 1-10 (2017).
- Su, T. -. W., Seo, S., Erlinger, A., Ozcan, A. High-throughput lensfree imaging and characterization of a heterogeneous cell solution on a chip. Biotechnol Bioeng. 102 (3), 856-868 (2009).
- Delacroix, R., et al. Cerebrospinal fluid lens-free microscopy: a new tool for the laboratory diagnosis of meningitis. Sci Rep. 7, 39893 (2017).
- Tinevez, J. -. Y., et al. TrackMate: an open and extensible platform for single-particle tracking. Methods. 115, 80-90 (2016).
- Popescu, G. Optical imaging of cell mass and growth dynamics. Am J Physiol Physiol. 295 (2), 538-544 (2008).
- Liu, P. Y., et al. Cell refractive index for cell biology and disease diagnosis: past, present and future. Lab Chip. 16, 634-644 (2016).
- Rapoport, D. H., Becker, T., Mamlouk, A. M., Schicktanz, S., Kruse, C. A novel validation algorithm allows for automated cell tracking and the extraction of biologically meaningful parameters. PLoS One. 6 (11), e27315 (2011).
- Al-Kofahi, O., et al. Automated cell lineage construction: A rapid method to analyze clonal development established with murine neural progenitor cells. Cell Cycle. 5 (3), 327-335 (2006).
- Meijering, E., Dzyubachyk, O., Smal, I., van Cappellen, W. A. Tracking in cell and developmental biology. Semin Cell Dev Biol. 20 (8), 894-902 (2009).
- Posakony, J. W., England, J. M., Attardi, G. Mitochondrial growth and division during the cell cycle in HeLa cells. J Cell Biol. 74 (2), 468-491 (1977).
- Zocchi, E., et al. Expression of CD38 Increases Intracellular Calcium Concentration and Reduces Doubling Time in HeLa and 3T3 Cells. J Biol Chem. 273 (14), 8017-8024 (1979).
- Reitzer, L. J., Wice, B. M., Kennell, D. Evidence that glutamine, not sugar, is the major energy source for cultured HeLa cells. J Biol Chem. 254 (8), 2669-2676 (1979).
- Benedetti, A. D. E., Joshi-barve, S., Rinker-Schaeffer, C., Rhoads, R. E. Expression of Antisense RNA against Initiation Factor eIF-4E mRNA in HeLa Cells Results in Lengthened Cell Division Times, Diminished Translation Rates, and Reduced Levels of Both eIF-4E and the p220 Component of eIF-4F. Mol Cell Biol. 11 (11), 5435-5445 (1991).
- Kumei, Y., Nakajima, T., Sato, A., Kamata, N., Enomoto, S. Reduction of G1 phase duration and enhancement of c-myc gene expression in HeLa cells at hypergravity. J Cell Sci. 93 (2), 221-226 (1989).
- Ginzberg, M. B., Kafri, R., Kirschner, M. On being the right (cell) size. Science. 348 (6236), 1245075 (2015).
- Mathieu, E., et al. Time-lapse lens-free imaging of cell migration in diverse physical microenvironments. Lab Chip. 16 (17), 3304-3316 (2016).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved