È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Lente-free Video microscopia per la dinamica e analisi quantitativa della coltura delle cellule aderenti
In questo articolo
Riepilogo
Lente-libero video microscopia permette di monitorare le colture cellulari direttamente all'interno dell'incubatrice. Qui descriviamo il protocollo completo utilizzato per acquisire e analizzare un'acquisizione di 2,7 giorni lunghi delle cellule coltivate di HeLa, che conduce a un dataset di 2.2 x 106 misurazioni della morfologia delle cellule individuali e ciclo cellulare 10584 tracce.
Abstract
Qui, dimostriamo che lente-libero video microscopia permette di catturare simultaneamente la cinetica di migliaia di cellule direttamente all'interno dell'incubatrice e che è possibile monitorare e quantificare singole cellule lungo diversi cicli cellulari. Descriviamo il protocollo completo utilizzato per monitorare e quantificare una coltura delle cellule HeLa per 2,7 giorni. In primo luogo, acquisizione di cultura cellulare viene eseguita con un microscopio obiettivo-libero video, e quindi i dati vengono analizzati seguendo un processo in quattro fasi: rilevazione delle cellule segmentazione e la divisione cellulare delle cellule-rilevamento, ricostruzione olografica di multi-lunghezza d'onda, algoritmi. Di conseguenza, mostriamo che è possibile raccogliere un dataset con più di 10.000 brani del ciclo cellulare e più di 2 x 106 cella misurazioni morfologiche.
Introduzione
Cellule di mammiferi coltivate nel corso di diversi cicli cellulari di monitoraggio e misurazione con precisione dimensioni delle celle e celle massa secca è un compito impegnativo. Diverse tecniche ottiche privo di etichetta sono in grado di eseguire questo compito1,2: uno spostamento di fase interferometria3, microscopia digitale oleografica (DHM)4,5,6, 7, quadriwave laterale taglio interferometria8,9 e fase quantitativa tomografia10,11. Questi metodi hanno portato a molte nuove idee per la comprensione del ciclo cellulare delle cellule di mammiferi. Tuttavia raramente sono accoppiati con cella automatica algoritmi di rilevamento e loro velocità effettiva resta limitato durante la misurazione delle cellule traiettorie massa1 (N < 20 rispettivamente3,4,5 , 6). quindi un nuovo metodo ottico è necessario misurare il traiettorie massa cellulare con grandi statistiche (N > 1000).
In questa carta, dimostriamo la capacità della lente-libero video microscopia di immagine contemporaneamente migliaia di cellule direttamente all'interno dell'incubatrice e quindi quantificare metriche di singola cellula lungo migliaia di tracce individuali ciclo cellulare. Privo di lente la microscopia è una fase quantitativa, tecnica che consente l'acquisizione dell'immagine di fase delle cellule densamente imballate su un campo visivo molto ampio di imaging (in genere diverse decine di mm2, qui 29,4 mm2)12,13 ,14,15. Diversi criteri di misurazione a livello di singola cellula sono determinati, ad es., zona di cella, cellulare massa secca, spessore della cella, lunghezza asse maggiore delle cellule e delle cellule di proporzioni12,15, da ogni immagine. Quindi, applicando un algoritmo di rilevamento delle cellule, queste caratteristiche possono essere tracciate per ogni singola cella come una funzione dell'esperimento tempo14,15. Inoltre, rilevando la presenza di divisioni cellulari nelle tracce delle cellule, è possibile estrarre altre informazioni importanti come la cella iniziale a secco massa (appena dopo la divisione delle cellule), la massa secca di cella finale (appena prima la divisione delle cellule) e la cella ciclo di durata, cioè, il tempo tra due divisioni consecutive15. Tutte queste misure possono essere computate con statistiche molto buone (N > 1000) perché l'ampio campo visivo in genere consentirebbe l'analisi di 200 a 10.000 cellule in una singola acquisizione privo di lente.
Per spiegare questa metodologia basata sulla lente-libero video microscopia, descriviamo il protocollo per monitorare e quantificare una coltura delle cellule HeLa per 2,7 giorni. Analisi dei dati è un processo in quattro fasi basato sulla ricostruzione olografica multi-lunghezza d'onda, cella-rilevamento, segmentazione delle cellule e algoritmi di divisione cellulare. Qui è indicato che la risoluzione spaziale e la frequenza di fotogrammi relativamente veloce (uno acquisizione ogni 10 minuti) ottenuti con questa configurazione obiettivo-libero video microscopia è compatibile con algoritmi di rilevamento delle cellule standard. L'analisi completa di questo dataset deriva nella misurazione di 10.584 cella tracce sopra cicli completi di cella.
Per riassumere, privo di lente video microscopia è un potente strumento per monitorare automaticamente migliaia di senza etichetta, non sincronizzato e le cellule non modificate ogni esperimento; ogni cella monitorata nel corso di diversi cicli cellulari. Le nostre misurazioni forniscono così il valore medio dei diversi parametri di cella, ma ancora più importante, la variabilità inter-cella sopra una grande popolazione di cellule.
Protocollo
1. cella cultura monitoraggio acquisizione
- Crescere le cellule HeLa in DMEM + glutammina (ad es., GlutaMAX) supplementato con 10% (v/v) fetale di vitello inattivato per calore del siero e l'1% penicillina e streptomicina.
- Cappotto lastre di cultura di fondo 6 pozzetti vetro con fibronectina (25 µ g/mL) per 1 h. Allora seed 2 x 104 cellule per pozzetto.
- Durante l'acquisizione, cambiare il mezzo ogni 3 giorni.
- Per l'acquisizione di time-lapse, utilizzare il video microscopio obiettivo-gratuita (disponibile in commercio).
Nota: Questo si basa la tecnica di imaging privo di lente computazionale come descritto da Ozcan et al. 16 che è stato modificato per eseguire il monitoraggio continuo in un incubatore a temperatura controllata di 37 ° C12,15. È dotato di un sensore di immagine del semiconductor (CMOS) di ossido di metallo complementare con un pixel pitch di 1,67 µm e un'area di imaging di 6.4 x 4.6 mm2. Più lunghezza d'onda illuminazione è fornita da un multichip luce che emettono diodo (LED) dispositivo che fornisce illuminazione rossa, verde e blu. Le lunghezze d'onda sono centrate, 636, 521 e 452 nm rispettivamente, con una larghezza di banda spettrale di 25, 45 e 25 nm rispettivamente. I LED (RGB) rosso, verde e blu si trovano sopra un pinhole 150 µm ad una distanza di circa 5 cm dalle cellule. Il potere della luce misurato vicino il LED è basso quanto 10 µW a ogni lunghezza d'onda e il tempo di illuminazione è solo un secondo per acquisizione. Quindi, non foto-tossicità sono previsti problemi quando colture cellulari sono osservati al microscopio video senza lente. - Mettere il contenitore di cultura cellulare messo in contatto con il sensore CMOS (Figura 1). Utilizzare un contenitore con un coprioggetto in vetro nella parte inferiore, che è importante per la qualità dell'acquisizione senza lente. Contenitori di plastica possono anche essere usati, ma l'acquisizione senza lente sarà degradata a causa della scarsa qualità ottica di questi contenitori.
- Controllare il lente senza microscopio video con il software di acquisizione (disponibile in commercio), che esegue sia l'acquisizione di time-lapse e la ricostruzione olografica. I parametri che devono essere immesse dall'utente nell'interfaccia del software sono il frame rate, la durata dell'esperimento e il tipo di coltura cellulare, cioè aderente celle o galleggiante.
- Impostare i parametri di input del software di acquisizione. Impostare il frame rate impostato su un'acquisizione ogni 10 minuti e impostare il tipo di coltura cellulare di 'seguace'.
Nota: Un frame rate di uno acquisizione ogni 10 minuti è un buon compromesso in quanto assicura una cella affidabile rilevamento mentre la dimensione del dataset risultante da analizzare è ancora ragionevole. Il più veloce frame rate ottenibile con questa configurazione di microscopia privo di lente è un'acquisizione ogni 5 minuti. Aumentando ulteriormente il framerate provoca eccessivo riscaldamento del sensore CMOS che possa influenzare la vitalità delle cellule in coltura. - Avviare l'acquisizione di time-lapse e l'algoritmo di ricostruzione olografica (disponibile in commercio) elaborerà automaticamente l'acquisizione senza lente per ottenere l'immagine di fase della coltura delle cellule. L'algoritmo di ricostruzione olografica si basa su un algoritmo di recupero multi-lunghezza d'onda fase17 e fornisce un'immagine ricostruita fase RGB nell'intervallo [-π, + π] per ogni singola cella sopra un grande campo di vista di 29,4 mm2. Il principio della lente-gratuita in linea olografia è quello di illuminare un campione sottile (z = 0) con un'onda piana (di ampiezza 1 dopo normalizzazione) e di rilevare su un sensore CMOS l'immagine di intensità di diffrazione successive a una breve distanza z = Z, in genere 1 mm dopo la campione. Nel nostro setup, l'illuminazione è passato in sequenza a 3 lunghezze d'onda (λ1= 0,450 µm, λ2= 0.540 µm, λ3= 0.647 µm) per misurare tre modelli di diffrazione del campione stesso.
2. cellula cultura dati analisi
- Per cella-tracking, è possibile utilizzare l'algoritmo di Trackmate, un plugin di Fiji open source per il rilevamento automatizzato delle singole particelle18. At in primo luogo, caricare l'acquisizione completa Time-lapse in Fiji (comando di sequenza di immagine di carico). A causa delle grandi dimensioni del set di dati, che presenta in genere 400 fotogrammi RGB di 29,7 Mb, è più veloce di caricarle in formato 8-bit. Successivamente il plugin Trackmate Fiji guida l'utente attraverso diverse fasi dell'algoritmo di rilevamento delle cellule, vale a dire una fase di rilevamento delle cellule, una fase di tracking cellulare e diversi filtri applicati per i rilevamenti di cella e le tracce computate. In ogni fase, gli algoritmi di configurare e visualizzare i risultati immediatamente in modo che, se necessario, si può facilmente navigare avanti e indietro per regolare le impostazioni. I vari menù dell'interfaccia Trackmate sono mostrati in Figura 2 con le impostazioni effettive utilizzate per l'esperimento di cellule HeLa.
- Impostare i parametri di input del software di acquisizione come segue (Vedi Figura 2). Impostare il diametro stimato blob di 15 pixel, la soglia di rivelatore a 0,25, la distanza massima di collegamento di 15 pixel, la distanza massima di gap-chiusura a 15 pixel e il numero di punti nelle tracce a 3.5.
Nota: Queste impostazioni variano ovviamente da un esperimento a altro, soprattutto il 'blob stimato diametro' che corrisponde alle dimensioni delle cellule (qui data in pixel di 1,67 µm). La stessa osservazione vale per la 'distanza max collegamento' che rappresentano la distanza massima percorsa da una cella all'interno di due fotogrammi consecutivi (qui dato in pixel di 1,67 µm). Il valore ottimo dipenderebbe sulla motilità cellulare ma anche sulla densità delle cellule. - Alla fine del processo di rilevamento delle cellule, generare i risultati sotto forma di tre file di testo ('Analisi' pulsante, Vedi Figura 2). Il file più utile, denominato 'Punti nelle statistiche di tracce', è costituito da una tabella che elenca rilevate tutte le celle con le loro posizioni rispettive (x, y) nell'acquisizione, il numero di telaio e il loro numero di traccia. Sulla base di questi dati, eseguire la segmentazione delle cellule e rilevare divisioni cellulari utilizzando algoritmi dedicati (vedere file di codice supplementare). Gli altri due file sono denominati 'Links nelle statistiche di tracce' e 'Tenere traccia delle statistiche' e includere tutti i risultati a che fare con le tracce, ad esempio traccia durate, numero di lacune rilevate, traccia fotogramma iniziale, ecc.
- Utilizzare l'algoritmo di segmentazione delle cellule (Vedi codice supplementare file) per estrarre automaticamente diversi parametri che descrivono la morfologia delle cellule dall'immagine ricostruita fase. Queste metriche sono l'area della superficie delle cellule (S), la lunghezza di asse maggiore della cella (L), la lunghezza di asse minore di cella (l) e le proporzioni di cella (L/l). La fase ha recuperata da microscopia privo di lente è proporzionale alla densità e spessore dello strato esemplare come discusso in precedenza15.
- Oltre alle caratteristiche morfologiche della cellula, estrarre misure (CDM) della massa secche quantitativa delle cellule dalla fase ricostruito13,19,20
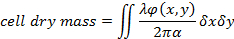 EQ. (1)
EQ. (1)
dove φ(x,y) è la fase ricostruito MAIUSC19, λ la lunghezza d'onda e α = 1,8 x 10-4 m3/kg l'indice di rifrazione specifico che riguarda la variazione dell'indice di rifrazione per asciugare la massa1, 19. - Valutare lo spessore di cella utilizzando una stima della differenza tra l'indice di rifrazione delle cellule e quella del terreno di coltura. La relazione tra la cella spessore h(x,y) e lo sfasamento misurato è dato da20:
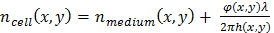 EQ. (2.1)
EQ. (2.1) EQ. (2,2)
EQ. (2,2)
con Δn(x,y) = ncella (x,y) - nmedia (x,y), la differenza di indice di rifrazione tra la cella e i terreni di coltura. In seguito, assumono un valore costante di Δn = 0.025, che è stimato dall'indice di rifrazione misurato nei nuclei delle cellule HeLa (n = 1.35511) e quella dei terreni di coltura di tampone fosfato salino (PBS) (n = 1.33). Anche se il valore di spessore di cella è solo indicativo in quanto si basa su un presupposto, sue relative variazioni sono significative e possono essere sfruttate per rilevare le cellule in divisione. - Utilizzare l'algoritmo dedicato (Vedi codice supplementare file) per rilevare la presenza di divisioni cellulari e quindi estrarre le tracce delle cellule che sono associate alle divisioni cellulari rilevate, al fine di rilevare una divisione cellulare, le cellule con un più grande di 8 µm di spessore misurato sono in primo luogo identificato, quindi verificare se una nuova cella apparirà strettamente in spazio e tempo. In questo modo, la rilevazione di divisioni cellulari si basa su due criteri robusti. Alternativa e più elaborati metodi per il rilevamento di divisione cellulare possono essere applicati al lensfree time-lapse acquisizione21,22,23.
Risultati
Per il processo di ricostruzione olografica, il campo di luce è descritta da un campo scalare A (dove 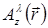 è il valore complesso di A sull'aereo a distanza z dalla posizione del campione e laterale
è il valore complesso di A sull'aereo a distanza z dalla posizione del campione e laterale 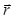 e a lunghezza d'onda λ). Propagazione della luce è modellato con la teoria di Huygens-Fresnel che fornisce un kernel propagatore
e a lunghezza d'onda λ). Propagazione della luce è modellato con la teoria di Huygens-Fresnel che fornisce un kernel propagatore
Discussione
In questa carta, indichiamo che lente-libero video microscopia può essere utilizzato all'interno di un'incubatrice per catturare la cinetica di migliaia di celle. Al fine di descrivere la metodologia globale che abbiamo spiegato come un'acquisizione di time-lapse 2,7 giorno delle cellule HeLa in cultura possa essere analizzati con algoritmi di rilevamento delle cellule standard. Il risultato è un set di dati con 2.2 x 106 cella misure e 10.584 tracce del ciclo cellulare. Le acquisizioni sono state effettuate...
Divulgazioni
Gli autori non hanno nulla a rivelare.
Riconoscimenti
Gli autori non hanno nulla a riconoscere.
Materiali
| Name | Company | Catalog Number | Comments |
| Cytonote lens-free video microscope | Iprasense | ||
| Horus acquisition software | Iprasense | ||
| 6-well glass bottom culture plates | MatTek corporation | Part No: P06G-0-14-F | |
| DMEM + GlutaMAX medium | Gibco | ||
| heat-inactivated fetal calf serum | Eurobio | ||
| penicillin and streptomycin | Gibco | ||
| Fibronectin | Sigma Aldrich | ||
| Matlab, image processing toolbox | Mathworks |
Riferimenti
- Zangle, T. A., Teitell, M. A. Live-cell mass profiling: an emerging approach in quantitative biophysics. Nat Methods. 11 (12), 1221-1228 (2014).
- Popescu, G., Park, K., Mir, M., Bashir, R. New technologies for measuring single cell mass. Lab Chip. 14 (4), 646-652 (2014).
- Reed, J., et al. Rapid, massively parallel single-cell drug response measurements via live cell interferometry. Biophys J. 101 (5), 1025-1031 (2011).
- Mir, M., et al. Optical measurement of cycle-dependent cell growth. Proc Natl Acad Sci. USA. 108 (32), 13124-13129 (2011).
- Girshovitz, P., Shaked, N. T. Generalized cell morphological parameters based on interferometric phase microscopy and their application to cell life cycle characterization. Biomed Opt Express. 3 (8), 1757-1773 (2012).
- Kemper, B., Bauwens, A., Vollmer, A., Ketelhut, S., Langehanenberg, P. Label-free quantitative cell division monitoring of endothelial cells by digital holographic microscopy. J Biomed Opt. 15 (3), (2010).
- Mir, M., Bergamaschi, A., Katzenellenbogen, B. S., Popescu, G. Highly sensitive quantitative imaging for monitoring single cancer cell growth kinetics and drug response. PLoS One. 9 (2), 1-8 (2014).
- Bon, P., Savatier, J., Merlin, M., Wattellier, B., Monneret, S. Optical detection and measurement of living cell morphometric features with single-shot quantitative phase microscopy. J Biomed Opt. 17 (7), (2012).
- Aknoun, S., et al. Living cell dry mass measurement usinq quantitative phase imaging with quadriwave lateral shearing interferometry: an accuracy and sensitivity discussion. J Biomed Opt. 20 (1), 1-4 (2015).
- Cotte, Y., et al. Marker-free phase nanoscopy. Nature Photonics. 7 (2), 113-117 (2013).
- Choi, W., et al. Tomographic phase microscopy. Nat Methods. 4 (9), 717-719 (2007).
- Kesavan, S. V., et al. High-throughput monitoring of major cell functions by means of lensfree video microscopy. Sci Rep. 4, 1-11 (2014).
- Zheng, G., Lee, S. A., Antebi, Y., Elowitz, M. B., Yang, C. The ePetri dish, an on-chip cell imaging platform based on subpixel perspective sweeping microscopy (SPSM). Proc Natl Acad Sci USA. 108 (41), 16889-16894 (2011).
- Pushkarsky, I., et al. Automated single-cell motility analysis on a chip using lensfree microscopy. Sci Rep. 4, 4717 (2014).
- Allier, C., et al. Imaging of dense cell cultures by multiwavelength lens-free video microscopy. Cytom Part A. 91 (5), 1-10 (2017).
- Su, T. -. W., Seo, S., Erlinger, A., Ozcan, A. High-throughput lensfree imaging and characterization of a heterogeneous cell solution on a chip. Biotechnol Bioeng. 102 (3), 856-868 (2009).
- Delacroix, R., et al. Cerebrospinal fluid lens-free microscopy: a new tool for the laboratory diagnosis of meningitis. Sci Rep. 7, 39893 (2017).
- Tinevez, J. -. Y., et al. TrackMate: an open and extensible platform for single-particle tracking. Methods. 115, 80-90 (2016).
- Popescu, G. Optical imaging of cell mass and growth dynamics. Am J Physiol Physiol. 295 (2), 538-544 (2008).
- Liu, P. Y., et al. Cell refractive index for cell biology and disease diagnosis: past, present and future. Lab Chip. 16, 634-644 (2016).
- Rapoport, D. H., Becker, T., Mamlouk, A. M., Schicktanz, S., Kruse, C. A novel validation algorithm allows for automated cell tracking and the extraction of biologically meaningful parameters. PLoS One. 6 (11), e27315 (2011).
- Al-Kofahi, O., et al. Automated cell lineage construction: A rapid method to analyze clonal development established with murine neural progenitor cells. Cell Cycle. 5 (3), 327-335 (2006).
- Meijering, E., Dzyubachyk, O., Smal, I., van Cappellen, W. A. Tracking in cell and developmental biology. Semin Cell Dev Biol. 20 (8), 894-902 (2009).
- Posakony, J. W., England, J. M., Attardi, G. Mitochondrial growth and division during the cell cycle in HeLa cells. J Cell Biol. 74 (2), 468-491 (1977).
- Zocchi, E., et al. Expression of CD38 Increases Intracellular Calcium Concentration and Reduces Doubling Time in HeLa and 3T3 Cells. J Biol Chem. 273 (14), 8017-8024 (1979).
- Reitzer, L. J., Wice, B. M., Kennell, D. Evidence that glutamine, not sugar, is the major energy source for cultured HeLa cells. J Biol Chem. 254 (8), 2669-2676 (1979).
- Benedetti, A. D. E., Joshi-barve, S., Rinker-Schaeffer, C., Rhoads, R. E. Expression of Antisense RNA against Initiation Factor eIF-4E mRNA in HeLa Cells Results in Lengthened Cell Division Times, Diminished Translation Rates, and Reduced Levels of Both eIF-4E and the p220 Component of eIF-4F. Mol Cell Biol. 11 (11), 5435-5445 (1991).
- Kumei, Y., Nakajima, T., Sato, A., Kamata, N., Enomoto, S. Reduction of G1 phase duration and enhancement of c-myc gene expression in HeLa cells at hypergravity. J Cell Sci. 93 (2), 221-226 (1989).
- Ginzberg, M. B., Kafri, R., Kirschner, M. On being the right (cell) size. Science. 348 (6236), 1245075 (2015).
- Mathieu, E., et al. Time-lapse lens-free imaging of cell migration in diverse physical microenvironments. Lab Chip. 16 (17), 3304-3316 (2016).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon