このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
レンズ無料のビデオ顕微鏡、付着性細胞培養の定量的解析
要約
レンズ無料ビデオ顕微鏡は、インキュベーターの中に直接培養細胞を監視することが出来ます。ここで集録し、解析を培養 HeLa 細胞の 2.7 日間の長い取得するために使用完全なプロトコルについて述べる、2.2 x 10 のデータセットにつながる個々 の細胞の形態や 10584 細胞周期の6測定を追跡します。
要約
レンズ無料ビデオを紹介ここでは、顕微鏡により直接インキュベーター内の細胞の何千もの速度を同時にキャプチャおよび監視し、いくつかの細胞周期に沿って単一セルを定量化することが可能であります。完全な監視し、2.7 日間の HeLa 細胞培養を定量化に使用プロトコルについて述べる。まず、レンズ無料ビデオ顕微鏡、細胞文化獲得して実行され、次の 4 段階のプロセス データを分析し、: 多波長ホログラフィック再構成、細胞追跡、セル分割と細胞分裂検出アルゴリズム。その結果、以上 10,000 の細胞周期トラックと 2 x 10 以上6細胞形態計測のデータセットの特徴を収集することが可能だと紹介します。
概要
いくつかの細胞周期を通じて培養細胞を監視し、正確に測定セルのサイズとセルの乾燥質量はやりがいのある仕事です。いくつかラベル無料光技術がこのタスク1,2を実行することができる: 位相シフト干渉法3、デジタル ホログラフィー顕微鏡法 (DHM)4,5,6,7quadriwave 横方向せん断干渉8,9定量位相トモグラフィ10,11。これらのメソッドは、哺乳動物細胞の細胞周期の理解に多くの新しい洞察力につながっています。しかし、彼らは、ほとんど追尾法自動セルと結合し、そのスループットは制限を測定するときセル大容量の軌跡1 (N < それぞれ3,4、5の 20,6). 光学手法は大規模な統計と細胞質量軌跡の測定に必要なため (N > 1000)。
本稿では、同時に数千、インキュベーターの中に直接細胞のイメージを作成し、単一のセル数千もの個々 の細胞周期トラックに沿って指標を定量化しレンズ無料ビデオ顕微鏡の機能を示します。顕微鏡のレンズ無料は定量位相イメージング技術の非常に広い視野で密集細胞の位相画像の取得を可能にする (通常数十 mm2、ここの 29.4 mm2)12,13 ,14,15。単一セルのレベルでいくつかのメトリックが決定されます、例えば領域をセル、携帯の乾燥質量、セル厚、セルの長軸の長さとセルの縦横比12,15, それぞれのイメージから。その後、細胞追跡アルゴリズムを適用すると、これらの機能をプロットできますすべての単一セルの実験時間14,15の関数として。さらに、セルのトラックで細胞分裂の発生を検出することにより初期の細胞乾燥質量 (細胞分裂) の直後に、(細胞分裂) の直前に最後のセルの乾燥質量とセルの他の重要な情報を抽出することが可能です。期間、すなわち、2 つの連続した分割15間の時間を周期します。これらのすべての測定値を計算する非常によい統計量 (N > 1000) 以来、ビューの大きなフィールドが通常単一レンズ無料取得 200 に 10,000 細胞の解析を許可。
レンズ無料ビデオ顕微鏡検査に基づいたこの方法を説明するために監視および 2.7 日間の HeLa 細胞培養を定量化するプロトコルについて述べる。データ解析、多波長ホログラフィック再構成、細胞追跡、セル分割と細胞分裂のアルゴリズムに基づいた 4 つのステップ プロセスです。ここで空間分解能とこのレンズ無料ビデオ顕微鏡のセットアップで得られた比較的高速なフレーム レート (10 分ごと 1 つ取得) の標準的な細胞追跡アルゴリズムと互換性があることを示します。このデータセットの完全な分析は完全な細胞周期上 10,584 セル トラックの測定結果します。
要約すれば、レンズ無料ビデオ顕微鏡は何千ものラベル、同期されていないと実験; ごとそのままセルを自動的に監視する強力なツール各セルは、いくつかの細胞周期を追跡されています。我々 の測定はこのように細胞の大規模な人口でセル間変動より重要なことは、しかし、いくつかのセルのパラメーターの平均値を提供します。
プロトコル
1. 買収の監視培養
- DMEM + グルタミン (例えばGlutaMAX) の HeLa 細胞を成長 10% (v/v) 熱不活化牛胎児血清および 1% ペニシリンとストレプトマイシン培。
- フィブロネクチン (25 μ g/mL) 1 h で 6 よくガラス底培養皿をコートします。ウェルあたり 2 x 10 の4セルをシードします。
- 買収は、中に 3 日おきに媒体を変更します。
- 時間経過の取得ビデオの無料のレンズ顕微鏡 (市販) を使用します。
注: これは、レンズ無料計算イメージング法に基づいて Ozcan et al.による記述で16 37 ° C12,15の温度制御でインキュベーターで継続的な監視を実行する変更されました。1.67 μ m の画素ピッチ 6.4 x 4.6 mm2のイメージング領域と相補的金属酸化物半導体 (CMOS) イメージ センサーを提供しています。複数の波長の照明は、マルチチップ光発光ダイオード (Led) デバイス、赤、緑、青の照明を提供するによって提供されます。波長が中心に、452、521, 636 nm それぞれ、25、45、25 のスペクトル帯域幅を持つ nm それぞれ。赤、緑、青 (RGB) Led は、セルから約 5 cm の距離で 150 μ m のピンホールの上にあります。LED に近い測定光は各波長で 10 μ と低く、照明時間は 1 秒当たりの取得。したがって、写真毒性の問題も、細胞培養、レンズ無料ビデオ顕微鏡下で観察すると必要ありません。 - CMOS センサー (図 1) と接触して置く細胞培養容器を入れなさい。レンズ無料取得の品質にとって重要である下部にガラス基板とコンテナーを使用します。プラスチック容器も使用できますが、レンズ無料獲得がこれらのコンテナーの光質の悪さにより低下します。
- ビデオ レンズ無料顕微鏡集録ソフトウェア (市販) が時間経過の取得とホログラフィック再構成を実行するを制御します。ソフトウェア インターフェイスでユーザーが入力する必要があるパラメーターがフレーム レート、実験、細胞培養、すなわち付着性のセルまたは浮遊細胞の種類の期間。
- 集録ソフトウェアの入力パラメーターを設定します。1 つ取得 10 分ごとに、「付着」に細胞培養型を設定フレーム レートを設定します。
注: 追跡分析する結果のデータセットのサイズはまだ合理的な信頼性の高いセルに、1 つ取得 10 分ごとのフレーム レートは良い妥協案です。この顕微鏡のレンズ無料セットアップで達成可能な最速のフレーム レートは、1 つの取得は 5 分ごとです。培養細胞の生存率に影響を与えることができる CMOS センサーの過剰加熱の結果、フレーム レートがさらに向上します。 - コマ撮りの集録を開始し、ホログラフィック再構成アルゴリズム (市販) は、細胞培養の位相画像を取得するレンズ無料取得を自動的に処理します。ホログラフィック再構成アルゴリズムの多波長位相検索アルゴリズム17に基づいておりの範囲で RGB 再構成画像を提供します [-π、π +] 29.4 mm2のビューの大きなフィールドを単一セルごとに。レンズ無料で線ホログラフィーの原理は薄いサンプルを照らす (z = 0) (振幅 1 の正規化後) の平面波と短い距離 z 以降の回折強度画像 CMOS センサーで検出する = Z 後、通常 1 mm、サンプルです。セットアップ時に照明が順番に 3 波長に切り替えられた (λ1= 0.450 μ、λ20.540 μ、λ3を = = 0.647 μ m) 同じサンプルの 3 つの回折パターンを測定します。
2. 細胞培養データ解析
- セルの追跡を単一粒子18の自動追跡のためのオープン ソース フィジー プラグイン Trackmate アルゴリズムを使用します。At はまず、フィジー (負荷イメージ シーケンス コマンド) にコマ撮り完全買収を読み込みます。29.7 Mb 通常 400 RGB フレームの機能は、データセットのサイズが大きいため 8 ビットの形式でそれらをロードするより高速です。次に Trackmate フィジー プラグインは、すなわち細胞検出期細胞トラッカー期、細胞検出および計算のトラックに適用されるいくつかのフィルター、細胞追跡アルゴリズムのいくつかの段階でユーザーをガイドします。すべての段階でアルゴリズムを構成し、結果を表示すぐにして、必要に応じて、1 つ簡単に移動できます前後設定を再調整します。Trackmate インタ フェースのさまざまなメニューは HeLa 細胞実験に使用される実際の設定で図 2のとおりです。
- (図 2参照) を次のよう入力ソフトウェアの入力パラメーターを設定します。15 ピクセル、15 ピクセルにギャップを閉じる最大距離と 3.5 にトラックでのスポットの数に 15 ピクセルに推定される blob 直径 0.25 検出しきい値、リンクの最大距離を設定します。
メモ: これらの設定が別の特に '推定 blob 直径」(ここでは 1.67 μ m のピクセルで与えられる) セル サイズに対応する異なる 1 つの実験から明らかに。同じ発言は (ここでは 1.67 μ m のピクセルで与えられる) 2 つの連続するフレーム内のセルで、最大距離を占める旅「リンク最大距離」に適用されます。最適な値は、細胞運動、細胞密度にも依存します。 - 細胞追跡プロセスの最後に、3 つのテキスト ファイルの形式で結果を生成 (「解析」ボタンを図 2を参照します).最も有用なファイル、'統計追跡のスポット' は、買収、フレーム数、トラック数 (x, y) それぞれの立場で検出されたすべてのセルの一覧で構成されます。このデータに基づいてセル分割を実行し、(サプルメンタリ コード ファイルを参照してください) 専用のアルゴリズムを使用して細胞分裂を検出します。他の 2 つのファイルがという名前の 'トラックの統計情報にリンク' と 'トラック' の統計情報とトラック、トラック期間など、検出されたギャップ数を扱うすべての結果を含む、最初のフレームなどを追跡。
- セル分割アルゴリズムを使用して、自動的に再構成画像からの細胞の形態を記述するいくつかの指標を抽出する (を参照してください補足コード ファイル)。これらの指標は、細胞表面積 (S)、細胞の長軸の長さ (L)、セルの短軸の長さ (l) とセルの縦横比 (l)。顕微鏡のレンズ無料から回復フェーズは、密度と前述のとおり15試料の層の厚さに比例です。
- 細胞の形態学的特徴、に加えて再構成13,19,20から定量細胞乾燥質量 (CDM) 測定を抽出します。
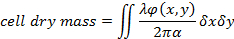 イコライザー (1)
イコライザー (1)
φ(x,y) が再建された位相シフト19波長 λ と α = 1.8 × 10-4 m3/kg乾燥質量1、屈折率の変化を関連している特定の屈折 19。 - セルの屈折と培地の違いの推定を用いたセル厚を評価します。セル厚h(x,y) と位相シフトの関係は、20で与えられます。
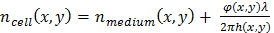 式 (2.1)
式 (2.1) 式 (2.2)
式 (2.2)
Δn(x,y) = nセル(x、y) - n培地(x、y)、細胞と培地との間の屈折率差。以下では、Δ の定数値を想定n = 0.025、HeLa 細胞核で測定した屈折率からと推定されている (n = 1.35511)、リン酸緩衝生理食塩水 (PBS) 文化メディアの (n = 1.33)。セルの厚さ値は仮定に基づいているように示して、その相対的な変化が意味のある、分裂細胞を検出するために悪用される可能性が。 - 専用のアルゴリズムを使用して測定した厚さ 8 μ m より大きいによる細胞の細胞分裂の発生を検出し、細胞分裂を検出するために検出された細胞の分裂に関連付けられているセルのトラックを抽出する (を参照してください補足コード ファイル)まず識別されます、確認してくださいかどうか新しい細胞が密接に空間し、時間。この方法で細胞分裂の検出は、2 つの堅牢な条件に依存します。Lensfree コマ獲得21,22,23に代替と細胞分裂の検出のためのより精巧な方法を適用できます。
結果
ホログラフィック再構成プロセスのライト フィールドは、スカラー場によって記述されます (、 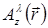 Aのサンプル、および横の位置から距離z平面上の複雑な値は、
Aのサンプル、および横の位置から距離z平面上の複雑な値は、 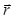 、波長 λ)。光の伝搬はプロパゲータ カーネルを提供する Huygens フレネル理論によるモ...
、波長 λ)。光の伝搬はプロパゲータ カーネルを提供する Huygens フレネル理論によるモ...
ディスカッション
本稿ではそのレンズ無料ビデオ顕微鏡は、細胞の何千もの速度をキャプチャするインキュベーター内使用ことができますを示す.全体的な方法論を記述するために、標準細胞追跡アルゴリズムの 2.7 日コマ撮りによる培養 HeLa 細胞の分析方法を説明しました。結果は、2.2 × 106セル測定と 10,584 細胞周期トラックを備えデータセットです。買収を行った比較的大規模な細胞間距離と Hela ?...
開示事項
著者が明らかに何もありません。
謝辞
著者が認めることを何もありません。
資料
| Name | Company | Catalog Number | Comments |
| Cytonote lens-free video microscope | Iprasense | ||
| Horus acquisition software | Iprasense | ||
| 6-well glass bottom culture plates | MatTek corporation | Part No: P06G-0-14-F | |
| DMEM + GlutaMAX medium | Gibco | ||
| heat-inactivated fetal calf serum | Eurobio | ||
| penicillin and streptomycin | Gibco | ||
| Fibronectin | Sigma Aldrich | ||
| Matlab, image processing toolbox | Mathworks |
参考文献
- Zangle, T. A., Teitell, M. A. Live-cell mass profiling: an emerging approach in quantitative biophysics. Nat Methods. 11 (12), 1221-1228 (2014).
- Popescu, G., Park, K., Mir, M., Bashir, R. New technologies for measuring single cell mass. Lab Chip. 14 (4), 646-652 (2014).
- Reed, J., et al. Rapid, massively parallel single-cell drug response measurements via live cell interferometry. Biophys J. 101 (5), 1025-1031 (2011).
- Mir, M., et al. Optical measurement of cycle-dependent cell growth. Proc Natl Acad Sci. USA. 108 (32), 13124-13129 (2011).
- Girshovitz, P., Shaked, N. T. Generalized cell morphological parameters based on interferometric phase microscopy and their application to cell life cycle characterization. Biomed Opt Express. 3 (8), 1757-1773 (2012).
- Kemper, B., Bauwens, A., Vollmer, A., Ketelhut, S., Langehanenberg, P. Label-free quantitative cell division monitoring of endothelial cells by digital holographic microscopy. J Biomed Opt. 15 (3), (2010).
- Mir, M., Bergamaschi, A., Katzenellenbogen, B. S., Popescu, G. Highly sensitive quantitative imaging for monitoring single cancer cell growth kinetics and drug response. PLoS One. 9 (2), 1-8 (2014).
- Bon, P., Savatier, J., Merlin, M., Wattellier, B., Monneret, S. Optical detection and measurement of living cell morphometric features with single-shot quantitative phase microscopy. J Biomed Opt. 17 (7), (2012).
- Aknoun, S., et al. Living cell dry mass measurement usinq quantitative phase imaging with quadriwave lateral shearing interferometry: an accuracy and sensitivity discussion. J Biomed Opt. 20 (1), 1-4 (2015).
- Cotte, Y., et al. Marker-free phase nanoscopy. Nature Photonics. 7 (2), 113-117 (2013).
- Choi, W., et al. Tomographic phase microscopy. Nat Methods. 4 (9), 717-719 (2007).
- Kesavan, S. V., et al. High-throughput monitoring of major cell functions by means of lensfree video microscopy. Sci Rep. 4, 1-11 (2014).
- Zheng, G., Lee, S. A., Antebi, Y., Elowitz, M. B., Yang, C. The ePetri dish, an on-chip cell imaging platform based on subpixel perspective sweeping microscopy (SPSM). Proc Natl Acad Sci USA. 108 (41), 16889-16894 (2011).
- Pushkarsky, I., et al. Automated single-cell motility analysis on a chip using lensfree microscopy. Sci Rep. 4, 4717 (2014).
- Allier, C., et al. Imaging of dense cell cultures by multiwavelength lens-free video microscopy. Cytom Part A. 91 (5), 1-10 (2017).
- Su, T. -. W., Seo, S., Erlinger, A., Ozcan, A. High-throughput lensfree imaging and characterization of a heterogeneous cell solution on a chip. Biotechnol Bioeng. 102 (3), 856-868 (2009).
- Delacroix, R., et al. Cerebrospinal fluid lens-free microscopy: a new tool for the laboratory diagnosis of meningitis. Sci Rep. 7, 39893 (2017).
- Tinevez, J. -. Y., et al. TrackMate: an open and extensible platform for single-particle tracking. Methods. 115, 80-90 (2016).
- Popescu, G. Optical imaging of cell mass and growth dynamics. Am J Physiol Physiol. 295 (2), 538-544 (2008).
- Liu, P. Y., et al. Cell refractive index for cell biology and disease diagnosis: past, present and future. Lab Chip. 16, 634-644 (2016).
- Rapoport, D. H., Becker, T., Mamlouk, A. M., Schicktanz, S., Kruse, C. A novel validation algorithm allows for automated cell tracking and the extraction of biologically meaningful parameters. PLoS One. 6 (11), e27315 (2011).
- Al-Kofahi, O., et al. Automated cell lineage construction: A rapid method to analyze clonal development established with murine neural progenitor cells. Cell Cycle. 5 (3), 327-335 (2006).
- Meijering, E., Dzyubachyk, O., Smal, I., van Cappellen, W. A. Tracking in cell and developmental biology. Semin Cell Dev Biol. 20 (8), 894-902 (2009).
- Posakony, J. W., England, J. M., Attardi, G. Mitochondrial growth and division during the cell cycle in HeLa cells. J Cell Biol. 74 (2), 468-491 (1977).
- Zocchi, E., et al. Expression of CD38 Increases Intracellular Calcium Concentration and Reduces Doubling Time in HeLa and 3T3 Cells. J Biol Chem. 273 (14), 8017-8024 (1979).
- Reitzer, L. J., Wice, B. M., Kennell, D. Evidence that glutamine, not sugar, is the major energy source for cultured HeLa cells. J Biol Chem. 254 (8), 2669-2676 (1979).
- Benedetti, A. D. E., Joshi-barve, S., Rinker-Schaeffer, C., Rhoads, R. E. Expression of Antisense RNA against Initiation Factor eIF-4E mRNA in HeLa Cells Results in Lengthened Cell Division Times, Diminished Translation Rates, and Reduced Levels of Both eIF-4E and the p220 Component of eIF-4F. Mol Cell Biol. 11 (11), 5435-5445 (1991).
- Kumei, Y., Nakajima, T., Sato, A., Kamata, N., Enomoto, S. Reduction of G1 phase duration and enhancement of c-myc gene expression in HeLa cells at hypergravity. J Cell Sci. 93 (2), 221-226 (1989).
- Ginzberg, M. B., Kafri, R., Kirschner, M. On being the right (cell) size. Science. 348 (6236), 1245075 (2015).
- Mathieu, E., et al. Time-lapse lens-free imaging of cell migration in diverse physical microenvironments. Lab Chip. 16 (17), 3304-3316 (2016).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved