Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Objektiv-kostenlose Videomikroskopie für die Dynamik und Quantitative Analyse der adhärente Zellkultur
In diesem Artikel
Zusammenfassung
Objektiv-freie Videomikroskopie ermöglicht es uns, Zellkulturen direkt in die Brutmaschine zu überwachen. Hier beschreiben wir die vollständige Protokoll zum erwerben und analysieren eine 2,7 Tage lang Übernahme von kultivierten HeLa-Zellen, was zu einem Dataset von 2,2 x 106 Messungen der einzelnen Zellmorphologie und 10584 Zellzyklus tracks.
Zusammenfassung
Hier zeigen wir, dass Objektiv-freie Video Mikroskopie ermöglicht es, gleichzeitig die Kinetik von Tausenden von Zellen direkt in die Brutmaschine zu erfassen und es ist möglich, zu überwachen und Einzelzellen entlang mehrere Zelle Zyklen zu quantifizieren. Wir beschreiben das vollständige Protokoll zur Überwachung und Quantifizierung einer HeLa Zellkultur für 2,7 Tage. Erstens Zelle Kultur Erwerb erfolgt mit einem Objektiv-freie video Mikroskop, und dann die Daten analysiert, nach einem vier-Stufen-Prozess: Multi-Wellenlänge holographischen Rekonstruktion, Handy-Tracking, Zelle Segmentierung und Zellteilung-Erkennung Algorithmen. Dadurch zeigen wir, dass es möglich ist, sammeln Sie ein Dataset mit mehr als 10.000 Zellzyklus-Tracks und mehr als 2 x 106 Zelle morphologischen Messungen.
Einleitung
Kultivierte Säugerzellen in mehrere Zelle Zyklen Überwachung und Messung genau Zelle Größe und Zelle Trockenmasse ist eine anspruchsvolle Aufgabe. Verschiedene optische markierungsfreie Techniken sind in der Lage, diese Aufgabe1,2: Phasenverschiebung Interferometrie3, digitaler holographischer Mikroskopie (DHM)4,5,6, 7, Quadriwave laterale shearing Interferometrie8,9 und quantitativen Phase Tomographie10,11. Diese Methoden führten zu viele neue Einblicke in das Verständnis des Zellzyklus von Säugerzellen. Aber sie sind selten gepaart mit Automatikzelle tracking-Algorithmen und deren Durchsatz begrenzt, bleibt bei der Messung von Zelle Masse Bahnen1 (N < 20 bzw.3,4,5 , 6). daher eine neuartige optische Methode erforderlich ist, um die Zelle Masse Trajektorien mit großen Statistiken messen (N > 1000).
In diesem Beitrag zeigen wir die Fähigkeit der Linse-freie Videomikroskopie, gleichzeitig Tausende von Zellen direkt in die Brutmaschine Bild, und dann einzelne Zelle Metriken an Tausende von einzelnen Zelle Radwege zu quantifizieren. Objektiv-freie Mikroskopie ist eine quantitative Phase bildgebende Technik, die Phase Bildaufnahme von dicht gepackten Zellen über ein sehr großes Sehfeld ermöglicht (in der Regel mehrere zehntausend mm2, hier 29,4 mm2)12,13 ,14,15. Werden mehrere Metriken auf der Ebene der einzelnen Zelle bestimmt, z.B.Handy-Bereich, Handy-Trockenmasse, Zelle Dicke, Zelle Hauptachse Länge und Zell-Seitenverhältnis12,15, aus jedem Bild. Dann können diese Funktionen durch die Anwendung eines Handy-Tracking-Algorithmus, für jede einzelne Zelle in Abhängigkeit von dem Experiment Mal14,15geplottet werden. Durch das Auftreten von Zellteilungen in der Zelle Spuren erkennen, ist es darüber hinaus möglich, weitere wichtige Informationen zu extrahieren, wie z. B. die erste Zelle trockene Masse (kurz nach der Zellteilung), die letzte Zelle Trockenmasse (kurz vor der Zellteilung) und die Zelle Dauer, d. h., die Zeit zwischen zwei aufeinander folgenden Divisionen15-Zyklus. All diese Messungen mit sehr gute Statistiken berechnet werden können (N > 1000) da das große Sichtfeld in der Regel die Analyse von 200 bis 10.000 Zellen in eine einzelne Linse-freie Erwerb ermöglichen würde.
Um diese Methode basiert auf Objektiv-freie Videomikroskopie erklären, beschreiben wir das Protokoll zur Überwachung und zur Quantifizierung einer HeLa Zellkultur für 2,7 Tage. Die Datenanalyse ist ein vier-Stufen-Prozess basierend auf Multi-Wellenlänge holographischen Rekonstruktion, Handy-Tracking, Zelle Segmentierung und Zellteilung Algorithmen. Hier wird gezeigt, dass die räumliche Auflösung und die relativ schnelle Bildrate (ein Erwerb alle 10 Minuten) erhalten mit diesem Objektiv-freie Videomikroskopie Setup kompatibel mit standard-Handy-Tracking-Algorithmen. Die vollständige Analyse dieses Datensatzes führt die Messung der 10.584 Zelle Tracks über vollständige Zelle Zyklen.
Zusammenfassend lässt sich sagen, ist Objektiv-free video-Mikroskopie ein leistungsfähiges Werkzeug automatisch Tausende von unbeschrifteten, unsynchronisiert und unveränderten Zellen pro Experiment zu überwachen; Jede Zelle, die über mehrere Zyklen der Zelle verfolgt. Unsere Messungen liefern somit den Mittelwert aus mehreren Zellen Parametern, aber noch wichtiger ist, die Inter Zelle Variabilität über eine große Population von Zellen.
Protokoll
(1) die Überwachung Erwerb Zellkultur
- HeLa-Zellen in DMEM + Glutamin (z.B.GlutaMAX) wachsen Medium mit 10 % (V/V) Hitze-inaktivierten fetalen Kalb Serum und 1 % Penicillin und Streptomycin ergänzt.
- 6-Well unten Kultur Glasplatten mit Fibronektin (25 µg/mL) für 1 h zu beschichten. Dann Samen Sie 2 x 104 Zellen pro Bohrloch.
- Schon beim Erwerb ändern Sie des Mediums alle 3 Tage.
- Verwenden Sie für den Zeitraffer Erwerb der Objektiv-freie Videomikroskop (im Handel erhältlich).
Hinweis: Dieses Objektiv-freie rechnerische bildgebendes Verfahren basiert auf wie von Ozcan Et Al. beschrieben 16 die auszuführenden kontinuierliche Überwachung in einem Inkubator bei einer kontrollierten Temperatur von 37 ° C12,15geändert wurde. Freuen Sie sich auf einen komplementäre Metalloxid-Halbleiter (CMOS)-Bild-Sensor mit einem Pixelabstand von 1,67 µm und eine imaging-Bereich von 6,4 x 4,6 mm2. Mehreren Wellenlänge Beleuchtung bietet eine multichip lichtemittierende Diode (LED) Vorrichtung, die roten, grünen und blauen Beleuchtung liefert. Die Wellenlängen sind zentriert, 636, 521 und 452 nm bzw. mit einer spektralen Bandbreite von 25, 45 und 25 nm bzw.. Die rot-grün-blau (RGB) LEDs befinden sich oberhalb einer 150 µm Lochkamera in einem Abstand von ca. 5 cm aus den Zellen. Die Lichtleistung gemessen in der Nähe der LED ist so niedrig wie 10 µW bei jeder Wellenlänge und die Leuchtdauer beträgt nur eine Sekunde pro Akquisition. Daher dürften keine Phototoxizität Probleme bei Zellkulturen unter dem Objektiv-freie video Mikroskop beobachtet werden. - Stellen Sie die Zelle-Kultur-Behälter setzen Sie in Kontakt mit dem CMOS-Sensor (Abbildung 1). Verwenden Sie einen Behälter mit einem Glas Deckgläschen an der Unterseite, die wichtig für die Qualität der Linse-freie Übernahme ist. Kunststoff-Behälter verwendet werden, aber die Objektiv-freie Übernahme wird aufgrund der schlechten optischen Qualität dieser Container abgebaut werden.
- Steuern Sie die Objektiv-freie Videomikroskop mit der Datenerfassungs-Software (im Handel erhältlich), die die Zeitraffer-Akquisition und der holographischen Rekonstruktion durchführt. Die Parameter, die vom Benutzer in der Softwareoberfläche eingegeben werden müssen sind die Frame-Rate, die Dauer des Experiments und die Art der Zellkultur, d.h. adhärente Zellen oder schwimmenden Zellen.
- Legen Sie die Eingabeparameter der Datenerfassungs-Software. Legen Sie die Frame-Rate auf einen Erwerb alle 10 Minuten und Kultur Zelltyp zu "Anhänger" eingestellt.
Hinweis: Eine Bildrate von einem Erwerb alle 10 Minuten ist ein guter Kompromiss, denn es eine zuverlässige Zelle verfolgen gewährleistet, während die Größe des resultierenden Datasets zu analysierenden noch zumutbar ist. Die schnellste Frame-Rate erreichen mit diesem Objektiv-freie Mikroskopie-Setup ist ein Erwerb alle 5 Minuten. Weitere Steigerung der Framerate führt zu übermäßiger Erwärmung des CMOS-Sensors, die die Lebensfähigkeit der Zellen in Kultur beeinflussen können. - Die Time-Lapse Übernahme starten, und die holographische Rekonstruktionsalgorithmus (im Handel erhältlich) wird automatisch verarbeiten die Objektiv-freie Übernahme um die Phase Bild der Zellkultur zu erhalten. Der holographischen Rekonstruktionsalgorithmus basiert auf einer Multi-Wellenlänge Phase Retrieval Algorithmus17 und bietet ein RGB-Bild rekonstruierten Phase im Bereich von [-π, + π] für jede einzelne Zelle über ein großes Sehfeld von 29,4 mm2. Das Prinzip der Objektiv-freie Inline-Holographie ist eine dünne Probe zu beleuchten (bei Z = 0) mit einer Ebenen Welle (von Amplitude 1 nach Normalisierung) und auf einem CMOS-Sensor erkennt das anschließende Beugung Intensität Bild bei einem kurzen Abstand Z = Z, in der Regel 1 mm nach der Probe. In unserem Setup wird die Beleuchtung auf 3 Wellenlängen sequenziell geschaltet (λ1= 0.450 µm, λ2= 0.540 µm, λ3= 0.647 µm), drei Beugungsmuster der gleichen Probe zu messen.
(2) Zelle Kultur Datenanalyse
- Verwenden Sie für Handy-Tracking Trackmate Algorithmus ", ein open-Source-Fidschi-Plugin für das automatisierte Tracking Einzelkörner18". At zuerst laden die volle Zeitraffer Übernahme in Fidschi (Load-Bild-Sequenz-Befehl). Aufgrund der Größe der Datensätze, die in der Regel 400 RGB-Bilder von 29,7 Mb verfügt, ist es schneller sie in 8-Bit-Format laden. Als nächstes führt das Trackmate Fidschi-Plugin den Anwender durch mehrere Phasen des Handy-Tracking-Algorithmus, nämlich eine Erkennung-Zell-Stadium, ein Tracker-Zell-Stadium und mehrere Filter angewendet, um die Zelle Erkennungen und die berechneten Bahnen. In jeder Phase die Algorithmen zu konfigurieren und die Ergebnisse anzeigen sofort damit, falls erforderlich, eine leicht hin und her navigieren kann, passen Sie die Einstellungen. Die verschiedenen Menüs der Trackmate Schnittstelle sind mit den aktuellen Einstellungen für die HeLa-Zellen-Experiment in Abbildung 2 dargestellt.
- Legen Sie die Eingabeparameter der Datenerfassungs-Software wie folgt (siehe Abbildung 2). Der geschätzte Blob Durchmesser 15 Pixel, die Detektor-Schwelle 0,25, verbindet maximale Entfernung auf 15 Pixel, die Lücke schließen max. Entfernung auf 15 Pixel und die Anzahl der Plätze in den Spuren auf 3,5 einstellen
Hinweis: Diese Einstellungen werden offensichtlich unterscheiden sich von einem Experiment zur anderen, vor allem der "geschätzte Blob Durchmesser", das entspricht der Größe der Zellen (hier in Pixel von 1,67 µm angegeben). Die gleiche Bemerkung bezieht sich auf die "Verknüpfung max zurückgelegte" welches Konto für die maximale Entfernung von einer Zelle in zwei aufeinander folgenden Frames (hier in Pixel von 1,67 µm angegeben). Der optimale Wert würde auf Zelle Motilität, sondern auch auf Zelldichte abhängen. - Am Ende des Prozesses Handy-Tracking, generieren die Ergebnisse in Form von drei Textdateien ("Analyse" Taste, siehe Abbildung 2). Die nützlichste Datei mit dem Namen "Spots in Spuren Statistik", besteht aus einer Tabelle alle erkannte Zellen mit ihren jeweiligen (x, y) Positionen in der Akquisition, ihre Frame-Nummer und die Track-Nummer. Auf Basis dieser Daten durchzuführen Sie Zelle Segmentierung und erkennen Sie Zellteilungen mit speziellen Algorithmen (siehe ergänzende Codedateien) zu. Die beiden anderen Dateien heißen "Links in der Titel-Statistik" und "Track Statistiken" und enthalten die Ergebnisse, die der Umgang mit den Spuren, z.B. Track Dauer, Anzahl der erkannten Lücken, erste Frame usw. zu verfolgen.
- Verwenden Sie die Zelle Segmentierungsalgorithmus (siehe ergänzende Codedatei), mehrere Kennzahlen beschreiben die Zellmorphologie aus dem rekonstruierten Phase Bild automatisch zu extrahieren. Diese Metriken sind die Zelle Oberfläche (S), die Zelle Hauptachse Länge (L), die Zelle Nebenachse Länge (l) und der Zelle Aspect Ratio (L/l). Die Phase von Objektiv-freie Mikroskopie erholt ist proportional zur Dichte und Dicke der Probe wie zuvor15besprochen.
- Neben den morphologischen Eigenschaften der Zelle quantitative Zelle trockenere Massenmessungen (CDM) Auszug aus der rekonstruierten Phase13,19,20
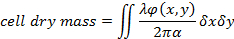 GL. (1)
GL. (1)
wo φ(x,y) ist das rekonstruierte Phase Shift19, λ die Wellenlänge und α = 1,8 x 10-4 m3/kg der spezifischen Brechungsindex steht die Variation der Brechungsindex Masse1, trocken 19. - Die Zelle Dicke über eine Schätzung der Differenz zwischen der Zelle Brechungsindex und derjenigen der Nährmedien zu bewerten. Das Verhältnis zwischen der Zelle Dicke h(X,y) und die gemessene Phasenverschiebung wird durch20gegeben:
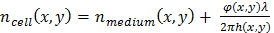 GL. (2.1)
GL. (2.1) GL. (2.2)
GL. (2.2)
mit Δn(X,y) = nZelle (X,y) - nMedium (X,y), der Brechungsindex Unterschied zwischen der Zelle und die Nährmedien. Im folgenden davon ausgehen einen konstanten Wert von Δn = 0,025, die Schätzungen von dem Brechungsindex gemessen in HeLa Zellkerne (n = 1.35511) und Phosphat-gepufferte Kochsalzlösung (PBS) Kultur Medien (n = 1,33). Obwohl der Wert der Zelle Dicke nur Richtwerte, da es auf der Annahme beruht, seine relative Variationen sind aussagekräftig und können genutzt werden, um teilenden Zellen zu erkennen. - Algorithmus "engagierten" verwenden (siehe ergänzende Codedatei) das Auftreten von Zellteilungen zu erkennen und dann extrahieren Zelle Spuren, die die erkannten Zellteilungen zugeordnet sind, um eine Zellteilung zu erkennen-Zellen mit einer gemessenen Dicke größer als 8 µm werden zuerst identifiziert, dann prüfen Sie ob eine neue Zelle erscheint eng in Raum und Zeit. Auf diese Weise setzt der Nachweis von Zellteilungen auf zwei robuste Kriterien. Alternative und aufwändigere Methoden zur Erkennung der Zellteilung können auf Lensfree Zeitraffer Erwerb21,22,23angewendet werden.
Ergebnisse
Für die holografische Wiederaufbauprozess Lichtfeldes anhand eines skalaren Feldes pro (wo 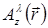 der komplexen Wert von A in das Flugzeug im Abstand Z von der Probe und seitliche Position
der komplexen Wert von A in das Flugzeug im Abstand Z von der Probe und seitliche Position 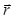 und Wellenlänge λ). Lichtausbreitung wird durch die Huygens-Fresnel-Theorie stellt einen Verbreiter Kernel modelliert
und Wellenlänge λ). Lichtausbreitung wird durch die Huygens-Fresnel-Theorie stellt einen Verbreiter Kernel modelliert
Diskussion
In diesem Beitrag zeigen wir, dass dieses Objektiv-freie Videomikroskopie in einer Brutmaschine verwendet werden kann, um die Kinetik von Tausenden von Zellen zu erfassen. Um zu beschreiben, der Gesamtmethodik haben wir erklärt, wie eine 2,7 Tage Zeitraffer Übernahme von HeLa-Zellen in Kultur mit standard Handy-Tracking-Algorithmen analysiert werden kann. Das Ergebnis ist ein Dataset mit 2,2 x 106 Zelle Messungen und 10.584 Zelle Radwege. Die Akquisitionen wurden durchgeführt, auf eine Kultur der Hela-Zelle...
Offenlegungen
Die Autoren haben nichts preisgeben.
Danksagungen
Die Autoren haben nichts zu erkennen.
Materialien
| Name | Company | Catalog Number | Comments |
| Cytonote lens-free video microscope | Iprasense | ||
| Horus acquisition software | Iprasense | ||
| 6-well glass bottom culture plates | MatTek corporation | Part No: P06G-0-14-F | |
| DMEM + GlutaMAX medium | Gibco | ||
| heat-inactivated fetal calf serum | Eurobio | ||
| penicillin and streptomycin | Gibco | ||
| Fibronectin | Sigma Aldrich | ||
| Matlab, image processing toolbox | Mathworks |
Referenzen
- Zangle, T. A., Teitell, M. A. Live-cell mass profiling: an emerging approach in quantitative biophysics. Nat Methods. 11 (12), 1221-1228 (2014).
- Popescu, G., Park, K., Mir, M., Bashir, R. New technologies for measuring single cell mass. Lab Chip. 14 (4), 646-652 (2014).
- Reed, J., et al. Rapid, massively parallel single-cell drug response measurements via live cell interferometry. Biophys J. 101 (5), 1025-1031 (2011).
- Mir, M., et al. Optical measurement of cycle-dependent cell growth. Proc Natl Acad Sci. USA. 108 (32), 13124-13129 (2011).
- Girshovitz, P., Shaked, N. T. Generalized cell morphological parameters based on interferometric phase microscopy and their application to cell life cycle characterization. Biomed Opt Express. 3 (8), 1757-1773 (2012).
- Kemper, B., Bauwens, A., Vollmer, A., Ketelhut, S., Langehanenberg, P. Label-free quantitative cell division monitoring of endothelial cells by digital holographic microscopy. J Biomed Opt. 15 (3), (2010).
- Mir, M., Bergamaschi, A., Katzenellenbogen, B. S., Popescu, G. Highly sensitive quantitative imaging for monitoring single cancer cell growth kinetics and drug response. PLoS One. 9 (2), 1-8 (2014).
- Bon, P., Savatier, J., Merlin, M., Wattellier, B., Monneret, S. Optical detection and measurement of living cell morphometric features with single-shot quantitative phase microscopy. J Biomed Opt. 17 (7), (2012).
- Aknoun, S., et al. Living cell dry mass measurement usinq quantitative phase imaging with quadriwave lateral shearing interferometry: an accuracy and sensitivity discussion. J Biomed Opt. 20 (1), 1-4 (2015).
- Cotte, Y., et al. Marker-free phase nanoscopy. Nature Photonics. 7 (2), 113-117 (2013).
- Choi, W., et al. Tomographic phase microscopy. Nat Methods. 4 (9), 717-719 (2007).
- Kesavan, S. V., et al. High-throughput monitoring of major cell functions by means of lensfree video microscopy. Sci Rep. 4, 1-11 (2014).
- Zheng, G., Lee, S. A., Antebi, Y., Elowitz, M. B., Yang, C. The ePetri dish, an on-chip cell imaging platform based on subpixel perspective sweeping microscopy (SPSM). Proc Natl Acad Sci USA. 108 (41), 16889-16894 (2011).
- Pushkarsky, I., et al. Automated single-cell motility analysis on a chip using lensfree microscopy. Sci Rep. 4, 4717 (2014).
- Allier, C., et al. Imaging of dense cell cultures by multiwavelength lens-free video microscopy. Cytom Part A. 91 (5), 1-10 (2017).
- Su, T. -. W., Seo, S., Erlinger, A., Ozcan, A. High-throughput lensfree imaging and characterization of a heterogeneous cell solution on a chip. Biotechnol Bioeng. 102 (3), 856-868 (2009).
- Delacroix, R., et al. Cerebrospinal fluid lens-free microscopy: a new tool for the laboratory diagnosis of meningitis. Sci Rep. 7, 39893 (2017).
- Tinevez, J. -. Y., et al. TrackMate: an open and extensible platform for single-particle tracking. Methods. 115, 80-90 (2016).
- Popescu, G. Optical imaging of cell mass and growth dynamics. Am J Physiol Physiol. 295 (2), 538-544 (2008).
- Liu, P. Y., et al. Cell refractive index for cell biology and disease diagnosis: past, present and future. Lab Chip. 16, 634-644 (2016).
- Rapoport, D. H., Becker, T., Mamlouk, A. M., Schicktanz, S., Kruse, C. A novel validation algorithm allows for automated cell tracking and the extraction of biologically meaningful parameters. PLoS One. 6 (11), e27315 (2011).
- Al-Kofahi, O., et al. Automated cell lineage construction: A rapid method to analyze clonal development established with murine neural progenitor cells. Cell Cycle. 5 (3), 327-335 (2006).
- Meijering, E., Dzyubachyk, O., Smal, I., van Cappellen, W. A. Tracking in cell and developmental biology. Semin Cell Dev Biol. 20 (8), 894-902 (2009).
- Posakony, J. W., England, J. M., Attardi, G. Mitochondrial growth and division during the cell cycle in HeLa cells. J Cell Biol. 74 (2), 468-491 (1977).
- Zocchi, E., et al. Expression of CD38 Increases Intracellular Calcium Concentration and Reduces Doubling Time in HeLa and 3T3 Cells. J Biol Chem. 273 (14), 8017-8024 (1979).
- Reitzer, L. J., Wice, B. M., Kennell, D. Evidence that glutamine, not sugar, is the major energy source for cultured HeLa cells. J Biol Chem. 254 (8), 2669-2676 (1979).
- Benedetti, A. D. E., Joshi-barve, S., Rinker-Schaeffer, C., Rhoads, R. E. Expression of Antisense RNA against Initiation Factor eIF-4E mRNA in HeLa Cells Results in Lengthened Cell Division Times, Diminished Translation Rates, and Reduced Levels of Both eIF-4E and the p220 Component of eIF-4F. Mol Cell Biol. 11 (11), 5435-5445 (1991).
- Kumei, Y., Nakajima, T., Sato, A., Kamata, N., Enomoto, S. Reduction of G1 phase duration and enhancement of c-myc gene expression in HeLa cells at hypergravity. J Cell Sci. 93 (2), 221-226 (1989).
- Ginzberg, M. B., Kafri, R., Kirschner, M. On being the right (cell) size. Science. 348 (6236), 1245075 (2015).
- Mathieu, E., et al. Time-lapse lens-free imaging of cell migration in diverse physical microenvironments. Lab Chip. 16 (17), 3304-3316 (2016).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten