A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
ללא עדשה מיקרוסקופ וידאו עבור הדינמיקה וניתוח כמותי של תרבית תאים חסיד
In This Article
Summary
וידאו ללא עדשה מיקרוסקופ מאפשר לנו לעקוב אחר תרביות תאים ישירות בתוך החממה. כאן נתאר את פרוטוקול מלא נהגה לרכוש וניתוח של 2.7 היום רכישת בתרבית תאים הלה, שמוביל dataset של 2.2 x 10 שירים6 מדידות של תא בודד ומורפולוגיה 10584 מחזור התא.
Abstract
כאן, נדגים את הסרטון ללא עדשה מיקרוסקופ מאפשרת לנו לתפוס בו זמנית את קינטיקה של אלפי תאים ישירות בתוך החממה וכי ניתן לפקח ולכמת תאים בודדים לאורך כמה מחזורים התא. אנו מתארים פרוטוקול מלא שמשמש כדי לעקוב אחר ולכמת תרבות הלה תא ימים 2.7. ראשית, תא תרבות הרכישה מתבצע באמצעות מיקרוסקופ וידאו ללא עדשה, ולאחר מכן הנתונים זה נותחו בעקבות תהליך ארבעה שלבים: גל מרובה שיקום הולוגרפי, תא-מעקב, תא זיהוי פילוח, חלוקת התא אלגוריתמים. כתוצאה מכך, אנו מראים שאפשר לאסוף של הנתונים (dataset) הכולל יותר מ 10,000 מחזור התא מסלולים ומדידות יותר מ 2 x 106 תאים מורפולוגי.
Introduction
בתרבית של תאים בתרבית במשך כמה מחזורים תא ומדידת במדויק תא וגודל תא יבש הוא משימה מאתגרת. מספר טכניקות ללא תווית אופטי מסוגלים לבצע את המשימה1,2: שלב-shifting אינטרפרומטריה3, מיקרוסקופ דיגיטלי הולוגרפי (סוג התושבות שלהם. מופיעים)4,5,6, 7, quadriwave לרוחב הטיה אינטרפרומטריה8,9 ו כמותיים שלב טומוגרפיה10,11. שיטות אלה הובילו תובנות חדשות רבות ההבנה של מחזור התא של תאי יונקים. אולם הם נמצאים לעתים רחוקות ביחד עם תא אוטומטי מעקב אלגוריתמים התפוקה שלהם נותר מוגבל כאשר מודדים את התא מסלולים מסה1 (N < 20 בהתאמה3,4,5 , 6). לפיכך שיטה אופטי יש צורך למדוד את התא מסלולים המוני עם סטטיסטיקה גדולים (N > 1000).
בנייר זה, נדגים את היכולת של וידאו ללא עדשה מיקרוסקופ בו-זמנית תמונה אלפי תאים ישירות בתוך החממה ולאחר מכן לכמת תא בודד מדדים לאורך אלפי שירים בודדים מחזור התא. ללא עדשה מיקרוסקופ הוא שלב כמותיים הדמיה טכניקה המאפשרת רכישת תמונת שלב של תאים בצפיפות מעל שדה ראייה גדול מאוד (בדרך כלל כמה עשרות של מ מ2, כאן 29.4 מ מ2)12,13 14, ,15. במספר מדדים ברמת תא בודד נקבע, למשל, תא שטח, תא בנפח גדול יבש, עובי התא, אורך ציר מרכזי תא ותא יחס גובה-רוחב12,15, של כל תמונה. לאחר מכן, על-ידי החלת אלגוריתם תא-מעקב, תכונות אלה יכולים להיות מותווים עבור כל תא ותא כפונקציה של ניסוי זמן14,15. יתר על כן, על ידי גילוי המופע של חלוקות תאים על הפסים תא, זה אפשרי לחלץ מידע חשוב אחר כגון התא הראשוני יבש בנפח גדול (רק לאחר חלוקת התא), המסה יבשות תא הסופי (ממש לפני חלוקת התא), התא מחזור משך, כלומר, הזמן בין שתי חטיבות ברציפות15. כל המדידות הללו יכול להיות מחושב עם סטטיסטיקה טובה מאוד (N > 1000) מאז שדה הראייה גדול יאפשר בדרך כלל הניתוח של 200 עד 10,000 תאים ברכישת יחידה ללא עדשה.
על מנת להסביר מתודולוגיה זו מבוסס על וידאו ללא עדשה מיקרוסקופ, נתאר את פרוטוקול כדי לפקח, לכמת תרבות הלה תא ימים 2.7. ניתוח נתונים הוא תהליך ארבעה שלבים בהתבסס על שחזור הולוגרפית גל רב, תא-מעקב, פילוח תא ואלגוריתמים חלוקת התא. כאן הוא הראה כי הרזולוציה המרחבית, את קצב המסגרות מהיר יחסית (רכישה אחת כל 10 דקות) שהושג עם תוכנית התקנה זו וידאו ללא עדשה מיקרוסקופ הוא תואם עם אלגוריתמים תא-מעקב רגיל. ניתוח מלא של ערכת נתונים זה תוצאות המדידה של רצועות תא 10,584 על מחזורי תא שלם.
לסיכום, וידאו ללא עדשה מיקרוסקופ הוא כלי רב עוצמה כדי לפקח באופן אוטומטי אלפי ללא תווית, שאינו מסונכרן ותאים שלא שונתה לכל ניסוי; כל תא במעקב במשך כמה מחזורים התא. המידות שלנו ובכך לספק את הערך הממוצע של מספר פרמטרים תא, אבל יותר חשוב, ההשתנות הבין-תאית על אוכלוסיה גדולה של תאים.
Protocol
1. תרבית תאים פיקוח על רכישת
- לגדול הלה בתאים DMEM + גלוטמין (למשל, GlutaMAX) בינוני בתוספת 10% (v/v) חום-לא פעיל עגל עוברית סרום ו 1% פניצילין, סטרפטומיצין.
- מעיל טוב 6 זכוכית תרבות לוחות עם fibronectin (25 µg/mL) לשעה. ואז זרע 2 x 10 תאים4 לכל טוב.
- במהלך רכישת, לשנות את המדיום כל 3 ימים.
- עבור רכישת זמן לשגות, השתמש וידאו ללא עדשה מיקרוסקופ (זמינים מסחרית).
הערה: זה מבוסס על טכניקת דימות ללא עדשה חישובית כפי שתואר על ידי. Ozcan et al. 16 אשר שונתה כדי לבצע ניטור רציף באינקובטור בטמפרטורה מבוקרת של 37 ° C12,15. הוא כולל חיישן התמונה משלימים תחמוצת מתכת מוליכים למחצה (CMOS) עם זריקה פיקסל של מיקרומטר 1.67 ואזור הדמיה של 6.4 x 4.6 מ מ2. תאורה גל מרובים מסופק על ידי multichip אור פולטות דיודות (נוריות) התקן, אשר מספק תאורה אדום, ירוק וכחול. אורכי הגל מרוכזים, על 636, 521, 452 nm בהתאמה, עם רוחב הפס ספקטרלי של 25, 45 ו- 25 ננומטר בהתאמה. אדום-ירוק-כחול (RGB) הנוריות ממוקמים מעל 150 הקדמוניות מיקרומטר ממרחק של 5 ס מ התאים. הכוח אור נמדד בקרבת ה-LED הוא נמוך כמו 10 µW-כל גל והוא הזמן תאורה רק שנייה אחת לכל רכישה. לפיכך, אין בעיות רעילות צילום צפויים כאשר התא תרבויות מובחנות תחת מיקרוסקופ וידאו ללא עדשה. - לשים את המיכל תרבות תא לשים בקשר עם חיישן CMOS (איור 1). השתמש מיכל עם coverslip זכוכית בתחתית, וזה חשוב על האיכות של הרכישה ללא עדשה. מיכלי פלסטיק יכול לשמש גם אבל הרכישה ללא עדשה תגרע בשל איכות אופטית ירודה של גורמים מכילים אלה.
- בקרת וידאו ללא עדשה מיקרוסקופ עם רכישת התוכנה (זמינים מסחרית), אשר מבצעת את רכישת זמן לשגות והן על שחזור הולוגרפי. הפרמטרים שצריכים להיות מוזנים על ידי המשתמש בממשק התוכנה הן קצב המסגרות, משך הזמן של הניסוי, ואת הסוג של תרבית תאים, תאים חסיד דהיינו או תאים צף.
- הגדר את פרמטרי קלט של רכישת התוכנה. הגדר את קצב המסגרות מוגדר אחד רכישת כל 10 דקות, להגדיר את סוג התרבות התא 'חסיד'.
הערה: קצב רכישת אחד כל 10 דקות היא פשרה טובה שהיא מבטיחה תא אמין מעקב בעוד גודל ערכת הנתונים וכתוצאה מכך שינותח הוא עדיין סביר. קצב המסגרות המהירה השגה עם תוכנית התקנה זו ללא עדשה מיקרוסקופ הוא אחד רכישת כל 5 דקות. להגדיל עוד יותר את קצב המסגרות תוצאות חימום יתר את חיישן CMOS אשר יכול להשפיע על הכדאיות של התאים בתרבות. - להתחיל את רכישת זמן לשגות, האלגוריתם שחזור הולוגרפי (זמינים מסחרית) יעבד באופן אוטומטי את הרכישה ללא עדשה כדי לקבל את התמונה שלב של התרבות תאים. האלגוריתם שחזור הולוגרפי מבוסס על אלגוריתם אחזור מרובת גל שלב17 והוא מספק תמונת שלב המשוחזרת RGB בטווח של [-π, + π] עבור כל תא יחיד מעל שדה ראייה גדול של 29.4 מ מ2. העיקרון של הולוגרפיה ללא עדשה בשורה הוא להאיר מדגם דק (ב- z = 0) עם גל מישורי (של משרעת 1 לאחר נורמליזציה) וכדי לזהות על חיישן CMOS התמונה בעוצמה עקיפה עוקבות במרחק קצר z = Z, בדרך כלל 1 מ מ לאחר לדוגמה. בהגדרת שלנו, ההארה הוא החליף ברצף ל 3 אורכי גל (λ1= מיקרומטר 0.450, λ2= 0.540 מיקרומטר, λ3= מיקרומטר 0.647) כדי למדוד את שלוש תבניות עקיפה באותה דגימת זרע.
2. התא תרבות ניתוח נתונים
- לשימוש התא-מעקב, האלגוריתם Trackmate, תוסף פיג'י קוד פתוח למעקב אוטומטי של חלקיקים יחיד18. At טען תחילה, רכישה מלא זמן לשגות פיג'י (טעינת התמונה רצף הפקודה). בשל גודלו של קבצי הנתונים, הכולל בדרך כלל 400 RGB מסגרות של 29.7 Mb, זה מהר יותר כדי לטעון אותם בתבנית 8 סיביות. הבא התוסף Trackmate פיג'י המדריכים למשתמש דרך מספר שלבים של האלגוריתם תא-מעקב, כלומר שלב זיהוי תא, שלב המעקב תא ו מספר מסננים המוחלים על גילויים של התא, המסילה מחושב. בכל שלב, להגדיר את האלגוריתמים ולהציג את התוצאות באופן מיידי כך, במידת הצורך, אחד יכול בקלות לנווט הלוך ושוב כדי להתאים מחדש את ההגדרות. התפריטים השונים של הממשק Trackmate מוצגים באיור 2 עם ההגדרות בפועל משמש הניסוי תאים הלה.
- הגדר את פרמטרי קלט של התוכנה רכישה בעקבות (ראה איור 2). הגדר את קוטר בועה משוער 15 פיקסלים, הסף גלאי כדי 0.25, המרחק המקסימלי המקשרים 15 פיקסלים, המרחק מקסימום הפער סגירה 15 פיקסלים, ואת מספר המקומות במסלולים ל 3.5.
הערה: הגדרות אלה יהיו שונים מן הסתם מהניסוי אחד למשנהו, במיוחד את 'בועה מוערך קוטר' התואם לגודל התא (מוענקת כאן בפיקסלים של 1.67 מיקרומטר). ההערה באותו חל "המקשרים מקסימום מרחק' באיזה חשבון עבור המרחק המקסימלי על ידי תא בתוך שתי מסגרות רצופים (מוענקת כאן בפיקסלים של 1.67 מיקרומטר). הערך האופטימלי שלו תלוי על תנועתיות תא אלא גם על צפיפות התאים. - בסופו של תהליך מעקב תא, להפיק את התוצאות בצורה של שלושה קבצי טקסט ('ניתוח' כפתור, ראה איור 2). הקובץ השימושי ביותר, בשם 'כתמים בסטטיסטיקה רצועות', מכיל טבלה לרישום תאים כל שזוהו עם העמדות שלהם בהתאמה (x, y) הרכישה, מספר מסגרת שלהם, את מספר המסלול שלהם. על בסיס נתונים אלה, לבצע פילוח תא ולגלות חלוקות תאים באמצעות אלגוריתמים ייעודי (ראה קובצי קוד משלים). שני הקבצים האחרים נקראים 'קישורים בסטטיסטיקה רצועות' ו 'סטטיסטיקת מעקב', כוללים את כל התוצאות התמודדות עם רצועות, למשל מסלול המשכים, מספר פערים שאותרו, לעקוב אחר מסגרת ראשונית, וכו '.
- השתמש האלגוריתם פילוח תא (ראה קובץ קוד משלים) באופן אוטומטי לחלץ במספר מדדים המתארים המורפולוגיה תא מהתמונה שלב המשוחזרת. מדדים אלה הם שטח הפנים של התא (S), אורך ציר מרכזי תא (L), אורך ציר קטין תאים (l), יחס הגובה-רוחב התא (L/l). השלב התאוששה ללא עדשה מיקרוסקופ הוא יחסי צפיפות, עובי השכבה הדגימה כפי שפורט קודם לכן15.
- בנוסף לתכונות מורפולוגי של התא, לחלץ מדידות כמותיים תא יבש (CDM) מסת מ ה19,13,המשוחזרת שלב20
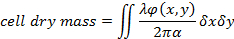 הציוד (1)
הציוד (1)
כאשר φ(x,y) הוא שלב המשוחזרת shift19, λ אורך הגל וα = 1.8 x-4 10 מ'3/ק ג שבירה מסוים אשר מגולל את הווריאציה של שבירה להתייבש מסה1, 19. - להעריך את עובי התא באמצעות הערכת ההבדל בין תא את מקדם שבירה של תרבות התקשורת. היחס בין את התא עובי h(x,y) משמרת שלב נמדד ניתנת על ידי20:
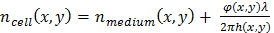 הציוד (2.1)
הציוד (2.1) הציוד (2.2)
הציוד (2.2)
עם Δn(x,y) = nתא (x,y) - nבינוני (x,y), ההבדל שבירה בין התא לבין התקשורת תרבות. בחלק זה, להניח ערך קבוע של Δn = 0.025, אשר מוערך מהאינדקס השבירה נמדד גרעין התא הלה (n = 1.35511) וזו של מדיה תרבות באגירה פוספט תמיסת מלח (PBS) (n = 1.33). למרות הערך עובי התא הוא רק מעיד ככל שהיא מבוססת על ההנחה, הווריאציות היחסי שלו משמעותיים, אפשר לנצל כדי לזהות תאים החלוקה. - השתמש האלגוריתם ייעודי (ראה קובץ קוד משלים) כדי לזהות את המופע של חלוקות תאים ולאחר מכן לחלץ רצועות תא שאינן משוייך לחטיבות תא שאותרו, כדי לזהות את חלוקת התא, תאים עם עובי נמדד שגודלם 8 מיקרומטר לראשונה ניתן לזהות, ולאחר מכן בדוק אם תא חדש תופיע באופן הדוק שהמרחב והזמן. בדרך זו, הגילוי של חלוקות תאים מסתמך על שני קריטריונים חזקים. ניתן להחיל חלופה ושיטות משוכלל יותר לגילוי חלוקת התא עד22,2321,רכישת זמן לשגות lensfree.
תוצאות
עבור תהליך שחזור הולוגרפי, השדה אור מתואר על ידי שדה סקלרי (איפה 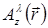 הוא הערך מורכבים של A על המטוס על מרחק z מהמיקום דוגמה ולאחר לרוחב
הוא הערך מורכבים של A על המטוס על מרחק z מהמיקום דוגמה ולאחר לרוחב 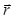 ו- גל λ). הפצת האור הוא עוצב בידי בתיאוריה Huygens-פרנל אש...
ו- גל λ). הפצת האור הוא עוצב בידי בתיאוריה Huygens-פרנל אש...
Discussion
בנייר זה, אנו מראים שאת מיקרוסקופ וידאו ללא עדשה יכול לשמש בתוך אינקובטור כדי ללכוד את קינטיקה של אלפי תאים. על מנת לתאר את המתודולוגיה הכללית הסברנו כיצד ניתן לנתח של רכישת זמן לשגות היום 2.7 של הלה תאים בתרבות עם אלגוריתמים תא-מעקב רגיל. התוצאה היא dataset שמציעות 2.2 x 10 מדידות תא6 רצועות מ...
Disclosures
המחברים אין לחשוף.
Acknowledgements
המחברים אין להכיר.
Materials
| Name | Company | Catalog Number | Comments |
| Cytonote lens-free video microscope | Iprasense | ||
| Horus acquisition software | Iprasense | ||
| 6-well glass bottom culture plates | MatTek corporation | Part No: P06G-0-14-F | |
| DMEM + GlutaMAX medium | Gibco | ||
| heat-inactivated fetal calf serum | Eurobio | ||
| penicillin and streptomycin | Gibco | ||
| Fibronectin | Sigma Aldrich | ||
| Matlab, image processing toolbox | Mathworks |
References
- Zangle, T. A., Teitell, M. A. Live-cell mass profiling: an emerging approach in quantitative biophysics. Nat Methods. 11 (12), 1221-1228 (2014).
- Popescu, G., Park, K., Mir, M., Bashir, R. New technologies for measuring single cell mass. Lab Chip. 14 (4), 646-652 (2014).
- Reed, J., et al. Rapid, massively parallel single-cell drug response measurements via live cell interferometry. Biophys J. 101 (5), 1025-1031 (2011).
- Mir, M., et al. Optical measurement of cycle-dependent cell growth. Proc Natl Acad Sci. USA. 108 (32), 13124-13129 (2011).
- Girshovitz, P., Shaked, N. T. Generalized cell morphological parameters based on interferometric phase microscopy and their application to cell life cycle characterization. Biomed Opt Express. 3 (8), 1757-1773 (2012).
- Kemper, B., Bauwens, A., Vollmer, A., Ketelhut, S., Langehanenberg, P. Label-free quantitative cell division monitoring of endothelial cells by digital holographic microscopy. J Biomed Opt. 15 (3), (2010).
- Mir, M., Bergamaschi, A., Katzenellenbogen, B. S., Popescu, G. Highly sensitive quantitative imaging for monitoring single cancer cell growth kinetics and drug response. PLoS One. 9 (2), 1-8 (2014).
- Bon, P., Savatier, J., Merlin, M., Wattellier, B., Monneret, S. Optical detection and measurement of living cell morphometric features with single-shot quantitative phase microscopy. J Biomed Opt. 17 (7), (2012).
- Aknoun, S., et al. Living cell dry mass measurement usinq quantitative phase imaging with quadriwave lateral shearing interferometry: an accuracy and sensitivity discussion. J Biomed Opt. 20 (1), 1-4 (2015).
- Cotte, Y., et al. Marker-free phase nanoscopy. Nature Photonics. 7 (2), 113-117 (2013).
- Choi, W., et al. Tomographic phase microscopy. Nat Methods. 4 (9), 717-719 (2007).
- Kesavan, S. V., et al. High-throughput monitoring of major cell functions by means of lensfree video microscopy. Sci Rep. 4, 1-11 (2014).
- Zheng, G., Lee, S. A., Antebi, Y., Elowitz, M. B., Yang, C. The ePetri dish, an on-chip cell imaging platform based on subpixel perspective sweeping microscopy (SPSM). Proc Natl Acad Sci USA. 108 (41), 16889-16894 (2011).
- Pushkarsky, I., et al. Automated single-cell motility analysis on a chip using lensfree microscopy. Sci Rep. 4, 4717 (2014).
- Allier, C., et al. Imaging of dense cell cultures by multiwavelength lens-free video microscopy. Cytom Part A. 91 (5), 1-10 (2017).
- Su, T. -. W., Seo, S., Erlinger, A., Ozcan, A. High-throughput lensfree imaging and characterization of a heterogeneous cell solution on a chip. Biotechnol Bioeng. 102 (3), 856-868 (2009).
- Delacroix, R., et al. Cerebrospinal fluid lens-free microscopy: a new tool for the laboratory diagnosis of meningitis. Sci Rep. 7, 39893 (2017).
- Tinevez, J. -. Y., et al. TrackMate: an open and extensible platform for single-particle tracking. Methods. 115, 80-90 (2016).
- Popescu, G. Optical imaging of cell mass and growth dynamics. Am J Physiol Physiol. 295 (2), 538-544 (2008).
- Liu, P. Y., et al. Cell refractive index for cell biology and disease diagnosis: past, present and future. Lab Chip. 16, 634-644 (2016).
- Rapoport, D. H., Becker, T., Mamlouk, A. M., Schicktanz, S., Kruse, C. A novel validation algorithm allows for automated cell tracking and the extraction of biologically meaningful parameters. PLoS One. 6 (11), e27315 (2011).
- Al-Kofahi, O., et al. Automated cell lineage construction: A rapid method to analyze clonal development established with murine neural progenitor cells. Cell Cycle. 5 (3), 327-335 (2006).
- Meijering, E., Dzyubachyk, O., Smal, I., van Cappellen, W. A. Tracking in cell and developmental biology. Semin Cell Dev Biol. 20 (8), 894-902 (2009).
- Posakony, J. W., England, J. M., Attardi, G. Mitochondrial growth and division during the cell cycle in HeLa cells. J Cell Biol. 74 (2), 468-491 (1977).
- Zocchi, E., et al. Expression of CD38 Increases Intracellular Calcium Concentration and Reduces Doubling Time in HeLa and 3T3 Cells. J Biol Chem. 273 (14), 8017-8024 (1979).
- Reitzer, L. J., Wice, B. M., Kennell, D. Evidence that glutamine, not sugar, is the major energy source for cultured HeLa cells. J Biol Chem. 254 (8), 2669-2676 (1979).
- Benedetti, A. D. E., Joshi-barve, S., Rinker-Schaeffer, C., Rhoads, R. E. Expression of Antisense RNA against Initiation Factor eIF-4E mRNA in HeLa Cells Results in Lengthened Cell Division Times, Diminished Translation Rates, and Reduced Levels of Both eIF-4E and the p220 Component of eIF-4F. Mol Cell Biol. 11 (11), 5435-5445 (1991).
- Kumei, Y., Nakajima, T., Sato, A., Kamata, N., Enomoto, S. Reduction of G1 phase duration and enhancement of c-myc gene expression in HeLa cells at hypergravity. J Cell Sci. 93 (2), 221-226 (1989).
- Ginzberg, M. B., Kafri, R., Kirschner, M. On being the right (cell) size. Science. 348 (6236), 1245075 (2015).
- Mathieu, E., et al. Time-lapse lens-free imaging of cell migration in diverse physical microenvironments. Lab Chip. 16 (17), 3304-3316 (2016).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved