A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
PCR الكمي من عاثية T7 من بيوبانينج
In This Article
Summary
ويرد وصف استنساخه، ودقيقة، والوقت كمي PCR (qPCR) طريقة فعالة لتعداد عاثية T7 هنا. البروتوكول يصف بوضوح بالعاثية المجينية الحمض النووي إعداد وإعداد رد فعل بكر وظروف ركوب قبكر وتحليل البيانات قبكر.
Abstract
ويصف هذا البروتوكول استخدام PCR الكمي (قبكر) لتعداد فاجيس T7 من تجارب اختيار بالعاثية (أي، "بيوبانينج"). قبكر نهج قائم على الأسفار التحديد الكمي للحمض النووي، وهنا، أنها تتكيف مع تحديد جينومات بالعاثية كوكيل للجسيمات بالعاثية. في هذا البروتوكول، هو وصف أسلوب إعداد الحمض النووي بالعاثية سهلة باستخدام تدفئة ارتفاع درجة الحرارة دون تنقية الحمض النووي الإضافي. وتحتاج الطريقة فقط كميات صغيرة من فاجيس ردات وكميات صغيرة من رد فعل قبكر. قبكر الفائق وسريعة وقادرة على معالجة والحصول على البيانات من لوحة 96-جيدا من ردود فعل في حاء 2 – 4 بالمقارنة مع إلى نهج التعداد بالعاثية أخرى، قبكر أكثر كفاءة من حيث الوقت. وهنا، يستخدم qPCR تعداد فاجيس T7 المحددة من بيوبانينج ضد في المختبر النموذجي مخاط مثل التليف الكيسي. يمكن توسيع الأسلوب qPCR لقياس فاجيس T7 من التجارب الأخرى، بما في ذلك أنواع أخرى من بيوبانينج (على سبيل المثال.، المعطل تداولها البروتين ملزمة، المجراة في فحص بالعاثية) ومصادر أخرى (مثل شبكات مياه أو سوائل الجسم). وباختصار، يمكن تعديل هذا البروتوكول لتحديد أي نوع من الفيروسات المغلفة الحمض النووي.
Introduction
عاثية (بالعاثية) عرض التكنولوجيا، وضعت من قبل جورج سميث في عام 1985، نهج قوية، الفائق لتحديد يغاندس الببتيد أو البروتين ضد أهداف أو مستقبلات من غشاء الخلية، ومولدات الأمراض، العضيات الخلوية أو محددة والأنسجة في العقدين الماضيين1،الماضي2. هنا، يتم عرض المكتبات عشوائي البروتينية أو الأجسام المضادة لسلسلة واحدة على البروتينات معطف من فاجيس (عادة M13 أو T7)، ويغاندس معينة يمكن تحديدها من الغسل ضد البروتينات المعطل تداولها في المختبر أو في الجسم الحي النظم البيولوجية من خلال عملية اختيار تكرارية. ثم، يمكن أن يقترن يغاندس مع وكلاء التصوير أو العلاجية لتشخيص وعلاج الأمراض3،4. من الأهمية بمكان لدقة تعداد فاجيس خلال خطوات متعددة من بيوبانينج: (1) لقياس فاجيس التي تربط إلى الركيزة و (2) لقياس فاجيس لتحديد ما إذا كان هناك إثراء مع كل جولة من التحديد (الإثراء بالعاثية يشير إلى بيوبانيد تقارب بالعاثية للهدف). معيار الذهب الحالية للقياس الكمي، اللوحة طبقة مزدوجة بالانزيم، تحديات متعددة؛ أنها شاقة ومرهقة، ويحتمل أن تكون غير دقيقة لعدد كبير من العينات. ولذلك، وضعت مجموعتنا أسلوب سلسلة من ردود فعل (qPCR) بوليميريز كمية الحساسة واستنساخه، ودقيقة وفعالة من حيث الوقت لتعداد الجسيمات بالعاثية M13 و T7 من بيوبانينج5.
qPCR أسلوباً جذاباً ومجديا لقياس دقة فاجيس T7 و M13. حيث يمكن أن تحتوي كل الجسيمات بالعاثية الفردية فقط نسخة واحدة من الحمض النووي (دسدنا أو ssDNA)، واحد بالعاثية الجينوم يساوي واحد بالعاثية الجسيمات؛ بالتحديد الكمي لعدد مورثات بالعاثية، أنه من الممكن تحديد عدد فاجيس. أثناء قبكر، مراسل الفلورية الأصباغ ربط بالعاثية المجينية الحمض النووي غير تحديداً أو على وجه التحديد عن طريق الإشعال الخاصة بالتسلسل خلال التضخيم PCR، والأسفار إشارة يزيد مع كل جولة من التضخيم. ويلاحظ عند وصول إشارة الأسفار إلى العتبة، أن الجولة/دورة التضخيم كدورة عتبة (Ct). يتم رسم تركيزات معروفة من مرجع بالعاثية الحمض النووي ضد قيمها Ct إنشاء منحنى قياسي. استخدام المنحنى القياسي مع القيم المقطعية لعينات من الحمض النووي، يمكن أن يكون محرف تركيزات فاجيس.
في حين تم سبق أن وضعت استراتيجيات عديدة وتستخدم على نطاق واسع لقياس فاجيس من بيوبانينج، كل واحد منهم لديه تحديات معينة. هو الأسلوب الأكثر شعبية وتقليدية المقايسة البلاك طبقة مزدوجة. هنا، المضيف البكتيريا مصابون فاجيس أعد في تخفيف المسلسل وهي مطلية على ركيزة صلبة أجار، ويتم مضافين مع أجار؛ يتم تعداد فاجيس مع عدد اللوحات شكلت (أي، الوحدات تشكيل اللوحة أو بفو) على اللوحة أجار. فحص اللوحة الحساسة ولكن شاقة وتستغرق وقتاً طويلاً وغير دقيقة، خاصة بالنسبة لعينات عديدة ومع تركيزات عالية5. بالإضافة إلى ذلك، جرى تكييف لتعداد M13 و T7 بالعاثية الجسيمات6،،من78فحوصات الممتز المرتبط بالانزيم (ELISA). هنا، هي ملزمة فاجيس في تخفيف مختلفة واستولت على الركازة صلبة (أي، ميكروسكوبية) وسبر مع الأجسام المضادة بالعاثية محددة، والكشف عنها بواسطة استخدام الصحفيين (مثلاً إنزيم الحساسة ركائز اللونية، fluorophores) إلى تحديد عدد الجسيمات بالعاثية الحالية. قراءات (مثلاً، الأسفار، وامتصاص) من عينات يمكن استخدامها لقياس تركيزات غير معروفة ضد المعايير بالعاثية بتركيزات معروفة. مختلفة وقد وضعت على أساس بالعاثية اليساس ولكن لديهم القيود المحتملة. ووضع الفريق واحد الورقية ساندويتش أليسا مع مجموعة القياس الكمي التي امتدت سبعة أوامر من حجم (2-10 10،9 بفو/mL)؛ ومع ذلك، هذا الأسلوب يتطلب خطوات متعددة من طلاء جسم وأخذت إلى يوم كامل ل الفحص8. مجموعتنا أيضا تطوير أسلوب أليسا لقياس الجسيمات بالعاثية M13 ولكن كان أقل حساسية الكشف عن مجموعة من 106 إلى بفو/مل11 105. تم وضع المسابر fluorescence لانثانيدات التحويل لتعداد M13 ويمكن الحصول على بيانات القياس الكمي في غضون 20 دقيقة؛ ومع ذلك، قد هذا التحليل مجموعة ديناميكية ضيقة من 109 إلى 1012 مل بفو/9. مجموعة واحدة استخدام مجهر القوة الذرية لتعداد M13 بالعاثية الجسيمات في الحل، ولكن هذا يتطلب الميكروسكوب الإلكتروني المتقدمة وعملت فقط ضمن نطاق ضيق من تركيزات10. دراسة أخرى تستخدم المستحلبات مونوديسبيرسي إلى فخ فاجيس مراسل M13 و T4 الفلورسنت وحساب فاجيس بعدد قطرات الفلورية؛ ومع ذلك، أظهر هذا النهج أيضا طائفة ضيقة الكمي من 102 إلى6 10 مل بفو/11. بينما الحبرية PCR الرقمية قد استخدمت لقياس فاجيس M13، أن هذا النهج غير قادر على التفريق بين العدد من الأمراض المعدية وغير المعدية بالعاثية الجسيمات12.
ووضعت مجموعتنا مؤخرا أساليب qPCR لتعداد فاجيس T7 و M13 وحدد من بيوبانينج ضد نموذج خلية حاجز الدم في الدماغ باستخدام الأصباغ مراسل الفلورسنت مختلفة اثنين5. مقارنة بأساليب القياس الكمي المشار إليها أعلاه، قمنا بتطوير أساليب qPCR كانت الفائق، وكفاءة من حيث الوقت لقياس عينات عديدة وقادرة على التفريق بين الأمراض المعدية وغير المعدية فاجيس مع الدناز أنا ما قبل المعالجة بالعاثية العينات. الأهم من ذلك، هذا النهج يمكن أن تكاثر ودقة قياس bacteriophages M13 و T7 من عينات بيوبانينج. لتوجيه الباحثين المبتدئين في مجال qPCR بالعاثية، هنا يصف لنا طريقة qPCR مفصلة لتعداد T7 بالعاثية الجسيمات من مكتبة مقيدة سيستين (CX7C) انتقادات ضد حاجز مخاط تليف الكيسي (CF) في المختبر . من هذا العمل، ويمكن تمديد الأسلوب قبكر تحديد حجم الجسيمات بالعاثية من أنواع أخرى من بيوبانينج ومن مصادر أخرى، بما في ذلك المياه والتربة، وسوائل الجسم.
Access restricted. Please log in or start a trial to view this content.
Protocol
1-التمهيدي تصميم وتحليل للحمض النووي T7 بالعاثية
- تصميم كبسولة تفجير للتضخيم T7 بالعاثية المجينية الحمض النووي.
ملاحظة: و (إلى الأمام) والإشعال R (عكس) (انظر الجدول للمواد) تضخيم تسلسل الحمض النووي T7 يقع المنبع المنطقة مكتبة متغير (الشكل 1). - اختر محلل التمهيدي مناسبة لتقييم المعلمات كبسولة تفجير، بما في ذلك درجة حرارة ذوبان (Tm)، المئة GC المحتوى (GC %)، مبلمر، تشكيل دبوس وملاءمة بكر (الشكل 1).
- تأمر النوكليوتيد التمهيدي (أوليجوس)، وعند وصولهم، ريسوسبيند أوليجوس المجففة بالتبريد في الدناز ورناسي المياه النقية فائقة مجاناً لجعل 100 ميكرومتر الأسهم تركيزات في أنبوب الطرد مركزي 1.5 مل، وتخزينها في-20 درجة مئوية لعدة أشهر.
- جعل عدة مختبرين (حوالي 50 ميليلتر في كل أنبوبة 1.5 مل) من الأسهم التمهيدي 100 ميكرومتر.
ملاحظة: يتم استخدام هذه الخطوة منع دورات تجميد أذاب متكررة.
2-إعداد معايير للقياس الكمي للحمض النووي T7 بالعاثية
- متسلسل تضعف إشارة 0.1 ميكروغرام/ميليلتر (1.0 × 108 جي/ميليلتر) T7 بالعاثية الحمض النووي (تم شراؤها؛ بتركيز معلوم من 0.1 ميكروغرام/ميليلتر، انظر المواد) إذ من 1.0 × 106 إلى 1، 0 ×0 10 جي/ميليلتر مع نقية فائقة ح2س في أنابيب PCR 0.2 مل. إعداد كل تركيز في إجمالي حجم 20 ميليلتر (الشكل 2).
- دوامة PCR أنابيب لضمان خلط دقيق في كل خطوة للتخفيف. أيضا، تغيير نصائح عند إضافة عينات الحمض النووي لتمييع التالية.
ملاحظة: من المستحسن إعداد الحل الجديد لمعايير الحمض النووي لمنحنى قياسي لكل دفعة من التجربة قبكر. - استخدام المعادلة 1 (أدناه) لتحويل للإشارة بالعاثية T7 تركيزات الحمض النووي من جي/ميليلتر لنسخ الجينوم (gc)/µL (الشكل 2).
- تركيز الحمض النووي بالعاثية T7 في gc/ميليلتر =
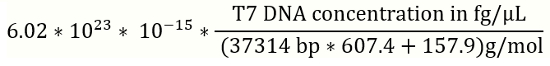 (1)
(1)
ملاحظة: بي بي: زوج قاعدي؛ مرجع T7 المجينية الحمض النووي هو 37314 بي بي.
3-قبل العلاج "بالعاثية عينات قبل" قبكر "إعداد رد فعل"
-
(الخيار 1) بيبيت 20 ميليلتر واحد T7 استنساخ (بالعاثية) مختارة من بيوبانينج ضد في المختبر التليف الكيسي (CF)-مثل نموذج المخاط إلى 1.5 مل أنابيب الطرد المركزي. إضافة وحدات 1.25 من الدناز الأول (0.5 ميليلتر) لكل عينة 20 ميليلتر.
ملاحظة: تم T7 بالعاثية سيستين هيبتابيبتيدي مقيدة مكتبة (CX7C) بيوبانيد ضد قوات التحالف مثل المخاط مغلفة بشأن إدراج غشاء 24-جيدا (انظر الجدول للمواد) لمدة 15 دقيقة. ترد تفاصيل أخرى في قسم النتائج. واختير واحد T7 استنساخ لإعداد نموذج qPCR من النذرة.- الخيار 2: بيبيت 200 ميليلتر T7 استنساخ في أنابيب 1.5 مل، وإضافة 5 وحدات من الدناز أنا
(2 ميليلتر) لكل عينة.
ملاحظة: يعتمد حجم العينة بالعاثية المختارة في هذه الخطوة على وحدة التخزين التي تم جمعها من بيوبانينج. بينما يركز البروتوكول على قبكر من بيوبانيد بالعاثية، الخطوات بيوبانينج مفصلة في البروتوكول المستخدم بالعاثية T7 كيت (انظر الجدول للمواد)13.
- الخيار 2: بيبيت 200 ميليلتر T7 استنساخ في أنابيب 1.5 مل، وإضافة 5 وحدات من الدناز أنا
- كاب الأنابيب العينة بالعاثية T7 (الخطوة 3.1) واحتضانها لهم في 37 درجة مئوية لمدة 10 دقيقة في حمام جافة ساخنة (أي كتلة الحرارة).
- احتضان أنابيب (من الخطوة 3، 2) عند 100 درجة مئوية لمدة 15 دقيقة في حمام جافة ساخنة (الشكل 3A).
تنبيه: أن التبخر والتكثيف يحدث أثناء الخطوة تدفئة، يجب التأكد من أن أنابيب الطرد المركزي 1.5 مل مغطاة تماما. - تدور فاجيس المستخرجة من الخطوة 3، 3 في س 18,534 ز للمزيج س. 10 فاجيس عن طريق وضع الأنبوب في دوامة خلاط تعيين في وضع لمسة في تفعيل 10 s، وتدور في س 18,534 ز ل 10 s مرة أخرى.
ملاحظة: دورة تدور-ميكس-سبين للتأكد من العينات بالعاثية ساخنة جيدا مختلطة. - تبريد الأنابيب العينة في درجة حرارة الغرفة (RT) بتركهم على مقاعد البدلاء التجربة أو المخزن في 4 درجات مئوية في ثلاجة بين عشية وضحاها.
ملاحظة: يمكن أن يكون مؤقتاً التجربة في هذه الخطوة.
4-إعداد ردود فعل قبكر
ملاحظة: رد فعل PCR واحد عينة واحدة. كل تفاعل PCR يحتوي على 5 ميليلتر سيد qPCR مزيج (انظر المواد)، 1 ميليلتر من 5 ميكرومتر التمهيدي زوج مزيج، 2 ميليلتر من ح2س و 2 ميليلتر عينة بالعاثية T7 ردات. للعديد من ردود الفعل بكر، هي ممزوجة مسبقاً جميع الكواشف استثناء بالعاثية العينة في أنبوب 1.5 مل واحد. وحدة التخزين لكل كاشف في مواده يعتمد على عدد العينات بالعاثية ل qPCR.
- إعداد مواده qPCR 10 عينات بالعاثية بيوبانيد تركيز غير معروف وعينات 7 من التركيزات القياسية في ثلاث نسخ. نتيجة لذلك هناك عينات إجمالي 51، ومن ثم، 51 من ردود فعل قبكر (أي رد فعل واحد كل عينة). لردود الفعل 51، إعداد مواده من 255 ميليلتر من مزيج الرئيسي قبكر (51 × 5 ميليلتر)، ميليلتر 51 كبسولة تفجير (51 × 1 ميليلتر)، و 102 ميليلتر من ح2س (51 × 2 ميليلتر).
ملاحظة: من المستحسن إعداد بضعة ردود PCR إضافي للتعويض عن فقدان وحدة التخزين أثناء اليقوتينج مزيج PCR في كل من لوحة قبكر 96-جيدا جيدا. - الكوة ميليلتر 8 من مواده بكر في كل بئر من لوحة قبكر 96-جيدا لما مجموعة 51 بئرا (أي 51 ردود فعل qPCR). ليست هناك حاجة لتغيير نصائح أثناء اليقوتينج.
- إضافة 2 ميليلتر من عينات بالعاثية T7 ردات (الخطوة 3، 4) أو مرجع بالعاثية T7 الحمض النووي (2.1 خطوة) لكل بئر (الشكل 3 (ب))؛ تغيير نصائح عند إضافة مختلفة من عينات الحمض النووي للبئر لمنع التلوث المتبادل. أيضا، الاستغناء عن عينات الحمض النووي ميليلتر 2 تحت سطح مواده قبكر (أي، في مواده بكر ميليلتر 8).
- إغلاق لوحة قبكر مع الفيلم لاصقة.
ملاحظة: هذه الخطوة الحاسمة، بغض النظر عن نوع لوحة qPCR. استخدام طقم الموصى بها لإغلاق اللوحة تماما؛ خلاف ذلك، أي منطقة غير مختومة في اللوحة سوف تسبب فقدان وحدة التخزين أثناء PCR ركوب الدراجات. - التفاف لوحة qPCR مع رقائق الألومنيوم إلى أدنى حد من فوتوبليتشينج الأسفار والمضي قدما لتشغيله على المعدات qPCR.
5-قبكر ظروف ركوب الدراجات
ملاحظة: شروط ركوب qPCR أقيمت على المعدات qPCR محددة (انظر الجدول للمواد).
- قم بتشغيل لوحة qPCR 96-جيدا على المعدات qPCR. على المعدات، حدد إعدادات من القائمة لإعداد الشروط التجريبية التالية (الشكل 3).
- إعداد الدراجات الشروط كما يلي لوحدة تخزين عينة من 10 ميليلتر: دورة واحدة في 50 درجة مئوية لمدة 2 دقيقة، دورة واحدة في 95 درجة مئوية لمدة 2 دقيقة، تليها دورات 40 (95 درجة مئوية لمدة 15 ثانية، 60 درجة مئوية لمدة 1 دقيقة). وبعد تشغيل الإعدادات منحنى تذوب: دورة واحدة من 95 درجة مئوية عن 15 ثانية، 60 درجة مئوية لمدة 1 دقيقة، و 95 درجة مئوية 15 s.
- الشروع في تحليل البيانات كما هو موضح في الخطوة 6.
6-تحليل qPCR "البيانات الخام" وتحويل قيمة الأشعة المقطعية إلى gc/ميليلتر من qPCR ل T7 Bacteriophages
- تحليل البيانات الخام قبكر وتقييم جودة البيانات من خلال تحليل قطع مميزة من البيانات الخام (الشكل 4).
ملاحظة: التضخيم مؤامرة ومؤامرة متعددة المكونات وتذوب منحنى الأرض هي المؤامرات أن السمة الرئيسية للتحقق من جودة البيانات qPCR. -
حساب قيم العتبة يعني دورة (Ct) من ثلاث عينات في تركيز كل من المنحنى المعياري (الخطوة 2، 1).
- ارسم يعني "الأشعة المقطعية" (ص) ضد سجل (gc/ميليلتر) (X) للحصول على المنحنى الخطي Y = kX + ب (ك هو ميل المنحنى الخطي، ب هو التقاطع).
- حساب معامل خطي14 ص2 (يجب أن يكون R2 > 0.99).
- استخدام المنحنى القياسي Y = kX + b لحساب عدد فاجيس T7 في عينات بيوبانينج (أي، تحديد ضد قوات التحالف مثل المخاط فاجيس) في gc/ميليلتر (الشكل 5).
ملاحظة: هو ك منحدر المنحنى المعياري. - حساب كفاءة التضخيم بالمعادلة التالية
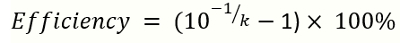 (2)
(2)
ملاحظة: هو ك منحدر المنحنى المعياري المتولدة من مرجع الحمض النووي قبكر. مجموعة مثالية من كفاءة التضخيم هو 90 – 110%.
Access restricted. Please log in or start a trial to view this content.
النتائج
يمكن استخدام أدوات التصميم التمهيدي مختلفة لتصميم كبسولة تفجير qPCR. بشكل عام، التمهيدي تصميم البرامج على خوارزميات المضمنة الخاصة بهم لحساب والتحقق من صحة معلمات مفتاح الإشعال، مثلاً، GC %، Tm، ديمر التمهيدي أو تشكيل دبوس، إلخ عموما، المعايير الأساسية التي مماثل...
Access restricted. Please log in or start a trial to view this content.
Discussion
قمنا بتطوير أساليب qPCR لقياس بالعاثية المجينية الحمض النووي5، وهنا يمكننا وصف وتكييف أسلوب qPCR لتعداد فاجيس T7 المحددة ضد حاجز مخاط مثل قوات التحالف. تم تكييف الحد الأدنى من المعلومات للمنشور من الكمية Real-Time PCR تجارب (ميقي) مبادئ توجيهية تطوير، والتحقق من صحة الأسلوب qPCR لتعداد T7 ف...
Access restricted. Please log in or start a trial to view this content.
Disclosures
الكتاب ليس لها علاقة بالكشف عن.
Acknowledgements
هذا العمل وأيده PhRMA مؤسسة أبحاث كاتب المنح والمنح الوطنية القلب والرئة، والدم معهد من "معاهد الصحة الوطنية" تحت رقم جائزة R01HL138251.
Access restricted. Please log in or start a trial to view this content.
Materials
| Name | Company | Catalog Number | Comments |
| Materials and Reagents | |||
| Primers for T7 genomic DNA | IDT | F: CCTCTTGGGAGGAAGAGATTTG R: TACGGGTCTCGTAGGACTTAAT | |
| T7 Select packaging control DNA | EMD Millipore | 69679-1UG | |
| MicroAmp optical 96-well reaction plate | ThermoFisher Scientific | N8010560 | |
| qPCR master mix--Power up SYBR Green master mix | Applied biosystems | A25742 | |
| MicroAmp optical adhesive film kit | ThermoFisher Scientific | 4313663 | |
| T7Select 415-1 Cloning Kit | EMD Millipore | 70015 | User protocols : http://www.emdmillipore.com/US/en/product/T7Select-415-1-Cloning-Kit,EMD_BIO-70015#anchor_USP |
| DNase I solution | ThermoFisher Scientific | 90083 | |
| 24-well transwell plate | Corning | 3472 | |
| UltraPure DNase/RNase-Free Distilled H2O | Invitrogen | 10977015 | |
| Phosphate Buffered Saline (1x) | Corning | 21040CV | |
| Name | Company | Catalog Number | Comments |
| Equipments | |||
| ViiA7 Real-Time PCR System with Fast 96-Well Block | ThermoFisher Scientific | 4453535 | |
| Heraeus Pico 21 Microcentrifuge | ThermoFisher Scientific | 75002415 | |
| Fisherbrand Digital Vortex Mixer | Fisher Scientific | 02-215-370 | |
| HERMO SCIENTIFIC Multi-Blok Heater | ThermoFisher Scientific | Model:2001 | |
| Sorvall Legend X1 Centrifuge | ThermoFisher Scientific | 75004220 | |
| M-20 Microplate Swinging Bucket Rotor | ThermoFisher Scientific | 75003624 | |
| Name | Company | Catalog Number | Comments |
| Software | |||
| QuantStudio Real-time PCR software | ThermoFisher Scientific | v1.2 | |
| Real-time qPCR primer design | IDT | ||
| OligoAnalyzer 3.1 | IDT |
References
- Smith, G. Filamentous fusion phage: novel expression vectors that display cloned antigens on the virion surface. Science. 228 (4705), 1315-1317 (1985).
- Smith, G. P., Petrenko, V. A. Phage Display. Chemical Reviews. 97 (96), 391-410 (1997).
- Sergeeva, A., Kolonin, M. G., Molldrem, J. J., Pasqualini, R., Arap, W. Display technologies: Application for the discovery of drug and gene delivery agents. Advanced Drug Delivery Reviews. 58 (15), 1622-1654 (2006).
- Dias-Neto, E., et al. Next-generation phage display: Integrating and comparing available molecular tools to enable costeffective high-throughput analysis. PLoS ONE. 4 (12), 1-11 (2009).
- Peng, X., Nguyen, A., Ghosh, D. Quantification of M13 and T7 bacteriophages by TaqMan and SYBR green qPCR. Journal of Virological Methods. 252 (June 2017), 100-107 (2017).
- Khan, M. S., Pande, T., Mvan de Ven, T. G. Qualitative and quantitative detection of T7 bacteriophages using paper based sandwich ELISA. Colloids and Surfaces B: Biointerfaces. 132, 264-270 (2015).
- Brasino, M., Lee, J. H., Cha, J. N. Creating highly amplified enzyme-linked immunosorbent assay signals from genetically engineered bacteriophage. Analytical Biochemistry. 470, 7-13 (2014).
- Yu, X., Burgoon, M., Shearer, A., Gilden, D. Characterization of phage peptide interaction with antibody using phage mediated immuno-PCR. J immunol Methods. 326 (1-2), 33-40 (2007).
- Lehmusvuori, A., Manninen, J., Huovinen, T., Soukka, T., Lamminmäki, U. Homogenous M13 bacteriophage quantification assay using switchable lanthanide fluorescence probes. BioTechniques. 53 (5), 301-303 (2012).
- Schlaman, H. R. M., et al. Analysis of interactions of signaling proteins with phage-displayed ligands by fluorescence correlation spectroscopy. Journal of Biomolecular Screening. 13 (8), 766-776 (2008).
- Tjhung, K. F., Burnham, S., Anany, H., Griffiths, M. W., Derda, R. Rapid enumeration of phage in monodisperse emulsions. Analytical Chemistry. 86 (12), 5642-5648 (2014).
- Reitinger, S., Petriv, O. I., Mehr, K., Hansen, C. L., Withers, S. G. Purification and quantitation of bacteriophage M13 using desalting spin columns and digital PCR. Journal of Virological Methods. 185 (1), 171-174 (2012).
- T7Select Biopanning Kit. , Millipore Sigma. Available from: https://www.emdmillipore.com/US/en/product/T7Select-Biopanning-Kit,EMD_BIO-70018#anchor_USP (2018).
- Schneider, A., Hommel, G., Blettner, M. Linear Regression Analysis. Deutsches Ärzteblatt international. 107 (44), 776-782 (2010).
- Bustin, S. A., Benes, V., Garson, J. A., Hellemans, J., Huggert, J. The MIQE Guidelines: Minimun information for publication of quantitative real-time PCR experiments. Clinical Chemitry. 55 (4), 611-622 (2009).
- Lock, M., Alvira, M. R., Chen, S. -J., Wilson, J. M. Absolute Determination of Single-Stranded and Self-Complementary Adeno-Associated Viral Vector Genome Titers by Droplet Digital PCR. Human Gene Therapy Methods. 25 (2), 115-125 (2014).
- Fittipaldi, M., et al. Discrimination of infectious bacteriophage T4 virus by propidium monoazide real-time PCR. Journal of Virological Methods. 168 (1-2), 228-232 (2010).
- Lodder, W. J., et al. Reduction of bacteriophage MS2 by filtration and irradiation determined by culture and quantitative real-time RT-PCR. Journal of Water and Health. 11 (2), 256-266 (2013).
- Brown-Jaque, M., et al. Antibiotic resistance genes in phage particles isolated from human feces and induced from clinical bacterial isolates. International Journal of Antimicrobial Agents. , (2017).
- Naveca, F. G., et al. Multiplexed reverse transcription real-time polymerase chain reaction for simultaneous detection of Mayaro, Oropouche, and oropouche-like viruses. Memorias do Instituto Oswaldo Cruz. 112 (7), 510-513 (2017).
- Jia, J., et al. Simultaneous detection and differentiation of human parvovirus B19 and human parvovirus 4 by an internally controlled multiplex quantitative real-time PCR. Molecular and Cellular Probes. 36, 50-57 (2017).
- Bliem, R., et al. A novel triplex quantitative pcr strategy for quantification of toxigenic and nontoxigenic Vibrio cholerae in aquatic environments. Applied and Environmental Microbiology. 81 (9), 3077-3085 (2015).
- Wan, Z., Goddard, N. L. Competition Between Conjugation and M13 Phage Infection in Escherichia coli in the Absence of Selection Pressure: A Kinetic Study. G3 Genes|Genomes|Genetics. 2 (10), 1137-1144 (2012).
Access restricted. Please log in or start a trial to view this content.
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved