JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
Biopanning에서 t 7 살 균 소의 양이 많은 PCR
요약
재현, 정확 하 고 시간이 효율적인 정량 PCR (정량) 방법 살 균 소 T7 열거 하는 여기에 설명 되어 있습니다. 프로토콜에는 살 균 소 게놈 DNA 준비, PCR 반응 준비, 정량 자전거 조건, 및 정량 데이터 분석 명확 하 게 설명합니다.
초록
이 프로토콜 T7 페이지 페이지 선택 실험 (즉, "biopanning")에서 열거 하 양이 많은 PCR (정량)의 사용을 설명 합니다. 정량은 DNA, 계량에 형광 기반의 접근 그리고 여기, 살 균 소 게놈 살 균 소 입자에 대 한 프록시로 계량에 적응. 이 프로토콜에서 추가적인 DNA 정화 없이 높은 온도 난방을 사용 하 여 손쉬운 살 균 소 DNA 준비 방법을 설명 합니다. 메서드를 사용 하면 열 처리 페이지의 작은 볼륨 및 정량 반응의 작은 볼륨에만 필요합니다. 정량은 높은 처리량 및 빠르고, 처리 하 고 다른 페이지 열거 방법에 2-4 헤 비교에에서 반응의 96 잘 접시에서 데이터를 가져올 수, 정량 시간 효율적입니다. 여기, 정량은 낭 성 섬유 증 같은 점액 모델 생체 외에서 biopanning에서 식별 T7 페이지를 열거 하는 데 사용 됩니다. 정량 방법 척도 biopanning의 다른 종류를 포함 하 여 다른 실험에서 T7 페이지를 확장할 수 있습니다 (예., 단백질 바인딩, vivo에서 움직일 수 살 균 소 심사) 및 기타 소스 (예: 시스템 또는 체액). 요약 하자면,이 프로토콜 DNA 캡슐화 바이러스 척도를 수정할 수 있습니다.
서문
살 균 소 (살 균 소) 디스플레이 기술, 1985 년, 조지 스미스에 의해 개발은 대상 또는 세포 막, 질병 항 원, 세포 세포 또는 특정 수용 체에 대 한 펩 티 드 또는 단백질 ligands를 식별 하는 강력한, 높은 처리량 접근 장기 고 지난 2 년간1,2조직. 여기, polypeptides 또는 단일 체인 항 체의 임의의 라이브러리 페이지 (일반적으로 M13 또는 T7)의 외 투 단백질에 표시 되 고 특정 ligands 고정된 단백질에 생체 외에서 또는 vivo에서 이동에서 확인할 수 있습니다. 반복 선택 과정을 통해 생물 시스템입니다. 다음, ligands 질병3,4의 진단 및 치료에 대 한 이미징 또는 치료 에이전트와 결합 될 수 있습니다. 그것은 정확 하 게 biopanning의 여러 단계 중 페이지를 열거 하는 중요 한: (1) 계량 기판에 바인딩할 페이지 (2) 하 고 선택의 각 라운드 농축 인지 확인 하려면 페이지를 계량 (살 균 소 농축 나타냅니다 biopanned 살 균 소 선호도 대상에 대 한)입니다. 정량화, 더블 레이어 패 분석 결과, 현재 금 표준에는 여러 과제가; 그것은 지루한, 성가신, 잠재적으로 많은 수의 샘플에 대 한 부정 확 한입니다. 따라서, 우리의 그룹 T7, M13 살 균 소 입자 biopanning5에서 열거 하는 민감한, 재현 하 고, 정확 하 고 효율적인 시간 정량적 중 합 효소 연쇄 반응 (정량) 방법을 개발 했습니다.
정량 Pcr T7 및 M13 페이지를 정확 하 게 계량 하는 매력적이 고 실현 가능한 방법 이다. 때문에 각 개별 살 균 소 입자 (dsDNA 또는 ssDNA) 게놈 DNA의 복사본을 하나 포함할 수 있습니다, 한 살 균 소 게놈은 한 살 균 소 입자; 살 균 소 게놈의 수를 측정, 페이지의 수를 정할 수 있다. 정량, 형광 기자 중 염료 비 특히 살 균 소 게놈 DNA 바인딩 또는 PCR 증폭, 동안 시퀀스 특정 뇌관 그리고 형광을 통해 구체적으로 신호 증폭의 각 라운드 증가. 형광 신호 그 라운드/사이클 확대의 임계값에 도달 하는 때 임계값 주기 (Ct)으로 유명 하다. 참조 살 균 소 DNA의 알려진된 농도 표준 곡선을 설정할 Ct 값에 대 한 구성 됩니다. DNA 샘플의 Ct 값으로 표준 곡선을 사용 하 여 페이지의 농도 보간됩니다 수 있습니다.
동안 수많은 전략 이전 개발 되었고 페이지 biopanning에서 척도를 광범위 하 게 사용, 그들 각각의 구체적인 과제는. 가장 인기 있는 하 고 기존의 방법은 더블 레이어 패 분석 결과입니다. 여기, 호스트 박테리아 감염 페이지 직렬 희석에 준비 된, 고체 agar 기판에 도금 및 천;와 겹쳐 페이지 수 플 라크 형성 (즉, 플 라크 형성 단위 또는 pfu) 한 천 배지에서의 열거 됩니다. 상 패 분석 결과 이며 민감한 하지만 지루한, 시간이 정확 하지 않은, 특히 많은 예제와 높은 농도5. 또한, 효소 연결 된 immunosorbent 분석 실험 (ELISA) T7, M13 살 균 소 입자6,7,8열거 적응 되었습니다. 여기, 페이지 다른 희석에 묶여있다 고 고체 기판 (즉, 한 미 판), 살 균 소 특정 항 체를 탐지 캡처하고 기자 (예를 들어, 효소 민감한 비 기판, fluorophores)를 사용 하 여 검색 현재 살 균 소 입자의 수를 결정 합니다. 샘플의 정보 (예를 들어, 형광, 흡 광도) 알려진된 농도에서 살 균 소 표준에 대 한 알 수 없는 농도 척도를 사용할 수 있습니다. 살 균 소 기반 ELISAs 개발 되었습니다 하지만 그들은 잠재적인 제한 다른. 한 그룹 개발 종이 기반 샌드위치 ELISA를 7 배나 (10-1029 pfu/mL); 부 량 범위 그러나,이 방법은 항 체 코팅의 여러 단계를 필요한 그리고8분석 결과 대 한 전체 하루에 최대 했다. 우리의 그룹 또한 M13 살 균 소 입자를 계량 하는 ELISA 방법 개발 했지만 10의 덜 민감한 감지 범위6 1011 pfu/mL5. M13 열거 하 전환 란타넘족 형광 프로브를 개발 하 고 20 분; 내 부 량 데이터를 얻을 수 있습니다. 그러나,이 분석 결과 10의 좁은 동적 범위 1012 pfu/mL99 . 한 그룹 솔루션, M13 살 균 소 입자를 열거할 원자 힘 현미경 검사 법을 사용 하지만이 고급 전자 현미경 필요 농도10의 좁은 범위 내에서 일했다. 또 다른 연구 형광 M13 및 T4 기자 페이지를 트랩 및 형광 방울; 수로 페이지를 단 분산 유화 제 사용 그러나,이 접근은 또한 10에서 좁은 부 량 범위 전시 106 pfu/mL112 . 드롭릿 디지털 PCR M13 페이지를 계량 하는 데 사용 되었습니다, 동안이 접근은 전염 성 및 비 전염 성 살 균 소 입자12의 번호를 수 없습니다.
우리의 그룹 최근 정량 방법을 열거 하는 두 개의 서로 다른 형광 기자 염료5를 사용 하 여 혈액-뇌 장벽 세포 모델에 대 한 biopanning에서 식별 T7 및 M13 페이지를 개발 했습니다. 상기 정량화 방법에 비해 정량 방법 개발 되었고 높은 처리량 및 수많은 샘플을 계량 시간 효율적인 DNase와 전염 성 및 비 전염 성 페이지 구분 나 전처리의 수는 살 균 소 샘플입니다. 중요 한 것은,이 이렇게 수 reproducibly 고 정확 하 게 계량 M13 및 T7 biopanning 샘플에서 살 균 소. 살 균 소 정량의 지역에 있는 연구원은 초보자 안내, 여기 우리가 T7 살 균 소 입자 시스테인 제한 된 라이브러리 (CX7C)에서 한 체 외에 낭 성 섬유 증 (CF) 점액 장벽에 대 한 패닝 열거 하는 상세한 정량 방법을 설명 합니다. 이 작품에서 정량 방법 살 균 소 입자와 물, 토양, 및 체액을 포함 하 여 다른 소스에서 biopanning의 다른 종류에서 계량을 확장할 수 있습니다.
프로토콜
1. 뇌관 디자인 및 T7 살 균 소 게놈 DNA에 대 한 분석
- T7 살 균 소 게놈 DNA의 확대를 위한 뇌관 디자인.
참고: F (앞으로)와 R (반전) 뇌관 ( 재료의 표참조) T7 DNA 순서를 증폭 라이브러리 변수 영역 (그림 1) 상류에 위치. - 용융 온도 (Tm), 백분율 GC 내용 (GC %), 뇌관 이합체, 머리 핀 형성 및 PCR 적합성 (그림 1)를 포함 한 뇌관의 매개 변수를 평가 하는 적절 한 뇌관 분석기를 선택 하십시오.
- 뇌관 oligonucleotides (oligos), 주문 그리고 도착 시 DNase과 RNase 무료 울트라 순수 물 1.5 mL 원심 분리기 튜브, 100 µ M 재고 농도 확인 하 고 몇 달 동안-20 ° C에서 저장에 동결 건조 된 oligos resuspend.
- 100 µ M 뇌관의 여러 aliquots (각 1.5 mL 튜브에 약 50 µ L)을 확인 합니다.
참고:이 단계는 자주 freeze-thaw 주기를 방지 하기 위해 사용 됩니다.
2. t 7 살 균 소 게놈 DNA의 정량화에 대 한 표준 준비
- 직렬로 0.1 µ g / µ L (1.0 x 108 fg / µ L) 참조 T7 살 균 소 DNA를 희석 (구입, 0.1 µ g / µ L의 알려진된 농도에 자료 참조) 10 1.0 x 10에서6 100 fg / µ L 울트라 순수 H2O 0.2 mL PCR 튜브에 x 1.0. 20 µ L (그림 2)의 총 볼륨의 각 농도 준비 합니다.
- 소용돌이 PCR 튜브 희석의 각 단계에서 철저 하 게 혼합 되도록 합니다. 또한, 다음 희석에 DNA 샘플을 추가할 때 팁을 변경 합니다.
참고: 그것은 정량 실험의 모든 일괄 처리에 대 한 표준 곡선에 대 한 DNA 표준의 새로운 솔루션을 준비 하 고 좋습니다. - 방정식 1 (아래)를 사용 하 여 변환할 T7 페이지 참조에 대 한 DNA 농도 fg / µ L에서 게놈 복사본 (gc) /µL (그림 2).
- T7 gc / µ L에서 살 균 소 DNA 농도 =
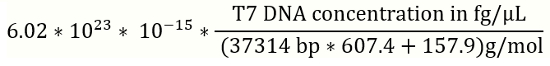 (1)
(1)
참고: bp: 기본 쌍; 참조 T7 genomic DNA는 37314 혈압.
3. 살 균 소 샘플 전에 정량 Pcr 반응 준비의 전처리
-
(옵션 1) 피펫으로 20 µ L 한 T7 클론 (살 균 소)의 낭 성 섬유 증 (CF) 생체 외에서 에 대 한 biopanning에서 선택-1.5 mL 원심 분리기 튜브로 점액 모델 처럼. 1.25 단위의 DNase 추가 나 (0.5 µ L) 각 20 µ L 샘플을.
참고: T7 페이지 시스테인 제한 heptapeptide 라이브러리 (CX7C)은 15 분 동안 24-잘 막 삽입 ( 재료의 표참조)에 코팅 하는 CF 같은 점액에 대 한 biopanned. 자세한 내용은 결과 섹션에서 제공 됩니다. T7 클론이 eluate에서 정량 샘플 준비를 위해 선택 되었다.- 옵션 2: 피펫으로 200 µ L는 t 7의 1.5 mL 튜브에 복제 하 고 5 단위의 DNase 추가 나
(2 µ L) 각 샘플을.
참고:이 단계에서 선택 하는 살 균 소 견본의 볼륨 biopanning에서 수집 된 볼륨에 따라 달라 집니다. Biopanned 살 균 소의 정량에는 프로토콜의 포커스를 실행 하는 동안 단계 biopanning T7 페이지의 사용자 프로토콜에 자세한 키트 ( 재료의 표참조)13.
- 옵션 2: 피펫으로 200 µ L는 t 7의 1.5 mL 튜브에 복제 하 고 5 단위의 DNase 추가 나
- T7 페이지 샘플 튜브 (3.1 단계)을 닫습니다 그리고 온수 건조 목욕 (즉, 열 블록)에 10 분 동안 37 ° C에서 그들을 품 어.
- 튜브 (3.2 단계)에서 열된 건조 목욕 (그림 3A)에서 15 분 100 ° C에서 품 어.
주의: 증발 및 응축 열 단계 동안 발생, 이후 1.5 mL 원심 분리기 튜브 완전히 덮인 있는지 확인 합니다. - 10 미 믹스 10 s, 및 18,534 x g 10에서 스핀 터치 활성화 모드와 동 믹서에 튜브를 삽입 하 여 페이지 설정에 대 한 18,534 x g 에서 3.3 단계에서 열 처리 페이지를 회전 s 다시.
참고:가 열된 살 균 소 샘플 잘 혼합 되도록 스핀-믹스-스핀 사이클이입니다. - 밤새 실험 벤치 또는 냉장고에서 4 ° C에서 저장소에 그들을 떠나는 하 여 실 온 (RT)에서 샘플 튜브를 식혀.
참고: 실험은이 단계에는 일시 중지 될 수 있습니다.
4. 정량 Pcr 반응 준비
참고: 한 PCR 반응 한 샘플입니다. 각 PCR 반응 포함 5 µ L 정량 마스터의 믹스 (자료 참조), 5 µ M 뇌관 쌍 믹스의 1 µ L, H2O 2 µ L, 2 µ L 열 처리 T7 페이지 샘플의. 다중 PCR 반응에 대 한 살 균 소 샘플을 제외 하 고 모든 시 약 한 1.5 mL 튜브에 혼합 됩니다. 각 시에는 프리 믹스의 볼륨 정량에 대 한 살 균 소 견본의 수에 따라 달라 집니다.
- 알 수 없는 농도의 10 biopanned 페이지 샘플 및 3 중에서 표준 농도의 7 샘플에 대 한 정량 프리 믹스를 준비 합니다. 그 결과, 51 총 샘플 및 따라서 51 정량 반응 (즉, 샘플 당 하나의 반응) 있다. 51 반응에 대 한 255 µ L 정량 마스터 믹스 (51 x 5 µ L), 뇌관 (51 x 1 µ L)의 51 µ L 및 H2O의 102 µ L의 (2 µ L x 51)의 프리 믹스를 준비 합니다.
참고: 그것 aliquoting 96 잘 정량 플레이트의 각 음에 PCR 혼합 하는 동안 볼륨 손실에 대 한 보상을 몇 가지 추가 PCR 반응 준비 하 고 좋습니다. - Aliquot 8 µ L 51 우물 (즉, 51 정량 반응)의 총에 대 한 정량 96 잘 접시의 각 음에 PCR 프리 믹스의. Aliquoting 중 팁을 변경할 필요가 있다.
- 열 처리 T7 페이지 샘플 (단계 3.4)의 2 µ L 또는 T7 페이지 참조 DNA (단계 2.1) 각 잘 (그림 3B);를 추가합니다 교차 오염을 방지 하기 위해 잘에 다른 DNA 샘플을 추가할 때 팁을 변경 합니다. 또한, 정량 프리 믹스 (즉, 8 µ L PCR 섞으로은)의 표면 아래에 2 µ L DNA 샘플 분배.
- 접착 필름으로 정량 접시를 봉인.
참고:이 단계는 중요 한 정량 접시의 유형에 관계 없이. 권장된 키트를 사용 하 여 완전히; 접시를 봉인 그렇지 않으면, 모든 봉인 지역 접시에 PCR 동안 볼륨 손실 하면 사이클링. - 형광 photobleaching를 최소화 하기 위해 알루미늄 호 일 정량 플레이트 랩 고 정량 장비에 그것을 실행.
5입니다. 정량 사이클링 조건
참고: 정량 자전거 조건 설정 했다 특정 정량 장비에 ( 재료의 표참조).
- 정량 Pcr 장비에서 정량 96 잘 접시를 실행 합니다. 장비, 다음과 같은 실험 조건 (그림 3C)를 설정 하는 메뉴에서 설정을 선택 합니다.
- 사이클링 다음과 같이 10 µ L의 샘플 볼륨에 대 한 조건 설정: 2 분 50 ° C에서 하나 주기 2 분 동안 95 ° C에서 하나 주기 다음의 40 주기 (15 95 ° C s, 1 분 동안 60 ° C). 용융 곡선 설정 후, 실행: 15 s, 1 분 동안 60 ° C 및 95 ° C 15 s 95 ° C의 1 개 주기.
- 6 단계에 설명 된 대로 데이터를 분석을 진행 합니다.
6. 정량 원시 데이터를 분석 하 고 변환 정량에서 Ct 값 gc / µ L을 t 7 살 균 소에 대 한
- 원시 정량 데이터를 분석 하 고 원시 데이터 (그림 4)의 독특한 플롯을 분석 하 여 데이터 품질을 평가 합니다.
참고: 증폭 플롯, multicomponent 줄거리 및 용융 곡선 작 키 특성을 정량의 데이터 품질을 검사할 플롯 됩니다. -
표준 곡선 (2.1 단계)의 각 농도에서 triplicate 샘플에서 평균 임계값 주기 (Ct) 값 계산 합니다.
- 평균 Ct (Y)에 대 한 로그 (gc / µ L) (X) Y 선형 곡선을 플롯 kX + b = (k는 선형 곡선의 기울기, b는 절편).
- 계산 하는 선형 계수14 R2 (R2 가 되어야 합니다 > 0.99).
- 사용 하는 표준 곡선 Y = kX + b gc / µ L (그림 5)에서 biopanning 샘플 (즉, 페이지 CF 같은 점액에 대 한 선택) T7 페이지의 수를 계산 하.
참고: k 표준 곡선의 기울기 이다. - 증폭 효율을 계산 하는 다음 수식으로
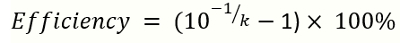 (2)
(2)
참고: k 참조 DNA 정량에서 생성 된 표준 곡선의 기울기 이다. 증폭 효율의 이상적인 범위 90-110%입니다.
결과
정량 Pcr 뇌관 디자인 다른 뇌관 디자인 도구를 사용할 수 있습니다. 일반적으로, 뇌관 디자인 프로그램은 그들의 자신의 내장 알고리즘을 계산 하 고 뇌관, 예를 들어, GC %, Tm, 뇌관 이합체 또는 머리 핀 형성 등 의 중요 매개 변수 일반적으로, 주요 기준은 다른 유사 뇌관 설계 도구, 및 뇌관 그들의 지침에 따라 설계할 수 있습니다. 뇌관 폭발 뇌관의 특이성을 ...
토론
우리 살 균 소 게놈 DNA5, 계량을 정량 방법을 개발 하 고 우리가 여기 설명 T7 페이지 CF 같은 점액 장벽에 대 한 선정을 열거 하는 정량 방법을 적응. 간행물의 양적 실시간 PCR 실험 (MIQE) 지침에 대 한 최소 정보 개발 T7 페이지15의 열거에 대 한 정량 방법의 유효성을 검사 하 적응 했다. 우리는 biopanning 실험에서 페이지 척도를 개발 하는 프로토콜은 시간 효율적, ?...
공개
저자는 공개 없다.
감사의 말
이 작품은 PhRMA 기초 연구 동기 부여에 의해 지원 되었다 고 국가 심 혼, 폐, 혈액 연구소 건강의 국가 학회의 보너스 번호 R01HL138251에서 부여 합니다.
자료
| Name | Company | Catalog Number | Comments |
| Materials and Reagents | |||
| Primers for T7 genomic DNA | IDT | F: CCTCTTGGGAGGAAGAGATTTG R: TACGGGTCTCGTAGGACTTAAT | |
| T7 Select packaging control DNA | EMD Millipore | 69679-1UG | |
| MicroAmp optical 96-well reaction plate | ThermoFisher Scientific | N8010560 | |
| qPCR master mix--Power up SYBR Green master mix | Applied biosystems | A25742 | |
| MicroAmp optical adhesive film kit | ThermoFisher Scientific | 4313663 | |
| T7Select 415-1 Cloning Kit | EMD Millipore | 70015 | User protocols : http://www.emdmillipore.com/US/en/product/T7Select-415-1-Cloning-Kit,EMD_BIO-70015#anchor_USP |
| DNase I solution | ThermoFisher Scientific | 90083 | |
| 24-well transwell plate | Corning | 3472 | |
| UltraPure DNase/RNase-Free Distilled H2O | Invitrogen | 10977015 | |
| Phosphate Buffered Saline (1x) | Corning | 21040CV | |
| Name | Company | Catalog Number | Comments |
| Equipments | |||
| ViiA7 Real-Time PCR System with Fast 96-Well Block | ThermoFisher Scientific | 4453535 | |
| Heraeus Pico 21 Microcentrifuge | ThermoFisher Scientific | 75002415 | |
| Fisherbrand Digital Vortex Mixer | Fisher Scientific | 02-215-370 | |
| HERMO SCIENTIFIC Multi-Blok Heater | ThermoFisher Scientific | Model:2001 | |
| Sorvall Legend X1 Centrifuge | ThermoFisher Scientific | 75004220 | |
| M-20 Microplate Swinging Bucket Rotor | ThermoFisher Scientific | 75003624 | |
| Name | Company | Catalog Number | Comments |
| Software | |||
| QuantStudio Real-time PCR software | ThermoFisher Scientific | v1.2 | |
| Real-time qPCR primer design | IDT | ||
| OligoAnalyzer 3.1 | IDT |
참고문헌
- Smith, G. Filamentous fusion phage: novel expression vectors that display cloned antigens on the virion surface. Science. 228 (4705), 1315-1317 (1985).
- Smith, G. P., Petrenko, V. A. Phage Display. Chemical Reviews. 97 (96), 391-410 (1997).
- Sergeeva, A., Kolonin, M. G., Molldrem, J. J., Pasqualini, R., Arap, W. Display technologies: Application for the discovery of drug and gene delivery agents. Advanced Drug Delivery Reviews. 58 (15), 1622-1654 (2006).
- Dias-Neto, E., et al. Next-generation phage display: Integrating and comparing available molecular tools to enable costeffective high-throughput analysis. PLoS ONE. 4 (12), 1-11 (2009).
- Peng, X., Nguyen, A., Ghosh, D. Quantification of M13 and T7 bacteriophages by TaqMan and SYBR green qPCR. Journal of Virological Methods. 252 (June 2017), 100-107 (2017).
- Khan, M. S., Pande, T., Mvan de Ven, T. G. Qualitative and quantitative detection of T7 bacteriophages using paper based sandwich ELISA. Colloids and Surfaces B: Biointerfaces. 132, 264-270 (2015).
- Brasino, M., Lee, J. H., Cha, J. N. Creating highly amplified enzyme-linked immunosorbent assay signals from genetically engineered bacteriophage. Analytical Biochemistry. 470, 7-13 (2014).
- Yu, X., Burgoon, M., Shearer, A., Gilden, D. Characterization of phage peptide interaction with antibody using phage mediated immuno-PCR. J immunol Methods. 326 (1-2), 33-40 (2007).
- Lehmusvuori, A., Manninen, J., Huovinen, T., Soukka, T., Lamminmäki, U. Homogenous M13 bacteriophage quantification assay using switchable lanthanide fluorescence probes. BioTechniques. 53 (5), 301-303 (2012).
- Schlaman, H. R. M., et al. Analysis of interactions of signaling proteins with phage-displayed ligands by fluorescence correlation spectroscopy. Journal of Biomolecular Screening. 13 (8), 766-776 (2008).
- Tjhung, K. F., Burnham, S., Anany, H., Griffiths, M. W., Derda, R. Rapid enumeration of phage in monodisperse emulsions. Analytical Chemistry. 86 (12), 5642-5648 (2014).
- Reitinger, S., Petriv, O. I., Mehr, K., Hansen, C. L., Withers, S. G. Purification and quantitation of bacteriophage M13 using desalting spin columns and digital PCR. Journal of Virological Methods. 185 (1), 171-174 (2012).
- . T7Select® Biopanning Kit Available from: https://www.emdmillipore.com/US/en/product/T7Select-Biopanning-Kit (2018)
- Schneider, A., Hommel, G., Blettner, M. Linear Regression Analysis. Deutsches Ärzteblatt international. 107 (44), 776-782 (2010).
- Bustin, S. A., Benes, V., Garson, J. A., Hellemans, J., Huggert, J. The MIQE Guidelines: Minimun information for publication of quantitative real-time PCR experiments. Clinical Chemitry. 55 (4), 611-622 (2009).
- Lock, M., Alvira, M. R., Chen, S. -. J., Wilson, J. M. Absolute Determination of Single-Stranded and Self-Complementary Adeno-Associated Viral Vector Genome Titers by Droplet Digital PCR. Human Gene Therapy Methods. 25 (2), 115-125 (2014).
- Fittipaldi, M., et al. Discrimination of infectious bacteriophage T4 virus by propidium monoazide real-time PCR. Journal of Virological Methods. 168 (1-2), 228-232 (2010).
- Lodder, W. J., et al. Reduction of bacteriophage MS2 by filtration and irradiation determined by culture and quantitative real-time RT-PCR. Journal of Water and Health. 11 (2), 256-266 (2013).
- Brown-Jaque, M., et al. Antibiotic resistance genes in phage particles isolated from human feces and induced from clinical bacterial isolates. International Journal of Antimicrobial Agents. , (2017).
- Naveca, F. G., et al. Multiplexed reverse transcription real-time polymerase chain reaction for simultaneous detection of Mayaro, Oropouche, and oropouche-like viruses. Memorias do Instituto Oswaldo Cruz. 112 (7), 510-513 (2017).
- Jia, J., et al. Simultaneous detection and differentiation of human parvovirus B19 and human parvovirus 4 by an internally controlled multiplex quantitative real-time PCR. Molecular and Cellular Probes. 36, 50-57 (2017).
- Bliem, R., et al. A novel triplex quantitative pcr strategy for quantification of toxigenic and nontoxigenic Vibrio cholerae in aquatic environments. Applied and Environmental Microbiology. 81 (9), 3077-3085 (2015).
- Wan, Z., Goddard, N. L. Competition Between Conjugation and M13 Phage Infection in Escherichia coli in the Absence of Selection Pressure: A Kinetic Study. G3 Genes|Genomes|Genetics. 2 (10), 1137-1144 (2012).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유