É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
PCR quantitativo do bacteriófago T7 de Biopanning
Neste Artigo
Resumo
Um tempo, preciso e reprodutível quantitativo PCR (qPCR) método eficiente para enumerar do bacteriófago T7 é descrito aqui. O protocolo descreve claramente a preparação de DNA genômica do fago, preparação de reação de PCR, qPCR ciclismo condições e análise de dados de qPCR.
Resumo
Este protocolo descreve o uso do PCR quantitativo (qPCR) para enumerar fago T7 de experimentos de seleção do fago (i.e., "biopanning"). qPCR é uma abordagem baseada em fluorescência para quantificação de DNA, e aqui, é adaptado para quantificar o genoma do fago como um proxy para as partículas do fago. Neste protocolo, um método de preparação do ADN do fago facile é descrito usando o aquecimento de alta temperatura sem purificação de DNA adicional. O método só precisa de pequenos volumes do fago tratado termicamente e pequenos volumes da reação qPCR. qPCR é alta produtividade e rápido, capaz de processar e obter dados de uma placa de 96 poços de reações em 2-4 h. comparado a outras abordagens de enumeração do fago, qPCR é mais eficiente em termos de tempo. Aqui, qPCR é usado para enumerar fago T7 identificado de biopanning contra o modelo de muco da fibrose cística, como in vitro . O método de qPCR pode ser estendido para quantificar o fago T7 de outros experimentos, incluindo outros tipos de biopanning (ex., triagem de fago imobilizou o emperramento da proteína, in vivo ) e outras fontes (por exemplo, sistemas de água ou fluidos corporais). Em resumo, este protocolo pode ser modificado para quantificar qualquer vírus DNA encapsulado.
Introdução
Tecnologia de exibição do bacteriófago (fago), desenvolvida por George Smith em 1985, é uma abordagem poderosa, elevado-throughput para identificar peptídeos ou proteínas ligantes contra alvos ou receptores da membrana celular, organelas celulares, antígenos da doença ou específicos órgãos e tecidos nas últimas duas décadas1,2. Aqui, bibliotecas aleatórias de polipeptídeos ou anticorpos de cadeia única são exibidas sobre as proteínas do casaco do fago (tipicamente M13 ou T7) e ligantes específicos podem ser identificados de garimpar contra proteínas imobilizadas em vitro ou in vivo sistemas biológicos, através de um processo de seleção iterativa. Então, ligantes podem ser acoplados com agentes terapêuticos ou de imagem para diagnóstico e tratamento de doenças3,4. É fundamental para enumerar com precisão o fago durante várias etapas de biopanning: (1) para quantificar o fago que ligam-se ao substrato e (2) para quantificar o fago para determinar se há enriquecimento com cada rodada de seleção (enriquecimento do fago indica biopanned afinidade do fago para o destino). O atual padrão de ouro para quantificação, ensaio de chapa de dupla camada, apresenta vários desafios; é tedioso, complexos e potencialmente impreciso para um grande número de amostras. Portanto, nosso grupo desenvolveu um método de (qPCR) reação em cadeia de polimerase quantitativo sensível, reprodutível, precisos e tempo-eficiente para enumerar as partículas do fago M13 e T7 de biopanning5.
qPCR é um método atraente e viável quantificar com precisão o fago T7 e M13. Desde que cada partícula individual do fago pode conter apenas uma cópia do DNA genômico (dsDNA ou ssDNA), um genoma do fago é equivalente a uma partícula fago; quantificando o número de genomas do fago, é possível quantificar o número de fago. Durante qPCR, repórter fluorescente corantes ligam o DNA genômico do fago não especìfica ou especificamente por meio de cartilhas específicas durante a amplificação por PCR e a fluorescência sinal aumenta com cada rodada de amplificação. Quando o sinal de fluorescência atinge o limite, que rodada/ciclo da amplificação é conhecido como o ciclo de limiar (Ct). As concentrações conhecidas de referência do fago DNA são plotadas contra seus valores de Ct para estabelecer uma curva padrão. Usando a curva padrão com os valores de Ct de amostras de DNA, as concentrações do fago podem ser interpoladas.
Enquanto inúmeras estratégias foram anteriormente desenvolvidas e usadas extensivamente para quantificar o fago de biopanning, cada um deles tem desafios específicos. O método mais popular e convencional é o ensaio da chapa de dupla camada. Aqui, o anfitrião bactérias estão infectadas com fago preparado em diluições em série, são banhadas em um substrato sólido ágar e são revestidas com ágar; fagos são enumerados com o número de placas formado (ou seja, placa formando unidades ou pfu) na placa de ágar. Ensaio de placa é sensível mas tedioso, demorado e impreciso, especialmente para numerosas amostras e com altas concentrações de5. Além disso, ensaios imunoenzimático (ELISA) foram adaptados para enumerar M13 e T7 fago partículas6,7,8. Aqui, o fago em diferentes diluições é vinculado e capturado em um substrato sólido (ou seja, uma microplaca), sondado com anticorpos específicos do fago e detectados por meio de repórteres (por exemplo, enzima sensível cromogênico substratos, fluorophores) para Determine o número de partículas do fago presentes. As leituras (por exemplo, fluorescência, absorbância) das amostras podem ser usadas para quantificar as concentrações desconhecidas contra normas do fago em concentrações conhecidas. Diferentes baseados no fago ELISAs têm sido desenvolvidas, mas têm limitações potenciais. Um grupo desenvolveu um sanduíche em papel ELISA com uma gama de quantificação que durou sete ordens de magnitude (102-109 pfu/mL); no entanto, esse método necessárias várias etapas de revestimento de anticorpo e levou para um dia inteiro para o ensaio de8. Nosso grupo também desenvolveu um método de ELISA para quantificar as partículas de fago M13, mas tinha um intervalo de detecção menos sensível do 106 a de pfu/mL 1011 5. Lantanídios comutável sondas de fluorescência foram desenvolvidas para enumerar M13 e dados de quantificação podem ser obtidos dentro de 20 min; no entanto, este ensaio tem uma estreita faixa dinâmica de 109 1012 pfu/mL9. Um grupo usado microscopia de força atômica para enumerar as partículas de fago M13 em solução, mas isso requer avançados de microscopia eletrônica e trabalhou apenas dentro de uma faixa estreita de concentrações10. Outro estudo usado monodisperso emulsões para prender fluorescente fago M13 e T4 do repórter e contar fago pelo número de gotículas fluorescentes; no entanto, esta abordagem também exibiu uma gama estreita de quantificação de 102 a 10 de pfu/mL6 11. Enquanto gotículas que digital PCR tem sido utilizada para quantificar o fago M13, esta abordagem é incapaz de diferenciar entre o número de partículas de fago infecciosas e não infecciosas12.
O nosso grupo desenvolveu recentemente qPCR métodos para enumerar fago T7 e M13 identificado de biopanning contra um modelo de célula barreira sangue - cérebro usando dois diferentes repórter fluorescente tinturas5. Em comparação com métodos de quantificação acima mencionados, os métodos de qPCR desenvolvemos foram elevado-throughput e tempo-eficiente para quantificar numerosas amostras e capaz de diferenciar entre fago infeccioso e não infecciosas com DNase eu pré-tratamento da amostras do fago. Importante, esta abordagem pode reproducibly e precisão quantificar bacteriófagos M13 e T7 de biopanning amostras. Para orientar pesquisadores novatos na área do fago qPCR, aqui descrevemos um método de qPCR detalhadas para enumerar as partículas do fago T7 de uma biblioteca de cisteína-restrita (CX7C) garimpadas contra uma barreira de muco de fibrose cística (CF) em vitro . Partir deste trabalho, método de qPCR pode ser estendido para quantificar as partículas do fago de outros tipos de biopanning e de outras fontes, incluindo água, solo e fluidos corporais.
Protocolo
1. primer Design e análise de DNA genômico do fago T7
- Desenho de primers para amplificação de DNA genômico do fago T7.
Nota: F (frente) e as primeiras demão R (reverso) (ver Tabela de materiais) amplificam a sequência de DNA T7 localizado a montante da região variável da biblioteca (Figura 1). - Escolha um analisador primário adequado para avaliar parâmetros dos primers, incluindo a temperatura de fusão (Tm), porcentagem conteúdo GC (GC %), dímero da primeira demão, formação de hairpin e adequação PCR (Figura 1).
- Encomendar os oligonucleotides cartilha (oligos) e na chegada, resuspenda os oligos liofilizado em água ultra-pura DNase e RNase livre para fazer as concentrações estoque de 100 µM em um tubo de centrífuga de 1,5 mL e armazenar a-20 ° C por vários meses.
- Faça várias alíquotas (cerca de 50 µ l em cada tubo de 1,5 mL) da unidade populacional de cartilha de 100 µM.
Nota: Este passo é utilizado para prevenir ciclos de congelamento-descongelamento frequentes.
2. preparar as normas para a quantificação de DNA genômico do fago T7
- Diluir em série referência 0,1 µ g / µ l (1.0 x 108 fg / µ l) ADN do fago T7 (comprado; em uma concentração conhecida de 0,1 µ g / µ l, consulte materiais) 10 vezes de 1,0 x 106 a 1,0 x 100 fg / µ l com ultra-pura H2O em cadeia da polimerase 0,2 mL. Prepare cada concentração em um volume total de 20 µ l (Figura 2).
- Vórtice da PCR tubos para assegurar uma mistura completa em cada etapa de diluição. Além disso, altere dicas ao adicionar a amostra de DNA para a diluição seguinte.
Nota: É aconselhável preparar nova solução de padrões de DNA para uma curva padrão para cada lote do experimento qPCR. - Use a equação 1 (abaixo) para converter para referência do fago T7 concentrações de DNA de fg / µ l de cópias do genoma (gc), /µL (Figura 2).
- Concentração de DNA do fago T7 em gc / µ l =
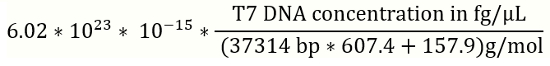 (1)
(1)
Nota: bp: pares de base; referência de DNA genômico de T7 é 37314 bp.
3. pré-tratamento de amostras antes de Phage qPCR preparação de reação
-
(Opção 1) Pipetar 20 µ l de um clone de T7 (fago) selecionados a partir de biopanning contra em vitro fibrose cística (CF)-como modelo de muco em tubos de centrífuga de 1,5 mL. Adicionar 1,25 unidades de DNase I (0,5 µ l) para cada 20 µ l da amostra.
Nota: A biblioteca (CX7C) heptapeptide restrita cisteína do fago T7 foi biopanned contra CF-como muco revestido em pastilhas de membrana 24-poço (ver Tabela de materiais) por 15 min. Mais detalhes são fornecidos na seção de resultados. Um clone de T7 foi escolhido para a preparação de amostra qPCR de eluído.- Opção 2: Pipetar 200 µ l de T7 a clonar um tubos de 1,5 mL e adicionar 5 unidades de DNase eu
(2 µ l) para cada amostra.
Nota: O volume da amostra do fago selecionado neste passo depende do volume coletado de biopanning. Enquanto o foco do protocolo é o qPCR do fago biopanned, as etapas para biopanning estão detalhadas no protocolo do usuário do fago T7 kit (veja a Tabela de materiais)13.
- Opção 2: Pipetar 200 µ l de T7 a clonar um tubos de 1,5 mL e adicionar 5 unidades de DNase eu
- Tampar os tubos de amostra do fago T7 (passo 3.1) e incube-os a 37 ° C por 10 min em um banho seco aquecido (ou seja, o bloco de calor).
- Incube os tubos (de passo 3.2) a 100 ° C por 15 min em um banho seco aquecido (Figura 3A).
Atenção: Desde que a evaporação e a condensação ocorrem durante a etapa de aquecimento, certifique-se que os tubos de centrífuga de 1,5 mL são tampados completamente. - Girar os fago tratado termicamente da etapa 3.3 a 18.534 x g por 10 s. Mix do fago, colocando o tubo no vortex definidos no modo de toque-ativação de 10 s e girar a 18.534 x g durante 10 s novamente.
Nota: O ciclo de mistura-spin-spin é ter certeza de que as amostras do fago aquecida estejam bem misturadas. - Arrefecer os tubos de amostra em temperatura ambiente (RT), deixando-os no experimento banco ou loja a 4 ° C no frigorífico durante a noite.
Nota: O experimento pode ser pausado para esta etapa.
4. prepare qPCR reações
Nota: Uma reação de PCR é para uma amostra. Cada reação de PCR contém 5 µ l de qPCR mestre da mistura (ver materiais), 1 µ l de 5 µM cartilha par mix, 2 µ l de H2O e 2 µ l da amostra de fago T7 tratado termicamente. Para várias reações de PCR, todos os reagentes, exceto a amostra do fago são pré-misturados em um tubo de 1,5 mL. O volume de cada reagente em pré-mistura depende do número de amostras do fago para qPCR.
- Prepare a pré-mistura de qPCR para 10 amostras de fago biopanned de concentração desconhecida e 7 amostras das concentrações padrão em triplicado. Como resultado, existem 51 amostras totais e, portanto, 51 qPCR reações (ou seja, uma reação por amostra). Para 51 reações, prepare um premix de µ l 255 de qPCR mestre mix (51 x 5 µ l), 51 µ l de primers (51 x 1 µ l) e 102 µ l de H2O (51 x 2 µ l).
Nota: É aconselhável preparar algumas reações de PCR extras para compensar a perda de volume durante a mistura PCR em cada poço da placa de 96 poços qPCR de alíquotas. - Alíquota de 8 µ l de pré-mistura PCR em cada poço da placa de 96 poços qPCR para um total de 51 poços (ou seja, 51 reações qPCR). Não há nenhuma necessidade de mudar dicas durante alíquotas.
- Adicionar 2 µ l das amostras de fago T7 tratado termicamente (passo 3.4) ou referência de fago T7 DNA (passo 2.1) a cada poço (Figura 3B); trocar dicas ao adicionar diferentes amostras de DNA para o bem para evitar a contaminação cruzada. Além disso, dispense as 2 amostras de DNA µ l debaixo da superfície da qPCR pré-mistura (i.e., em pré-mistura PCR 8 µ l).
- A placa de qPCR com película adesiva do selo.
Nota: Este passo é fundamental, independentemente do tipo de placa de qPCR. Use o kit recomendado para selar a placa completamente; caso contrário, qualquer região não seladas na placa causará perda de volume durante a PCR ciclismo. - Embrulhe a placa qPCR com folha de alumínio para minimizar o fotobranqueamento fluorescência e prosseguir para executá-lo no equipamento qPCR.
5. qPCR condições de ciclismo
Nota: condições de ciclismo qPCR foram criadas no equipamento qPCR específicas (ver Tabela de materiais).
- Execute a placa de 96 poços qPCR no equipamento qPCR. No equipamento, selecione configurações do menu para configurar as seguintes condições experimentais (Figura 3).
- Configurar as condições da seguinte forma para um volume de amostra de 10 µ l de ciclismo: um ciclo a 50 ° C por 2 min, um ciclo a 95 ° C por 2 min, seguido de 40 ciclos de (95 ° C por 15 s, 60 ° C por 1 min). Depois, execute as configurações de curva de derretimento: um ciclo de 95 ° C por 15 s, 60 ° C por 1 min e 95 ° C 15 s.
- Proceda para analisar os dados, conforme descrito na etapa 6.
6. analisar o dados brutos qPCR e converter de qPCR Ct valor para gc / µ l para bacteriófagos T7
- Analisar os dados brutos qPCR e avaliar a qualidade dos dados, analisando as parcelas características dos dados brutos (Figura 4).
Nota: Amplificação enredo, trama multicomponente e derreter curva enredo são que a característica chave parcelas para validar a qualidade dos dados de qPCR. -
Calcule os média limiares de ciclo (TC) de amostras de três vias em cada concentração de curva padrão (passo 2.1).
- Parcela média Ct (Y) contra log (gc / µ l) (X) para obter a curva linear Y = kX + b (k é o declive da curva linear, b é o intercepto).
- Calcular o coeficiente linear14 R2 (R2 deve ser > 0,99).
- Use a curva padrão Y = kX + b para calcular o número de fago T7 nas amostras biopanning (i.e., fago selecionado contra CF-como muco) no gc / µ l (Figura 5).
Nota: k é o declive da curva padrão. - Calcular a eficiência de amplificação pela seguinte equação
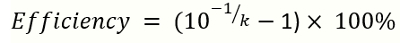 (2)
(2)
Nota: k é o declive da curva padrão gerado a partir de referência DNA qPCR. A faixa ideal de eficiência de amplificação é 90-110%.
Resultados
Ferramentas de design diferente da primeira demão podem ser usadas para projetar primers qPCR. Normalmente, o projeto da primeira demão programas têm seus próprios algoritmos internos para calcular e validar os parâmetros chaves do cartilhas, por exemplo, % de GC, Tm, dímero da primeira demão ou formação de grampo de cabelo, etc. em geral, os critérios-chave são semelhantes em diferentes ferramentas de projeto da primeira demão e primers podem ser projetados ...
Discussão
Desenvolvemos métodos de qPCR para quantificação de DNA genômico do fago5, e aqui descrevemos e adaptou um método de qPCR para enumerar fago T7 selecionado contra uma barreira de muco tipo CF. Informações mínimas para diretrizes de publicação do quantitativo real-time PCR experimentos (MIQE) foram adaptados para desenvolver e validar o método de qPCR para enumeração de T7 fago15. O protocolo que desenvolvemos para quantificar o fago de experimentos biopanning ?...
Divulgações
Os autores não têm nada para divulgar.
Agradecimentos
Este trabalho foi apoiado por PhRMA Foundation Research Grant Starter e nacional do coração, pulmão e Instituto de sangue do institutos nacionais da saúde concedem sob número prêmio R01HL138251.
Materiais
| Name | Company | Catalog Number | Comments |
| Materials and Reagents | |||
| Primers for T7 genomic DNA | IDT | F: CCTCTTGGGAGGAAGAGATTTG R: TACGGGTCTCGTAGGACTTAAT | |
| T7 Select packaging control DNA | EMD Millipore | 69679-1UG | |
| MicroAmp optical 96-well reaction plate | ThermoFisher Scientific | N8010560 | |
| qPCR master mix--Power up SYBR Green master mix | Applied biosystems | A25742 | |
| MicroAmp optical adhesive film kit | ThermoFisher Scientific | 4313663 | |
| T7Select 415-1 Cloning Kit | EMD Millipore | 70015 | User protocols : http://www.emdmillipore.com/US/en/product/T7Select-415-1-Cloning-Kit,EMD_BIO-70015#anchor_USP |
| DNase I solution | ThermoFisher Scientific | 90083 | |
| 24-well transwell plate | Corning | 3472 | |
| UltraPure DNase/RNase-Free Distilled H2O | Invitrogen | 10977015 | |
| Phosphate Buffered Saline (1x) | Corning | 21040CV | |
| Name | Company | Catalog Number | Comments |
| Equipments | |||
| ViiA7 Real-Time PCR System with Fast 96-Well Block | ThermoFisher Scientific | 4453535 | |
| Heraeus Pico 21 Microcentrifuge | ThermoFisher Scientific | 75002415 | |
| Fisherbrand Digital Vortex Mixer | Fisher Scientific | 02-215-370 | |
| HERMO SCIENTIFIC Multi-Blok Heater | ThermoFisher Scientific | Model:2001 | |
| Sorvall Legend X1 Centrifuge | ThermoFisher Scientific | 75004220 | |
| M-20 Microplate Swinging Bucket Rotor | ThermoFisher Scientific | 75003624 | |
| Name | Company | Catalog Number | Comments |
| Software | |||
| QuantStudio Real-time PCR software | ThermoFisher Scientific | v1.2 | |
| Real-time qPCR primer design | IDT | ||
| OligoAnalyzer 3.1 | IDT |
Referências
- Smith, G. Filamentous fusion phage: novel expression vectors that display cloned antigens on the virion surface. Science. 228 (4705), 1315-1317 (1985).
- Smith, G. P., Petrenko, V. A. Phage Display. Chemical Reviews. 97 (96), 391-410 (1997).
- Sergeeva, A., Kolonin, M. G., Molldrem, J. J., Pasqualini, R., Arap, W. Display technologies: Application for the discovery of drug and gene delivery agents. Advanced Drug Delivery Reviews. 58 (15), 1622-1654 (2006).
- Dias-Neto, E., et al. Next-generation phage display: Integrating and comparing available molecular tools to enable costeffective high-throughput analysis. PLoS ONE. 4 (12), 1-11 (2009).
- Peng, X., Nguyen, A., Ghosh, D. Quantification of M13 and T7 bacteriophages by TaqMan and SYBR green qPCR. Journal of Virological Methods. 252 (June 2017), 100-107 (2017).
- Khan, M. S., Pande, T., Mvan de Ven, T. G. Qualitative and quantitative detection of T7 bacteriophages using paper based sandwich ELISA. Colloids and Surfaces B: Biointerfaces. 132, 264-270 (2015).
- Brasino, M., Lee, J. H., Cha, J. N. Creating highly amplified enzyme-linked immunosorbent assay signals from genetically engineered bacteriophage. Analytical Biochemistry. 470, 7-13 (2014).
- Yu, X., Burgoon, M., Shearer, A., Gilden, D. Characterization of phage peptide interaction with antibody using phage mediated immuno-PCR. J immunol Methods. 326 (1-2), 33-40 (2007).
- Lehmusvuori, A., Manninen, J., Huovinen, T., Soukka, T., Lamminmäki, U. Homogenous M13 bacteriophage quantification assay using switchable lanthanide fluorescence probes. BioTechniques. 53 (5), 301-303 (2012).
- Schlaman, H. R. M., et al. Analysis of interactions of signaling proteins with phage-displayed ligands by fluorescence correlation spectroscopy. Journal of Biomolecular Screening. 13 (8), 766-776 (2008).
- Tjhung, K. F., Burnham, S., Anany, H., Griffiths, M. W., Derda, R. Rapid enumeration of phage in monodisperse emulsions. Analytical Chemistry. 86 (12), 5642-5648 (2014).
- Reitinger, S., Petriv, O. I., Mehr, K., Hansen, C. L., Withers, S. G. Purification and quantitation of bacteriophage M13 using desalting spin columns and digital PCR. Journal of Virological Methods. 185 (1), 171-174 (2012).
- . T7Select® Biopanning Kit Available from: https://www.emdmillipore.com/US/en/product/T7Select-Biopanning-Kit (2018)
- Schneider, A., Hommel, G., Blettner, M. Linear Regression Analysis. Deutsches Ärzteblatt international. 107 (44), 776-782 (2010).
- Bustin, S. A., Benes, V., Garson, J. A., Hellemans, J., Huggert, J. The MIQE Guidelines: Minimun information for publication of quantitative real-time PCR experiments. Clinical Chemitry. 55 (4), 611-622 (2009).
- Lock, M., Alvira, M. R., Chen, S. -. J., Wilson, J. M. Absolute Determination of Single-Stranded and Self-Complementary Adeno-Associated Viral Vector Genome Titers by Droplet Digital PCR. Human Gene Therapy Methods. 25 (2), 115-125 (2014).
- Fittipaldi, M., et al. Discrimination of infectious bacteriophage T4 virus by propidium monoazide real-time PCR. Journal of Virological Methods. 168 (1-2), 228-232 (2010).
- Lodder, W. J., et al. Reduction of bacteriophage MS2 by filtration and irradiation determined by culture and quantitative real-time RT-PCR. Journal of Water and Health. 11 (2), 256-266 (2013).
- Brown-Jaque, M., et al. Antibiotic resistance genes in phage particles isolated from human feces and induced from clinical bacterial isolates. International Journal of Antimicrobial Agents. , (2017).
- Naveca, F. G., et al. Multiplexed reverse transcription real-time polymerase chain reaction for simultaneous detection of Mayaro, Oropouche, and oropouche-like viruses. Memorias do Instituto Oswaldo Cruz. 112 (7), 510-513 (2017).
- Jia, J., et al. Simultaneous detection and differentiation of human parvovirus B19 and human parvovirus 4 by an internally controlled multiplex quantitative real-time PCR. Molecular and Cellular Probes. 36, 50-57 (2017).
- Bliem, R., et al. A novel triplex quantitative pcr strategy for quantification of toxigenic and nontoxigenic Vibrio cholerae in aquatic environments. Applied and Environmental Microbiology. 81 (9), 3077-3085 (2015).
- Wan, Z., Goddard, N. L. Competition Between Conjugation and M13 Phage Infection in Escherichia coli in the Absence of Selection Pressure: A Kinetic Study. G3 Genes|Genomes|Genetics. 2 (10), 1137-1144 (2012).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados