Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Quantitative PCR von Bakteriophagen T7 aus Biopanning
In diesem Artikel
Zusammenfassung
Eine reproduzierbare und genaue Zeit effizientere quantitative PCR (qPCR) Methode aufzuzählenden T7 Bakteriophagen wird hier beschrieben. Das Protokoll beschreibt klar Phagen genomische DNA-Vorbereitung, PCR Reaktion Vorbereitung, qPCR Radsport Bedingungen und qPCR Datenanalyse.
Zusammenfassung
Dieses Protokoll beschreibt den Einsatz von quantitativen PCR (qPCR) aufzuzählenden Phagen T7 von Phagen Auswahl Experimente (z.B. "Biopanning"). qPCR ist eine Fluoreszenz basierenden Ansatz, DNA zu quantifizieren, und es hier angepasst, Phagen Genome als Proxy für den Phagen Partikel zu quantifizieren. In diesem Protokoll wird eine einfache Phagen DNA Vorbereitung Methode beschrieben mit Hochtemperatur-Heizung ohne zusätzliche DNA-Reinigung. Die Methode benötigt nur kleine Mengen an wärmebehandelten Phagen und Kleinmengen der qPCR Reaktion. qPCR ist Hochdurchsatz- und schnell, in der Lage, zu verarbeiten und erhalten Daten von einer 96-Well-Platte von Reaktionen im 2 – 4 h im Vergleich zu anderen Ansätzen Phagen-Enumeration, qPCR ist Zeit effizienter. Hier dient der qPCR aufzuzählenden T7 Phagen aus Biopanning gegen in-vitro- Mukoviszidose-artigen Schleim Modell identifiziert. Die qPCR-Methode kann ausgedehnt werden um Phagen T7 aus anderen Experimenten, einschließlich andere Arten von Biopanning zu quantifizieren (zB., immobilisiert Proteinbindung, in-vivo -Phagen-Screening) und anderen Quellen (z. B. Wasser-Systeme oder Körperflüssigkeiten). Zusammenfassend kann dieses Protokoll geändert werden, um DNA-gekapselte Viren quantifizieren.
Einleitung
Bakteriophagen (Phagen)-Display-Technologie, entwickelt von George Smith 1985 ist ein leistungsfähiges, Hochdurchsatz-Ansatz Peptid oder Protein Liganden gegen Ziele oder Rezeptoren von der Zellmembran, Krankheit Antigene, zellulare Organellen oder spezifische identifizieren Organe und Gewebe in den vergangenen zwei Jahrzehnten1,2. Hier zufällige Bibliotheken der Polypeptide oder einzelne Kette Antikörper auf die Hüllproteine des Phagen (in der Regel M13 oder T7) angezeigt werden, und spezifische Liganden identifiziert werden, von panning gegen immobilisierten Proteine in Vitro oder in vivo biologischen Systemen durch einen iterativen Auswahlprozess. Dann können die Liganden mit bildgebenden oder therapeutischen Wirkstoffen für Diagnose und Behandlung von Krankheiten3,4gekoppelt werden. Ist es wichtig, genau Auflisten von Phagen in mehrere Stufen der Biopanning: (1), Phagen zu quantifizieren, die an das Substrat gebunden und (2) zu quantifizieren Phagen um festzustellen, ob Anreicherung mit jeder Runde Auswahl (Phagen Anreicherung zeigt Biopanned Phagen-Affinität für das Ziel). Der derzeitige Goldstandard für Quantifizierung, Doppelschicht-Plaque-Assay, Herausforderungen mehrere; Es ist langwierig, umständlich und möglicherweise ungenau für eine große Anzahl von Proben. Daher hat unsere Fraktion eine sensible, reproduzierbare, präzise und zeitsparend quantitative Polymerase Kettenreaktion (qPCR) Methode, um M13 und T7 Phagen Partikel aus Biopanning5aufzählen entwickelt.
qPCR ist eine attraktive und machbare Methode, T7 und M13 Phagen genau zu quantifizieren. Da jedes einzelnen Phagen Partikel nur eine Kopie der genomischen DNA (DsDNA oder SsDNA) enthalten kann, entspricht einem Phagen-Genom ein Phagen Partikel; durch die Zahl der Phagen Genome zu quantifizieren, ist es möglich, die Zahl der Phagen zu quantifizieren. Während qPCR, fluoreszierende Reporter Farbstoffe binden Phagen genomischer DNA unspezifisch oder speziell durch Sequenz-spezifische Primer während PCR Verstärkung und die Fluoreszenz Signal erhöht sich mit jeder Runde der Verstärkung. Wenn das Fluoreszenzsignal erreicht die Schwelle, die Runde/Zyklus der Verstärkung wird als der Schwellenwert-Zyklus (Ct) vermerkt. Die bekannten Konzentrationen von Referenz-Phagen DNA werden gegen ihre Ct-Werte herstellen eine Standardkurve geplottet. Mit der Ct-Werte von DNA-Proben der Standardkurve, können die Konzentrationen von Phagen interpoliert werden.
Während zahlreiche Strategien wurden zuvor entwickelt und intensiv genutzt, um Phagen aus Biopanning zu quantifizieren, hat jeder von ihnen spezifischen Herausforderungen. Die beliebtesten und konventionelle Methode ist Doppelschicht-Plaque-Assay. Hier, Host Bakterien mit Phagen vorbereitet in Verdünnungsreihen infiziert sind, sind auf einem festen Nährboden Substrat beschichtet und werden mit Agar überlagert; Phagen sind mit der Anzahl von Plaques gebildet (d. h. Plaque bildende Einheiten oder Pfu) auf der Agarplatte aufgezählt. Plaque-Assay ist sensibel aber mühsam, zeitaufwendig und ungenau, vor allem für zahlreiche Proben und mit hohen Konzentrationen5. Darüber hinaus wurden Enzym-linked Immunosorbentprobe Assays (ELISA) M13 und T7 Phagen Partikel6,7,8aufzählen angepasst. Hier werden Phagen bei verschiedenen Verdünnungen gebunden und auf ein festes Substrat (d. h. einer Mikrotestplatte) gebannt, sondiert mit Phagen-spezifische Antikörper und mithilfe von Reportern (z. B. Enzym empfindliche chromogene Substrate, Fluorophore) erkannt bestimmen Sie die Anzahl der Phagen Partikel vorhanden. Die anzeigen (z. B. Fluoreszenz, Absorption) der Proben können verwendet werden, zu unbekannte Konzentrationen gegen Phagen Standards bei bekannten Konzentrationen zu quantifizieren. Verschiedenen Phagen-basierte ELISAs entwickelt worden, aber sie haben mögliche Einschränkungen. Eine Gruppe entwickelte eine Papier-basierten Sandwich ELISA mit einer Quantifizierung-Palette, die überspannte sieben Größenordnungen (102-109 Pfu/mL); jedoch diese Methode erforderte mehrere Schritte der Antikörper-Beschichtung und dauerte bis einen ganzen Tag für den Assay-8. Unsere Fraktion auch entwickelt eine ELISA-Methode zur Quantifizierung der M13 Phagen Partikel aber hatte ein unempfindlicher Erfassungsbereich 106 bis 1011 Pfu/mL5. Schaltbare lanthanid Fluoreszenz Sonden wurden entwickelt, um M13 aufzählen und Quantifizierung Daten innerhalb von 20 min gewonnen werden; Dieser Test hat jedoch einen engen Dynamikbereich von 109 1012 Pfu/mL9. Eine Gruppe verwendet Rasterkraftmikroskopie M13 Phagen Partikel in Lösung aufzählen, aber dies erfordert erweiterte Elektronenmikroskopie und funktionierte nur in einem engen Bereich von Konzentrationen10. Eine weitere Studie zur trap fluoreszierender M13 und T4 Reporter Phagen und Phagen zählen, indem Sie die Anzahl der fluoreszierenden Tröpfchen Monodisperse Emulsionen; jedoch stellte dieser Ansatz auch eine schmale Quantifizierung reichen von 102 bis 106 Pfu/mL11. Während Tröpfchen, die digitale PCR verwendet wurde, um M13 Phagen zu quantifizieren, ist dieser Ansatz nicht in der Lage, zwischen der Anzahl der infektiösen und nicht infektiösen Phagen Partikel12zu unterscheiden.
Unsere Gruppe hat vor kurzem qPCR Methoden zum Auflisten von T7 und M13 Phagen aus Biopanning gegen ein Blut - Hirn-Schranke-Zelle-Modell mit zwei verschiedenen fluoreszierenden Reporter Farbstoffe5identifiziert entwickelt. Im Vergleich zu oben genannten Quantifizierungsmethoden, qPCR-Methoden, die wir entwickelt wurden Hochdurchsatz- und zeiteffizient zu zahlreichen Proben zu quantifizieren und in der Lage zu differenzieren zwischen infektiösen und nicht infektiösen Phagen mit DNase ich Vorbehandlung von der Phagen Proben. Wichtig ist, kann dieser Ansatz reproduzierbar und präzise M13 und T7 Bakteriophagen von Biopanning Proben quantifizieren. Um Anfänger Forscher auf dem Gebiet des Phagen qPCR zu leiten, beschreiben wir hier eine detaillierte qPCR-Methode, um T7 Phagen Partikel aus einer Cystein eingeschränkt Bibliothek (CX7C) gegen eine in-vitro- zystische Fibrose (CF) Schleim Barriere verrissen aufzählen. Aus dieser Arbeit kann qPCR-Methode erweitert werden um Phagen Partikel aus anderen Arten von Biopanning und aus anderen Quellen, einschließlich Wasser, Boden und Körperflüssigkeiten zu quantifizieren.
Access restricted. Please log in or start a trial to view this content.
Protokoll
(1) besser gekleideteres Design und Analyse für T7 Phagen genomischer DNA
- Primer für die Amplifikation der T7 Phagen genomischer DNA zu entwerfen.
Hinweis: F (vorwärts) und R (rückwärts) Primer (siehe Tabelle der Materialien) verstärken die T7 DNA-Sequenz befindet sich oberhalb der Bibliothek Variable Region (Abbildung 1). - Wählen Sie einen geeignete Grundierung-Analysator, Parameter der Primer, einschließlich Schmelztemperatur (Tm), prozentuale GC-Gehalt (GC %), Grundierung Dimere, Haarnadel Bildung und PCR-Eignung (Abbildung 1) zu bewerten.
- Bestellen Sie die Grundierung Oligonucleotides (Oligos), und bei der Ankunft, Aufschwemmen Sie lyophilisierter Oligos in DNase und RNase frei Ultrareines Wasser zu 100 µM Lager Konzentrationen in einem 1,5 mL Zentrifugenröhrchen, mehrere Monate bei-20 ° C lagern.
- Machen Sie mehrere Aliquote (rund 50 µL in jedem 1,5 mL-Röhrchen) von 100 µM-Grundierung-Lager.
Hinweis: Dieser Schritt dient zur häufigen Frost-Tau-wechseln zu verhindern.
2. bereiten Sie Standards für die Quantifizierung der T7 Phagen genomischer DNA
- Verdünnen Sie Seriell 0,1 µg/µL (1,0 x 108 fg/µL) Referenz T7-Phagen-DNA (gekauft, bei einer bekannten Konzentration von 0,1 µg/µL, siehe Materialien) 10-divisibel von 1,0 x 106 bis 1.0 x 100 fg/µL mit hochreinem H2O in 0,2 mL PCR-Röhrchen. Bereiten Sie jede Konzentration in einem Gesamtvolumen von 20 µL (Abbildung 2).
- Vortex-Röhren die PCR dafür ein gründliches mischen bei jedem Schritt der Verdünnung. Ändern Sie auch Tipps, wenn Sie die folgenden Verdünnung DNA-Probe hinzufügen.
Hinweis: Es wird empfohlen, neue Lösung von DNA-Standards für eine Standardkurve für jede Charge des Experiments qPCR vorzubereiten. - Verwenden Sie die Gleichung 1 (unten), konvertieren Sie zu Referenzzwecken T7 Phagen DNA-Konzentrationen von fg/µL Genom Kopien (gc) /µL (Abbildung 2).
- T7-Phagen-DNA-Konzentration im gc/µL =
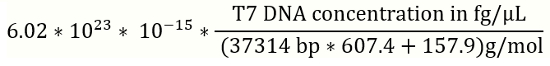 (1)
(1)
Hinweis: bp: Basenpaar; Referenz T7 genomischer DNA ist 37314 bp.
(3) vor der Behandlung des Phagen Proben vor qPCR Reaktion Vorbereitung
-
(Option 1) Pipettieren 20 µL einer T7 Klon (Phagen) ausgewählt aus Biopanning gegen in-vitro- zystische Fibrose (CF)-wie Schleim-Modell in 1,5 mL Zentrifuge Röhren. Hinzufügen von 1,25 Einheiten der DNase I (0,5 µL) zu jeweils 20 µL Probe.
Hinweis: T7 Phagen Cystein eingeschränkte Heptapeptid Bibliothek (CX7C) war Biopanned gegen CF-artigen Schleim beschichtet 24 Wohlen Membran Einsätze (siehe Tabelle der Materialien) für 15 Minuten. Weitere Details sind in den Abschnitt "Ergebnisse" zur Verfügung gestellt. Eine T7 Klon wurde für qPCR Probenvorbereitung aus Eluat gewählt.- Option 2: Pipettieren 200 µL T7 Klonen in einem 1,5 mL-Tuben, und fügen Sie 5 Einheiten der DNase ich
(2 µL) zu jeder Probe.
Hinweis: Die Lautstärke des Phagen Probe ausgewählt bei diesem Schritt richtet sich nach den Sammelband von Biopanning. Während des Protokolls auf der qPCR Biopanned Phagen konzentriert, kit denn Biopanning sind detailliert in der Benutzer-Protokoll von den Phagen T7 die Schritte (siehe Tabelle der Materialien)13.
- Option 2: Pipettieren 200 µL T7 Klonen in einem 1,5 mL-Tuben, und fügen Sie 5 Einheiten der DNase ich
- Verschließe die Phagen T7 Probenröhrchen (Schritt 3.1) und inkubieren sie bei 37 ° C für 10 min in einem beheizten Trockenbad (d. h. Heizblock).
- Inkubieren Sie Röhren (von Schritt 3.2) bei 100 ° C für 15 min in einem beheizten Trockenbad (Abb. 3A).
Achtung: Da Verdampfung und Kondensation während der Heizung Schritt auftreten, stellen Sie sicher, dass die 1,5 mL Zentrifuge Rohre komplett verschlossen werden. - Drehen der wärmebehandelten Phagen aus Schritt 3.3 18.534 x g für 10 S. Mix Phagen, indem das Rohr auf einem Vortex-Mixer eingestellt am Touch-Aktivierungsmodus für 10 s und Spin 18.534 x g für 10 s wieder.
Hinweis: Der Spin-Mix-Schleuder ist um sicherzustellen, dass die beheizten Phagen Proben gut vermischt sind. - Die Probenröhrchen bei Raumtemperatur (RT) indem man sie auf den Experiment-Bank oder Store bei 4 ° C im Kühlschrank über Nacht abkühlen.
Hinweis: Experiment kann an dieser Stelle angehalten werden.
4. bereiten Sie qPCR Reaktionen
Hinweis: Eine PCR Reaktion ist für eine Probe. Jeder PCR-Reaktion enthält 5 µL qPCR Master mix (siehe Material), 1 µL 5 µM Primer paar Mix, H2O 2 µL und 2 µL wärmebehandelte T7-Phagen-Probe. Für mehrere PCR-Reaktionen sind alle Reagenzien mit Ausnahme von Phagen Probe in eine 1,5 mL Tube vorgemischt. Das Volumen der einzelnen Reagenz in der Vormischung hängt von der Anzahl der Phagen Proben für qPCR.
- 10 Biopanned Phagen unbekannter Konzentration und 7 Proben von standard-Konzentrationen in dreifacher Ausfertigung der qPCR-Premix vorbereiten. Infolgedessen gibt es 51 gesamt Proben und daher 51 qPCR Reaktionen (d. h. eine Reaktion je Probe). 51 Reaktionen zu bereiten eine Vormischung von 255 µL qPCR master-Mix (51 x 5 µL), 51 µL von Primern (51 x 1 µL) und H2O 102 µL (51 x 2 µL).
Hinweis: Es wird empfohlen, ein paar zusätzliche PCR-Reaktionen zum Ausgleich der Volumenverlust während Aliquotierung des PCR-Mixes in jede Vertiefung der qPCR 96-Well-Platte vorzubereiten. - Aliquoten 8 µL des PCR-Vormischung in jede Vertiefung der qPCR 96-Well-Platte für eine Gesamtmenge von 51 Brunnen (d. h. 51 qPCR Reaktionen). Es gibt keine Spitzenwechsel während Aliquotierung.
- Fügen Sie 2 µL der wärmebehandelten T7 Phagen Proben (Schritt 3.4) oder T7 Phagen Referenz DNA (Schritt 2.1) in jede Vertiefung (Abb. 3 b); Tipps zu ändern, wenn Sie verschiedene DNA-Proben zum Brunnen, um Kreuzkontaminationen zu vermeiden. Darüber hinaus verzichten Sie die 2 µL DNA-Proben unter der Oberfläche der qPCR Premix (d. h. in den 8 µL PCR Premix).
- Die qPCR Dichtplatte mit Klebefilm.
Hinweis: Dieser Schritt ist wichtig, unabhängig von der Art der qPCR-Platte. Verwenden Sie das empfohlene Kit, um die Platte vollständig zu verschließen; Andernfalls wird eine nicht versiegelte Region in der Platte Volumenverlust führen, während der PCR Radfahren. - Wickeln Sie die qPCR-Platte mit Aluminium-Folie, Immunofluoreszenz Fluoreszenz zu minimieren und fahren Sie mit auf die qPCR Ausrüstung ausgeführt.
5. qPCR Cycling Bedingungen
Hinweis: qPCR Radsport Bedingungen wurden auf die spezifischen qPCR Ausrüstung eingerichtet (siehe Tabelle der Materialien).
- Die qPCR 96-Well-Platte auf die qPCR-Ausrüstung ausgeführt. Am Gerät wählen Sie Einstellungen aus dem Menü zum Einrichten der folgenden experimentellen Bedingungen (Abbildung 3).
- Bedingungen wie folgt für ein Probenvolumen von 10 µL Radfahren eingerichtet: ein Zyklus bei 50 ° C für 2 min, ein Zyklus bei 95 ° C für 2 min, gefolgt von 40 Zyklen (95 ° C für 15 s, 60 ° C für 1 min). Danach laufen die Schmelze Kurveneinstellungen: ein Zyklus von 95 ° C für 15 s, 1 min, 60 ° C und 95 ° C 15 s.
- Fahren Sie fort, um die Daten zu analysieren, wie in Schritt 6 beschrieben.
6. der qPCR Rohdaten analysieren und Konvertieren von qPCR Ct Wert auf gc/µL für T7 Bakteriophagen
- Analysieren Sie die rohen qPCR-Daten und bewerten Sie die Qualität der Daten durch die Analyse der charakteristischen Grundstücke der raw-Daten (Abbildung 4).
Hinweis: Amplification Plot, Mehrkomponenten Plot und Schmelze Kurve Handlung sind das Hauptmerkmal-plots, um die Datenqualität der qPCR validiert. -
Berechnen Sie die mittlere Zyklus (Ct) Schwellenwerte von dreifacher Proben bei jeder Konzentration der Standardkurve (Schritt 2.1).
- Plot-mittlere Ct (Y) gegen Log (gc/µL) (X) zu den linearen Kurve Y = kX + b (k ist die Steigung der linearen Kurve, b ist der Schnittpunkt).
- Berechnen Sie die lineare Koeffizient14 R2 (R2 muss sein > 0,99).
- Verwenden die Standardkurve Y = kX + b, um die Anzahl der Phagen T7 in den Biopanning Proben (z.B. Phagen gewählt gegen CF-artigen Schleim) im gc/µL (Abbildung 5) zu berechnen.
Hinweis: k ist die Steigung der Standardkurve. - Die Effizienz der Verstärkung durch die folgende Gleichung berechnen
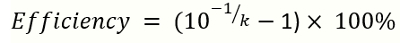 (2)
(2)
Hinweis: k ist die Steigung der Standardkurve generiert aus Referenz DNA qPCR. Die ideale Verstärkung Effizienz ist 90 – 110 %.
Access restricted. Please log in or start a trial to view this content.
Ergebnisse
Verschiedene Primer-Design-Tools lässt sich qPCR Primer zu entwerfen. In der Regel ähneln besser gekleideteres Design Programme haben ihre eigenen eingebauten Algorithmen berechnen und validieren die Schlüsselparameter für die Grundierungen, z. B. GC %, Tm, Grundierung Dimer oder Haarnadel Bildung usw. in der Regel, die wichtigsten Kriterien in verschiedenen Grundierung-Design-Tools und Primer ist gestaltbar nach ihren Anweisungen. Grundierung BLAST kann verwendet we...
Access restricted. Please log in or start a trial to view this content.
Diskussion
Wir qPCR-Methoden zur Quantifizierung von genomische DNA-Phagen5entwickelt, und wir hier beschrieben und eine qPCR-Methode zum Auflisten der T7 Phagen gewählt gegen eine CF-wie Schleim-Barriere angepasst. Mindestangaben für die Veröffentlichung der Quantitative Real-Time PCR Experimente (MIQE) Leitlinien wurden angepasst, um Entwicklung und Validierung der qPCR-Methode für die Enumeration von T7 Phagen15. Das Protokoll, die wir entwickelt, um die Phagen aus Biopanning E...
Access restricted. Please log in or start a trial to view this content.
Offenlegungen
Die Autoren haben nichts preisgeben.
Danksagungen
Diese Arbeit wurde von PhRMA Stiftung Forschungsstipendium Starter unterstützt und das National Heart, Lung and Blood Institute der National Institutes of Health unter prämiennummer R01HL138251 gewähren.
Access restricted. Please log in or start a trial to view this content.
Materialien
| Name | Company | Catalog Number | Comments |
| Materials and Reagents | |||
| Primers for T7 genomic DNA | IDT | F: CCTCTTGGGAGGAAGAGATTTG R: TACGGGTCTCGTAGGACTTAAT | |
| T7 Select packaging control DNA | EMD Millipore | 69679-1UG | |
| MicroAmp optical 96-well reaction plate | ThermoFisher Scientific | N8010560 | |
| qPCR master mix--Power up SYBR Green master mix | Applied biosystems | A25742 | |
| MicroAmp optical adhesive film kit | ThermoFisher Scientific | 4313663 | |
| T7Select 415-1 Cloning Kit | EMD Millipore | 70015 | User protocols : http://www.emdmillipore.com/US/en/product/T7Select-415-1-Cloning-Kit,EMD_BIO-70015#anchor_USP |
| DNase I solution | ThermoFisher Scientific | 90083 | |
| 24-well transwell plate | Corning | 3472 | |
| UltraPure DNase/RNase-Free Distilled H2O | Invitrogen | 10977015 | |
| Phosphate Buffered Saline (1x) | Corning | 21040CV | |
| Name | Company | Catalog Number | Comments |
| Equipments | |||
| ViiA7 Real-Time PCR System with Fast 96-Well Block | ThermoFisher Scientific | 4453535 | |
| Heraeus Pico 21 Microcentrifuge | ThermoFisher Scientific | 75002415 | |
| Fisherbrand Digital Vortex Mixer | Fisher Scientific | 02-215-370 | |
| HERMO SCIENTIFIC Multi-Blok Heater | ThermoFisher Scientific | Model:2001 | |
| Sorvall Legend X1 Centrifuge | ThermoFisher Scientific | 75004220 | |
| M-20 Microplate Swinging Bucket Rotor | ThermoFisher Scientific | 75003624 | |
| Name | Company | Catalog Number | Comments |
| Software | |||
| QuantStudio Real-time PCR software | ThermoFisher Scientific | v1.2 | |
| Real-time qPCR primer design | IDT | ||
| OligoAnalyzer 3.1 | IDT |
Referenzen
- Smith, G. Filamentous fusion phage: novel expression vectors that display cloned antigens on the virion surface. Science. 228 (4705), 1315-1317 (1985).
- Smith, G. P., Petrenko, V. A. Phage Display. Chemical Reviews. 97 (96), 391-410 (1997).
- Sergeeva, A., Kolonin, M. G., Molldrem, J. J., Pasqualini, R., Arap, W. Display technologies: Application for the discovery of drug and gene delivery agents. Advanced Drug Delivery Reviews. 58 (15), 1622-1654 (2006).
- Dias-Neto, E., et al. Next-generation phage display: Integrating and comparing available molecular tools to enable costeffective high-throughput analysis. PLoS ONE. 4 (12), 1-11 (2009).
- Peng, X., Nguyen, A., Ghosh, D. Quantification of M13 and T7 bacteriophages by TaqMan and SYBR green qPCR. Journal of Virological Methods. 252 (June 2017), 100-107 (2017).
- Khan, M. S., Pande, T., Mvan de Ven, T. G. Qualitative and quantitative detection of T7 bacteriophages using paper based sandwich ELISA. Colloids and Surfaces B: Biointerfaces. 132, 264-270 (2015).
- Brasino, M., Lee, J. H., Cha, J. N. Creating highly amplified enzyme-linked immunosorbent assay signals from genetically engineered bacteriophage. Analytical Biochemistry. 470, 7-13 (2014).
- Yu, X., Burgoon, M., Shearer, A., Gilden, D. Characterization of phage peptide interaction with antibody using phage mediated immuno-PCR. J immunol Methods. 326 (1-2), 33-40 (2007).
- Lehmusvuori, A., Manninen, J., Huovinen, T., Soukka, T., Lamminmäki, U. Homogenous M13 bacteriophage quantification assay using switchable lanthanide fluorescence probes. BioTechniques. 53 (5), 301-303 (2012).
- Schlaman, H. R. M., et al. Analysis of interactions of signaling proteins with phage-displayed ligands by fluorescence correlation spectroscopy. Journal of Biomolecular Screening. 13 (8), 766-776 (2008).
- Tjhung, K. F., Burnham, S., Anany, H., Griffiths, M. W., Derda, R. Rapid enumeration of phage in monodisperse emulsions. Analytical Chemistry. 86 (12), 5642-5648 (2014).
- Reitinger, S., Petriv, O. I., Mehr, K., Hansen, C. L., Withers, S. G. Purification and quantitation of bacteriophage M13 using desalting spin columns and digital PCR. Journal of Virological Methods. 185 (1), 171-174 (2012).
- T7Select Biopanning Kit. , Millipore Sigma. Available from: https://www.emdmillipore.com/US/en/product/T7Select-Biopanning-Kit,EMD_BIO-70018#anchor_USP (2018).
- Schneider, A., Hommel, G., Blettner, M. Linear Regression Analysis. Deutsches Ärzteblatt international. 107 (44), 776-782 (2010).
- Bustin, S. A., Benes, V., Garson, J. A., Hellemans, J., Huggert, J. The MIQE Guidelines: Minimun information for publication of quantitative real-time PCR experiments. Clinical Chemitry. 55 (4), 611-622 (2009).
- Lock, M., Alvira, M. R., Chen, S. -J., Wilson, J. M. Absolute Determination of Single-Stranded and Self-Complementary Adeno-Associated Viral Vector Genome Titers by Droplet Digital PCR. Human Gene Therapy Methods. 25 (2), 115-125 (2014).
- Fittipaldi, M., et al. Discrimination of infectious bacteriophage T4 virus by propidium monoazide real-time PCR. Journal of Virological Methods. 168 (1-2), 228-232 (2010).
- Lodder, W. J., et al. Reduction of bacteriophage MS2 by filtration and irradiation determined by culture and quantitative real-time RT-PCR. Journal of Water and Health. 11 (2), 256-266 (2013).
- Brown-Jaque, M., et al. Antibiotic resistance genes in phage particles isolated from human feces and induced from clinical bacterial isolates. International Journal of Antimicrobial Agents. , (2017).
- Naveca, F. G., et al. Multiplexed reverse transcription real-time polymerase chain reaction for simultaneous detection of Mayaro, Oropouche, and oropouche-like viruses. Memorias do Instituto Oswaldo Cruz. 112 (7), 510-513 (2017).
- Jia, J., et al. Simultaneous detection and differentiation of human parvovirus B19 and human parvovirus 4 by an internally controlled multiplex quantitative real-time PCR. Molecular and Cellular Probes. 36, 50-57 (2017).
- Bliem, R., et al. A novel triplex quantitative pcr strategy for quantification of toxigenic and nontoxigenic Vibrio cholerae in aquatic environments. Applied and Environmental Microbiology. 81 (9), 3077-3085 (2015).
- Wan, Z., Goddard, N. L. Competition Between Conjugation and M13 Phage Infection in Escherichia coli in the Absence of Selection Pressure: A Kinetic Study. G3 Genes|Genomes|Genetics. 2 (10), 1137-1144 (2012).
Access restricted. Please log in or start a trial to view this content.
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten