Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
PCR cuantitativa de bacteriófago T7 de Biopanning
En este artículo
Resumen
Reproducible, exacto y tiempo eficiente cuantitativo PCR (qPCR) describe un método para enumerar bacteriófago T7 es aquí. El protocolo describe claramente la preparación de ADN genómico de fago, preparación de reacción de PCR, qPCR ciclismo condiciones y análisis de datos qPCR.
Resumen
Este protocolo describe el uso de la PCR cuantitativa (qPCR) para enumerar fagos T7 de experimentos de selección de fagos (es decir, "biopanning"). qPCR es un enfoque basado en la fluorescencia para cuantificar ADN, y aquí, se adapta a cuantificar genomas de fagos como proxy para las partículas de fago. En este protocolo, se describe un método de preparación de DNA de fago fácil usando calefacción de alta temperatura sin purificación adicional de ADN. El método sólo necesita pequeñas cantidades de phages tratada térmicamente y volúmenes pequeños de la reacción de la qPCR. qPCR es alto rendimiento y rápido, capaz de procesar y obtener los datos de una placa de 96 pocillos de reacciones en 2 – 4 h. en comparación a otros métodos de enumeración de fagos, qPCR es más eficiente en tiempo. Aquí, qPCR se utiliza para enumerar fagos T7 identificadas biopanning contra vitro moco de la fibrosis quística como modelo. El método de qPCR puede ampliarse para cuantificar fagos T7 de otros experimentos, incluyendo otros tipos de biopanning (e.g., proyección de fago inmovilizada la Unión a proteínas, in vivo ) y otras fuentes (por ejemplo, los sistemas de agua u otros líquidos corporales). En Resumen, este protocolo puede modificarse para cuantificar cualquier virus ADN encapsulado.
Introducción
Tecnología de visualización de bacteriófagos (fagos), desarrollada por George Smith en 1985, es un enfoque potente y alto rendimiento para identificar péptidos o proteínas ligandos contra objetivos o receptores de la membrana celular, antígenos de la enfermedad, organelos celulares o específicos órganos y tejidos en los últimos dos décadas1,2. Aquí, al azar bibliotecas de polipéptidos o anticuerpos de cadena simple se muestran en las proteínas de la capa de phages (típicamente M13 o T7) y ligandos específicos pueden ser identificados de bateo contra proteínas inmovilizadas en vitro o en vivo sistemas biológicos a través de un proceso iterativo de selección. Entonces, los ligandos pueden ser juntados con agentes terapéuticos o proyección de imagen para el diagnóstico y tratamiento de enfermedades3,4. Es importante enumerar con precisión los phages durante varios pasos de biopanning: (1) cuantificar fagos que se unen al sustrato y (2) cuantificar los phages para determinar si hay enriquecimiento con cada ronda de selección (enriquecimiento phage indica biopanned afinidad de fagos para el destino). El estándar de oro actual para la cuantificación, ensayo de placa de doble capa, presenta múltiples desafíos; es tedioso, engorroso y potencialmente inexacto para un gran número de muestras. Por lo tanto, nuestro grupo ha desarrollado un método de (qPCR) la reacción en cadena de la polimerasa cuantitativa sensible, reproducible, precisa y eficiente para enumerar las partículas de fago M13 y T7 de biopanning5.
qPCR es un método atractivo y factible de cuantificar con precisión fagos T7 y M13. Puesto que cada partícula de fago individual sólo puede contener una copia de la DNA genomic (dsDNA o ssDNA), un genoma phage es equivalente a una partícula de fago; al cuantificar el número de genomas de fagos, es posible cuantificar el número de fagos. Durante qPCR, reportero fluorescente tintes atan phage de la DNA genomic no específicamente o específicamente a través de cebadores específicos de secuencia durante la amplificación por PCR y la fluorescencia señal aumenta con cada ronda de amplificación. Cuando la señal de fluorescencia alcanza el umbral, que ronda/ciclo de amplificación se observa como el ciclo umbral (Ct). Las concentraciones conocidas de fago ADN de referencia se trazan contra sus valores de Ct para establecer una curva estándar. Usando la curva estándar con los valores de Ct de muestras de ADN, las concentraciones de fagos pueden ser interpoladas.
Mientras que numerosas estrategias han sido previamente desarrollados y ampliamente utilizado para cuantificar los phages de biopanning, cada uno de ellos tiene retos específicos. El método más popular y convencional es el ensayo de placa de doble capa. Aquí, host bacterias infectan con phages preparados en diluciones seriadas, se platean en un sustrato de agar sólido y son overlaid con agar; los phages son enumerados con el número de las placas formadas (es decir, unidades formadoras de placa o pfu) en la placa de agar. Ensayo de placa es sensible pero tedioso, lento e inexacto, especialmente para muestras numerosas y con altas concentraciones de5. Además, los análisis enzima-ligado del inmunosorbente (ELISA) han sido adaptados para enumerar M13 y T7 fagos partículas6,7,8. Aquí, están obligados los phages en diferentes diluciones y capturado en un sustrato sólido (es decir, una microplaca), sondeado con anticuerpos específicos del fago y detectado usando Reporteros (p. ej., enzimas sustratos cromogénicos sensible, fluoróforos) para determinar el número de partículas de fago presente. Las lecturas (por ejemplo, fluorescencia, absorbancia) de muestras pueden utilizarse para cuantificar las concentraciones desconocidas contra estándares de fago en concentraciones conocidas. Se han desarrollado ELISAs basados en fagos diferentes pero tienen limitaciones potenciales. Un grupo desarrolló un sándwich en papel ELISA con un rango de cuantificación que abarcó siete órdenes de magnitud (102-109 pfu/mL); sin embargo, este método requiere varios pasos de la capa de anticuerpo y tomó hasta un día entero para el ensayo8. Nuestro grupo también desarrolló un método de ELISA para cuantificar las partículas de fago M13 pero tenía un alcance de detección menos sensible de 106 1011 pfu/mL5. Han desarrollado sondas de fluorescencia de lantánido conmutable para enumerar M13 y podrían obtenerse datos de cuantificación dentro de 20 minutos; sin embargo, este ensayo tiene un rango dinámico estrecho de 109 1012 pfu/mL9. Un grupo utilizó microscopía de fuerza atómica enumerar las partículas de fago M13 en solución, pero esto requiere microscopia electrónica avanzada y sólo trabajó dentro de un estrecho rango de concentraciones10. Otro estudio utilizado emulsiones monodispersa phages de reportero fluorescentes M13 y T4 y phages de la cuenta por el número de gotas fluorescentes; sin embargo, este enfoque también exhibió una gama estrecha de la cuantificación de 102 106 pfu/mL11. Mientras gota que PCR digital se ha utilizado para cuantificar los phages M13, este enfoque es incapaz de distinguir entre el número de partículas de fago infecciosas y no infecciosas12.
Nuestro grupo ha desarrollado métodos de qPCR para enumerar fagos T7 y M13 identificadas biopanning contra un modelo de celular de la barrera blood - brain con dos diferentes reportero fluorescente tintes5. Comparado con métodos de cuantificación ya mencionado, los métodos de qPCR desarrollamos fueron alto rendimiento y tiempo-eficiente para cuantificar muestras numerosas y capaces de diferenciar entre phages infecciosos y no infecciosas con DNasa I tratamiento previo de la muestras de fago. Lo importante, este enfoque puede reproducible y precisa cuantificar bacteriófagos M13 y T7 de muestras biopanning. Para guiar a los investigadores noveles en el área de qPCR de fago, aquí se describe un método detallado de qPCR para enumerar las partículas de fago T7 en una biblioteca con restricciones de cisteína (CX7C) panorámica contra una barrera de moco en vitro la fibrosis quística (FQ). De este trabajo, método de qPCR puede ampliarse para cuantificar las partículas de fago de otros tipos de biopanning y de otras fuentes, incluyendo agua, suelo y fluidos corporales.
Protocolo
1. primer diseño y análisis de ADN genómico de fago T7
- Diseño de cebadores para la amplificación de la DNA genomic de fago T7.
Nota: F (delantero) y R (reversa) cartillas (véase Tabla de materiales) amplifican la secuencia de la DNA T7 situado aguas arriba de la región variable de la biblioteca (figura 1). - Elegir un analizador de la cartilla apropiada para evaluar los parámetros de los iniciadores, incluyendo punto de fusión (Tm), por ciento contenido de GC (% GC), dímeros de primer, horquilla formación y adecuación PCR (figura 1).
- Los oligonucleótidos de cartilla (oligos) de la orden y a su llegada, resuspender los oligos liofilizado en agua ultra puro libre DNasa y Rnasa a concentraciones comunes de 100 μm en un tubo de centrífuga de 1.5 mL y almacenar a-20 ° C durante varios meses.
- Hacer varias alícuotas (alrededor de 50 μL en cada tubo 1,5 mL) de las acciones de la cartilla de 100 μm.
Nota: Este paso se utiliza para evitar frecuentes de congelación y descongelación.
2. preparar las normas para la cuantificación de ADN genómico de fago T7
- Diluir en serie referencia de 0,1 μg/μl (1.0 x 108 fg/μL) ADN de fago T7 (compró, en una concentración conocida de 0,1 μg/μl, ver materiales) 10 veces de 1.0 x 106 a 1.0 x 100 fg/μl con pureza de H2O en tubos PCR de 0,2 mL. Preparar cada concentración en un volumen total de 20 μl (figura 2).
- Vórtice el PCR los tubos para asegurar la mezcla cuidadosa en cada paso de dilución. También, cambiar consejos al agregar la muestra de ADN a la siguiente dilución.
Nota: Se recomienda preparar nueva solución de estándares de ADN para una curva estándar para cada lote del experimento qPCR. - Utilizar la ecuación 1 (abajo) para convertir para referencia del fago T7 concentraciones de ADN de fg/μl a copias de genoma (gc) /µL (figura 2).
- Concentración de DNA de fago T7 en gc/μl =
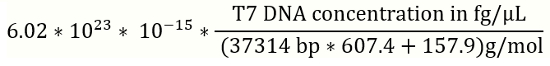 (1)
(1)
Nota: bp: pares; referencia T7 DNA genomic es 37314 bp.
3. pretratamiento de las muestras antes de Phage qPCR preparación de reacción
-
(Opción 1) Pipetear 20 μl de un clon de T7 (fago) de biopanning contra en vitro la fibrosis quística (CF)-como modelo moco en tubos de centrífuga de 1.5 mL. Añadir 1,25 unidades de DNasa I (0,5 μl) a cada muestra de 20 μl.
Nota: T7 phage cisteína heptapéptido con restricciones (CX7C) fue biopanned contra CF-como moco revestida en insertos de membrana de 24 pocillos (véase Tabla de materiales) durante 15 minutos. Más detalles se proporcionan en la sección de resultados. Un clon de T7 fue elegido para qPCR preparación de la muestra desde el efluente.- Opción 2: Pipetee 200 μL de la T7 clon en un tubos de 1.5 mL y agregar 5 unidades de la ADNsa I
(2 μL) a cada muestra.
Nota: El volumen de muestra de fago seleccionado en este paso depende del volumen recogido de biopanning. Mientras que el enfoque del protocolo es la qPCR de phage biopanned, los pasos para biopanning se detallan en el protocolo de usuario del fago T7 kit (véase Tabla de materiales)13.
- Opción 2: Pipetee 200 μL de la T7 clon en un tubos de 1.5 mL y agregar 5 unidades de la ADNsa I
- Tapa los tubos de muestras de fago T7 (paso 3.1) e incúbelos a 37 ° C durante 10 min en un baño seco calentado (es decir, bloque de calor).
- Incubar los tubos (paso 3.2) a 100 ° C durante 15 min en un baño seco caliente (Figura 3A).
PRECAUCIÓN: Puesto que la evaporación y la condensación ocurren durante la etapa de calentamiento, asegúrese de que los tubos de centrífuga de 1.5 mL están capped completamente. - Spin los phages con tratamiento térmico de paso 3.3 a 18.534 x g por 10 s. mezcla los phages colocando el tubo en un mezclador de tipo vórtex establecer en modo de activación por contacto para 10 s y vuelta a 18.534 x g durante 10 s otra vez.
Nota: El ciclo de centrifugado-mezcla-centrifugado es para asegurarse de que las muestras de fago climatizada estén bien mezcladas. - Enfriar los tubos de muestras a temperatura ambiente (RT) dejando en el Banco de experimento o de a 4 ° C en un refrigerador durante la noche.
Nota: El experimento se puede pausarse en este paso.
4. preparar reacciones de qPCR
Nota: Una reacción de PCR es para una muestra. Cada reacción de PCR contiene 5 μl de qPCR master de la mezcla (ver materiales), 1 μl de 5 μm primer par mezcla, 2 μl de H2O y 2 μl de la muestra de fago T7 tratada térmicamente. Para múltiples reacciones de PCR, todos los reactivos excepto la muestra de fagos son premezclados en un tubo de 1,5 mL. El volumen de cada reactivo en la premezcla depende del número de muestras de fago para qPCR.
- Preparar la premezcla de qPCR para 10 biopanned fago muestras de concentración desconocida y 7 muestras de concentraciones estándar por triplicado. Como resultado, hay 51 muestras total y por lo tanto, 51 qPCR reacciones (es decir, uno por muestra). Para 51 reacciones, preparar una mezcla preparada de antemano de 255 μl de qPCR master mix (51 x 5 μl), 51 μl de primers (51 x 1 μl) y 102 μl de H2O (51 x 2 μL).
Nota: Se recomienda preparar unas reacciones de PCR extras para compensar la pérdida de volumen en tomar la mezcla PCR en cada pocillo de la placa de 96 pocillos qPCR. - Alícuota 8 μl de premezcla de PCR en cada pocillo de la placa de 96 pocillos qPCR para un total de 51 pozos (es decir, 51 reacciones de qPCR). No hay necesidad para cambiar consejos durante Alicuotar.
- Añadir 2 μl de las muestras tratada térmicamente T7 phage (paso 3.4) o referencia del fago T7 DNA (paso 2.1) a cada pozo (figura 3B); cambiar consejos al agregar muestras de ADN en el pozo para evitar la contaminación cruzada. Además, dispensar las muestras de ADN de μl 2 debajo de la superficie de la premezcla de qPCR (es decir, en la premezcla PCR 8 μL).
- La placa de qPCR con la película adhesiva del sello.
Nota: Este paso es crítico, independientemente del tipo de placa de qPCR. Utilice el kit recomendado para sellar la placa completamente; de lo contrario, ninguna región no selladas en la placa causará pérdida de volumen durante la polimerización en cadena bicicleta. - Envolver la placa qPCR con papel de aluminio para minimizar el fotoblanqueo de fluorescencia y proceder a ejecutarlo en el equipo de qPCR.
5. qPCR ciclismo condiciones
Nota: condiciones ciclismo qPCR fueron instaladas en el equipo de qPCR específicos (véase Tabla de materiales).
- Ejecutar la placa de 96 pocillos qPCR en el equipo de qPCR. En el equipo, seleccione Configuración en el menú para configurar las siguientes condiciones experimentales (figura 3).
- Configurar ciclismo condiciones como sigue para un volumen de 10 μl de muestra: un ciclo a 50 ° C por 2 min, un ciclo a 95 ° C por 2 min, seguido de 40 ciclos de (95 ° C por 15 s, 60 ° C por 1 min). Después, ejecute la configuración de la curva de fusión: un ciclo de 95 ° C durante 15 s, 60 ° C por 1 min y 95 ° C 15 r.
- Proceder a analizar los datos como se describe en el paso 6.
6. analizar la qPCR datos sin procesar y convertir de qPCR Ct valor gc/μL para bacteriófagos T7
- Analizar los datos raw qPCR y evaluar la calidad de los datos mediante el análisis de los diagramas característicos de los datos en bruto (figura 4).
Nota: Amplificación parcela, parcela multicomponente y curva de fusión trama son que la característica clave de parcelas para validar la calidad de los datos de la qPCR. -
Calcular los valores de umbral promedio de ciclo (Ct) de las muestras por triplicado en cada concentración de la curva estándar (paso 2.1).
- Parcela media Ct (Y) contra el registro (gc/μL) (X) para obtener la curva lineal Y = kX + b (k es la pendiente de la curva lineal, b es el intercepto).
- Calcular el coeficiente lineal14 R2 (R2 debe ser > 0,99).
- Utilice la curva estándar Y = kX + b para calcular el número de fagos T7 en las muestras de biopanning (es decir, phages seleccionados contra CF-como moco) en gc/μl (figura 5).
Nota: k es la pendiente de la curva estándar. - Calcular la eficiencia de amplificación por la siguiente ecuación
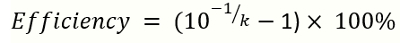 (2)
(2)
Nota: k es la pendiente de la curva estándar generada de qPCR de ADN de referencia. El rango ideal de eficiencia de amplificación es 90 – 110%.
Resultados
Herramientas de diseño de la cartilla diferentes pueden utilizarse para diseñar primers qPCR. Por lo general, primer diseño programas tienen sus propios incorporados algoritmos para calcular y validar los parámetros clave de cartillas, por ejemplo, GC %, Tm, dimer de cartilla o formación de horquilla, etc. generalmente, los criterios claves son similares en diferentes herramientas de diseño de la cartilla y cartillas pueden diseñarse siguiendo sus instrucciones. P...
Discusión
Desarrollamos métodos de qPCR para cuantificar phage genoma ADN5y aquí describimos y adaptado un método de qPCR para enumerar T7 phages seleccionados contra una barrera de moco similar a CF. Información mínima para las directrices de publicación de cuantitativa en tiempo real PCR experimentos (MIQE) fueron adaptados para desarrollar y validar el método de qPCR para enumeración de fagos T715. El protocolo que hemos desarrollado para cuantificar los phages de biopanni...
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Este trabajo fue financiado por beca de PhRMA Foundation investigación arranque y el National Heart, Lung and Blood Institute de los institutos nacionales de salud otorgan el número de concesión R01HL138251.
Materiales
| Name | Company | Catalog Number | Comments |
| Materials and Reagents | |||
| Primers for T7 genomic DNA | IDT | F: CCTCTTGGGAGGAAGAGATTTG R: TACGGGTCTCGTAGGACTTAAT | |
| T7 Select packaging control DNA | EMD Millipore | 69679-1UG | |
| MicroAmp optical 96-well reaction plate | ThermoFisher Scientific | N8010560 | |
| qPCR master mix--Power up SYBR Green master mix | Applied biosystems | A25742 | |
| MicroAmp optical adhesive film kit | ThermoFisher Scientific | 4313663 | |
| T7Select 415-1 Cloning Kit | EMD Millipore | 70015 | User protocols : http://www.emdmillipore.com/US/en/product/T7Select-415-1-Cloning-Kit,EMD_BIO-70015#anchor_USP |
| DNase I solution | ThermoFisher Scientific | 90083 | |
| 24-well transwell plate | Corning | 3472 | |
| UltraPure DNase/RNase-Free Distilled H2O | Invitrogen | 10977015 | |
| Phosphate Buffered Saline (1x) | Corning | 21040CV | |
| Name | Company | Catalog Number | Comments |
| Equipments | |||
| ViiA7 Real-Time PCR System with Fast 96-Well Block | ThermoFisher Scientific | 4453535 | |
| Heraeus Pico 21 Microcentrifuge | ThermoFisher Scientific | 75002415 | |
| Fisherbrand Digital Vortex Mixer | Fisher Scientific | 02-215-370 | |
| HERMO SCIENTIFIC Multi-Blok Heater | ThermoFisher Scientific | Model:2001 | |
| Sorvall Legend X1 Centrifuge | ThermoFisher Scientific | 75004220 | |
| M-20 Microplate Swinging Bucket Rotor | ThermoFisher Scientific | 75003624 | |
| Name | Company | Catalog Number | Comments |
| Software | |||
| QuantStudio Real-time PCR software | ThermoFisher Scientific | v1.2 | |
| Real-time qPCR primer design | IDT | ||
| OligoAnalyzer 3.1 | IDT |
Referencias
- Smith, G. Filamentous fusion phage: novel expression vectors that display cloned antigens on the virion surface. Science. 228 (4705), 1315-1317 (1985).
- Smith, G. P., Petrenko, V. A. Phage Display. Chemical Reviews. 97 (96), 391-410 (1997).
- Sergeeva, A., Kolonin, M. G., Molldrem, J. J., Pasqualini, R., Arap, W. Display technologies: Application for the discovery of drug and gene delivery agents. Advanced Drug Delivery Reviews. 58 (15), 1622-1654 (2006).
- Dias-Neto, E., et al. Next-generation phage display: Integrating and comparing available molecular tools to enable costeffective high-throughput analysis. PLoS ONE. 4 (12), 1-11 (2009).
- Peng, X., Nguyen, A., Ghosh, D. Quantification of M13 and T7 bacteriophages by TaqMan and SYBR green qPCR. Journal of Virological Methods. 252 (June 2017), 100-107 (2017).
- Khan, M. S., Pande, T., Mvan de Ven, T. G. Qualitative and quantitative detection of T7 bacteriophages using paper based sandwich ELISA. Colloids and Surfaces B: Biointerfaces. 132, 264-270 (2015).
- Brasino, M., Lee, J. H., Cha, J. N. Creating highly amplified enzyme-linked immunosorbent assay signals from genetically engineered bacteriophage. Analytical Biochemistry. 470, 7-13 (2014).
- Yu, X., Burgoon, M., Shearer, A., Gilden, D. Characterization of phage peptide interaction with antibody using phage mediated immuno-PCR. J immunol Methods. 326 (1-2), 33-40 (2007).
- Lehmusvuori, A., Manninen, J., Huovinen, T., Soukka, T., Lamminmäki, U. Homogenous M13 bacteriophage quantification assay using switchable lanthanide fluorescence probes. BioTechniques. 53 (5), 301-303 (2012).
- Schlaman, H. R. M., et al. Analysis of interactions of signaling proteins with phage-displayed ligands by fluorescence correlation spectroscopy. Journal of Biomolecular Screening. 13 (8), 766-776 (2008).
- Tjhung, K. F., Burnham, S., Anany, H., Griffiths, M. W., Derda, R. Rapid enumeration of phage in monodisperse emulsions. Analytical Chemistry. 86 (12), 5642-5648 (2014).
- Reitinger, S., Petriv, O. I., Mehr, K., Hansen, C. L., Withers, S. G. Purification and quantitation of bacteriophage M13 using desalting spin columns and digital PCR. Journal of Virological Methods. 185 (1), 171-174 (2012).
- . T7Select® Biopanning Kit Available from: https://www.emdmillipore.com/US/en/product/T7Select-Biopanning-Kit (2018)
- Schneider, A., Hommel, G., Blettner, M. Linear Regression Analysis. Deutsches Ärzteblatt international. 107 (44), 776-782 (2010).
- Bustin, S. A., Benes, V., Garson, J. A., Hellemans, J., Huggert, J. The MIQE Guidelines: Minimun information for publication of quantitative real-time PCR experiments. Clinical Chemitry. 55 (4), 611-622 (2009).
- Lock, M., Alvira, M. R., Chen, S. -. J., Wilson, J. M. Absolute Determination of Single-Stranded and Self-Complementary Adeno-Associated Viral Vector Genome Titers by Droplet Digital PCR. Human Gene Therapy Methods. 25 (2), 115-125 (2014).
- Fittipaldi, M., et al. Discrimination of infectious bacteriophage T4 virus by propidium monoazide real-time PCR. Journal of Virological Methods. 168 (1-2), 228-232 (2010).
- Lodder, W. J., et al. Reduction of bacteriophage MS2 by filtration and irradiation determined by culture and quantitative real-time RT-PCR. Journal of Water and Health. 11 (2), 256-266 (2013).
- Brown-Jaque, M., et al. Antibiotic resistance genes in phage particles isolated from human feces and induced from clinical bacterial isolates. International Journal of Antimicrobial Agents. , (2017).
- Naveca, F. G., et al. Multiplexed reverse transcription real-time polymerase chain reaction for simultaneous detection of Mayaro, Oropouche, and oropouche-like viruses. Memorias do Instituto Oswaldo Cruz. 112 (7), 510-513 (2017).
- Jia, J., et al. Simultaneous detection and differentiation of human parvovirus B19 and human parvovirus 4 by an internally controlled multiplex quantitative real-time PCR. Molecular and Cellular Probes. 36, 50-57 (2017).
- Bliem, R., et al. A novel triplex quantitative pcr strategy for quantification of toxigenic and nontoxigenic Vibrio cholerae in aquatic environments. Applied and Environmental Microbiology. 81 (9), 3077-3085 (2015).
- Wan, Z., Goddard, N. L. Competition Between Conjugation and M13 Phage Infection in Escherichia coli in the Absence of Selection Pressure: A Kinetic Study. G3 Genes|Genomes|Genetics. 2 (10), 1137-1144 (2012).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados