Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
Количественная ПЦР бактериофага Т7 от Biopanning
В этой статье
Резюме
Воспроизводимость, точность и время эффективный количественный PCR (ПЦР) метод для перечисления бактериофага Т7 описано здесь. Протокол четко описывает подготовка геномной ДНК фага, подготовка реакции PCR, Велоспорт условия ПЦР и ПЦР анализа данных.
Аннотация
Этот протокол описывает использование количественного PCR (ПЦР) для перечисления T7 бактериофаги от фага выбор экспериментов (т.е., «biopanning»). ПЦР является подход, основанный на флуоресценции, для количественного определения ДНК, и здесь, он приспособлен для количественного определения Фаговые геномов как прокси для Фаговые частицы. В этом протоколе метод подготовки ДНК снисходительный Фаговые описывается с помощью высокой температуры Отопление без дополнительной очистки ДНК. Этот метод должен только небольшие объемы термообработанная бактериофаги и небольшие объемы реакции ПЦР. ПЦР высокой пропускной способностью и быстрый, возможность обработки и получения данных от 96-луночных пластины реакций в 2 – 4 ч. по сравнению с другими подходами перечисления ФАГ, ПЦР более эффективным с точки зрения времени. Здесь ПЦР используется для перечисления T7 бактериофаги от biopanning в vitro модели муковисцидоза как слизи. Метод ПЦР может быть расширен для количественного определения T7 бактериофаги от других экспериментов, включая другие виды biopanning (например., иммобилизованных связывания белков, в естественных условиях скрининг ФАГ) и другие источники (например, систем водоснабжения или жидкостями организма). Таким образом этот протокол могут быть изменены для количественного определения любых инкапсулированные ДНК вирусов.
Введение
Бактериофага (ФАГ) дисплей технология, разработанная Джордж Смит в 1985 году, представляет собой мощный, высок объём подход к идентификации пептид или белок лигандов против целей или рецепторов клеточной мембраны, болезни антигены, клеточных органелл или конкретных органов и тканей в последние два десятилетия1,2. Здесь случайные библиотек полипептиды или одной цепи антитела отображаются на пальто белки бактериофаги (обычно M13 или T7), и конкретные лигандов могут быть определены из панорамирование против подвижности белков в vitro или в естественных условиях биологические системы через итеративный отбора. Затем лиганды может быть в сочетании с изображений или терапевтических агентов для диагностики и лечения заболеваний3,4. Очень важно точно перечислять бактериофаги во время нескольких шагов biopanning: (1) количественно бактериофаги, привязанные к подложке и (2) для количественной оценки бактериофаги, чтобы определить, если есть обогащения с каждом раунде отбора (ФАГ обогащения указывает biopanned Фаговые сродство для целевого объекта). Текущий золотой стандарт для количественной оценки, assay металлической пластинкы-двухслойная, представляет несколько проблем; Это утомительно, громоздкой и потенциально неточным для большого числа проб. Таким образом наша группа разработала чувствительных, воспроизводимость, точной и время эффективных количественных полимеразной цепной реакции (ПЦР) метод для перечисления M13 и Т7 Фаговые частицы от biopanning5.
ПЦР является привлекательным и осуществимым методом точного количественного определения T7 и M13 бактериофаги. Поскольку каждый индивидуальный фаговых частиц может содержать только одну копию геномной ДНК (dsDNA или ssDNA), один Фаговые генома эквивалентна одной Фаговые частицы; путем определения количественных показателей числа Фаговые геномов, целесообразно определить количество бактериофаги. Во время ПЦР, флуоресцентный репортер красители привязать геномной ДНК фага неспецифически или специально через последовательность специфические праймеры во время ПЦР-амплификации и флуоресценции сигнала увеличивается с каждым раундом амплификации. Когда сигнал флуоресценции достигает порога, что раунд/цикл амплификации отмечается как порог цикла (Ct). Известной концентрации ссылка Фаговые ДНК заговор против их Ct значения для установления стандартной кривой. Использование стандартной кривой с КТ значения образцов ДНК, может путем интерполяции концентрации бактериофаги.
В то время как ранее разработаны и широко используется для количественного определения бактериофаги от biopanning многочисленные стратегии, каждая из них имеет конкретные проблемы. Метод наиболее популярных и обычных является assay металлической пластинкы двойного слоя. Здесь, принимающей бактерии заражены бактериофаги, подготовленных на серийных разведений, покрытие на подложке твердых агар и обложил с агар; Бактериофаги перечисляются с количество металлических пластинк сформирована (то есть, доска формирования подразделений или ОРП) на табличке агар. Assay металлической пластинкы чувствительных, но утомительный, длительным и неточной, особенно для многочисленных образцов и с высокой концентрации5. Кроме того энзим соединенный иммуноферментного анализа (ИФА) были адаптированы для перечисления M13 и Т7 Фаговые частицы6,,78. Здесь, связаны бактериофагов в различных разведениях и захвачен на твердом субстрате (т.е., микроплита), исследовали с ФАГ специфические антитела и обнаружить с помощью Репортеры (например, фермент чувствительных хромогенных субстратов, флуорофоров) для Определите количество фаговых частиц настоящего. Надписи (например, флюоресценция, поглощения) образцов может использоваться для количественного определения неизвестных концентрациях против Фаговые стандартов в известных концентрациях. Были разработаны различные ELISAs ФАГ основе, но они имеют потенциальные ограничения. Одна группа разработала бумажных сэндвич ELISA с диапазоном количественной оценки, которые охватывали семь порядков (102-109 ОРП/мл); Однако этот метод требует несколько шагов антитела покрытия и take up к целый день для пробирного8. Наша группа также разработала метод ИФА для количественного определения M13 Фаговые частицы, но был менее чувствительных дальность обнаружения 106 - 1011 ОРП/мл5. Переключаемый лантаноиды флуоресценции зондов были разработаны для перечисления M13 и количественной оценке данные могут быть получены в течение 20 мин; Однако, этот assay имеет узкий динамический диапазон из 109 1012 ОРП/мл9. Одна группа использовала атомно-силовой микроскопии для перечисления M13 Фаговые частицы в растворе, но это требует передовых электронной микроскопии и работал только в узком диапазоне концентраций10. Другое исследование используется монодисперсных эмульсии для улавливания флуоресцентные M13 и T4 репортер бактериофаги и рассчитывать бактериофаги, количество флуоресцентные капель; Однако, этот подход также выставлены узкие количественной оценки варьируются от 102 -106 ОРП/мл11. При этом капельки цифровой PCR были использованы для количественной оценки M13 бактериофаги этот подход не может различать количество инфекционных и неинфекционных Фаговые частицы12.
Наша группа недавно разработала методы ПЦР для перечисления T7 и M13 бактериофаги, определены от biopanning против blood - brain барьер ячеечная модель с использованием двух различных флуоресцентные репортер красителей5. По сравнению с вышеупомянутой количественной оценки методов, мы разработали методы ПЦР были высокой пропускной способности и времени эффективным количественно оценить многочисленные образцы и возможность различать инфекционных и неинфекционных бактериофаги с DNase I предварительной обработки Фаговые образцов. Важно отметить, что этот подход может можно воспроизвести и точно количественно M13 и Т7 бактериофаги от biopanning образцов. Для начинающих исследователей в области Фаговые ПЦР, здесь мы опишем подробный ПЦР метод для перечисления T7 Фаговые частицы из хвоща ограничены библиотеки (CX7C) приготовлена в vitro барьер слизь кистозный фиброз (CF). От этой работы метод ПЦР может быть продлен для количественного определения Фаговые частицы от других видов biopanning и из других источников, включая воду, почву и жидкости организма.
протокол
1. грунт проектирование и анализ для геномной ДНК фага Т7
- Праймеры дизайн для амплификации геномной ДНК фага Т7.
Примечание: F (вперед) и R (реверс) грунты (см. Таблицу материалы) усилить последовательности ДНК T7 вверх по течению расположена библиотека переменной региона (рис. 1). - Выберите соответствующий грунт анализатора для оценки параметров праймеров, включая температуру плавления (Tm), процентное содержание GC (GC %), Димеры праймера, формирование шпильки и пригодности ПЦР (рис. 1).
- Заказать грунт олигонуклеотиды (oligos) и по прибытии, Ресуспензируйте лиофилизированные oligos в DNase и РНКазы бесплатно ультра-чистой воды, чтобы сделать 100 мкм запасов концентрации в пластиковых пробирок 1.5 мл и хранить при температуре от-20 ° C в течение нескольких месяцев.
- Сделайте несколько аликвоты (около 50 мкл в пробирки 1,5 мл) акций грунт 100 мкм.
Примечание: Этот шаг используется для предотвращения циклов часто замораживания оттаивания.
2. разработать стандарты для количественного определения геномной ДНК фага Т7
- Серийно разбавить 0,1 мкг/мкл (1,0 x 10-8 fg/мкл) ссылка ДНК фага Т7 (приобрели; в известной концентрации 0,1 мкг/мкл, смотрите материалы) десятикратного от 1,0 x 106 1,0 x 100 fg/мкл с ультра-чистой H2O в 0.2 мл ПЦР пробирок. Подготовка каждого концентрация в общем объеме 20 мкл (рис. 2).
- Вихревой ПЦР пробирок для обеспечения тщательного перемешивания на каждом шаге разведение. Кроме того измените советы при добавлении следующих разбавления образца ДНК.
Примечание: Рекомендуется подготовить новое решение ДНК стандартов для стандартной кривой для каждого пакета ПЦР эксперимента. - Используйте уравнение 1 (ниже) для преобразования ссылки фага Т7 ДНК концентрации от fg/мкл копии генома (gc) µл (рис. 2).
- Концентрация ДНК фага Т7 в gc/мкл =
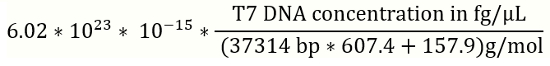 (1)
(1)
Примечание: bp: пары; Ссылка T7 геномной ДНК является 37314 bp.
3. Предварительная обработка Фаговые образцы перед ПЦР реакции подготовка
-
(Вариант 1) Накапайте 20 мкл один клон T7 (ФАГ) из biopanning против в vitro кистозный фиброз (CF)-как модель слизи в 1,5 мл пробирок. Добавить 1,25 единиц DNase I (0.5 мкл) к каждой 20 мкл пример.
Примечание: T7 Фаговые хвоща ограничением меланокортину библиотека (CX7C) был biopanned против CF-как слизью наносится на 24-ну мембраны вставки (см. Таблицу материалы) для 15 мин. Дополнительная информация представлена в разделе результаты. Один клон T7 был выбран для ПЦР пробоподготовки от элюата.- Вариант 2: Пипетки 200 мкл T7 клонировать в 1,5 мл пробирок и добавить 5 единиц DNase I
(2 мкл) для каждого образца.
Примечание: Объем выборки фага, выбранного на этом этапе зависит от объема собранных от biopanning. В то время как протокол посвящен ПЦР biopanned ФАГ, шаги для biopanning, подробно изложены в протоколе пользователя фага Т7 комплект (см. Таблицу материалы)13.
- Вариант 2: Пипетки 200 мкл T7 клонировать в 1,5 мл пробирок и добавить 5 единиц DNase I
- Cap пробоотборные трубки фага Т7 (шаг 3.1) и инкубировать их при 37 ° C 10 мин в ванне с подогревом сухой (т.е., блок тепла).
- Инкубируйте трубы (от шага 3.2) на 100 ° C в течение 15 мин в ванне с подогревом сухой (рис. 3A).
Предупреждение: Поскольку испарение и конденсация происходит во время шага Отопление, убедитесь, полностью максимум 1,5 мл пробирок. - Спина термообработанная бактериофаги от шага 3.3 на 18,534 x g 10 s. смеси бактериофагов, поставив трубку на вихревой смеситель установлен на режим сенсорного 10 s, и спина на 18,534 x g 10 s снова.
Примечание: Спин микс-отжима, чтобы убедиться, что с подогревом Фаговые образцы хорошо смешиваются. - Охладить пробоотборные трубки при комнатной температуре (RT), оставляя их на скамейке эксперимент или хранить при 4 ° C в холодильник на ночь.
Примечание: Эксперимент может быть приостановлена на этот шаг.
4. Подготовьте реакций ПЦР
Примечание: Одна реакция PCR предназначен для одного образца. Каждый реакции PCR содержит 5 мкл ПЦР мастер смесь (см. материалы), 1 мкл смеси пара грунтовка 5 мкм, H2O 2 мкл и 2 мкл термообработанная образца фага Т7. Для нескольких реакций PCR все реагенты, за исключением Фаговые образца смешиванием в одном 1,5 мл. Объем каждого реагента в премикс зависит количество Фаговые выборок для ПЦР.
- Подготовьте ПЦР премикс для 10 проб biopanned ФАГ неизвестной концентрации и 7 стандартной концентрации в трех экземплярах. В результате есть 51 всего образцы и, следовательно, 51 реакций ПЦР (то есть, Одна реакция на сэмпл). Для 51 реакций Подготовьте премикс 255 мкл ПЦР главный микс (51 x 5 мкл), 51 мкл праймеров (51 x 1 мкл) и 102 мкл H2O (51 x 2 мкл).
Примечание: Рекомендуется подготовить несколько дополнительных ПЦР-реакции для компенсации потери объема при aliquoting смесь ПЦР в каждой скважине пластину 96-луночных ПЦР. - Аликвота 8 мкл ПЦР премикса на каждой скважине 96-луночных ПЦР пластины для в общей сложности 51 скважин (т.е., 51 реакций ПЦР). Существует не нужно менять советы во время aliquoting.
- Добавить 2 мкл Фаговые образцов термообработанная T7 (шаг 3.4) или ссылку фага Т7 ДНК (шаг 2.1) для каждой скважины (рис. 3B); изменение подсказки при добавлении различные образцы ДНК к колодцу, чтобы избежать перекрестного загрязнения. Кроме того отказаться от 2 мкл образцы ДНК под поверхность премикс ПЦР (то есть, в премикс ПЦР 8 мкл).
- Уплотнение пластину ПЦР с липкой пленкой.
Примечание: Этот шаг очень важен, независимо от типа плиты ПЦР. Используйте рекомендованные комплект для герметизации плита полностью; в противном случае, любые незапечатанным региона в пластине приведет к потере тома во время ПЦР Велоспорт. - Оберните пластину ПЦР с алюминиевой фольгой, чтобы свести к минимуму флуоресценции Фотообесцвечивание и перейдите к запустите его на оборудование для ПЦР.
5. ПЦР Велоспорт условия
Примечание: ПЦР Велоспорт условия были созданы на оборудовании конкретного ПЦР (см. Таблицу материалы).
- Запустите пластину 96-луночных ПЦР на оборудование для ПЦР. На оборудовании выберите настройки из меню, чтобы настроить следующие экспериментальных условий (рис. 3 c).
- Настройка Велоспорт условий следующим объем 10 мкл пример: один цикл при 50 ° C за 2 мин, один цикл при 95 ° C на 2 мин, затем 40 циклов (95 ° C в течение 15 сек, 60 ° C в течение 1 мин). После запуска параметры кривой расплава: один цикл 95 ° c 15 s, 60 ° C в течение 1 мин и 95 ° C 15 s.
- Перейти для анализа данных, как описано в шаге 6.
6. анализ ПЦР необработанных данных и конвертировать из ПЦР Ct значение в gc/мкл для бактериофагов T7
- Анализ данных сырье ПЦР и оценивать качество данных путем анализа характерных участков необработанных данных (Рисунок 4).
Примечание: Усиление сюжет, многокомпонентных участок и участок кривой расплава являются ключевой характеристикой участки для проверки качества данных ПЦР. -
Вычислить среднее пороговые значения цикла (Ct) из трех экземплярах образцов на каждом концентрации стандартной кривой (шаг 2.1).
- Участок средней Ct (Y) против журнала (gc/мкл) (X), чтобы получить линейную кривую Y = kX + b (k является наклон линейной кривой, b отсекаемый отрезок).
- Вычислить коэффициент линейного14 R2 (R2 должна быть > 0,99).
- Используйте стандартные кривая Y = kX + b, чтобы вычислить количество T7 бактериофагов в biopanning образцах (т.е. бактериофаги выбран против CF-как слизь) в gc/мкл (рис. 5).
Примечание: k-наклон калибровочной кривой. - Рассчитать эффективность усиления по следующей формуле
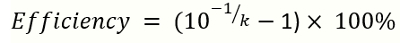 (2)
(2)
Примечание: k-наклон стандартной кривой, генерируемые ссылки ПЦР ДНК. Идеальный диапазон усиления эффективности составляет 90-110%.
Результаты
Инструменты для конструирования праймера различные могут быть использованы для разработки ПЦР праймеры. Как правило грунтовки дизайн программы имеют свои собственные встроенные алгоритмы для расчета и проверки ключевых параметров грунты, например, GC %, ТМ, диме?...
Обсуждение
Мы разработали методы ПЦР для количественного определения геномной ДНК фага5, и здесь мы описали и адаптировать метод ПЦР для перечисления T7 бактериофаги выбран слизь CF-как барьер. Минимальная информация для публикации о количественных Real-Time PCR экспериментов (MIQE) руководящ...
Раскрытие информации
Авторы не имеют ничего сообщать.
Благодарности
Эта работа была поддержана стартера Грант PhRMA фонда исследований и Национальный сердца, легких и крови института национальных институтов здравоохранения предоставить под номером R01HL138251 премии.
Материалы
| Name | Company | Catalog Number | Comments |
| Materials and Reagents | |||
| Primers for T7 genomic DNA | IDT | F: CCTCTTGGGAGGAAGAGATTTG R: TACGGGTCTCGTAGGACTTAAT | |
| T7 Select packaging control DNA | EMD Millipore | 69679-1UG | |
| MicroAmp optical 96-well reaction plate | ThermoFisher Scientific | N8010560 | |
| qPCR master mix--Power up SYBR Green master mix | Applied biosystems | A25742 | |
| MicroAmp optical adhesive film kit | ThermoFisher Scientific | 4313663 | |
| T7Select 415-1 Cloning Kit | EMD Millipore | 70015 | User protocols : http://www.emdmillipore.com/US/en/product/T7Select-415-1-Cloning-Kit,EMD_BIO-70015#anchor_USP |
| DNase I solution | ThermoFisher Scientific | 90083 | |
| 24-well transwell plate | Corning | 3472 | |
| UltraPure DNase/RNase-Free Distilled H2O | Invitrogen | 10977015 | |
| Phosphate Buffered Saline (1x) | Corning | 21040CV | |
| Name | Company | Catalog Number | Comments |
| Equipments | |||
| ViiA7 Real-Time PCR System with Fast 96-Well Block | ThermoFisher Scientific | 4453535 | |
| Heraeus Pico 21 Microcentrifuge | ThermoFisher Scientific | 75002415 | |
| Fisherbrand Digital Vortex Mixer | Fisher Scientific | 02-215-370 | |
| HERMO SCIENTIFIC Multi-Blok Heater | ThermoFisher Scientific | Model:2001 | |
| Sorvall Legend X1 Centrifuge | ThermoFisher Scientific | 75004220 | |
| M-20 Microplate Swinging Bucket Rotor | ThermoFisher Scientific | 75003624 | |
| Name | Company | Catalog Number | Comments |
| Software | |||
| QuantStudio Real-time PCR software | ThermoFisher Scientific | v1.2 | |
| Real-time qPCR primer design | IDT | ||
| OligoAnalyzer 3.1 | IDT |
Ссылки
- Smith, G. Filamentous fusion phage: novel expression vectors that display cloned antigens on the virion surface. Science. 228 (4705), 1315-1317 (1985).
- Smith, G. P., Petrenko, V. A. Phage Display. Chemical Reviews. 97 (96), 391-410 (1997).
- Sergeeva, A., Kolonin, M. G., Molldrem, J. J., Pasqualini, R., Arap, W. Display technologies: Application for the discovery of drug and gene delivery agents. Advanced Drug Delivery Reviews. 58 (15), 1622-1654 (2006).
- Dias-Neto, E., et al. Next-generation phage display: Integrating and comparing available molecular tools to enable costeffective high-throughput analysis. PLoS ONE. 4 (12), 1-11 (2009).
- Peng, X., Nguyen, A., Ghosh, D. Quantification of M13 and T7 bacteriophages by TaqMan and SYBR green qPCR. Journal of Virological Methods. 252 (June 2017), 100-107 (2017).
- Khan, M. S., Pande, T., Mvan de Ven, T. G. Qualitative and quantitative detection of T7 bacteriophages using paper based sandwich ELISA. Colloids and Surfaces B: Biointerfaces. 132, 264-270 (2015).
- Brasino, M., Lee, J. H., Cha, J. N. Creating highly amplified enzyme-linked immunosorbent assay signals from genetically engineered bacteriophage. Analytical Biochemistry. 470, 7-13 (2014).
- Yu, X., Burgoon, M., Shearer, A., Gilden, D. Characterization of phage peptide interaction with antibody using phage mediated immuno-PCR. J immunol Methods. 326 (1-2), 33-40 (2007).
- Lehmusvuori, A., Manninen, J., Huovinen, T., Soukka, T., Lamminmäki, U. Homogenous M13 bacteriophage quantification assay using switchable lanthanide fluorescence probes. BioTechniques. 53 (5), 301-303 (2012).
- Schlaman, H. R. M., et al. Analysis of interactions of signaling proteins with phage-displayed ligands by fluorescence correlation spectroscopy. Journal of Biomolecular Screening. 13 (8), 766-776 (2008).
- Tjhung, K. F., Burnham, S., Anany, H., Griffiths, M. W., Derda, R. Rapid enumeration of phage in monodisperse emulsions. Analytical Chemistry. 86 (12), 5642-5648 (2014).
- Reitinger, S., Petriv, O. I., Mehr, K., Hansen, C. L., Withers, S. G. Purification and quantitation of bacteriophage M13 using desalting spin columns and digital PCR. Journal of Virological Methods. 185 (1), 171-174 (2012).
- . T7Select® Biopanning Kit Available from: https://www.emdmillipore.com/US/en/product/T7Select-Biopanning-Kit (2018)
- Schneider, A., Hommel, G., Blettner, M. Linear Regression Analysis. Deutsches Ärzteblatt international. 107 (44), 776-782 (2010).
- Bustin, S. A., Benes, V., Garson, J. A., Hellemans, J., Huggert, J. The MIQE Guidelines: Minimun information for publication of quantitative real-time PCR experiments. Clinical Chemitry. 55 (4), 611-622 (2009).
- Lock, M., Alvira, M. R., Chen, S. -. J., Wilson, J. M. Absolute Determination of Single-Stranded and Self-Complementary Adeno-Associated Viral Vector Genome Titers by Droplet Digital PCR. Human Gene Therapy Methods. 25 (2), 115-125 (2014).
- Fittipaldi, M., et al. Discrimination of infectious bacteriophage T4 virus by propidium monoazide real-time PCR. Journal of Virological Methods. 168 (1-2), 228-232 (2010).
- Lodder, W. J., et al. Reduction of bacteriophage MS2 by filtration and irradiation determined by culture and quantitative real-time RT-PCR. Journal of Water and Health. 11 (2), 256-266 (2013).
- Brown-Jaque, M., et al. Antibiotic resistance genes in phage particles isolated from human feces and induced from clinical bacterial isolates. International Journal of Antimicrobial Agents. , (2017).
- Naveca, F. G., et al. Multiplexed reverse transcription real-time polymerase chain reaction for simultaneous detection of Mayaro, Oropouche, and oropouche-like viruses. Memorias do Instituto Oswaldo Cruz. 112 (7), 510-513 (2017).
- Jia, J., et al. Simultaneous detection and differentiation of human parvovirus B19 and human parvovirus 4 by an internally controlled multiplex quantitative real-time PCR. Molecular and Cellular Probes. 36, 50-57 (2017).
- Bliem, R., et al. A novel triplex quantitative pcr strategy for quantification of toxigenic and nontoxigenic Vibrio cholerae in aquatic environments. Applied and Environmental Microbiology. 81 (9), 3077-3085 (2015).
- Wan, Z., Goddard, N. L. Competition Between Conjugation and M13 Phage Infection in Escherichia coli in the Absence of Selection Pressure: A Kinetic Study. G3 Genes|Genomes|Genetics. 2 (10), 1137-1144 (2012).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеThis article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены