A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
مجهريتتبع جزيء واحد - أداة لتحديد الدول المنافية للجزيئات الخلوية
In This Article
Summary
يتم استخدام الفحص المجهري لتوطين جزيء واحد ثلاثي الدمنل لسبر المواقف المكانية ومسارات الحركة للبروتينات ذات العلامات الفلورية في الخلايا البكتيرية الحية. يحدد بروتوكول تحليل البيانات التجريبية الموضحة هنا السلوكيات المشتتة السائدة للبروتينات الخلوية استناداً إلى مسارات أحادية الجزيء مجمعة.
Abstract
يسبر الفحص المجهري لتوطين جزيء واحد موضع وحركات الجزيئات الفردية في الخلايا الحية مع عشرات النانومتر المكاني والاستبانة الزمنية المليثانية. هذه القدرات تجعل من جزيء واحد التعريب المجهري ة مناسبة بشكل مثالي لدراسة الوظائف البيولوجية على المستوى الجزيئي في البيئات ذات الصلة من الناحية الفسيولوجية. هنا، نقوم بتوضيح بروتوكول متكامل لكل من الحصول على ومعالجة/تحليل بيانات تتبع جزيء واحد لاستخراج مختلف الدول المشتتة التي قد تظهر البروتين ذات الأهمية. ويمكن استخدام هذه المعلومات لتحديد كمية تشكيل المعقدة الجزيئية في الخلايا الحية. نحن نقدم وصفا مفصلا لتجربة توطين جزيء واحد 3D المستندة إلى الكاميرا، فضلا عن خطوات معالجة البيانات اللاحقة التي تسفر عن مسارات الجزيئات الفردية. ثم يتم تحليل هذه المسارات باستخدام إطار تحليل عددي لاستخراج الحالات الانقباحية السائدة للجزيئات التي تحمل علامة الفلورسنت والوفرة النسبية لهذه الدول. ويستند إطار التحليل إلى عمليات محاكاة استوكاستك لمسارات الانتشار البنيداخل الخلايا التي تقتصر مكانياً على هندسة الخلايا التعسفية. استناداً إلى مسارات محاكاة، يتم إنشاء الصور الخام جزيء واحد وتحليلها بنفس الطريقة مثل الصور التجريبية. وبهذه الطريقة، فإن القيود التجريبية على الدقة والدقة، التي يصعب معايرة تجريبياً، تُدمج صراحة في سير عمل التحليل. يتم تحديد معامل الانتشار والكسور السكانية النسبية للحالات المشتتة السائدة عن طريق تركيب توزيعات القيم التجريبية باستخدام تركيبات خطية من التوزيعات المحاكاة. نحن نبرهن على فائدة بروتوكولنا من خلال حل الحالات المنافية للبروتين الذي يعرض حالات مختلفة من عدم الاكتراث عند تشكيل المجمعات المتجانسة والمتغايّرة القلة في السيتوسول من مسببات الأمراض البكتيرية.
Introduction
دراسة السلوك المختلف للجزيئات الحيوية يوفر نظرة ثاقبة في وظائفها البيولوجية. أصبحت التقنيات المستندة إلى التنظير المجهري الفلوري أدوات قيمة لمراقبة الجزيئات الحيوية في بيئة الخلايا الأصلية الخاصة بها. الانتعاش الفلوري بعد تبييض الصور (FRAP) ومطياف الارتباط الفلوري (FCS)1 توفير السلوكيات الفرقية متوسط الفرقة. وعلى العكس من ذلك، فإن الفحص المجهري لتوطين جزيء واحد يتيح مراقبة الجزيئات الفردية ذات العلامات الفلورية ذات الدقة المكانية والزمنية العالية2و3و4. مراقبة الجزيئات الفردية مفيد لأن البروتين من الفائدة قد توجد في حالات مختلفة من عدم الأهمية. على سبيل المثال، تنشأ دولتان مختلفتان يمكن تمييزهما بسهولة عندما ينتشر منظم النسخ، مثل CueR في الإشريكية القولونية، بحرية في السيتوسول أو يرتبط بتسلسل الحمض النووي ويصبح معطلة على المقياس الزمني للقياس5 . يوفر تتبع جزيء واحد أداة لمراقبة هذه الدول المختلفة مباشرة، وليس هناك حاجة إلى تحليلات متطورة لحلها. ومع ذلك، يصبح من الصعب أكثر حل حالات متعددة غير مُقابَّلة وكسور سكانها في الحالات التي تكون فيها معدلاتها المشتتة أكثر تشابهاً. على سبيل المثال، بسبب الاعتماد على حجم معامل الانتشار، تظهر حالات القلة المختلفة للبروتين نفسها كحالات مختلفة غير مُقَدِّمة6و7و8و9 , 10- وتتطلب هذه الحالات نهجا متكاملا من حيث الحصول على البيانات وتجهيزها وتحليلها.
ومن العوامل الحاسمة التي تؤثر على المعدلات المختلفة للجزيئات الكليوم أثر الحبس بحدود الخلية. وتتسبب القيود المفروضة على الحركة الجزيئية من قبل حد الخلية البكتيرية في أن يظهر معدل الانتشار المقاس للجزيئات الكلوسالية أبطأ مما لو كان نفس الحركة قد حدث في مساحة غير محصورة. بالنسبة للجزيئات التي تنتشر ببطء شديد، فإن تأثير الحبس الخلوي لا يكاد يذكر بسبب عدم وجود تصادمات مع الحدود. في مثل هذه الحالات، قد يكون من الممكن حل الحالات المشتتة بدقة عن طريق تركيب توزيعات الإزاحات الجزيئية، ص،أو معاملات الانتشار الظاهرة، D*، باستخدام نماذج تحليلية تستند إلى معادلات الحركة البراونية ( نشر عشوائي)11،12،13. ومع ذلك، لنشر الجزيئات الخلوية بسرعة، والتوزيعات التجريبية لم تعد تشبه تلك التي تم الحصول عليها للحركة براونيان غير المحصورة بسبب اصطدام الجزيئات الناشرة مع حدود الخلية. يجب حساب آثار الحبس لتحديد معاملات الانتشار غير المحصورة للجزيئات التي تحمل علامة الفلورسنت بدقة. وقد وضعت مؤخرا عدة نهج لحساب آثار الحبس إما (شبه)تحليليا 5،14،15،16 أو عدديا من خلال محاكاة مونتي كارلو من نشر براونيان6،10،16،17،18،19.
هنا، نحن نقدم بروتوكول متكامل لجمع وتحليل البيانات المجهرية توطين جزيء واحد مع التركيز بشكل خاص على تتبع جزيء واحد. الهدف النهائي للبروتوكول هو حل الحالات المنافية للبروتينات السيتوفورية التي تحمل علامة الفلوروس داخل، في هذه الحالة، الخلايا البكتيرية على شكل قضيب. ويستند عملنا على بروتوكول سابق لتتبع جزيء واحد، الذي تم تبين أن بوليميراز الحمض النووي، PolI، موجودة في حالة الحمض النووي ملزمة وغير منضمة عن طريق تحليل نشر20. هنا، نقوم بتوسيع تحليل تتبع جزيء واحد إلى قياسات 3D وإجراء محاكاة حسابية أكثر واقعية لحل وقياس حالات مختلفة في وقت واحد موجودة في الخلايا. يتم الحصول على البيانات باستخدام مجهر الفلورة فائقة الدقة 3D بنيت في المنزل والتي هي قادرة على تحديد موقف 3D من باعثات الفلورسنت عن طريق التصوير مع مزدوج الحلزون نقطة انتشار وظيفة (DHPSF)21،22. تتم معالجة الصور الخام ذات الجزيء الواحد باستخدام برامج مكتوبة حسب الطلب لاستخراج التعريب ثلاثي الأبعاد أحادي الجزيء، والتي يتم دمجها بعد ذلك في مسارات أحادية الجزيء. يتم تجميع الآلاف من المسارات لتوليد توزيعات معاملات الانتشار الظاهرة. في خطوة أخيرة, تتناسب التوزيعات تجريبيّة مع عدديّا ينتج توزيعات ينال من خلال [مونت-كرلو] محاكاة من حركة [بروونين] في حجم يحصر. نحن نطبق هذا البروتوكول لحل حالات عدم الانتشار من نوع 3 إفراز نظام البروتين YscQ في العيش Yersinia enterocolitica. نظرًا لطبيعته النمطية، فإن بروتوكولنا ينطبق بشكل عام على أي نوع من تجارب تتبع الجزيئات الواحدة أو الجسيمات الواحدة في الجيومتريات الخلوية التعسفية.
Protocol
1. مزدوج الحلزون نقطة انتشار وظيفة المعايرة 2.
ملاحظة: يتم الحصول على الصور الموضحة في هذا القسم والأقسام التالية باستخدام مجهر الفلورة المقلوب المبني ة حسب الطلب، كما هو موضح في Rocha et al.23. نفس الإجراء ينطبق على تطبيقات المجهر المختلفة المصممة لتوطين جزيء واحد وتتبع المجهرية2،3،4. تتوفر كافة البرامج الخاصة بالحصول على الصور ومعالجة البيانات الموضحة في هذه المقالة (https://github.com/GahlmannLab2014/Single-Molecule-Tracking-Analysis.git).
- إعداد منصات أغاروز لتركيب عينات على زلات غطاء زجاجي ليتم عرضها تحت المجهر.
- إضافة 1.5-2٪ من الوزن منخفضة ذوبان نقطة أجاروز إلى 5 مل من M2G العازلة (4.9 مليون متر Na2HPO4،3.1 m KH2PO4،7.5 m NH4Cl، 0.5 mM MgSO4،10 μM FeSO4،0.5 M CaCl2 و 0.2٪ الجلوكوز). الميكروويف لعدة ثوان حتى يذوب كل أجاروز. لا تدع الحل يغلي.
- دع محلول الأجاروز يبرد لبضع دقائق (2-3 دقائق).
- ماصة 600 ميكرولتر من محلول أجاروز على غطاء زجاجي (22 مم × 22 مم). وضع بلطف غطاء زجاجي الثاني زلة على رأس أغاروز. وهذا يخلق رقيقة (~ 0.5 ملم) وسادة هلام أغاروز بين اثنين من زلات غطاء الزجاج.
- دع منصات أجاروز تتماسك لمدة 20 دقيقة تقريبًا.
- افصل وافصل زلات الغطاء الزجاجي برفق. وسادة الأجاروز سوف تلتصق بواحد منهم.
- باستخدام شفرة الحلاقة، قطع وسادة أغاروز إلى أربعة أقسام مربعة من حجم متساو. يمكن استخدام كل مقطع مربع لعينة واحدة.
- ماصة 1.5 ميكرولتر من محلول حبة الفلورسنت على وسادة واحدة أغاروز. حل حبة هو تخفيف 1/100000 من حل الأسهم في M2G (انظر جدول المواد).
- عكس وسادة أغاروز ووضعها على غطاء زجاجي زلة التي تم تنظيفها في منظف الأوزون لمدة 30 دقيقة. يتم اختيار وقت التنظيف للقضاء على أي جزيئات الفلورسنت الملتصقة بالأغطية.
- جبل الزجاج غطاء عينة على حامل العينة من مجهر الفلورة المقلوب وتأمينه في مكان باستخدام شريط لاصق أو الربيع تحميل مقاطع حامل العينة.
- إضافة قطرة من زيت الغمر على هدف المجهر.
- ضع حامل العينة على المجهر وتأمينه في مكانه.
- تهيئة واجهة المستخدم الرسومية (GUI) للتحكم في كاميرا المجهر، مرحلة العينة، وأشعة الليزر الإثارة. هنا، يتم استخدام البرمجيات المكتوبة حسب الطلب في MATLAB للتحكم في الآلات(الشكل التكميلي 1).
- تهيئة برنامج الكاميرا HCImage لايف. في علامة التبويب التقاط ضمن قسم التحكم في الكاميرا، قم بتعيين وقت التعرض إلى 0.03 s. انقر فوق Live لبدء بث مباشر للكاميرا.
- قم بتشغيل الليزر بالنقر فوق فتح ليزر 514 نانومتر على واجهة المستخدم الرسومية لإثارة الخرز الفلورسنت على الوسادة وعرض انبعاث الفلورة على الكاميرا باستخدام وضع البث المباشر (أي لا توجد بيانات محفوظة على القرص).
- ضبط مواقف X و Y من مرحلة المجهر عن طريق النقر على الأسهم 'XY-نقاط البيع' تحت قسم "مرحلة تحديد المواقع الصغيرة" من واجهة المستخدم الرسومية لوضع حبة الفلورسنت واحد على الأقل في وسط مجال الرؤية (FOV). يمكن تغيير حجم الخطوة بالنقر فوق المربع المنسدل أسفل الأسهم.
- ضبط موقف Z من مرحلة المجهر عن طريق النقر فوق الأسهم Z-نقاط تحت قسم مرحلة تحديد المواقع نانو من واجهة المستخدم الرسومية. قم بتعيين اتجاه وظيفة انتشار نقطة اللولب المزدوج (DHPSF) لحبة الفلورسنت لتكون عمودية. يتم تعريف هذا الاتجاه العمودي كنقطة بداية في معايرة Z. يمكن تغيير حجم الخطوة بالنقر فوق المربع المنسدل أسفل الأسهم.
- في HCImage لايف، تحت التقاط | وسائط الزناد، والسرعة والتسجيل،وتغيير وضع الزناد من الداخلية إلى الخارجية المستوى الزناد. سيسمح هذا واجهة المستخدم الرسومية MATLAB للتحكم في الكاميرا.
- تحت التسلسل | إعدادات المسح الضوئي، تغيير عدد عدد الإطارات إلى 1200. اختر مجلد حفظ الوجهة بالنقر على الزر المسمى .... وأخيراً انقر فوق ابدأ. يتم تعيين عدد عدد الإطارات إلى 1200 بحيث يمكن تجميع 10 إطارات لكل من خطوات الموضع Z 120.
- المسح الضوئي من خلال مجموعة من المواضع Z (30 خطوة فوق وتحت موضع Z عند 50 نانومتر) وتسجيل 10 إطارات في كل خطوة باستخدام وقت التعرض 0.03 s. بدء العملية التلقائية بالنقر فوق GO ضمن قسم Z-معايرة واجهة المستخدم الرسومية.
ملاحظة: يمكن تعديل معلمات المعايرة، بما في ذلك طول الخطوة وعدد الخطوات ووقت تعرض الكاميرا وعدد الإطارات لكل خطوة هنا أيضًا. حوالي 105 فوتون يمكن الحصول عليها من حبة الفلورسنت في إطار واحد باستخدام 0.03 ق أوقات التعرض مما أدى إلى x، y، z دقة التعريب من حوالي 1 نانومتر. - قم بإيقاف تشغيل إضاءة الليزر بالنقر فوق إغلاق ليزر 515 نانومتر في واجهة المستخدم الرسومية. في HCImage لايف، تحت التقاط | وسائط الزناد، والسرعة والتسجيل،وتغيير وضع الزناد مرة أخرى إلى الداخلية. تحت التسلسل | تغيير إعدادات المسح الضوئي عدد عدد الإطارات إلى 200. اختر مجلد حفظ الوجهة بالنقر على الزر المسمى .... انقر فوق ابدأ لجمع 200 إطار من الصور الداكنة مع وقت تعرض 0.03 s.
ملاحظة: حتى في حالة سقوط الضوء على الكاشف، سيقرأ كل بكسل رقمًا موجبًا (يشار إليه باسم قيمة الإزاحة الداكنة)، والذي يمكن أن يختلف قليلاً بين البيكسلات. قد تتغير قيمة الإزاحة الداكنة مع مرور الوقت. لذلك، فمن الضروري جمع إطارات داكنة لكل معايرة. - تناسب DHPSF مع نموذج غاوسي مزدوجباستخدام برنامج Easy-DHPSF24 للحصول على مواقف X و Y من حبة، فضلا عن زاوية مقابل منحنى معايرة Z.
- تهيئة برنامج Easy-DHPSF في MATLAB. ضمن الإعداد، قم بتعيين القناة إلى G وتعيين طريقة التركيب إلى MLE مع نموذج DG. G يشير إلى كاميرا القناة الخضراء، كما البروتين الفلورسنت المستخدمة للحصول على البيانات تنبعث مع الأطوال الموجية الخضراء. MLE مع نموذج DG يشير إلى تقدير الاحتمال الأقصى مع نموذج غاوسي مزدوج.
ملاحظة: يعتمد حجم البكسل وربح التحويل على الإعداد البصري المحدد وقد تحتاج إلى تغيير. - ضمن المقطع معايرة DHPSF ، انقر فوق تشغيل. انقر فوق موافق في الإطار المنبثق التالي للاحتفاظ بالإعدادات الافتراضية.
- حدد مكدس الصور المحفوظة في الخطوات 1.13-1.14. بعد ذلك، حدد مكدس الصور مع الخلفية الداكنة المحفوظة في الخطوة 1.15. وأخيراً، حدد الملف .txt الذي تم حفظه تلقائياً أثناء الخطوة 1.14. يحتوي هذا الملف على موضع Z للمرحلة خلال عملية المسح الضوئي.
- في النافذة المنبثقة التالية، إذا لم يتم استخدام شريحة الكاميرا الكاملة لمجال العرض، قم بإدخال مواضع البداية x0 وy0 على الشريحة. وإلا، يمكن العثور على الإدخال x0 = 1 و y0 = 1.This المعلومات في HCImage Live ضمن قسم Binning و SubArrary.
- في الإطار التالي، قم بتغيير حجم وتغيير موضع المربع الذي يظهر فوق صورة الخرز الفلورسنت بحيث يكون حوالي 100 × 100 بكسل في الحجم وتوسيط فوق إشارة DHPSF الفلورسنت واحدة. ثم انقر نقراً مزدوجاً فوق للمتابعة.
ملاحظة: يجب عزل DHPSF المختار ة عن إشارات DHPSF الأخرى ويكون مثاليًا ألمع حبة ممكنة. - انقر في وسط إشارة DHPSF، بين الفصوص اثنين، ثم اضغط Enter. يظهر الإطار التالي طريقة عرض تكبير من DHPSF المختار. اختر بدقة أكبر موقع مركز DHPSF بالنقر فوق.
ملاحظة: سيقوم البرنامج بعد ذلك باحتواء DHPSF وعرض الصورة الخام وإعادة الإنشاء من صالح. كما سيتم إخراج صور قالب من معايرة Z المقابلة المقطع العرضي DHPSF في مواضع Z مختلفة. وسوف تستخدم هذه في وقت لاحق لتركيب البيانات التجريبية. سيقوم البرنامج بإخراج موضع X وY وZ المقدر في كل إطار. في نظام بصري محاذاة بشكل جيد، X و Y يجب أن تتغير قليلا جدا (~ 30 نانومتر الانحراف) كما يتغير موقف Z. إذا كان اختلاف الإخراج أكبر من 30 نانومتر، يجب إعادة محاذاة قناع المرحلة، الموجود في مستوى فورييه لنظام التصوير(الشكل 1)،وتكرار الخطوات 1.9-1.16. - حفظ واجهة المستخدم الرسومية سهلة DHPSF عن طريق النقر على رمز حفظ في الزاوية اليسرى العليا من واجهة المستخدم الرسومية. يمكن تحميل هذا لاحقاً بالنقر فوق رمز التحميل في واجهة المستخدم الرسومية.
ملاحظة: يجب إجراء إجراء معايرة Z في كل يوم من أيام التجربة لمراعاة تغييرات المحاذاة في المجهر التي قد تكون حدثت بسبب تقلبات درجة الحرارة أو الاهتزازات الميكانيكية.
- تهيئة برنامج Easy-DHPSF في MATLAB. ضمن الإعداد، قم بتعيين القناة إلى G وتعيين طريقة التركيب إلى MLE مع نموذج DG. G يشير إلى كاميرا القناة الخضراء، كما البروتين الفلورسنت المستخدمة للحصول على البيانات تنبعث مع الأطوال الموجية الخضراء. MLE مع نموذج DG يشير إلى تقدير الاحتمال الأقصى مع نموذج غاوسي مزدوج.
2. إعداد الثقافة البكتيرية
- إعداد وسائل الإعلام الثقافية التي تدعم نمو الخلايا البكتيرية. لY. enterocolitica، استخدم 5 مل من BHI (ضخ قلب الدماغ) المرق الذي يحتوي على حمض nalidixic (35 ميكروغرام / مل) و 2،6-حمض diaminopimelic (80 ميكروغرام / مل). هنا، يتم استخدام سلالة Y. enterocolitica التي لديها البروتين YscQ الموسومة بالبروتين الفلورسنت eYFP23.
- تلقيح الوسائط مع الثقافات البكتيرية من مخزونات الفريزر أو الثقافات لوحة.
- تنمو الثقافة في 28 درجة مئوية مع الهز بين عشية وضحاها.
- تخفيف كمية صغيرة (~ 250 درجة مئوية) من الثقافة المشبعة بين عشية وضحاها إلى 5 مل باستخدام وسائل الإعلام ثقافة جديدة.
- تنمو الثقافة في 28 درجة مئوية مع الهز لمدة 60-90 دقيقة.
- حث التعبير عن بروتين الانصهار الفلورسنت. لY. enterocolitica، صدمة الحرارة الخلايا إلى 37 درجة مئوية في شاكر الماء للحث على regulon yop.
- احتضان الخلايا لمدة 3 ساعة إضافية في 37 درجة مئوية مع الهز.
- الطرد المركزي 1 مل من الثقافة في 5000 × ز لمدة 3 دقائق في درجة حرارة الغرفة. تجاهل الـ supernatant.
- غسل بيليه 3 مرات مع 1 مل من وسائل الإعلام M2G.
- إعادة تعليق البكتيريا الكريات في ~ 250 درجة مئوية من وسائل الإعلام M2G.
- إضافة حبات الفلورسنت كعلامات fiducial. وينبغي إضافة محلول حبة الفلورسنت في كميات مخففة بشكل مناسب، بحيث يكون هناك فقط 1-2 الخرز في FOV عندما ينظر إليها في المجهر.
- ماصة بلطف أو دوامة التعليق لفصل الخلايا المجمعة.
- لوحة 1.5 درجة مئوية من التعليق على وسادة أجاروز 1.5-2٪ مصنوعة من M2G.
- عكس وسادة أغاروز ووضعها على غطاء المجهر تنظيفها بالأوزون زلة. وينبغي وضع قسيمة الغطاء في منظف الأوزون لمدة 30 دقيقة للحد من أي خلفية الفلورة المتأصلة.
3 - الحصول على البيانات
- جبل الزجاج غطاء عينة على حامل العينة من مجهر الفلورة المقلوب وتأمينه في مكان باستخدام شريط لاصق أو الربيع تحميل مقاطع حامل العينة.
- إضافة قطرة من زيت الغمر على هدف المجهر، ثم وضع حامل العينة على المجهر وتأمينه في مكانه.
- تهيئة واجهة المستخدم الرسومية (GUI) للتحكم في كاميرا المجهر، مرحلة العينة، وأشعة الليزر الإثارة. هنا، يتم استخدام البرامج المكتوبة حسب الطلب في MATLAB للتحكم في الآلات.
- تهيئة برنامج الكاميرا HCImage لايف. في علامة التبويب التقاط ضمن قسم التحكم في الكاميرا، قم بتعيين وقت التعرض إلى 0.025 s. انقر فوق Live لبدء بث مباشر للكاميرا.
- ضبط مواقف X و Y من مرحلة المجهر عن طريق النقر على الأسهم XY-نقاط تحت قسم مرحلة تحديد المواقع الدقيقة من واجهة المستخدم الرسومية لمسح حول العينة والعثور على FOV مع عدد كثيف بشكل مناسب من الخلايا البكتيرية.
ملاحظة: لزيادة الإنتاجية البيانات، يجب أن تكون الخلايا كثيفة قدر الإمكان، دون تداخل أو لمس الخلايا. وينبغي أن تشمل FOV أيضا على الأقل 1 حبة الفلورسنت لاستخدامها كعلامة fiducial، ويفضل وضعها في زاوية من FOV. - ضبط موقف Z من مرحلة المجهر عن طريق النقر على الأسهم Z-نقاط تحت قسم مرحلة تحديد المواقع نانو من واجهة المستخدم الرسومية، بحيث الفصوص DHPSF حبة الفلورسنت عمودي.
- تحت التسلسل | إعدادات المسح الضوئي، تغيير عدد عدد الإطارات إلى 20,000. اختر مجلد حفظ الوجهة بالنقر على الزر المسمى .... وأخيرا انقر فوق ابدأ لجمع ما يصل إلى 20،000 إطارات الكاميرا باستخدام وقت التعرض القصير من 0.025 s. يتم بدء eYFP photoblinking باستخدام ضوء الإثارة عالية الكثافة في 514 نانومتر19،25.
ملاحظة: هنا، يتم استخدام كثافة الليزر من ~ 350 W/cm2 في المستوى البؤري للتبييض الأولي والتصوير اللاحق لجزيئات EYFP واحدة. لم يتم استخدام التنشيط الضوئي لجزيئات eYFP في أطوال موجية الأشعة فوق البنفسجية أثناء التصوير. يجب أن يكون هناك على الأكثر إشارة جزيء واحد لكل خلية بكتيرية. إذا كانت كثافة إشارة جزيء واحد عالية جدا في البداية، استمر في إلقاء الضوء حتى يحدث ما يكفي من تبييض الصور قبل البدء في الحصول على البيانات. - قم بإيقاف تشغيل إضاءة الليزر بالنقر فوق إغلاق ليزر 515 نانومتر في واجهة المستخدم الرسومية. جمع 200 لقطة من الصور الداكنة باستخدام نفس وقت التعرض.
- في واجهة المستخدم الرسومية، حدد المربع بجوار Thorlabs LED وانقر فوق تبديل النسخ المتطابق . سيؤدي ذلك إلى تحويل المسار من مسار الفلورة إلى مسار تباين المرحلة.
- تهيئة برنامج الحصول على البيانات IC Capture 2.4. يتحكم هذا في الكاميرا في مسار تباين المرحلة. اضغط على الزر شاشة البدء/التوقف المباشر لعرض بث مباشر من الكاميرا. انقر فوق التقاط | حفظ صورة لتجميع صورة تباين المرحلة للخلايا في حقل العرض.
- كرر الخطوات من 3.5 إلى 3.10 لFOVs إضافية. هنا، يتم استخدام البيانات التي تم الحصول عليها من حوالي 500 خلية بكتيرية في عشرة FOVs مختلفة لزيادة عدد مسارات جزيء واحد المتاحة للتحليل.
تحذير: الخلايا المثبتة على منصات أجاروز لفترات طويلة من الزمن قد يتصرف بشكل مختلف عن الخلايا المثبتة حديثا. بالإضافة إلى ذلك، قد تفقد لوحة أغاروز سلامتها بعد مرور بعض الوقت، مما قد يؤثر سلباً على جودة البيانات. عادة، على الأكثر 3 FOV (~ 30 دقيقة على المجهر) وتستخدم في الشريحة عينة.
4 - تجهيز البيانات
ملاحظة: يتم استخدام إصدار معدل من برنامج Easy-DHPSF24 في MATLAB لتحليل إطارات الكاميرا الخام لاستخراج التعريب جزيء واحد. يستخدم Easy-DHPSF على وجه التحديد لتناسب تعريب DHPSF في التصوير أحادي الجزيء. تم إجراء تغييرات مخصصة لتنفيذ روتين التركيب المستند إلى تقدير الاحتمال الأقصى (MLE) الذي يفسر خصائص الضوضاء المعتمدة على البكسل لكاميرات sCMOS الحديثة26. كما تم تعديله لقبول إخراج نوع ملف الصورة من برنامج HCImage Live (.dcimg). للحصول على شرح أكثر تفصيلا ً للبرنامج والخطوات الفردية، يرجى الاطلاع على ليو وآخرون24
- تهيئة واجهة المستخدم الرسومية سهلة DHPSF في MATLAB(الشكل التكميلي 2). تحميل في الملف الذي تم حفظه في الخطوة 1.16.8.
- تحديد قيم العتبة لكل إخراج قوالب 7 في الخطوة 1.16.7
- ضمن المقطع معايرة تعريف SM ، انقر فوق تشغيل. انقر فوق موافق على النوافذ المنبثقة جهازي التالية للاحتفاظ بالإعدادات الافتراضية.
- افتح مكدس الصور الذي يحتوي على البيانات من FOV الأول عند المطالبة.
- اختر مجموعة صغيرة من الإطارات لمطابقة القوالب. عادة ما تستخدم إطارات 1001-2000 لتجنب إشارات متداخلة كثيفة في أول عدة مئات من الإطارات. انقر فوق موافق في الإطار المنبثق التالي للاحتفاظ بالإعدادات الافتراضية. انقر فوق إلغاء الأمر عند مطالبتك بملف سجل التسلسل في الإطار التالي.
- افتح مكدس الصور مع الخلفية الداكنة المحفوظة في الخطوة 3.8. انقر فوق "موافق" في الإطار المنبثق التالي لترك المعلمات لتقدير الخلفية تعيينها إلى افتراضي. الافتراضي هو تقدير الخلفية باستخدام عامل تصفية متوسط27 تغطي 100 إطارات الكاميرا اللاحقة حول الإطار الحالي.
- في الإطار التالي، قم بتغيير حجم المربع المراكب على الصورة لتغطية FOV الكامل، ثم انقر نقراً مزدوجاً للمتابعة.
- حدد المنطقة ذات الأهمية بالنقر فوق عدة نقاط على الصورة لإنشاء مضلع. يجب أن تشمل المنطقة ذات الاهتمام أكبر قدر ممكن من مجال الرؤية، مع ضمان عدم وجود أي حبات فلورسنت (كائنات مشرقة جداً) في الصورة داخل المضلع، ثم انقر نقرًا مزدوجًا للمتابعة.
ملاحظة: سيحاول البرنامج بعد ذلك مطابقة القوالب إلى الصورة ثم سيتم عرض صورة مع التطابقات المحتملة دائرة. - عند توقف البرنامج، فإنه سيتم حفظ العديد من الصور من مطابقة القالب وجدت وعرض قيمة العتبة المقابلة في مجلد محدد مسبقا. قيمة عتبة أعلى يتوافق مع تطابق أفضل. لكل رقم قالب، قم بفحص تطابق المثال وتحديد العتبة الأدنى التي تعرض صورة DHPSF. إدخال هذه العتبات لكل من القوالب 7 ضمن قسم معايرة تعريف SM في واجهة المستخدم الرسومية سهلة DHPSF.
ملاحظة: سيحاول البرنامج تلقائياً اختيار وإدخال عتبات ومع ذلك، غالباً ما تكون هذه غير موثوق بها ويجب التحقق يدوياً. يتم اختيار العتبات بحيث يتم تفويت عدد قليل من الإشارات الحقيقية ذات الجزيء الواحد، ولكن عدد المرشحين الإيجابيين المزيفين للتركيب لا يزال قابلاً للإدارة من الناحية الحسابية. - حفظ واجهة المستخدم الرسومية سهلة DHPSF مرة أخرى عن طريق النقر على أيقونة حفظ في الزاوية اليسرى العليا.
- تركيب حبة الفلورسنت في FOV لاستخدامها كعلامة fiducial
- ضمن قسم تتبع التوكيلات من واجهة المستخدم الرسومية سهلة DHPSF ، انقر فوق تشغيل. انقر فوق موافق على النوافذ المنبثقة جهازي التالية للاحتفاظ بالإعدادات الافتراضية.
- اسحب المربع المُرَصّع على الصورة ومركزه فوق إشارة DHPSF من حبة الفلورسنت، ثم انقر نقرًا مزدوجًا.
- انقر في وسط إشارة DHPSF، في نقطة الوسط بين الفصوص اثنين، ثم ضرب أدخل. انقر فوق إلغاء الأمر عند مطالبتك بملف سجل التسلسل في الإطار التالي.
ملاحظة: البرنامج سوف تناسب DHPSF في جميع إطارات الكاميرا، وعرض الصورة الخام والصورة التي أعيد بناؤها. عند الانتهاء من البرنامج، فإنه سيتم إخراج الأرقام مع X، Y، وZ مواقف من حبة الفلورسنت على مدى مدة الحصول على الصورة. - حدد المربع بجوار استخدام الجهات الائتمانية وحفظ واجهة المستخدم الرسومية سهلة DHPSF مرة أخرى بالنقر فوق رمز حفظ في الزاوية العلوية اليسرى.
- البحث عن كافة عمليات الترجمة واحتواؤها في كافة إطارات الكاميرا باستخدام عتبات القالب التي تم الحصول عليها في الخطوة 4.2.
- ضمن المقطع ترجمة DHPSF SMs من واجهة المستخدم الرسومية سهلة DHPSF ، انقر فوق تشغيل. انقر فوق موافق على الإطارات المنبثقة التالية للاحتفاظ بالإعدادات الافتراضية. انقر فوق إلغاء الأمر عند مطالبتك بملف سجل التسلسل في الإطار التالي.
ملاحظة: سوف تجد البرنامج وتناسب DHPSF باستخدام نموذج غاوسي مزدوج إذا كانت جودة المطابقة أعلى من العتبة المعرفة من قبل المستخدم. فإنه سيتم عرض الصورة الخام مع دوائر حول مباريات قالب، فضلا عن صورة لDHPSF المعاد بناؤها يناسب. - حفظ واجهة المستخدم الرسومية سهلة DHPSF مرة أخرى عن طريق النقر على أيقونة حفظ في الزاوية اليسرى العليا.
- ضمن المقطع ترجمة DHPSF SMs من واجهة المستخدم الرسومية سهلة DHPSF ، انقر فوق تشغيل. انقر فوق موافق على الإطارات المنبثقة التالية للاحتفاظ بالإعدادات الافتراضية. انقر فوق إلغاء الأمر عند مطالبتك بملف سجل التسلسل في الإطار التالي.
- عرض التعريب أحادي الجزيء وتصفية التعريب غير المرغوب فيه أو غير الموثوق به
- ضمن المقطع ترجمة DHPSF الإخراج ، انقر فوق إخراج عامل التصفية.
- انقر فوق موافق في الإطارات الثلاثة التالية لإجراء استيفاء المواضع x و Y و Z fiducial مع مرور الوقت. في معظم الحالات، تكون الخيارات الافتراضية كافية. إذا كان الخط الأسود الاستيفاء لا يعكس الاستيفاء المعقول لخط الموضع الأحمر، قم بتغيير معلمات الاستيفاء في النافذة المنبثقة.
ملاحظة: يتم استخدام الخط المحرف لتصحيح التعريب أحادي الجزيء. - افتح صورة تباين المرحلة المقابلة لFOV التي يتم تحليلها عند المطالبة. انقر فوق موافق على النوافذ المنبثقة جهازي التالية للاحتفاظ بالإعدادات الافتراضية.
- في النوافذ المنبثقة اثنين التالية، تغيير قيم عامل التصفية للسماح لمتطلبات توطين جزيء واحد أكثر صرامة أو أكثر تساهلا، ثم انقر فوق موافق.
- في الإطار الذي يظهر، اسحب أو قم بتغيير حجم المربع المراكب على الصور لعرض المنطقة المرغوبة من الاهتمام وانقر نقراً مزدوجاً للمتابعة.
- يتم عرض إعادة بناء ثلاثية الدُعد لتوطين الجزيء الواحد. استخدام أدوات الشكل للتعامل مع إعادة الإعمار (تدوير، التكبير، الخ). انقر فوق متابعة لعرض مربع حوار آخر يسأل'هل تريد إعادة رسم مع مجموعة معلمة مختلفة؟'. إذا كانت النتائج مرضية، انقر فوق "لا". إذا لم تكن كذلك، انقر فوق نعم.
ملاحظة: تتضمن الأسباب الشائعة للنتائج غير المرضية صورة تباين المرحلة غير متراكبة بشكل صحيح مع بيانات الترجمة أو عتبات القالب الأولي كانت منخفضة جداً إنشاء العديد من عمليات التعريب الإيجابية الكاذبة. إذا تم تحديد "نعم"، سيعود البرنامج إلى الخطوة 4.5.4 بحيث يمكن تعريف قيم المعلمات الجديدة. إذا لم يتم تحديد أي، سيتم حفظ النتائج.
5 - تجهيز البيانات بعد التجهيز
- باستخدام البرامج المكتوبة حسب الطلب في MATLAB، قم باقتصاص صورة تباين المرحلة بحيث تبقى المنطقة التي تحتوي على الخلايا التي تم تصويرها تحت المجهر الفلوري فقط. هذه الخطوة ضرورية لأن صورة تباين المرحلة تغطي منطقة أكبر بكثير من صورة الفلورة. تبسط قص الصورة الخطوة التالية.
- تقسيم الخلايا الفردية عن طريق معالجة صورة تباين المرحلة مع برنامج OUFTI28 (الشكل التكميلي 3)
- تهيئة OUFTI في MATLAB. قم بتحميل صورة تباين المرحلة المقطوعة من الخطوة السابقة بالنقر على مرحلة التحميل.
- انقر فوق ملف لاختيار وتسمية موقع حفظ لملف الإخراج.
- حدد إطارات مستقلة ضمن عنوان الكشف والتحليل.
- انقر فوق معلمات التحميل لتحميل معلمات الكشف عن الخلايا. تتضمن أمثلة المعلمات منطقة الخلية المقبولة وعرض الخلية وعتبة تقسيم الخلية.
ملاحظة: يجب ضبط كافة هذه المعلمات لزيادة الأداء لأحجام الخلايا المحددة وجودة الصورة المستخدمة. الأهم من ذلك، يجب تعيين معلمة الخوارزمية إلى subpixel للسماح بقياس دقيق للخطوط العريضة للخلية. - انقر فوق هذا الإطار لبدء تجزئة الخلايا. ستظهر الخطوط العريضة للخلية عبر صورة تباين المرحلة عند انتهاء العملية.
- باستخدام عناصر التحكم ضمن المقطع يدوي تقسيم الخلايا أو إضافة خلايا أو تحسين مخططات الخلايا للحصول على مخططات تفصيلية للخلايا التي تم تقسيمها بشكل غير دقيق أثناء العملية التلقائية.
- إخراج الخطوط العريضة للخلية بالنقر فوق حفظ التحليل.
- استخدم البرامج المكتوبة حسب الطلب في MATLAB لتراكب الخطوط العريضة التي تم الحصول عليها في الخطوة السابقة باستخدام عمليات التعريب ذات الجزيء الواحد. توضح الخطوات الفرعية التالية خطوات البرنامج بالتفصيل.
- حدد يدويًا 5 أزواج نقاط تحكم في النافذة المنبثقة عن طريق تقدير موضع أعمدة الخلايا لنفس الخلايا الخمس والنقر عليها تقريبًا في كل من بيانات تعريب الجزيء الواحد والخطوط العريضة للخلايا، التي تم إنشاؤها في الخطوة السابقة. ويمكن تقدير موقف عمود الخلية تقريبا عن طريق رسم عقليا بدن محدب حول توطين جزيء واحد ينتمي إلى خلية واحدة واختيار نقطة من أعلى انحناء(الشكل التكميلي 4).
- إنشاء وظيفة تحويل affine 2D باستخدام وظيفة cp2tform في MATLAB واستخدامها لتوليد تراكب الخام من الخطوط العريضة للخلية وتوطين جزيء واحد.
- حذف الخلايا التي تحتوي على أقل من 10 تعريب وإزالة الخلايا التي تم وضعها جزئيا خارج حقل العرض. حذف يدويا أي خلايا إضافية غير مرغوب فيها في النافذة المنبثقة عن طريق النقر داخل مخطط الخلية الخاصة بهم(الشكل التكميلي 5).
- استخدام مركز الكتلة لجميع الخطوط العريضة الخلية المتبقية وتوطين جزيء واحد داخلها لتشكيل مجموعة أكبر من أزواج نقطة التحكم، وإعادة حساب وظيفة التحويل 2D، واستخدامها لتوليد تراكب النهائي من الخطوط العريضة الخلية و توطين جزيء واحد.
- تعيين التعريب الموجودة داخل حدود مخطط الخلايا إلى تلك الخلية. تجاهل أي توطينات غير موجودة ضمن أي مخطط تفصيلي للخلية(الشكل 2أ، الشكل التكميلي 6).
6. تتبع جزيء واحد
ملاحظة: يتم إكمال المقطع التالي باستخدام البرامج المكتوبة حسب الطلب في MATLAB. يصف هذا القسم الخطوات التي ينفذها البرنامج.
- بالنسبة للتعريب المعين لنفس الخلية وفي إطارات الكاميرا اللاحقة، قم بحساب المسافة الإقليدية بين التعريب. إذا كانت المسافة بين التعريب أقل من عتبة 2.5 ميكرومتر، قم بربط التعريب عن طريق تعيينها إلى نفس مسار جزيء واحد.
ملاحظة: من المهم فقط النظر في التعريب داخل خلية واحدة بحيث لا يتم ربط التعريب في الخلايا المجاورة التي تحدث لتلبية متطلبات العتبة المكانية والزمنية. تم اختيار عتبة 2.5 ميكرومتر كمسافة قصوى يمكن لجزيء سريع جدا (30 ميكرومتر2/ثانية) السفر في طول وقت التعرض (0.025 ثانية) بالإضافة إلى المخزن المؤقت 20٪. - تجاهل مسارات أقصر من 4 عمليات تعريب. إذا كان اثنان أو أكثر من التعريب (أي اثنين أو أكثر من باعثات الفلور) موجودة في وقت واحد في خلية، تجاهل المسارات المرتبطة بها. يتيح تعيين طول المسار على الأقل إلى 4 تعريبات أن تكون قياسات المسافة العديدة متوسطة مما يؤدي إلى تقدير أكثر دقة لمعاملات الانتشار.
- حساب متوسط الإزاحة التربيعية (MSD) لمسار معين بواسطة:
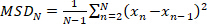 (1)
(1)
حيث N هو العدد الإجمالي للتعريب في المسار و xn هو موضع الجزيء في الوقتn نقطة . - حساب معامل الانتشار الظاهر، D* بواسطة
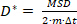 (2)
(2)
حيث m = 2 أو 3 هو بعد القياس وΔt هو وقت التعرض للكاميرا.
ملاحظة: سوف تنتج تجربة نموذجية ما بين 5,000-100,000 مسار في المجموع، مما يؤدي إلى توزيع معامل الانتشار الظاهر مع هذا العدد الكبير.
7. محاكاة مونتي كارلو للحركة براونيان في حجم ضيق
ملاحظة: إنشاء مكتبات لتوزيعات معامل الانتشار الظاهرة المحاكاة عن طريق إجراء محاكاة مونتي كارلو للحركة البراونية تقتصر على حجم أسطواني، باستخدام 64 قيمة في نطاق 0.05-20 ميكرومتر2/s كمعلمات إدخال (برنامج متاح من أصحاب البلاغ عند الطلب). وقد تم اختيار هذا النطاق لتغطية مجموعة من معاملات الانتشار المقدرة سابقا من البروتينات الفلورية (الانصهار) في البكتيريا. يتم استخدام 64 معامل انتشار لأخذ عينات من هذا النطاق بما فيه الكفاية. يتم تنفيذ هذا القسم باستخدام برامج مكتوبة حسب الطلب في MATLAB ويصف الخطوات التي يتخذها البرنامج تلقائيًا. يتم تقريب خلايا Y. enterocolitica على شكل قضيب المستخدمة هنا بحجم أسطواني من الطول l = 5 ميكرومتر وقطر d = 0.8 ميكرومتر.
- بدء مسارات فردية في وضع عشوائي في الحجم الأسطواني، ومحاكاة خطوات الانتشار العشوائية (أي Brownian) باستخدام فاصل زمني قدره 100 ns (ينبغي أن يكون الفاصل الزمني أقصر بكثير من وقت التعرض للكاميرا لتمكين كافية أخذ العينات من الموقف على مدى إطار الكاميرا). أخذ عينة من كل خطوة إزاحة لإدخال D من وظيفة التوزيع الغوسية المقابلة عن طريق إعادة ترتيب Eqn. 2 وإضافتها إلى الموضع السابق:
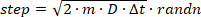 (3)
(3)
حيث الدالة راندن في MATLAB عينات رقم عشوائي من توزيع عادي. إذا كانت خطوة تسبب الجزيء إلى إزاحة خارج حجم الاسطوانة، تعكس جزيء مرة أخرى داخل الاسطوانة في زاوية عشوائية. - لكل فاصل زمني، قم بإنشاء صورة DHPSF التي تتوافق مع موضع x وy و z الفوري للمرسل المحاكي.
- لمطابقة الظروف التجريبية، قم بمحاكاة الصور التي تحتوي على 1000 فوتون لكل توطين، مع خلفية ليزر ية من 13 فوتون لكل بكسل، وضوضاء بواسون. بالإضافة إلى ذلك، أضف إزاحة داكنة من حوالي 50 فوتونًا لكل بكسل وضوضاء قراءة غاوسي (حوالي 1.5 فوتون)، بما يتفق مع قياسات معايرة الكاميرا التجريبية. وأخيرا، ضرب الصورة من قبل كسب البكسل قياس تجريبيا من الكاميرا sCMOS للحصول على الصورة في وحدات من التهم كاشف.
ملاحظة: بعد هذه المناورات، نسبة الإشارة إلى الضوضاء للصورة النهائية ~ 2. - توليد الصور غير واضحة الحركة التي تعكس الموقف المتغير للجزيئات عن طريق جمع 50 صورة DHPSF محاكاة خلال وقت التعرض المستخدمة أثناء الحصول على البيانات التجريبية. وللحد من المصروفات الحسابية، تم اختيار 50 وظيفة فقط يتم أخذ عينات منها بشكل دوري لإنشاء صورة (بدلاً من جميع المراكز الـ 250,000 التي تم أخذ عينات منها خلال وقت التعرض 0.025 s في فاصل أخذ العينات 100 ns).
ملاحظة: ينبغي أن يتطابق طول (عدد الإطارات) للمسارات المحاكاة مع متوسط طول المسارات التجريبية. في هذه الحالة، عدد الإطارات لكل مسار هو 6.
- لمطابقة الظروف التجريبية، قم بمحاكاة الصور التي تحتوي على 1000 فوتون لكل توطين، مع خلفية ليزر ية من 13 فوتون لكل بكسل، وضوضاء بواسون. بالإضافة إلى ذلك، أضف إزاحة داكنة من حوالي 50 فوتونًا لكل بكسل وضوضاء قراءة غاوسي (حوالي 1.5 فوتون)، بما يتفق مع قياسات معايرة الكاميرا التجريبية. وأخيرا، ضرب الصورة من قبل كسب البكسل قياس تجريبيا من الكاميرا sCMOS للحصول على الصورة في وحدات من التهم كاشف.
- توليد 5000 مسار لكل معامل من معاملات نشر المدخلات المحاكاة الـ 64.
- تحليل إطارات الكاميرا المحاكية كما هو موضح في قسم معالجة البيانات.
- ربط التعريب في المسارات كما هو موضح في قسم تتبع جزيء واحد.
- استيفاء وظيفة التوزيع التراكمي (CDF) لكل توزيع محاكاة باستخدام الاستيفاء B-spline من أجل 25. ومن الضروري إجراء عمليات توزيع محرفة بحيث يمكن الاستفسار عنها في نقاط تعسفية.
- الاستيفاء المنحنيات B-spline الناتجة على طول المحور D(D هو معامل الانتشار غير المحصور الحقيقي الذي يحكم حركة الباعثين المحاكيين) باستخدام وظيفة المحاكاة المتناثرة في MATLAB (حدد ' الطبيعية ' طريقة الاستيفاء). وهذا يوفر وظيفة 2D مستمرة يمكن من خلالها الاستعلام عن أي توزيع واضح لمعامل الانتشار يقابلقيمة معامل انتشار حقيقي في نطاق 0.05-20 ميكرومتر 2/ثانية.
8- تركيب معامل الانتشار الظاهر التجريبي
ملاحظة: ملائمة التوزيعات المقاسة تجريبياً لمعاملات الانتشار الظاهرة باستخدام تركيبات خطية من التوزيعات المحاكاة التي تم إنشاؤها في القسم السابق(محاكاة مونتي كارلو لحركة براونيان في مجلد ضيق). يتم تنفيذ هذا القسم باستخدام برامج مكتوبة حسب الطلب في MATLAB ويصف الخطوات التي يتخذها البرنامج تلقائيًا. لمزيد من المعلومات والأمثلة على التطبيق، يرجى الاطلاع على روشا وآخرون29
- تنفيذ تناسب المربعات الخطية الأقل مقيدة (باستخدام الدالة lsqlin في MATLAB) من CDF التجريبية باستخدام صفيف عينات دورية من CDFs محاكاة من المكتبة التي تم إنشاؤها في القسم 7. ناتج هذه الخطوة هو متجه معلمة تحتوي على معاملات الانتشار والكسور السكانية للحالات المشتتة السائدة في التوزيع التجريبي.
- الجمع بين حالات عدم الانتشار مع قيم معامل الانتشار داخل 20٪ من بعضها البعض في حالة واحدة من عدم الانتشار حسب متوسط الوزن على أساس الكسور السكانية النسبية. هذا هو متجه معلمة البداية.
ملاحظة: لتقليل تعقيد النموذج، يمكن أن تكون الحالات المشتتة مع معاملات الانتشار أقل من 0.5 ميكرومتر2/ثانيةثابتة أثناء كافة الخطوات التالية. - إنشاء صفائف من ناقلات المعلمة المناسب التجريبي مع أرقام مختلفة من حالات عدم الاستخدام، تتراوح من حالة واحدة غير إلزامية إلى الحد الأقصى لعدد الحالات المعرفة من قبل المستخدم.
- باستخدام متجه معلمة البداية، الجمع بين الدول المتاخمة المجاورة من خلال المتوسط المرجح وتقسيم حالات الانقسام إلى دولتين مع كسور السكان المتساوية ومعاملات الانتشار 20٪ فوق وأسفل القيمة الأصلية. كرر لجميع مجموعة الدولة وتقسيم الاحتمالات.
- استخدم كل متجه معلمة تركيب تجريبي لتهيئة تركيب مربع غير خطي أقل من 5 مجموعات فرعية منفصلة من البيانات (باستخدام الدالة fmincon في MATLAB). تحديد جودة الاحتواء عن طريق العثور على مجموع المربعات المتبقية بين الاحتواء والتوزيع المقابل ة للمجموعات الفرعية المتبقية (التحقق من صحة البيانات عبر).
- استخدام متوسط المبلغ المتبقي من المربعات من 5 التجهيزات منفصلة لكل ناقلات المحاكمة كنوعية عامة من صالح, تحديد ناقلات المحاكمة مع أفضل نوعية من صالح لكل عدد من الدول الناشر.
- تحديد العدد الأمثل من الحالات عن طريق تحديد متجه المحاكمة التي إضافة حالة إضافية لا يؤدي إلى تحسن 5% على الأقل في نوعية صالح.
- استخدم هذا المتجه التجريبي لتهيئة المربعات الأقل غير الخطية المناسبة لمجموعة البيانات الكاملة.
- تقدير الخطأ للمعلمات الفردية عن طريق إعادة أخذ عينات من البيانات عن طريق ربط bootstrapping وإعادة تركيب نفس متجه التجربة.
النتائج
في ظل الظروف التجريبية الموصوفة هنا (20،000 لقطة، طول المسار الحد الأدنى من 4 التعريب) واعتمادا على مستويات التعبير من البروتينات الانصهار المسمى الفلورسنت، ما يقرب من 200-3،000 التعريب تسفر عن 10-150 يمكن إنشاء مسارات لكل خلية(الشكل 2a،ب). وهناك عدد كبير م...
Discussion
والعامل الحاسم للتطبيق الناجح للبروتوكول المعروض هو ضمان فصل إشارات الجزيء الواحد بشكل جيد عن بعضها البعض (أي أنها تحتاج إلى أن تكون متفرقة في الفضاء وفي الوقت المناسب(Supplementary Mov. 1)). إذا كان هناك أكثر من جزيء مفلور واحد في خلية في نفس الوقت، ثم يمكن تعيين التعريب بشكل غير صحيح إلى م...
Disclosures
وليس لدى أصحاب البلاغ ما يكشفون عنه.
Acknowledgements
نشكر أليسيا أكيموفيتش وتينغ يان على قراءتهما النقدية للمخطوطة. نشكر إد هول، كبير علماء الموظفين في مجموعة خدمات الحوسبة البحثية المتقدمة في جامعة فرجينيا، على المساعدة في إعداد إجراءات التحسين المستخدمة في هذا العمل. وقدمت جامعة فرجينيا التمويل اللازم لهذا العمل.
Materials
| Name | Company | Catalog Number | Comments |
| 2,6-diaminopimelic acid | Chem Impex International | 5411 | Necessary for growth of Y. enterocolitica cells used. |
| 4f lenses | Thorlabs | AC508-080-A | f = 80mm, 2" |
| 514 nm laser | Coherent | Genesis MX514 MTM | Use for fluorescence excitation |
| agarose | Inivtrogen | 16520100 | Used to make gel pads to mount liquid bacterial sample on microscope. |
| ammonium chloride | Sigma Aldrich | A9434 | M2G ingredient. |
| bandpass filter | Chroma | ET510/bp | Excitation pathway. |
| Brain Heart Infusion | Sigma Aldrich | 53286 | Growth media for Y. enterocolitica. |
| calcium chloride | Sigma Aldrich | 223506 | M2G ingredient. |
| camera | Imaging Source | DMK 23UP031 | Camera for phase contrast imaging. |
| dielectric phase mask | Double Helix, LLC | N/A | Produces DHPSF signal. |
| disodium phosphate | Sigma Aldrich | 795410 | M2G ingredient. |
| ethylenediaminetetraacetic acid | Fisher Scientific | S311-100 | Chelates Ca2+. Induces secretion in the T3SS. |
| flip mirror | Newport | 8892-K | Allows for switching between fluorescence and phase contrast pathways. |
| fluospheres | Invitrogen | F8792 | Fluorescent beads. 540/560 exication and emission wavelengths. 40 nm diameter. |
| glass cover slip | VWR | 16004-302 | #1.5, 22mmx22mm |
| glucose | Chem Impex International | 811 | M2G ingredient. |
| immersion oil | Olympus | Z-81025 | Placed on objective lens. |
| iron(II) sulfate | Sigma Aldrich | F0518 | M2G ingredient. |
| long pass filter | Semrock | LP02-514RU-25 | Emission pathway. |
| magnesium sulfate | Fisher Scientific | S25414A | M2G ingredient. |
| microscope platform | Mad City Labs | custom | Platform for inverted microscope. |
| nalidixic acid | Sigma Aldrich | N4382 | Y. enterocolitica cells used are resistant to nalidixic acid. |
| objective lens | Olympus | 1-U2B991 | 60X, 1.4 NA |
| Ozone cleaner | Novascan | PSD-UV4 | Used to eliminate background fluorescence on glass cover slips. |
| potassium phosphate | Sigma Aldrich | 795488 | M2G ingredient. |
| Red LED | Thorlabs | M625L3 | Illuminates sample for phase contrast imaging. 625nm. |
| sCMOS camera | Hamamatsu | ORCA-Flash 4.0 V2 | Camera for fluorescence imaging. |
| short pass filter | Chroma | ET700SP-2P8 | Emission pathway. |
| Tube lens | Thorlabs | AC508-180-A | f=180 mm, 2" |
| Yersinia enterocolitica dHOPEMTasd | N/A | N/A | Strain AD4442, eYFP-YscQ |
| zero-order quarter-wave plate | Thorlabs | WPQ05M-514 | Excitation pathway. |
References
- Kapanidis, A. N., Uphoff, S., Stracy, M. Understanding Protein Mobility in Bacteria by Tracking Single Molecules. Journal of Molecular Biology. , (2018).
- Betzig, E., et al. Imaging intracellular fluorescent proteins at nanometer resolution. Science. 313 (5793), 1642-1645 (2006).
- Hess, S. T., Girirajan, T. P. K., Mason, M. D. Ultra-high resolution imaging by fluorescence photoactivation localization microscopy. Biophysical Journal. 91 (11), 4258-4272 (2006).
- Rust, M. J., Bates, M., Zhuang, X. W. Sub-diffraction-limit imaging by stochastic optical reconstruction microscopy (STORM). Nature Methods. 3 (10), 793-795 (2006).
- Chen, T. Y., et al. Quantifying Multistate Cytoplasmic Molecular Diffusion in Bacterial Cells via Inverse Transform of Confined Displacement Distribution. Journal of Physical Chemistry B. 119 (45), 14451-14459 (2015).
- Mohapatra, S., Choi, H., Ge, X., Sanyal, S., Weisshaar, J. C. Spatial Distribution and Ribosome-Binding Dynamics of EF-P in Live Escherichia coli. mBio. 8 (3), (2017).
- Stracy, M., et al. Single-molecule imaging of UvrA and UvrB recruitment to DNA lesions in living Escherichia coli. Nature Communications. 7, 12568 (2016).
- Persson, F., Lindén, M., Unoson, C., Elf, J. Extracting intracellular diffusive states and transition rates from single-molecule tracking data. Nature Methods. 10 (3), 265-269 (2013).
- Bakshi, S., Choi, H., Weisshaar, J. C. The spatial biology of transcription and translation in rapidly growing Escherichia coli. Frontiers in Microbiology. 6, 636 (2015).
- Mustafi, M., Weisshaar, J. C. Simultaneous Binding of Multiple EF-Tu Copies to Translating Ribosomes in Live Escherichia coli. mBio. 9 (1), (2018).
- Michalet, X., Berglund, A. J. Optimal diffusion coefficient estimation in single-particle tracking. Physical Review E. 85 (6), (2012).
- Michalet, X. Mean square displacement analysis of single-particle trajectories with localization error: Brownian motion in an isotropic medium. Physical Review E Statistical, Nonlinear, and Soft Matter Physics. 82 (4 Pt 1), 041914 (2010).
- Backlund, M. P., Joyner, R., Moerner, W. E. Chromosomal locus tracking with proper accounting of static and dynamic errors. Physical Review E Statistical, Nonlinear, and Soft Matter Physics. 91 (6), 062716 (2015).
- Stracy, M., et al. Live-cell superresolution microscopy reveals the organization of RNA polymerase in the bacterial nucleoid. Proceedings of the National Academy of Sciences of the United States of America. 112 (32), E4390-E4399 (2015).
- Plochowietz, A., Farrell, I., Smilansky, Z., Cooperman, B. S., Kapanidis, A. N. In vivo single-RNA tracking shows that most tRNA diffuses freely in live bacteria. Nucleic Acids Research. 45 (2), 926-937 (2017).
- Koo, P. K., Mochrie, S. G. Systems-level approach to uncovering diffusive states and their transitions from single-particle trajectories. Physical Review E. 94 (5-1), 052412 (2016).
- Uphoff, S., Reyes-Lamothe, R., Garza de Leon, F., Sherratt, D. J., Kapanidis, A. N. Single-molecule DNA repair in live bacteria. Proceedings of the National Academy of Sciences of the United States of America. 110 (20), 8063-8068 (2013).
- Bakshi, S., Bratton, B. P., Weisshaar, J. C. Subdiffraction-Limit Study of Kaede Diffusion and Spatial Distribution in Live Escherichia coli. Biophysical Journal. 101 (10), 2535-2544 (2011).
- Bakshi, S., Siryaporn, A., Goulian, M., Weisshaar, J. C. Superresolution imaging of ribosomes and RNA polymerase in live Escherichia coli cells. Molecular Microbiology. 85 (1), 21-38 (2012).
- Uphoff, S., Sherratt, D. J., Kapanidis, A. N. Visualizing Protein-DNA Interactions in Live Bacterial Cells Using Photoactivated Single-molecule Tracking. JoVE. (85), e51177 (2014).
- Pavani, S. R. P., Piestun, R. Three dimensional tracking of fluorescent microparticles using a photon-limited double-helix response system. Optics Express. 16 (26), 22048-22057 (2008).
- Pavani, S. R. P., et al. Three-dimensional, single-molecule fluorescence imaging beyond the diffraction limit by using a double-helix point spread function. Proceedings of the National Academy of Sciences of the United States of America. 106 (9), 2995-2999 (2009).
- Rocha, J. M., et al. Single-molecule tracking in live Yersinia enterocolitica reveals distinct cytosolic complexes of injectisome subunits. Integrative Biology. 10 (9), 502-515 (2018).
- Lew, M. D., von Diezmann, A. R. S., Moerner, W. E. Easy-DHPSF open-source software for three-dimensional localization of single molecules with precision beyond the optical diffraction limit. Protocol Exchange. , (2013).
- Biteen, J. S., et al. Super-resolution imaging in live Caulobacter crescentus cells using photoswitchable EYFP. Nature Methods. 5 (11), 947-949 (2008).
- Huang, F., et al. Video-rate nanoscopy using sCMOS camera-specific single-molecule localization algorithms. Nature Methods. 10 (7), 653-658 (2013).
- Hoogendoorn, E., et al. The fidelity of stochastic single-molecule super-resolution reconstructions critically depends upon robust background estimation. Scientific Reports. 4, 3854 (2014).
- Paintdakhi, A., et al. Oufti: An integrated software package for high-accuracy, high-throughput quantitative microscopy analysis. Molecular microbiology. 99 (4), 767-777 (2016).
- Rocha, J. M., Corbitt, J., Yan, T., Richardson, C., Gahlmann, A. Resolving Cytosolic Diffusive States in Bacteria by Single-Molecule Tracking. bioRxiv. , 483321 (2018).
- Lee, A., Tsekouras, K., Calderon, C., Bustamante, C., Presse, S. Unraveling the Thousand Word Picture: An Introduction to Super-Resolution Data Analysis. Chemical Reviews. 117 (11), 7276-7330 (2017).
- Los, G. V., et al. HaloTag: A Novel Protein Labeling Technology for Cell Imaging and Protein Analysis. ACS Chemical Biology. , (2008).
- Gautier, A., et al. An Engineered Protein Tag for Multiprotein Labeling in Living Cells. Chemistry & Biology. 15 (2), 128-136 (2008).
- Bisson-Filho, A. W., et al. Treadmilling by FtsZ filaments drives peptidoglycan synthesis and bacterial cell division. Science. 355 (6326), 739-743 (2017).
- Douglass, K. M., Sieben, C., Archetti, A., Lambert, A., Manley, S. Super-resolution imaging of multiple cells by optimised flat-field epi-illumination. Nature Photonics. 10 (11), 705-708 (2016).
- Zhao, Z., Xin, B., Li, L., Huang, Z. L. High-power homogeneous illumination for super-resolution localization microscopy with large field-of-view. Optics Express. 25 (12), 13382-13395 (2017).
- Yan, T., Richardson, C. J., Zhang, M., Gahlmann, A. Computational Correction of Spatially-Variant Optical Aberrations in 3D Single Molecule Localization Microscopy. bioRxiv. , 504712 (2018).
- Gahlmann, A., Moerner, W. E. Exploring bacterial cell biology with single-molecule tracking and super-resolution imaging. Nature Reviews Microbiology. 12 (1), 9-22 (2014).
- Liu, Z., Lavis, L. D., Betzig, E. Imaging live-cell dynamics and structure at the single-molecule level. Mol Cell. 58 (4), 644-659 (2015).
- Berglund, A. J. Statistics of camera-based single-particle tracking. Physical Review E. 82 (1), 011917 (2010).
- Parry, B. R., et al. The bacterial cytoplasm has glass-like properties and is fluidized by metabolic activity. Cell. 156 (1-2), 183-194 (2014).
- English, B. P., et al. Single-molecule investigations of the stringent response machinery in living bacterial cells. Proceedings of the National Academy of Sciences of the United States of America. 108 (31), E365-E373 (2011).
- Niu, L. L., Yu, J. Investigating intracellular dynamics of FtsZ cytoskeleton with photoactivation single-molecule tracking. Biophysical Journal. 95 (4), 2009-2016 (2008).
- Coquel, A. S., et al. Localization of protein aggregation in Escherichia coli is governed by diffusion and nucleoid macromolecular crowding effect. PLoS Computational Biology. 9 (4), e1003038 (2013).
- Nenninger, A., Mastroianni, G., Mullineaux, C. W. Size Dependence of Protein Diffusion in the Cytoplasm of Escherichia coli. Journal of Bacteriology. 192 (18), 4535-4540 (2010).
- Dix, J. A., Verkman, A. S. Crowding effects on diffusion in solutions and cells. Annual Review of Biophysics. 37, 247-263 (2008).
- Elliott, L. C., Barhoum, M., Harris, J. M., Bohn, P. W. Trajectory analysis of single molecules exhibiting non-brownian motion. Physical Chemistry Chemical Physics. 13 (10), 4326-4334 (2011).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved