A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
מיקרוסקופ מעקב חד-מולקולה-כלי לקביעת המדינות התפוצבניות של מולקולות ציטוסולג
In This Article
Summary
3D מיקרוסקופ לוקליזציה יחיד מולקולה מנוצל כדי לחקור את המיקומים מרחבי ומסלולי תנועה של חלבונים המסומנים באנרגיה בתאי בקטריאלי חיים. פרוטוקול ניתוח הנתונים הניסיוני והמידע המתואר להלן קובע את ההתנהגויות התפוצזות הנפוצות של חלבונים ציטוסולמים בהתבסס על מסלולים בעלי מולקולה בודדת.
Abstract
מיקרוסקופ חד-מולקולות לוקליזציה מטפל בעמדה ובתנועות של מולקולות בודדות בתאים חיים עם עשרות ברזולוציה מרחבית ומאלפית שניה של נאנמטר. יכולות אלה הופכים מיקרוסקופ חד-מולקולות לוקליזציה מתאים באופן אידיאלי לחקר פונקציות ביולוגיות ברמה המולקולרית בסביבות רלוונטיות מבחינה פיזיולוגית. כאן, אנו מדגימים פרוטוקול משולב עבור רכישת ועיבוד/ניתוח של מולקולה בודדת מעקב נתונים כדי לחלץ את המצבים המפזרים שונים חלבון הריבית עשוי להפגין. מידע זה ניתן להשתמש כדי לכמת היווצרות קומפלקס מולקולרי בתאי החיים. אנו מספקים תיאור מפורט של ניסוי לוקליזציה של מולקולה בודדת תלת-ממד המבוסס על המצלמה, כמו גם את הצעדים הבאים עיבוד נתונים התשואה מסלולים של מולקולות בודדות. מסלולים אלה נותחו לאחר מכן באמצעות מסגרת ניתוח מספרית כדי לחלץ את המצבים המפזרים הנפוצים של מולקולות המסומנות באמצעות פלואורוסקופים ואת השפע היחסי של מצבים אלה. מסגרת הניתוח מתבססת על סימולציות סטוכסטיים של מסלולים בראוניים מתחום הדיפוזיה התאית, אשר מוגבלים בגיאומטריה של תאים שרירותיים. בהתבסס על מסלולים מדומים, תמונות raw מולקולה אחת נוצרות ונותחו באותו אופן כמו תמונות ניסיוני. בדרך זו, מגבלות דיוק ודיוק נסיוניות, שקשה לכייל את הניסוי, משולבים באופן מפורש בתהליך הניתוח. מקדם הדיפוזיה ושברי האוכלוסיה היחסיים של מדינות הפיזור הנפוצות נקבעים על-ידי התאמת הפצות הערכים הניסיוניים באמצעות שילובים ליניאריים של הפצות מדומה. אנו מדגימים את השירות של הפרוטוקול שלנו על ידי פתרון המצבים המפזרים של חלבון אשר מציג מצבים מפזרים שונים על יצירת הומו-והטרוסקסואל מתחמים oligomeric ב ציטוסול של פתוגן חיידקי.
Introduction
בחינת ההתנהגות הbiomolecules מספקת תובנה לתפקודים הביולוגיים. מיקרוסקופ קרינה פלואורסצנטית מבוססי הפכו כלים יקרי ערך להתבוננות biomolecules בסביבת התא יליד שלהם. התאוששות פלואורסצנטית לאחר photobleaching לבנה (FRAP) ו ספקטרוסקופיית מתאם פלואורסצנטית (FCS)1 לספק התנהגויות ממוצעים של הרכב. לעומת זאת, מיקרוסקופ חד-מולקולות לוקליזציה מאפשר התבוננות של מולקולות מתויגות בודדות עם שתייגת מרחבית גבוהה וברזולוציה טמפורלית2,3,4. התבוננות במולקולות בודדות היא יתרון מאחר שחלבון של עניין עשוי להתקיים במצבים מפזרים שונים. לדוגמה, שני מצבים מפזרים בקלות מופיעים כאשר מווסת משתנה, כגון CueR ב- es,מפזר באופן חופשי בציטוזול, או נקשר לרצף דנ א והופך להיות מאוגד על ציר הזמן של מדידה5 . מעקב באמצעות מולקולה בודדת מספק כלי להתבוננות ישירה במצבים שונים אלה, וניתוחים מתוחכמים אינם נדרשים לפתרונן. עם זאת, הוא הופך ליותר מאתגר לפתור מצבים מפזרים מרובים ושברי האוכלוסייה שלהם במקרים שבהם שיעורי התפוצתם דומים יותר. לדוגמה, בשל התלות בגודל של מקדם הדיפוזיה, מדינות האויגמרים שונות של חלבון מתגלשות את עצמן כמצבים מפזרים שונים6,7,8,9 , 10. מקרים כאלה דורשים גישה משולבת במונחים של רכישת נתונים, עיבוד וניתוח.
גורם קריטי המשפיע על שיעורי התפוצו של מולקולות ציטוסולג היא השפעת הכליאה בגבול התא. ההגבלות המוטלות על תנועה מולקולרית על ידי גבול תא חיידקי לגרום מולקולות ציטוסולריות נמדד שיעור דיפוזיה להופיע איטי יותר מאשר אם התנועה אותה התרחשה בחלל לא סגור. עבור מולקולות מאוד איטיות, ההשפעה של הכליאה התאית היא זניחה עקב חוסר התנגשויות עם הגבול. במקרים כאלה, ייתכן שיהיה אפשר לפתור במדויק מצבים מפזרים על ידי התאמת ההפצות של displacements מולקולרית, rאו מקדמי דיפוזיה לכאורה, D *, באמצעות מודלים אנליטיים המבוססים על המשוואות לתנועה בראונית ( דיפוזיה אקראית)11,12,13. עם זאת, לצורך הפצת מולקולות ציטוסוליות מהירות, ההפצות הנסיוניות כבר אינן דומות לאלה שהתקבלו לתנועה בראונית לא מוגבלת עקב התנגשויות של מולקולות מדומות עם גבולות התא. יש לבדוק את השפעות הכליאה כדי לקבוע במדויק את מקדמי הדיפוזיה הבלתי מוגבל של המולקולות המסומנות בתווית הפלואורוסקופים. מספר גישות פותחו לאחרונה לחשבון עבור הכליאה השפעות או (semi-) באופן אנליסטי 5,14,15,16 או מספרית דרך סימולציות מונטה קרלו של דיפוזיה בראונית6,10,16,17,18,19.
כאן, אנו מספקים פרוטוקול משולב לאיסוף וניתוח נתוני מיקרוסקופ לוקליזציה של מולקולה אחת בדגש מסוים על מעקב יחיד-מולקולה. המטרה הסופית של הפרוטוקול היא לפתור מצבים מפזרים של חלבונים ציטוסקופים בעלי שם התווית בתוך, במקרה זה, תאים חיידקיים בצורת מוט. העבודה שלנו בונה על פרוטוקול קודם עבור מעקב חד מולקולות, שבו DNA פולימראז, PolI, הוכח להתקיים ב-DNA מאוגד ומצב לא מאוגד על ידי ניתוח דיפוזיה20. כאן, אנו מרחיבים ניתוח מעקב חד-מולקולות למדידות תלת-ממדית ומבצעים סימולציות חישוביות ריאליות יותר כדי לפתור ולכמת מספר רב של מצבים מפזרים בו המצויים בתאים. הנתונים נרכשים באמצעות ביתי בנוי 3d ברזולוציה גבוהה מיקרוסקופ פלורסנט אשר מסוגל לקבוע את מיקום 3d של הפולטים פלואורסצנט על ידי הדמיה עם הסליל כפול הפריסה-פונקציה (dhpsf)21,22. התמונות raw-מולקולה בודדת מעובדות באמצעות תוכנה כתובה מותאמת אישית כדי לחלץ את 3D מולקולה חד-ממדית לוקליזציה, אשר משולבים אז לתוך המולקולה יחיד מסלולים. אלפי מסלולים ממאגר להפקת הפצות של מקדמי דיפוזיה לכאורה. בשלב האחרון, ההפצות הנסיוניות מתאימות להפצות שהופקו באמצעות מספרים שהתקבלו באמצעות סימולציות מונטה-קרלו של תנועה בראונית בכרך מוגבל. אנו מיישמים פרוטוקול זה כדי לפתור את המצבים המפזרים של הפרשה סוג 3 חלבון מערכת YscQ בחיים Yersinia בידור. בשל אופיו המודולרי, הפרוטוקול שלנו מתאים בדרך כלל לכל סוג של ניסוי מעקב יחיד או חלקיק בודד, באמצעות מערכות שרירותיות המתאימות לתאים.
Protocol
1. כיול כפולה של נקודת-הכפולה
הערה: התמונות המתוארות בסעיף זה והסעיפים הבאים נרכשים באמצעות מיקרוסקופ הפוך מותאם אישית מובנה, כפי שמתואר ברוצ'ה ואח '23. אותו הליך חל על יישומי מיקרוסקופ שונים המיועדים ללוקליזציה של מולקולה אחת ולמעקב אחר מיקרוסקופ2,3,4. כל תוכנה לרכישת תמונות ועיבוד נתונים המתוארים במאמר זה זמינה (https://github.com/GahlmannLab2014/Single-Molecule-Tracking-Analysis.git).
- הכנת רפידות agarose עבור דגימות הרכבה על כיסוי זכוכית שוברי לצפות מתחת למיקרוסקופ.
- להוסיף 1.5-2% על ידי משקל נקודת התכה נמוכה agarose ל 5 מ מ של M2G מאגר (4.9 mM Na2hpo4, 3.1 mM KH2PO4, 7.5 Mm NH4קלרנית, 0.5 Mm mgso4, 10 μm feso4, 0.5 mm cacl2 ו 0.2% גלוקוז). מיקרוגל למשך כמה שניות עד התפרקה כל. אל תתנו לפתרון לרתוח.
- תנו לפתרון האגקם להתקרר לכמה דקות (2-3 דקות).
- פיפטה 600 μL של הפתרון על גבי מכסה זכוכית (22 מ"מ x 22 מ"מ). הנח בעדינות על מכסה הזכוכית השניה על גבי הצמח. זה יוצר משטח ג'ל דק (~ 0.5 מ"מ) שעלה בין שתי פתקי כיסוי זכוכית.
- תנו לתחבושות האגריות לשבת. ולחזק במשך ~ 20 דקות
- הפרד בעדינות את פתקי כיסוי הזכוכית. . כרית הצמח תיצמד לאחד מהם
- באמצעות להב גילוח, לחתוך את הלוח האגקם לתוך ארבעה חלקים רבועים בגודל שווה. ניתן להשתמש בכל מקטע מרובע עבור דגימה אחת.
- פיפטה 1.5 μL של הפתרון חרוז פלורסנט על כרית אחת agarose. הפתרון חרוז הוא 1/100000 דילול של פתרון המניה ב M2G (ראה טבלת חומרים).
- היפוך כרית agarose והמקום על שובר כיסוי זכוכית כי ניקה בניקוי האוזון עבור 30 דקות. זמן ניקוי נבחר לחסל כל מולקולות פלורסנט מחסיד הכיסויים.
- הר את מכסה את הזכוכית לדוגמה על מחזיק המדגם של מיקרוסקופ פלואורסצנטית הפוכה ולאבטח אותו במקום באמצעות דבק סלוטייפ או קפיץ טעון לדוגמה קליפים.
- הוסיפו טיפת שמן טבילה אל מטרת המיקרוסקופ.
- הנח את מחזיק הדגימה על מיקרוסקופ ואבטח אותו במקומו.
- אתחל את ממשק המשתמש הגרפי (GUI) כדי לשלוט על המצלמה של המיקרוסקופ, לדוגמה שלב, ולייזרים עירור. כאן, תוכנה כתובה מותאמת אישית ב-MATLAB משמשת לבקרת מכשירים (איור משלים 1).
- אתחל את תוכנת המצלמה HCImage לחיות. בכרטיסיה לכידה תחת המקטע פקד מצלמה , קבע את זמן החשיפה ל-0.03 s. לחץ על Live כדי להתחיל בשידור חי מהמצלמה.
- הפעל את הלייזר על-ידי לחיצה על פתח לייזר nm 514 על ממשק GUI כדי להלהיב את חרוזי פלורסנט על כרית ולהציג את פליטת הניאון על המצלמה באמצעות חי-זרם מצב (כלומר, אין נתונים שנשמרו בדיסק).
- כוונן את מיקומי X ו-Y של שלב המיקרוסקופ על-ידי לחיצה על החצים ' XY-Pos ' תחת ' שלב מיקרו-מיצוב ' של GUI כדי למקם לפחות חרוז פלורסנט אחד במרכז שדה התצוגה (FOV). גודל השלב ניתן לשינוי על-ידי לחיצה על התיבה הנפתחת מתחת לחיצים.
- כוונן את מיקום Z של שלב המיקרוסקופ על-ידי לחיצה על חיצי z-Pos תחת המקטע מיצוב ננו של GUI. הגדר את הכיוון של הסליל הכפול של נקודת הכפולה (DHPSF) של חרוז פלורסנט להיות אנכי. אוריינטציה אנכית זו מוגדרת כנקודת ההתחלה בכיול Z. גודל השלב ניתן לשינוי על-ידי לחיצה על התיבה הנפתחת מתחת לחיצים.
- ב HCImage לחיות, תחת לכידת | ההדק מצבים, מהירות ורישום, לשנות את מצב ההדק מבפנים אל ההדק ברמה החיצונית. זה יאפשר MATLAB GUI כדי לשלוט על המצלמה.
- תחת רצף | סריקה הגדרות, לשנות את מספר ספירות המסגרת ל 1200. בחר תיקיית היעד שמור על ידי לחיצה על הכפתור המסומנת ... לבסוף לחץ על התחל. מספר ספירות המסגרות מוגדר ל- 1200 כך שניתן יהיה לאסוף 10 מסגרות עבור כל אחד משלבי המיקום של 120 Z.
- סריקה באמצעות מגוון של מיקומי Z (30 שלבים מעל ומתחת לתנוחת Z החל ב-50 בהפרשים קבועים של ננומטר) ולהקליט 10 מסגרות בכל שלב באמצעות זמן חשיפה של 0.03 s. הפעל את התהליך האוטומטי על-ידי לחיצה על עבור תחת המקטע כיול Z של GUI.
הערה: הפרמטרים עבור הכיול, כולל אורך שלב, מספר שלבים, זמן חשיפה למצלמה ומספר מסגרות בכל שלב ניתן לכוונן גם כאן. כ 105 פוטונים ניתן לרכוש מתוך מסגרת פלורסנט במסגרת אחת באמצעות 0.03 s זמני חשיפה וכתוצאה מכך x, y, z לוקליזציה בערך של 1 ננומטר. - כבה את התאורה לייזר על ידי לחיצה על סגור 515 ננומטר לייזר ב GUI. ב HCImage לחיות, תחת לכידת | ההדק מצבי, מהירות ורישום, לשנות את מצב ההדק חזרה לפנים. תחת רצף | הגדרות סריקה משנות את מספר ספירות המסגרת ל-200. בחר תיקיית היעד שמור על ידי לחיצה על הכפתור המסומנת ... לחץ על התחל כדי לאסוף 200 מסגרות של תמונות כהות עם זמן חשיפה של 0.03 s.
הערה: גם בהיעדר אור הנופל על הגלאי, כל פיקסל יקרא מספר חיובי (הנקרא ערך היסט כהה), העשוי להשתנות מעט בין הפיקסלים. ערך ההיסט הכהה עשוי להשתנות עם הזמן. לכן, יש צורך לאסוף מסגרות כהות עבור כל כיול. - התאם את ה-DHPSF במודל כפול-גאוס באמצעות תוכנת Easy-DHPSF24 כדי להשיג את מיקומי X ו- Y של החרוז, כמו גם עקומת כיול זווית לעומת Z.
- אתחל את התוכנה Easy-DHPSF ב MATLAB. תחת התקנה, הגדר ערוץ ל- G והגדר שיטת התאמה ל- mle עם מודל DG. G מתייחס למצלמה ערוץ ירוק, כמו חלבון פלורסנט המשמש לרכישת נתונים פולט עם אורכי גל ירוק. Mle עם מודל DG מתייחס להערכת סבירות מרבית עם דגם כפול-גאוס.
הערה: גודל הפיקסל ורווח ההמרה תלויים בכיוונון האופטי הספציפי, וייתכן שיהיה צורך לשנותם. - תחת המקטע כיול DHPSF , לחץ על הפעלה. לחץ על אישור בחלון המוקפץ הבא כדי לשמור על הגדרות ברירת המחדל.
- בחר את מחסנית התמונה שנשמרה בשלבים 1.13-1.14. לאחר מכן, בחר את מחסנית התמונה עם הרקע הכהה שנשמר בשלב 1.15. לבסוף, בחר את קובץ ה-. txt שנשמר באופן אוטומטי במהלך שלב 1.14. קובץ זה מכיל את מיקום Z של השלב לאורך תהליך הסריקה.
- בחלון המוקפץ הבא, אם לא נעשה שימוש בשבב המצלמה המלא עבור שדה התצוגה, הקלט את המיקומים המתחילים x0 ו-y0 בשבב. אחרת, קלט x0 = 1 ו-y0 = 1. מידע זה ניתן למצוא ב HCImage Live תחת המקטע Binning ו-SubArrary .
- בחלון הבא, לשנות את גודל ולמקם מחדש את התיבה המופיעה מעל התמונה של חרוזי פלורסנט כך שהוא כ 100 x 100 פיקסלים בגודל וממורכז על אות פלורסנט אחד DHPSF. לאחר מכן, לחץ פעמיים כדי להמשיך.
הערה: DHPSF נבחר צריך להיות מבודד מאותות DHPSF אחרים ובאופן אידיאלי להיות חרוז המבריק ביותר האפשרי. - לחץ במרכז האות DHPSF, בין שתי האונות, ואז להכות Enter. החלון הבא מציג תצוגה מוגדלת של ה-DHPSF הנבחר. בדיוק לבחור את המיקום של מרכז DHPSF על ידי לחיצה.
הערה: התוכנית תתאים לאחר מכן ל-DHPSF, ותציג את התמונה הגולמית ואת השחזור מההתאמה. זה יהיה גם פלט תמונות תבנית מכיול Z המתאים לחתך DHPSF במיקומים שונים Z. אלה ישמשו מאוחר יותר להתאמת הנתונים הניסיוניים. התוכנית פלט את המיקום X, Y ו-Z המשוער בכל מסגרת. במערכת אופטית מיושרת היטב, X ו-Y אמורים לשנות מעט מאוד (~ 30 סטיית nm) כשינויי מיקום Z. אם וריאציה הפלט גדול יותר 30 ננומטר, מסכת הפאזה, הממוקם במישור פורייה של מערכת ההדמיה (איור 1), צריך להיות ינה ושלבים 1.9-1.16 חוזרות ונשנות. - שמור את הGUI Easy-DHPSF על ידי לחיצה על סמל שמור בפינה השמאלית העליונה של GUI. זה יכול להיות נטען מאוחר יותר על ידי לחיצה על סמל עומס ב GUI.
הערה: הליך כיול Z צריך להתבצע בכל יום של ניסוי לחשבון עבור שינויי יישור במיקרוסקופ שייתכן שהתרחשו עקב תנודות בטמפרטורה או תנודות מכניות.
- אתחל את התוכנה Easy-DHPSF ב MATLAB. תחת התקנה, הגדר ערוץ ל- G והגדר שיטת התאמה ל- mle עם מודל DG. G מתייחס למצלמה ערוץ ירוק, כמו חלבון פלורסנט המשמש לרכישת נתונים פולט עם אורכי גל ירוק. Mle עם מודל DG מתייחס להערכת סבירות מרבית עם דגם כפול-גאוס.
2. הכנת התרבות החיידקית
- הכן מדיה תרבותית התומכת בצמיחת תאים חיידקיים. עבור Y. בידור, השתמש 5 מ ל של Bhi (אינפוזיה לב המוח) מרק המכיל nalidixic חומצה (35 μg/ml) ו-2, 6-diaminopimelic חומצה (80 Μg/ml). כאן, זן Y. enterocolitica משמש כי יש חלבון YscQ מתויג עם חלבון פלורסנט eyfp23.
- איחסן מדיה עם תרבויות חיידקית ממניות המקפיא או מתרביות הצלחות.
- לגדל את התרבות ב -28 ° c עם טלטול בן לילה.
- לדלל כמות קטנה (~ 250 μL) של תרבות לילה רווי ל-5 מ ל באמצעות מדיית תרבות טרייה.
- לגדל את התרבות ב 28 ° צ' עם טלטול עבור 60-90 דקות.
- לגרום לביטוי של חלבון היתוך פלורסנט. עבור Y. בידור, החום לחשמל את התאים 37 מעלות צלזיוס בשייקר מים כדי לגרום ל- yop רגלון.
- מודדים את התאים בתוספת של 3 h ב 37 ° צ' עם טלטול.
- צנטריפוגה 1 מ ל של תרבות ב 5,000 x g עבור 3 דקות בטמפרטורת החדר. . מחק את הסופרנטאנט
- שטוף את הגלולה 3 פעמים עם 1 מ ל של M2G מדיה.
- מחדש להשעות את החיידקים הפלטאד ב ~ 250 μL של M2G Media.
- הוסף חרוזי פלורסנט כfiducial סמנים. הפתרון חרוז פלורסנט יש להוסיף כמויות מדולל כראוי, כך יש רק 1-2 חרוזים לכל FOV כאשר מוצג במיקרוסקופ.
- בעדינות פיפטה או מערבולת את ההשעיה כדי להפריד תאים צבורים.
- צלחת 1.5 μL של השעיה על כרית 1.5-2% agarose שנעשו עם M2G.
- להפוך את כרית agarose ולמקם אותו על שובר מיקרוסקופ ניקה האוזון. שובר הכיסוי צריך להיות ממוקם בניקוי האוזון במשך 30 דקות כדי להפחית את כל הרקע הפנימי של הזריחה.
3. רכישת נתונים
- הר את מכסה את הזכוכית לדוגמה על מחזיק המדגם של מיקרוסקופ פלואורסצנטית הפוכה ולאבטח אותו במקום באמצעות דבק סלוטייפ או קפיץ טעון לדוגמה קליפים.
- להוסיף טיפה של שמן טבילה על מטרת המיקרוסקופ, ואז למקם את המחזיק לדוגמה על מיקרוסקופ ולאבטח אותו במקום.
- אתחל ממשק משתמש גרפי (GUI) כדי לשלוט על המצלמה של המיקרוסקופ, לדוגמה שלב, ולייזרים עירור. כאן, תוכנה כתובה מותאמת אישית ב-MATLAB משמשת לבקרת מכשירים.
- אתחל את תוכנת המצלמה HCImage לחיות. בכרטיסיה לכידה תחת המקטע פקד מצלמה , קבע את זמן החשיפה ל-0.025 s. לחץ על Live כדי להתחיל בשידור חי מהמצלמה.
- להתאים את מיקומי X ו-Y של שלב המיקרוסקופ על ידי לחיצה על החצים XY-Pos תחת המקטע מיקרו מיצוב שלב של GUI לסרוק סביב המדגם למצוא fov עם אוכלוסיה צפופה כראוי של תאים חיידקיים.
הערה: כדי למקסם את תפוקת הנתונים, על התאים להיות דחוסים ככל האפשר, ללא תאים חופפים או נוגעים למגע. FOV צריך גם לכלול לפחות 1 חרוז פלורסנט לשמש סמן fiducial, רצוי ממוקם בפינה של FOV. - כוונן את מיקום Z של שלב המיקרוסקופ על-ידי לחיצה על חיצי z-Pos תחת המקטע מיצוב ננו של GUI, כך האונות dhpsf של חרוז הפלורסנט הם אנכיים.
- תחת רצף | סריקה הגדרות, לשנות את מספר ספירות המסגרת ל 20,000. בחר תיקיית היעד שמור על ידי לחיצה על הכפתור המסומנת ... לבסוף לחץ להתחיל לאסוף עד 20,000 מסגרות מצלמה באמצעות זמן חשיפה קצר של 0.025 s. eyfp צילום מהבהב הוא יזם באמצעות אור עירור גבוה בעוצמה ב 514 ננומטר19,25.
הערה: כאן, עוצמת לייזר של ~ 350 W/cm2 במישור המוקד משמש הלבנה הראשונית הדמיה הבאים של מולקולות eyfp יחיד. הפעלת פוטומולקולות eYFP באורכי גל UV במהלך ההדמיה לא נעשה שימוש. צריך להיות ברוב אות אחד מולקולה אחת לכל תא חיידקי. אם הצפיפות של אות מולקולה בודדת גבוהה מדי בתחילה, המשך להאיר עד שתתרחש הלבנה מספקת לפני התחלת רכישת הנתונים. - כבה את התאורה לייזר על ידי לחיצה על סגור 515 ננומטר לייזר ב GUI. לאסוף 200 מסגרות של תמונות כהות באמצעות זמן חשיפה זהה.
- ב GUI, לבדוק את התיבה ליד LED ת'ורמעבדות לחץ על החלף מראה למעלה. זה יהיה להחליף את השביל מנתיב הזריחה למסלול ניגודיות שלב.
- אתחל את תוכנת רכישת הנתונים IC לכידת 2.4. זה שולט במצלמה במסלול הניגוד שלב. לחץ על לחצן התחל/הפסק בתצוגת live כדי להציג שידור חי מהמצלמה. לחץ על לכידת | שמור תמונה כדי לאסוף תמונת חדות פאזה של התאים בשדה התצוגה.
- חזור על שלבים 3.5-3.10 עבור FOVs נוספים. כאן, נתונים שנרכשו מ-~ 500 תאים חיידקיים בעשרה FOVs שונים משמש כדי להגדיל את מספר מסלולים חד מולקולה זמין לניתוח.
התראה: תאים הנטענים על גבי הרפידות האגריות לפרקי זמן ארוכים עשויים להתנהג בצורה שונה מאשר תאים שנטענו באופן טרי. בנוסף, המשטח העולה עלול לאבד את שלמות לאחר זמן מה, דבר שעלול להשפיע לרעה על איכות הנתונים. בדרך כלל, לכל היותר 3 FOV (~ 30 דקות על המיקרוסקופ) משמשים לכל שקופית לדוגמה.
4. עיבוד נתונים
הערה: גירסה ששונתה של תוכנת Easy-DHPSF24 משמש MATLAB לניתוח של מסגרות המצלמה raw לחלץ לוקליזציה מולקולה אחת. Easy-DHPSF משמש במיוחד כדי להתאים לוקליזציה DHPSF בדימות יחיד מולקולה. בוצעו שינויים מותאמים אישית כדי ליישם שגרת התאמה מרבית (MLE) המבוססת על שגרה המתאימה למאפייני רעש תלויי פיקסל של מצלמות sCMOS המודרנית26. הוא שונה גם כדי לקבל את סוג קובץ התמונה פלט מהתוכנית HCImage Live (. dcimg). להסבר מפורט יותר על התוכנה והצעדים הבודדים, אנא ראו את לו ואח '24
- אתחל את הMATLAB קל-DHPSF ב (איור משלים 2). טען בקובץ שנשמר בשלב 1.16.8.
- קביעת ערכי הסף עבור כל אחד מתוך 7 תבניות פלט בשלב 1.16.7
- תחת מקטע הזיהוי של כיול SM , לחץ על הפעלה. לחץ על אישור בשני החלונות המוקפצים הבאים כדי לשמור על הגדרות ברירת המחדל.
- פתח את אוסף התמונות המכיל את הנתונים מ-FOV הראשון כשתתבקש.
- בחר מגוון קטן של מסגרות כדי להתאים לתבניות. בדרך כלל מסגרות 1001-2000 משמשות כדי למנוע אותות חופפים צפוף במספר מאות המסגרות הראשונות. לחץ על אישור בחלון המוקפץ הבא כדי לשמור על הגדרות ברירת המחדל. לחץ על ביטול כאשר תתבקש לבצע את קובץ יומן הרישום בחלון הבא.
- פתח את מחסנית התמונה עם הרקע הכהה שנשמר בשלב 3.8. לחץ על 'אישור' בחלון המוקפץ הבא כדי להשאיר את הפרמטרים עבור שערוך הרקע המוגדר כברירת מחדל. ברירת המחדל היא להעריך את הרקע באמצעות מסנן חציון27 כיסוי 100 מסגרות המצלמה הבאות סביב המסגרת הנוכחית.
- בחלון הבא, שנה את גודל התיבה הנמצאת בתמונה כדי לכסות את ה-FOV המלא, ולאחר מכן לחץ פעמיים כדי להמשיך.
- הגדר את אזור הריבית על-ידי לחיצה על מספר נקודות בתמונה כדי ליצור מצולע. אזור העניין צריך לכלול את שטח התצוגה ככל האפשר, תוך הקפדה על כל חרוזי פלורסנט (עצמים בהירים מאוד) בתמונה לא לשכב בתוך המצולע, ולאחר מכן ללחוץ פעמיים כדי להמשיך.
הערה: התוכנה תנסה להתאים את התבניות לתמונה, ותציג תמונה עם התאמות אפשריות בעיגול. - כשהתוכנה עצרה, היא תשמור תמונות רבות של התאמות תבנית שנמצאו ותציג את ערך הסף המתאים בתיקיה שהוגדרה מראש. ערך סף גבוה יותר תואם להתאמה טובה יותר. עבור כל מספר תבנית, בדוק את הדוגמה המתאימה וקבע את הסף הנמוך ביותר המוצגים בתמונה של DHPSF. קלט את ספי הנתונים עבור כל אחת מ -7 התבניות תחת המקטע כיול ה-SM ב-EASY-DHPSF GUI.
הערה: התוכנית תנסה לבחור באופן אוטומטי ספי קלט, עם זאת, אלה הם בדרך כלל לא אמין ויש לבדוק באופן ידני. מספר הסף נבחר כך שמספר האותות האמיתיים של המולקולה החד מחמיץ, אך מספרם של המועמדים החיוביים השקרית לצורך התאמה מבחינה מבצעית ניתן לניהול. - שמור את הGUI Easy-DHPSF שוב על ידי לחיצה על סמל שמור בפינה השמאלית העליונה.
- מתאים את חרוז פלורסנט ב FOV להשתמש כמו סמן fiducial
- תחת המקטע Track fiduciaries של הממשק הקל-DHPSF, לחץ על הפעל. לחץ על אישור בשני החלונות המוקפצים הבאים כדי לשמור על הגדרות ברירת המחדל.
- גררו את התיבה המצופים בתמונה ומרכוז אותה מעל לאות DHPSF מחרוז הפלורסנט, ולאחר מכן לחץ פעמיים.
- לחץ במרכז האות DHPSF, בנקודת האמצע בין שתי האונות, ואז להכות להיכנס. לחץ על ביטול כאשר תתבקש לבצע את קובץ יומן הרישום בחלון הבא.
הערה: התוכנה תתאים ל-DHPSF בכל מסגרות המצלמה, ויציגו את התמונה הגולמית ואת התמונה המשוחזרת. כאשר התוכנה סיימה, זה יהיה פלט דמויות עם X, Y, ו Z מיקומים של מפרק פלורסנט על משך הרכישה של התמונה. - בדוק את התיבה ליד להשתמש fiduciaries ולשמור את הGUI EASY-DHPSF שוב על ידי לחיצה על סמל שמור בפינה השמאלית העליונה.
- חפש והתאם את כל הלוהתאמות בכל מסגרות המצלמה באמצעות ספי התבנית שהתקבלו בשלב 4.2.
- תחת מקטע SMs לוקליזציה המקטע של EASY-DHPSF GUI, לחץ על הפעל. לחץ על אישור בחלונות המוקפצים הבאים כדי לשמור על הגדרות ברירת המחדל. לחץ על ביטול כאשר תתבקש לבצע את קובץ יומן הרישום בחלון הבא.
הערה: התוכנה תמצא ותתאים ל-DHPSF במודל כפול-גאוס אם איכות ההתאמה מעל לסף המוגדר על-ידי המשתמש. זה יציג את התמונה raw עם עיגולים סביב גפרורים התבנית, כמו גם התמונה של DHPSF שוחזר מתאים. - שמור את הGUI Easy-DHPSF שוב על ידי לחיצה על סמל שמור בפינה השמאלית העליונה.
- תחת מקטע SMs לוקליזציה המקטע של EASY-DHPSF GUI, לחץ על הפעל. לחץ על אישור בחלונות המוקפצים הבאים כדי לשמור על הגדרות ברירת המחדל. לחץ על ביטול כאשר תתבקש לבצע את קובץ יומן הרישום בחלון הבא.
- הצגת לוקליזציה של מולקולה בודדת וסינון מגרסאות לא רצויות או לא מהימנות
- במקטע הלוקליזציה של הפלט של DHPSF , לחץ על מסנן פלט.
- לחץ על אישור בשלושת החלונות הבאים כדי לבצע אינטרפולציה של המיקומים fiducial X, Y ו-Z לאורך זמן. ברוב המקרים, אפשרויות ברירת המחדל הן מספיקות. אם קו האינטרפולציה השחור אינו משקף אינטרפולציה סבירה של קו המיקום האדום, שנה את פרמטרי האינטרפולציה בחלון המוקפץ.
הערה: קו האינטרפולציה משמש להסחף הבמה ומתקן את הלוקליזציה של המולקולה היחידה. - פתח את תמונת הניגודיות של הפאזה המתאימה לניתוח FOV כשתתבקש. לחץ על אישור בשני החלונות המוקפצים הבאים כדי לשמור על הגדרות ברירת המחדל.
- בשני החלונות המוקפצים הבאים, שנה את ערכי המסנן כדי לאפשר דרישות לוקליזציה מחמירות יותר או מקילות יותר, ולאחר מכן לחץ על אישור.
- בחלון שמופיע, גרור או שנה את גודל התיבה המוצגת בתמונות כדי להציג את אזור הריבית הרצוי ולחץ פעמיים כדי להמשיך.
- מוצג שחזור תלת-ממדי של לוקליזציה של מולקולה אחת. השתמש בכלי איור כדי לטפל בשחזור (לסובב, זום, וכו '). לחץ על ' המשך ' כדי להציג תיבת דו-שיח אחרת ששואלת האם ברצונךלהוסיף מחדש עם ערכת פרמטרים שונה?'. אם התוצאות משביעות רצון, לחץ על לא. אם לא, לחץ על כן.
הערה: סיבות נפוצות לתוצאות משביעות רצון כוללות את תמונת הניגודיות של הפאזה שאינה מצופה כראוי עם נתוני הלוקליזציה או שספי התבנית הראשוניים היו נמוכים מדי ביצירת לוקליזציה חיוביים שקריים רבים. אם האפשרות Yes נבחרה, התוכנה תחזור לשלב 4.5.4 כך שניתן יהיה להגדיר ערכי פרמטרים חדשים. אם לא נבחר, התוצאות יישמרו.
5. עיבוד לאחר נתונים
- באמצעות תוכנה שנכתבה בהתאמה אישית ב-MATLAB, חתוך את תמונת הניגודיות של הפאזה כך שרק האזור המכיל תאים שנוצרו באמצעות תמונות מתחת למיקרוסקופ הזריחה. שלב זה הכרחי מכיוון שתמונת הניגודיות של השלב מכסה שטח הרבה יותר גדול מהתמונה הפלואורסצנטית. חיתוך התמונה מפשט את השלב הבא.
- פלח תאים בודדים על-ידי עיבוד תמונת הניגודיות של השלב עם התוכנה OUFTI28 (איור משלים 3)
- אתחל OUFTI ב-MATLAB. טען את תמונת הניגודיות של השלב החתוך מהשלב הקודם על-ידי לחיצה על שלב הטעינה.
- לחץ על קובץ כדי לבחור ולקרוא למיקום שמירה עבור קובץ הפלט.
- בחר מסגרות עצמאיות תחת הכותרת זיהוי וניתוח .
- לחץ על טען פרמטרים כדי לטעון פרמטרים עבור זיהוי תאים. דוגמאות לפרמטרים כוללות שטח תא קביל, רוחב תא ותא פיצול תא.
הערה: יש לכוונן את כל הפרמטרים הללו כדי למקסם את הביצועים עבור גודלי התא ואיכות התמונה הספציפיים הנמצאים בשימוש. חשוב להניח שפרמטר האלגוריתם צריך להיות מוגדר לפיקסל משנה כדי לאפשר מדידה מדויקת של קווי מתאר של תאים. - לחץ על מסגרת זו כדי להתחיל בפילוח של תאים. קווי מתאר של תאים יופיעו מעל תמונת הניגודיות של השלב עם סיום התהליך.
- שימוש בפקדים תחת המקטע הידני , פיצול תאים, הוספת תאים או עידון קווי מתאר של תאים כדי לקבל קווי מתאר עבור תאים שהיו מפוצלים באופן לא מדויק במהלך התהליך האוטומטי.
- הקלט את קווי המתאר של התא על-ידי לחיצה על שמור ניתוח.
- השתמש בתוכנה הכתובה בצורה מותאמת אישית ב-MATLAB לכיסוי מדויק של קווי המתאר המתקבלים בשלב הקודם באמצעות לוקליזציה של מולקולה בודדת. שלבי המשנה הבאים מפרטים את השלבים של התוכנה.
- בחר באופן ידני 5 זוגות של נקודות בקרה בחלון המוקפץ על-ידי הערכה ולחיצה על המיקום של עמודי התאים של אותם חמישה תאי התאמה לשפות לוקליזציה של המולקולה היחידה וקווי המתאר של התא, שנוצרו בשלב הקודם. המיקום של מוט התא יכול להיות מוערך בערך על ידי ציור נפשי גוף קמור סביב המולקולה יחיד מולקולה השייכת תא אחד ובחירת הנקודה של העקמומיות הגבוהה ביותר (איור משלים 4).
- צור פונקציית טרנספורמציה דו-ממדית באמצעות הפונקציה cp2tform ב-MATLAB והשתמש בה כדי ליצור כיסוי מחוספס של קווי המתאר של התא ולוקליזציה של מולקולה אחת.
- מחק תאים המכילים פחות מ-10 לוקליזציה והסר תאים הממוקמים חלקית מחוץ לשדה התצוגה. מחק באופן ידני תאים לא רצויים נוספים בחלון המוקפץ על-ידי לחיצה על החלק הפנימי של קו המתאר של התא (איור משלים 5).
- השתמש במרכז המסה עבור כל קווי המתאר של התא הנותרים ולוקליזציה של מולקולה בודדת בתוכם כדי ליצור קבוצה גדולה יותר של זוגות של נקודות בקרה, לחשב מחדש את פונקציית ההמרה הדו ולהשתמש בה כדי ליצור כיסוי סופי של קווי המתאר של התא וה לוקליזציה של מולקולה אחת.
- הקצאת לוקליזציה השוכנים בגבולות החלוקה לרמות של תאים לתא זה. התעלם מכל החנויות שאינן ממוקמות בתוך חלוקה לרמות של תא (איור 2a, איור משלים 6).
6. מעקב יחיד-מולקולה
הערה: הסעיף הבא מושלם באמצעות תוכנה מותאמת אישית ב-MATLAB. סעיף זה מתאר את השלבים שהתוכנה מבצעת.
- לאיתור לוקליזציה המוקצים לאותו תא ובמסגרות מצלמה עוקבות, חשב את המרחק האוקלידי בין הלוקליזציה. אם המרחק בין לוקליזציה נמצא מתחת לסף של 2.5 μm, קשר את הלוקליזציה על-ידי הקצאתם לאותו נתיב של מולקולה יחידה.
הערה: חשוב לשקול רק לוקליזציה בתוך תא בודד, כך שלוקליזציה בתאים סמוכים שבמקרה עומדות בדרישות הסף המרחבי והזמני אינן מקושרות. סף 2.5 יקרומטר נבחר כמרחק המקסימלי מולקולה מהירה מאוד (30 יקרומטר2/s) יכול לנוע במשך זמן החשיפה (0.025 s) ועוד 20% מאגר. - התעלם מהמסלולים קצרים מ-4 לוקליזציה. אם שתי לוקליזציה או יותר (כלומר, שניים או יותר פולטים רואה) נמצאים בו בתא, למחוק את המסלולים המשויכים. הגדרת אורך המסלול המינימלי ל-4 לוקליזציה מאפשרת למספר מדידות מרחוק להיות בממוצע מניב הערכה מדויקת יותר של מקדמי הדיפוזיה.
- חישוב ההזחה הממוצע בריבוע (MSD) עבור מסלול נתון על-ידי:
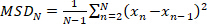 1
1
כאשר N הוא המספר הכולל של לוקליזציה במסלול ו- xN הוא מיקום המולקולה בנקודת זמן N. - חשב את מקדם הדיפוזיה לכאורה, D * על-ידי
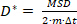 2
2
כאשר m = 2 או 3 הוא הממד של המדידה ו Δt הוא זמן חשיפה למצלמה.
הערה: ניסוי אופייני יפיק ~ 5000-100,000 מסלולים בסך הכל, וכתוצאה מכך התפלגות מקדם דיפוזיה לכאורה עם סעיפים רבים אלה.
7. מונטה קרלו הדמיה של תנועה בראונית בכרך סגור
הערה: צור ספריות של הפצות הדמיה מדומה לכאורה של מקדם דיפוזיה על-ידי ביצוע סימולציות של מונטה קרלו בתנועה בראונית התחומה בנפח גלילי, תוך שימוש ב-64 ערכים בטווח 0.05 – 20 יקרומטר2/s כפרמטרי קלט (תוכנה זמין מהמחברים על פי בקשה). טווח זה נבחר לכסות את הטווח של מקדמי דיפוזיה בעבר מוערך של חלבונים פלורסנט (פיוז'ן) בחיידקים. 64 מקדמי דיפוזיה משמשים לדגימת טווח זה בצורה מספקת. סעיף זה מבוצע באמצעות תוכנות מותאמות אישית ב-MATLAB ומתארת את השלבים שהתוכנה מקבלת באופן אוטומטי. מוט בצורת Y. enterocolitica התאים המשמשים כאן מתקרבת על ידי נפח גלילי של אורך l = 5 יקרומטר וקוטר d = 0.8 יקרומטר.
- הפעלת מסלולים בודדים במיקום אקראי בכרך הגלילי, והדמיית שלבי הפיזור האקראיים (כלומר, בראוניאן) באמצעות מרווח זמן של 100 ns (מרווח זמן צריך להיות קצר בהרבה מזמן חשיפת המצלמה כדי לאפשר מספיק דיגום התנוחה במסגרת מסגרת מצלמה). דגום כל צעד של הזחה עבור קלט D מפונקציית ההתפלגות של גאוס התואמת על-ידי סידור מחדש של אקורן .2 והוסף אותו למיקום הקודם:
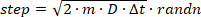 3
3
כאשר הפונקציה randn ב-MATLAB מדגימה מספר אקראי מהתפלגות נורמלית. אם שלב גורם להמולקולה להיות מנעקרו מחוץ לנפח הצילינדר, משקפים את המולקולה בחזרה לתוך הצילינדר בזווית אקראית. - עבור כל מרווח זמן, צור תמונת DHPSF המתאימה למיקום המיידי x, y, z של פולט הסימולציה.
- כדי להתאים את התנאים הניסיוניים, הדמיית תמונות המכילות 1,000 פוטונים לכל לוקליזציה, עם רקע לייזר של 13 פוטונים לפיקסל, ורעש של פואסון. בנוסף, הוסף היסט כהה של ~ 50 פוטונים לפיקסל לקרוא את גאוס (σ ~ 1.5 פוטונים), עקבי עם מדידות כיול ניסיוני מצלמה. בסופו של דבר, להכפיל את התמונה על ידי הרווח הנמדד בפיקסל ממדד של המצלמה sCMOS להשיג את התמונה ביחידות של ספירות גלאי.
הערה: לאחר המניפולציות הללו, היחס בין האות לרעש של התמונה הסופית הוא ~ 2. - ליצור תנועה מטושטשת תמונות המשקפים את המיקום המשתנה של המולקולות על ידי סיכום 50 DHPSF תמונות מדומה בזמן החשיפה בשימוש במהלך רכישת נתונים ניסיוניים. כדי להגביל את ההוצאות החישוביות, רק 50 מיקומים שנדגמו מדי פעם נבחרו כדי ליצור תמונה (במקום כל 250,000 המיקומים שנדגמו במהלך זמן חשיפה של 0.025 במרווח דגימה של 100 ns).
הערה: אורך (מספר המסגרות) של מסלולים מדומים צריך להתאים לאורך הממוצע של מסלולים ניסיוניים. במקרה זה, מספר המסגרות בכל מסלול הוא 6.
- כדי להתאים את התנאים הניסיוניים, הדמיית תמונות המכילות 1,000 פוטונים לכל לוקליזציה, עם רקע לייזר של 13 פוטונים לפיקסל, ורעש של פואסון. בנוסף, הוסף היסט כהה של ~ 50 פוטונים לפיקסל לקרוא את גאוס (σ ~ 1.5 פוטונים), עקבי עם מדידות כיול ניסיוני מצלמה. בסופו של דבר, להכפיל את התמונה על ידי הרווח הנמדד בפיקסל ממדד של המצלמה sCMOS להשיג את התמונה ביחידות של ספירות גלאי.
- צור 5,000 מסלולים עבור כל אחד 64 מקדמי דיפוזיה הקלט מדומה.
- ניתוח מסגרות מצלמה מדומה כמתואר בסעיף עיבוד נתונים .
- קישור לוקליזציה לתוך מסלולים כמתואר בסעיף ' מעקב אחר מולקולה אחת '.
- מבצעת אינטרפולציה על פונקציית ההתפלגות המצטברת (CDF) עבור כל התפלגות מדומה באמצעות אינטרפולציה באמצעות B-spline של הזמנה 25. הפצות שעברו אינטרפולציה נחוצות כדי שניתן יהיה לבצע עליהן שאילתה בנקודות שרירותיות.
- האינטרפולציה המתקבלת עקומות ב-spline לאורך ה- d-ציר (d הוא מקדם דיפוזיה האמיתי ללא מוגבל השולט על התנועה של הפולטים מדומה) תוך שימוש בפונקציה מלאה האינטרפולציה ב MATLAB (לציין את " שיטת האינטרפולציה הטבעית). זה מספק פונקציה דו-ממדית מתמשכת שממנה כל התפלגות מקדם דיפוזיה לכאורה התואמת לערך מקדם דיפוזיה אמיתי בטווח 0.05-20 יקרומטר2/s ניתן לבצע שאילתה.
8. התאמת הפצת מקדם דיפוזיה לכאורה
הערה: התאמה להפצות המסיביות הניכרות של מקדמי דיפוזיה לכאורה באמצעות צירופים ליניאריים של ההפצות המדומים שנוצרו בסעיף הקודם (הדמיית מונטה-קרלו של תנועה בראונית באמצעי אחסון מוגבל). סעיף זה מבוצע באמצעות תוכנות מותאמות אישית ב-MATLAB ומתארת את השלבים שהתוכנה מקבלת באופן אוטומטי. למידע נוסף ודוגמאות לאפליקציה, אנא ראו את רוצ'ה ואח '29
- בצע התאמה מוגבלת של ריבועים ליניאריים לפחות (באמצעות הפונקציה lsqlin ב-MATLAB) של cdf הנסיוני באמצעות מערך שנדגם מדי פעם של cdf מדומה מהספריה שנוצרה בסעיף 7. הפלט של שלב זה הוא וקטור פרמטר המכיל את מקדמי הדיפוזיה ושברי האוכלוסיה של מצבים מפזרים נפוצים בהתפלגות הניסיונית.
- שילוב מצבים מפזרים עם ערכי מקדם דיפוזיה בתוך 20% אחד מהשני למצב פיזור יחיד על-ידי חישוב משקל בהתאם לשבר האוכלוסיה היחסי. זהו וקטור הפרמטר ההתחלתי.
הערה: כדי להפחית את מורכבות המודלים, המצבים הדיפוזיה עם מקדמי הדיפוזיה מתחת ל-0.5 יקרומטר2/s יכולים להיות קבועים במהלך כל השלבים הבאים. - צור מערכים של וקטורים של פרמטרים המתאימים לניסיון עם מספר שונה של מצבים מפזרים, החל ממצב משתנה יחיד למספר מרבי של מצבים המוגדרים על-ידי המשתמש.
- באמצעות וקטור הפרמטר ההתחלתי, שלב מצבים מפזרים סמוכים באמצעות חישוב בממוצע ופיצול של מצבים מפוצלים לשתי מדינות עם שברי אוכלוסיה שווים ומקדמי דיפוזיה של 20% מעל ומתחת לערך המקורי. חזור על כל שילוב המצב ופיצול אפשרויות.
- השתמש בכל וקטור פרמטרים של התאמת ניסיון כדי לאתחל התאמה מרובעת לא ליניארית של 5 ערכות מידע נפרדות של הנתונים (באמצעות הפונקציה fmincon ב-MATLAB). קבע את איכות ההתאמה על-ידי מציאת סכום הריבועים השיורי בין ההתאמה להתפלגות המתאימה לערכות המשנה הנותרות (אימות הנתונים החוצה).
- השתמש בסכום השיורי הממוצע של ריבועים של 5 האביזרים הנפרדים עבור כל וקטור הניסוי כאיכות ההתאמה הכוללת, לקבוע את וקטור המשפט עם האיכות הטובה ביותר של התאמה לכל מספר של מצבים מפזרים.
- קבע את מספר המצבים האופטימלי על-ידי זיהוי וקטור הניסיון שהוספת מצב נוסף אינו מהווה לפחות שיפור של 5% באיכות ההתאמה.
- השתמש בווקטור ניסיון זה כדי לאתחל את התאמת הריבועים הבלתי-ליניאריים לפחות של ערכת הנתונים המלאה.
- הערכת שגיאה עבור הפרמטרים הבודדים על-ידי דגימה מחדש של הנתונים על-ידי אתחול והתאמה מחודשת של אותו וקטור ניסוי.
תוצאות
תחת התנאים הניסיוניים המתוארים כאן (20,000 מסגרות, אורך מסלול מינימום של 4 לוקליזציה) ובהתאם לרמות הביטוי של חלבונים היתוך בעלי תוויות פיוז'ן, כ 200-3000 לוקליזציה מניב 10-150 ניתן ליצור מסלולים לכל תא (איור 2a, b). יש צורך במספר רב של מסלולים כדי ליצור התפלגות...
Discussion
גורם קריטי ליישום המוצלח של הפרוטוקול המוצג הוא לוודא שאותות מולקולות בודדות מופרדים היטב זה מזה (כלומר, הם צריכים להיות דלילים בחלל ובזמן (Mov משלימים 1)). אם יש יותר ממולקולה רואה אחת בתא באותו זמן, לוקליזציה ניתן להקצות באופן שגוי מסלול אחר של מולקולות. הדבר נקרא בעיית הקישור
Disclosures
. למחברים אין מה לגלות
Acknowledgements
אנו מודים אלישה אחמוביץ ' וטינג יאן על הקריאה הקריטית של כתב היד. אנו מודים לאד הול, מדען סגל בכיר בקבוצת שירותי המחשוב המתקדם באוניברסיטת וירג, לקבלת עזרה בהקמת רוטינות האופטימיזציה המשמשות בעבודה זו. המימון לעבודה זו סופק על ידי אוניברסיטת וירג.
Materials
| Name | Company | Catalog Number | Comments |
| 2,6-diaminopimelic acid | Chem Impex International | 5411 | Necessary for growth of Y. enterocolitica cells used. |
| 4f lenses | Thorlabs | AC508-080-A | f = 80mm, 2" |
| 514 nm laser | Coherent | Genesis MX514 MTM | Use for fluorescence excitation |
| agarose | Inivtrogen | 16520100 | Used to make gel pads to mount liquid bacterial sample on microscope. |
| ammonium chloride | Sigma Aldrich | A9434 | M2G ingredient. |
| bandpass filter | Chroma | ET510/bp | Excitation pathway. |
| Brain Heart Infusion | Sigma Aldrich | 53286 | Growth media for Y. enterocolitica. |
| calcium chloride | Sigma Aldrich | 223506 | M2G ingredient. |
| camera | Imaging Source | DMK 23UP031 | Camera for phase contrast imaging. |
| dielectric phase mask | Double Helix, LLC | N/A | Produces DHPSF signal. |
| disodium phosphate | Sigma Aldrich | 795410 | M2G ingredient. |
| ethylenediaminetetraacetic acid | Fisher Scientific | S311-100 | Chelates Ca2+. Induces secretion in the T3SS. |
| flip mirror | Newport | 8892-K | Allows for switching between fluorescence and phase contrast pathways. |
| fluospheres | Invitrogen | F8792 | Fluorescent beads. 540/560 exication and emission wavelengths. 40 nm diameter. |
| glass cover slip | VWR | 16004-302 | #1.5, 22mmx22mm |
| glucose | Chem Impex International | 811 | M2G ingredient. |
| immersion oil | Olympus | Z-81025 | Placed on objective lens. |
| iron(II) sulfate | Sigma Aldrich | F0518 | M2G ingredient. |
| long pass filter | Semrock | LP02-514RU-25 | Emission pathway. |
| magnesium sulfate | Fisher Scientific | S25414A | M2G ingredient. |
| microscope platform | Mad City Labs | custom | Platform for inverted microscope. |
| nalidixic acid | Sigma Aldrich | N4382 | Y. enterocolitica cells used are resistant to nalidixic acid. |
| objective lens | Olympus | 1-U2B991 | 60X, 1.4 NA |
| Ozone cleaner | Novascan | PSD-UV4 | Used to eliminate background fluorescence on glass cover slips. |
| potassium phosphate | Sigma Aldrich | 795488 | M2G ingredient. |
| Red LED | Thorlabs | M625L3 | Illuminates sample for phase contrast imaging. 625nm. |
| sCMOS camera | Hamamatsu | ORCA-Flash 4.0 V2 | Camera for fluorescence imaging. |
| short pass filter | Chroma | ET700SP-2P8 | Emission pathway. |
| Tube lens | Thorlabs | AC508-180-A | f=180 mm, 2" |
| Yersinia enterocolitica dHOPEMTasd | N/A | N/A | Strain AD4442, eYFP-YscQ |
| zero-order quarter-wave plate | Thorlabs | WPQ05M-514 | Excitation pathway. |
References
- Kapanidis, A. N., Uphoff, S., Stracy, M. Understanding Protein Mobility in Bacteria by Tracking Single Molecules. Journal of Molecular Biology. , (2018).
- Betzig, E., et al. Imaging intracellular fluorescent proteins at nanometer resolution. Science. 313 (5793), 1642-1645 (2006).
- Hess, S. T., Girirajan, T. P. K., Mason, M. D. Ultra-high resolution imaging by fluorescence photoactivation localization microscopy. Biophysical Journal. 91 (11), 4258-4272 (2006).
- Rust, M. J., Bates, M., Zhuang, X. W. Sub-diffraction-limit imaging by stochastic optical reconstruction microscopy (STORM). Nature Methods. 3 (10), 793-795 (2006).
- Chen, T. Y., et al. Quantifying Multistate Cytoplasmic Molecular Diffusion in Bacterial Cells via Inverse Transform of Confined Displacement Distribution. Journal of Physical Chemistry B. 119 (45), 14451-14459 (2015).
- Mohapatra, S., Choi, H., Ge, X., Sanyal, S., Weisshaar, J. C. Spatial Distribution and Ribosome-Binding Dynamics of EF-P in Live Escherichia coli. mBio. 8 (3), (2017).
- Stracy, M., et al. Single-molecule imaging of UvrA and UvrB recruitment to DNA lesions in living Escherichia coli. Nature Communications. 7, 12568 (2016).
- Persson, F., Lindén, M., Unoson, C., Elf, J. Extracting intracellular diffusive states and transition rates from single-molecule tracking data. Nature Methods. 10 (3), 265-269 (2013).
- Bakshi, S., Choi, H., Weisshaar, J. C. The spatial biology of transcription and translation in rapidly growing Escherichia coli. Frontiers in Microbiology. 6, 636 (2015).
- Mustafi, M., Weisshaar, J. C. Simultaneous Binding of Multiple EF-Tu Copies to Translating Ribosomes in Live Escherichia coli. mBio. 9 (1), (2018).
- Michalet, X., Berglund, A. J. Optimal diffusion coefficient estimation in single-particle tracking. Physical Review E. 85 (6), (2012).
- Michalet, X. Mean square displacement analysis of single-particle trajectories with localization error: Brownian motion in an isotropic medium. Physical Review E Statistical, Nonlinear, and Soft Matter Physics. 82 (4 Pt 1), 041914 (2010).
- Backlund, M. P., Joyner, R., Moerner, W. E. Chromosomal locus tracking with proper accounting of static and dynamic errors. Physical Review E Statistical, Nonlinear, and Soft Matter Physics. 91 (6), 062716 (2015).
- Stracy, M., et al. Live-cell superresolution microscopy reveals the organization of RNA polymerase in the bacterial nucleoid. Proceedings of the National Academy of Sciences of the United States of America. 112 (32), E4390-E4399 (2015).
- Plochowietz, A., Farrell, I., Smilansky, Z., Cooperman, B. S., Kapanidis, A. N. In vivo single-RNA tracking shows that most tRNA diffuses freely in live bacteria. Nucleic Acids Research. 45 (2), 926-937 (2017).
- Koo, P. K., Mochrie, S. G. Systems-level approach to uncovering diffusive states and their transitions from single-particle trajectories. Physical Review E. 94 (5-1), 052412 (2016).
- Uphoff, S., Reyes-Lamothe, R., Garza de Leon, F., Sherratt, D. J., Kapanidis, A. N. Single-molecule DNA repair in live bacteria. Proceedings of the National Academy of Sciences of the United States of America. 110 (20), 8063-8068 (2013).
- Bakshi, S., Bratton, B. P., Weisshaar, J. C. Subdiffraction-Limit Study of Kaede Diffusion and Spatial Distribution in Live Escherichia coli. Biophysical Journal. 101 (10), 2535-2544 (2011).
- Bakshi, S., Siryaporn, A., Goulian, M., Weisshaar, J. C. Superresolution imaging of ribosomes and RNA polymerase in live Escherichia coli cells. Molecular Microbiology. 85 (1), 21-38 (2012).
- Uphoff, S., Sherratt, D. J., Kapanidis, A. N. Visualizing Protein-DNA Interactions in Live Bacterial Cells Using Photoactivated Single-molecule Tracking. JoVE. (85), e51177 (2014).
- Pavani, S. R. P., Piestun, R. Three dimensional tracking of fluorescent microparticles using a photon-limited double-helix response system. Optics Express. 16 (26), 22048-22057 (2008).
- Pavani, S. R. P., et al. Three-dimensional, single-molecule fluorescence imaging beyond the diffraction limit by using a double-helix point spread function. Proceedings of the National Academy of Sciences of the United States of America. 106 (9), 2995-2999 (2009).
- Rocha, J. M., et al. Single-molecule tracking in live Yersinia enterocolitica reveals distinct cytosolic complexes of injectisome subunits. Integrative Biology. 10 (9), 502-515 (2018).
- Lew, M. D., von Diezmann, A. R. S., Moerner, W. E. Easy-DHPSF open-source software for three-dimensional localization of single molecules with precision beyond the optical diffraction limit. Protocol Exchange. , (2013).
- Biteen, J. S., et al. Super-resolution imaging in live Caulobacter crescentus cells using photoswitchable EYFP. Nature Methods. 5 (11), 947-949 (2008).
- Huang, F., et al. Video-rate nanoscopy using sCMOS camera-specific single-molecule localization algorithms. Nature Methods. 10 (7), 653-658 (2013).
- Hoogendoorn, E., et al. The fidelity of stochastic single-molecule super-resolution reconstructions critically depends upon robust background estimation. Scientific Reports. 4, 3854 (2014).
- Paintdakhi, A., et al. Oufti: An integrated software package for high-accuracy, high-throughput quantitative microscopy analysis. Molecular microbiology. 99 (4), 767-777 (2016).
- Rocha, J. M., Corbitt, J., Yan, T., Richardson, C., Gahlmann, A. Resolving Cytosolic Diffusive States in Bacteria by Single-Molecule Tracking. bioRxiv. , 483321 (2018).
- Lee, A., Tsekouras, K., Calderon, C., Bustamante, C., Presse, S. Unraveling the Thousand Word Picture: An Introduction to Super-Resolution Data Analysis. Chemical Reviews. 117 (11), 7276-7330 (2017).
- Los, G. V., et al. HaloTag: A Novel Protein Labeling Technology for Cell Imaging and Protein Analysis. ACS Chemical Biology. , (2008).
- Gautier, A., et al. An Engineered Protein Tag for Multiprotein Labeling in Living Cells. Chemistry & Biology. 15 (2), 128-136 (2008).
- Bisson-Filho, A. W., et al. Treadmilling by FtsZ filaments drives peptidoglycan synthesis and bacterial cell division. Science. 355 (6326), 739-743 (2017).
- Douglass, K. M., Sieben, C., Archetti, A., Lambert, A., Manley, S. Super-resolution imaging of multiple cells by optimised flat-field epi-illumination. Nature Photonics. 10 (11), 705-708 (2016).
- Zhao, Z., Xin, B., Li, L., Huang, Z. L. High-power homogeneous illumination for super-resolution localization microscopy with large field-of-view. Optics Express. 25 (12), 13382-13395 (2017).
- Yan, T., Richardson, C. J., Zhang, M., Gahlmann, A. Computational Correction of Spatially-Variant Optical Aberrations in 3D Single Molecule Localization Microscopy. bioRxiv. , 504712 (2018).
- Gahlmann, A., Moerner, W. E. Exploring bacterial cell biology with single-molecule tracking and super-resolution imaging. Nature Reviews Microbiology. 12 (1), 9-22 (2014).
- Liu, Z., Lavis, L. D., Betzig, E. Imaging live-cell dynamics and structure at the single-molecule level. Mol Cell. 58 (4), 644-659 (2015).
- Berglund, A. J. Statistics of camera-based single-particle tracking. Physical Review E. 82 (1), 011917 (2010).
- Parry, B. R., et al. The bacterial cytoplasm has glass-like properties and is fluidized by metabolic activity. Cell. 156 (1-2), 183-194 (2014).
- English, B. P., et al. Single-molecule investigations of the stringent response machinery in living bacterial cells. Proceedings of the National Academy of Sciences of the United States of America. 108 (31), E365-E373 (2011).
- Niu, L. L., Yu, J. Investigating intracellular dynamics of FtsZ cytoskeleton with photoactivation single-molecule tracking. Biophysical Journal. 95 (4), 2009-2016 (2008).
- Coquel, A. S., et al. Localization of protein aggregation in Escherichia coli is governed by diffusion and nucleoid macromolecular crowding effect. PLoS Computational Biology. 9 (4), e1003038 (2013).
- Nenninger, A., Mastroianni, G., Mullineaux, C. W. Size Dependence of Protein Diffusion in the Cytoplasm of Escherichia coli. Journal of Bacteriology. 192 (18), 4535-4540 (2010).
- Dix, J. A., Verkman, A. S. Crowding effects on diffusion in solutions and cells. Annual Review of Biophysics. 37, 247-263 (2008).
- Elliott, L. C., Barhoum, M., Harris, J. M., Bohn, P. W. Trajectory analysis of single molecules exhibiting non-brownian motion. Physical Chemistry Chemical Physics. 13 (10), 4326-4334 (2011).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved