Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
Одномолекулярная слежение за микроскопией - инструмент для определения диффузивных состояний цитосолильных молекул
В этой статье
Резюме
3D одномолекулярная микроскопия локализации используется для зондирования пространственных положений и траекторий движения флуоресцентно обозначенных белков в живых бактериальных клетках. Экспериментальный протокол анализа данных, описанный в данном документе, определяет распространенное диффузное поведение цитосолильных белков на основе объединенных одномолекулярных траекторий.
Аннотация
Одномолекулярная локализационная микроскопия зондирует положение и движения отдельных молекул в живых клетках с десятками нанометров пространственного и миллисекундного височного разрешения. Эти возможности делают одномолекулярную микроскопию локализации идеально подходящей для изучения биологических функций молекулярного уровня в физиологически значимых средах. Здесь мы демонстрируем интегрированный протокол для приобретения и обработки/анализа данных отслеживания одной молекулы для извлечения различных диффузивных состояний, которые может проявлять сявряющий белок. Эта информация может быть использована для количественной оценки формирования молекулярного комплекса в живых клетках. Мы предоставляем подробное описание эксперимента по локализации на основе 3D-молекулы на основе камеры, а также последующих шагов по обработке данных, которые дают траектории отдельных молекул. Эти траектории затем анализируются с помощью численного анализа для извлечения распространенных диффузивных состояний флуоресцентно обозначенных молекул и относительного изобилия этих состояний. Основа анализа основана на стохастической моделировании внутриклеточных броуновских диффузионных траекторий, которые пространственно ограничены произвольной геометрией клеток. На основе смоделированных траекторий, сырые одномолекулярные изображения генерируются и анализируются так же, как экспериментальные изображения. Таким образом, экспериментальные ограничения точности и точности, которые трудно откалибровать экспериментально, явно включены в аналитический рабочий процесс. Коэффициент диффузии и относительные фракции населения в преобладающих диффузионных состояниях определяются путем установки распределения экспериментальных значений с использованием линейных комбинаций смоделированных распределений. Мы демонстрируем полезность нашего протокола путем разрешения диффузивных состояний белка, который проявляет различные диффузионные состояния при формировании гомо- и гетеро-олигомерных комплексов в цитозоле бактериального патогена.
Введение
Изучение диффузивного поведения биомолекул дает представление об их биологических функциях. Методы, основанные на флуоресценции, стали ценными инструментами для наблюдения за биомолекулами в их родной клеточной среде. Восстановление флуоресценции после фотоотрубения (FRAP) и флуоресцентной корреляции спектроскопии (FCS)1 обеспечивают ансамбль-средний диффузивного поведения. И наоборот, одномолекулярная локализация микроскопии позволяет наблюдать за отдельными флуоресцентно отмеченными молекулами с высоким пространственным и временным разрешением2,3,4. Наблюдение отдельных молекул выгодно, так как интересующий белок может существовать в различных диффузивных состояниях. Например, два легко различимых диффузионных состояния возникают, когда транскрипционный регулятор, такой как CueR в Escherichia coli,свободно рассеивается в цитозоле или связывается с последовательностью ДНК и становится обездвижемым по шкале времени измерения5 . Одномолекулярное слежение обеспечивает инструмент для непосредственного наблюдения за этими различными состояниями, и для их разрешения не требуется сложных анализов. Тем не менее, становится все более сложным для решения нескольких диффузивных государств и их населения фракций в тех случаях, когда их диффузионные ставки более похожи. Например, из-за зависимости размера коэффициента диффузии различные состояния олигомеризации белка проявляются как различные диффузионные состояния6,7,8,9 , 10. Такие случаи требуют комплексного подхода с точки зрения сбора, обработки и анализа данных.
Критическим фактором, влияющим на диффузивные темпы цитосолильных молекул, является эффект заточения клеточной границы. Ограничения, наложенные на молекулярное движение границами бактериальных клеток, приводят к тому, что измеренная скорость диффузии цитосолильных молекул выглядит медленнее, чем если бы то же самое движение происходило в неограниченном пространстве. Для очень медленно рассеивающихся молекул эффект клеточного заточения незначителен из-за отсутствия столкновений с границей. В таких случаях, это может быть возможным для точного решения диффузионных состояний путем установки распределения молекулярных смещений, r, или очевидной диффузии коэффициенты, D ,с помощью аналитических моделей, основанных на уравнениях для броуновского движения ( случайная диффузия)11,12,13. Однако для быстро распространяющих цитосолильных молекул экспериментальные распределения больше не напоминают те, которые получены для неограниченного броуновского движения из-за столкновений диффузионных молекул с границами клеток. Эффекты заключения должны быть учтены для точного определения неограниченных коэффициентов диффузии флуоресцентно обозначенных молекул. Несколько подходов были недавно разработаны для учета последствий заключения либо (полу-) аналитически 5,14,15,16 или численно через Монте-Карло моделирования Броуновская диффузия6,10,16,17,18,19.
Здесь мы предоставляем интегрированный протокол для сбора и анализа данных одномолекулярной локализации микроскопии с особым акцентом на одномолекулярное отслеживание. Конечная цель протокола заключается в разрешении диффузивных состояний флуоресцентно обозначенных цитосолильных белков внутри, в данном случае, бактериальных клеток в форме стержня. Наша работа основывается на предыдущем протоколе для одномолекулярной слежения, в котором полимераза ДНК, PolI, было показано, существуют в ДНК связаны и несвязанное состояние диффузии анализа20. Здесь мы расширяем одномолекулярный анализ слежения до 3D-измерений и выполняем более реалистичные вычислительные симуляции для разрешения и количественной оценки нескольких диффузивных состояний, одновременно присутствующих в клетках. Данные приобретаются с помощью домашнего 3D супер-разрешение флуоресценции микроскоп, который способен определить 3D положение флуоресцентных излучателей с помощью изображения с двойной селипс точки распространения-функции (DHPSF)21,22. Необработанные одномолекулярные изображения обрабатываются с помощью специально написанного программного обеспечения для извлечения 3D одномолекулярных локализаций, которые затем объединяются в одномолекулярные траектории. Тысячи траекторий объединяются для генерации распределения явных коэффициентов диффузии. На последнем этапе экспериментальные дистрибутивы соответствуют численно генерируемым дистрибутивам, полученным с помощью моделирования броуновского движения Монте-Карло в ограниченном объеме. Мы применяем этот протокол для решения диффузивных состояний белка секреционной системы типа 3 Ysc, в живых Yersinia enterocolitica. Из-за своей модульной природы, наш протокол, как правило, применим к любому типу одномолекулярных или одночастиц слежения эксперимента в произвольной геометрии клеток.
протокол
1. Двухсексовая точечная калибровка
ПРИМЕЧАНИЕ: Изображения, описанные в этом и следующих разделах приобретаются с помощью специально голой перевернутой флуоресценции микроскоп, как описано в Rocha и др.23. Та же процедура применима к различным реализациям микроскопа, предназначенным для одномолекулярной локализации и отслеживания микроскопии2,3,4. Все программное обеспечение для получения изображений и обработки данных, описанное в этой статье, доступно (https://github.com/GahlmannLab2014/Single-Molecule-Tracking-Analysis.git).
- Приготовление агарозных прокладок для монтажа образцов на стеклянных крышках скользит для просмотра под микроскопом.
- Добавьте 1,5-2% по весу низкой точки плавления агароуз до 5 мл буфера M2G (4,9 мМ Na 2 HPO4, 3,1 мМ KH2PO4, 7,5 мМ NH4Cl, 0,5 мМ MgSO4, 10 мкм FeSO4, 0,5 мм На2HPO4 и 0,2% глюкозы). Микроволновая печь в течение нескольких секунд, пока все агарозы растворяется. Не дайте раствору закипеть.
- Дайте раствору агарозы остыть в течение нескольких минут (2-3 мин).
- Пипетка 600 л раствора агарозы на стеклянную крышку скольжения (22 мм х 22 мм). Аккуратно поместите второй стеклянный покров скольжения на верхней части агарозы. Это создает тонкий (0,5 мм) агарозный гель площадку между двумя стеклянными крышками скользит.
- Пусть агароуз колодки сидеть, чтобы затвердеть в течение 20 минут.
- Аккуратно отделите стеклянную крышку скользит. Агарозная подушечка будет прилипать к одному из них.
- Используя лезвие бритвы, разрежьте агарозную площадку на четыре квадратных секции одинакового размера. Каждая квадратная секция может быть использована для одного образца.
- Пипетка 1,5 л флуоресцентного раствора биса на одной агарозной площадке. Решение bead является 1/100000 разбавления фондового раствора в M2G (см. Таблица материалов).
- Перевернуть агарозную подушечку и поместите на стеклянную крышку скольжения, который был очищен в очиститель озона в течение 30 минут. Время очистки выбрано для устранения любых флуоресцентных молекул, придерживающихся обложек.
- Установите стекло крышки образца на держатель образца перевернутого флуоресценции микроскопа и закрепите его в месте используя клейкую ленту или зажимы держателя образца загружаемого образца.
- Добавьте каплю масла погружения на цель микроскопа.
- Поместите держатель образца на микроскоп и закрепите его на месте.
- Инициализировать графический пользовательский интерфейс (GUI) для управления камерой микроскопа, этапом образца и возбуждающим лазером. Здесь специально написанное программное обеспечение в MATLAB используется для управления инструментами(Дополнительная диаграмма 1).
- Инициализация программного обеспечения камеры HCImage Live. Во вкладке Захват в разделе "Управление камерой" установите время экспозиции до 0,03 с. Нажмите в прямом эфире, чтобы начать прямую трансляцию камеры.
- Включите лазер, нажав Открытый 514 нм лазера на интерфейсе GUI, чтобы возбудить флуоресцентные бусы на площадке и просматривать флуоресценции выбросов на камеру с помощью режима живой трансляции (т.е., нет данных, сохраненных на диске).
- Отрегулируйте позиции X и Y на этапе микроскопа, нажав на стрелки 'XY-Pos' в разделе 'Micro-Positioning Stage' GUI, чтобы позиционировать по крайней мере одну флуоресцентную бису в центре поля зрения (FOV). Размер шага можно изменить, нажав на выпадающую коробку ниже стрелок.
- Отрегулируйте положение в стадии микроскопа, нажав на стрелки «З-Пос» под разделом Nano-Positioning Stage GUI. Установите ориентацию двойной селиповой точки распространения-функции (DHPSF) флуоресцентной бисовой бытной точки, чтобы быть вертикальной. Эта вертикальная ориентация определяется как отправная точка в калибровке. Размер шага можно изменить, нажав на выпадающую коробку ниже стрелок.
- В HCImage Live, под захватом (ru) Режимы триггера, скорость и регистрация,измените режим триггера с внутреннего на внешний триггер уровня. Это позволит GUI MATLAB управлять камерой.
- Под последовательностью Настройки сканирования,изменение числа подсчетов кадров до 1200. Выберите папку назначения сохранения, нажав на кнопку с пометкой ... . Наконец нажмите Начало. Количество количество кадров устанавливается до 1200, так что 10 кадров могут быть собраны для каждого из 120 шагов позиции.
- Сканирование через ряд позиций (30 шагов выше и ниже стартового положения с шагом 50 нм) и запись 10 кадров на каждом шагу, используя время экспозиции 0,03 с. Начните автоматизированный процесс, нажав GO под разделом калибровки GUI.
ПРИМЕЧАНИЕ: Параметры для калибровки, включая длину шага, количество шагов, время экспозиции камеры и количество кадров на шаг, также могут быть скорректированы здесь. Около 105 фотонов могут быть приобретены из флуоресцентной бисера в одном кадре, используя 0,03 с время экспозиции в результате чего x, y, z точность локализации около 1 нм. - Выключите лазерное освещение, нажав Закрыть 515 нм лазера в GUI. В HCImage Live, под захватом (ru) Режимы триггера, скорость и регистрация,измените режим триггера обратно на внутренний. Под последовательностью Настройки сканирования меняют количество подсчетов кадров до 200. Выберите папку назначения сохранения, нажав на кнопку с пометкой ... . Нажмите "Начать" для сбора 200 кадров темных изображений с 0,03 с выдержкой времени.
ПРИМЕЧАНИЕ: Даже при отсутствии света, падающего на детектор, каждый пиксель будет зачитывать положительное число (называемое темным смещением), которое может незначительно отличаться между пикселями. Темное смещение значение может меняться с течением времени. Поэтому необходимо собирать темные кадры для каждой калибровки. - Fit DHPSF с двойной гауссианской модели с использованием программного обеспечения Easy-DHPSF24 для получения X и Y позиции биса, а также угол против кривой калибровки.
- Инициализируем программное обеспечение Easy-DHPSF в MATLAB. Под настройкой,установить канал g и установить метод установки mLE с моделью DG. G относится к зеленой камере канала, так как флуоресцентный белок, используемый для получения данных, излучает с зелеными длинами волн. MLE с dG Модель относится к максимальной оценки вероятности с двойной гауссианской модели.
ПРИМЕЧАНИЕ: Размер пикселя и увеличение преобразования зависят от конкретной оптической настройки, и, возможно, потребуется изменить. - Под разделом Калибра DHPSF нажмите Run. Нажмите OK на следующем всплывающем окне, чтобы сохранить настройки по умолчанию.
- Выберите стек изображения, сохраненный в шагах 1.13-1.14. Затем выберите стек изображения с темным фоном, сохраненным в шаге 1.15. Наконец, выберите файл .txt, который был автоматически сохранен во время шага 1.14. Этот файл содержит положение сцены на протяжении всего процесса сканирования.
- В следующем всплывающем окне, если чип полной камеры не был использован для поля зрения, ввинь те стартовые позиции x0 и y0 на чипе. В противном случае, входные данные x0 и y0 - 1.Эта информация может быть найдена в HCImage Live в разделе Binning и SubArrary.
- В следующем окне, изменить размер и изменить положение окна, которое появляется над изображением флуоресцентных бусин так, что это примерно 100 х 100 пикселей в размере и по центру одного флуоресцентного сигнала DHPSF. Затем, дважды нажмите, чтобы продолжить.
ПРИМЕЧАНИЕ: Выбранный DHPSF должен быть изолирован от других сигналов DHPSF и в идеале быть самым ярким бисиней. - Нажмите в центре сигнала DHPSF, между двумя долей, а затем нажмите Enter. Следующее окно показывает увеличенное представление выбранного DHPSF. Точнее, выберите расположение центра DHPSF, нажав.
ПРИМЕЧАНИЕ: Программа будет соответствовать DHPSF, и отображать сырое изображение и реконструкции из припадка. Он также выводит образы шаблонов из калибровки, соответствующей сечению DHPSF на разных позициях. Они будут использованы позже для установки экспериментальных данных. Программа будет вывести X, Y, и я позиции оценивается в каждом кадре. В хорошо выровненная оптическая система, X и Y должны измениться очень мало (отклонение в 30 нм) по мере изменения положения. Если выходная вариация больше 30 нм, фазовая маска, расположенная в плоскости Фурье системы визуализации(рисунок 1),должна быть перестроена и шаги 1,9-1,16 повторяются. - Сохраните графический интерфейс Easy-DHPSF, нажав на значок Сохранить в верхнем левом углу графического интерфейса. Это может быть загружено, нажав на значок нагрузки в графическом интерфейсе.
ПРИМЕЧАНИЕ: Процедура калибровки должна проводиться каждый день эксперимента, чтобы учитывать изменения выравнивания в микроскопе, которые могли произойти из-за колебаний температуры или механических вибраций.
- Инициализируем программное обеспечение Easy-DHPSF в MATLAB. Под настройкой,установить канал g и установить метод установки mLE с моделью DG. G относится к зеленой камере канала, так как флуоресцентный белок, используемый для получения данных, излучает с зелеными длинами волн. MLE с dG Модель относится к максимальной оценки вероятности с двойной гауссианской модели.
2. Подготовка бактериальной культуры
- Подготовьте культурные средства массовой информации, поддерживающие рост бактериальных клеток. Для Y. enterocolitica,используйте 5 мл bhI (Инфузия сердца мозга) бульон, содержащий налидиксовую кислоту (35 мкг/мл) и 2,6-диаминопимелиловую кислоту (80 мкг/мл). Здесь используется штамм Y. enterocolitica, который имеет белок Ysc, помеченный флуоресцентным белком eYFP23.
- Прививать средства массовой информации с бактериальными культурами из морозильной камеры запасов или пластины культур.
- Расти культуры на 28 градусов по Цельсию с встряхивания на ночь.
- Разбавить небольшое количество (250 евро) насыщенной ночной культуры до 5 мл с помощью свежих культурных носителей.
- Выращивайте культуру при 28 градусах Цельсия при встряхивании в течение 60-90 мин.
- Индуцировать экспрессию флуоресцентного белка синтеза. Для Y. enterocolitica, тепловой шок клеток до 37 градусов по Цельсию в шейкере воды, чтобы вызвать yop regulon.
- Инкубировать клетки еще 3 ч при 37 градусах Цельсия с встряхиванием.
- Центрифуга 1 мл культуры при 5000 х г в течение 3 мин при комнатной температуре. Отбросьте супернатант.
- Вымойте гранулы 3 раза с 1 мл M2G средств массовой информации.
- Повторно приостановить гранулированных бактерий в 250 зл ИЛ M2G Media.
- Добавить флуоресцентные бусы в качестве фидуциальных маркеров. Раствор флуоресцентного шарика следует добавлять в надлежащим образом разбавленных количествах, так что есть только 1-2 бусины на FOV при просмотре в микроскоп.
- Аккуратно пипетка или вихрь подвески для отдельных агрегированных ячеек.
- Плита 1,5 л подвески на 1,5-2% агарозной площадке, сделанной с Помощью M2G.
- Перевернуть агарозную подушечку и поместите ее на озоновый микроскоп крышку скольжения. Крышка скольжения должны быть помещены в очиститель озона в течение 30 минут, чтобы уменьшить любой присущий фон флуоресценции.
3. Приобретение данных
- Установите стекло крышки образца на держатель образца перевернутого флуоресценции микроскопа и закрепите его в месте используя клейкую ленту или зажимы держателя образца загружаемого образца.
- Добавьте каплю масла погружения на цель микроскопа, затем поместите держатель образца на микроскоп и закрепите его на месте.
- Инициализировать графический пользовательский интерфейс (GUI) для управления камерой микроскопа, этапом образца и возбуждающим лазером. Здесь специально написанное программное обеспечение в MATLAB используется для управления инструментами.
- Инициализация программного обеспечения камеры HCImage Live. Во вкладке Захват в разделе "Управление камерой" установите время экспозиции до 0,025 с. Нажмите в прямом эфире, чтобы начать прямую трансляцию камеры.
- Отрегулируйте положения X и Y стадии микроскопа, нажав на стрелки XY-Pos под разделом Micro-Positioning Stage GUI, чтобы сканировать образец и найти FOV с соответствующей плотной популяцией бактериальных клеток.
ПРИМЕЧАНИЕ: Для максимизации пропускной записи данных ячейки должны быть как можно плотнее, без перекрытия или касания ячеек. FOV должен также включать по крайней мере 1 флуоресцентный бис, который будет использоваться в качестве фидуциального маркера, предпочтительно расположен в углу FOV. - Отрегулируйте положение в стадии микроскопа, нажав на стрелки З-Поса под разделом Nano-Positioning Stage GUI, чтобы флуоресцентные доли DHPSF были вертикальными.
- Под последовательностью Настройки сканирования,изменение количества кадров до 20 000. Выберите папку назначения сохранения, нажав на кнопку с пометкой ... . Наконец нажмите Кнопка Начните собирать до 20000 кадров камеры, используя короткое время экспозиции 0,025 с. eYFP фотомикинг инициируется с использованием высокой интенсивности возбуждения света на 514 нм19,25.
ПРИМЕЧАНИЕ: Здесь лазерная интенсивность в размере 350 Вт/см2 в фокусной плоскости используется для первоначального отбеливания и последующего изображения одной молекулы EYFP. Фотоактивация молекул eYFP на уф-волнах во время визуализации не использовалась. На бактериальную клетку должен быть не более одного одномолекулярного сигнала. Если плотность одномолекулярного сигнала на начальном этапе слишком высока, продолжайте освещать до тех пор, пока не произойдет достаточное фотоотбеление до начала получения данных. - Выключите лазерное освещение, нажав Закрыть 515 нм лазера в GUI. Соберите 200 кадров темных изображений, используя то же время экспозиции.
- В GUI, проверить окно рядом с Thorlabs led и нажмите Кнопка Toggle Зеркало Up. Это позволит переключить путь от флуоресценции путь к фазе контрастного пути.
- Инициализация программного обеспечения для сбора данных IC Capture 2.4. Это контролирует камеру в фазе контрастного пути. Нажмите кнопку Start/Stop Live Display, чтобы просмотреть прямую трансляцию с камеры. Нажмите Захват (ru) Сохранить изображение для сбора фазового контрастного изображения ячеек в поле зрения.
- Повторите шаги 3.5-3.10 для дополнительных FOVs. Здесь данные, полученные из 500 бактериальных клеток в десяти различных FOVs используется для увеличения числа одномолекулярных траекторий, доступных для анализа.
ВНИМАНИЕ: Клетки, установленные на агарозных прокладки в течение длительных периодов времени, могут вести себя иначе, чем только что установленные клетки. Кроме того, агарозная площадка может потерять свою целостность через некоторое время, что может негативно сказаться на качестве данных. Как правило, не более 3 FOV (30 мин на микроскоп) используются на один образец слайда.
4. Обработка данных
ПРИМЕЧАНИЕ: Модифицированная версия программного обеспечения Easy-DHPSF24 используется в MATLAB для анализа необработанных кадров камеры для извлечения одномолекулярных локализаций. Easy-DHPSF используется специально для пригонки локализации DHPSF в одномолекулярной визуализации. Пользовательские изменения были внесены для реализации максимальная оценка вероятности (MLE) на основе установки рутины, которая учитывает пиксель-зависимых шумовых характеристик современных камер SCMOS26. Он также был изменен, чтобы принять вывод типа файла изображения из программы HCImage Live (.dcimg). Более подробное объяснение программного обеспечения и отдельных шагов, пожалуйста, см.
- Инициализация легкого DHPSF GUI в MATLAB(Дополнительная диаграмма 2). Загрузка в файле сохранена в шаге 1.16.8.
- Определение пороговых значений для каждого из 7 шаблонов вывода в шаге 1.16.7
- Под разделом идентификации калибровки SM нажмите Run. Нажмите OK на следующих двух всплывающих окон, чтобы сохранить настройки по умолчанию.
- Откройте стек изображения, содержащий данные из первого FOV по запросу.
- Выберите небольшой диапазон кадров в соответствии с шаблонами. Обычно кадры 1001-2000 используются для избежания плотных перекрывающихся сигналов в первых нескольких сотнях кадров. Нажмите OK на следующем всплывающем окне, чтобы сохранить настройки по умолчанию. Нажмите Отмена при запросе для файла журнала последовательности в следующем окне.
- Откройте стек изображения с темным фоном, сохраненным в шаге 3.8. Нажмите 'OK'в следующем всплывающем окне, чтобы оставить параметры для фоновой оценки, установленной по умолчанию. По умолчанию необходимо оценить фон с помощью медианного фильтра27, охватывающего 100 последующих кадров камеры вокруг текущего кадра.
- В следующем окне, изменить размер окна накладывается на изображение, чтобы покрыть полный FOV, а затем дважды нажмите, чтобы продолжить.
- Определите область интереса, нажав несколько точек на изображении, чтобы создать полигон. Область интереса должна включать как можно больше поля зрения, как это возможно, обеспечивая при этом, что любые флуоресцентные бусы (очень яркие объекты) на изображении не лежат в пределах полигона, а затем дважды нажмите, чтобы продолжить.
ПРИМЕЧАНИЕ: Программное обеспечение будет пытаться сопоставить шаблоны с изображением, и будет отображать изображение с возможными совпадениями кружили. - Когда программное обеспечение остановилось, оно сохранит множество изображений найденных совпадений шаблонов и отобразит соответствующее значение порога в заранее определенной папке. Более высокое пороговое значение соответствует лучшему соответствию. Для каждого номера шаблона просмотрите совпадения примера и определите самый нижний порог, на который изображен DHPSF. Ввиньте эти пороговые значения для каждого из 7 шаблонов в разделе Калибрование SM Identification в графическом интерфейсе Easy-DHPSF.
ПРИМЕЧАНИЕ: Программа будет пытаться автоматически выбирать и вводимых порогов, однако, они часто ненадежны и должны быть проверены вручную. Пороги выбираются таким образом, что несколько истинных одномолекулярных сигналов пропущены, но количество ложноположительных кандидатов на установку остается вычислительно управляемым. - Сохранить Easy-DHPSF GUI снова, нажав на значок Сохранить в верхнем левом углу.
- Установка флуоресцентной бисы в FOV для использования в качестве фидуциального маркера
- В разделе Трек фидуциарных Easy-DHPSF GUI, нажмите Run. Нажмите OK на следующих двух всплывающих окон, чтобы сохранить настройки по умолчанию.
- Перетащите поле накладной на изображение и центр его над сигналом DHPSF от флуоресцентной бисовой, а затем дважды нажмите кнопку.
- Нажмите в центре сигнала DHPSF, в средней точке между двумя долей, а затем нажмите введите. Нажмите Отмена при запросе для файла журнала последовательности в следующем окне.
ПРИМЕЧАНИЕ: Программное обеспечение будет соответствовать DHPSF во всех кадрах камеры, и отображать сырое изображение и реконструированное изображение. Когда программное обеспечение будет закончено, он будет выход цифры с X, Y, и й позиции флуоресцентной бисовой в течение всего срока приобретения изображения. - Проверьте поле рядом с фидуциерами использования и сохранить Easy-DHPSF GUI снова, нажав на значок Сохранить в верхнем левом углу.
- Найдите и уместите все локализации во всех кадрах камеры, используя пороги шаблона, полученные в шаге 4.2.
- В разделе Локализация DHPSF SMs в графическом интерфейсе Easy-DHPSF нажмите Run. Нажмите OK на следующих всплывающих окон, чтобы сохранить настройки по умолчанию. Нажмите Отмена при запросе для файла журнала последовательности в следующем окне.
ПРИМЕЧАНИЕ: Программное обеспечение найдет и подходит DHPSF с использованием двойной гауссианской модели, если качество матча выше пользовательского порога. Он будет отображать сырое изображение с кругами вокруг шаблона матчей, а также изображение реконструированных DHPSF подходит. - Сохранить Easy-DHPSF GUI снова, нажав на значок Сохранить в верхнем левом углу.
- В разделе Локализация DHPSF SMs в графическом интерфейсе Easy-DHPSF нажмите Run. Нажмите OK на следующих всплывающих окон, чтобы сохранить настройки по умолчанию. Нажмите Отмена при запросе для файла журнала последовательности в следующем окне.
- Просмотр одномолекулярных локализаций и фильтрация нежелательных или ненадежных локализаций
- Под разделом локализации output DHPSF SM нажмите Кнопка Выход фильтра.
- Нажмите OK в следующих трех окнах, чтобы выполнить интерполяцию фидуциальных Позиций X,Y и No с течением времени. В большинстве случаев достаточно вариантов по умолчанию. Если черная интерполированная линия не отражает разумной интерполяции красной линии положения, измените параметры интерполяции во всплывающем окне.
ПРИМЕЧАНИЕ: Интерполированная линия используется для коррекции одномолекулярных локализаций. - Откройте соответствующее фазовое контрастное изображение для анализа FOV при запросе. Нажмите OK на следующих двух всплывающих окон, чтобы сохранить настройки по умолчанию.
- В следующих двух всплывающих окнах, изменить значения фильтра, чтобы обеспечить более строгие или более мягкие одномолекулярные требования к локализации, а затем нажмите OK.
- В окне, которое появляется, перетащите или изменить размер окна, наложенного на изображения, чтобы просмотреть желаемую область интереса и дважды щелкните, чтобы продолжить.
- Отображается 3D-реконструкция одномолекулярных локализаций. Используйте инструменты фигуры для управления реконструкцией (поворот, масштабирование и т.д.). Нажмите Продолжить отобразить другой диалог поле с просьбой "Вы хотели бы переусмотреть с другим набором параметров?'. Если результаты удовлетворительны, нажмите Нет. Если это не так, нажмите Да.
ПРИМЕЧАНИЕ: Общие причины неудовлетворительных результатов включают фазовое контрастное изображение, не правильно наложенное данными локализации, или пороги исходного шаблона были слишком низкими, создавая множество ложноположительных локализаций. Если Yes был выбран, программное обеспечение вернется к шагу 4.5.4, так что новые значения параметров могут быть определены. Если нет был выбран, результаты будут сохранены.
5. После обработки данных
- Используя специально написанное программное обеспечение в MATLAB, обрезать фазовые контрастные изображения так, что только область, которая содержит клетки, которые были изображены под флуоресцентным микроскопом остается. Этот шаг необходим, потому что фазовое контрастное изображение охватывает область, намного большую, чем флуоресценция изображения. Обрезка изображения упрощает следующий шаг.
- Сегмент отдельных ячеек путем обработки фазового контрастного изображения с программным обеспечением OUFTI28 (Дополнительный рисунок 3)
- Инициализация OUFTI в MATLAB. Загрузите обрезанное изображение контрастного фазы от предыдущего шага, нажав на фазу нагрузки.
- Нажмите файл, чтобы выбрать и назвать место сохранения для выходного файла.
- Выберите независимые кадры под заголовком «Обнаружение и анализ».
- Нажмите Параметры нагрузки для загрузки параметров для обнаружения ячейки. Примеры параметров включают приемлемую область ячейки, ширину ячейки и порог расщепления ячейки.
ПРИМЕЧАНИЕ: Все эти параметры должны быть скорректированы, чтобы максимизировать производительность для конкретных размеров ячейки и качества изображения используется. Важно отметить, что параметр алгоритма должен быть установлен на субпиксель, чтобы обеспечить точное измерение контуров клеток. - Нажмите на эту рамку, чтобы начать сегментацию ячеек. Контуры клеток будут отображаться над изображением фазового контраста, когда процесс закончен.
- Используя элементы управления в разделе Руководство, разделить ячейки, добавить ячейки, или уточнить контуры клеток для получения контуров для ячеек, которые были неточно сегментированы во время автоматизированного процесса.
- Выход ячейки очертания, нажав Сохранить анализ.
- Используйте специально написанное программное обеспечение в MATLAB для точного наложения контуров, полученных на предыдущем этапе, с одномолекулярными локализациями. Следующие подшаги подробно шаги программного обеспечения.
- Вручную выберите 5 пар контрольных точек во всплывающем окне, грубо оценив аяии и нажав на положение клеточных полюсов тех же пяти клеток как в одномолекулярных данных локализации, так и в очертаниях клеток, созданных на предыдущем этапе. Положение клеточного полюса можно приблизительно оценить, мысленно нарисовав выпуклый корпус вокруг одномолекулярных локализаций, принадлежащих одной клетке, и выбрав точку наибольшей кривизны(Дополнительная диаграмма 4).
- Создайте функцию преобразования 2D-аффин с использованием функции cp2tform в MATLAB и используйте ее для создания грубой накладки клеточных контуров и одномолекулярных локализаций.
- Удалить ячейки, содержащие менее 10 локализаций, и удалить ячейки, которые расположены частично за пределами поля зрения. Вручную удалите любые дополнительные нежелательные ячейки во всплывающем окне, нажав внутри их контура ячейки(Дополнительная диаграмма 5).
- Используйте центр массы для всех оставшихся клеточных контуров и одномолекулярных локализаций внутри них, чтобы сформировать больший набор пар контрольной точки, перевычислить функцию 2D-трансформации и использовать ее для создания окончательного наложения контуров клеток и одномолекулярные локализации.
- Присвоить локализации, которые находятся в пределах границы контура ячейки, этой ячейке. Откажитесь от любых локализаций, не расположенных в пределах каких-либо контуров клеток(рисунок 2a, Дополнительная рисунок 6).
6. Одномолекулярное слежение
ПРИМЕЧАНИЕ: Следующий раздел завершается с помощью пользовательского программного обеспечения в MATLAB. В этом разделе описаны шаги, выполняемые программным обеспечением.
- Для локализации, назначенных в одну и ту же ячейку, и в последующих кадрах камеры вычислите евклидовое расстояние между локализациями. Если расстояние между локализациями ниже порога в 2,5 мкм, свяжите локализацию, назначив их на одну и ту же одномолекулярную траекторию.
ПРИМЕЧАНИЕ: Важно учитывать только локализации в одной ячейке, чтобы локализации в соседних ячейках, которые отвечают требованиям пространственного и временного порога, не были связаны между собой. Порог в 2,5 мкм был выбран в качестве максимальногорасстояния, в результате которого очень быстрая молекула (30 мкм 2/с) могла перемещаться в течение времени экспозиции (0,025 с) плюс 20% буфера. - Откажитесь от траекторий короче 4 локализаций. Если две или более локализаций (т.е. два или более флуоресцентных излучателей) одновременно присутствуют в клетке, отбросьте связанные траектории. Установка минимума длины трассы до 4 локализаций позволяет усреднеть несколько измерений расстояния, что дает более точную оценку коэффициентов диффузии.
- Рассчитайте среднее смещение квадрата (MSD) для заданной траектории:
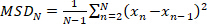 (1)
(1)
где N является общее количество локализаций в траектории и xn является положение молекулы в точке времени n. - Вычислить кажущийся коэффициент диффузии, D
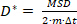 (2)
(2)
где м 2 или 3 - это размерность измерения, а это время экспозиции камеры.
ПРИМЕЧАНИЕ: Типичный эксперимент будет производить 5000-100000 траекторий в общей сложности, в результате чего очевидное распределение коэффициента диффузии с таким количеством рассчитывает.
7. Монте-Карло моделирования броуновского движения в ограниченном объеме
ПРИМЕЧАНИЕ: Создание библиотек смоделированных видимых дистрибутивов коэффициента диффузии путем выполнения моделирования Броуновского движения Монте-Карло, ограниченного цилиндрическим объемом, используя 64 значения в диапазоне 0,05-20 мкм2/с в качестве входных параметров (программное обеспечение доступны от авторов по запросу). Этот диапазон был выбран для покрытия диапазона ранее оцененных коэффициентов диффузии флуоресцентных (фьюжн) белков в бактериях. 64 коэффициента диффузии используются для достаточной пробы этогодиапазона. Этот раздел выполняется с использованием пользовательского программного обеспечения в MATLAB и описывает шаги, которые программное обеспечение автоматически принимает. Используемые здесь клетки в форме стержня Y. enterocolitica приближены к цилиндрическому объему длины l 5 мкм и диаметру d 0,8 мкм.
- Инициировать отдельные траектории в случайном положении в цилиндрическом объеме и имитировать их случайные (т.е. броуновские) шаги диффузии, используя временной интервал в 100 нс (временной интервал должен быть намного короче времени экспозиции камеры, чтобы обеспечить достаточное количество выборка положения в течение кадра камеры). Пример каждого шага перемещения для входного D из соответствующей гауссийской функции распределения путем перестановки Eqn. 2 и добавьте его к предыдущему положению:
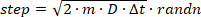 (3)
(3)
где рандн функции в MATLAB образцов случайное число из нормального распределения. Если шаг приводит к смещению молекулы за пределы объема цилиндра, отражайте молекулу обратно внутри цилиндра под случайным углом. - Для каждого интервала времени, генерировать изображение DHPSF, которое соответствует мгновенной х,y,z положение смоделированного излучателя.
- Чтобы соответствовать экспериментальным условиям, моделируйте изображения, содержащие 1000 фотонов на локализацию, с лазерным фоном 13 фотонов на пиксель и шумом Пуассона. Кроме того, добавьте темное смещение в размере 50 фотонов на пиксель и гауссианский считывательный шум (1,5 фотоны), соответствующие экспериментальным измерениям калибровки камеры. Наконец, умножьте изображение на экспериментально измеренное пиксельно-зависимое увеличение камеры sCMOS для получения изображения в единицах отсчета детекторов.
ПРИМЕЧАНИЕ: После этих манипуляций соотношение сигнала к шуму конечного изображения составляет 2 евро. - Создание размытых движениями изображений, которые отражают изменение положения молекул, подводя 50 изображений DHPSF, смоделированных во время экспозиции, используемой во время экспериментального сбора данных. Для ограничения вычислительных расходов для создания изображения было выбрано только 50 периодически отобранных позиций (вместо всех 250 000 позиций, отобранных в течение времени экспозиции 0,025 с интервалом выборки в 100 нч).
ПРИМЕЧАНИЕ: Длина (количество кадров) смоделированных траекторий должна соответствовать средней длине экспериментальных траекторий. В этом случае количество кадров на траекторию составляет 6.
- Чтобы соответствовать экспериментальным условиям, моделируйте изображения, содержащие 1000 фотонов на локализацию, с лазерным фоном 13 фотонов на пиксель и шумом Пуассона. Кроме того, добавьте темное смещение в размере 50 фотонов на пиксель и гауссианский считывательный шум (1,5 фотоны), соответствующие экспериментальным измерениям калибровки камеры. Наконец, умножьте изображение на экспериментально измеренное пиксельно-зависимое увеличение камеры sCMOS для получения изображения в единицах отсчета детекторов.
- Создайте 5000 траекторий для каждого из 64 смоделированных коэффициентов диффузии ввода.
- Проанализируйте смоделированные кадры камеры, описанные в разделе Обработка данных.
- Связать локализации в траектории, описанные в разделе слежения за одномолекуляцией.
- Интерполировать функцию совокупного распределения (CDF) для каждого смоделированного распределения с помощью B-spline интерполяции порядка 25. Интерполированные распределения необходимы для того, чтобы их можно было запрашивать в произвольных точках.
- Интерполировать результирующие B-сплайн кривые вдоль D-оси (D является истинным неограниченным коэффициентом диффузии, который регулирует движение смоделированных излучателей) с помощью разрозненных функций Интерполята в MATLAB (укажите ' метод омовения природы). Это обеспечивает непрерывную 2D функцию, из которой можно запрашивать любое очевидное распределение коэффициента диффузии,соответствующее истинному коэффициенту диффузии в диапазоне 0,05-20 мкм 2/с.
8. Экспериментальный коэффициент распространения диффузии
ПРИМЕЧАНИЕ: Fit экспериментально измеренных распределений очевидных коэффициентов диффузии с использованием линейных комбинаций смоделированных распределений, генерируемых в предыдущем разделе (Монте-Карло моделирования броуновского движения в ограниченном объеме). Этот раздел выполняется с использованием пользовательского программного обеспечения в MATLAB и описывает шаги, которые программное обеспечение автоматически принимает. Для получения дополнительной информации и примеров применения, пожалуйста, см.
- Выполните ограниченные линейные наименьшие квадраты ( с использованием функции lsqlin в MATLAB) экспериментального CDF, используя периодически отобранный массив смоделированных CD-файлов из библиотеки, созданной в разделе 7. Выход этого шага является паравектором, содержащим коэффициенты диффузии и демографические фракции распространенных диффузных состояний в экспериментальном распределении.
- Объедините диффузионные состояния с диффузионными значениями коэффициента в пределах 20% друг от друга в единое диффузионное состояние по усреднению веса на основе относительной фракции населения. Это вектор исходного параметра.
ПРИМЕЧАНИЕ: Чтобы уменьшить сложность модели, диффузионные состояния с коэффициентами диффузии ниже 0,5 мкм2/с могут быть удерживаться постоянными в течение всех следующих шагов. - Создавайте массивы векторов параметра пробной установки с различными числами диффузионных состояний, начиная от одного диффузного состояния и кончаясь максимального количества состояний.
- Используя вектор исходного параметра, объедините смежные диффузивные состояния через взвешенное усреднение и разделение диффузионных состояний на два состояния с равными фракциями населения и коэффициентами диффузии на 20% выше и ниже исходного значения. Повторите для всех возможностей комбинации состояния и разделения.
- Используйте каждый вектор параметра пробной установки для инициализации нелинейной наименьшей квадратной установки 5 отдельных подмножества данных (с использованием функции fmincon в MATLAB). Определите качество пригонки, найдя остаточную сумму квадратов между подгонкой и распределением, соответствующим оставшимся подмм (перекрестная проверка данных).
- Используйте среднюю остаточную сумму квадратов 5 отдельных фитингов для каждого пробного вектора в качестве общего качества подгонки, определите пробный вектор с лучшим качеством подходящих для каждого числа диффузионных состояний.
- Определите оптимальное количество состояний путем определения пробного вектора, для которого добавление дополнительного состояния не приводит к улучшению качества подгонки на 5%.
- Используйте этот пробный вектор, чтобы инициализировать нелинейные наименьшие квадраты, подходящие из полного набора данных.
- Оцените ошибку отдельных параметров путем повторного отбора данных путем загрузки и переоборудования с тем же вектором проб.
Результаты
В описанных здесь экспериментальных условиях (20 000 кадров, длина траектории минимум 4 локализации) и в зависимости от уровня экспрессии флуоресцентно обозначенных термоядерных белков, приблизительно 200-3000 локализаций дают 10-150 траектории могут быть сгенерированы на яч...
Обсуждение
Критическим фактором для успешного применения представленного протокола является обеспечение того, чтобы одномолекулярные сигналы были хорошо отделены друг от друга (т.е. они должны быть редкими в пространстве и во времени(Дополнительный Mov. 1)). Если в клетке одновременно нах?...
Раскрытие информации
Авторам нечего раскрывать.
Благодарности
Мы благодарим Алеся Ахимович и Тингя Нан за критическое прочтение рукописи. Мы благодарим Эда Холла (Ed Hall), старшего научного сотрудника группы Advanced Research Computing Services в Университете Вирджинии, за помощь в настройке процедур оптимизации, используемых в этой работе. Финансирование этой работы было предоставлено Университетом Вирджинии.
Материалы
| Name | Company | Catalog Number | Comments |
| 2,6-diaminopimelic acid | Chem Impex International | 5411 | Necessary for growth of Y. enterocolitica cells used. |
| 4f lenses | Thorlabs | AC508-080-A | f = 80mm, 2" |
| 514 nm laser | Coherent | Genesis MX514 MTM | Use for fluorescence excitation |
| agarose | Inivtrogen | 16520100 | Used to make gel pads to mount liquid bacterial sample on microscope. |
| ammonium chloride | Sigma Aldrich | A9434 | M2G ingredient. |
| bandpass filter | Chroma | ET510/bp | Excitation pathway. |
| Brain Heart Infusion | Sigma Aldrich | 53286 | Growth media for Y. enterocolitica. |
| calcium chloride | Sigma Aldrich | 223506 | M2G ingredient. |
| camera | Imaging Source | DMK 23UP031 | Camera for phase contrast imaging. |
| dielectric phase mask | Double Helix, LLC | N/A | Produces DHPSF signal. |
| disodium phosphate | Sigma Aldrich | 795410 | M2G ingredient. |
| ethylenediaminetetraacetic acid | Fisher Scientific | S311-100 | Chelates Ca2+. Induces secretion in the T3SS. |
| flip mirror | Newport | 8892-K | Allows for switching between fluorescence and phase contrast pathways. |
| fluospheres | Invitrogen | F8792 | Fluorescent beads. 540/560 exication and emission wavelengths. 40 nm diameter. |
| glass cover slip | VWR | 16004-302 | #1.5, 22mmx22mm |
| glucose | Chem Impex International | 811 | M2G ingredient. |
| immersion oil | Olympus | Z-81025 | Placed on objective lens. |
| iron(II) sulfate | Sigma Aldrich | F0518 | M2G ingredient. |
| long pass filter | Semrock | LP02-514RU-25 | Emission pathway. |
| magnesium sulfate | Fisher Scientific | S25414A | M2G ingredient. |
| microscope platform | Mad City Labs | custom | Platform for inverted microscope. |
| nalidixic acid | Sigma Aldrich | N4382 | Y. enterocolitica cells used are resistant to nalidixic acid. |
| objective lens | Olympus | 1-U2B991 | 60X, 1.4 NA |
| Ozone cleaner | Novascan | PSD-UV4 | Used to eliminate background fluorescence on glass cover slips. |
| potassium phosphate | Sigma Aldrich | 795488 | M2G ingredient. |
| Red LED | Thorlabs | M625L3 | Illuminates sample for phase contrast imaging. 625nm. |
| sCMOS camera | Hamamatsu | ORCA-Flash 4.0 V2 | Camera for fluorescence imaging. |
| short pass filter | Chroma | ET700SP-2P8 | Emission pathway. |
| Tube lens | Thorlabs | AC508-180-A | f=180 mm, 2" |
| Yersinia enterocolitica dHOPEMTasd | N/A | N/A | Strain AD4442, eYFP-YscQ |
| zero-order quarter-wave plate | Thorlabs | WPQ05M-514 | Excitation pathway. |
Ссылки
- Kapanidis, A. N., Uphoff, S., Stracy, M. Understanding Protein Mobility in Bacteria by Tracking Single Molecules. Journal of Molecular Biology. , (2018).
- Betzig, E., et al. Imaging intracellular fluorescent proteins at nanometer resolution. Science. 313 (5793), 1642-1645 (2006).
- Hess, S. T., Girirajan, T. P. K., Mason, M. D. Ultra-high resolution imaging by fluorescence photoactivation localization microscopy. Biophysical Journal. 91 (11), 4258-4272 (2006).
- Rust, M. J., Bates, M., Zhuang, X. W. Sub-diffraction-limit imaging by stochastic optical reconstruction microscopy (STORM). Nature Methods. 3 (10), 793-795 (2006).
- Chen, T. Y., et al. Quantifying Multistate Cytoplasmic Molecular Diffusion in Bacterial Cells via Inverse Transform of Confined Displacement Distribution. Journal of Physical Chemistry B. 119 (45), 14451-14459 (2015).
- Mohapatra, S., Choi, H., Ge, X., Sanyal, S., Weisshaar, J. C. Spatial Distribution and Ribosome-Binding Dynamics of EF-P in Live Escherichia coli. mBio. 8 (3), (2017).
- Stracy, M., et al. Single-molecule imaging of UvrA and UvrB recruitment to DNA lesions in living Escherichia coli. Nature Communications. 7, 12568 (2016).
- Persson, F., Lindén, M., Unoson, C., Elf, J. Extracting intracellular diffusive states and transition rates from single-molecule tracking data. Nature Methods. 10 (3), 265-269 (2013).
- Bakshi, S., Choi, H., Weisshaar, J. C. The spatial biology of transcription and translation in rapidly growing Escherichia coli. Frontiers in Microbiology. 6, 636 (2015).
- Mustafi, M., Weisshaar, J. C. Simultaneous Binding of Multiple EF-Tu Copies to Translating Ribosomes in Live Escherichia coli. mBio. 9 (1), (2018).
- Michalet, X., Berglund, A. J. Optimal diffusion coefficient estimation in single-particle tracking. Physical Review E. 85 (6), (2012).
- Michalet, X. Mean square displacement analysis of single-particle trajectories with localization error: Brownian motion in an isotropic medium. Physical Review E Statistical, Nonlinear, and Soft Matter Physics. 82 (4 Pt 1), 041914 (2010).
- Backlund, M. P., Joyner, R., Moerner, W. E. Chromosomal locus tracking with proper accounting of static and dynamic errors. Physical Review E Statistical, Nonlinear, and Soft Matter Physics. 91 (6), 062716 (2015).
- Stracy, M., et al. Live-cell superresolution microscopy reveals the organization of RNA polymerase in the bacterial nucleoid. Proceedings of the National Academy of Sciences of the United States of America. 112 (32), E4390-E4399 (2015).
- Plochowietz, A., Farrell, I., Smilansky, Z., Cooperman, B. S., Kapanidis, A. N. In vivo single-RNA tracking shows that most tRNA diffuses freely in live bacteria. Nucleic Acids Research. 45 (2), 926-937 (2017).
- Koo, P. K., Mochrie, S. G. Systems-level approach to uncovering diffusive states and their transitions from single-particle trajectories. Physical Review E. 94 (5-1), 052412 (2016).
- Uphoff, S., Reyes-Lamothe, R., Garza de Leon, F., Sherratt, D. J., Kapanidis, A. N. Single-molecule DNA repair in live bacteria. Proceedings of the National Academy of Sciences of the United States of America. 110 (20), 8063-8068 (2013).
- Bakshi, S., Bratton, B. P., Weisshaar, J. C. Subdiffraction-Limit Study of Kaede Diffusion and Spatial Distribution in Live Escherichia coli. Biophysical Journal. 101 (10), 2535-2544 (2011).
- Bakshi, S., Siryaporn, A., Goulian, M., Weisshaar, J. C. Superresolution imaging of ribosomes and RNA polymerase in live Escherichia coli cells. Molecular Microbiology. 85 (1), 21-38 (2012).
- Uphoff, S., Sherratt, D. J., Kapanidis, A. N. Visualizing Protein-DNA Interactions in Live Bacterial Cells Using Photoactivated Single-molecule Tracking. JoVE. (85), e51177 (2014).
- Pavani, S. R. P., Piestun, R. Three dimensional tracking of fluorescent microparticles using a photon-limited double-helix response system. Optics Express. 16 (26), 22048-22057 (2008).
- Pavani, S. R. P., et al. Three-dimensional, single-molecule fluorescence imaging beyond the diffraction limit by using a double-helix point spread function. Proceedings of the National Academy of Sciences of the United States of America. 106 (9), 2995-2999 (2009).
- Rocha, J. M., et al. Single-molecule tracking in live Yersinia enterocolitica reveals distinct cytosolic complexes of injectisome subunits. Integrative Biology. 10 (9), 502-515 (2018).
- Lew, M. D., von Diezmann, A. R. S., Moerner, W. E. Easy-DHPSF open-source software for three-dimensional localization of single molecules with precision beyond the optical diffraction limit. Protocol Exchange. , (2013).
- Biteen, J. S., et al. Super-resolution imaging in live Caulobacter crescentus cells using photoswitchable EYFP. Nature Methods. 5 (11), 947-949 (2008).
- Huang, F., et al. Video-rate nanoscopy using sCMOS camera-specific single-molecule localization algorithms. Nature Methods. 10 (7), 653-658 (2013).
- Hoogendoorn, E., et al. The fidelity of stochastic single-molecule super-resolution reconstructions critically depends upon robust background estimation. Scientific Reports. 4, 3854 (2014).
- Paintdakhi, A., et al. Oufti: An integrated software package for high-accuracy, high-throughput quantitative microscopy analysis. Molecular microbiology. 99 (4), 767-777 (2016).
- Rocha, J. M., Corbitt, J., Yan, T., Richardson, C., Gahlmann, A. Resolving Cytosolic Diffusive States in Bacteria by Single-Molecule Tracking. bioRxiv. , 483321 (2018).
- Lee, A., Tsekouras, K., Calderon, C., Bustamante, C., Presse, S. Unraveling the Thousand Word Picture: An Introduction to Super-Resolution Data Analysis. Chemical Reviews. 117 (11), 7276-7330 (2017).
- Los, G. V., et al. HaloTag: A Novel Protein Labeling Technology for Cell Imaging and Protein Analysis. ACS Chemical Biology. , (2008).
- Gautier, A., et al. An Engineered Protein Tag for Multiprotein Labeling in Living Cells. Chemistry & Biology. 15 (2), 128-136 (2008).
- Bisson-Filho, A. W., et al. Treadmilling by FtsZ filaments drives peptidoglycan synthesis and bacterial cell division. Science. 355 (6326), 739-743 (2017).
- Douglass, K. M., Sieben, C., Archetti, A., Lambert, A., Manley, S. Super-resolution imaging of multiple cells by optimised flat-field epi-illumination. Nature Photonics. 10 (11), 705-708 (2016).
- Zhao, Z., Xin, B., Li, L., Huang, Z. L. High-power homogeneous illumination for super-resolution localization microscopy with large field-of-view. Optics Express. 25 (12), 13382-13395 (2017).
- Yan, T., Richardson, C. J., Zhang, M., Gahlmann, A. Computational Correction of Spatially-Variant Optical Aberrations in 3D Single Molecule Localization Microscopy. bioRxiv. , 504712 (2018).
- Gahlmann, A., Moerner, W. E. Exploring bacterial cell biology with single-molecule tracking and super-resolution imaging. Nature Reviews Microbiology. 12 (1), 9-22 (2014).
- Liu, Z., Lavis, L. D., Betzig, E. Imaging live-cell dynamics and structure at the single-molecule level. Mol Cell. 58 (4), 644-659 (2015).
- Berglund, A. J. Statistics of camera-based single-particle tracking. Physical Review E. 82 (1), 011917 (2010).
- Parry, B. R., et al. The bacterial cytoplasm has glass-like properties and is fluidized by metabolic activity. Cell. 156 (1-2), 183-194 (2014).
- English, B. P., et al. Single-molecule investigations of the stringent response machinery in living bacterial cells. Proceedings of the National Academy of Sciences of the United States of America. 108 (31), E365-E373 (2011).
- Niu, L. L., Yu, J. Investigating intracellular dynamics of FtsZ cytoskeleton with photoactivation single-molecule tracking. Biophysical Journal. 95 (4), 2009-2016 (2008).
- Coquel, A. S., et al. Localization of protein aggregation in Escherichia coli is governed by diffusion and nucleoid macromolecular crowding effect. PLoS Computational Biology. 9 (4), e1003038 (2013).
- Nenninger, A., Mastroianni, G., Mullineaux, C. W. Size Dependence of Protein Diffusion in the Cytoplasm of Escherichia coli. Journal of Bacteriology. 192 (18), 4535-4540 (2010).
- Dix, J. A., Verkman, A. S. Crowding effects on diffusion in solutions and cells. Annual Review of Biophysics. 37, 247-263 (2008).
- Elliott, L. C., Barhoum, M., Harris, J. M., Bohn, P. W. Trajectory analysis of single molecules exhibiting non-brownian motion. Physical Chemistry Chemical Physics. 13 (10), 4326-4334 (2011).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеThis article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены