このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
単一分子トラッキング顕微鏡 - 細胞細胞分子の拡散状態を決定するツール
要約
3D単分子局在顕微鏡は、生きた細菌細胞における蛍光標識タンパク質の空間位置および運動軌道をプローブするために利用される。本明細書に記載される実験およびデータ分析プロトコルは、プールされた単一分子軌道に基づいてサイトソリックタンパク質の一般的な拡散挙動を決定する。
要約
単一分子局在顕微鏡は、数十ナノメートルの空間的およびミリ秒の時間分解能を有する生細胞における個々の分子の位置と動きを調べた。これらの機能により、単分子局在顕微鏡は、生理学的に関連する環境での分子レベルの生物学的機能の研究に最適です。ここでは、単一分子トラッキングデータの取得と処理/分析の両方のための統合プロトコルを示し、目的のタンパク質が示す可能性のある異なる拡散状態を抽出する。この情報は、生細胞における分子複合体形成を定量化するために使用することができる。カメラベースの3D単一分子ローカリゼーション実験の詳細な説明と、個々の分子の軌跡を生み出すその後のデータ処理ステップについて説明します。これらの軌道は、数値解析フレームワークを使用して分析され、蛍光標識分子の一般的な拡散状態とこれらの状態の相対的な豊富さを抽出します。解析フレームワークは、任意のセル ジオメトリによって空間的に限定される細胞内ブラウン拡散軌道の確率シミュレーションに基づいています。シミュレートされた軌道に基づいて、生の単一分子画像が実験画像と同じように生成され、解析されます。このように、実験的に校正するのが難しい実験精度と精度の制限が、解析ワークフローに明示的に組み込まれます。一般的な拡散状態の拡散係数と相対母率は、シミュレートされた分布の線形組み合わせを使用して実験値の分布を適合することによって決定されます。我々は、細菌病原体のサイトソールにホモとヘテロオリゴマー複合体を形成する際に異なる拡散状態を示すタンパク質の拡散状態を解明することにより、我々のプロトコルの有用性を実証する。
概要
生体分子の拡散的な挙動を調べることは、その生物学的機能に関する洞察を提供する。蛍光顕微鏡ベースの技術は、生体分子をネイティブの細胞環境で観察するための貴重なツールとなっています。光漂白後の蛍光回収(FRAP)および蛍光相関分光法(FCS)1は、アンサンブル平均拡散行動を提供する。逆に、単一分子局在化顕微鏡は、高い空間および時間分解能2、3、4を有する個々の蛍光タグ付き分子の観察を可能にする。目的のタンパク質が異なる拡散状態に存在する可能性があるため、個々の分子を観察することは有利です。例えば、大腸菌のCueRのような転写レギュレータがサイトゾルで自由に拡散したり、DNA配列に結合したりすると、2つの容易に区別可能な拡散状態が生じ、測定5のタイムスケールで固定化される5.単一分子トラッキングは、これらの異なる状態を直接観察するためのツールを提供し、それらを解決するために高度な分析は必要ありません。ただし、拡散率が類似している場合は、複数の拡散状態とその母集団の割合を解決することがより困難になります。例えば、拡散係数の大きさ依存性のために、タンパク質の異なるオリゴマー化状態は、異なる拡散状態6、7、8、9として現れる,10.このような場合は、データの取得、処理、分析の面で統合されたアプローチが必要です。
サイトソリック分子の拡散率に影響を与える重要な因子は、細胞境界による閉じ込めの影響である。細菌細胞境界によって分子運動に対する制限により、細胞細胞分子の測定拡散速度は、同じ運動が閉じ込められていない空間で発生した場合よりも遅く見える。非常にゆっくりと拡散する分子の場合、細胞閉じ込めの効果は、境界との衝突の欠如のためにごくわずかです。このような場合、ブラウン運動の方程式に基づく解析モデルを使用して、分子変位、r、または明らかな拡散係数D*の分布を合わせることによって、拡散状態を正確に解決することができるかもしれません(ランダム拡散)11,12,13.しかし、細胞細胞分子の拡散が速い場合、実験分布は、細胞境界との拡散分子の衝突による非限定ブラウン運動のために得られたものに似なくなりました。蛍光標識分子の非限定拡散係数を正確に決定するためには、閉じ込め効果を考慮する必要があります。いくつかのアプローチは、最近、(半)解析的に5、14、15、16、またはモンテカルロシミュレーションを通じて、閉じ込め効果を説明するために開発されました。 ブラウン拡散 6,10,16,17,18,19.
ここでは、単一分子追跡に特に焦点を当てた単一分子局在顕微鏡データを収集および分析するための統合プロトコルを提供する。このプロトコルの最終目標は、内部の蛍光標識細胞質タンパク質の拡散状態を解決する(この場合は、棒状の細菌細胞)。我々の研究は、単一分子追跡のための以前のプロトコルに基づいて構築され、DNAポリメラーゼPolIは、拡散分析20によってDNA結合および非結合状態に存在することが示された。ここでは、単分子トラッキング解析を3D測定に拡張し、よりリアルな計算シミュレーションを実行して、細胞内に同時に存在する複数の拡散状態を解決・定量化します。このデータは、二重らせん点広がり関数(DHPSF)21,22を用いて撮像することにより蛍光発光器の3D位置を決定することができる自家製の3D超解像蛍光顕微鏡を用いて取得される。生の単一分子画像は、カスタム書き込みソフトウェアを使用して3D単分子ローカリゼーションを抽出して処理され、単一分子軌道に結合されます。何千もの軌道がプールされ、明らかな拡散係数の分布が生成されます。最後のステップでは、実験分布は、限られた体積でブラウン運動のモンテカルロシミュレーションを介して得られた数値生成分布に適合します。このプロトコルを適用して、生きているエルシニア・エンテロコリチカにおけるタイプ3分泌系タンパク質YscQの拡散状態を解決する。そのモジュラー性質のために、私達のプロトコルは、一般的に任意の細胞形状の単一分子または単一粒子追跡実験の任意のタイプに適用可能です。
プロトコル
1. 二重らせんポイントスプレッド関数キャリブレーション
注:本および以下のセクションで説明する画像は、Rocha et al.23に記載されているように、カスタム構築された反転蛍光顕微鏡を使用して取得される。同じ手順は、単一分子局在化および追跡顕微鏡検査2、3、4用に設計された異なる顕微鏡実装に適用可能である。この資料に記載されている画像取得およびデータ処理用のすべてのソフトウェアが利用可能です (https://github.com/GahlmannLab2014/Single-Molecule-Tracking-Analysis.git)
- 顕微鏡下で見られるガラスカバースリップにサンプルを取り付けるためのアガロースパッドの準備。
- M2Gバッファーの5mLに重量低融点アガローズ1.5-2%を加えます(4.9 mM Na2HPO 4、3.1 mM KH2PO4、7.5mM NH4Cl、0.5 mM MgSO 4、10 μM FeSO4、0.5mM CaCl2および0.2%グルコース)。すべてのアガロースが溶解するまで数秒間電子レンジ。溶液を沸騰させないようにしてください。
- アガロース溶液を数分間冷やします(2-3分)。
- ガラスカバースリップ(22 mm x 22 mm)にアガロース溶液のピペット600 μL。アガロースの上に2枚目のガラスカバースリップをそっと置きます。これは2つのガラスカバーのスリップの間に薄い(約0.5 mm)アガロースのゲルパッドを作成する。
- アガロースパッドを20分間固めるために座らせます。
- ガラスカバーのスリップをそっと切り離します。アガロースパッドは、そのうちの1つに固執します。
- かみそり刃を使用して、同じサイズの4つの正方形のセクションにアガロースパッドをカットします。各正方形のセクションは、単一のサンプルに使用できます。
- 1つのアガロースパッド上の蛍光ビーズ溶液のピペット1.5 μL。ビーズ溶液は、M2Gのストック溶液の1/100000希釈である(材料の表を参照)。
- アガロースパッドを反転させ、オゾンクリーナーで30分間洗浄したガラスカバースリップの上に置きます。洗浄時間は、カバースリップに付着する蛍光分子を排除するために選択されます。
- サンプルカバーガラスを反転蛍光顕微鏡のサンプルホルダーに取り付け、粘着テープまたはスプリングロードサンプルホルダークリップを使用して所定の位置に固定します。
- 顕微鏡の目的に浸漬油の滴を追加します。
- サンプルホルダーを顕微鏡の上に置き、所定の位置に固定します。
- グラフィカルユーザーインターフェイス(GUI)を初期化して、顕微鏡のカメラ、サンプルステージ、励起レーザーを制御します。ここでは、MATLABのカスタム記述ソフトウェアを計測器制御に使用しています(補足図1)。
- カメラソフトウェアHCImage Liveを初期化します。[カメラ コントロール]セクションの [キャプチャ]タブで、露出時間を 0.03 s に設定し、[ライブ]をクリックしてカメラのライブ フィードを開始します。
- GUIインターフェイスのOpen 514 nmレーザーをクリックしてレーザーをオンにすると、パッド上の蛍光ビーズを励起し、ライブストリームモード(ディスクに保存されたデータがない)を使用してカメラ上の蛍光放射を表示します。
- GUIの「マイクロポジショニングステージ」セクションの下にある「XY-Pos」矢印をクリックして、視野の中央に少なくとも1つの蛍光ビード(FOV)を配置して、顕微鏡ステージのXとYの位置を調整します。ステップサイズは、矢印の下にあるドロップダウンボックスをクリックして変更できます。
- GUIのナノポジショニングステージセクションの下にあるZ-Pos矢印をクリックして、顕微鏡ステージのZ位置を調整します。蛍光ビーズの二重らせん点広げ関数(DHPSF)の向きを垂直に設定します。この垂直方向は、Z キャリブレーションの開始点として定義されます。ステップサイズは、矢印の下にあるドロップダウンボックスをクリックして変更できます。
- HCImage Live では、キャプチャの下 |トリガーモード、速度と登録、トリガモードを内部から外部レベルトリガーに変更します。これにより、MATLAB GUI がカメラを制御できます。
- シーケンスの下 |[設定をスキャン]をクリックすると、フレーム数の数を1200に変更します。ラベルの付いたボタンをクリックして、保存先フォルダを選択します。最後に [開始]をクリックします。フレーム数は1200に設定され、Z 位置ステップ 120 個ごとに 10 フレームを収集できます。
- Z位置の範囲(50 nm刻みで開始Z位置の上下30ステップ)をスキャンし、0.03秒の露出時間を使用して各ステップで10フレームを記録し、Z-キャリブレーションセクションの下にあるGOをクリックして自動化プロセスを開始します。GUI。
注:ステップ長、歩数、カメラの露出時間、ステップあたりのフレーム数など、キャリブレーションのパラメータもここで調整できます。約105個の光子は、0.03sの露光時間を使用して、単一フレーム内の蛍光ビードから取得することができ、その結果、約1nmのx,y,z局在精度が得られます。 - GUI で[515 nm レーザーを閉じる]をクリックして、レーザー イルミネーションをオフにします。HCImage Live では、キャプチャの下 |トリガーモード、速度と登録、トリガモードを内部に戻します。シーケンスの下 |[スキャンの設定]をクリックすると、フレーム数が 200 に変更されます。ラベルの付いたボタンをクリックして、保存先フォルダを選択します。[開始]をクリックして、0.03 秒の露出時間で 200 フレームの暗い画像を収集します。
注: 検出器に光が落ちない場合でも、各ピクセルは正の数 (ダーク オフセット値と呼ばれます) を読み取ります。暗いオフセット値は、時間の経過と同じ時間の経過と同時に変化する可能性があります。したがって、キャリブレーションごとに暗いフレームを収集する必要があります。 - イージーDHPSFソフトウェア24を使用して、ビーズのXとYの位置、および角度対Zキャリブレーション曲線を使用して、DHPSFをダブルガウスモデルに合わせます。
- MATLAB で簡単 DHPSF ソフトウェアを初期化します。[セットアップ]で、[チャネル]をGに設定し、DG モデルを使用して[フィッティング方法]をMLEに設定します。Gは、データ集録に使用される蛍光タンパク質が緑色の波長を発する緑色チャネルカメラを指す。DG モデルを使用した MLEは、倍精度浮動小数モデルによる最大尤度推定を指します。
注: ピクセルサイズと変換ゲインは、特定の光学式設定に依存するため、変更が必要になる場合があります。 - [DHPSF のキャリブレーション]セクションで、[実行]をクリックします。次のポップアップ ウィンドウで[OK]をクリックして、既定の設定を維持します。
- 手順 1.13-1.14 に保存されているイメージ スタックを選択します。次に、手順 1.15 で暗い背景を保存したイメージ スタックを選択します。最後に、手順 1.14 の間に自動的に保存された .txt ファイルを選択します。このファイルには、スキャンプロセス全体を通じてステージの Z 位置が含まれています。
- 次のポップアップウィンドウで、フルカメラチップが視野に使用されていない場合は、チップ上の開始x0とy0の位置を入力します。それ以外の場合は、入力 x0 = 1 および y0 = 1.この情報は、ビンインセクションとサブアラリーセクションの下の HCImage Live で見つけることができます。
- 次のウィンドウで、蛍光ビーズの画像の上に表示されるボックスのサイズを変更し、サイズが約 100 x 100 ピクセルになり、単一の蛍光 DHPSF 信号の上に中央に配置します。次に、ダブルクリックして続行します。
注: 選択した DHPSF は、他の DHPSF 信号から分離し、理想的には可能な限り最も明るいビーズである必要があります。 - DHPSF 信号の中央をクリックし、2 つのローブの間をクリックし、Enterを押します。次のウィンドウは、選択した DHPSF の拡大表示を示しています。クリックして、DHPSF の中心の位置をより正確に選択します。
注:プログラムは、DHPSFにフィットし、生の画像とフィットからの再構築を表示します。また、異なるZ位置のDHPSF断面に対応するZキャリブレーションからテンプレート画像を出力します。これらは、後で実験データのフィッティングに使用されます。プログラムは、各フレームで推定される X、Y、および Z の位置を出力します。整列した光学系では、Z位置が変化するにつれて、XとYはほとんど変化しない(~30nm偏差)。出力変動が 30 nm より大きい場合、撮像システムのフーリエ平面にある位相マスク (図 1)を再配置し、ステップ 1.9~1.16 を繰り返す必要があります。 - GUI の左上隅にある[保存]アイコンをクリックして、簡単に DHPSF GUI を保存します。これは、GUI の[読み込み]アイコンをクリックして後で読み込むことができます。
注:Zキャリブレーション手順は、温度変動や機械的振動のために発生した可能性のある顕微鏡の位置合わせの変化を考慮して、実験の各日に行う必要があります。
- MATLAB で簡単 DHPSF ソフトウェアを初期化します。[セットアップ]で、[チャネル]をGに設定し、DG モデルを使用して[フィッティング方法]をMLEに設定します。Gは、データ集録に使用される蛍光タンパク質が緑色の波長を発する緑色チャネルカメラを指す。DG モデルを使用した MLEは、倍精度浮動小数モデルによる最大尤度推定を指します。
2. 細菌培養製剤
- 細菌細胞増殖を支持する培養培養培養剤を調剤する。Y.エンテロコリチカの場合は、ナリジキシン酸(35μg/mL)と2,6-ジアミノピメル酸(80μg/mL)を含むBHI(脳心注入)スープの5mLを使用します。ここで、Y.エンテロコリチカ株は、蛍光タンパク質eYFP23とタグ付けされたタンパク質YscQを有するY.エンテロコリチカ株を用いる。
- 冷凍ストックまたはプレート培養物から細菌培養物で培地を接種する。
- 一晩振って28°Cで培養を成長させる。
- 飽和一晩培養量を少量(約250μL)に希釈し、新鮮な培養培養を用いて5mLに希釈する。
- 60-90分間振りながら28°Cで培養を成長させる。
- 蛍光融合タンパク質の発現を誘導する。Y.エンテロコリチカの場合、細胞を水シェーカーで37°Cにヒートショックさせ、ヨップレグロンを誘導する。
- 37°Cでさらに3時間、振って細胞をインキュベートします。
- 室温で3分間5,000 x gの培養の遠心分離機1 mL。上清を捨てます。
- M2G培中の1mLでペレットを3回洗います。
- M2G培中の約250μLでペレット菌を再停止します。
- 蛍光ビーズをフィデューシャルマーカーとして追加します。蛍光ビーズ溶液は、顕微鏡で見たときにFOVあたり1〜2ビーズが存在するので、適切に希釈された量で添加する必要があります。
- 穏やかにピペットまたは渦は、凝集細胞を分離するために懸濁液を。
- M2Gで作られた1.5-2%のアガロースパッドの懸濁液の版1.5 μL。
- アガロースパッドを反転し、オゾン洗浄顕微鏡カバースリップの上に置きます。カバースリップは、固有の蛍光の背景を減らすために30分間オゾンクリーナーに置かれるべきです。
3. データ取得
- サンプルカバーガラスを反転蛍光顕微鏡のサンプルホルダーに取り付け、粘着テープまたはスプリングロードサンプルホルダークリップを使用して所定の位置に固定します。
- 顕微鏡の目的に浸漬油の滴を追加し、顕微鏡上にサンプルホルダーを置き、所定の位置に固定します。
- グラフィカルユーザーインターフェイス(GUI)を初期化して、顕微鏡のカメラ、サンプルステージ、励起レーザーを制御します。ここでは、MATLABのカスタム書き込みソフトウェアを計測器制御に使用します。
- カメラソフトウェアHCImage Liveを初期化します。[カメラ コントロール]セクションの [キャプチャ]タブで、露出時間を 0.025 s に設定します。
- GUIのマイクロポジショニングステージセクションの下にあるXY-Pos矢印をクリックして顕微鏡ステージのXとYの位置を調整し、サンプルの周りをスキャンし、細菌細胞の適切に密集した集団を持つFOVを見つけます。
注: データスループットを最大化するには、セルが重なり合ったり触れたりすることなく、セルの密度を可能な限り高くする必要があります。FOVはまた、FIDucialマーカーとして使用される少なくとも1つの蛍光ビーズを含むべきであり、好ましくはFOVの角に配置される。 - 蛍光ビーズのDHPSFローブが垂直になるように、GUIのナノポジショニングステージセクションの下にあるZ-Pos矢印をクリックして、顕微鏡ステージのZ位置を調整します。
- シーケンスの下 |[設定をスキャン]フレーム数を 20,000 に変更します。ラベルの付いたボタンをクリックして、保存先フォルダを選択します。最後に[スタート]をクリックして、0.025秒の短い露光時間を使用して最大20,000枚のカメラフレームを収集し、514 nm19,25の高輝度励起光を使用してeYFPフォトブリンクを開始します。
注:ここでは、焦点面で〜350 W/cm2のレーザー強度が単一のEYFP分子の初期漂白およびその後のイメージングに使用される。撮像中のUV波長におけるeYFP分子の光活性化は用いられなかった。細菌細胞につき、単一分子シグナルが1個まで存在する必要があります。単分子信号の密度が最初に高すぎる場合は、データ集録を開始する前に十分な光漂白が起こるまで点灯し続ける。 - GUI で[515 nm レーザーを閉じる]をクリックして、レーザー イルミネーションをオフにします。同じ露出時間を使用して暗い画像の200フレームを収集します。
- GUIで、Thorlabs LEDの横にあるチェックボックスをオンにし、[ミラーアップを切り替える]をクリックします。これにより、蛍光経路から相コントラスト経路への経路が切り替わる。
- データ取得ソフトウェア ICキャプチャ 2.4を初期化します。これは、位相コントラスト経路のカメラを制御します。ライブ表示の開始/停止ボタンを押すと、カメラからのライブフィードが表示されます。[キャプチャ]をクリックします |[イメージを保存]を使用して、視野内のセルの位相コントラスト イメージを収集します。
- 追加の FOV については、手順 3.5~3.10 を繰り返します。ここでは、10種類のFOVで約500個の細菌細胞から取得したデータを用いて、分析に利用できる単分子軌道の数を増やすために使用される。
注意: アガロースパッドに長時間取り付けられたセルは、新しく取り付けられたセルとは動作が異なる場合があります。さらに、アガロースパッドは、しばらくすると完全性を失い、データ品質に悪影響を及ぼす可能性があります。典型的には、サンプルスライドごとに最高3FOV(顕微鏡上で〜30分)が使用されます。
4. データ処理
注:Easy-DHPSFソフトウェア24の修正版は、単一分子のローカリゼーションを抽出するために生のカメラフレームの分析のためにMATLABで使用されます。容易なDHPSFは単一分子イメージ投射のDHPSFの局在化に合うためにとりわけ使用される。カスタム変更は、現代のsCMOSカメラ26のピクセル依存ノイズ特性を説明する最大尤度推定(MLE)ベースのフィッティングルーチンを実装するために行われました。また、HCImage Live プログラム (.dcimg) からのイメージ ファイルタイプ出力を受け入れるように変更されました。ソフトウェアと個々の手順の詳細については、Lew etal. 24を参照してください。
- MATLAB で簡単 DHPSF GUI を初期化します (補足図 2)。ステップ 1.16.8 に保存されたファイルを読み込みます。
- ステップ 1.16.7 で出力される 7 つのテンプレートのそれぞれのしきい値の決定
- [SM のキャリブレーション]セクションで、[実行]をクリックします。次の 2 つのポップアップ ウィンドウで[OK]をクリックして、既定の設定を維持します。
- プロンプトが表示された場合は、最初の FOV からのデータを含むイメージ スタックを開きます。
- テンプレートに一致するフレームの小さな範囲を選択します。通常、フレーム 1001 から 2000 は、最初の数百フレームで高密度の重なり合う信号を避けるために使用されます。次のポップアップ ウィンドウで[OK]をクリックして、既定の設定を維持します。次のウィンドウでシーケンス ログ ファイルの入力を求められたときに[キャンセル]をクリックします。
- 手順 3.8 で保存された暗い背景を使用してイメージ スタックを開きます。次のポップアップ ウィンドウで[OK]をクリックすると、背景推定のパラメータが既定のままになります。デフォルトでは、現在のフレームの周囲に 100 の後続のカメラ フレームをカバーする中央値フィルター27を使用して背景を推定します。
- 次のウィンドウで、画像上に重なっているボックスのサイズを変更して完全な FOV をカバーし、ダブルクリックして続行します。
- 画像上の複数のポイントをクリックして、ポリゴンを作成して、対象領域を定義します。対象領域には、画像内の蛍光ビーズ (非常に明るいオブジェクト) がポリゴン内に置かれていないことを確認しながら、可能な限り多くの視野を含め、ダブルクリックして続行する必要があります。
注:ソフトウェアは、画像にテンプレートを一致させようとし、可能な一致が丸で表示されます。 - ソフトウェアが停止すると、見つかったテンプレートの多くの画像が一致し、対応するしきい値が定義済みのフォルダに表示されます。しきい値が大きいほど、より良い一致に対応します。テンプレート番号ごとに、一致例を調べて、DHPSF のイメージを示す最小しきい値を決定します。[簡単 DHPSF GUI] の[SM 識別の調整]セクションの下にある 7 つのテンプレートごとにこれらのしきい値を入力します。
注: プログラムは自動的にしきい値を選択して入力しようとしますが、これらは信頼性が低く、手動でチェックする必要があります。しきい値は、真の単分子シグナルがほとんど見逃せないような値が選択されますが、フィッティングの偽陽性候補の数は計算上管理可能なままです。 - 左上隅の[保存]アイコンをクリックして、簡単にDHPSF GUIを再度保存します。
- FOVに蛍光ビーズを取り付け、フィデューシャルマーカーとして使用する
- イージー DHPSF GUI の[追跡受託者]セクションの下にある [実行]をクリックします。次の 2 つのポップアップ ウィンドウで[OK]をクリックして、既定の設定を維持します。
- 画像上に重なったボックスをドラッグし、蛍光ビーズからDHPSF信号の上に中央にドラッグし、ダブルクリックします。
- DHPSF 信号の中央をクリックし、2 つのローブの中間点で Enter キーを押します。次のウィンドウでシーケンス ログ ファイルの入力を求められたときに[キャンセル]をクリックします。
注:ソフトウェアは、すべてのカメラフレームにDHPSFにフィットし、生の画像と再構築された画像を表示します。ソフトウェアが終了すると、画像取得の期間中に蛍光ビーズのX、Y、Z位置で数値が出力されます。 - [受託者を使用]の横にあるチェックボックスをオンにし、左上隅にある [保存] アイコンをクリックして、Easy-DHPSF GUI をもう一度保存します。
- ステップ 4.2 で取得したテンプレートしきい値を使用して、すべてのカメラ フレームのすべてのローカリゼーションを検索して適合させます。
- [簡単に DHPSF GUI] の[DHPSF SM のローカライズ]セクションで、[実行]をクリックします。次のポップアップ ウィンドウで[OK]をクリックして、既定の設定を維持します。次のウィンドウでシーケンス ログ ファイルの入力を求められたときに[キャンセル]をクリックします。
注: 一致の品質がユーザー定義のしきい値を超えている場合、ソフトウェアはダブルガウスモデルを使用して DHPSF を検索して適合します。テンプレートの一致の周りに円を持つ生の画像と、再構築された DHPSF フィットのイメージが表示されます。 - 左上隅の[保存]アイコンをクリックして、簡単にDHPSF GUIを再度保存します。
- [簡単に DHPSF GUI] の[DHPSF SM のローカライズ]セクションで、[実行]をクリックします。次のポップアップ ウィンドウで[OK]をクリックして、既定の設定を維持します。次のウィンドウでシーケンス ログ ファイルの入力を求められたときに[キャンセル]をクリックします。
- 単一分子のローカリゼーションを表示し、不要または信頼性の低いローカリゼーションを除外する
- [出力 DHPSF SM ローカリゼーション]セクションで、[出力をフィルター処理] をクリックします。
- 次の 3 つのウィンドウで[OK]をクリックして、時間の経過に伴う図 X、Y、および Z の位置の補間を実行します。ほとんどの場合、既定のオプションで十分です。黒の補間線が赤い位置線の妥当な補間を反映していない場合は、ポップアップ ウィンドウの補間パラメータを変更します。
注: 補間された線は、単一分子のローカリゼーションを補正するステージドリフトに使用されます。 - プロンプトが表示されたときに分析される FOV の対応する位相コントラスト イメージを開きます。次の 2 つのポップアップ ウィンドウで[OK]をクリックして、既定の設定を維持します。
- 次の 2 つのポップアップ ウィンドウで、フィルタ値を変更して、より厳密なまたはより寛大な単一分子のローカリゼーション要件を許可し、[OK]をクリックします。
- 表示されるウィンドウで、画像上に重なっているボックスをドラッグまたはサイズ変更して、目的の領域を表示し、ダブルクリックして続行します。
- 単一分子局在化の3D再構成が表示されます。図形ツールを使用して、再構成 (回転、ズームなど) を操作します。[続行]をクリックして、別のパラメータ セットを使用して再印刷を行う場合は、[もう一度] をクリックします。結果が満足できる場合は、[いいえ]をクリックします。そうでない場合は、[はい]をクリックします。
注: 不十分な結果の一般的な理由として、位相コントラストイメージがローカリゼーション データと正しくオーバーレイされていないか、初期テンプレートのしきい値が低すぎて、多くの誤検知ローカリゼーションが作成されたりします。[はい]が選択されている場合、ソフトウェアはステップ 4.5.4 に戻り、新しいパラメータ値を定義できます。[いいえ]が選択されている場合は、結果が保存されます。
5. データ後処理
- MATLABでカスタム作成されたソフトウェアを使用して、蛍光顕微鏡下で画像化された細胞を含む領域だけが残るように位相コントラスト画像をトリミングします。位相コントラスト画像は蛍光画像よりもはるかに大きな領域をカバーするため、このステップが必要です。イメージをトリミングすると、次の手順が簡略化されます。
- OUFTI28ソフトウェアで位相コントラスト画像を処理して個々の細胞をセグメント化する(補足図3)
- MATLAB で OUFTI を初期化します。[ロード フェーズ]をクリックして、前の手順からトリミングされた位相コントラスト イメージを読み込みます。
- [ファイル]をクリックして、出力ファイルの保存場所を選択して名前を付けます。
- [検出と分析]見出しの下にある[独立したフレーム]を選択します。
- [パラメータの読み込み]をクリックして、セル検出用のパラメータを読み込みます。パラメータの例としては、許容可能なセル領域、セル幅、セル分割しきい値などがあります。
注: これらのパラメータはすべて、使用する特定のセル サイズと画質のパフォーマンスを最大化するように調整する必要があります。重要なのは、セルのアウトラインを正確に測定できるように、アルゴリズムパラメータをサブピクセルに設定する必要があります。 - セルセグメンテーションを開始するには、このフレームをクリックします。プロセスが完了すると、セルのアウトラインが位相コントラストイメージの上に表示されます。
- [手動]セクションの下のコントロールを使用して、セルを分割、セルの追加、またはセルのアウトラインをリファインして、自動化されたプロセス中に不正確にセグメント化されたセルのアウトラインを取得します。
- [分析を保存]をクリックしてセルのアウトラインを出力します。
- MATLAB のカスタム作成ソフトウェアを使用して、前の手順で取得したアウトラインを単一分子のローカリゼーションに正確にオーバーレイします。次のサブステップでは、ソフトウェアの手順について詳しく説明します。
- 前のステップで生成された単一分子ローカリゼーションデータと細胞アウトラインの両方で、同じ5つの細胞のセル極の位置を大まかに推定してクリックすることにより、ポップアップウィンドウで5つのコントロールポイントペアを手動で選択します。細胞極の位置は、1つの細胞に属する単一分子局在化の周りに対凸体を描き、最も高い曲率の点を選択することによって、おおよそ推定することができる(補足図4)。
- MATLABのcp2tform関数を使用して2Dアフィン変換関数を生成し、それを使用して細胞の輪郭と単一分子のローカリゼーションの大まかなオーバーレイを生成します。
- 10 未満のローカリゼーションを含むセルを削除し、視野の外側に部分的に配置されているセルを削除します。そのセルのアウトラインの内側をクリックして、ポップアップウィンドウ内の他の不要なセルを手動で削除します (補足図 5)。
- 残りのすべての細胞アウトラインと単一分子のローカリゼーションに対して質量の中心を使用して、より大きな制御点ペアを形成し、2D変換関数を再計算し、それを使用してセルアウトラインとセルのアウトラインの最終的なオーバーレイを生成します。単一分子のローカリゼーション。
- セルの境界内にあるローカリゼーションをそのセルのアウトラインに割り当てます。セルのアウトライン内にないローカリゼーションを破棄します (図 2a、補足図 6)。
6. 単分子トラッキング
注: MATLAB のカスタム作成ソフトウェアを使用して、次のセクションを完了します。このセクションでは、ソフトウェアが実行する手順について説明します。
- 同じセルと後続のカメラ フレームに割り当てられたローカリゼーションの場合は、ローカリゼーション間のユークリッド距離を計算します。ローカリゼーション間の距離が 2.5 μm のしきい値を下回っている場合は、同じ単一分子軌道に割り当ててローカリゼーションをリンクします。
注: 空間的および時間的しきい値の要件を満たす隣接セルのローカリゼーションがリンクされていないように、単一のセル内のローカリゼーションのみを考慮することが重要です。2.5 μmの閾値は、非常に速い分子(30 μm2/s)が露光時間(0.025s)プラス20%のバッファの長さで移動できる最大距離として選択されました。 - 4 つのローカリゼーションより短い軌道を破棄します。2つ以上のローカリゼーション(すなわち、2つ以上の蛍光エミッタ)が同時にセル内に存在する場合は、関連する軌道を破棄します。トラックの長さの最小値を 4 つのローカリゼーションに設定すると、複数の距離測定を平均化して、拡散係数のより正確な推定値を得られます。
- 特定の軌道の平均平方変位(MSD)を次のように計算します。
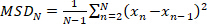 (1)
(1)
ここで、Nは軌道内のローカリゼーションの総数であり、x nは時点nにおける分子の位置である。 - 見かけの拡散係数D* を計算します。
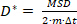 (2)
(2)
ここでm =2または3は測定の次元であり、Δtはカメラの露出時間である。
注:典型的な実験では、合計で約5,000~100,000の軌道が生成され、その数が多い明らかな拡散係数分布が得られます。
7. 限られた容積におけるブラウン運動のモンテカルロシミュレーション
注:0.05~20 μm2/sの範囲の64の値を入力パラメータ(ソフトウェア)として使用して、円筒体積に限定されたブラウンモーションのモンテカルロシミュレーションを実行することにより、シミュレートされた明らかな拡散係数分布のライブラリを作成します。)は、要求に応じて著者から入手可能です)。この範囲は、細菌中の蛍光(融合)タンパク質の以前に推定された拡散係数の範囲をカバーするために選択されました。64の拡散係数は、この範囲を十分にサンプリングするために使用される。このセクションでは、MATLAB のカスタム作成ソフトウェアを使用して実行し、ソフトウェアが自動的に実行する手順について説明します。ここで使用される棒状Y.エンテロコリチカ細胞は、長さl= 5 μmおよび直径d=0.8 μmの円筒体積によって近似される。
- 円筒体積のランダムな位置で個々の軌道を開始し、100 nsの時間間隔を使用してランダム(すなわち、ブラウン)拡散ステップをシミュレートします(時間間隔は、十分に可能にするために、カメラの露出時間よりもはるかに短くする必要があります)カメラフレームのコース上の位置のサンプリング)。Eqn. 2 の再配置によって対応するガウス分布関数からの入力 D の各変位ステップをサンプリングし、前の位置に追加します。
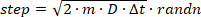 (3)
(3)
ここで、MATLAB のrandn関数は正規分布から乱数をサンプルします。ステップによって分子が円柱の体積の外側に変位する場合は、分子を円柱内にランダムな角度で反射します。 - 時間間隔ごとに、シミュレートされたエミッタの瞬間x、Y、z位置に対応する DHPSF イメージを生成します。
- 実験条件に合わせて、ローカリゼーションあたり 1,000 個のフォトンを含む画像をシミュレートし、レーザーの背景はピクセルあたり 13 個、ポアソン ノイズをシミュレートします。さらに、実験カメラのキャリブレーション測定と一致して、ピクセルあたり約50フォトンの暗いオフセットとガウス読み取りノイズ(σ~1.5フォトン)を追加します。最後に、sCMOSカメラの実験的に測定されたピクセル依存ゲインによって画像を乗算し、検出器数の単位で画像を得る。
注:これらの操作の後、最終的な画像の信号対雑音比は~2です。 - 実験データ取得時に使用される露光時間中にシミュレートされた50枚のDHPSF画像を合計することにより、分子の位置の変化を反映するモーションブラー画像を生成します。計算コストを制限するために、画像を生成するために定期的にサンプリングされた位置は 50 つだけ選択されました (サンプリング間隔 100 ns で 0.025 s 露光時間中にサンプリングされた 250,000 の位置すべてではなく)。
注: シミュレートされた軌道の長さ (フレーム数) は、実験軌道の平均長さと一致する必要があります。この場合、軌道あたりのフレーム数は 6 です。
- 実験条件に合わせて、ローカリゼーションあたり 1,000 個のフォトンを含む画像をシミュレートし、レーザーの背景はピクセルあたり 13 個、ポアソン ノイズをシミュレートします。さらに、実験カメラのキャリブレーション測定と一致して、ピクセルあたり約50フォトンの暗いオフセットとガウス読み取りノイズ(σ~1.5フォトン)を追加します。最後に、sCMOSカメラの実験的に測定されたピクセル依存ゲインによって画像を乗算し、検出器数の単位で画像を得る。
- 64のシミュレートされた入力拡散係数のそれぞれに対して5,000の軌道を生成します。
- 「データ処理」セクションで説明されているように、シミュレートされたカメラ フレームを分析します。
- 「単一分子トラッキング」セクションで説明されているように、ローカリゼーションを軌道にリンクします。
- 順序 25 の B スプライン補間を使用して、シミュレートされた分布ごとに累積分布関数 (CDF) を補間します。補間された分布は、任意のポイントで照会できるように必要です。
- MATLAB の散布されたインターポラント関数を使用して、D軸に沿って得られる B スプライン 曲線を補間します(Dは、シミュレートされたエミッタのモーションを支配する真の非限定拡散係数です)。自然'補間方法)。これにより、0.05~20 μm 2 /sの範囲で真の拡散係数値に対応する明らかな拡散係数分布を照会できる連続的な2D関数が提供されます。
8. 実験的明らかな拡散係数分布フィッティング
注:前のセクションで生成されたシミュレートされた分布の線形組み合わせを使用して、見かけの拡散係数の分布を実験的に測定しました(限られた体積のブラウン運動のモンテカルロシミュレーション)。このセクションでは、MATLAB のカスタム作成ソフトウェアを使用して実行し、ソフトウェアが自動的に実行する手順について説明します。アプリケーションの詳細と例については、Rocha et al.29を参照してください。
- セクション7で作成されたライブラリからシミュレートされたCdFの定期的にサンプリングされた配列を使用して、実験CDFの拘束された線形最小二乗適合を実行します(MATLABのlsqlin関数を使用)。このステップの出力は、実験分布における一般的な拡散状態の拡散係数と母集団分数を含むパラメータベクトルです。
- 相対母率に基づくウェイト平均化により、互いに 20% 以内の拡散係数値を持つ拡散状態を 1 つの拡散状態に結合します。これは開始パラメータ ベクトルです。
注: モデルの複雑さを軽減するために、拡散係数が 0.5 μm2/s 未満の拡散状態は、次のすべての手順で一定に保つことができます。 - 単一の拡散状態からユーザー定義の最大数の状態に至るまで、異なる数の拡散状態を持つ試行的なフィッティング パラメータ ベクトルの配列を作成します。
- 開始パラメータ ベクトルを使用して、加重平均を使用して隣接する拡散状態を結合し、拡散状態を 2 つの状態に分割し、母集団の分数を同じ値と拡散係数を元の値の上下 20% に分割します。すべての状態の組み合わせと分割の可能性についても、この手順を繰り返します。
- 各試用フィッティングパラメータベクトルを使用して、データの5つの別々のサブセットの非線形最小二乗継手を初期化します(MATLABのfmincon関数を使用)。適合値と残りのサブセットに対応する分布 (データクロス検証) の間の残りの平方和を見つけることで、適合の品質を決定します。
- フィット感の全体的な品質として、各トライアルベクトルの5つの別々の継手の平均残量平方和を使用し、拡散状態の数ごとに最適な適合品質でトライアルベクトルを決定します。
- 追加の状態を追加してもフィットの品質が少なくとも 5% 向上しないトライアル ベクトルを特定して、最適な状態数を決定します。
- このトライアルベクトルを使用して、完全なデータ・セットの非線形最小二乗継を初期化します。
- ブートストラップと同じトライアルベクトルでの再フィットによってデータをリサンプリングすることにより、個々のパラメータの誤差を推定します。
結果
ここで説明する実験条件(20,000フレーム、軌道長最小4局在)および蛍光標識融合タンパク質の発現レベルに応じて、約200〜3,000の局在化が10〜150を生み出す軌道は、セルごとに生成することができます (図 2a, b)。見かけの拡散係数の十分にサンプリングされた分布を生成するには、多数の軌道が必要です。ここで収集されるFOVのサイズ?...
ディスカッション
提示されたプロトコルの正常な適用のための重要な要因は、単一分子信号が互いに十分に分離されていることを確認することです(すなわち、それらは空間と時間内にまばらである必要があります(補足Mov. 1)。細胞内に複数の蛍光分子が同時に存在する場合、局在化が別の分子の軌道に誤って割り当てられる可能性があります。これをリンク問題30と呼ぶ。タンパ...
開示事項
著者は何も開示していない。
謝辞
私たちは、アレシア・アチモビッチとティン・ヤンに原稿を批判的に読んでくれたことに感謝します。バージニア大学の先端研究コンピューティングサービスグループのシニアスタッフサイエンティストであるエド・ホール氏に、この研究で使用される最適化ルーチンの設定を支援してくださったことに感謝します。この研究のための資金は、バージニア大学によって提供されました.
資料
| Name | Company | Catalog Number | Comments |
| 2,6-diaminopimelic acid | Chem Impex International | 5411 | Necessary for growth of Y. enterocolitica cells used. |
| 4f lenses | Thorlabs | AC508-080-A | f = 80mm, 2" |
| 514 nm laser | Coherent | Genesis MX514 MTM | Use for fluorescence excitation |
| agarose | Inivtrogen | 16520100 | Used to make gel pads to mount liquid bacterial sample on microscope. |
| ammonium chloride | Sigma Aldrich | A9434 | M2G ingredient. |
| bandpass filter | Chroma | ET510/bp | Excitation pathway. |
| Brain Heart Infusion | Sigma Aldrich | 53286 | Growth media for Y. enterocolitica. |
| calcium chloride | Sigma Aldrich | 223506 | M2G ingredient. |
| camera | Imaging Source | DMK 23UP031 | Camera for phase contrast imaging. |
| dielectric phase mask | Double Helix, LLC | N/A | Produces DHPSF signal. |
| disodium phosphate | Sigma Aldrich | 795410 | M2G ingredient. |
| ethylenediaminetetraacetic acid | Fisher Scientific | S311-100 | Chelates Ca2+. Induces secretion in the T3SS. |
| flip mirror | Newport | 8892-K | Allows for switching between fluorescence and phase contrast pathways. |
| fluospheres | Invitrogen | F8792 | Fluorescent beads. 540/560 exication and emission wavelengths. 40 nm diameter. |
| glass cover slip | VWR | 16004-302 | #1.5, 22mmx22mm |
| glucose | Chem Impex International | 811 | M2G ingredient. |
| immersion oil | Olympus | Z-81025 | Placed on objective lens. |
| iron(II) sulfate | Sigma Aldrich | F0518 | M2G ingredient. |
| long pass filter | Semrock | LP02-514RU-25 | Emission pathway. |
| magnesium sulfate | Fisher Scientific | S25414A | M2G ingredient. |
| microscope platform | Mad City Labs | custom | Platform for inverted microscope. |
| nalidixic acid | Sigma Aldrich | N4382 | Y. enterocolitica cells used are resistant to nalidixic acid. |
| objective lens | Olympus | 1-U2B991 | 60X, 1.4 NA |
| Ozone cleaner | Novascan | PSD-UV4 | Used to eliminate background fluorescence on glass cover slips. |
| potassium phosphate | Sigma Aldrich | 795488 | M2G ingredient. |
| Red LED | Thorlabs | M625L3 | Illuminates sample for phase contrast imaging. 625nm. |
| sCMOS camera | Hamamatsu | ORCA-Flash 4.0 V2 | Camera for fluorescence imaging. |
| short pass filter | Chroma | ET700SP-2P8 | Emission pathway. |
| Tube lens | Thorlabs | AC508-180-A | f=180 mm, 2" |
| Yersinia enterocolitica dHOPEMTasd | N/A | N/A | Strain AD4442, eYFP-YscQ |
| zero-order quarter-wave plate | Thorlabs | WPQ05M-514 | Excitation pathway. |
参考文献
- Kapanidis, A. N., Uphoff, S., Stracy, M. Understanding Protein Mobility in Bacteria by Tracking Single Molecules. Journal of Molecular Biology. , (2018).
- Betzig, E., et al. Imaging intracellular fluorescent proteins at nanometer resolution. Science. 313 (5793), 1642-1645 (2006).
- Hess, S. T., Girirajan, T. P. K., Mason, M. D. Ultra-high resolution imaging by fluorescence photoactivation localization microscopy. Biophysical Journal. 91 (11), 4258-4272 (2006).
- Rust, M. J., Bates, M., Zhuang, X. W. Sub-diffraction-limit imaging by stochastic optical reconstruction microscopy (STORM). Nature Methods. 3 (10), 793-795 (2006).
- Chen, T. Y., et al. Quantifying Multistate Cytoplasmic Molecular Diffusion in Bacterial Cells via Inverse Transform of Confined Displacement Distribution. Journal of Physical Chemistry B. 119 (45), 14451-14459 (2015).
- Mohapatra, S., Choi, H., Ge, X., Sanyal, S., Weisshaar, J. C. Spatial Distribution and Ribosome-Binding Dynamics of EF-P in Live Escherichia coli. mBio. 8 (3), (2017).
- Stracy, M., et al. Single-molecule imaging of UvrA and UvrB recruitment to DNA lesions in living Escherichia coli. Nature Communications. 7, 12568 (2016).
- Persson, F., Lindén, M., Unoson, C., Elf, J. Extracting intracellular diffusive states and transition rates from single-molecule tracking data. Nature Methods. 10 (3), 265-269 (2013).
- Bakshi, S., Choi, H., Weisshaar, J. C. The spatial biology of transcription and translation in rapidly growing Escherichia coli. Frontiers in Microbiology. 6, 636 (2015).
- Mustafi, M., Weisshaar, J. C. Simultaneous Binding of Multiple EF-Tu Copies to Translating Ribosomes in Live Escherichia coli. mBio. 9 (1), (2018).
- Michalet, X., Berglund, A. J. Optimal diffusion coefficient estimation in single-particle tracking. Physical Review E. 85 (6), (2012).
- Michalet, X. Mean square displacement analysis of single-particle trajectories with localization error: Brownian motion in an isotropic medium. Physical Review E Statistical, Nonlinear, and Soft Matter Physics. 82 (4 Pt 1), 041914 (2010).
- Backlund, M. P., Joyner, R., Moerner, W. E. Chromosomal locus tracking with proper accounting of static and dynamic errors. Physical Review E Statistical, Nonlinear, and Soft Matter Physics. 91 (6), 062716 (2015).
- Stracy, M., et al. Live-cell superresolution microscopy reveals the organization of RNA polymerase in the bacterial nucleoid. Proceedings of the National Academy of Sciences of the United States of America. 112 (32), E4390-E4399 (2015).
- Plochowietz, A., Farrell, I., Smilansky, Z., Cooperman, B. S., Kapanidis, A. N. In vivo single-RNA tracking shows that most tRNA diffuses freely in live bacteria. Nucleic Acids Research. 45 (2), 926-937 (2017).
- Koo, P. K., Mochrie, S. G. Systems-level approach to uncovering diffusive states and their transitions from single-particle trajectories. Physical Review E. 94 (5-1), 052412 (2016).
- Uphoff, S., Reyes-Lamothe, R., Garza de Leon, F., Sherratt, D. J., Kapanidis, A. N. Single-molecule DNA repair in live bacteria. Proceedings of the National Academy of Sciences of the United States of America. 110 (20), 8063-8068 (2013).
- Bakshi, S., Bratton, B. P., Weisshaar, J. C. Subdiffraction-Limit Study of Kaede Diffusion and Spatial Distribution in Live Escherichia coli. Biophysical Journal. 101 (10), 2535-2544 (2011).
- Bakshi, S., Siryaporn, A., Goulian, M., Weisshaar, J. C. Superresolution imaging of ribosomes and RNA polymerase in live Escherichia coli cells. Molecular Microbiology. 85 (1), 21-38 (2012).
- Uphoff, S., Sherratt, D. J., Kapanidis, A. N. Visualizing Protein-DNA Interactions in Live Bacterial Cells Using Photoactivated Single-molecule Tracking. JoVE. (85), e51177 (2014).
- Pavani, S. R. P., Piestun, R. Three dimensional tracking of fluorescent microparticles using a photon-limited double-helix response system. Optics Express. 16 (26), 22048-22057 (2008).
- Pavani, S. R. P., et al. Three-dimensional, single-molecule fluorescence imaging beyond the diffraction limit by using a double-helix point spread function. Proceedings of the National Academy of Sciences of the United States of America. 106 (9), 2995-2999 (2009).
- Rocha, J. M., et al. Single-molecule tracking in live Yersinia enterocolitica reveals distinct cytosolic complexes of injectisome subunits. Integrative Biology. 10 (9), 502-515 (2018).
- Lew, M. D., von Diezmann, A. R. S., Moerner, W. E. Easy-DHPSF open-source software for three-dimensional localization of single molecules with precision beyond the optical diffraction limit. Protocol Exchange. , (2013).
- Biteen, J. S., et al. Super-resolution imaging in live Caulobacter crescentus cells using photoswitchable EYFP. Nature Methods. 5 (11), 947-949 (2008).
- Huang, F., et al. Video-rate nanoscopy using sCMOS camera-specific single-molecule localization algorithms. Nature Methods. 10 (7), 653-658 (2013).
- Hoogendoorn, E., et al. The fidelity of stochastic single-molecule super-resolution reconstructions critically depends upon robust background estimation. Scientific Reports. 4, 3854 (2014).
- Paintdakhi, A., et al. Oufti: An integrated software package for high-accuracy, high-throughput quantitative microscopy analysis. Molecular microbiology. 99 (4), 767-777 (2016).
- Rocha, J. M., Corbitt, J., Yan, T., Richardson, C., Gahlmann, A. Resolving Cytosolic Diffusive States in Bacteria by Single-Molecule Tracking. bioRxiv. , 483321 (2018).
- Lee, A., Tsekouras, K., Calderon, C., Bustamante, C., Presse, S. Unraveling the Thousand Word Picture: An Introduction to Super-Resolution Data Analysis. Chemical Reviews. 117 (11), 7276-7330 (2017).
- Los, G. V., et al. HaloTag: A Novel Protein Labeling Technology for Cell Imaging and Protein Analysis. ACS Chemical Biology. , (2008).
- Gautier, A., et al. An Engineered Protein Tag for Multiprotein Labeling in Living Cells. Chemistry & Biology. 15 (2), 128-136 (2008).
- Bisson-Filho, A. W., et al. Treadmilling by FtsZ filaments drives peptidoglycan synthesis and bacterial cell division. Science. 355 (6326), 739-743 (2017).
- Douglass, K. M., Sieben, C., Archetti, A., Lambert, A., Manley, S. Super-resolution imaging of multiple cells by optimised flat-field epi-illumination. Nature Photonics. 10 (11), 705-708 (2016).
- Zhao, Z., Xin, B., Li, L., Huang, Z. L. High-power homogeneous illumination for super-resolution localization microscopy with large field-of-view. Optics Express. 25 (12), 13382-13395 (2017).
- Yan, T., Richardson, C. J., Zhang, M., Gahlmann, A. Computational Correction of Spatially-Variant Optical Aberrations in 3D Single Molecule Localization Microscopy. bioRxiv. , 504712 (2018).
- Gahlmann, A., Moerner, W. E. Exploring bacterial cell biology with single-molecule tracking and super-resolution imaging. Nature Reviews Microbiology. 12 (1), 9-22 (2014).
- Liu, Z., Lavis, L. D., Betzig, E. Imaging live-cell dynamics and structure at the single-molecule level. Mol Cell. 58 (4), 644-659 (2015).
- Berglund, A. J. Statistics of camera-based single-particle tracking. Physical Review E. 82 (1), 011917 (2010).
- Parry, B. R., et al. The bacterial cytoplasm has glass-like properties and is fluidized by metabolic activity. Cell. 156 (1-2), 183-194 (2014).
- English, B. P., et al. Single-molecule investigations of the stringent response machinery in living bacterial cells. Proceedings of the National Academy of Sciences of the United States of America. 108 (31), E365-E373 (2011).
- Niu, L. L., Yu, J. Investigating intracellular dynamics of FtsZ cytoskeleton with photoactivation single-molecule tracking. Biophysical Journal. 95 (4), 2009-2016 (2008).
- Coquel, A. S., et al. Localization of protein aggregation in Escherichia coli is governed by diffusion and nucleoid macromolecular crowding effect. PLoS Computational Biology. 9 (4), e1003038 (2013).
- Nenninger, A., Mastroianni, G., Mullineaux, C. W. Size Dependence of Protein Diffusion in the Cytoplasm of Escherichia coli. Journal of Bacteriology. 192 (18), 4535-4540 (2010).
- Dix, J. A., Verkman, A. S. Crowding effects on diffusion in solutions and cells. Annual Review of Biophysics. 37, 247-263 (2008).
- Elliott, L. C., Barhoum, M., Harris, J. M., Bohn, P. W. Trajectory analysis of single molecules exhibiting non-brownian motion. Physical Chemistry Chemical Physics. 13 (10), 4326-4334 (2011).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved