Bu içeriği görüntülemek için JoVE aboneliği gereklidir. Oturum açın veya ücretsiz deneme sürümünü başlatın.
Method Article
Tek Moleküllü İzleme Mikroskobu - Sitosolik Moleküllerin Diffüz Durumlarını Belirlemede Bir Araç
Bu Makalede
Özet
3D tek moleküllü lokalizasyon mikroskopisi, yaşayan bakteri hücrelerinde floresan olarak etiketlenmiş proteinlerin mekansal konumlarını ve hareket yörüngelerini araştırmak için kullanılır. Burada açıklanan deneysel ve veri analizi protokolü, sitosolik proteinlerin havuzhalinde ki tek moleküllü yörüngelere dayalı yaygın diffüzif davranışlarını belirler.
Özet
Tek moleküllü lokalizasyon mikroskopisi, onlarca nanometre uzamsal ve milisaniye zamansal çözünürlükle canlı hücrelerdeki tek tek moleküllerin pozisyonunu ve hareketlerini inceler. Bu yetenekler, fizyolojik olarak ilgili ortamlarda moleküler düzeydebiyolojik fonksiyonları incelemek için tek moleküllü lokalizasyon mikroskobu ideal hale getirmektedir. Burada, ilgi çekici bir proteinin sergileneebileceği farklı difüzif durumları ayıklamak için tek moleküllü izleme verilerinin hem elde edilmesi hem de işlenmesi/analizi için entegre bir protokol gösteriyoruz. Bu bilgiler canlı hücrelerdeki moleküler karmaşık oluşumu ölçmek için kullanılabilir. Kamera tabanlı 3D tek moleküllü yerelleştirme deneyinin ayrıntılı bir açıklamasını ve tek tek moleküllerin yörüngelerini sağlayan sonraki veri işleme adımlarını salıyoruz. Bu yörüngeler daha sonra floresan etiketli moleküllerin yaygın diffusive durumları ve bu devletlerin göreceli bolluk ayıklamak için sayısal bir analiz çerçevesi kullanılarak analiz edilir. Analiz çerçevesi, rasgele hücre geometrisi ile uzaysal olarak sınırlı olan hücre içi Browndifüzyon yörüngelerinin stokakstik simülasyonlarına dayanmaktadır. Simüle edilen yörüngelere dayanarak, ham tek moleküllü görüntüler deneysel görüntülerle aynı şekilde üretilir ve analiz edilir. Bu şekilde, deneysel olarak kalibre edilmesi zor olan deneysel hassasiyet ve doğruluk sınırlamaları, analiz iş akışına açıkça dahil edilir. Yaygın difüzyon katsayısı ve yaygın diffüzif durumların göreli popülasyon fraksiyonları, simüle edilmiş dağılımların doğrusal kombinasyonları kullanılarak deneysel değerlerin dağılımları uygun hale alınarak belirlenir. Bakteriyel bir patojenin sitosolunda homo- ve hetero-oligomerik kompleksler oluşturarak farklı diffusive durumları sergileyen bir proteinin diffüzif durumlarını çözerek protokolümüzün yararını gösteriyoruz.
Giriş
Biyomoleküllerin diffüzif davranışlarının incelenmesi biyolojik işlevlerihakkında bilgi sağlar. Floresan mikroskobu tabanlı teknikler kendi doğal hücre ortamında biyomolekülleri gözlemlemek için değerli araçlar haline gelmiştir. Fotobeyazrlama (FRAP) ve floresan korelasyon spektroskopisi (FCS) 1 sonrası floresan iyileşme1 topluluk ortalama difüzif davranışlar sağlar. Tersine, tek moleküllü lokalizasyon mikroskopisi yüksek uzamsal ve zamansal çözünürlük2,3,4ile bireysel floresan etiketli moleküllerin gözlem sağlar. İlgi çekici bir protein farklı diffüzive durumlarda var olabileceğinden, moleküllerin tek tek gözlemlemesi avantajlıdır. Örneğin, escherichia coli'dekiCueR gibi transkripsiyonel regülatörün sitosolda serbestçe dağıldığında veya DNA dizisine bağlandığında ve ölçüm zaman ölçeğinde hareketsiz hale geldiğinde, kolayca ayırt edilebilir iki difüzif durum ortaya çıkar5 . Tek moleküllü izleme, bu farklı durumları doğrudan gözlemlemek için bir araç sağlar ve bunları çözmek için karmaşık analizler gerekmez. Ancak, diffusive oranları daha benzer olduğu durumlarda birden fazla diffusive durumları ve nüfus fraksiyonları çözmek için daha zor hale gelir. Örneğin, difüzyon katsayısının boyut bağımlılığı nedeniyle, bir proteinin farklı oligomerizasyon durumları farklı diffusive durumları olarak kendini gösterir6,7,8,9 , 10. Bu gibi durumlarda veri toplama, işleme ve analiz açısından entegre bir yaklaşım gerektirir.
Sitosolik moleküllerin diffüzive oranlarını etkileyen kritik bir faktör hücre sınırı tarafından hapsedilmesinin etkisidir. Bir bakteri hücresi sınırının moleküler hareketine konan kısıtlamalar, sitosolik moleküllerin ölçülen difüzyon hızının, aynı hareketin sınırlandırılmamış bir alanda meydana gelmemiş olduğundan daha yavaş görünmesine neden olur. Çok yavaş difüzyon molekülleriçin, hücresel hapsi etkisi sınır ile çarpışma eksikliği nedeniyle ihmal edilebilir. Bu gibi durumlarda, brown hareketi denklemlerine dayalı analitik modeller kullanarak moleküler yer değiştirme, rveya belirgin difüzyon katsayıları, D*dağılımlarını uygun olarak diffüz durumlarını doğru bir şekilde çözmek mümkün olabilir ( rastgele difüzyon)11,12,13. Ancak, hızlı difüzyon sitosolik moleküller için, deneysel dağılımları artık hücre sınırları ile difüzyon moleküllerin çarpışmaları nedeniyle sınırsız Brownhareketi için elde edilen benzer. Floresan etiketli moleküllerin sınırlandırılmamış difüzyon katsayılarını doğru bir şekilde belirlemek için hapsedilme etkileri göz önünde bulundurulmalıdır. Çeşitli yaklaşımlar son zamanlarda hapsi etkileri ya (yarı-) analitik 5,14,15,16 veya sayısal Monte Carlo simülasyonları aracılığıyla hesap geliştirilmiştir Browndifüzyon6,10,16,17,18,19.
Burada, tek moleküllü lokalizasyon mikroskobu verilerinin toplanması ve analiz iã§in tek moleküllü izleme ã1/4zerinde durularak entegre bir protokol saÄ lık edeÄ izdir. Protokolün nihai amacı, bu durumda, çubuk şeklindeki bakteri hücrelerinin içinde floresan etiketli sitosolik proteinlerin diffüzif durumları çözmektir. Çalışmalarımız tek moleküllü izleme için bir önceki protokol üzerine inşa, hangi bir DNA polimeraz, PolI, difüzyon analizi20ile DNA bağlı ve bağlı olmayan durumda var olduğu gösterilmiştir. Burada, tek moleküllü izleme analizini 3B ölçümlere genişletiyoruz ve hücrelerde aynı anda bulunan birden fazla diffüzive durumu çözmek ve ölçmek için daha gerçekçi hesaplamalı simülasyonlar gerçekleştiriyoruz. Veriler çift sarmal nokta-yayma fonksiyonu (DHPSF)21,22ile görüntüleme ile floresan yayıcıların 3D konumunu belirleme yeteneğine sahip bir ev yapımı 3D süper çözünürlüklü floresan mikroskop kullanılarak elde edilir. Ham tek moleküllü görüntüler, 3B tek moleküllü yerelleştirmeleri ayıklamak için özel yazılmış yazılım kullanılarak işlenir ve bunlar daha sonra tek moleküllü yörüngelerde birleştirilir. Binlerce yörünge, görünürdidifüzyon katsayıları dağılımları oluşturmak için bir araya toplandı. Son bir adımda, deneysel dağılımlar sınırlı bir hacimde Brown hareketinin Monte-Carlo simülasyonları yoluyla elde edilen sayısal olarak oluşturulan dağılımlarla uyum sağlar. Biz yaşayan Yersinia enterocoliticaTip 3 salgı sistemi protein YscQ diffüzif durumları çözmek için bu protokolü uygulamak. Modüler yapısı nedeniyle, protokolümüz genellikle rasgele hücre geometrilerinde her türlü tek moleküllü veya tek parçacık izleme deneyi için geçerlidir.
Protokol
1. Çift sarlak noktası-yayma fonksiyonu kalibrasyonu
NOT: Bu ve aşağıdaki bölümlerde açıklanan görüntüler, Rocha ve ark.23'teaçıklandığı gibi, özel olarak oluşturulmuş ters floresan mikroskobu kullanılarak elde edilir. Aynı prosedür tek moleküllü lokalizasyon ve izleme mikroskobu 2,3,4için tasarlanmış farklı mikroskop uygulamaları için geçerlidir. Bu makalede açıklanan görüntü toplama ve veri işleme için tüm yazılımlar mevcuttur (https://github.com/GahlmannLab2014/Single-Molecule-Tracking-Analysis.git).
- Mikroskop altında izlenecek cam kapak lı numunelerin montajı için agarose pedlerin hazırlanması.
- M2G tampon (4.9 mM Na2HPO4, 3.1 mM KH2PO4, 7.5 mM NH4Cl, 0.5 mM MgSO 4 , 10 μM FeSO4, 0.5 mM CaCl 2 ve 0.2% glikoz) 5 mL ağırlık düşük erime noktası agarose tarafından% 1.5-2 ekleyin. Mikrodalga birkaç saniye tüm agarose çözülene kadar. Çözeltinin kaynamasına izin vermeyin.
- Agarose çözeltisi birkaç dakika soğumaya bırakın (2-3 dk).
- Bir cam kapak kayma (22 mm x 22 mm) üzerine agarose çözeltisi Pipet 600 μL. Yavaşça agarose üstüne ikinci bir cam kapak kayma yerleştirin. Bu iki cam kapak fişleri arasında ince (~ 0,5 mm) agarose jel ped oluşturur.
- Agarose pedleri ~ 20 dakika için katılaşmak için oturalım.
- Cam kapak fişlerini yavaşça ayırın. Agarose ped onlardan birine yapışacak.
- Bir jilet kullanarak, eşit boyutta dört kare bölümlerhalinde agarose ped kesti. Her kare bölüm tek bir örnek için kullanılabilir.
- Pipet 1.5 μL floresan boncuk çözeltisi tek bir agarose ped üzerine. Boncuk çözeltisi, M2G'deki stok çözeltisinin 1/100000 seyreltilmesidir (bkz.
- 30 dakika boyunca bir ozon temizleyici temizlenmiş bir cam kapak kayma ve agarose ped ters. Temizleme süresi, kapaklara yapışan floresan molekülleri ortadan kaldırmak için seçilir.
- Örnek kapak camı ters floresan mikroskobunun numune tutucuya monte edin ve yapışkan bant veya yay yüklü numune tutucu klipsleri kullanarak yerine sabitleyin.
- Mikroskop hedefi üzerine daldırma yağı bir damla ekleyin.
- Numune tutucuyu bir mikroskoba yerleştirin ve yerine sabitedin.
- Mikroskobun kamerasını, örnek sahnesini ve uyarma lazerlerini kontrol etmek için grafik kullanıcı arabirimini (GUI) başlangıç olarak verin. Burada, MATLAB'da özel olarak yazılmış yazılımlar enstrüman kontrolü için kullanılır (Ek Şekil 1).
- Kamera yazılımı HCImage Live'ı hayata geçirin. Kamera Denetimi bölümünün altındaki Yakalama sekmesinde, kameranın canlı yayınına başlamak için Canlı'yı tıklatın.
- Pad'deki floresan boncukları heyecanlandırmak ve canlı akış modunu kullanarak kameradaki floresan emisyonunu görüntülemek için GUI arabiriminde Aç 514 nm lazeri tıklatarak lazeri açın (yani diske kaydedilmiş veri yok).
- GUI'nin 'Mikro Konumlandırma Aşaması' bölümünün altındaki 'XY-Pos' oklarını tıklayarak mikroskop aşamasının X ve Y pozisyonlarını ayarlayarak görüş alanının (FOV) merkezinde en az bir floresan boncuk yerleştirin. Adım boyutu okların altındaki açılır kutuya tıklayarak değiştirilebilir.
- GUI'nin Nano Konumlandırma Aşaması bölümünün altındaki Z-Pos oklarını tıklatarak mikroskop aşamasının Z konumunu ayarlayın. Floresan boncuğun çift saraklı nokta yayma fonksiyonunun (DHPSF) yönünü dikey olarak ayarlayın. Bu dikey yönlendirme Z kalibrasyonunda başlangıç noktası olarak tanımlanır. Adım boyutu okların altındaki açılır kutuya tıklayarak değiştirilebilir.
- HCImage Live'da, Yakalama altında | TetikLeme Modları, Hız ve Kayıt,İç Ten Dış Düzey Tetiklemeiçin tetikleme modunu değiştirin. Bu, MATLAB GUI'nin kamerayı kontrol etmesini sağlar.
- Sıraaltında | Ayarları tama,çerçeve sayısı sayısını 1200olarak değiştirin. Etiketli düğmeye tıklayarak hedef kaydet klasörünü seçin .... Son olarak Başlat'ıtıklatın. Kare sayısı 1200'e ayarlanır, böylece 120 Z konum adımlarının her biri için 10 kare toplanabilir.
- Z pozisyonlarını (50 nm'lik artışlarla başlangıç Z pozisyonunun 30 adım yukarısı ve altında) tarayın ve her adımda 0,03 s'lik pozlama süresini kullanarak 10 kare kaydedin. Z-Kalibrasyon bölümü altında GO'ya tıklayarak otomatik işlemi başlatın GUI.
NOT: Adım uzunluğu, adım sayısı, kamera pozlama süresi ve adım başına kare sayısı da dahil olmak üzere kalibrasyon parametreleri burada da ayarlanabilir. Yaklaşık 105 foton, tek bir çerçevede bir floresan boncuktan 0,03 s pozlama süreleri kullanılarak elde edilebilir ve bu da yaklaşık 1 nm x,y,z lokalizasyon hassasiyeti sağlar. - GUI'deki Close 515 nm lazer'e tıklayarak lazer aydınlatmasını kapatın. HCImage Live'da, Yakalama altında | Tetik Modları, Hız ve Kayıt,tetikleme modunu Internalolarak değiştirin. Sıraaltında | Tscan Ayarları çerçeve sayısı sayısını 200 olarak değiştirir. Etiketli düğmeye tıklayarak hedef kaydet klasörünü seçin .... 0,03'lük pozlama süresiyle 200 kare koyu renkli görüntü toplamak için Başlat'ı tıklatın.
NOT: Dedektöre Düşen ışık olmasa bile, her piksel pikseller arasında biraz farklılık gösterebilen pozitif bir sayı (koyu ofset değeri olarak adlandırılır) okur. Koyu ofset değeri zaman içinde değişebilir. Bu nedenle, her kalibrasyon için koyu çerçeveler toplamak için gereklidir. - Boncuk X ve Y pozisyonları yanı sıra bir açı vs Z kalibrasyon eğrisi elde etmek için Easy-DHPSF yazılımı24 kullanarak bir çift Gaussian modeli ile DHPSF uygun.
- MATLAB'da Easy-DHPSF yazılımLarını başlangıç olarak karşıdan ala. Kurulumaltında, Kanal'ı G'ye ayarlayın ve DG modeliyle Montaj Yöntemini MLE olarak ayarlayın. G, veri toplamada kullanılan floresan proteinin yeşil dalga boylarıyla yayıldısı nedeniyle yeşil kanal lı kameraya atıfta bulunur. DG Modeli ile MLE, Çift Gaussian modeliile Maksimum Olasılık Tahmini anlamına gelir.
NOT: Piksel boyutu ve dönüşüm kazancı belirli optik kuruluma bağlıdır ve değiştirilmesi gerekebilir. - DHPSF Kalibrasyon bölümü altında Çalıştır'ıtıklatın. Varsayılan ayarları tutmak için aşağıdaki açılır pencerede Tamam'ı tıklatın.
- 1.13-1.14 adımlarında kaydedilen görüntü yığınını seçin. Ardından, adım 1.15'te kaydedilmiş koyu arka planlı görüntü yığınını seçin. Son olarak, adım 1.14 sırasında otomatik olarak kaydedilen .txt dosyasını seçin. Bu dosya, tarama işlemi boyunca sahne nin Z konumunu içerir.
- Bir sonraki açılır pencerede, tam kamera çipi görüş alanı için kullanılmamışsa, fiş üzerindeki başlangıç x0 ve y0 pozisyonlarını girdi. Aksi takdirde, x0 = 1 ve y0 = 1.Bu bilgileri Binning ve SubArrary bölümü altında HCImage Live'da bulabilirsiniz.
- Aşağıdaki pencerede, floresan boncukların görüntüsü üzerinde görünen kutuyu yeniden boyutlandırın ve yeniden konumlandırın, böylece yaklaşık 100 x 100 piksel boyutunda dır ve tek bir floresan DHPSF sinyali üzerinde ortalanır. Ardından, devam etmek için çift tıklatın.
NOT: Seçilen DHPSF diğer DHPSF sinyallerinden izole edilmeli ve ideal olarak mümkün olan en parlak boncuk olmalıdır. - DHPSF sinyalinin ortasına tıklayın, iki lob arasında, sonra Entertuşuna basın. Aşağıdaki pencere, seçilen DHPSF'nin yakınlaştırılmış görünümünü gösterir. Daha doğrusu tıklayarak DHPSF merkezinin konumunu seçin.
NOT: Program daha sonra DHPSF uyacak ve uygun gelen ham görüntü ve rekonstrüksiyon görüntüler. Ayrıca, farklı Z konumlarında bir DHPSF kesitine karşılık gelen Z kalibrasyonundan şablon görüntüler çıkaracaktır. Bunlar daha sonra deneysel verilerin montajı için kullanılacaktır. Program, her karede tahmin edilen X,Y ve Z konumunu çıkaracaktır. İyi hizalanmış bir optik sistemde, Z konumu değiştikçe X ve Y çok az (~30 nm sapma) değiştirmelidir. Çıkış değişimi 30 nm'den büyükse, görüntüleme sisteminin Fourier düzleminde bulunan faz maskesi(Şekil 1),yeniden hizalanmalı ve 1.9-1.16 adımları tekrarlanmalıdır. - GUI'nin sol üst köşesindeki Kaydet simgesine tıklayarak Easy-DHPSF GUI'yi kaydedin. Bu daha sonra GUI'deki Yük simgesine tıklayarak yüklenebilir.
NOT: Sıcaklık dalgalanmaları veya mekanik titreşimler nedeniyle oluşmuş olabilecek mikroskoptaki hizalama değişikliklerini hesaba katmak için bir deneyin her gününde Z kalibrasyon işlemi yapılmalıdır.
- MATLAB'da Easy-DHPSF yazılımLarını başlangıç olarak karşıdan ala. Kurulumaltında, Kanal'ı G'ye ayarlayın ve DG modeliyle Montaj Yöntemini MLE olarak ayarlayın. G, veri toplamada kullanılan floresan proteinin yeşil dalga boylarıyla yayıldısı nedeniyle yeşil kanal lı kameraya atıfta bulunur. DG Modeli ile MLE, Çift Gaussian modeliile Maksimum Olasılık Tahmini anlamına gelir.
2. Bakteri kültürü hazırlama
- Bakteri hücresi büyümesini destekleyen kültür ortamını hazırlayın. Y. enterokoliticaiçin, nalidixic asit (35 g/mL) ve 2,6-diaminopimelik asit (80 g/mL) içeren 5 mL BHI (Beyin Kalp İnfüzyonu) et suyu kullanın. Burada, bir Y. enterokolitika suşu floresan protein eYFP23ile etiketlenmiş protein YscQ vardır kullanılır.
- Dondurucu stoklarında veya plaka kültürlerinden bakteri kültürleriyle ortam aşılayın.
- Bir gecede sallayarak 28 °C'de kültürü büyütün.
- Taze kültür ortamı nı kullanarak az miktarda (~250 μL) doymuş gece kültürünü 5 mL'ye seyreltin.
- 60-90 dakika sallayarak 28 °C'de kültürü büyütün.
- Floresan füzyon proteininin ekspresyonu. Y. enterocoliticaiçin , ısı şok hücreleri 37 °C bir su çalkalayıcı yop regulon indüklemek için.
- Hücreleri 37 °C'de 37 °C'de sallayarak ek bir 3 saat için kuluçkaya yatırın.
- Oda sıcaklığında 3 dk için 5.000 x g kültür santrifüj 1 mL. Supernatant atın.
- Peleti 1 mL M2G ortam ile 3 kez yıkayın.
- M2G Media~250 μL'lik peletli bakterileri yeniden askıya alın.
- Fiducial belirteçler olarak floresan boncuk ekleyin. Floresan boncuk çözeltisi uygun seyreltilmiş miktarlarda eklenmelidir, böylece mikroskopta bakıldığında FOV başına sadece 1-2 boncuk vardır.
- Yavaşça pipet veya girdap toplu hücreleri ayırmak için süspansiyon.
- Plaka 1.5 μL süspansiyon 1.5-2% agarose ped M2G ile yapılan.
- Agarose pedini ters çevirin ve ozonla temizlenmiş mikroskop kapağı nın üzerine yerleştirin. Kapak fişi herhangi bir doğal floresan arka plan azaltmak için 30 dakika için bir ozon temizleyici yerleştirilmelidir.
3. Veri toplama
- Örnek kapak camı ters floresan mikroskobunun numune tutucuya monte edin ve yapışkan bant veya yay yüklü numune tutucu klipsleri kullanarak yerine sabitleyin.
- Mikroskop hedefi üzerine daldırma yağı bir damla ekleyin, sonra mikroskop üzerine örnek tutucu yerleştirin ve yerine güvenli.
- Mikroskobun kamerasını, örnek sahnesini ve uyarma lazerlerini kontrol etmek için grafik kullanıcı arabirimini (GUI) başlangıç olarak verin. Burada, MATLAB'da özel olarak yazılmış yazılımlar enstrüman kontrolü için kullanılmaktadır.
- Kamera yazılımı HCImage Live'ı hayata geçirin. Kamera Denetimi bölümünün altındaki Yakalama sekmesinde, kameranın canlı yayınına başlamak için Canlı'yı tıklatın.
- Örnek etrafında tatmak ve bakteri hücrelerinin uygun yoğun nüfusa sahip bir FOV bulmak için GUI Mikro Konumlandırma Aşaması bölümü altında XY-Pos okları tıklayarak mikroskop aşamasıx ve Y pozisyonlarını ayarlayın.
NOT: Veri veri kullanımını en üst düzeye çıkarmak için, hücreler çakışma veya dokunmadan mümkün olduğunca yoğun olmalıdır. FOV da en az 1 floresan boncuk bir fiducial marker olarak kullanılmak üzere içermelidir, tercihen FOV bir köşesinde konumlandırılmış. - Floresan boncuk DHPSF loblar dikey böylece GUI Nano-Konumlandırma Sahne bölümü altında Z-Pos okları tıklayarak mikroskop aşamasının Z konumunu ayarlayın.
- Sıraaltında | Ayarları tama,kare sayısı sayısını 20.000 olarak değiştirin. Etiketli düğmeye tıklayarak hedef kaydet klasörünü seçin .... Son olarak 0,025 s. eYFP photoblinking kısa pozlama süresi kullanarak 20.000 kamera kareleri kadar toplamak için Başlat'ı tıklatın 514 nm19,25yüksek yoğunluklu uyarma ışığı kullanılarak başlatılır .
NOT: Burada, odak düzleminde ~350 W/cm2 lazer yoğunluğu ilk ağartma ve tek EYFP moleküllerinin sonraki görüntülemeiçin kullanılır. Görüntüleme sırasında UV dalga boylarında eYFP moleküllerinin fotoaktivasyonu kullanılmadı. Bakteri hücresi başına en fazla bir moleküllü sinyal olmalıdır. Başlangıçta tek moleküllü sinyalin yoğunluğu çok yüksekse, veri edinmeye başlamadan önce yeterli fotobeyaztasyon meydana gelene kadar aydınlatmaya devam edin. - GUI'deki Close 515 nm lazer'e tıklayarak lazer aydınlatmasını kapatın. Aynı pozlama süresini kullanarak 200 kare karanlık görüntü toplayın.
- GUI'de Thorlabs LED'inin yanındaki kutuyu işaretleyin ve Ayna Yık'ıtıklatın. Bu faz kontrast yolu floresan yolu geçiş olacaktır.
- Veri toplama yazılımı IC Capture 2.4'ünbaşlatılmasını önleyin. Bu, faz kontrast ı yolundaki kamerayı kontrol eder. Kameradan canlı yayını görüntülemek için Canlı Görüntüle düğmesine basın. Capture'e tıklayın | Görüntü alanındaki hücrelerin faz kontrast görüntüsünü toplamak için Görüntüyü kaydedin.
- Ek FOV'lar için 3.5-3.10 adımlarını tekrarlayın. Burada, on farklı FOV'da ~500 bakteri hücrelerinden elde edilen veriler analiz için kullanılabilir tek moleküllü yörüngelerin sayısını artırmak için kullanılır.
DİkKAT: Agarose pedlerine uzun süre monte edilen hücreler, yeni monte edilmiş hücrelerden farklı davranabilir. Ayrıca, agarose pad veri kalitesini olumsuz etkileyebilir, bir süre sonra bütünlüğünü kaybedebilir. Tipik olarak, örnek slayt başına en fazla 3 FOV (mikroskop üzerinde~30 dk) kullanılır.
4. Veri işleme
NOT: MATLAB'da tek moleküllü yerelleştirmeleri ayıklamak için ham kamera çerçevelerinin analizi için Easy-DHPSF yazılımı24'ün değiştirilmiş bir sürümü kullanılır. Kolay-DHPSF tek moleküllü görüntüleme DHPSF lokalizasyonları sığdırmak için özel olarak kullanılır. Modern sCMOS kameraların piksele bağımlı gürültü özelliklerini hesaba karayan Maksimum Olasılık Tahmini (MLE) tabanlı montaj yordamını uygulamak için özel değişiklikler yapıldı26. Ayrıca HCImage Live programından (.dcimg) görüntü dosyası türü çıktısını kabul etmek üzere değiştirilmiştir. Yazılımın ve bireysel adımların daha ayrıntılı bir açıklaması için lütfen Lew ve ark.24
- MATLAB'daki Easy-DHPSF GUI'yi initialize edin (Ek Şekil 2). Adım 1.16.8'de kaydedilen dosyaya yük le.
- Adım 1.16.7'deki 7 şablonçıktısının her biri için eşik değerlerinin belirlenmesi
- SM tanımlamayı kalibre edin bölümünde Çalıştır'ıtıklatın. Varsayılan ayarları tutmak için aşağıdaki iki açılır pencerede Tamam'ı tıklatın.
- İstendiğinde ilk FOV'dan gelen verileri içeren görüntü yığınını açın.
- Şablonlarla eşleşmek için küçük bir çerçeve aralığı seçin. Genellikle 1001-2000 çerçeveleri ilk birkaç yüz kare yoğun çakışan sinyalleri önlemek için kullanılır. Varsayılan ayarları tutmak için aşağıdaki açılır pencerede Tamam'ı tıklatın. Aşağıdaki pencerede sıra günlüğü dosyası için istendiğinde İptal'i tıklatın.
- 3.8 adımda kaydedilmiş koyu arka planile görüntü yığınını açın. Arka plan tahmini parametrelerini varsayılan olarak bırakmak için aşağıdaki açılır pencerede 'Tamam' seçeneğini tıklatın. Varsayılan değer, geçerli çerçevenin etrafındaki 100 sonraki kamera çerçevesini kapsayan ortanca filtre27'yi kullanarak arka planı tahmin etmektir.
- Bir sonraki pencerede, tam FOV'yu kapsayacak şekilde resmin üzerine konan kutuyu yeniden boyutlandırın ve devam etmek için çift tıklatın.
- Çokgen oluşturmak için görüntünün birkaç noktasını tıklatarak ilgi çekici bölgeyi tanımlayın. İlgi alanı mümkün olduğunca çok görüş alanı içermelidir, görüntüde herhangi bir floresan boncuk (çok parlak nesneler) çokgen içinde yatıyordu değil, sonra devam etmek için çift tıklayın sağlarken.
NOT: Yazılım daha sonra şablonları görüntüyle eşleştirmeye çalışır ve daire içine alınmış olası eşleşmeleri olan bir görüntü görüntüler. - Yazılım durdurulduğunda, bulunan şablon eşleşmelerinin birçok görüntüsünü kaydeder ve ilgili eşik değerini önceden tanımlanmış bir klasörde görüntüler. Daha yüksek bir eşik değeri daha iyi bir eşleşmeye karşılık gelir. Her şablon numarası için, örnek eşleşmeleri inceleyin ve bir DHPSF görüntüsünü gösteren en düşük eşiği belirleyin. Easy-DHPSF GUI'deki SM Tanımlamayı Kalibrasyon bölümü altındaki 7 şablonun her biri için bu eşikleri girin.
NOT: Program otomatik olarak seçmek ve eşikleri giriş, ancak, bu genellikle güvenilmez ve el ile kontrol edilmelidir çalışır. Eşikler öyle seçilir ki, birkaç gerçek tek moleküllü sinyal kaçırılır, ancak montaj için yanlış pozitif adayların sayısı hesaplamalı olarak yönetilebilir kalır. - Sol üst köşedeki Kaydet simgesine tıklayarak Easy-DHPSF GUI'yi tekrar kaydedin.
- FOV floresan boncuk bir fiducial marker olarak kullanmak için uydurma
- Easy-DHPSF GUI'nin Track fiduciaries bölümünün altında Çalıştır'ıtıklatın. Varsayılan ayarları tutmak için aşağıdaki iki açılır pencerede Tamam'ı tıklatın.
- Görüntünün üzerine böşenen kutuyu sürükleyin ve floresan boncuktan gelen DHPSF sinyalinin üzerine ortalayın, ardından çift tıklatın.
- DHPSF sinyalinin ortasına tıklayın, iki lob arasındaki orta noktada, sonra enter tuşuna basın. Aşağıdaki pencerede sıra günlüğü dosyası için istendiğinde İptal'i tıklatın.
NOT: Yazılım, dhpsf'yi tüm kamera çerçevelerine sığdıracak ve ham görüntüyü ve yeniden oluşturulmuş görüntüyü görüntüler. Yazılım tamamlandığında, görüntü edinimi süresince floresan boncuğun X,Y ve Z konumları ile rakamlar çıkaracaktır. - Fiduciaries'i kullan yanındaki kutuyu işaretleyin ve sol üst köşedeki Kaydet simgesine tıklayarak Easy-DHPSF GUI'yi tekrar kaydedin.
- Adım 4.2'de elde edilen şablon eşiklerini kullanarak tüm kamera karelerinde tüm yerelleştirmeleri bulun ve sığdırın.
- Easy-DHPSF GUI'nin Yerelleştir DHPSF SM'leri bölümünün altında Çalıştır'ıtıklatın. Varsayılan ayarları tutmak için aşağıdaki açılır pencerelerde Tamam'ı tıklatın. Aşağıdaki pencerede sıra günlüğü dosyası için istendiğinde İptal'i tıklatın.
NOT: Eşleşmenin kalitesi kullanıcı tarafından tanımlanan eşiğin üzerindeyse, yazılım çift Gaussian modeli kullanarak DHPSF'yi bulur ve sığdıracaktır. Bu şablon eşleşmeleri etrafında daireler ile ham görüntü yanı sıra yeniden inşa DHPSF uygun bir görüntü görüntüler. - Sol üst köşedeki Kaydet simgesine tıklayarak Easy-DHPSF GUI'yi tekrar kaydedin.
- Easy-DHPSF GUI'nin Yerelleştir DHPSF SM'leri bölümünün altında Çalıştır'ıtıklatın. Varsayılan ayarları tutmak için aşağıdaki açılır pencerelerde Tamam'ı tıklatın. Aşağıdaki pencerede sıra günlüğü dosyası için istendiğinde İptal'i tıklatın.
- Tek moleküllü yerelleştirmeleri görüntüleme ve istenmeyen veya güvenilmez yerelleştirmeleri filtreleme
- Output DHPSF SM yerelleştirmeleri bölümü altında Filtre Çıktısı'nıtıklatın.
- Zaman içinde fiducial X,Y ve Z konumlarının enterpolasyonunu gerçekleştirmek için aşağıdaki üç pencerede Tamam'ı tıklatın. Çoğu durumda, varsayılan seçenekler yeterlidir. Siyah enterpolasyonlu çizgi kırmızı konum çizgisinin makul bir enterpolasyonunu yansıtmıyorsa, açılan penceredeki enterpolasyon parametrelerini değiştirin.
NOT: İnterpolasyonlu hat, tek moleküllü lokalizasyonları düzeltmek için kademe kayması için kullanılır. - İstendiğinde analiz edilen FOV için karşılık gelen faz kontrast görüntüsünü açın. Varsayılan ayarları tutmak için aşağıdaki iki açılır pencerede Tamam'ı tıklatın.
- Aşağıdaki iki açılır pencerede, filtre değerlerini değiştirilerek daha katı veya daha yumuşak tek moleküllü yerelleştirme gereksinimlerine izin verin, ardından Tamam'ıtıklatın.
- Görüntülenen pencerede, istenen ilgi alanı görüntülemek için görüntülerin üzerine konan kutuyu sürükleyin veya yeniden boyutlandırın ve devam etmek için çift tıklatın.
- Tek moleküllü yerelleştirmelerin 3Boyutlu rekonstrüksiyonu görüntülenir. Yeniden yapılandırma (döndürme, yakınlaştırma, vb) işlemek için şekil araçlarını kullanın. Başka bir diyalog kutusunu görüntülemek için Devam'ı tıklatın ve'Farklı bir parametre kümesiyle yeniden çizmek ister misiniz? Sonuçlar tatmin ediciyse, No'yutıklatın. Değilse, Evet'itıklatın.
NOT: Tatmin edici olmayan sonuçların yaygın nedenleri arasında faz kontrastı görüntüsünün yerelleştirme verileriyle doğru şekilde örtüşmemesi veya ilk şablon eşiklerinin çok düşük olması ve birçok yanlış pozitif yerelleştirme oluşturması yer almaktadır. Evet seçilirse, yazılım yeni parametre değerlerinin tanımlanabilmesi için adım 4.5.4'e geri döner. Hayır seçilirse, sonuçlar kaydedilir.
5. Veri işleme sonrası
- MATLAB'da özel yazılmış yazılım kullanarak, faz kontrast görüntüsünü yalnızca floresan mikroskobu altında görüntülenmiş hücreleri içeren bölgenin kalması için kırpın. Faz kontrastı görüntüsü floresan görüntüden çok daha büyük bir alanı kapsadığından, bu adım gereklidir. Görüntüyü kırpmak bir sonraki adımı basitleştirir.
- FAZ kontrast görüntüsünü OUFTI28 yazılımı ile işleyerek tek tek hücreleri segmentleyin (Ek Şekil 3)
- MATLAB'da OUFTI'yi başlangıç olarak algıla. Yük aşamasınatıklayarak kırpılmış faz kontrast görüntüsünü önceki adımdan yükleyin.
- Çıktı dosyası için bir kaydet konumunu seçmek ve adlandırmak için Dosya'yı tıklatın.
- Algılama ve Analiz başlığı altında Bağımsız çerçeveleri seçin.
- Hücre algılama parametrelerini yüklemek için Yük Parametreleri'ni tıklatın. Parametrelere örnek olarak kabul edilebilir hücre alanı, hücre genişliği ve hücre bölme eşiği verilebilir.
NOT: Bu parametrelerin tümü, kullanılan belirli hücre boyutları ve görüntü kalitesi için performansı en üst düzeye çıkarmak için ayarlanmalıdır. Daha da önemlisi, algoritma parametresi hücre anahatlarının kesin ölçümü için alt piksel olarak ayarlanmalıdır. - Hücre segmentasyonunu başlatmak için bu çerçeveyi tıklatın. İşlem tamamlandığında hücre anahatları faz kontrastı görüntüsü üzerinde görünür.
- El Kitabı bölümünün altındaki denetimleri kullanarak, hücreleri böl, hücreleri ekleyin veya otomatik işlem sırasında yanlış bölümlenen hücrelerin anahatlarını elde etmek için hücre anahatlarını hassaslaştırın.
- Hücre anahatlarını Kaydet çözümlemesi'nitıklatarak çıktı.
- Tek moleküllü yerelleştirmelerle önceki adımda elde edilen ana hatları tam olarak kaplamak için MATLAB'da özel yazılmış yazılım kullanın. Aşağıdaki alt adımlar yazılımın adımlarını ayrıntılı olarak ayrıntıya girer.
- Önceki adımda oluşturulan tek moleküllü yerelleştirme verilerinde ve hücre anahatlarında aynı beş hücrenin hücre kutuplarının konumunu kabaca tahmin ederek ve tıklayarak açılır penceredeki 5 kontrol noktası çiftini el ile seçin. Hücre kutbunun konumu kabaca bir hücreye ait tek moleküllü lokalizasyonlar etrafında bir konveks gövde çizim ve en yüksek eğrilik noktası seçerek tahmin edilebilir(Ek Şekil 4).
- MATLAB'daki cp2tform işlevini kullanarak bir 2B affine dönüştürme işlevi oluşturun ve hücre anahatlarının ve tek moleküllü yerelleştirmelerin kaba bir bindirmesini oluşturmak için kullanın.
- 10'dan az yerelleştirme içeren hücreleri silin ve görüş alanının dışında kısmen konumlandırılmış hücreleri kaldırın. Açılır penceredeki diğer istenmeyen hücreleri hücre anahattının içine tıklayarak el ile silin(Ek Şekil 5).
- Kalan tüm hücre anahatları ve içlerindeki tek moleküllü yerelleştirmeler için kütle merkezini kullanarak daha büyük bir kontrol noktası çifti kümesi oluşturmak, 2B dönüşüm işlevini yeniden hesaplamak ve hücre anahatlarının son bir bindirmesini oluşturmak için kullanın ve tek moleküllü lokalizasyonlar.
- Bir hücreanahattı nın sınırları içinde bulunan yerelleştirmeleri atayın. Herhangi bir hücre anahattı içinde bulunmayan tüm yerelleştirmeleri atın(Şekil 2a, Ek Şekil 6).
6. Tek moleküllü izleme
NOT: Aşağıdaki bölüm MATLAB'da özel olarak yazılmış yazılım kullanılarak tamamlanmıştır. Bu bölümde, yazılımın gerçekleştirdiği adımlar açıklanmaktadır.
- Aynı hücreye ve sonraki kamera karelerinde atanan yerelleştirmeler için, yerelleştirmeler arasındaki Öklid uzaklığı hesaplayın. Yerelleştirmeler arasındaki mesafe 2,5 μm'lik bir eşiğin altındaysa, yerelleştirmeleri aynı tek moleküllü yörüngeye atayarak bağlayın.
NOT: Mekansal ve zamansal eşik gereksinimlerini karşılayan bitişik hücrelerdeki yerelleştirmelerin birbirine bağlı olmaması için yalnızca tek bir hücreiçindeki yerelleştirmeleri göz önünde bulundurmak önemlidir. 2.5 μm eşik, çok hızlı bir molekülün (30μm 2/s) maruz kalma süresi (0.025 s) ve %20 tampon luk uzunluğu yla seyahat edebileceği maksimum mesafe olarak seçildi. - 4 yerelleştirmeden daha kısa yörüngeleri atın. Bir hücrede aynı anda iki veya daha fazla yerelleştirme (yani iki veya daha fazla floresan yayıcısı) varsa, ilişkili yörüngeleri atın. Pist uzunluğunun en az 4 yerelleştirmeye ayarlanması, çeşitli mesafe ölçümlerinin ortalamasını oluşturarak difüzyon katsayıları hakkında daha doğru bir tahmin sağlar.
- Belirli bir yörünge için ortalama kare lisi (MSD) hesaplayın:
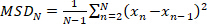 (1)
(1)
n yörüngedeki toplam lokalizasyon sayısı dır ve xn molekülün nnoktasındaki konumudur. - Görünürdidifüzyon katsayısını hesaplamak, D* tarafından
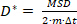 (2)
(2)
m = 2 veya 3 ölçümün boyutsallığı ve Δt kameranın pozlama süresidir.
NOT: Tipik bir deney toplamda ~5.000-100.000 yörünge üretecek ve bu da çok sayıda difüzyon katsayısı dağılımıyla sonuçlanır.
7. Sınırlı bir hacimde Brown hareketinin Monte-Carlo simülasyonu
NOT: 0,05-20 μm2/saralığında 64 değer kullanarak silindirik bir hacimle sınırlı Brown hareketinin Monte Carlo simülasyonlarını gerçekleştirerek simüle edilmiş görünür difüzyon katsayısı dağılımlarının kitaplıklarını oluşturun (yazılım istek üzerine yazarlardan temin edilebilir). Bu aralık, bakterilerde daha önce tahmin edilen floresan (füzyon) proteinlerinin difüzyon katsayıları aralığını kapsayacak şekilde seçilmiştir. Bu aralığın yeterince örneklemi için 64 difüzyon katsayısıkullanılmaktadır. Bu bölüm MATLAB'da özel yazılmış yazılım kullanılarak gerçekleştirilir ve yazılımın otomatik olarak attığı adımları açıklar. Burada kullanılan çubuk şeklindeki Y. enterokolitica hücreleri silindirik hacim l = 5 μm ve çapı d = 0.8 m olarak tahmin edilmektedir.
- Silindirik hacimde rastgele bir konumda tek tek yörüngeleri başlatın ve 100 ns'lik bir zaman aralığı kullanarak rastgele (yani Brownian) difüzyon adımlarını simüle edin (yeterli sağlamak için zaman aralığı kameranın maruz kalma süresinden çok daha kısa olmalıdır bir kamera çerçevesi boyunca pozisyonun örneklemesi). Eqn. 2'nin yeniden düzenlenmesiyle ilgili Gaussdağıtım işlevinden bir giriş D girişi için her yer değiştirme adımını örnekle ve önceki konuma ekleyin:
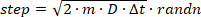 (3)
(3)
Burada MATLAB'daki randn fonksiyonu normal bir dağılımdan rastgele bir sayı örnekleri ver. Eğer bir adım molekülün silindirhacminin dışında yer değiştirmesine neden oluyorsa, molekülü silindirin içinde rastgele bir açıyla yansıtır. - Her zaman aralığı için, simüle edilen yayımlayıcının anlık x,y,z konumuna karşılık gelen bir DHPSF görüntüsü oluşturun.
- Deneysel koşulları eşleştirmek için, yerelleştirme başına 1.000 foton içeren görüntüleri, piksel başına 13 foton ve Poisson gürültüsüiçeren lazer arka planıyla simüle edin. Buna ek olarak, deneysel kamera kalibrasyon ölçümleri ile tutarlı piksel başına ~ 50 foton ve Gaussian okuma gürültü (σ ~ 1,5 foton) koyu ofset ekleyin. Son olarak, görüntüyü dedektör sayıları birimlerinde elde etmek için sCMOS kameranın deneysel olarak ölçülen piksele bağımlı kazancıyla çarpın.
NOT: Bu manipülasyonlardan sonra, son görüntünün sinyal-gürültü oranı ~2'dir. - Deneysel veri toplama sırasında kullanılan pozlama süresi boyunca simüle edilen 50 DHPSF görüntüsünü bir birime kten sonra moleküllerin değişen konumunu yansıtan hareket bulanıklığı görüntüleri oluşturun. Hesaplama giderini sınırlamak için, görüntü oluşturmak için yalnızca 50 periyodik olarak örneklenmiş pozisyon seçilmiştir (100 ns'lik bir örnekleme aralığında 0,025 s pozlama süresi boyunca örneklenmiş 250.000 pozisyonun tümü yerine).
NOT: Simüle edilmiş yörüngelerin uzunluğu (kare sayısı) deneysel yörüngelerin ortalama uzunluğuyla eşleşmelidir. Bu durumda, yörünge başına kare sayısı 6'dır.
- Deneysel koşulları eşleştirmek için, yerelleştirme başına 1.000 foton içeren görüntüleri, piksel başına 13 foton ve Poisson gürültüsüiçeren lazer arka planıyla simüle edin. Buna ek olarak, deneysel kamera kalibrasyon ölçümleri ile tutarlı piksel başına ~ 50 foton ve Gaussian okuma gürültü (σ ~ 1,5 foton) koyu ofset ekleyin. Son olarak, görüntüyü dedektör sayıları birimlerinde elde etmek için sCMOS kameranın deneysel olarak ölçülen piksele bağımlı kazancıyla çarpın.
- 64 simüle girdi difüzyon katsayısının her biri için 5.000 yörünge oluşturun.
- Veri İşleme bölümünde açıklandığı gibi simüle edilmiş kamera çerçevelerini analiz edin.
- Yerelleştirmeleri Tek Moleküllü İzleme bölümünde açıklandığı gibi yörüngelere bağla.
- 25. siparişin B-spline enterpolasyonunu kullanarak her simüle dağıtım için kümülatif dağıtım işlevini (CDF) enterpolasyona tabi atın. İnterpolated dağılımları, rasgele noktalarda sorgulanabilmeleri için gereklidir.
- Ortaya çıkan B-spline eğrilerini Dekseni boyunca enterpolasyon(D, simüle edilen yayıcıların hareketini yöneten gerçek sınırlandırMa katsayısıdır) MATLAB'daki dağınık Interpolant işlevini kullanarak (' doğal' enterpolasyon yöntemi). Bu, 0,05-20 μm2/s aralığında gerçek bir difüzyon katsayısı değerine karşılık gelen herhangi bir belirgin difüzyon katsayısı dağılımının sorgulanabildiği sürekli bir 2B fonksiyonu sağlar.
8. Deneysel görünür difüzyon katsayısı dağılımı montajı
NOT: Önceki bölümde oluşturulan simüle edilmiş dağılımların doğrusal kombinasyonlarını kullanarak görünür difüzyon katsayılarının deneysel olarak ölçülen dağılımlarını(sınırlı hacimdeki Brown hareketinin Monte-Carlo simülasyonu)sığdırın. Bu bölüm MATLAB'da özel yazılmış yazılım kullanılarak gerçekleştirilir ve yazılımın otomatik olarak attığı adımları açıklar. Daha fazla bilgi ve uygulama örnekleri için lütfen Rocha ve ark.29
- Bölüm 7'de oluşturulan kütüphaneden periyodik olarak örneklenmiş bir dizi simüle CDF kullanarak deneysel CDF'nin (MATLAB'daki lsqlin işlevini kullanarak) sınırlandırılmış doğrusal en küçük kareler gerçekleştirin. Bu adımın çıktısı, deneysel dağılımda yaygın diffüz durumların difüzyon katsayılarını ve popülasyon fraksiyonlarını içeren bir parametre vektörüdür.
- Difüzyon katsayısı değerleri ile difüzyon katsayısı değerlerini% 20'lik bir oranla tek bir diffusive durumuna göre göreli popülasyon fraksiyonuna göre ortalama ağırlık la birleştirin. Bu başlangıç parametre vektörüdür.
NOT: Model karmaşıklığını azaltmak için, difüzyon katsayıları 0,5 μm2/s'nin altında olan diffüzif durumlar aşağıdaki adımların tümlerinde sabit tutulabilir. - Tek bir diffusive durumundan kullanıcı tarafından tanımlanan maksimum durum sayısına kadar farklı sayıda diffusive durumiçeren deneme donatım parametre vektörleri dizileri oluşturun.
- Başlangıç parametre vektörünü kullanarak, bitişik difüzyon durumlarını ağırlıklı ortalama ve bölünmüş difusive durumları eşit popülasyon fraksiyonları ve difüzyon katsayıları orijinal değerin %20 üzerinde ve altında iki eyalete ayırın. Tüm durum birleşimi ve bölme olasılıkları için tekrarlayın.
- Verilerin 5 ayrı alt kümesinin doğrusal olmayan en küçük kare montajını (MATLAB'daki fmincon işlevini kullanarak) başlatması için her deneme uygun parametre vektörü kullanın. Sığdırma ve kalan alt kümelere karşılık gelen dağılım arasındaki karelerin artık toplamını bularak uyumun kalitesini belirleyin (veri çapraz doğrulama).
- Her deneme vektörü için 5 ayrı bağlantı nın ortalama kare sayısını genel uyum kalitesi olarak kullanın, her diffusive durum sayısı için en uygun şekilde deneme vektörü belirleyin.
- Ek bir durum eklemenin uyum un kalitesinde en az %5'lik bir iyileşmeye yol açmayan deneme vektörünü belirleyerek en uygun durum sayısını belirleyin.
- Tam veri kümesinin doğrusal olmayan en küçük karelerini başlatmayı sağlamak için bu deneme vektörü kullanın.
- Aynı deneme vektörü ile bootstrapping ve refitting tarafından verileri yeniden örnekleme tarafından tek tek parametreler için hata tahmin.
Sonuçlar
Burada açıklanan deneysel koşullar altında (20.000 kare, yörünge uzunluğu en az 4 lokalizasyon) ve floresan etiketli füzyon proteinlerinin ifade düzeylerine bağlı olarak, yaklaşık 200-3.000 lokalizasyon 10-150 verim yörüngeler hücre başına oluşturulabilir (Şekil 2a,b). Belirgin difüzyon katsayıları iyi örneklenmiş bir dağılım üretmek için çok sayıda yörünge gereklidir. Burada toplanan FOV boyutu ~ 55 x 55 μm...
Tartışmalar
Sunulan protokolün başarılı bir şekilde uygulanması için kritik bir faktör, tek moleküllü sinyallerin birbirinden iyi ayrılmasını sağlamaktır (yani, uzayda ve zamanda seyrek olmaları gerekir(Ek Mov. 1)). Bir hücrede aynı anda birden fazla floresan molekül varsa, lokalizasyon başka moleküllerin yörüngesine yanlış atanabilir. Bu bağlantı sorunu30olarak adlandırılır. Bağlanma problemini önlemek için protein ekspresyonu düzeyleri ve uyarma lazer yoğu...
Açıklamalar
Yazarların açıklayacak bir şeyi yok.
Teşekkürler
El yazmasının eleştirel okuması için Alecia Achimovich ve Ting Yan'a teşekkür ederiz. Biz Ed Hall, Virginia Üniversitesi'nde İleri Araştırma Bilgi İşlem Hizmetleri grubunda kıdemli personel bilim adamı, bu çalışmada kullanılan optimizasyon rutinleri kurma konusunda yardım için teşekkür ederiz. Bu çalışma için finansman Virginia Üniversitesi tarafından sağlanmıştır.
Malzemeler
| Name | Company | Catalog Number | Comments |
| 2,6-diaminopimelic acid | Chem Impex International | 5411 | Necessary for growth of Y. enterocolitica cells used. |
| 4f lenses | Thorlabs | AC508-080-A | f = 80mm, 2" |
| 514 nm laser | Coherent | Genesis MX514 MTM | Use for fluorescence excitation |
| agarose | Inivtrogen | 16520100 | Used to make gel pads to mount liquid bacterial sample on microscope. |
| ammonium chloride | Sigma Aldrich | A9434 | M2G ingredient. |
| bandpass filter | Chroma | ET510/bp | Excitation pathway. |
| Brain Heart Infusion | Sigma Aldrich | 53286 | Growth media for Y. enterocolitica. |
| calcium chloride | Sigma Aldrich | 223506 | M2G ingredient. |
| camera | Imaging Source | DMK 23UP031 | Camera for phase contrast imaging. |
| dielectric phase mask | Double Helix, LLC | N/A | Produces DHPSF signal. |
| disodium phosphate | Sigma Aldrich | 795410 | M2G ingredient. |
| ethylenediaminetetraacetic acid | Fisher Scientific | S311-100 | Chelates Ca2+. Induces secretion in the T3SS. |
| flip mirror | Newport | 8892-K | Allows for switching between fluorescence and phase contrast pathways. |
| fluospheres | Invitrogen | F8792 | Fluorescent beads. 540/560 exication and emission wavelengths. 40 nm diameter. |
| glass cover slip | VWR | 16004-302 | #1.5, 22mmx22mm |
| glucose | Chem Impex International | 811 | M2G ingredient. |
| immersion oil | Olympus | Z-81025 | Placed on objective lens. |
| iron(II) sulfate | Sigma Aldrich | F0518 | M2G ingredient. |
| long pass filter | Semrock | LP02-514RU-25 | Emission pathway. |
| magnesium sulfate | Fisher Scientific | S25414A | M2G ingredient. |
| microscope platform | Mad City Labs | custom | Platform for inverted microscope. |
| nalidixic acid | Sigma Aldrich | N4382 | Y. enterocolitica cells used are resistant to nalidixic acid. |
| objective lens | Olympus | 1-U2B991 | 60X, 1.4 NA |
| Ozone cleaner | Novascan | PSD-UV4 | Used to eliminate background fluorescence on glass cover slips. |
| potassium phosphate | Sigma Aldrich | 795488 | M2G ingredient. |
| Red LED | Thorlabs | M625L3 | Illuminates sample for phase contrast imaging. 625nm. |
| sCMOS camera | Hamamatsu | ORCA-Flash 4.0 V2 | Camera for fluorescence imaging. |
| short pass filter | Chroma | ET700SP-2P8 | Emission pathway. |
| Tube lens | Thorlabs | AC508-180-A | f=180 mm, 2" |
| Yersinia enterocolitica dHOPEMTasd | N/A | N/A | Strain AD4442, eYFP-YscQ |
| zero-order quarter-wave plate | Thorlabs | WPQ05M-514 | Excitation pathway. |
Referanslar
- Kapanidis, A. N., Uphoff, S., Stracy, M. Understanding Protein Mobility in Bacteria by Tracking Single Molecules. Journal of Molecular Biology. , (2018).
- Betzig, E., et al. Imaging intracellular fluorescent proteins at nanometer resolution. Science. 313 (5793), 1642-1645 (2006).
- Hess, S. T., Girirajan, T. P. K., Mason, M. D. Ultra-high resolution imaging by fluorescence photoactivation localization microscopy. Biophysical Journal. 91 (11), 4258-4272 (2006).
- Rust, M. J., Bates, M., Zhuang, X. W. Sub-diffraction-limit imaging by stochastic optical reconstruction microscopy (STORM). Nature Methods. 3 (10), 793-795 (2006).
- Chen, T. Y., et al. Quantifying Multistate Cytoplasmic Molecular Diffusion in Bacterial Cells via Inverse Transform of Confined Displacement Distribution. Journal of Physical Chemistry B. 119 (45), 14451-14459 (2015).
- Mohapatra, S., Choi, H., Ge, X., Sanyal, S., Weisshaar, J. C. Spatial Distribution and Ribosome-Binding Dynamics of EF-P in Live Escherichia coli. mBio. 8 (3), (2017).
- Stracy, M., et al. Single-molecule imaging of UvrA and UvrB recruitment to DNA lesions in living Escherichia coli. Nature Communications. 7, 12568 (2016).
- Persson, F., Lindén, M., Unoson, C., Elf, J. Extracting intracellular diffusive states and transition rates from single-molecule tracking data. Nature Methods. 10 (3), 265-269 (2013).
- Bakshi, S., Choi, H., Weisshaar, J. C. The spatial biology of transcription and translation in rapidly growing Escherichia coli. Frontiers in Microbiology. 6, 636 (2015).
- Mustafi, M., Weisshaar, J. C. Simultaneous Binding of Multiple EF-Tu Copies to Translating Ribosomes in Live Escherichia coli. mBio. 9 (1), (2018).
- Michalet, X., Berglund, A. J. Optimal diffusion coefficient estimation in single-particle tracking. Physical Review E. 85 (6), (2012).
- Michalet, X. Mean square displacement analysis of single-particle trajectories with localization error: Brownian motion in an isotropic medium. Physical Review E Statistical, Nonlinear, and Soft Matter Physics. 82 (4 Pt 1), 041914 (2010).
- Backlund, M. P., Joyner, R., Moerner, W. E. Chromosomal locus tracking with proper accounting of static and dynamic errors. Physical Review E Statistical, Nonlinear, and Soft Matter Physics. 91 (6), 062716 (2015).
- Stracy, M., et al. Live-cell superresolution microscopy reveals the organization of RNA polymerase in the bacterial nucleoid. Proceedings of the National Academy of Sciences of the United States of America. 112 (32), E4390-E4399 (2015).
- Plochowietz, A., Farrell, I., Smilansky, Z., Cooperman, B. S., Kapanidis, A. N. In vivo single-RNA tracking shows that most tRNA diffuses freely in live bacteria. Nucleic Acids Research. 45 (2), 926-937 (2017).
- Koo, P. K., Mochrie, S. G. Systems-level approach to uncovering diffusive states and their transitions from single-particle trajectories. Physical Review E. 94 (5-1), 052412 (2016).
- Uphoff, S., Reyes-Lamothe, R., Garza de Leon, F., Sherratt, D. J., Kapanidis, A. N. Single-molecule DNA repair in live bacteria. Proceedings of the National Academy of Sciences of the United States of America. 110 (20), 8063-8068 (2013).
- Bakshi, S., Bratton, B. P., Weisshaar, J. C. Subdiffraction-Limit Study of Kaede Diffusion and Spatial Distribution in Live Escherichia coli. Biophysical Journal. 101 (10), 2535-2544 (2011).
- Bakshi, S., Siryaporn, A., Goulian, M., Weisshaar, J. C. Superresolution imaging of ribosomes and RNA polymerase in live Escherichia coli cells. Molecular Microbiology. 85 (1), 21-38 (2012).
- Uphoff, S., Sherratt, D. J., Kapanidis, A. N. Visualizing Protein-DNA Interactions in Live Bacterial Cells Using Photoactivated Single-molecule Tracking. JoVE. (85), e51177 (2014).
- Pavani, S. R. P., Piestun, R. Three dimensional tracking of fluorescent microparticles using a photon-limited double-helix response system. Optics Express. 16 (26), 22048-22057 (2008).
- Pavani, S. R. P., et al. Three-dimensional, single-molecule fluorescence imaging beyond the diffraction limit by using a double-helix point spread function. Proceedings of the National Academy of Sciences of the United States of America. 106 (9), 2995-2999 (2009).
- Rocha, J. M., et al. Single-molecule tracking in live Yersinia enterocolitica reveals distinct cytosolic complexes of injectisome subunits. Integrative Biology. 10 (9), 502-515 (2018).
- Lew, M. D., von Diezmann, A. R. S., Moerner, W. E. Easy-DHPSF open-source software for three-dimensional localization of single molecules with precision beyond the optical diffraction limit. Protocol Exchange. , (2013).
- Biteen, J. S., et al. Super-resolution imaging in live Caulobacter crescentus cells using photoswitchable EYFP. Nature Methods. 5 (11), 947-949 (2008).
- Huang, F., et al. Video-rate nanoscopy using sCMOS camera-specific single-molecule localization algorithms. Nature Methods. 10 (7), 653-658 (2013).
- Hoogendoorn, E., et al. The fidelity of stochastic single-molecule super-resolution reconstructions critically depends upon robust background estimation. Scientific Reports. 4, 3854 (2014).
- Paintdakhi, A., et al. Oufti: An integrated software package for high-accuracy, high-throughput quantitative microscopy analysis. Molecular microbiology. 99 (4), 767-777 (2016).
- Rocha, J. M., Corbitt, J., Yan, T., Richardson, C., Gahlmann, A. Resolving Cytosolic Diffusive States in Bacteria by Single-Molecule Tracking. bioRxiv. , 483321 (2018).
- Lee, A., Tsekouras, K., Calderon, C., Bustamante, C., Presse, S. Unraveling the Thousand Word Picture: An Introduction to Super-Resolution Data Analysis. Chemical Reviews. 117 (11), 7276-7330 (2017).
- Los, G. V., et al. HaloTag: A Novel Protein Labeling Technology for Cell Imaging and Protein Analysis. ACS Chemical Biology. , (2008).
- Gautier, A., et al. An Engineered Protein Tag for Multiprotein Labeling in Living Cells. Chemistry & Biology. 15 (2), 128-136 (2008).
- Bisson-Filho, A. W., et al. Treadmilling by FtsZ filaments drives peptidoglycan synthesis and bacterial cell division. Science. 355 (6326), 739-743 (2017).
- Douglass, K. M., Sieben, C., Archetti, A., Lambert, A., Manley, S. Super-resolution imaging of multiple cells by optimised flat-field epi-illumination. Nature Photonics. 10 (11), 705-708 (2016).
- Zhao, Z., Xin, B., Li, L., Huang, Z. L. High-power homogeneous illumination for super-resolution localization microscopy with large field-of-view. Optics Express. 25 (12), 13382-13395 (2017).
- Yan, T., Richardson, C. J., Zhang, M., Gahlmann, A. Computational Correction of Spatially-Variant Optical Aberrations in 3D Single Molecule Localization Microscopy. bioRxiv. , 504712 (2018).
- Gahlmann, A., Moerner, W. E. Exploring bacterial cell biology with single-molecule tracking and super-resolution imaging. Nature Reviews Microbiology. 12 (1), 9-22 (2014).
- Liu, Z., Lavis, L. D., Betzig, E. Imaging live-cell dynamics and structure at the single-molecule level. Mol Cell. 58 (4), 644-659 (2015).
- Berglund, A. J. Statistics of camera-based single-particle tracking. Physical Review E. 82 (1), 011917 (2010).
- Parry, B. R., et al. The bacterial cytoplasm has glass-like properties and is fluidized by metabolic activity. Cell. 156 (1-2), 183-194 (2014).
- English, B. P., et al. Single-molecule investigations of the stringent response machinery in living bacterial cells. Proceedings of the National Academy of Sciences of the United States of America. 108 (31), E365-E373 (2011).
- Niu, L. L., Yu, J. Investigating intracellular dynamics of FtsZ cytoskeleton with photoactivation single-molecule tracking. Biophysical Journal. 95 (4), 2009-2016 (2008).
- Coquel, A. S., et al. Localization of protein aggregation in Escherichia coli is governed by diffusion and nucleoid macromolecular crowding effect. PLoS Computational Biology. 9 (4), e1003038 (2013).
- Nenninger, A., Mastroianni, G., Mullineaux, C. W. Size Dependence of Protein Diffusion in the Cytoplasm of Escherichia coli. Journal of Bacteriology. 192 (18), 4535-4540 (2010).
- Dix, J. A., Verkman, A. S. Crowding effects on diffusion in solutions and cells. Annual Review of Biophysics. 37, 247-263 (2008).
- Elliott, L. C., Barhoum, M., Harris, J. M., Bohn, P. W. Trajectory analysis of single molecules exhibiting non-brownian motion. Physical Chemistry Chemical Physics. 13 (10), 4326-4334 (2011).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır