Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Single-Molecule Tracking-Mikroskopie - Ein Werkzeug zur Bestimmung der diffusiven Zustände von zytosolischen Molekülen
In diesem Artikel
Zusammenfassung
Die 3D-Einzelmolekül-Lokalisationsmikroskopie wird verwendet, um die räumlichen Positionen und Bewegungsbahnen fluoreszierend markierter Proteine in lebenden Bakterienzellen zu untersuchen. Das hier beschriebene experimentelle und Datenanalyseprotokoll bestimmt das vorherrschende diffusive Verhalten von zytosolischen Proteinen auf der Grundlage gepoolter Einzelmolekülbahnen.
Zusammenfassung
Die Einzelmolekül-Lokalisationsmikroskopie untersucht die Position und Bewegungen einzelner Moleküle in lebenden Zellen mit einer räumlichen und Millisekunden-Zeitlichen Auflösung von Zehnmetern Nanometer. Diese Fähigkeiten machen die Einmolekül-Lokalisationsmikroskopie ideal geeignet, um biologische Funktionen auf molekularer Ebene in physiologisch relevanten Umgebungen zu untersuchen. Hier zeigen wir ein integriertes Protokoll zur Erfassung und Verarbeitung/Analyse von Single-Molekül-Tracking-Daten, um die verschiedenen diffusiven Zustände zu extrahieren, die ein Protein von Interesse aufweisen kann. Diese Informationen können verwendet werden, um molekulare komplexe Bildung in lebenden Zellen zu quantifizieren. Wir bieten eine detaillierte Beschreibung eines kamerabasierten 3D-Einzelmolekül-Lokalisierungsexperiments sowie der nachfolgenden Datenverarbeitungsschritte, die die Bahnen einzelner Moleküle ergeben. Diese Bahnen werden dann mit hilfe eines numerischen Analyserahmens analysiert, um die vorherrschenden diffusiven Zustände der fluoreszierend markierten Moleküle und die relative Häufigkeit dieser Zustände zu extrahieren. Das Analyseframework basiert auf stochastischen Simulationen intrazellulärer Brownianer Diffusionsbahnen, die durch eine beliebige Zellgeometrie räumlich begrenzt sind. Basierend auf den simulierten Flugbahnen werden rohe Einzelmolekülbilder auf die gleiche Weise erzeugt und analysiert wie experimentelle Bilder. Auf diese Weise werden experimentelle Präzisions- und Genauigkeitseinschränkungen, die experimentell schwer zu kalibrieren sind, explizit in den Analyse-Workflow integriert. Der Diffusionskoeffizient und die relativen Bevölkerungsfraktionen der vorherrschenden diffusiven Zustände werden bestimmt, indem die Verteilungen der experimentellen Werte mithilfe linearer Kombinationen simulierter Verteilungen einzupassen sind. Wir demonstrieren den Nutzen unseres Protokolls, indem wir die diffusiven Zustände eines Proteins auflösen, das bei der Bildung von homo- und heterooligomeren Komplexen im Zytosol eines bakteriellen Erregers unterschiedliche diffusive Zustände aufweist.
Einleitung
Die Untersuchung des diffusiven Verhaltens von Biomolekülen gibt Einblick in ihre biologischen Funktionen. Fluoreszenzmikroskopie-basierte Techniken sind zu wertvollen Werkzeugen für die Beobachtung von Biomolekülen in ihrer nativen Zellumgebung geworden. Fluoreszenzrückgewinnung nach Photobleichung (FRAP) und Fluoreszenzkorrelationsspektroskopie (FCS)1 bieten ensemble-gemittelte diffusive Verhaltensweisen. Umgekehrt ermöglicht die Einzelmolekül-Lokalisationsmikroskopie die Beobachtung einzelner fluoreszierend markierter Moleküle mit hoher räumlicher und zeitlicher Auflösung2,3,4. Die Beobachtung einzelner Moleküle ist von Vorteil, da ein Protein von Interesse in verschiedenen diffusiven Zuständen existieren kann. Beispielsweise entstehen zwei leicht zu unterscheidende diffusive Zustände, wenn ein Transkriptionsregulator, wie CueR in Escherichia coli,frei im Zytosol diffundiert oder an eine DNA-Sequenz bindet und auf der Zeitskala der Messung immobilisiert wird5 . Ein-Molekül-Tracking bietet ein Werkzeug, um diese verschiedenen Zustände direkt zu beobachten, und anspruchsvolle Analysen sind nicht erforderlich, um sie zu lösen. Allerdings wird es schwieriger, mehrere diffusive Staaten und ihre Bevölkerungsfraktionen in Fällen zu lösen, in denen ihre diffusiven Raten ähnlicher sind. Aufgrund der Größenabhängigkeit des Diffusionskoeffizienten manifestieren sich z. B. unterschiedliche Oligomerisierungszustände eines Proteins als verschiedene diffusive Zustände6,7,8,9 , 10. Solche Fälle erfordern einen integrierten Ansatz in Bezug auf Datenerfassung, -verarbeitung und -analyse.
Ein kritischer Faktor, der die diffusiven Raten von zytosolischen Molekülen beeinflusst, ist die Wirkung der Eingrenzung durch die Zellgrenze. Die Beschränkungen für die molekulare Bewegung durch eine bakterielle Zellgrenze führen dazu, dass die gemessene Diffusionsrate eines zytosolischen Moleküls langsamer erscheint, als wenn dieselbe Bewegung in einem nicht begrenzten Raum aufgetreten wäre. Bei sehr langsam diffundierenden Molekülen ist die Wirkung der zellulären Einschließung aufgrund fehlender Kollisionen mit der Grenze vernachlässigbar. In solchen Fällen kann es möglich sein, diffusive Zustände genau aufzulösen, indem die Verteilungen von molekularen Verschiebungen, roder scheinbaren Diffusionskoeffizienten, D*,mithilfe analytischer Modelle, die auf den Gleichungen für die Brownsche Bewegung basieren ( zufällige Diffusion)11,12,13. Für schnell diffundierende zytosolische Moleküle ähneln die experimentellen Verteilungen jedoch nicht mehr denen, die aufgrund von Kollisionen von diffundierenden Molekülen mit den Zellgrenzen für eine unbeschränkte Brownsche Bewegung erhalten wurden. Die Eingrenzungseffekte müssen berücksichtigt werden, um die nicht begrenzten Diffusionskoeffizienten der fluoreszierend markierten Moleküle genau zu bestimmen. In jüngster Zeit wurden mehrere Ansätze entwickelt, um Die eingrenzende Wirkung entweder (halb-)analytisch 5,14,15,16 oder numerisch durch Monte-Carlo-Simulationen von Braune Diffusion6,10,16,17,18,19.
Hier bieten wir ein integriertes Protokoll zum Sammeln und Analysieren von Single-Molekül-Lokalisierungsmikroskopiedaten mit besonderem Fokus auf Single-Molekül-Tracking. Das Endziel des Protokolls ist es, diffusive Zustände von fluoreszierend markierten zytosolischen Proteinen in diesem Fall stabförmigen Bakterienzellen aufzulösen. Unsere Arbeit baut auf einem früheren Protokoll zur Einzelmolekülverfolgung auf, in dem eine DNA-Polymerase, PolI, durch Diffusionsanalyse20in einem DNA-gebundenen und ungebundenen Zustand nachgewiesen wurde. Hier erweitern wir die Einzelmolekül-Tracking-Analyse auf 3D-Messungen und führen realistischere Rechensimulationen durch, um mehrere diffusive Zustände gleichzeitig in Zellen aufzulösen und zu quantifizieren. Die Daten werden mit einem selbst gebauten 3D-Superauflösungsfluoreszenzmikroskop erfasst, das in der Lage ist, die 3D-Position von Fluoreszenzstrahlern durch Bildgebung mit der Doppelhelix-Punkt-Spread-Funktion (DHPSF)21,22zu bestimmen. Die rohen Einzelmolekülbilder werden mit individueller Software verarbeitet, um die 3D-Einzelmolekül-Lokalisationen zu extrahieren, die dann zu Einmolekül-Trajektorien kombiniert werden. Tausende von Flugbahnen werden gebündelt, um Verteilungen von scheinbaren Diffusionskoeffizienten zu erzeugen. In einem letzten Schritt passen die experimentellen Verteilungen zu numerisch generierten Verteilungen, die durch Monte-Carlo-Simulationen der Brownschen Bewegung in einem begrenzten Volumen erhalten werden. Wir wenden dieses Protokoll an, um die diffusiven Zustände des Typ-3-Sekretionssystemproteins YscQ in lebenden Yersinia enterocoliticazu lösen. Aufgrund seines modularen Charakters ist unser Protokoll allgemein auf jede Art von Einzelmolekül- oder Einzelpartikel-Tracking-Experiment in beliebigen Zellgeometrien anwendbar.
Protokoll
1. Doppelhelix Punkt-Spread-Funktionskalibrierung
HINWEIS: Die in diesem und den folgenden Abschnitten beschriebenen Bilder werden mit einem kundenspezifischen invertierten Fluoreszenzmikroskop, wie in Rocha et al.23beschrieben, erstellt. Das gleiche Verfahren gilt für verschiedene Mikroskop-Implementierungen für die Ein-Molekül-Lokalisierung und Tracking-Mikroskopie2,3,4. Alle in diesem Artikel beschriebenen Software zur Bildaufnahme und -verarbeitung ist verfügbar (https://github.com/GahlmannLab2014/Single-Molecule-Tracking-Analysis.git).
- Herstellung von Agarose-Pads zur Montage von Proben auf Glasabdeckungsscheinen, die unter dem Mikroskop zu betrachten sind.
- Fügen Sie 1,5-2 Gewichtsniedriger Schmelzpunkt Agarose zu 5 ml M2G Puffer hinzu (4,9 mM Na2HPO4, 3,1 mM KH2PO4, 7,5 mM NH4Cl, 0,5 mM MgSO4, 10 m FeSO4, 0,5 mM CaCl2 und 0,2% Glukose). Mikrowelle für einige Sekunden, bis sich alle Agarose aufgelöst hat. Lassen Sie die Lösung nicht kochen.
- Lassen Sie die Agarose-Lösung für ein paar Minuten abkühlen (2-3 min).
- Pipette 600 l Agaroselösung auf einen Glasdeckelschlupf (22 mm x 22 mm). Legen Sie vorsichtig einen zweiten Glasdeckel auf die Agarose. Dadurch entsteht ein dünnes Agarose-Gelpad (ca. 0,5 mm) zwischen den beiden Glasabdeckungsschlüpfern.
- Lassen Sie die Agarose-Pads sitzen, um für 20 min zu erstarren.
- Trennen Sie die Glasabdeckung vorsichtig. Das Agarose-Pad klebt an einem von ihnen.
- Schneiden Sie das Agarose-Pad mit einer Rasierklinge in vier quadratische Abschnitte gleicher Größe. Jeder quadratische Abschnitt kann für eine einzelne Probe verwendet werden.
- Pipette 1,5 l fluoreszierende Perlenlösung auf einem Agarosepad. Die Perlenlösung ist eine Verdünnung der Lagerlösung in M2G (siehe Materialtabelle).
- Invertieren Sie das Agarose-Pad und legen Sie es auf einen Glasdeckel,der 30 min in einem Ozonreiniger gereinigt wurde. Die Reinigungszeit wird gewählt, um alle fluoreszierenden Moleküle zu beseitigen, die an den Abdeckungen haften.
- Montieren Sie das Probenabdeckungsglas auf den Probenhalter eines invertierten Fluoreszenzmikroskops und sichern Sie es mit Klebeband oder federbelasteten Probenhalterclips an Ort und Stelle.
- Fügen Sie einen Tropfen Tauchöl auf das Mikroskopobjektiv.
- Stellen Sie den Probenhalter auf ein Mikroskop und stellen Sie ihn sicher.
- Initialisieren Sie die grafische Benutzeroberfläche (GUI), um die Kamera, die Probenphase und die Anregungslaser des Mikroskops zu steuern. Hier wird kundenspezifische Software in MATLAB zur Instrumentensteuerung eingesetzt (Ergänzende Abbildung 1).
- Initialisieren Sie die Kamerasoftware HCImage Live. Legen Sie auf der Registerkarte Aufnahme unter dem Abschnitt Kamerasteuerung die Belichtungszeit auf 0,03 s fest. Klicken Sie auf Live, um einen Live-Feed der Kamera zu starten.
- Schalten Sie den Laser ein, indem Sie auf open 514 nm Laser auf der GUI-Schnittstelle klicken, um die fluoreszierenden Perlen auf dem Pad zu anregen und die Fluoreszenzemission auf der Kamera im Live-Stream-Modus anzuzeigen (d. h. keine Daten auf der Festplatte gespeichert).
- Passen Sie die X- und Y-Positionen der Mikroskopstufe an, indem Sie auf die Xy-Pos-Pfeile unter dem Abschnitt "Mikropositionierungsstufe" der GUI klicken, um mindestens eine fluoreszierende Perle in der Mitte des Sichtfeldes (FOV) zu positionieren. Die Schrittgröße kann geändert werden, indem Sie auf das Dropdown-Feld unter den Pfeilen klicken.
- Passen Sie die Z-Position der Mikroskopstufe an, indem Sie auf die Z-Pos-Pfeile unter dem Abschnitt Nano-Positionierungsstufe der GUI klicken. Legen Sie die Ausrichtung der Doppelhelix-Punkt-Spread-Funktion (DHPSF) der fluoreszierenden Perle vertikal fest. Diese vertikale Ausrichtung wird als Ausgangspunkt in der Z-Kalibrierung definiert. Die Schrittgröße kann geändert werden, indem Sie auf das Dropdown-Feld unter den Pfeilen klicken.
- In HCImage Live unter Capture | Trigger-Modi, Geschwindigkeit und Registrierung, ändern Sie den Trigger-Modus von Internal to External Level Trigger. Dadurch kann die MATLAB-GUI die Kamera steuern.
- Unter Sequenz | Scaneinstellungen, ändern Sie die Anzahl der Frame-Zahlen auf 1200. Wählen Sie einen Speicher-Zielordner aus, indem Sie auf die Schaltfläche mit der Bezeichnung ...klicken. Klicken Sie schließlich auf Start. Die Anzahl der Rahmenzählungen ist auf 1200 festgelegt, so dass 10 Frames für jeden der 120 Z Positionsschritte gesammelt werden können.
- Scannen Sie durch eine Reihe von Z-Positionen (30 Schritte über und unter der Start-Z-Position in 50 nm Schritten) und zeichnen Sie 10 Frames bei jedem Schritt mit einer Belichtungszeit von 0,03 s auf. Beginnen Sie den automatisierten Prozess, indem Sie unter dem Abschnitt Z-Kalibrierung von die GUI.
HINWEIS: Die Parameter für die Kalibrierung, einschließlich Schrittlänge, Anzahl der Schritte, Belichtungszeit der Kamera und Anzahl der Bilder pro Schritt, können auch hier angepasst werden. Etwa 105 Photonen können von einer fluoreszierenden Perle in einem einzigen Frame mit 0,03 s Belichtungszeiten erfasst werden, was zu xy,y,z Lokalisationsgenauigkeiten von ca. 1 nm führt. - Deaktivieren Sie die Laserbeleuchtung, indem Sie in der GUI auf 515 nm Laser schließen klicken. In HCImage Live unter Capture | Trigger-Modi, Geschwindigkeit und Registrierung,ändern Sie den Trigger-Modus zurück in Internal. Unter Sequenz | Die Scaneinstellungen ändern die Anzahl der Frameanzahl auf 200. Wählen Sie einen Speicher-Zielordner aus, indem Sie auf die Schaltfläche mit der Bezeichnung ...klicken. Klicken Sie auf Start, um 200 Bilder mit dunklen Bildern mit einer Belichtungszeit von 0,03 s zu sammeln.
HINWEIS: Selbst wenn kein Licht auf den Detektor fällt, liest jedes Pixel eine positive Zahl aus (als dunkler Offsetwert bezeichnet), die zwischen den Pixeln leicht variieren kann. Der dunkle Offsetwert kann sich im Laufe der Zeit ändern. Daher ist es notwendig, dunkle Rahmen für jede Kalibrierung zu sammeln. - Passen Sie den DHPSF mit einem Doppel-Gauß-Modell mit der Easy-DHPSF Software24 an, um die X- und Y-Positionen der Perle sowie eine Winkel-Gegen-Z-Kalibrierungskurve zu erhalten.
- Initialisieren Sie easy-DHPSF Software in MATLAB. Legen Sie unter Setup Kanal auf G fest und passen Sie die Fitting-Methode mit DG-Modellauf MLE . G bezieht sich auf die grüne Kanalkamera, da das fluoreszierende Protein, das für die Datenerfassung verwendet wird, mit grünen Wellenlängen emittiert. MLE mit DG-Modell bezieht sich auf Die maximale Wahrscheinlichkeitsschätzung mit Demassian-Modell.
HINWEIS: Die Pixelgröße und die Konvertierungsverstärkung hängen von der spezifischen optischen Einrichtung ab und müssen möglicherweise geändert werden. - Klicken Sie im Abschnitt DHPSF kalibrieren auf Ausführen. Klicken Sie im folgenden Popupfenster auf OK, um die Standardeinstellungen beizubehalten.
- Wählen Sie den in den Schritten 1.13-1.14 gespeicherten Bildstapel aus. Wählen Sie als Nächstes den Bildstapel mit dem dunklen Hintergrund aus, der in Schritt 1.15 gespeichert ist. Wählen Sie schließlich die .txt-Datei aus, die während Schritt 1.14 automatisch gespeichert wurde. Diese Datei enthält die Z-Position der Bühne während des gesamten Scanvorgangs.
- Wenn im nächsten Pop-up-Fenster der vollständige Kamerachip nicht für das Sichtfeld verwendet wurde, geben Sie die Startpositionen x0 und y0 auf dem Chip ein. Andernfalls finden Sie die Eingabe x0 = 1 und y0 = 1.Diese Informationen finden Sie in HCImage Live unter dem Abschnitt Binning und SubArrary.
- Ändern Sie im folgenden Fenster die Größe und Position des Feldes, das über dem Bild der fluoreszierenden Perlen angezeigt wird, so, dass es etwa 100 x 100 Pixel groß ist und über ein einzelnes fluoreszierendes DHPSF-Signal zentriert ist. Doppelklicken Sie dann, um fortzufahren.
HINWEIS: Die gewählte DHPSF sollte von den anderen DHPSF-Signalen isoliert werden und idealerweise die hellste Perle sein. - Klicken Sie in die Mitte des DHPSF-Signals, zwischen den beiden Lappen, und klicken Sie dann auf Enter. Das folgende Fenster zeigt eine vergrößerte Ansicht des ausgewählten DHPSF. Wählen Sie den Standort der Mitte des DHPSF genauer aus, indem Sie darauf klicken.
HINWEIS: Das Programm passt dann in die DHPSF und zeigt das Rohbild und die Rekonstruktion von der Passform an. Es gibt auch Vorlagenbilder aus der Z-Kalibrierung aus, die einem DHPSF-Querschnitt an verschiedenen Z-Positionen entsprechen. Diese werden später für die Anpassung der experimentellen Daten verwendet. Das Programm gibt die in jedem Frame geschätzte X-, Y- und Z-Position aus. In einem gut ausgerichteten optischen System sollten sich X und Y sehr wenig ändern (30 nm Abweichung), wenn sich die Z-Position ändert. Wenn die Ausgabevariation größer als 30 nm ist, sollte die Phasenmaske, die sich in der Fourier-Ebene des Bildgebungssystems befindet (Abbildung 1), neu ausgerichtet und die Schritte 1.9-1.16 wiederholt werden. - Speichern Sie die Easy-DHPSF GUI, indem Sie auf das Symbol Speichern in der oberen linken Ecke der GUI klicken. Dies kann später durch Klicken auf das Ladesymbol in der GUI geladen werden.
HINWEIS: Das Z-Kalibrierungsverfahren sollte an jedem Tag eines Experiments durchgeführt werden, um Ausrichtungsänderungen im Mikroskop zu berücksichtigen, die aufgrund von Temperaturschwankungen oder mechanischen Vibrationen aufgetreten sein können.
- Initialisieren Sie easy-DHPSF Software in MATLAB. Legen Sie unter Setup Kanal auf G fest und passen Sie die Fitting-Methode mit DG-Modellauf MLE . G bezieht sich auf die grüne Kanalkamera, da das fluoreszierende Protein, das für die Datenerfassung verwendet wird, mit grünen Wellenlängen emittiert. MLE mit DG-Modell bezieht sich auf Die maximale Wahrscheinlichkeitsschätzung mit Demassian-Modell.
2. Bakterielle Kulturzubereitung
- Bereiten Sie Kulturmedien vor, die das Wachstum von Bakterienzellen unterstützen. Für Y. enterocoliticaverwenden Sie 5 ml BHI (Brain Heart Infusion) Brühe, die Nalidixsäure (35 g/ml) und 2,6-Diaminopimelsäure (80 g/ml) enthält. Hier wird ein Y. enterocolitica Stamm verwendet, der das Protein YscQ mit dem fluoreszierenden Protein eYFP23markiert hat.
- Impfmedien mit Bakterienkulturen aus Gefriervorräten oder Plattenkulturen.
- Wachsen Sie die Kultur bei 28 °C mit Schütteln über Nacht.
- Verdünnen Sie eine kleine Menge (ca. 250 L) gesättigter Nachtkultur auf 5 ml mit frischen Kulturmedien.
- Wachsen Sie die Kultur bei 28 °C mit Schütteln für 60-90 min.
- Induzieren Sie die Expression von fluoreszierendem Fusionsprotein. Bei Y. enterocoliticaschockieren die Zellen in einem Wassershaker auf 37 °C, um das Yop-Regulon zu induzieren.
- Inkubieren Sie die Zellen für weitere 3 h bei 37 °C mit Schütteln.
- Zentrifuge 1 ml Kultur bei 5.000 x g für 3 min bei Raumtemperatur. Entsorgen Sie den Überstand.
- Waschen Sie das Pellet 3 mal mit 1 ml M2G-Medien.
- Setzen Sie die pelletierten Bakterien in M2G Media um 250 L wieder aus.
- Fügen Sie fluoreszierende Perlen als Treuhandmarker hinzu. Fluoreszierende Perlenlösung sollte in entsprechend verdünnten Mengen zugesetzt werden, so dass es nur 1-2 Perlen pro FOV gibt, wenn man sie im Mikroskop betrachtet.
- Sanft Pipette oder Wirbel die Suspension, um aggregierte Zellen zu trennen.
- Platte 1,5 l Aufhängung auf einem 1,5-2% Agarose-Pad aus M2G.
- Invertieren Sie das Agarose-Pad und legen Sie es auf einen ozongereinigten Mikroskopdeckel. Der Deckbedeckungsschlupf sollte 30 min in einen Ozonreiniger gelegt werden, um den inhärenten Fluoreszenzhintergrund zu reduzieren.
3. Datenerfassung
- Montieren Sie das Probenabdeckungsglas auf den Probenhalter eines invertierten Fluoreszenzmikroskops und sichern Sie es mit Klebeband oder federbelasteten Probenhalterclips an Ort und Stelle.
- Fügen Sie einen Tropfen Tauchöl auf das Mikroskopobjektiv, legen Sie dann den Probenhalter auf das Mikroskop und sichern Sie ihn an Ort und Stelle.
- Initialisieren Sie die grafische Benutzeroberfläche (GUI), um die Kamera, die Probenphase und die Anregungslaser des Mikroskops zu steuern. Hier wird kundenspezifische Software in MATLAB zur Gerätesteuerung eingesetzt.
- Initialisieren Sie die Kamerasoftware HCImage Live. Legen Sie auf der Registerkarte Aufnahme unter dem Abschnitt Kamerasteuerung die Belichtungszeit auf 0,025 s fest. Klicken Sie auf Live, um einen Live-Feed der Kamera zu starten.
- Passen Sie die X- und Y-Positionen der Mikroskopstufe an, indem Sie auf die XY-Pos-Pfeile unter dem Abschnitt Mikro-Positionierungsstufe der GUI klicken, um die Probe zu scannen und einen FOV mit einer entsprechend dichten Population von Bakterienzellen zu finden.
HINWEIS: Um den Datendurchsatz zu maximieren, sollten Zellen so dicht wie möglich sein, ohne sich zu überlappen oder Zellen zu berühren. Der FOV sollte auch mindestens 1 fluoreszierende Perle enthalten, die als Treuhandmarker verwendet werden soll, vorzugsweise in einer Ecke des FOV positioniert. - Passen Sie die Z-Position der Mikroskopstufe an, indem Sie auf die Z-Pos-Pfeile unter dem Abschnitt Nano-Positionierungsstufe der GUI klicken, sodass die DHPSF-Lappen der fluoreszierenden Perle vertikal sind.
- Unter Sequenz | Scaneinstellungen, ändern Sie die Anzahl der Frame-Zahlen auf 20.000. Wählen Sie einen Speicher-Zielordner aus, indem Sie auf die Schaltfläche mit der Bezeichnung ...klicken. Klicken Sie schließlich auf Start, um bis zu 20.000 Kamerabilder mit einer kurzen Belichtungszeit von 0,025 s zu sammeln. eYFP photoblinking wird mit hochintensivem Anregungslicht bei 514 nm19,25initiiert.
HINWEIS: Hier bei der ErstenBleiche und anschließenden Abbildung einzelner EYFP-Moleküle wird eine Laserintensität von 350 W/cm2 an der Brennebene verwendet. Die Photoaktivierung von eYFP-Molekülen bei UV-Wellenlängen während der Bildgebung wurde nicht verwendet. Es sollte höchstens ein Einzelnemolekülsignal pro Bakterienzelle geben. Wenn die Dichte des Einmolekülsignals zunächst zu hoch ist, leuchten Sie weiter, bis vor Beginn der Datenerfassung genügend Photobleichungen auftreten. - Deaktivieren Sie die Laserbeleuchtung, indem Sie in der GUI auf 515 nm Laser schließen klicken. Sammeln Sie 200 Bilder mit dunkler Belichtungszeit.
- Aktivieren Sie in der GUI das Kontrollkästchen neben Thorlabs LED, und klicken Sie auf Nach oben umschalten. Dadurch wird der Weg vom Fluoreszenzweg zum Phasenkontrastweg umgestellt.
- Initialisieren Sie die Datenerfassungssoftware IC Capture 2.4. Dadurch wird die Kamera im Phasenkontrastweg gesteuert. Drücken Sie die Schaltfläche Start/Stop Live Display, um einen Live-Feed von der Kamera anzuzeigen. Klicken Sie auf Capture | Bild speichern, um ein Phasenkontrastbild der Zellen im Sichtfeld zu sammeln.
- Wiederholen Sie die Schritte 3.5-3.10 für zusätzliche FOVs. Hier werden Daten aus 500 Bakterienzellen in zehn verschiedenen FOVs verwendet, um die Anzahl der für die Analyse verfügbaren Einzelmolekül-Trajektorien zu erhöhen.
VORSICHT: Zellen, die über einen längeren Zeitraum auf den Agarose-Pads montiert sind, verhalten sich möglicherweise anders als frisch montierte Zellen. Darüber hinaus kann das Agarose-Pad nach einiger Zeit seine Integrität verlieren, was sich negativ auf die Datenqualität auswirken kann. Typischerweise werden pro Probenschlitten höchstens 3 FOV (ca. 30 min am Mikroskop) verwendet.
4. Datenverarbeitung
HINWEIS: Eine modifizierte Version der Easy-DHPSF Software24 wird in MATLAB für die Analyse der rohen Kamerarahmen verwendet, um Einzelmolekül-Lokalisierungen zu extrahieren. Easy-DHPSF wird speziell für DHPSF-Lokalisierungen in die Einzelmolekül-Bildgebung eingesetzt. Benutzerdefinierte Änderungen wurden vorgenommen, um die Aufpasserungsroutine (Maximum Likelihood Estimation, MLE) zu implementieren, die die pixelabhängigen Rauscheigenschaften moderner sCMOS-Kamerasberücksichtigt 26. Es wurde auch geändert, um die Ausgabe des Bilddateityps aus dem HCImage Live-Programm (.dcimg) zu akzeptieren. Eine ausführlichere Erläuterung der Software und der einzelnen Schritte finden Sie unter Lew et al.24
- Initialisieren Sie die Easy-DHPSF GUI in MATLAB (Ergänzende Abbildung 2). Laden Sie die in Schritt 1.16.8 gespeicherte Datei.
- Bestimmen von Schwellenwerten für jede der 7 Vorlagen, die in Schritt 1.16.7 ausgegeben werden
- Klicken Sie im Abschnitt SM-Identifikation kalibrieren auf Ausführen. Klicken Sie in den folgenden beiden Popupfenstern auf OK, um die Standardeinstellungen beizubehalten.
- Öffnen Sie den Image-Stack, der die Daten des ersten FOV enthält, wenn Sie dazu aufgefordert werden.
- Wählen Sie eine kleine Auswahl an Frames aus, um Vorlagen zu entsprechen. Typischerweise werden Frames 1001-2000 verwendet, um dichte überlappende Signale in den ersten mehreren hundert Bildern zu vermeiden. Klicken Sie im folgenden Popupfenster auf OK, um die Standardeinstellungen beizubehalten. Klicken Sie auf Abbrechen, wenn Sie im folgenden Fenster zur Eingabe der Sequenzprotokolldatei aufgefordert werden.
- Öffnen Sie den Bildstapel mit dem dunklen Hintergrund, der in Schritt 3.8 gespeichert ist. KlickenSie im folgenden Popup-Fenster auf OK,, um die Parameter für die Hintergrundschätzung auf Standard festgelegt zu lassen. Standardmäßig wird der Hintergrund mit einem Medianfilter27 geschätzt, der 100 nachfolgende Kamerabilder um den aktuellen Frame abdeckt.
- Ändern Sie im nächsten Fenster die Größe des Auflagenfelds auf dem Bild, um den vollständigen FOV abzudecken, und doppelklicken Sie dann, um fortzufahren.
- Definieren Sie den Interessenbereich, indem Sie auf mehrere Punkte im Bild klicken, um ein Polygon zu erstellen. Der Interessenbereich sollte so viel wie möglich vom Sichtfeld umfassen und gleichzeitig sicherstellen, dass alle fluoreszierenden Perlen (sehr helle Objekte) im Bild nicht innerhalb des Polygons liegen, und dann doppelklicken, um fortzufahren.
HINWEIS: Die Software versucht dann, die Vorlagen mit dem Bild abzugleichen, und zeigt ein Bild mit möglichen Übereinstimmungen umkreist an. - Wenn die Software beendet wurde, werden viele Bilder gefundener Vorlagenübereinstimmungen gespeichert und der entsprechende Schwellenwert in einem vordefinierten Ordner angezeigt. Ein höherer Schwellenwert entspricht einer besseren Übereinstimmung. Untersuchen Sie für jede Vorlagennummer die Beispielübereinstimmungen, und bestimmen Sie den niedrigsten Schwellenwert, der ein Bild eines DHPSF aufweist. Geben Sie diese Schwellenwerte für jede der 7 Vorlagen unter dem Abschnitt SM-Identifikation kalibrieren in der Easy-DHPSF-GUI ein.
HINWEIS: Das Programm versucht automatisch, Schwellenwerte auszuwählen und einzugeben, diese sind jedoch oft unzuverlässig und sollten manuell überprüft werden. Schwellenwerte werden so gewählt, dass nur wenige echte Einmolekülsignale übersehen werden, aber die Anzahl der falsch positiven Kandidaten für die Anpassung bleibt rechnerisch überschaubar. - Speichern Sie die Easy-DHPSF GUI erneut, indem Sie auf das Symbol Speichern in der oberen linken Ecke klicken.
- Einbau der fluoreszierenden Perle im FOV zur Verwendung als Treuhandmarker
- Klicken Sie im Abschnitt Track-Treuhandprofile der Easy-DHPSF GUI auf Ausführen. Klicken Sie in den folgenden beiden Popupfenstern auf OK, um die Standardeinstellungen beizubehalten.
- Ziehen Sie die Box überlagert auf dem Bild und zentrieren Sie es über das DHPSF-Signal von der fluoreszierenden Perle, und doppelklicken Sie dann.
- Klicken Sie in die Mitte des DHPSF-Signals, in der Mitte zwischen den beiden Lappen, und drücken Sie dann enter. Klicken Sie auf Abbrechen, wenn Sie im folgenden Fenster zur Eingabe der Sequenzprotokolldatei aufgefordert werden.
HINWEIS: Die Software passt in alle Kamerarahmen in den DHPSF und zeigt das Rohbild und das rekonstruierte Bild an. Wenn die Software fertig ist, gibt sie Zahlen mit X-, Y- und Z-Positionen der fluoreszierenden Perle über die Dauer der Bildaufnahme aus. - Aktivieren Sie das Kontrollkästchen neben Treuhänder verwenden und speichern Sie die Easy-DHPSF GUI erneut, indem Sie auf das Symbol Speichern in der oberen linken Ecke klicken.
- Suchen und Passen Allerinschreibungen in allen Kamerabildern mithilfe der in Schritt 4.2 erhaltenen Vorlagenschwellenwerte.
- Klicken Sie im Abschnitt Lokalisieren von DHPSF-SMs der Easy-DHPSF-GUI auf Ausführen. Klicken Sie in den folgenden Popupfenstern auf OK, um die Standardeinstellungen beizubehalten. Klicken Sie auf Abbrechen, wenn Sie im folgenden Fenster zur Eingabe der Sequenzprotokolldatei aufgefordert werden.
HINWEIS: Die Software findet und passt den DHPSF mit einem Doppel-Gauß-Modell, wenn die Qualität der Übereinstimmung über dem benutzerdefinierten Schwellenwert liegt. Es zeigt das Rohbild mit Kreisen um Vorlagenübereinstimmungen sowie ein Bild der rekonstruierten DHPSF Passt. - Speichern Sie die Easy-DHPSF GUI erneut, indem Sie auf das Symbol Speichern in der oberen linken Ecke klicken.
- Klicken Sie im Abschnitt Lokalisieren von DHPSF-SMs der Easy-DHPSF-GUI auf Ausführen. Klicken Sie in den folgenden Popupfenstern auf OK, um die Standardeinstellungen beizubehalten. Klicken Sie auf Abbrechen, wenn Sie im folgenden Fenster zur Eingabe der Sequenzprotokolldatei aufgefordert werden.
- Anzeigen von Einzelmolekül-Lokalisierungen und Herausfiltern unerwünschter oder unzuverlässiger Lokalisierungen
- Klicken Sie im Abschnitt Ausgabe-DHPSF-SM-Lokalisierungen auf Ausgabe filtern.
- Klicken Sie in den folgenden drei Fenstern auf OK, um eine Interpolation der fiduzialen X-, Y- und Z-Positionen im Zeitverlauf durchzuführen. In den meisten Fällen sind die Standardoptionen ausreichend. Wenn die schwarze interpolierte Linie keine vernünftige Interpolation der roten Positionslinie widerspiegelt, ändern Sie die Interpolationsparameter im Popupfenster.
HINWEIS: Die interpolierte Linie wird zur Stufendriftkorrektur der Einzelmoleküllokalisierungen verwendet. - Öffnen Sie das entsprechende Phasenkontrastbild für den FOV, der analysiert wird, wenn Sie dazu aufgefordert werden. Klicken Sie in den folgenden beiden Popupfenstern auf OK, um die Standardeinstellungen beizubehalten.
- Ändern Sie in den folgenden beiden Popupfenstern die Filterwerte, um strengere oder nachsichtigere Lokalisierungsanforderungen für einzelne Moleküle zu ermöglichen, und klicken Sie dann auf OK.
- Ziehen oder ändern Sie in dem angezeigten Fenster das Feld, das auf den Bildern überlagert wird, um den gewünschten Interessenbereich anzuzeigen, und doppelklicken Sie, um fortzufahren.
- Eine 3D-Rekonstruktion von Einzelmolekül-Lokalisationen wird angezeigt. Verwenden Sie die Figur-Werkzeuge, um die Rekonstruktion zu manipulieren (drehen, zoomen, etc.). Klicken Sie auf Weiter, um ein weiteres Dialogfeld anzuzeigen, in dem sie fragt: MöchtenSie mit einem anderen Parametersatz neu plotieren?'. Wenn die Ergebnisse zufriedenstellend sind, klicken Sie auf Nein. Wenn dies nicht der Fall ist, klicken Sie auf Ja.
HINWEIS: Häufige Gründe für unbefriedigende Ergebnisse sind das Phasenkontrastbild, das nicht korrekt mit den Lokalisierungsdaten überlagert wurde, oder die anfänglichen Vorlagenschwellenwerte waren zu niedrig, wodurch viele falsch positive Lokalisierungen erstellt wurden. Wenn Ja ausgewählt wurde, kehrt die Software zu Schritt 4.5.4 zurück, damit neue Parameterwerte definiert werden können. Wenn Nein ausgewählt wurde, werden die Ergebnisse gespeichert.
5. Datennachbearbeitung
- Mit benutzerdefinierten Software in MATLAB, schneiden Sie das Phasenkontrastbild, so dass nur der Bereich, der Zellen enthält, die unter dem Fluoreszenzmikroskop abgebildet wurden, bleibt. Dieser Schritt ist notwendig, da das Phasenkontrastbild einen Bereich abdeckt, der viel größer ist als das Fluoreszenzbild. Das Zuschneiden des Bildes vereinfacht den nächsten Schritt.
- Segmentieren Einzelner Zellen durch Verarbeitung des Phasenkontrastbildes mit der Software OUFTI28 (Ergänzende Abbildung 3)
- Initialisieren Sie OUFTI in MATLAB. Laden Sie das zugeschnittene Phasenkontrastbild aus dem vorherigen Schritt, indem Sie auf Ladephaseklicken.
- Klicken Sie auf Datei, um einen Speicherort für die Ausgabedatei auszuwählen und zu benennen.
- Wählen Sie unabhängige Frames unter der Überschrift Erkennung und Analyse aus.
- Klicken Sie auf Parameter laden, um Parameter für die Zellerkennung zu laden. Beispiele für Parameter sind akzeptable Zellfläche, Zellenbreite und ein Zellaufteilungsschwellenwert.
HINWEIS: Alle diese Parameter sollten angepasst werden, um die Leistung für die verwendeten spezifischen Zellengrößen und Bildqualität enden zu können. Wichtig ist, dass der Algorithmusparameter auf Subpixel festgelegt werden sollte, um eine präzise Messung von Zellumrissen zu ermöglichen. - Klicken Sie auf Diesen Rahmen, um mit der Zellsegmentierung zu beginnen. Zellumrisse werden über dem Phasenkontrastbild angezeigt, wenn der Prozess abgeschlossen ist.
- Mithilfe der Steuerelemente unter dem Abschnitt Manuell teilen Sie Zellen, fügen Sie Zellen hinzu oder verfeinern Sie Zellumrisse, um Umrisse für Zellen zu erhalten, die während des automatisierten Prozesses ungenau segmentiert wurden.
- Geben Sie die Zellumrisse aus, indem Sie auf Analyse speichernklicken.
- Verwenden Sie in MATLAB kundenspezifische Software, um die im vorherigen Schritt erhaltenen Umrisse mit den Einmolekül-Lokalisierungen präzise zu überlagern. In den folgenden Unterschritten werden die Schritte der Software detailliert beschrieben.
- Wählen Sie manuell 5 Kontrollpunktpaare im Pop-up-Fenster aus, indem Sie die Position der Zellpole derselben fünf Zellen sowohl in den im vorherigen Schritt generierten Einzelmolekül-Lokalisierungsdaten als auch in den Zellumrissen grob schätzen und darauf klicken. Die Position des Zellpols kann grob geschätzt werden, indem man mental einen konvexen Rumpf um die einzelnen Molekül-Lokalisationen zieht, die zu einer Zelle gehören, und den Punkt der höchsten Krümmung auswählt (Ergänzende Abbildung 4).
- Generieren Sie eine 2D-affine Transformationsfunktion mit der cp2tform-Funktion in MATLAB und verwenden Sie sie, um eine grobe Überlagerung der Zellumrisse und der Einzelmolekül-Lokalisierungen zu erzeugen.
- Löschen Sie Zellen mit weniger als 10 Lokalisierungen, und entfernen Sie Zellen, die teilweise außerhalb des Sichtfelds positioniert sind. Löschen Sie manuell alle zusätzlichen unerwünschten Zellen im Popupfenster, indem Sie in die Zellumrisslinie klicken (Ergänzende Abbildung 5).
- Verwenden Sie den Massenmittelpunkt für alle verbleibenden Zellumrisse und Einzelmolekül-Lokalisierungen in ihnen, um einen größeren Satz von Kontrollpunktpaaren zu bilden, die 2D-Transformationsfunktion neu zu berechnen und sie zu verwenden, um eine endgültige Überlagerung der Zellumrisse und der Einzelmolekül-Lokalisierungen.
- Weisen Sie dieser Zelle Lokalisierungen zu, die sich innerhalb der Begrenzung einer Zellumrisslinie befinden. Verwerfen Sie alle Lokalisierungen, die sich nicht innerhalb einer Zellumrisslinie befinden (Abbildung 2a, Ergänzende Abbildung 6).
6. Einmolekül-Tracking
HINWEIS: Der folgende Abschnitt wird mit benutzerdefinierten Software in MATLAB abgeschlossen. In diesem Abschnitt werden die Schritte beschrieben, die die Software ausführt.
- Berechnen Sie für Lokalisierungen, die derselben Zelle und in nachfolgenden Kamerabildern zugewiesen sind, den euklidischen Abstand zwischen den Lokalisierungen. Wenn der Abstand zwischen den Lokalisierungen unter einem Schwellenwert von 2,5 m liegt, verknüpfen Sie die Lokalisierungen, indem Sie sie derselben einmolekularen Flugbahn zuweisen.
HINWEIS: Es ist wichtig, nur Lokalisierungen innerhalb einer einzelnen Zelle zu berücksichtigen, damit Lokalisierungen in benachbarten Zellen, die zufällig die räumlichen und zeitlichen Schwellenwertanforderungen erfüllen, nicht verknüpft sind. Als maximale Entfernung wurde der Schwellenwert von 2,5 m gewählt,in dem ein sehr schnelles Molekül (30 m 2/s) in der Dauer der Belichtungszeit (0,025 s) plus einem 20%-Puffer wandern konnte. - Entsorgen Sie Flugbahnen, die kürzer als 4 Lokalisierungen sind. Wenn zwei oder mehr Lokalisationen (d. h. zwei oder mehr Fluoreszenzstrahler) gleichzeitig in einer Zelle vorhanden sind, entsorgen Sie die zugehörigen Flugbahnen. Wenn Sie die Spurlänge auf mindestens 4 Lokalisierungen festlegen, können mehrere Entfernungsmessungen gemittelt werden, was zu einer genaueren Schätzung der Diffusionskoeffizienten führt.
- Berechnen Sie die mittlere quadrierte Verschiebung (MSD) für eine gegebene Flugbahn, indem Sie:
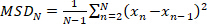 (1)
(1)
wobei N die Gesamtzahl der Lokalisierungen in der Flugbahn und xn die Position des Moleküls zum Zeitpunkt nist. - Berechnen Sie den scheinbaren Diffusionskoeffizienten D* nach
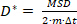 (2)
(2)
wobei m = 2 oder 3 die Dimensionalität der Messung ist und die Belichtungszeit der Kamera.
HINWEIS: Ein typisches Experiment erzeugt insgesamt 5.000-100.000 Flugbahnen, was zu einer scheinbaren Diffusionskoeffizientenverteilung mit so vielen Zählungen führt.
7. Monte-Carlo-Simulation der Brownschen Bewegung in einem begrenzten Volumen
ANMERKUNG: Erstellen Sie Bibliotheken simulierter scheinbarer Diffusionskoeffizientenverteilungen, indem Sie Monte-Carlo-Simulationen der Brownschen Bewegung durchführen, die auf ein zylindrisches Volumen beschränkt sind, indem 64 Werte im Bereich von 0,05–20 m2/s als Eingabeparameter (Software auf Anfrage bei den Autoren erhältlich). Dieser Bereich wurde ausgewählt, um den Bereich der zuvor geschätzten Diffusionskoeffizienten von fluoreszierenden (Fusions-)Proteinen in Bakterien abzudecken. 64 Diffusionskoeffizienten werden verwendet, um diesen Bereich ausreichend zubeproben. Dieser Abschnitt wird mit benutzerdefinierten Software in MATLAB durchgeführt und beschreibt die Schritte, die die Software automatisch ausführt. Die hier verwendeten stabförmigen Y. enterocolitica-Zellen werden durch ein zylindrisches Volumen der Länge l = 5 m und den Durchmesser d = 0,8 m angenähert.
- Initiieren Sie einzelne Flugbahnen an einer zufälligen Position im zylindrischen Volumen, und simulieren Sie ihre zufälligen (d. h. Brownian) Diffusionsschritte mit einem Zeitintervall von 100 ns (Zeitintervall sollte viel kürzer sein als die Belichtungszeit der Kamera, um eine ausreichende Abtasten der Position über den Verlauf eines Kamerarahmens). Probieren Sie jeden Verschiebungsschritt für einen Eingang D aus der entsprechenden Gaußschen Verteilungsfunktion durch Neuanordnung von Eqn. 2 und fügen Sie ihn der vorherigen Position hinzu:
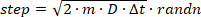 (3)
(3)
wobei die Randnfunktion in MATLAB eine Zufallszahl aus einer Normalverteilung abgibt. Wenn ein Schritt bewirkt, dass das Molekül außerhalb des Volumens des Zylinders verschoben wird, reflektieren Sie das Molekül in einem zufälligen Winkel zurück in den Zylinder. - Generieren Sie für jedes Zeitintervall ein DHPSF-Bild, das der momentanen x,y,z-Position des simulierten Emitters entspricht.
- Um experimentellen Bedingungen zu entsprechen, simulieren Sie Bilder mit 1.000 Photonen pro Lokalisation mit einem Laserhintergrund von 13 Photonen pro Pixel und Poisson-Rauschen. Fügen Sie außerdem einen dunklen Offset von 50 Photonen pro Pixel und Gaußsche Lesegeräusche (1,5 Photonen) hinzu, die mit experimentellen Kamerakalibrierungsmessungen übereinstimmen. Schließlich multiplizieren Sie das Bild mit der experimentell gemessenen pixelabhängigen Verstärkung der sCMOS-Kamera, um das Bild in Einheiten der Detektoranzahl zu erhalten.
ANMERKUNG: Nach diesen Manipulationen beträgt das Signal-Rausch-Verhältnis des endgültigen Bildes 2 . - Generieren Sie bewegungsverschwommene Bilder, die die sich ändernde Position der Moleküle widerspiegeln, indem 50 DHPSF-Bilder summiert werden, die während der Belichtungszeit simuliert werden, die während der experimentellen Datenerfassung verwendet wird. Um den Rechenaufwand zu begrenzen, wurden nur 50 periodisch abgetastete Positionen ausgewählt, um ein Bild zu erzeugen (anstelle aller 250.000 Positionen, die während einer Belichtungszeit von 0,025 s bei einem Abtastintervall von 100 ns abgetastet wurden).
HINWEIS: Die Länge (Anzahl der Frames) der simulierten Flugbahnen sollte der durchschnittlichen Länge der experimentellen Flugbahnen entsprechen. In diesem Fall beträgt die Anzahl der Frames pro Flugbahn 6.
- Um experimentellen Bedingungen zu entsprechen, simulieren Sie Bilder mit 1.000 Photonen pro Lokalisation mit einem Laserhintergrund von 13 Photonen pro Pixel und Poisson-Rauschen. Fügen Sie außerdem einen dunklen Offset von 50 Photonen pro Pixel und Gaußsche Lesegeräusche (1,5 Photonen) hinzu, die mit experimentellen Kamerakalibrierungsmessungen übereinstimmen. Schließlich multiplizieren Sie das Bild mit der experimentell gemessenen pixelabhängigen Verstärkung der sCMOS-Kamera, um das Bild in Einheiten der Detektoranzahl zu erhalten.
- Generieren Sie 5.000 Flugbahnen für jeden der 64 simulierten Eingangsdiffusionskoeffizienten.
- Analysieren Sie simulierte Kamerabilder, wie im Abschnitt Datenverarbeitung beschrieben.
- Verknüpfen Sie Lokalisierungen in Flugbahnen, wie im Abschnitt Single-Molecule Tracking beschrieben.
- Interpolieren Sie die kumulative Verteilungsfunktion (CDF) für jede simulierte Verteilung unter Verwendung der B-Spline-Interpolation der Ordnung 25. Interpolierte Verteilungen sind notwendig, damit sie an beliebigen Stellen abgefragt werden können.
- Interpolieren Sie die resultierenden B-Spline-Kurven entlang der D-Achse(D ist der wahre nicht begrenzte Diffusionskoeffizient, der die Bewegung der simulierten Emitter steuert) mit der streunendenInterpolant-Funktion in MATLAB (Angabe ' natural' Interpolationsmethode). Dies bietet eine kontinuierliche 2D-Funktion, von der aus eine scheinbare Diffusionskoeffizientenverteilung, die einem tatsächlichen Diffusionskoeffizientenwert im Bereich von 0,05-20 m2/sentspricht, abgefragt werden kann.
8. Experimentelle scheinbare Diffusionskoeffizientenverteilung
ANMERKUNG: Passen Sie experimentell gemessene Verteilungen der scheinbaren Diffusionskoeffizienten mithilfe linearer Kombinationen der simulierten Verteilungen an, die im vorherigen Abschnitt erzeugt wurden (Monte-Carlo-Simulation der Brownschen Bewegung in einem begrenzten Volumen). Dieser Abschnitt wird mit benutzerdefinierten Software in MATLAB durchgeführt und beschreibt die Schritte, die die Software automatisch ausführt. Weitere Informationen und Anwendungsbeispiele finden Sie unter Rocha et al.29
- Führen Sie eine eingeschränkte lineare Anpassung der kleinsten Quadrate (mit der lsqlin-Funktion in MATLAB) des experimentellen CDF mithilfe eines periodisch abgetasteten Arrays simulierter CDFs aus der in Abschnitt 7 erstellten Bibliothek aus. Die Ausgabe dieses Schritts ist ein Parametervektor, der die Diffusionskoeffizienten und Populationsfraktionen der vorherrschenden diffusiven Zustände in der experimentellen Verteilung enthält.
- Kombinieren Sie diffusive Zustände mit Diffusionskoeffizientenwerten innerhalb von 20 % voneinander in einem einzelnen diffusiven Zustand nach Gewichtsmittelung basierend auf dem relativen Bevölkerungsanteil. Dies ist der Startparametervektor.
ANMERKUNG: Um die Modellkomplexität zu reduzieren, können die diffusiven Zustände mit Diffusionskoeffizienten unter 0,5 m2/swährend aller folgenden Schritte konstant gehalten werden. - Erstellen Sie Arrays von Testanpassungsparametervektoren mit unterschiedlicher Anzahl diffusiver Zustände, die von einem einzelnen diffusiven Zustand bis zu einer benutzerdefinierten maximalen Anzahl von Zuständen reichen.
- Kombinieren Sie mit dem Anfangsparametervektor benachbarte diffusive Zustände durch gewichtete Mittelung und Aufteilung diffusiver Zustände in zwei Zustände mit gleichen Bevölkerungsfraktionen und Diffusionskoeffizienten 20 % über und unter dem ursprünglichen Wert. Wiederholen Sie dies für alle Zustandskombinations- und Splittingmöglichkeiten.
- Verwenden Sie jeden Parametervektor für die Versuchsanpassung, um ein nichtlineares kleinstes Quadrat von 5 separaten Teilmengen der Daten zu initialisieren (mit der fmincon-Funktion in MATLAB). Bestimmen Sie die Qualität der Anpassung, indem Sie die Restsumme der Quadrate zwischen der Anpassung und der Verteilung ermitteln, die den verbleibenden Teilmengen entspricht (Datenkreuzvalidierung).
- Verwenden Sie die durchschnittliche Restsumme der Quadrate der 5 separaten Fittings für jeden Versuchsvektor als Gesamtqualität der Anpassung, bestimmen Sie den Versuchsvektor mit der besten Qualität der Passung für jede Anzahl von diffusiven Zuständen.
- Bestimmen Sie die optimale Anzahl von Zuständen, indem Sie den Testvektor identifizieren, für den das Hinzufügen eines zusätzlichen Zustands nicht zu einer mindestens 5% Verbesserung der Anpassungsqualität führt.
- Verwenden Sie diesen Testvektor, um den nichtlinearen kleinsten Quadrate-Anpassung des vollständigen Datensatzes zu initialisieren.
- Schätzen Sie den Fehler für die einzelnen Parameter, indem Sie die Daten neu sampeln, indem Sie mit demselben Testvektor nachrüsten.
Ergebnisse
Unter den hier beschriebenen Experimentellen Bedingungen (20.000 Frames, Trajektollänge mindestens 4 Lokalisationen) und je nach Expressionsniveau der fluoreszierend markierten Fusionsproteine, ergeben ca. 200-3.000 Lokalisationen 10-150 Pro Zelle können Flugbahnen erzeugt werden (Abbildung 2a,b). Eine große Anzahl von Flugbahnen ist notwendig, um eine gut beprobte Verteilung der scheinbaren Diffusionskoeffizienten zu erzeugen. Die Größe...
Diskussion
Ein entscheidender Faktor für die erfolgreiche Anwendung des vorgestellten Protokolls ist es, sicherzustellen, dass einmolekulare Signale gut voneinander getrennt sind (d.h. sie müssen räumlich und zeitlich spärlich sein (Ergänzende Mov. 1)). Wenn es mehr als ein Fluoreszenzmolekül in einer Zelle gleichzeitig gibt, dann könnte die Lokalisierung fälschlicherweise der Flugbahn eines anderen Moleküls zugeordnet werden. Dies wird als Verknüpfungsproblem30bezeichnet. Experime...
Offenlegungen
Die Autoren haben nichts zu verraten.
Danksagungen
Wir danken Alecia Achimovich und Ting Yan für die kritische Lektüre des Manuskripts. Wir danken Ed Hall, Senior Staff Scientist in der Advanced Research Computing Services Gruppe an der University of Virginia, für die Hilfe bei der Einrichtung der Optimierungsroutinen, die bei dieser Arbeit verwendet werden. Die Finanzierung dieser Arbeit wurde von der University of Virginia bereitgestellt.
Materialien
| Name | Company | Catalog Number | Comments |
| 2,6-diaminopimelic acid | Chem Impex International | 5411 | Necessary for growth of Y. enterocolitica cells used. |
| 4f lenses | Thorlabs | AC508-080-A | f = 80mm, 2" |
| 514 nm laser | Coherent | Genesis MX514 MTM | Use for fluorescence excitation |
| agarose | Inivtrogen | 16520100 | Used to make gel pads to mount liquid bacterial sample on microscope. |
| ammonium chloride | Sigma Aldrich | A9434 | M2G ingredient. |
| bandpass filter | Chroma | ET510/bp | Excitation pathway. |
| Brain Heart Infusion | Sigma Aldrich | 53286 | Growth media for Y. enterocolitica. |
| calcium chloride | Sigma Aldrich | 223506 | M2G ingredient. |
| camera | Imaging Source | DMK 23UP031 | Camera for phase contrast imaging. |
| dielectric phase mask | Double Helix, LLC | N/A | Produces DHPSF signal. |
| disodium phosphate | Sigma Aldrich | 795410 | M2G ingredient. |
| ethylenediaminetetraacetic acid | Fisher Scientific | S311-100 | Chelates Ca2+. Induces secretion in the T3SS. |
| flip mirror | Newport | 8892-K | Allows for switching between fluorescence and phase contrast pathways. |
| fluospheres | Invitrogen | F8792 | Fluorescent beads. 540/560 exication and emission wavelengths. 40 nm diameter. |
| glass cover slip | VWR | 16004-302 | #1.5, 22mmx22mm |
| glucose | Chem Impex International | 811 | M2G ingredient. |
| immersion oil | Olympus | Z-81025 | Placed on objective lens. |
| iron(II) sulfate | Sigma Aldrich | F0518 | M2G ingredient. |
| long pass filter | Semrock | LP02-514RU-25 | Emission pathway. |
| magnesium sulfate | Fisher Scientific | S25414A | M2G ingredient. |
| microscope platform | Mad City Labs | custom | Platform for inverted microscope. |
| nalidixic acid | Sigma Aldrich | N4382 | Y. enterocolitica cells used are resistant to nalidixic acid. |
| objective lens | Olympus | 1-U2B991 | 60X, 1.4 NA |
| Ozone cleaner | Novascan | PSD-UV4 | Used to eliminate background fluorescence on glass cover slips. |
| potassium phosphate | Sigma Aldrich | 795488 | M2G ingredient. |
| Red LED | Thorlabs | M625L3 | Illuminates sample for phase contrast imaging. 625nm. |
| sCMOS camera | Hamamatsu | ORCA-Flash 4.0 V2 | Camera for fluorescence imaging. |
| short pass filter | Chroma | ET700SP-2P8 | Emission pathway. |
| Tube lens | Thorlabs | AC508-180-A | f=180 mm, 2" |
| Yersinia enterocolitica dHOPEMTasd | N/A | N/A | Strain AD4442, eYFP-YscQ |
| zero-order quarter-wave plate | Thorlabs | WPQ05M-514 | Excitation pathway. |
Referenzen
- Kapanidis, A. N., Uphoff, S., Stracy, M. Understanding Protein Mobility in Bacteria by Tracking Single Molecules. Journal of Molecular Biology. , (2018).
- Betzig, E., et al. Imaging intracellular fluorescent proteins at nanometer resolution. Science. 313 (5793), 1642-1645 (2006).
- Hess, S. T., Girirajan, T. P. K., Mason, M. D. Ultra-high resolution imaging by fluorescence photoactivation localization microscopy. Biophysical Journal. 91 (11), 4258-4272 (2006).
- Rust, M. J., Bates, M., Zhuang, X. W. Sub-diffraction-limit imaging by stochastic optical reconstruction microscopy (STORM). Nature Methods. 3 (10), 793-795 (2006).
- Chen, T. Y., et al. Quantifying Multistate Cytoplasmic Molecular Diffusion in Bacterial Cells via Inverse Transform of Confined Displacement Distribution. Journal of Physical Chemistry B. 119 (45), 14451-14459 (2015).
- Mohapatra, S., Choi, H., Ge, X., Sanyal, S., Weisshaar, J. C. Spatial Distribution and Ribosome-Binding Dynamics of EF-P in Live Escherichia coli. mBio. 8 (3), (2017).
- Stracy, M., et al. Single-molecule imaging of UvrA and UvrB recruitment to DNA lesions in living Escherichia coli. Nature Communications. 7, 12568 (2016).
- Persson, F., Lindén, M., Unoson, C., Elf, J. Extracting intracellular diffusive states and transition rates from single-molecule tracking data. Nature Methods. 10 (3), 265-269 (2013).
- Bakshi, S., Choi, H., Weisshaar, J. C. The spatial biology of transcription and translation in rapidly growing Escherichia coli. Frontiers in Microbiology. 6, 636 (2015).
- Mustafi, M., Weisshaar, J. C. Simultaneous Binding of Multiple EF-Tu Copies to Translating Ribosomes in Live Escherichia coli. mBio. 9 (1), (2018).
- Michalet, X., Berglund, A. J. Optimal diffusion coefficient estimation in single-particle tracking. Physical Review E. 85 (6), (2012).
- Michalet, X. Mean square displacement analysis of single-particle trajectories with localization error: Brownian motion in an isotropic medium. Physical Review E Statistical, Nonlinear, and Soft Matter Physics. 82 (4 Pt 1), 041914 (2010).
- Backlund, M. P., Joyner, R., Moerner, W. E. Chromosomal locus tracking with proper accounting of static and dynamic errors. Physical Review E Statistical, Nonlinear, and Soft Matter Physics. 91 (6), 062716 (2015).
- Stracy, M., et al. Live-cell superresolution microscopy reveals the organization of RNA polymerase in the bacterial nucleoid. Proceedings of the National Academy of Sciences of the United States of America. 112 (32), E4390-E4399 (2015).
- Plochowietz, A., Farrell, I., Smilansky, Z., Cooperman, B. S., Kapanidis, A. N. In vivo single-RNA tracking shows that most tRNA diffuses freely in live bacteria. Nucleic Acids Research. 45 (2), 926-937 (2017).
- Koo, P. K., Mochrie, S. G. Systems-level approach to uncovering diffusive states and their transitions from single-particle trajectories. Physical Review E. 94 (5-1), 052412 (2016).
- Uphoff, S., Reyes-Lamothe, R., Garza de Leon, F., Sherratt, D. J., Kapanidis, A. N. Single-molecule DNA repair in live bacteria. Proceedings of the National Academy of Sciences of the United States of America. 110 (20), 8063-8068 (2013).
- Bakshi, S., Bratton, B. P., Weisshaar, J. C. Subdiffraction-Limit Study of Kaede Diffusion and Spatial Distribution in Live Escherichia coli. Biophysical Journal. 101 (10), 2535-2544 (2011).
- Bakshi, S., Siryaporn, A., Goulian, M., Weisshaar, J. C. Superresolution imaging of ribosomes and RNA polymerase in live Escherichia coli cells. Molecular Microbiology. 85 (1), 21-38 (2012).
- Uphoff, S., Sherratt, D. J., Kapanidis, A. N. Visualizing Protein-DNA Interactions in Live Bacterial Cells Using Photoactivated Single-molecule Tracking. JoVE. (85), e51177 (2014).
- Pavani, S. R. P., Piestun, R. Three dimensional tracking of fluorescent microparticles using a photon-limited double-helix response system. Optics Express. 16 (26), 22048-22057 (2008).
- Pavani, S. R. P., et al. Three-dimensional, single-molecule fluorescence imaging beyond the diffraction limit by using a double-helix point spread function. Proceedings of the National Academy of Sciences of the United States of America. 106 (9), 2995-2999 (2009).
- Rocha, J. M., et al. Single-molecule tracking in live Yersinia enterocolitica reveals distinct cytosolic complexes of injectisome subunits. Integrative Biology. 10 (9), 502-515 (2018).
- Lew, M. D., von Diezmann, A. R. S., Moerner, W. E. Easy-DHPSF open-source software for three-dimensional localization of single molecules with precision beyond the optical diffraction limit. Protocol Exchange. , (2013).
- Biteen, J. S., et al. Super-resolution imaging in live Caulobacter crescentus cells using photoswitchable EYFP. Nature Methods. 5 (11), 947-949 (2008).
- Huang, F., et al. Video-rate nanoscopy using sCMOS camera-specific single-molecule localization algorithms. Nature Methods. 10 (7), 653-658 (2013).
- Hoogendoorn, E., et al. The fidelity of stochastic single-molecule super-resolution reconstructions critically depends upon robust background estimation. Scientific Reports. 4, 3854 (2014).
- Paintdakhi, A., et al. Oufti: An integrated software package for high-accuracy, high-throughput quantitative microscopy analysis. Molecular microbiology. 99 (4), 767-777 (2016).
- Rocha, J. M., Corbitt, J., Yan, T., Richardson, C., Gahlmann, A. Resolving Cytosolic Diffusive States in Bacteria by Single-Molecule Tracking. bioRxiv. , 483321 (2018).
- Lee, A., Tsekouras, K., Calderon, C., Bustamante, C., Presse, S. Unraveling the Thousand Word Picture: An Introduction to Super-Resolution Data Analysis. Chemical Reviews. 117 (11), 7276-7330 (2017).
- Los, G. V., et al. HaloTag: A Novel Protein Labeling Technology for Cell Imaging and Protein Analysis. ACS Chemical Biology. , (2008).
- Gautier, A., et al. An Engineered Protein Tag for Multiprotein Labeling in Living Cells. Chemistry & Biology. 15 (2), 128-136 (2008).
- Bisson-Filho, A. W., et al. Treadmilling by FtsZ filaments drives peptidoglycan synthesis and bacterial cell division. Science. 355 (6326), 739-743 (2017).
- Douglass, K. M., Sieben, C., Archetti, A., Lambert, A., Manley, S. Super-resolution imaging of multiple cells by optimised flat-field epi-illumination. Nature Photonics. 10 (11), 705-708 (2016).
- Zhao, Z., Xin, B., Li, L., Huang, Z. L. High-power homogeneous illumination for super-resolution localization microscopy with large field-of-view. Optics Express. 25 (12), 13382-13395 (2017).
- Yan, T., Richardson, C. J., Zhang, M., Gahlmann, A. Computational Correction of Spatially-Variant Optical Aberrations in 3D Single Molecule Localization Microscopy. bioRxiv. , 504712 (2018).
- Gahlmann, A., Moerner, W. E. Exploring bacterial cell biology with single-molecule tracking and super-resolution imaging. Nature Reviews Microbiology. 12 (1), 9-22 (2014).
- Liu, Z., Lavis, L. D., Betzig, E. Imaging live-cell dynamics and structure at the single-molecule level. Mol Cell. 58 (4), 644-659 (2015).
- Berglund, A. J. Statistics of camera-based single-particle tracking. Physical Review E. 82 (1), 011917 (2010).
- Parry, B. R., et al. The bacterial cytoplasm has glass-like properties and is fluidized by metabolic activity. Cell. 156 (1-2), 183-194 (2014).
- English, B. P., et al. Single-molecule investigations of the stringent response machinery in living bacterial cells. Proceedings of the National Academy of Sciences of the United States of America. 108 (31), E365-E373 (2011).
- Niu, L. L., Yu, J. Investigating intracellular dynamics of FtsZ cytoskeleton with photoactivation single-molecule tracking. Biophysical Journal. 95 (4), 2009-2016 (2008).
- Coquel, A. S., et al. Localization of protein aggregation in Escherichia coli is governed by diffusion and nucleoid macromolecular crowding effect. PLoS Computational Biology. 9 (4), e1003038 (2013).
- Nenninger, A., Mastroianni, G., Mullineaux, C. W. Size Dependence of Protein Diffusion in the Cytoplasm of Escherichia coli. Journal of Bacteriology. 192 (18), 4535-4540 (2010).
- Dix, J. A., Verkman, A. S. Crowding effects on diffusion in solutions and cells. Annual Review of Biophysics. 37, 247-263 (2008).
- Elliott, L. C., Barhoum, M., Harris, J. M., Bohn, P. W. Trajectory analysis of single molecules exhibiting non-brownian motion. Physical Chemistry Chemical Physics. 13 (10), 4326-4334 (2011).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten