È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Microscopia di tracciamento a singola molecola - Uno strumento per determinare gli stati diffusivi delle molecole citosoliche
In questo articolo
Riepilogo
La microscopia di localizzazione 3D a molecola singola viene utilizzata per sondare le posizioni spaziali e le traiettorie di movimento delle proteine fluorescenti etichettate nelle cellule batteriche viventi. Il protocollo sperimentale e di analisi dei dati descritto nel presente documento determina i comportamenti diffusivi prevalenti delle proteine citosoliche basate su traiettorie a singola molecola raggruppate.
Abstract
La microscopia di localizzazione a singola molecola sonda la posizione e i movimenti delle singole molecole nelle cellule viventi con decine di nanometri di risoluzione spaziale e temporale al millisecondo. Queste capacità rendono la microscopia di localizzazione a singola molecola ideale per studiare le funzioni biologiche a livello molecolare in ambienti fisiologicamente rilevanti. Qui, dimostriamo un protocollo integrato per l'acquisizione e l'elaborazione/analisi di dati di tracciamento a singola molecola per estrarre i diversi stati diffusive che una proteina di interesse può mostrare. Queste informazioni possono essere utilizzate per quantificare la formazione di complessi molecolari nelle cellule viventi. Forniamo una descrizione dettagliata di un esperimento di localizzazione 3D a singola molecola basato su telecamera, così come le successive fasi di elaborazione dei dati che producono le traiettorie delle singole molecole. Queste traiettorie vengono quindi analizzate utilizzando un quadro di analisi numerica per estrarre gli stati diffusi prevalenti delle molecole fluorescenti etichettate e l'abbondanza relativa di questi stati. Il quadro di analisi si basa su simulazioni stocastiche di traiettorie di diffusione browniana intracellulari che sono confinate spazialmente da una geometria cellulare arbitraria. Sulla base delle traiettorie simulate, le immagini a singola molecola grezze vengono generate e analizzate allo stesso modo delle immagini sperimentali. In questo modo, le limitazioni sperimentali di precisione e precisione, che sono difficili da calibrare sperimentalmente, sono esplicitamente incorporate nel flusso di lavoro di analisi. Il coefficiente di diffusione e le frazioni di popolazione relativa degli stati diffusi sono determinati adattando le distribuzioni di valori sperimentali utilizzando combinazioni lineari di distribuzioni simulate. Dimostriamo l'utilità del nostro protocollo risolvendo gli stati diffusivi di una proteina che presenta diversi stati diffusi dopo la formazione di complessi omo- ed etero-oligomerici nel citosol di un patogeno batterico.
Introduzione
L'esame del comportamento diffuso delle biomolecole fornisce informazioni sulle loro funzioni biologiche. Le tecniche basate sulla microscopia a fluorescenza sono diventate strumenti preziosi per osservare le biomolecole nel loro ambiente cellulare nativo. Il recupero della fluorescenza dopo la fotobleaching (FRAP) e la spettroscopia di correlazione della fluorescenza (FCS)1 forniscono comportamenti diffusischi con la media dell'insieme. Al contrario, la microscopia di localizzazione a singola molecola consente l'osservazione di singole molecole fluorescenti con alta risoluzione spaziale e temporale2,3,4. Osservare singole molecole è vantaggioso poiché una proteina di interesse può esistere in diversi stati diffusivi. Ad esempio, due stati diffusivi facilmente distinguibili sorgono quando un regolatore trascrizionale, come CueR in Escherichia coli, si diffonde liberamente nel citosol o si lega a una sequenza di DNA e diventa immobilizzato sulla scala cronologica della misurazione5 . Il tracciamento a singola molecola fornisce uno strumento per osservare direttamente questi diversi stati e non sono necessarie analisi sofisticate per risolverli. Tuttavia, diventa più difficile risolvere più stati diffusi e le loro frazioni di popolazione nei casi in cui i loro tassi di diffusione sono più simili. Ad esempio, a causa della dipendenza dalle dimensioni del coefficiente di diffusione, i diversi stati di oligomerizzazione di una proteina si manifestano come diversi stati diffusivi6,7,8,9 , 10. Tali casi richiedono un approccio integrato in termini di acquisizione, trattamento e analisi dei dati.
Un fattore critico che influenza i tassi diffusivi delle molecole citosoliche è l'effetto del confinamento attraverso il confine cellulare. Le restrizioni imposte al movimento molecolare da un confine cellulare batterico fanno sì che il tasso di diffusione misurato di una molecola citononica appaia più lenta rispetto a quando lo stesso movimento si fosse verificato in uno spazio non confinato. Per le molecole che si difcizzano molto lentamente, l'effetto del confinamento cellulare è trascurabile a causa della mancanza di collisioni con il confine. In questi casi, può essere possibile risolvere con precisione gli stati diffusivi adattando le distribuzioni di spostamenti molecolari, ro coefficienti di diffusione apparente, D ,utilizzando modelli analitici basati sulle equazioni per il moto browniano ( diffusione casuale)11,12,13. Tuttavia, per la rapida comfazione delle molecole citosoliche, le distribuzioni sperimentali non assomigliano più a quelle ottenute per il moto browniano non confinato a causa di collisioni di molecole che diffondevano i confini cellulari. Gli effetti di confinamento devono essere presi in considerazione per determinare con precisione i coefficienti di diffusione non confinati delle molecole fluorescenti. Recentemente sono stati sviluppati diversi approcci per tenere conto degli effetti di confinamento (semi-)analiticamente 5,14,15,16 o numericamente attraverso simulazioni Monte Carlo di Diffusione browniana6,10,16,17,18,19.
Qui, forniamo un protocollo integrato per la raccolta e l'analisi dei dati di microscopia di localizzazione a singola molecola con particolare attenzione al tracciamento a singola molecola. L'obiettivo finale del protocollo è quello di risolvere gli stati diffusivi di proteine citosoliche fluorescenti all'interno, in questo caso, cellule batteriche a forma di asta. Il nostro lavoro si basa su un protocollo precedente per il tracciamento a singola molecola, in cui una polimerasi di DNA, PolI, ha dimostrato di esistere in uno stato legato al DNA e non legato dall'analisi di diffusione20. Qui, espandiamo l'analisi di tracciamento a singola molecola a misurazioni 3D ed eseguiamo simulazioni computazionali più realistiche per risolvere e quantificare più stati diffusi contemporaneamente presenti nelle cellule. I dati vengono acquisiti utilizzando un microscopio a fluorescenza a super-risoluzione 3D costruito in casa che è in grado di determinare la posizione 3D degli emettitori fluorescenti mediante l'imaging con la funzione di diffusione del punto a doppia elica (DHPSF)21,22. Le immagini a singola molecola grezze vengono elaborate utilizzando un software scritto su misura per estrarre le localizzazioni 3D a singola molecola, che vengono poi combinate in traiettorie a singola molecola. Migliaia di traiettorie sono raggruppate per generare distribuzioni di coefficienti di diffusione apparente. In una fase finale, le distribuzioni sperimentali sono in sintonia con distribuzioni generate numericamente ottenute attraverso simulazioni Monte-Carlo di moto browniano in un volume limitato. Applichiamo questo protocollo per risolvere gli stati diffusidella proteina YscQ del sistema di secrezione di tipo 3 nella Yersinia enterocoliticavivente. A causa della sua natura modulare, il nostro protocollo è generalmente applicabile a qualsiasi tipo di esperimento di tracciamento a singola molecola o a particella singola in geometrie cellulari arbitrarie.
Protocollo
1. Calibrazione a doppia elica point-spread-function
NOTA: le immagini descritte in questa e nelle sezioni seguenti vengono acquisite utilizzando un microscopio a fluorescenza invertito personalizzato, come descritto in Rocha et al.23. La stessa procedura è applicabile a diverse implementazioni al microscopio progettate per la localizzazione di una singola molecola e la microscopia di tracciamento2,3,4. Tutto il software per l'acquisizione di immagini e l'elaborazione dei dati descritto in questo articolo è disponibile (https://github.com/GahlmannLab2014/Single-Molecule-Tracking-Analysis.git).
- Preparazione di cuscinetti agarose per il montaggio di campioni su vetrini di copertura da visualizzare al microscopio.
- Aggiungere l'1,5-2% peso basso punto di fusione agarose a 5 mL di buffer M2G (4,9 mM Na2HPO4, 3,1 mM KH2PO4, 7,5 mM NH4Cl, 0,5 mM MgSO4, 10 M FeSO4, 0,5 mM CaCl2 e 0,2 glucosio). Microonde per alcuni secondi fino a quando tutte le agarose si dissolvono. Non far bollire la soluzione.
- Lasciare raffreddare la soluzione di agarose per qualche minuto (2-3 min).
- Pipette 600 - L di soluzione agarose su uno scivolo di copertura di vetro (22 mm x 22 mm). Posizionare delicatamente un secondo coperchio di vetro sulla parte superiore dell'agarose. Questo crea un sottile tampone di gel di agarose (0,5 mm) tra i due coperchi di copertura in vetro.
- Lasciate che le pastiglie di agarose sedersi a solidificare per 20 dollari min.
- Separare delicatamente gli scivoli di copertura del vetro. Il cuscinetto di agarose si attaccherà a uno di loro.
- Utilizzando una lama di rasoio, tagliare il pad agarose in quattro sezioni quadrate di uguale dimensione. Ogni sezione quadrata può essere utilizzata per un singolo campione.
- Pipetta 1,5 - Soluzione di perline fluorescenti su un cuscinetto di agarose. La soluzione perline è una diluizione 1/100000 della soluzione stock in M2G (vedere Tabella dei materiali).
- Invertire il pad agarose e posizionare su uno scivolo di copertura di vetro che è stato pulito in un detergente per ozono per 30 min. Il tempo di pulizia viene scelto per eliminare eventuali molecole fluorescenti aderenti ai copricoperture.
- Montare il vetro di copertura del campione sul supporto del campione di un microscopio a fluorescenza invertito e fissarlo in posizione utilizzando nastri adesivi o clip portacampioni caricate a molla.
- Aggiungere una goccia di olio di immersione sull'obiettivo del microscopio.
- Posizionare il supporto del campione su un microscopio e fissarlo in posizione.
- Inizializzare l'interfaccia utente grafica (GUI) per controllare la fotocamera, lo stage campione e i laser di eccitazione del microscopio. In questo caso, il software scritto su misura in MATLAB viene utilizzato per il controllo dello strumento (Supplementary Figure 1).
- Inizializzare il software della fotocamera HCImage Live. Nella scheda Acquisizione nella sezione Controllo videocamera, impostate il tempo di esposizione su 0,03 s. Fare clic su Live per iniziare un feed live della videocamera.
- Accendere il laser facendo clic su Apri laser 514 nm sull'interfaccia GUI per eccitare le perline fluorescenti sul pad e visualizzare l'emissione di fluorescenza sulla fotocamera utilizzando la modalità live-stream (cioè nessun dato salvato su disco).
- Regolare le posizioni X e Y dello stage del microscopio facendo clic sulle frecce 'XY-Pos' nella sezione 'Stadio di micro-posizionamento' della GUI per posizionare almeno una perla fluorescente al centro del campo visivo (FOV). La dimensione del passo può essere modificata facendo clic sulla casella a discesa sotto le frecce.
- Regolare la posizione del microscopio facendo clic sulle frecce zo-pos nella sezione Nano-Position stage della GUI. Impostare l'orientamento della funzione DHPSF (Double-helix point-spread-) del tallone fluorescente in verticale. Questo orientamento verticale è definito come il punto di partenza della calibrazione z. La dimensione del passo può essere modificata facendo clic sulla casella a discesa sotto le frecce.
- In HCImage Live, in Acquisizione . Modalità di trigger, velocità e registrazione, modificare la modalità di trigger da Interno a Trigger livello esterno. Ciò consentirà alla GUI MATLAB di controllare la telecamera.
- In Sequenza Impostazioni scansione, modificare il numero di conteggi dei frame in 1200. Scegliere una cartella di destinazione di salvataggio facendo clic sul pulsante con l'etichetta .... Infine, fare clic su Avvia. Il numero di conteggi dei fotogrammi è impostato su 1200 in modo che sia possibile raccogliere 10 fotogrammi per ciascuno dei 120 gradi di posizione.
- Eseguire la scansione di una serie di posizioni di z (30 passi sopra e sotto la posizione iniziale in incrementi di 50 nm) e registrare 10 fotogrammi ad ogni passo utilizzando un tempo di esposizione di 0,03 s. Iniziare il processo automatizzato facendo clic su GO nella sezione l'interfaccia utente grafica.
NOTA: i parametri per la calibrazione, tra cui la lunghezza del passo, il numero di passi, il tempo di esposizione della fotocamera e il numero di fotogrammi per passo possono essere regolati anche qui. Circa 105 fotoni possono essere acquisiti da una perlina fluorescente in un singolo fotogramma utilizzando 0,03 s tempi di esposizione con conseguente x,y,z precisione di localizzazione di circa 1 nm. - Disattivare l'illuminazione laser facendo clic su Chiudi laser a 515 nm nella GUI. In HCImage Live, in Acquisizione . Modalità trigger, Velocità e Registrazione, modificare la modalità trigger in Interno. In Sequenza Le impostazioni di scansione modificano il numero di conteggi dei fotogrammi in 200. Scegliere una cartella di destinazione di salvataggio facendo clic sul pulsante con l'etichetta .... Fare clic su Avvia per raccogliere 200 fotogrammi di immagini scure con un tempo di esposizione di 0,03 s.
NOTA: anche in assenza di luce che cade sul rilevatore, ogni pixel leggerà un numero positivo (denominato valore di offset scuro), che può variare leggermente tra i pixel. Il valore di scostamento scuro può cambiare nel tempo. Pertanto, è necessario raccogliere cornici scure per ogni calibrazione. - Montare il DHPSF con un modello a doppia gaussia utilizzando il software Easy-DHPSF24 per ottenere le posizioni X e Y del tallone, nonché una curva di calibrazione angolo contro calibrazione.
- Inizializzare il software Easy-DHPSF in MATLAB. In Configurazione, impostare Canale su G e impostare Metodo di adattamento su MLE con modello DG. G si riferisce alla telecamera del canale verde, poiché la proteina fluorescente utilizzata per l'acquisizione dei dati emette lunghezze d'onda verdi. MLE con modello DG si riferisce alla stima della massima probabilità con il modello a doppia gaussiana.
NOTA: le dimensioni in pixel e il guadagno di conversione dipendono dalla configurazione ottica specifica e potrebbero dover essere modificati. - Nella sezione Calibrate DHPSF fare clic su Esegui. Fare clic su OK nella seguente finestra popup per mantenere le impostazioni predefinite.
- Selezionare la pila di immagini salvata nei passaggi 1.13-1.14. Quindi, selezionare la pila di immagini con lo sfondo scuro salvato nel passaggio 1.15. Infine, selezionare il file con estensione txt salvato automaticamente durante il passaggio 1.14. Questo file contiene la posizione z della fase durante il processo di scansione.
- Nella finestra pop-up successiva, se il chip della fotocamera completo non è stato utilizzato per il campo visivo, inserire le posizioni x0 e y0 iniziali sul chip. In caso contrario, l'input x0 è 1 e y0 - 1.Queste informazioni sono disponibili in HCImage Live nella sezione Binning and SubArrary.
- Nella finestra seguente, ridimensionare e riposizionare la casella visualizzata sopra l'immagine delle perline fluorescenti in modo che abbia una dimensione di circa 100 x 100 pixel e centrata su un singolo segnale DHPSF fluorescente. Quindi, fare doppio clic per continuare.
NOTA: il DHPSF scelto deve essere isolato dagli altri segnali DHPSF e idealmente essere il tallone più luminoso possibile. - Fare clic al centro del segnale DHPSF, tra i due lobi, quindi premere Invio. La finestra seguente mostra una visualizzazione ingrandita del DHPSF scelto. Più precisamente scegliere la posizione del centro del DHPSF cliccando.
NOTA: Il programma si adatta al DHPSF e visualizza l'immagine raw e la ricostruzione dall'adattamento. Emetterà anche immagini modello dalla calibrazione di z corrispondente a una sezione trasversale DHPSF in diverse posizioni. Questi saranno utilizzati in seguito per l'adattamento dei dati sperimentali. Il programma emetterà la posizione X, Y e s stimata in ogni fotogramma. In un sistema ottico ben allineato, X e Y dovrebbero cambiare pochissima (deviazione di 30 nm) man mano che cambia la posizione di z. Se la variazione di output è maggiore di 30 nm, la maschera di fase, situata nel piano di Fourier del sistema di imaging (Figura 1), deve essere riallineata e ripetere i passaggi 1.9-1.16. - Salvare la GUI Easy-DHPSF facendo clic sull'icona Salva nell'angolo in alto a sinistra della GUI. Questo può essere caricato in seguito facendo clic sull'icona Carica nella GUI.
NOTA: Ogni giorno di un esperimento deve essere condotta la procedura di calibrazione z per tenere conto dei cambiamenti di allineamento nel microscopio che potrebbero essersi verificati a causa di fluttuazioni di temperatura o vibrazioni meccaniche.
- Inizializzare il software Easy-DHPSF in MATLAB. In Configurazione, impostare Canale su G e impostare Metodo di adattamento su MLE con modello DG. G si riferisce alla telecamera del canale verde, poiché la proteina fluorescente utilizzata per l'acquisizione dei dati emette lunghezze d'onda verdi. MLE con modello DG si riferisce alla stima della massima probabilità con il modello a doppia gaussiana.
2. Preparazione della coltura batterica
- Preparare i mezzi di coltura a sostegno della crescita delle cellule batteriche. Per Y. enterocolitica, utilizzare 5 mL di BHI (Brain Heart Infusion) brodo contenente acido nalidissico (35 g/mL) e 2,6-diaminopimelico acido (80 g/mL). Qui, viene utilizzato un ceppo Y. enterocolitica che ha la proteina YscQ etichettata con la proteina fluorescente eYFP23.
- Inoclate i media con colture batteriche provenienti da scorte di congelatori o colture di placche.
- Far crescere la coltura a 28 gradi centigradi agitando durante la notte.
- Diluire una piccola quantità (250 dollari l) di cultura notturna satura a 5 mL utilizzando supporti di coltura fresca.
- Far crescere la coltura a 28 gradi centigradi con agitazione per 60-90 min.
- Indurre l'espressione della proteina di fusione fluorescente. Per Y. enterocolitica, il calore scuote le cellule a 37 gradi centigradi in uno shaker d'acqua per indurre il reguron yop.
- Incubare le cellule per un ulteriore 3 h a 37 s.C con agitazione.
- Centrifuga 1 mL di coltura a 5.000 x g per 3 min a temperatura ambiente. Scartare il super-attardato.
- Lavare il pellet 3 volte con 1 mL di supporti M2G.
- Sospendere nuovamente i batteri pelleta in 250 dollari ll di M2G Media.
- Aggiungere perline fluorescenti come marcatori fiduciari. La soluzione di perline fluorescenti deve essere aggiunta in quantità opportunamente diluite, in modo che ci siano solo 1-2 perline per FOV quando visualizzate al microscopio.
- Pipette o vortice le sospensioni per separare le cellule aggregate.
- Piastra 1,5 -L di sospensione su un pad agarose 1.5-2% realizzato con M2G.
- Invertire il cuscinetto di agarose e posizionarlo su uno scivolo di copertura del microscopio pulito con ozono. Lo slittamento di copertura deve essere collocato in un detergente per ozono per 30 min per ridurre qualsiasi sfondo di fluorescenza intrinseco.
3. Acquisizione dei dati
- Montare il vetro di copertura del campione sul supporto del campione di un microscopio a fluorescenza invertito e fissarlo in posizione utilizzando nastri adesivi o clip portacampioni caricate a molla.
- Aggiungere una goccia di olio di immersione sull'obiettivo del microscopio, quindi posizionare il supporto del campione al microscopio e fissarlo in posizione.
- Inizializzare l'interfaccia utente grafica (GUI) per controllare la fotocamera, lo stadio campione e i laser di eccitazione del microscopio. Qui, il software scritto su misura in MATLAB viene utilizzato per il controllo dello strumento.
- Inizializzare il software della fotocamera HCImage Live. Nella scheda Acquisizione nella sezione Controllo videocamera, impostate il tempo di esposizione su 0,025 s. Fare clic su Live per iniziare un feed live della videocamera.
- Regolare le posizioni X e Y dello stadio del microscopio facendo clic sulle frecce XY-Pos nella sezione Micro-Positioning Stage della GUI per eseguire la scansione intorno al campione e trovare un FOV con una popolazione opportunamente densa di cellule batteriche.
NOTA: per ottimizzare la velocità effettiva dei dati, le celle devono essere il più dense possibile, senza sovrapporre o toccare le celle. Il FOV dovrebbe inoltre includere almeno 1 perline fluorescente da utilizzare come marcatore fiduciario, preferibilmente posizionato in un angolo del FOV. - Regolare la posizione del microscopio facendo clic sulle frecce zo-pos sotto la sezione Nano-Position stage della GUI, in modo che i lobi DHPSF del tallone fluorescente siano verticali.
- In Sequenza Impostazioni scansione, modificare il numero di conteggi dei fotogrammi in 20.000. Scegliere una cartella di destinazione di salvataggio facendo clic sul pulsante con l'etichetta .... Infine, fare clic su Start per raccogliere fino a 20.000 fotogrammi della fotocamera utilizzando un breve tempo di esposizione di 0.025 s. eYFP photoblinking viene avviato utilizzando la luce di eccitazione ad alta intensità a 514 nm19,25.
NOTA: Qui, un'intensità laser di 350 W/cm2 sul piano focale viene utilizzata per lo sbiancamento iniziale e la successiva imaging di singole molecole EYFP. La fotoattivazione delle molecole eYFP a lunghezze d'onda UV durante l'imaging non è stata utilizzata. Dovrebbe esserci al massimo un segnale a singola molecola per cellula batterica. Se la densità del segnale a singola molecola è inizialmente troppo alta, continuare a illuminare fino a quando non si verifica un fotosbiancamento sufficiente prima di iniziare l'acquisizione dei dati. - Disattivare l'illuminazione laser facendo clic su Chiudi laser a 515 nm nella GUI. Raccogli 200 fotogrammi di immagini scure utilizzando lo stesso tempo di esposizione.
- Nella GUI, selezionare la casella accanto a Thorlabs LED e fare clic su Toggle Mirror Up. Questo cambierà il percorso dal percorso di fluorescenza al percorso di contrasto di fase.
- Inizializzare il software di acquisizione dati IC Capture 2.4. Questo controlla la fotocamera nel percorso di contrasto di fase. Premi il pulsante Avvia/Interrompi Live Display per visualizzare un feed live dalla fotocamera. Fare clic su Acquisizione . Salva immagine per raccogliere un'immagine a contrasto di fase delle celle nel campo visivo.
- Ripetere i passaggi da 3.5-3.10 per ulteriori FOV. In questo caso, i dati acquisiti da 500 cellule batteriche in dieci diversi FOV vengono utilizzati per aumentare il numero di traiettorie a singola molecola disponibili per l'analisi.
LEGGI: Le cellule montate sui cuscinetti di agarose per lunghi periodi di tempo possono comportarsi in modo diverso rispetto alle cellule appena montate. Inoltre, il pad agarose può perdere la sua integrità dopo qualche tempo, che può influenzare negativamente la qualità dei dati. In genere, al massimo 3 FOV (30 min al microscopio) vengono utilizzati per ogni vetrino campione.
4. Trattamento dei dati
NOTA: Una versione modificata del software Easy-DHPSF24 viene utilizzata in MATLAB per l'analisi dei fotogrammi della fotocamera raw per estrarre le localizzazioni a singola molecola. Easy-DHPSF è usato specificamente per adattarsi alle localizzazioni DHPSF nell'imaging a singola molecola. Sono state apportate modifiche personalizzate per implementare la routine di adattamento basata su MLE (Maximum Likelihood Estimation) che tiene conto delle caratteristiche di rumore dipendenti dai pixel delle moderne telecamere sCMOS26. È stato anche modificato per accettare l'output del tipo di file di immagine dal programma HCImage Live (.dcimg). Per una spiegazione più dettagliata del software e dei singoli passaggi, consultare Lew etal.
- Inizializzare la GUI Easy-DHPSF in MATLAB (Supplementary Figure 2). Caricare il file salvato nel passaggio 1.16.8.Load in the file saved in step 1.16.8.
- Determinazione dei valori di soglia per ognuno dei 7 modelli restituiti nel passaggio 1.16.7Determining threshold values for each of the 7 templates output in step 1.16.7
- Nella sezione Calibratura identificazione SM fare clic su Esegui. Fare clic su OK nelle due finestre popup seguenti per mantenere le impostazioni predefinite.
- Quando richiesto, aprire la pila di immagini contenente i dati del primo FOV.
- Scegliere una piccola gamma di cornici per abbinare i modelli. Tipicamente i fotogrammi 1001-2000 vengono utilizzati per evitare segnali sovrapposti densi nelle prime diverse centinaia di fotogrammi. Fare clic su OK nella seguente finestra popup per mantenere le impostazioni predefinite. Fare clic su Annulla quando viene richiesto il file di registro di sequenza nella finestra seguente.
- Aprire la pila di immagini con lo sfondo scuro salvato nel passaggio 3.8. Fare clic su 'OK' nella seguente finestra popup per lasciare i parametri per la stima in background impostati sul valore predefinito. L'impostazione predefinita prevede la stima dello sfondo utilizzando un filtro mediano27 che copre 100 fotogrammi successivi intorno al fotogramma corrente.
- Nella finestra successiva, ridimensiona la casella sovrapposta all'immagine per coprire l'intero FOV, quindi fai doppio clic per continuare.
- Definire l'area di interesse facendo clic su diversi punti dell'immagine per creare un poligono. La regione di interesse dovrebbe includere il più possibile il campo visivo, assicurando al contempo che le perline fluorescenti (oggetti molto luminosi) nell'immagine non si trovano all'interno del poligono, quindi fare doppio clic per continuare.
NOTA: il software tenterà quindi di abbinare i modelli all'immagine e visualizzerà un'immagine con possibili corrispondenze cerchiate. - Quando il software si è fermato, salverà molte immagini di corrispondenze del modello trovato e visualizzerà il valore di soglia corrispondente in una cartella predefinita. Un valore di soglia più alto corrisponde a una corrispondenza migliore. Per ogni numero di modello, esaminare le corrispondenze di esempio e determinare la soglia più bassa che presenta un'immagine di un DHPSF. Immettere queste soglie per ciascuno dei 7 modelli nella sezione Calibrate SM Identification nella GUI Easy-DHPSF.
NOTA: Il programma tenterà di scegliere automaticamente e di inserire soglie, tuttavia, queste sono spesso inaffidabili e dovrebbero essere controllate manualmente. Le soglie sono scelte in modo tale che pochi segnali a singola molecola vengano persi, ma il numero di candidati falsi positivi per il raccordo rimane computazionalmente gestibile. - Salvare nuovamente la GUI Easy-DHPSF facendo clic sull'icona Salva nell'angolo in alto a sinistra.
- Montaggio del tallone fluorescente nel FOV da utilizzare come marcatore fiduciario
- Nella sezione Track fiduciaries della GUI Easy-DHPSF, fare clic su Esegui. Fare clic su OK nelle due finestre popup seguenti per mantenere le impostazioni predefinite.
- Trascinare la casella sovrapposta all'immagine e centrarla sul segnale DHPSF dal tallone fluorescente, quindi fare doppio clic.
- Fare clic al centro del segnale DHPSF, nel punto medio tra i due lobi, quindi premere invio. Fare clic su Annulla quando viene richiesto il file di registro di sequenza nella finestra seguente.
NOTA: Il software si adatta al DHPSF in tutte le cornici della fotocamera e visualizza l'immagine raw e l'immagine ricostruita. Quando il software è terminato, emetterà cifre con le posizioni X,Y, e s del tallone fluorescente per tutta la durata dell'acquisizione dell'immagine. - Seleziona la casella accanto a Usa fiduciarie e salva di nuovo la GUI Easy-DHPSF facendo clic sull'icona Salva nell'angolo in alto a sinistra.
- Trova e adatta tutte le localizzazioni in tutti i fotogrammi della fotocamera utilizzando le soglie del modello ottenute nel passaggio 4.2.
- Nella sezione Localizzare Le SM DHPSF della GUI Easy-DHPSF, fare clic su Esegui. Fare clic su OK nelle seguenti finestre popup per mantenere le impostazioni predefinite. Fare clic su Annulla quando viene richiesto il file di registro di sequenza nella finestra seguente.
NOTA: Il software troverà e si adatta al DHPSF utilizzando un modello doppio-gaussiano se la qualità della partita è superiore alla soglia definita dall'utente. Visualizzerà l'immagine grezza con cerchi intorno alle corrispondenze del modello e un'immagine delle vestibilità DHPSF ricostruite. - Salvare nuovamente la GUI Easy-DHPSF facendo clic sull'icona Salva nell'angolo in alto a sinistra.
- Nella sezione Localizzare Le SM DHPSF della GUI Easy-DHPSF, fare clic su Esegui. Fare clic su OK nelle seguenti finestre popup per mantenere le impostazioni predefinite. Fare clic su Annulla quando viene richiesto il file di registro di sequenza nella finestra seguente.
- Visualizzazione delle localizzazioni a singola molecola e filtrazione delle localizzazioni indesiderate o inaffidabili
- Nella sezione Localizzazioni SM DHPSF output fare clic su Output filtro.
- Fare clic su OK nelle tre finestre seguenti per eseguire un'interpolazione delle posizioni fiduciarie X, Y e z nel tempo. Nella maggior parte dei casi, le opzioni predefinite sono sufficienti. Se la linea interpolata nera non riflette un'interpolazione ragionevole della linea di posizione rossa, modificate i parametri di interpolazione nella finestra pop-up.
NOTA: la linea interpolata viene utilizzata per correggere le localizzazioni a singola molecola. - Aprire l'immagine di contrasto di fase corrispondente per il FOV analizzato quando richiesto. Fare clic su OK nelle due finestre popup seguenti per mantenere le impostazioni predefinite.
- Nelle due finestre popup seguenti, modificare i valori del filtro per consentire requisiti di localizzazione a singola molecola più rigorosi o più indulgenti, quindi fare clic su OK.
- Nella finestra visualizzata, trascinare o ridimensionare la casella sovrapposta alle immagini per visualizzare l'area di interesse desiderata e fare doppio clic per continuare.
- Viene visualizzata una ricostruzione 3D delle localizzazioni a singola molecola. Utilizzare gli strumenti figura per manipolare la ricostruzione (rotazione, zoom, ecc). Fare clic su Continua per visualizzare un'altra finestra di dialogo in cui viene chiesto 'Desideri ritracciare con un setdi parametri diverso? '. Se i risultati sono soddisfacenti, fare clic su No. In caso contrario, fare clic su Sì.
NOTA: i motivi più comuni per i risultati insoddisfacenti includono l'immagine a contrasto di fase che non viene sovrapposta correttamente ai dati di localizzazione o le soglie iniziali del modello erano troppo basse creando molte localizzazioni false positive. Se è stato selezionato Sì, il software tornerà al passaggio 4.5.4 in modo che sia possibile definire nuovi valori di parametro. Se è stato selezionato No, i risultati verranno salvati.
5. Post-elaborazione dei dati
- Utilizzando un software scritto su misura in MATLAB, ritagliare l'immagine a contrasto di fase in modo che rimanga solo la regione che contiene le cellule che sono state immagini al microscopio a fluorescenza. Questo passaggio è necessario perché l'immagine a contrasto di fase copre un'area molto più grande dell'immagine a fluorescenza. Il ritaglio dell'immagine semplifica il passaggio successivo.
- Segmentare singole celle elaborando l'immagine a contrasto di fase con il software OUFTI28 (Figura supplementare 3)
- Inizializzare OUFTI in MATLAB. Caricare l'immagine a contrasto di fase ritagliata dal passaggio precedente facendo clic su Carica fase.
- Fare clic su File per scegliere e assegnare un nome a un percorso di salvataggio per il file di output.
- Selezionare Frame indipendenti sotto l'intestazione Rilevamento e analisi.
- Fare clic su Carica parametri per caricare i parametri per il rilevamento delle celle. Esempi di parametri includono l'area accettabile della cella, la larghezza della cella e una soglia di divisione delle celle.
NOTA: Tutti questi parametri devono essere regolati per massimizzare le prestazioni specifiche delle celle e della qualità dell'immagine in uso. È importante sottolineare che il parametro dell'algoritmo deve essere impostato su subpixel per consentire una misurazione precisa dei contorni delle celle. - Fare clic su Questo fotogramma per iniziare la segmentazione delle celle. I contorni delle celle appariranno sopra l'immagine di contrasto di fase al termine del processo.
- Utilizzando i controlli nella sezione Manuale, dividere le celle, aggiungere celle o perfezionare i contorni delle celle segmentate in modo impreciso durante il processo automatizzato.
- Eseguire l'output dei contorni delle celle facendo clic su Salva analisi.
- Utilizzare un software personalizzato in MATLAB per sovrapporre con precisione i contorni ottenuti nel passaggio precedente con le localizzazioni a singola molecola. I seguenti passaggi secondari illustrano in dettaglio i passaggi del software.
- Selezionare manualmente 5 coppie di punti di controllo nella finestra pop-up stimando approssimativamente e facendo clic sulla posizione dei poli cellulari delle stesse cinque celle sia nei dati di localizzazione a singola molecola che nei profili delle celle, generati nel passaggio precedente. La posizione del polo cellulare può essere stimata approssimativamente disegnando mentalmente uno scafo convesso intorno alle localizzazioni a singola molecola appartenenti a una cella e selezionando il punto di curvatura più alta (Figura supplementare 4).
- Generare una funzione di trasformazione affine 2D utilizzando la funzione cp2tform in MATLAB e utilizzarla per generare una sovrapposizione approssimativa dei contorni delle cellule e delle localizzazioni a singola molecola.
- Eliminare le celle contenenti meno di 10 localizzazioni e rimuovere le celle posizionate parzialmente all'esterno del campo di visualizzazione. Eliminare manualmente eventuali celle indesiderate aggiuntive nella finestra popup facendo clic all'interno del contorno della cella (Figura supplementare 5 ).
- Utilizzare il centro di massa per tutti i contorni di celle rimanenti e le localizzazioni a singola molecola al loro interno per formare un insieme più ampio di coppie di punti di controllo, ricalcolare la funzione di trasformazione 2D e utilizzarla per generare una sovrapposizione finale dei contorni delle celle e localizzazioni a singola molecola.
- Assegnare le localizzazioni che si trovano all'interno del contorno di una cella delineare a tale cella. Eliminare tutte le localizzazioni che non si trovano all'interno di alcuna struttura di cella (Figura 2a, Figura supplementare 6).
6. Tracciamento a singola molecola
NOTA: la sezione seguente viene completata utilizzando software personalizzato in MATLAB. Questa sezione descrive i passaggi eseguiti dal software.
- Per le localizzazioni assegnate alla stessa cella e nei fotogrammi successivi della videocamera, calcolate la distanza euclidea tra le localizzazioni. Se la distanza tra le localizzazioni è al di sotto di una soglia di 2,5 m, collegare le localizzazioni assegnandole alla stessa traiettoria a singola molecola.
NOTA: è importante considerare solo le localizzazioni all'interno di una singola cella, in modo che le localizzazioni nelle celle adiacenti che soddisfano i requisiti di soglia spaziale e temporale non siano collegate. La soglia di 2,5 m è stata scelta come distanza massima che una molecola molto veloce (30 m2/s) poteva percorrere per tutta la durata del tempo di esposizione (0,025 s) più un buffer del 20%. - Scarta traiettorie più brevi di 4 localizzazioni. Se due o più localizzazioni (cioè due o più emettitori fluorescianti) sono presenti contemporaneamente in una cella, eliminare le traiettorie associate. L'impostazione della lunghezza minima della traccia su 4 localizzazioni consente di calcolare la media di diverse misurazioni della distanza, producendo una stima più accurata dei coefficienti di diffusione.
- Calcolare lo spostamento medio al quadrato (MSD) per una determinata traiettoria:
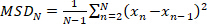 (1)
(1)
dove N è il numero totale di localizzazioni nella traiettoria e xn è la posizione della molecola nel punto temporale n. - Calcolare il coefficiente di diffusione apparente, d
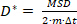 (2)
(2)
dove m è 2 o 3 è la dimensionalità della misura e il valore di z è il tempo di esposizione della fotocamera.
NOTA: Un esperimento tipico produrrà 5.000-100.000 dollari di traiettorie in totale, con conseguente distribuzione apparente coefficiente di diffusione con quel numero di conteggi.
7. Simulazione Monte-Carlo del moto browniano in un volume limitato
NOTA: Creare librerie di distribuzioni di coefficienti di diffusione apparente simulata eseguendo simulazioni Monte Carlo di movimento browniano confinate in un volume cilindrico, utilizzando 64 valori compresi nell'intervallo di 0,05-20m2/s come parametri di input (software disponibili su richiesta dagli autori). Questa gamma è stata scelta per coprire la gamma di coefficienti di diffusione stimati in precedenza delle proteine fluorescenti (fusione) nei batteri. 64 coefficienti di diffusione sono utilizzati per campionare sufficientemente questa gamma. Questa sezione viene eseguita utilizzando software su misura in MATLAB e descrive i passaggi eseguiti automaticamente dal software. Le cellule Y. enterocolitica a forma di asta utilizzate qui sono approssimate da un volume cilindrico di lunghezza l - 5 m e diametro d .
- Avviare singole traiettorie in una posizione casuale nel volume cilindrico e simularne le fasi casuali di diffusione (ad esempio Brownian) utilizzando un intervallo di tempo di 100 ns (l'intervallo di tempo dovrebbe essere molto più breve del tempo di esposizione della telecamera per consentire un tempo sufficiente la posizione nel corso di una cornice della fotocamera). Assaggia ogni passo di spostamento per un ingresso D dalla funzione di distribuzione gaussiana corrispondente ricomponendoe l'Eqn. 2 e aggiungilo alla posizione precedente:
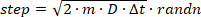 (3)
(3)
in cui la funzione randn in MATLAB campiona un numero casuale da una distribuzione normale. Se un passo fa sì che la molecola venga spostata all'esterno del volume del cilindro, riflettere la molecola all'interno del cilindro con un angolo casuale. - Per ogni intervallo di tempo, generare un'immagine DHPSF che corrisponde alla posizione istantanea x,y,z dell'emettitore simulato.
- Per soddisfare le condizioni sperimentali, simulare immagini contenenti 1.000 fotoni per localizzazione, con uno sfondo laser di 13 fotoni per pixel e il disturbo di Poisson. Inoltre, aggiungi l'offset scuro di 50 fotoni per pixel e il rumore di lettura gaussiano (1,5 fotoni), coerente con le misurazioni sperimentali della calibrazione della fotocamera. Infine, moltiplicare l'immagine per il guadagno dipendente dai pixel misurato sperimentalmente della fotocamera sCMOS per ottenere l'immagine in unità di conteggi del rivelatore.
NOTA: Dopo queste manipolazioni, il rapporto segnale-rumore dell'immagine finale è di 2 USD. - Genera immagini sfocate dal movimento che riflettano la mutevole posizione delle molecole sommando 50 immagini DHPSF simulate durante il tempo di esposizione utilizzato durante l'acquisizione di dati sperimentali. Per limitare le spese di calcolo, sono state scelte solo 50 posizioni campionate periodicamente per generare un'immagine (invece di tutte le 250.000 posizioni campionate durante un tempo di esposizione di 0,025 s ad un intervallo di campionamento di 100 ns).
NOTA: la lunghezza (numero di fotogrammi) delle traiettorie simulate deve corrispondere alla lunghezza media delle traiettorie sperimentali. In questo caso, il numero di fotogrammi per traiettoria è 6.
- Per soddisfare le condizioni sperimentali, simulare immagini contenenti 1.000 fotoni per localizzazione, con uno sfondo laser di 13 fotoni per pixel e il disturbo di Poisson. Inoltre, aggiungi l'offset scuro di 50 fotoni per pixel e il rumore di lettura gaussiano (1,5 fotoni), coerente con le misurazioni sperimentali della calibrazione della fotocamera. Infine, moltiplicare l'immagine per il guadagno dipendente dai pixel misurato sperimentalmente della fotocamera sCMOS per ottenere l'immagine in unità di conteggi del rivelatore.
- Genera 5.000 traiettorie per ciascuno dei 64 coefficienti simulati di diffusione dell'input.
- Analizzare i fotogrammi della fotocamera simulati come descritto nella sezione Elaborazione dati.
- Collegare le localizzazioni nelle traiettorie come descritto nella sezione Monitoraggio a singola molecola.
- Interpolare la funzione di distribuzione cumulativa (CDF) per ogni distribuzione simulata utilizzando l'interpolazione B-spline dell'ordine 25. Le distribuzioni interpolate sono necessarie per poter essere interrogate in punti arbitrari.
- Interpolare le curve B-spline risultanti lungo l'asse D(D è il vero coefficiente di diffusione non confinata che governa il movimento degli emettitori simulati) utilizzando la funzione scatteredInterpolant in MATLAB (specificare il ' metodo di interpolazione naturale). Ciò fornisce una funzione 2D continua da cui è possibile eseguire una query su qualsiasi distribuzione del coefficiente di diffusione apparente corrispondente a un valore di coefficiente di diffusione reale compreso nell'intervallo di 0,05-20 m2/s.
8. Raccordo di distribuzione coefficiente di diffusione apparente sperimentale
NOTA: Adattare distribuzioni misurate sperimentalmente di coefficienti di diffusione apparente utilizzando combinazioni lineari delle distribuzioni simulate generate nella sezione precedente (simulazione Monte-Carlo del moto browniano in un volume limitato). Questa sezione viene eseguita utilizzando software su misura in MATLAB e descrive i passaggi eseguiti automaticamente dal software. Per ulteriori informazioni ed esempi di applicazione, vedere Rocha etal.
- Eseguire un adattamento lineare limitato dei minimi quadrati (utilizzando la funzione lsqlin in MATLAB) del CDF sperimentale utilizzando una matrice campionata periodicamente di CF simulate dalla libreria creata nella sezione 7. L'output di questo passaggio è un vettore di parametro contenente i coefficienti di diffusione e le frazioni di popolazione degli stati diffusi prevalenti nella distribuzione sperimentale.
- Combinare gli stati diffusivi con i valori dei coefficienti di diffusione entro il 20% l'uno dell'altro in un unico stato diffuso in base alla media del peso in base alla frazione di popolazione relativa. Questo è il vettore di parametro iniziale.
NOTA: per ridurre la complessità del modello, gli stati diffusivi con coefficienti di diffusione inferiori a 0,5 m2/s possono essere mantenuti costanti durante tutti i passaggi seguenti. - Creare matrici di vettori di parametri di raccordo di prova con diversi numeri di stati diffusivi, che vanno da un singolo stato di diffusivo a un numero massimo di stati definito dall'utente.
- Utilizzando il vettore parametro iniziale, combinare gli stati diffusivi adiacenti attraverso la media ponderata e dividere gli stati diffusi in due stati con frazioni di popolazione uguali e coefficienti di diffusione del 20% sopra e sotto il valore originale. Ripetere l'operazione per tutte le possibilità di combinazione di stato e di suddivisione.
- Utilizzare ogni vettore di parametro di raccordo di prova per inizializzare un raccordo quadrato minimo non lineare di 5 sottoinsiemi separati di dati (utilizzando la funzione fmincon in MATLAB). Determinare la qualità dell'adattamento trovando la somma residua dei quadrati tra l'adattamento e la distribuzione corrispondente ai sottoinsiemi rimanenti (convalida incrociata dei dati).
- Utilizzare la somma residua media dei quadrati dei 5 raccordi separati per ogni vettore di prova come qualità complessiva della vestibilità, determinare il vettore di prova con la migliore qualità di adattamento per ogni numero di stati diffusivi.
- Determinare il numero ottimale di stati identificando il vettore di prova per il quale l'aggiunta di uno stato aggiuntivo non comporta un miglioramento di almeno il 5% della qualità della vestibilità.
- Utilizzare questo vettore di prova per inizializzare l'adattamento dei minimi quadrati non lineari del set di dati completo.
- Stimare l'errore per i singoli parametri ricampionando i dati eseguendo il bootstrap e il refitting con lo stesso vettore di prova.
Risultati
Nelle condizioni sperimentali qui descritte (20.000 fotogrammi, lunghezza minima di 4 localizzazioni) e a seconda dei livelli di espressione delle proteine di fusione fluorescenti, circa 200-3.000 localizzazioni che producono 10-150 le traiettorie possono essere generate per cella (Figura 2a,b). Un gran numero di traiettorie è necessario per produrre una distribuzione ben campionata di coefficienti di diffusione apparente. La dimensione del ...
Discussione
Un fattore critico per la corretta applicazione del protocollo presentato è quello di garantire che i segnali a singola molecola siano ben separati l'uno dall'altro (cioè, devono essere sparse nello spazio e nel tempo (Supplementary Mov. 1)). Se c'è più di una molecola fluorente in una cellula allo stesso tempo, la localizzazione potrebbe essere erroneamente assegnata alla traiettoria di un'altra molecola. Questo è indicato come il problema di collegamento30. Per evitare il p...
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Ringraziamo Alecia Achimovich e Ting Yan per la lettura critica del manoscritto. Ringraziamo Ed Hall, senior staff scientist del gruppo Advanced Research Computing Services presso l'Università della Virginia, per l'impostazione delle routine di ottimizzazione utilizzate in questo lavoro. I finanziamenti per questo lavoro sono stati forniti dall'Università della Virginia.
Materiali
| Name | Company | Catalog Number | Comments |
| 2,6-diaminopimelic acid | Chem Impex International | 5411 | Necessary for growth of Y. enterocolitica cells used. |
| 4f lenses | Thorlabs | AC508-080-A | f = 80mm, 2" |
| 514 nm laser | Coherent | Genesis MX514 MTM | Use for fluorescence excitation |
| agarose | Inivtrogen | 16520100 | Used to make gel pads to mount liquid bacterial sample on microscope. |
| ammonium chloride | Sigma Aldrich | A9434 | M2G ingredient. |
| bandpass filter | Chroma | ET510/bp | Excitation pathway. |
| Brain Heart Infusion | Sigma Aldrich | 53286 | Growth media for Y. enterocolitica. |
| calcium chloride | Sigma Aldrich | 223506 | M2G ingredient. |
| camera | Imaging Source | DMK 23UP031 | Camera for phase contrast imaging. |
| dielectric phase mask | Double Helix, LLC | N/A | Produces DHPSF signal. |
| disodium phosphate | Sigma Aldrich | 795410 | M2G ingredient. |
| ethylenediaminetetraacetic acid | Fisher Scientific | S311-100 | Chelates Ca2+. Induces secretion in the T3SS. |
| flip mirror | Newport | 8892-K | Allows for switching between fluorescence and phase contrast pathways. |
| fluospheres | Invitrogen | F8792 | Fluorescent beads. 540/560 exication and emission wavelengths. 40 nm diameter. |
| glass cover slip | VWR | 16004-302 | #1.5, 22mmx22mm |
| glucose | Chem Impex International | 811 | M2G ingredient. |
| immersion oil | Olympus | Z-81025 | Placed on objective lens. |
| iron(II) sulfate | Sigma Aldrich | F0518 | M2G ingredient. |
| long pass filter | Semrock | LP02-514RU-25 | Emission pathway. |
| magnesium sulfate | Fisher Scientific | S25414A | M2G ingredient. |
| microscope platform | Mad City Labs | custom | Platform for inverted microscope. |
| nalidixic acid | Sigma Aldrich | N4382 | Y. enterocolitica cells used are resistant to nalidixic acid. |
| objective lens | Olympus | 1-U2B991 | 60X, 1.4 NA |
| Ozone cleaner | Novascan | PSD-UV4 | Used to eliminate background fluorescence on glass cover slips. |
| potassium phosphate | Sigma Aldrich | 795488 | M2G ingredient. |
| Red LED | Thorlabs | M625L3 | Illuminates sample for phase contrast imaging. 625nm. |
| sCMOS camera | Hamamatsu | ORCA-Flash 4.0 V2 | Camera for fluorescence imaging. |
| short pass filter | Chroma | ET700SP-2P8 | Emission pathway. |
| Tube lens | Thorlabs | AC508-180-A | f=180 mm, 2" |
| Yersinia enterocolitica dHOPEMTasd | N/A | N/A | Strain AD4442, eYFP-YscQ |
| zero-order quarter-wave plate | Thorlabs | WPQ05M-514 | Excitation pathway. |
Riferimenti
- Kapanidis, A. N., Uphoff, S., Stracy, M. Understanding Protein Mobility in Bacteria by Tracking Single Molecules. Journal of Molecular Biology. , (2018).
- Betzig, E., et al. Imaging intracellular fluorescent proteins at nanometer resolution. Science. 313 (5793), 1642-1645 (2006).
- Hess, S. T., Girirajan, T. P. K., Mason, M. D. Ultra-high resolution imaging by fluorescence photoactivation localization microscopy. Biophysical Journal. 91 (11), 4258-4272 (2006).
- Rust, M. J., Bates, M., Zhuang, X. W. Sub-diffraction-limit imaging by stochastic optical reconstruction microscopy (STORM). Nature Methods. 3 (10), 793-795 (2006).
- Chen, T. Y., et al. Quantifying Multistate Cytoplasmic Molecular Diffusion in Bacterial Cells via Inverse Transform of Confined Displacement Distribution. Journal of Physical Chemistry B. 119 (45), 14451-14459 (2015).
- Mohapatra, S., Choi, H., Ge, X., Sanyal, S., Weisshaar, J. C. Spatial Distribution and Ribosome-Binding Dynamics of EF-P in Live Escherichia coli. mBio. 8 (3), (2017).
- Stracy, M., et al. Single-molecule imaging of UvrA and UvrB recruitment to DNA lesions in living Escherichia coli. Nature Communications. 7, 12568 (2016).
- Persson, F., Lindén, M., Unoson, C., Elf, J. Extracting intracellular diffusive states and transition rates from single-molecule tracking data. Nature Methods. 10 (3), 265-269 (2013).
- Bakshi, S., Choi, H., Weisshaar, J. C. The spatial biology of transcription and translation in rapidly growing Escherichia coli. Frontiers in Microbiology. 6, 636 (2015).
- Mustafi, M., Weisshaar, J. C. Simultaneous Binding of Multiple EF-Tu Copies to Translating Ribosomes in Live Escherichia coli. mBio. 9 (1), (2018).
- Michalet, X., Berglund, A. J. Optimal diffusion coefficient estimation in single-particle tracking. Physical Review E. 85 (6), (2012).
- Michalet, X. Mean square displacement analysis of single-particle trajectories with localization error: Brownian motion in an isotropic medium. Physical Review E Statistical, Nonlinear, and Soft Matter Physics. 82 (4 Pt 1), 041914 (2010).
- Backlund, M. P., Joyner, R., Moerner, W. E. Chromosomal locus tracking with proper accounting of static and dynamic errors. Physical Review E Statistical, Nonlinear, and Soft Matter Physics. 91 (6), 062716 (2015).
- Stracy, M., et al. Live-cell superresolution microscopy reveals the organization of RNA polymerase in the bacterial nucleoid. Proceedings of the National Academy of Sciences of the United States of America. 112 (32), E4390-E4399 (2015).
- Plochowietz, A., Farrell, I., Smilansky, Z., Cooperman, B. S., Kapanidis, A. N. In vivo single-RNA tracking shows that most tRNA diffuses freely in live bacteria. Nucleic Acids Research. 45 (2), 926-937 (2017).
- Koo, P. K., Mochrie, S. G. Systems-level approach to uncovering diffusive states and their transitions from single-particle trajectories. Physical Review E. 94 (5-1), 052412 (2016).
- Uphoff, S., Reyes-Lamothe, R., Garza de Leon, F., Sherratt, D. J., Kapanidis, A. N. Single-molecule DNA repair in live bacteria. Proceedings of the National Academy of Sciences of the United States of America. 110 (20), 8063-8068 (2013).
- Bakshi, S., Bratton, B. P., Weisshaar, J. C. Subdiffraction-Limit Study of Kaede Diffusion and Spatial Distribution in Live Escherichia coli. Biophysical Journal. 101 (10), 2535-2544 (2011).
- Bakshi, S., Siryaporn, A., Goulian, M., Weisshaar, J. C. Superresolution imaging of ribosomes and RNA polymerase in live Escherichia coli cells. Molecular Microbiology. 85 (1), 21-38 (2012).
- Uphoff, S., Sherratt, D. J., Kapanidis, A. N. Visualizing Protein-DNA Interactions in Live Bacterial Cells Using Photoactivated Single-molecule Tracking. JoVE. (85), e51177 (2014).
- Pavani, S. R. P., Piestun, R. Three dimensional tracking of fluorescent microparticles using a photon-limited double-helix response system. Optics Express. 16 (26), 22048-22057 (2008).
- Pavani, S. R. P., et al. Three-dimensional, single-molecule fluorescence imaging beyond the diffraction limit by using a double-helix point spread function. Proceedings of the National Academy of Sciences of the United States of America. 106 (9), 2995-2999 (2009).
- Rocha, J. M., et al. Single-molecule tracking in live Yersinia enterocolitica reveals distinct cytosolic complexes of injectisome subunits. Integrative Biology. 10 (9), 502-515 (2018).
- Lew, M. D., von Diezmann, A. R. S., Moerner, W. E. Easy-DHPSF open-source software for three-dimensional localization of single molecules with precision beyond the optical diffraction limit. Protocol Exchange. , (2013).
- Biteen, J. S., et al. Super-resolution imaging in live Caulobacter crescentus cells using photoswitchable EYFP. Nature Methods. 5 (11), 947-949 (2008).
- Huang, F., et al. Video-rate nanoscopy using sCMOS camera-specific single-molecule localization algorithms. Nature Methods. 10 (7), 653-658 (2013).
- Hoogendoorn, E., et al. The fidelity of stochastic single-molecule super-resolution reconstructions critically depends upon robust background estimation. Scientific Reports. 4, 3854 (2014).
- Paintdakhi, A., et al. Oufti: An integrated software package for high-accuracy, high-throughput quantitative microscopy analysis. Molecular microbiology. 99 (4), 767-777 (2016).
- Rocha, J. M., Corbitt, J., Yan, T., Richardson, C., Gahlmann, A. Resolving Cytosolic Diffusive States in Bacteria by Single-Molecule Tracking. bioRxiv. , 483321 (2018).
- Lee, A., Tsekouras, K., Calderon, C., Bustamante, C., Presse, S. Unraveling the Thousand Word Picture: An Introduction to Super-Resolution Data Analysis. Chemical Reviews. 117 (11), 7276-7330 (2017).
- Los, G. V., et al. HaloTag: A Novel Protein Labeling Technology for Cell Imaging and Protein Analysis. ACS Chemical Biology. , (2008).
- Gautier, A., et al. An Engineered Protein Tag for Multiprotein Labeling in Living Cells. Chemistry & Biology. 15 (2), 128-136 (2008).
- Bisson-Filho, A. W., et al. Treadmilling by FtsZ filaments drives peptidoglycan synthesis and bacterial cell division. Science. 355 (6326), 739-743 (2017).
- Douglass, K. M., Sieben, C., Archetti, A., Lambert, A., Manley, S. Super-resolution imaging of multiple cells by optimised flat-field epi-illumination. Nature Photonics. 10 (11), 705-708 (2016).
- Zhao, Z., Xin, B., Li, L., Huang, Z. L. High-power homogeneous illumination for super-resolution localization microscopy with large field-of-view. Optics Express. 25 (12), 13382-13395 (2017).
- Yan, T., Richardson, C. J., Zhang, M., Gahlmann, A. Computational Correction of Spatially-Variant Optical Aberrations in 3D Single Molecule Localization Microscopy. bioRxiv. , 504712 (2018).
- Gahlmann, A., Moerner, W. E. Exploring bacterial cell biology with single-molecule tracking and super-resolution imaging. Nature Reviews Microbiology. 12 (1), 9-22 (2014).
- Liu, Z., Lavis, L. D., Betzig, E. Imaging live-cell dynamics and structure at the single-molecule level. Mol Cell. 58 (4), 644-659 (2015).
- Berglund, A. J. Statistics of camera-based single-particle tracking. Physical Review E. 82 (1), 011917 (2010).
- Parry, B. R., et al. The bacterial cytoplasm has glass-like properties and is fluidized by metabolic activity. Cell. 156 (1-2), 183-194 (2014).
- English, B. P., et al. Single-molecule investigations of the stringent response machinery in living bacterial cells. Proceedings of the National Academy of Sciences of the United States of America. 108 (31), E365-E373 (2011).
- Niu, L. L., Yu, J. Investigating intracellular dynamics of FtsZ cytoskeleton with photoactivation single-molecule tracking. Biophysical Journal. 95 (4), 2009-2016 (2008).
- Coquel, A. S., et al. Localization of protein aggregation in Escherichia coli is governed by diffusion and nucleoid macromolecular crowding effect. PLoS Computational Biology. 9 (4), e1003038 (2013).
- Nenninger, A., Mastroianni, G., Mullineaux, C. W. Size Dependence of Protein Diffusion in the Cytoplasm of Escherichia coli. Journal of Bacteriology. 192 (18), 4535-4540 (2010).
- Dix, J. A., Verkman, A. S. Crowding effects on diffusion in solutions and cells. Annual Review of Biophysics. 37, 247-263 (2008).
- Elliott, L. C., Barhoum, M., Harris, J. M., Bohn, P. W. Trajectory analysis of single molecules exhibiting non-brownian motion. Physical Chemistry Chemical Physics. 13 (10), 4326-4334 (2011).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon