A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
دراسة ديناميكيات البروتين عبر التحليل الطيفي للصدى النيوتروني الدوار
In This Article
Summary
يصف هذا البروتوكول طرقا للتحقيق في بنية وديناميات اثنين من البروتينات النموذجية التي لها دور مهم في صحة الإنسان. تجمع هذه التقنية بين التوصيف الفيزيائي الحيوي على مقاعد البدلاء والتحليل الطيفي لصدى الدوران النيوتروني للوصول إلى الديناميكيات في مقاييس الوقت والطول ذات الصلة بحركات البروتين بين المجالات.
Abstract
يرتبط نشاط ووظائف معظم بروتينات جسم الإنسان بالتغيرات التكوينية للمجالات الفرعية بأكملها داخل البنية البلورية للبروتين. تبني الهياكل البلورية الأساس لأي عملية حسابية تصف بنية أو ديناميكيات البروتين ، في معظم الأحيان مع قيود هندسية قوية. ومع ذلك ، فإن هذه القيود من البنية البلورية غير موجودة في المحلول. قد تختلف بنية البروتينات في المحلول عن البلورة بسبب إعادة ترتيب الحلقات أو المجالات الفرعية على مقياس بيكو إلى نانو ثانية (أي النظام الزمني الداخلي لديناميكيات البروتين). يصف هذا العمل كيف يمكن الوصول إلى الحركات البطيئة على نطاقات زمنية لعدة عشرات من النانو ثانية باستخدام تشتت النيوترونات. وعلى وجه الخصوص، فإن التوصيف الديناميكي لبروتينين بشريين رئيسيين، وهو بروتين مضطرب في جوهره يفتقر إلى بنية ثانوية محددة جيدا وبروتين كلاسيكي للأجسام المضادة، يتم تناوله بواسطة التحليل الطيفي للصدى النيوتروني المغزلي (NSE) جنبا إلى جنب مع مجموعة واسعة من طرق التوصيف المختبرية. تم تحقيق المزيد من الأفكار حول ديناميكيات مجال البروتين باستخدام النمذجة الرياضية لوصف بيانات النيوترون التجريبية وتحديد التقاطع بين حركات البروتين المنتشرة والداخلية مجتمعة. إن استخراج المساهمة الديناميكية الداخلية في وظيفة التشتت الوسيطة التي تم الحصول عليها من NSE ، بما في ذلك الجدول الزمني للحركات المختلفة ، يسمح بمزيد من الرؤية في الخصائص الميكانيكية للبروتينات المفردة ونعومة البروتينات في بيئتها الطبيعية تقريبا في محلول البروتين المزدحم.
Introduction
استكشاف ديناميكيات المادة اللينة باستخدام النيوترونات
يعد التحقيق في الخصائص الديناميكية للبروتينات والببتيدات جزءا رئيسيا من الأبحاث الفيزيائية الحيوية ، وهناك العديد من الطرق المتطورة اليوم للوصول إلى مجموعة واسعة من المناظر الطبيعية للطاقة1. إن ربط ديناميكيات البروتينات المكتشفة تجريبيا بوظيفتها البيولوجية مهمة أكثر صعوبة بكثير ، حيث تتطلب نماذج رياضية معقدة ومحاكاة ديناميكية بمساعدة الكمبيوتر. تم التأكيد على أهمية التحليل الطيفي النيوتروني لتحليل حركات البروتين في العديد من الدراسات التي لاقت استحسانا جيدا والمعترف بها على نطاق واسع1،2،3،4،5. قبل استكشاف مشهد الطاقة المتنوع لديناميكيات البروتين الداخلية ، يلزم تقديم نظرة عامة قصيرة على العمليات الديناميكية في المادة اللينة وكيف يمكن للنيوترونات الوصول إليها.
إن حساسية النيوترونات للتكوين النظيري ونوع التفاعلات التي تعرضها مع المادة اللينة تجعل تشتت النيوترون أحد أكثر تقنيات التحقيق تنوعا6. هناك مجموعة واسعة من مقاييس طول الارتباط وأوقات الارتباط التي يمكن للنيوترونات الوصول إليها ، من الإثارة النووية والاهتزازات الذرية إلى الحركات الجماعية وعمليات الاسترخاء البطيئة مثل الدوران متساوي الخواص والحركات المنتشرة. عند التحقيق في النيوترونات المتناثرة لنقل الطاقة الخاصة بها ، يمكن التمييز بين ثلاثة تفاعلات رئيسية: التشتت المرن ، حيث لا يوجد تبادل للطاقة بين النيوترون الوارد والجسيمات في العينة. التشتت غير المرن ، مع تبادل طاقة كبير قابل للقياس الكمي بين النيوترون والجسيمات ؛ والحالة الغريبة للتشتت شبه المرن الذي يعين نقل طاقة صغير جدا مقارنة بالطاقة النيوترونية الحادثة 1,7. توفر هذه التفاعلات معلومات دقيقة حول المواد التي تم التحقيق فيها وتشكل الأساس النظري لمجموعة واسعة من تقنيات تشتت النيوترونات.
في التشتت المرن ، يسجل الكاشف اتجاهات النيوترونات كنمط حيود ، والذي يوضح موقع ذرات العينة بالنسبة لبعضها البعض. يتم الحصول على معلومات حول ارتباطات المواقع الذرية (أي الكثافة المتكاملة S(Q) فيما يتعلق بنقل الزخم Q ، والذي يتعلق بالمعلومات الهيكلية وحدها). يشكل هذا المبدأ أساس الحيود النيوتروني8.
ينشأ التعقيد عندما لا يكون نقل الطاقة صفرا بسبب الإثارة والتقلبات الداخلية في مادة العينة. وهذا يشكل أساس التحليل الطيفي النيوتروني، حيث يتم التحقيق في النيوترونات المتناثرة كدالة لكل من نقل الطاقة E ونقل الزخم Q. يتم الحصول على المعلومات الديناميكية والهيكلية. يقيس التحليل الطيفي النيوتروني نفس الكثافة المتكاملة S(Q) لنقل الطاقة (أي تغير سرعة النيوترونات بسبب تشتت العينة، S(Q,ω) = S(Q, E)، والذي يشار إليه أيضا باسم عامل البنية الديناميكية)9.
لحساب التشتت من مادة ما ، من الأنسب استخدام دالة ارتباط الزوج 7,10. في حالة الحيود ، تعطي دالة ارتباط الزوج الثابت G(r) احتمال إيجاد مركز جسيم على مسافة معينة r من مركز جسيم آخر. يعمم التحليل الطيفي دالة ارتباط الزوج الثابت ويتضمن الطاقة/التردد/الوقت في معادلة التشتت. تصبح دالة ارتباط الزوج G(r) دالة للوقت G(r, t) ، والتي يمكن أن تتحلل إلى دالة ارتباط زوج ذرة متميزة G D(r ، t) ، ودالة ارتباط ذاتي GS(r, t). تصف هذه نوعين من الارتباطات: الحركات المترابطة بين الأزواج للذرات التي تحكم التشتت المتماسك ، والارتباط الذاتي الذي يحكم التشتت غير المتماسك10.
التشتت المتماسك هو التشتت من "المتوسط" ويعتمد على المرحلة النسبية للموجات المتناثرة. في نظام التشتت صغير الزاوية ، تتداخل الموجات النيوترونية المتناثرة من مراكز تشتت مختلفة (ذرات مختلفة) بشكل بناء (لها مراحل مماثلة) ، وتلاحظ الحركة الجماعية للذرات مع تعزيز كثافة قوية. يصف التشتت المتماسك بشكل أساسي تشتت نيوترون واحد من جميع النوى في العينة10.
عندما لا يحدث أي تداخل بناء بين الموجات النيوترونية المتناثرة من مراكز مختلفة ، يتم اتباع ذرة واحدة في الوقت المناسب ، ويتم ملاحظة الارتباط الذاتي بين موضع الذرة في الوقت t = 0 ونفس الذرة في الوقت t. وبالتالي ، يتم فقدان المعلومات المتعلقة بالمواقع النسبية للذرات ، وينصب التركيز فقط على التقلبات المحلية. التشتت من التقلبات المحلية يحكم التشتت غير المتماسك. التشتت غير المتماسك متساوي الخواص ، ويساهم في إشارة الخلفية ، ويحط من الإشارة إلى الضوضاء10,11.
بالجمع بين كل ما سبق ، نميز أربع عمليات تشتت نيوترونية رئيسية10: (1) متماسكة مرنة (تقيس ارتباطات المواقع الذرية) ، (2) متماسكة غير مرنة (تقيس الحركات الجماعية للذرات) ، (3) مرنة غير متماسكة (تساهم في الخلفية ، وتقلل من شدة التشتت بواسطة عامل Debye-Waller (DWF) وتقيس عامل البنية المرنة غير المتماسكة (EISF) ، واصفا هندسة الحركات المنتشرة في الهندسة المحصورة ، و (4) غير متماسك غير مرن (يقيس ديناميكيات الذرة الواحدة والارتباط الذاتي).
تتراوح العمليات الديناميكية التي يمكن للنيوترونات الوصول إليها في علم الأحياء من تخميد الاهتزازات الذرية والجزيئية منخفضة التردد ، وتفاعل جزيئات المذيبات مع الأسطح الحيوية ، وعمليات الانتشار في طبقة الترطيب للجزيئات الكبيرة والهندسة المحصورة ، إلى الحركات المنتشرة الانتقالية والدورانية والهابطة قصيرة المدى ، ومجالات البروتين والحركات الألوستريكية1 . يعتمد التنوع الواسع في طرق وأدوات النيوترون لقياس ديناميكيات البروتين على كيفية تحقيق الشعاع النيوتروني الحادث أو الصادر وكيفية إجراء تحليل الطاقة للنيوترونات المتناثرة. من المحاور الثلاثية إلى وقت الطيران والتشتت الخلفي ومطياف الصدى المغزلي ، يمكن للمرء استكشاف العمليات الديناميكية ذات الأوقات المميزة بين 1 × 10-14 ثانية و 1 × 10-6 ثانية (الفيمتو ثانية إلى ميكروثانية)12.
يحتوي مختبر أوك ريدج الوطني ، بمصدريه النيوترونيين المشهورين ، مصدر نيوترون الانتشار - SNS13 ومفاعل تدفق النظائر العالية - HFIR14 ، على واحد من أفضل أجنحة أجهزة قياس الطيف للتحقيق في الديناميكيات في المواد الحيوية. بعض الأمثلة الأكثر بلاغة تشمل استخدام مطياف المروحية النيوترونية الباردة (CNCS) في SNS15 للتحقيق في الاضطراب الديناميكي لمياه الترطيب حول بروتين الفلورسنت الأخضر في المحلول 16 أو الاهتزازات الجماعية دون بيكو ثانية للعديد من البروتينات17. تتمثل إحدى المشكلات المتكررة لتحقيقات تشتت النيوترونات غير المرنة في أن بعض العمليات البيولوجية بطيئة للغاية بحيث لا يمكن ملاحظتها. بدون إعدادات متطرفة تؤدي إلى خسارة كبيرة في كثافة النيوترون ، تقتصر مطياف وقت الطيران على دقة طاقة 10 μeV ، المقابلة لمقياس زمني أقصى يبلغ ~ 200 ps10,11. هذا لا يكفي لمراقبة الحركات واسعة النطاق في البروتينات. لذلك ، غالبا ما تكون هناك حاجة إلى أدوات ذات دقة طاقة أعلى مثل مطياف التشتت الخلفي. وقد أثبت الجمع بين تقنيات وقت الطيران والتشتت الخلفي قوته في التحقيق في التغير في الديناميكيات الداخلية للسيتوكروم P450cam (CYP101)، وهو إنزيم يحفز كافور الهيدروكسيل18.
كان الانتشار المجهري الذي تم قياسه بواسطة مطياف التشتت الخلفي في SNS-BASIS19 محددا بشكل جيد بشكل مدهش ويمكن فصله إلى انتشار الماء (الترطيب ، السيتوبلازم ، والماء الشبيه بالكتلة) وانتشار مكونات الخلايا في الديدان المسطحة المستوية ، وهو أول حي يتم دراسته بواسطة تشتت النيوترون20 . التشتت الخلفي هو تقنية طيفية عالية الدقة ، ولكنه يقتصر أيضا على عدة μeV = عدة نانو ثانية ، في حين أن الديناميكيات البطيئة في المواد الحيوية تظهر أيضا كوقت بقاء للارتباط بين الموقع الذري أو اتجاهات الدوران (على سبيل المثال ، عمليات الاسترخاء ، والتي تحدث بانتظام في النطاق الزمني من عشرة إلى مئات من النانو ثانية).
التحليل الطيفي لصدى الدوران النيوتروني (NSE) هو تقنية التشتت النيوتروني الوحيدة التي تصل إلى هذه الدقة العالية. على عكس تقنيات النيوترونات الأخرى ، لا يتطلب NSE التدرج اللوني للحزمة لأنه يستخدم المرحلة الميكانيكية الكمومية للنيوترونات ، وهي لحظاتها المغناطيسية. يسمح التلاعب باللحظات المغناطيسية باستخدام توزيع الطول الموجي العريض لحزمة النيوترون ، في حين أن التقنية حساسة لتغيرات سرعة النيوترونات الصغيرة جدا في ترتيب 1 × 10-4. تم استخدام NSE بنجاح للتحقيق في الديناميكيات البطيئة للبروتينات في محلول للعديد من البروتينات. من بين هذه الدراسات الرائدة العديدة ، نعترف بدراسة المرونة القطاعية للغلوبولين المناعي للخنزير21 ؛ حركات المجال المقترن في Taq polymerase22 ؛ حركات المجال في رباعي نازعة هيدروجيناز الكحول الخميرة23 ؛ تغيير التشكيل في كيناز الفوسفوغليسيرات عند ربط الركيزة3 ؛ تنشيط حركات المجال والانتشار الديناميكي للإشارات الألوستريك في بروتين العامل المساعد التنظيمي 1 (NHERF1)لتبادل Na + / H (NHERF1) 4،24،25 ؛ ديناميات الحالة المدمجة لاختزال أيون الزئبق26 ؛ وانتشار الهيموجلوبين في خلايا الدم الحمراء27. كشفت دراستان حديثتان في ديناميكيات البروتين عن مرونة الجسم المضاد البشري الغلوبولين المناعي G (IgG) كربيع إنتروبي28 وخصائص مساهمة المذيبات في ديناميكيات بروتين المايلين الأساسي المضطرب جوهريا (MBP)5.
تشرح هذه المقالة المبادئ الأساسية ل NSE ، والطرق التحضيرية المتعددة الموصى بها لإجراء تحقيق شامل في ديناميكيات البروتين ، بالإضافة إلى المنهجية والبروتوكول التجريبي للحصول على بيانات NSE في مطياف NSE في SNS ، SNS-NSE. يميز البروتوكول بروتينين: IgG ، وهو بروتين جسم مضاد بشري منتظم ، والبروتين المضطرب جوهريا MBP. وتناقش بإيجاز الآثار البيوفيزيائية، والأهمية البحثية للأمثلة، والقيود المفروضة على هذه التقنية.
التحليل الطيفي NSE ، طريقة قياسات الديناميكيات البطيئة
NSE هي تقنية مستقطبة تستخدم وقت الطيران النيوتروني لقياس تبادل الطاقة (فقدان الاستقطاب) بسبب التفاعل شبه المرن بين النيوترونات والذرات في العينة. في قلب التحليل الطيفي NSE يكمن مبدأان أساسيان: (1) قدرة الدوران النيوتروني على التفوق في المجال المغناطيسي بتردد يتناسب مع القوة 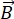 المغناطيسية ، أي تردد Larmor29 ، و (ب) صدى الدوران أو صدى هان ، الذي يمثل التلاعب وإعادة تركيز إشارة الاستقطاب عند تطبيق سلسلة من نبضات الترددات الراديوية30.
المغناطيسية ، أي تردد Larmor29 ، و (ب) صدى الدوران أو صدى هان ، الذي يمثل التلاعب وإعادة تركيز إشارة الاستقطاب عند تطبيق سلسلة من نبضات الترددات الراديوية30.
يمكن تلخيص أساسيات عملية NSE في بضع خطوات بسيطة 6,11 باستخدام الشكل 1. (1) يتم استقطاب الحزمة النيوترونية التي ينتجها المصدر (الموضع 1) (الموضع 2) ، وتوجيهها ، ونقلها (الموضع 3) ، وتصل إلى مدخل مطياف NSE ، حيث يتم تدويرها بمقدار 90 درجة بواسطة الزعانف النصف الأول من pi-half (الموضع 4). (2) يصبح الشعاع المستقطب (على سبيل المثال ، اللحظات المغناطيسية النيوترونية) عموديا على خطوط المجال المغناطيسي للمغناطيس الأول (منطقة الركود الأولى ، الموضع 5) ويبدأ في الاستباق. (3) في نهاية المغناطيس ، تتراكم الدوران النيوتروني زاوية ركود معينة تتناسب مع قوة المجال المغناطيسي ووقت الطيران الذي يقضيه في الداخل (يتناسب عكسيا بشكل أساسي مع سرعة النيوترون). يتم ترميز السرعات النيوترونية الفردية داخل زاوية الركود الخاصة بها في نهاية منطقة الركود الأولى. (4) بالقرب من موضع العينة ، يعكس pi-flipper (الموضع 6) اتجاه الدوران بمقدار 180 درجة ، مما يغير علامة زاوية الركود. (5) تتفاعل النيوترونات مع جزيئات العينة (الموضع 7) وتتناثر. (6) تدخل النيوترونات المتناثرة وتسبقها في منطقة الركود الثانية (الموضع 8) ولكنها تصبح معكوسة المنحى. (7) يستخدم زعانف نصف pi أخرى (الموضع 9) لتدوير اتجاه الدوران من الاتجاه العمودي إلى الاتجاه الأفقي. سيؤدي ذلك إلى إيقاف الركود ، وترجمة زاوية الركود φ إلى استقطاب يتناسب مع cos (φ). (8) يختار المحلل (الموضع 10) النيوترونات بناء على اتجاه واحد. إذا كان التفاعل مع العينة مرنا، فلن تتغير سرعة النيوترون. ستقضي النيوترونات قدرا مماثلا من الوقت في الطيران في منطقتي الركود الأولى والثانية ، ويتم استرداد زوايا الركود المتراكمة بالكامل. يتم استعادة الاستقطاب الكامل على الكاشف (الموضع 11) كصدى للاستقطاب الأصلي (أي صدى الدوران). (9) ومع ذلك ، في NSE ، يكون التشتت شبه مرن ، لذا فإن تبادل الطاقة الصغير بين النيوترونات وجزيئات العينة يؤدي إلى سرعات نيوترونية مختلفة بعد تشتت العينة. نظرا للسرعات المختلفة ، ستقضي النيوترونات وقتا إضافيا في الطيران عبر منطقة الركود الثانية ولن تكون قد استعادت زاوية الركود بشكل صحيح. يتم استرداد الاستقطاب الجزئي على الكاشف ، ويتناسب فقدان الاستقطاب بسبب استرخاء الدوران مع تحويل كوس-فورييه للدالة الطيفية S(Q ، ω) ، دالة التشتت الوسيطة F (Q ، t). (10) تتناسب المعلمة الزمنية للدالة F(Q, t) مع قوة المجال المغناطيسي للركود. وبالتالي ، فإن مسح فقدان الاستقطاب كدالة لقوة المجال المغناطيسي ينتج عنه وظيفة استرخاء تعتمد على العمليات الديناميكية داخل العينة.

الشكل 1: صورة فوتوغرافية لمقياس الطيف NSE في SNS (SNS-NSE) ومخطط مسار الذبابة النيوترونية مع أهم المكونات الوظيفية. من اليمين إلى اليسار: 1 = مصدر النيوترون. 2 = المروحيات-بندر-المستقطب-نظام الغالق الثانوي; 3 = أدلة النقل شعاع; 4 = pi / 2 زعانف لأول 90 درجة دوران ؛ 5 = منطقة الركود الأولى; 6 = pi flipper ل 180 ° دوران الدوران ؛ 7 = منطقة العينة وبيئة العينة (هنا ، يتم عرض فرن التبريد) ؛ 8 = منطقة الركود الثانية ؛ 9 = pi / 2 زعانف للدوران الثاني 90 درجة ؛ 10 = محلل ؛ 11 = كاشف. (لاحظ أن أجزاء من 3 ، وكذلك 2 و 1 ، تقع خلف الجدار الأزرق داخل التدريع ؛ يتم استبدال المروحيات بمحدد السرعة ل NSE القائم على المفاعل). يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
Protocol
يميز العمل الحالي بروتينين: بروتين الأجسام المضادة البشرية العادية IgG ، و MBP المضطرب في جوهره. تم الحصول على الشكل المجفف بالتجميد للبروتينات من مصادر تجارية (انظر جدول المواد).
1. إعداد عينة البروتين
- قم بإعداد فوسفات الصوديوم 50 mM + 0.1 M NaCl العازلة عن طريق وزن وإذابة الكواشف الصلبة المعنية في الماء الثقيل (D2O) (انظر جدول المواد). هذا هو المذيب العازل المحايد ل IgG.
- اضبط الرقم الهيدروجيني لمحلول المخزن المؤقت إلى 6.6.
- قم بإعداد فوسفات الصوديوم 20 mM + 6 M اليوريا العازلة عن طريق وزن وإذابة المكونات الصلبة المعنية في الماء الثقيل (D2O). هذا هو المذيب العازل المحايد ل MBP.
- اضبط الرقم الهيدروجيني لمحلول المخزن المؤقت إلى 4.7.
- قم بتصفية المذيبات العازلة باستخدام مرشحات بحجم المسام 0.2 ميكرومتر (انظر جدول المواد).
- وزن وإذابة مساحيق البروتين النقي المجفف بالتجميد في المذيبات المخصية للبروتينات المعنية (الخطوات 1.1.-1.2) بتركيز بروتين عال (~ 50 ملغ / مل).
- قم بتحميل محلول البروتين في سلال الدياليز بأغشية غسيل الكلى من 3.5 K MWCO ، وادلل ضد المخزن المؤقت المصفى لمدة 24 ساعة عند 10 درجات مئوية ل MBP و 25 درجة مئوية ل IgG (انظر جدول المواد) عن طريق هز الأنابيب قليلا لإنشاء تدرج انتشار.
- قم بتخفيف محلول البروتين باستخدام المخزن المؤقت لغسيل الكلى في سلسلة من التركيزات: 1 و 2 و 5 و 10 و 50 مجم / مل.
- تحديد التركيزات الدقيقة باستخدام مقياس الطيف الضوئي Nanodrop (انظر جدول المواد).
2. توصيف العينة الأولية عن طريق تشتت الضوء الديناميكي (DLS)
- قم بتحميل 80 ميكرولتر من كل محلول بروتين من سلسلة التركيز المحضرة أعلاه (الخطوة 1.6) في الخلية التي يمكن التخلص منها DLS (انظر جدول المواد) وحدد معاملات الانتشار ، بمتوسط أكثر من 10 عمليات استحواذ.
- ارسم معاملات الانتشار الانتقالي كدالة لتركيز البروتين واستقراء التركيز إلى الصفر.
- قم بتحميل كل محلول بروتين من سلسلة التركيز في الأنابيب الشعرية لمقياس اللزوجة (انظر جدول المواد) وقم بقياس اللزوجة الديناميكية.
- ارسم اللزوجة الديناميكية المقاسة كدالة لتركيز البروتين واستقراء التركيز إلى الصفر.
ملاحظة: إن استقراء انتشار DLS إلى تركيز صفري يعطي قيمة الانتشار الانتقالي لبروتين واحد. يجب أن يؤدي استقراء اللزوجة الديناميكية إلى التركيز الصفري إلى قيمة اللزوجة الديناميكية المقاسة تجريبيا للحل العازل.
3. جمع التشتت بزاوية صغيرة (النيوترون أو الأشعة السينية)
- قياس تشتت النيوترونات صغير الزاوية (SANS) و/أو تشتت الأشعة السينية صغير الزاوية (SAXS) (انظر جدول المواد) على أربعة تركيزات بروتينية، ويفضل أن تكون 2 ملغم/مل، و5 ملغم/مل، و10 ملغم/مل، و50 ملغم/مل.
- تطبيع أطياف SANS و SAXS عن طريق تركيز البروتين.
- قم بملاءمة عامل شكل البروتين P(Q) مع أطياف SANS و SAXS باستخدام برنامج تحسين المجموعة31 و / أو SasView32 .
- احسب عامل البنية S(Q, c) بقسمة إشارة SANS و SAXS مع P(Q) لكل تركيز.
ملاحظة: يتم تشجيع القراء المهتمين بكيفية قياس وتفسير بيانات التشتت ذات الزاوية الصغيرة كدعم لقياسات NSE على الرجوع بدقة إلى المراجع23،28،31،32،33.
4. قياس NSE
- قم بإعداد العينة وقم بتركيبها باتباع الخطوات أدناه.
- حدد سمك الخلية لتحميل العينة بناء على تركيز محلول البروتين ودرجة الحرارة اللازمة للقياس وكمية المحلول المتاح.
ملاحظة: استخدمت هذه الدراسة حاويات كوارتز شفافة للودر العلوي بسعة 40 مم × 30 مم × 4 مم. - نظف الخلية بشكل متكرر ، بالتناوب بين منظفات الأطباق الخالية من الفوسفات (انظر جدول المواد) ، والماء منزوع الأيونات ، والإيثانول بنسبة 70٪.
- تجفيف الخلية في فرن الحمل الحراري. لا تتجاوز 80 درجة مئوية لخلايا الكوارتز.
- تحميل 4 مل من محلول البروتين في الخلية وإغلاق مع قبعات. استخدم فيلم الشمع أو أي مادة مانعة للتسرب (انظر جدول المواد) لإغلاق خلايا العينة.
ملاحظة: في هذه الدراسة ، تم استخدام 4.8 مل من المحلول عند ~ 50 ملغ / مل للحصول على كثافة تشتت كافية. - قم بتحميل 4 مل من المخزن المؤقت لغسيل الكلى في حاوية مماثلة لعينة البروتين وختمها.
- انقل العينات إلى خط الشعاع ، وأغلق الغالق ، وأدخل منطقة كهف حاوية مقياس الطيف34.
- قم بتركيب خلية العينة على حامل عينة الألومنيوم عن طريق تشديد البراغي وألواح الإمساك (الشكل 2 ، اللوحة اليسرى).
ملاحظة: يمكن للمرء تركيب خليتين للعينات في نفس الوقت ، نظرا لأن هناك حاجة إلى نفس بروتوكول القياس لجميع العينات. - قم بتركيب عينة الجرافيت و / أو عينة مسحوق Al2O3 المحملة في حاوية مماثلة لعينة البروتين. هذه هي المعايير التي يوفرها دعم خط شعاع SNS-NSE.
- ضع حامل العينة عن طريق تحريكه برفق في علبة نظام فرض درجة الحرارة (TFS، انظر جدول المواد).
ملاحظة: TFS هي بيئة العينة الأكثر استخداما في SNS-NSE وتضخ الهواء الجاف في علبة العينة لتحقيق درجة الحرارة المطلوبة (الشكل 2 ، الألواح الوسطى واليمنى). - أغلق غطاء TFS واضبط درجة الحرارة على القيمة المطلوبة من خلال الوصول إلى الشاشة التفاعلية ل TFS.
- قم بتركيب الكاميرا النيوترونية (انظر جدول المواد) لمحاذاة العينات مع الحزمة.
- قم بكنس حاوية الجهاز وقم بالإخلاء وإغلاق الأبواب وفتح مصراع الشعاع.
ملاحظة: يتم توفير خلايا العينة بواسطة دعم خط شعاع SNS-NSE. للحصول على خلايا العينة المتوفرة ومكتبة بيئة العينات، يرجى الرجوع إلى صفحة ويب خط شعاع SNS-NSE 7,35.
- حدد سمك الخلية لتحميل العينة بناء على تركيز محلول البروتين ودرجة الحرارة اللازمة للقياس وكمية المحلول المتاح.
- اجمع بيانات NSE باتباع الخطوات أدناه.
- قم بمحاذاة العينة في الحزمة النيوترونية باستخدام الكاميرا النيوترونية وفتحات العينة الأربعة المستقلة.
- افتح برنامج جمع البيانات SNS-NSE36 وجمع إحصائيات العينات عن طريق تشغيل عمليات فحص الحيود لزوايا التشتت المطلوبة والطول الموجي.
- قم بإعداد معلمات القياس استنادا إلى الإحصاءات التي تم جمعها لكل عينة عن طريق تحرير وحدات ماكرو القياس التي يقدمها عالم الأدوات المساعد.
- ابدأ المسح الضوئي عن طريق كتابة اسم البروتوكول في موجه الأوامر واحصل على أصداء للعينة.
- ابدأ المسح الضوئي واحصل على أصداء أيضا للمرجع المرن والمذيب المؤقت. قم بإجراء عملية غالق شعاع متقطعة لتغيير العينة.

الشكل 2: نظام قياس NSE. اللوحة اليسرى: عينات من محلول البروتين في حاوية كوارتز مثبتة بمسامير وألواح على حامل عينات الألومنيوم (Al). يوفر حامل العينة Al إمكانية تركيب عينتين في وقت واحد داخل بيئة العينة. اللوحة الوسطى: يمكن تركيب عينة نظام تأثير درجة الحرارة (TFS) في مرحلة العينة التي تكون فيها نافذة شعاع النيوترون على اليمين ، بينما تكون الكاميرا النيوترونية المستخدمة للمحاذاة مرئية على الجانب الأيسر. اللوحة اليمنى: يمكن أن يعمل وضع حامل العينة مع عينتين في العينة مع نوافذ السيليكون (Si). يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
5. تقليل بيانات NSE
ملاحظة: تم تجهيز SNS-NSE ببرنامج مخصص يسمى DrSpine (تقليل البيانات ل spin-ECHO)37,38 متوفر في مجموعة التحليل عن بعد لعلوم النيوترون ORNL ، ودليل مستخدم سريع ، ودعم مساعدة مدمج.
- سجل الدخول إلى مجموعة التحليل عن بعد لعلوم النيوترون (انظر جدول المواد) باستخدام بيانات اعتماد مستخدم ORNL، واضغط على الزر تشغيل الجلسة .
- قم بإعداد برنامج تقليل البيانات باتباع الخطوات أدناه.
- في دليل المستخدم، افتح نافذة طرفية واكتب: source/SNS/software/nse/etc/setup_nse.sh.
- بعد ذلك، اكتب: drspine_create_env.sh.
- قم بإنشاء مجلد لتقليل البيانات في الدليل الرئيسي وانسخ البرامج النصية ووحدات الماكرو المتوفرة من الدليل المشترك.
- تحرير وإعادة تسمية وحفظ ماكرو التخفيض المقدم وفقا لذلك.
- اكتب drspine في موجه الأوامر واضغط على Enter لبدء بيئة تقليل البرامج.
- اكتب "اسم ماكرو التخفيض" الذي تم تحريره في الخطوة 5.4. في موجه الأوامر داخل بيئة البرنامج واضغط على مفتاح الإدخال Enter.
6. تركيب بيانات NSE
- قم بتحرير البرنامج النصي للثعبان "stapler-drspine.py" ، الذي قدمه عالم الأدوات المساعد ، مع أسماء بيانات الملف المخفضة.
ملاحظة: البرنامج النصي بايثون متاح مجانا لمستخدمي الأداة. - قم بتحرير الوظيفة لتناسب من المكتبة المتوفرة.
- اكتب اسم البرنامج النصي الذي تم تحريره "stapler-drspine.py" في موجه الأوامر واضغط على Enter لقراءة بيانات NSE المخفضة وملاءمتها ورسمها.
ملاحظة: سيوفر عالم الأدوات نموذجا لماكرو الاختزال والبرنامج النصي "stapler-drspine.py" للثعبان يمكنه قراءة بيانات NSE المخفضة وملاءمتها. بيانات NSE النهائية المخفضة هي بتنسيق ASCII ويمكن قراءتها بواسطة العديد من البرامج المفضلة.
النتائج
تم إعادة تشكيل بروتين IgG من المصل البشري وبروتينات MBP البقرية بتركيزات عالية (~ 50 ملغ / مل) في المخازن المؤقتة D2O-base. نظرا لأن البروتينات كانت مذابة بتركيزات عالية ، فإن المحاليل التي تم الحصول عليها كانت محاليل بروتينات مزدحمة. تعاني الديناميكيات التي تم التحقيق فيها باستخدام NSE من الب?...
Discussion
يوفر التحليل الطيفي NSE رؤية فريدة ومفصلة لديناميكيات البروتينات ، والتي لا تستطيع التقنيات الطيفية الأخرى إنتاجها. توفر القياسات على نطاق زمني ممتد ملاحظات لكل من الانتشار الانتقالي والدوراني للبروتينات ، كما هو موضح هنا. تكشف الديناميكيات القطاعية والتذبذبات الداخلية الأخرى عن نفسها ع...
Disclosures
ولا يعلن صاحب البلاغ عن وجود مصالح مالية متنافسة أو تضارب في المصالح. يعتمد محتوى المخطوطة على المحاضرة التي قدمها المؤلف في تطبيقات تشتت النيوترون HANDS-Neutron في مدرسة البيولوجيا الهيكلية بين عامي 2019-2021.
Acknowledgements
استخدم هذا البحث موارد في مصدر النيوترون التشتت (BL-15 و BL-6 ومختبرات البيولوجيا والكيمياء) ، وهو مكتب تابع لوزارة الطاقة تابع لمرفق مستخدم العلوم الذي يديره مختبر أوك ريدج الوطني. استخدم هذا البحث أيضا موارد في مفاعل MLZ-FRM2 Garching (KWS-2 ، Phoenix-J-NSE) و JCNS1 في Forschungszentrum Jülich GmbH ، ألمانيا. يعترف المؤلف بالدكتور رالف بيهل والدكتور أندرياس ستادلر لمساعدتهما في النمذجة ومساهمتهما في كل من أبحاث بروتينات IgG و MBP ، والدكتور بيوتر أ. Żołnierczuk لدعم تقليل بيانات NSE ، والدكتور Changwoo Do للدعم مع قياسات SANS ، و Rhonda Moody و Dr. Kevin Weiss لدعم مختبر الكيمياء الحيوية SNS.
Materials
| Name | Company | Catalog Number | Comments |
| Bovine MBP protein solution | Sigma-Aldrich | M1891 | lyophilized powder reconstituted in D2O |
| D2O - heavy water | Sigma-Aldrich | Product No. 151882 | liquid |
| Dionized water | in house | - | for washing / cleanning cells |
| DLS instrument | Zetasizer Nano ZS, FZ-Jülich | - | dynamic light scattering instrument |
| Elastic scattering standards | SNS-NSE, ORNL | - | Al2O3 and Graphite powders |
| Ethanol | Sigma-Aldrich | 65350-M | 70% ethanol for cleaning cells |
| IgG protein solution | Sigma-Aldrich | I4506 | lyophilized powder reconstituted in D2O |
| KWS-2 instrument | JCNS outstation at the MLZ, Garching, Germany | - | small angle neutron instrument |
| Liquinox dish detergent | Alconox | - | Phosphate-free liquid lab glassware cleaner |
| Na2HPO4·7H2O | Sigma-Aldrich | Product No.S9390 | disodium phosphate heptahydrate salt |
| NaCl | Sigma-Aldrich | Product No.S9888 | sodium chloride salt |
| NaH2PO4·H2O | Sigma-Aldrich | Product No. S9638 | monosodium phosphate monohydrate salt |
| Nanodrop spectrophotometer | Thermo Scientific | Catalog number: ND-2000 | NanoDrop 2000/2000c Spectrophotometer |
| Neutron alignment camera | NeutronOptics, Grenoble | NOG210222 | 100 x 100 mm camera with Sony IMX249 CMOS sensor |
| Parafilm M - wax parafilm | Bemis | Parafilm M - 5259-04LC PM996 | all-purpose laboratory film in cardboard dispenser |
| Phoenix-J-NSE Spectrometer | JCNS outstation at the MLZ, Garching, Germany | - | neutron spectrometer |
| SasView | https://www.sasview.org/ | ||
| SAXSpace, Anton Paar instrument | FZ-Jülich | - | small angle x-ray instrument |
| Slide-A-Lyzer dialysis membranes | Thermo Scientific | 88400-88405 | Slide-A-Lyzer mini dialysis devices tubes of 3.5 K MWCO |
| SNS Remote Analysis Cluster | Neutron Science Remote Analysis (sns.gov) | https://analysis.sns.gov | |
| SNS-NSE spectrometer | ORNL, Oak Ridge, TN, USA | - | neutron spectrometer |
| Sterile syringe filters | VWR | N.A. PN:28145-501 | 0.2 µm pore size filters |
| Temperature Forcing System (TFS) | SP Scientific | Part Number 100004055 | sample environment equipment |
| Urea -d4 | Sigma-Aldrich | Product No. 176087 | deuterated Urea salt |
| Viscometer | FZ-Jülich | - | falling ball viscometer |
References
- Fitter, J., Gutberlet, T., Katsaras, J. . Neutron Scattering in Biology: Techniques and Applications. , (2006).
- Stadler, A., Monkenbusch, M., Biehl, R., Richter, D., Ollivier, J. Neutron spin-echo and TOF reveals protein dynamics in solution. Journal of the Physical Society of Japan. 82, (2013).
- Inoue, R. Large domain fluctuations on 50-ns timescale enable catalytic activity in phosphoglycerate kinase. Biophysical Journal. 99 (7), 2309-2317 (2010).
- Callaway, D. J. E., et al. Controllable activation of nanoscale dynamics in a disordered protein alters binding kinetics. Journal of Molecular Biology. 429 (7), 987-998 (2017).
- Stingaciu, L. R., Biehl, R., Changwoo, D., Richter, D., Stadler, A. M. Reduced internal friction by osmolyte interaction in intrinsically disordered myelin basic protein. Journal of Physical Chemistry Letters. 11 (1), 292-296 (2020).
- Monkenbusch, M., Richter, D. High resolution neutron spectroscopy-a tool for the investigation of dynamics of polymers and soft matter. Comptes Rendus Physique. , (2007).
- Richter, D., Monkenbusch, M., Schwahn, D. Neutron Scattering. Polymer Science: A Comprehensive Reference, 10 Volume Set. , (2012).
- Wilson, C. C. . Single Crystal Neutron Diffraction From Molecular Materials. , (2000).
- Marshall, W. . Theory of thermal neutron scattering. , (1971).
- . Roger Pynn Introduction & Neutron Scattering "Theory" Available from: https://neutrons.ornl.gov/sites/default/files/intro_to_neutron_scattering.pdf (2004)
- Richter, D. Neutron scattering in polymer physics. Physica B: Condensed Matter. 276-278, 22-29 (2000).
- Harroun, T. A., Wignall, G. D., Katsaras, J. Neutron scattering for biology. Neutron Scattering in Biology. , (2006).
- . SNS Available from: https://neutrons.ornl.gov/sna (2020)
- . HFIR Available from: https://neutrons.ornl.gov/hfir (2020)
- . CNCS Available from: https://neutrons.ornl.gov/cncs (2020)
- Perticaroli, S., et al. Description of hydration water in protein (green fluorescent protein) solution. Journal of the American Chemical Society. 139 (3), 1098-1105 (2017).
- Perticaroli, S., Nickels, J. D., Ehlers, G., Sokolov, A. P. Rigidity, secondary structure, and the universality of the boson peak in proteins. Biophysical Journal. 106 (12), 2667-2674 (2014).
- Miao, Y., et al. Coupled flexibility change in cytochrome p450cam substrate binding determined by neutron scattering, NMR, and molecular dynamics simulation. Biophysical Journal. 103 (10), 2167-2176 (2012).
- Mamontov, E., Zamponi, M., Hammons, S., Keener, W. S., Hagen, M., Herwig, K. W. BASIS: A new backscattering spectrometer at the SNS. Neutron News. 19 (3), 22-24 (2008).
- Mamontov, E. Microscopic diffusion processes measured in living planarians. Scientific Reports. 9, 8708 (2018).
- Alpert, Y., Cser, L., Faragó, B., Franěk, F., Mezei, F., Ostanevich, Y. M. Segmental flexibility in pig immunoglobulin G studied by neutron spin-echo technique. Biopolymers. 24 (9), 1769-1784 (1985).
- Bu, Z., Biehl, R., Monkenbusch, M., Richter, D., Callaway, D. J. E. Coupled protein domain motion in Taq polymerase revealed by neutron spin-echo spectroscopy. Proceedings of the National Academy of Sciences of the United States of America. 102 (49), 17646-17651 (2005).
- Biehl, R., et al. Direct observation of correlated interdomain motion in alcohol dehydrogenase. Physical Review Letters. 101, 138102 (2008).
- Farago, B., Li, J., Cornilescu, G., Callaway, D. J. E., Bu, Z. Activation of nanoscale allosteric protein domain motion revealed by neutron spin echo spectroscopy. Biophysical Journal. 99 (10), 3473-3482 (2010).
- Bu, Z., Callaway, D. J. E. Dynamic propagation of long-range allosteric signals by nanoscale protein domain motion revealed by neutron spin echo spectroscopy. Biophysical Journal. 100 (3), 223 (2011).
- Hong, L., et al. Structure and dynamics of a compact state of a multidomain protein, the mercuric ion reductase. Biophysical Journal. 107 (2), 393-400 (2014).
- Longeville, S., Stingaciu, L. -. R. Hemoglobin diffusion and the dynamics of oxygen capture by red blood cells. Scientific Reports. 7, 10448 (2017).
- Stingaciu, L. R., Ivanova, O., Ohl, M., Biehl, R., Richter, D. Fast antibody fragment motion: Flexible linkers act as entropic spring. Scientific Reports. 6, 22148 (2016).
- Hahn, E. L. Nuclear induction due to free larmor precession. Physical Review. 77, 297 (1950).
- Hahn, E. L. Spin echoes. Physical Review. 80, 580 (1950).
- Svergun, D. I., Koch, M. H. J. Small-angle scattering studies of biological macromolecules in solution. Reports on Progress in Physics. 66 (10), 1735-1782 (2003).
- . Sasview Available from: https://www.sasview.org (2020)
- Zhao, J. K., Gao, C. Y., Liu, D. The extended Q-range small-angle neutron scattering diffractometer at the SNS. Journal of Applied Crystallography. 43, 1068-1077 (2010).
- Ohl, M., et al. The high-resolution neutron spin-echo spectrometer for the SNS with τ ≥ 1 µs. Physica B: Condensed Matter. 350, 147-150 (2004).
- . SNS-NSE web page Available from: https://neutrons.ornl.gov/nse (2022)
- Ohl, M., et al. The spin-echo spectrometer at the Spallation Neutron Source (SNS). Nuclear Instruments and Methods in Physics Research, Section A: Accelerators, Spectrometers, Detectors and Associated Equipment. 696, 85-99 (2012).
- Zolnierczuk, P. A., Holderer, O., Pasini, S., Kozielewski, T., Stingaciu, L. R., Monkenbusch, M. Efficient data extraction from neutron time-of-flight spin-echo raw data. Journal of Applied Crystallography. 52 (5), 1022-1034 (2019).
- . Dr Spine hub Available from: https://jugit.fz-juelich.de/nse/drspine (2022)
- Stadler, A. M., et al. Internal nanosecond dynamics in the intrinsically disordered myelin basic protein. Journal of the American Chemical Society. 136 (19), (2014).
- . FZJ Available from: https://www.fz-juelich.de/portal/EN/AboutUs (2022)
- . KWS2 Available from: https://miz-garching.de/kws-2 (2022)
- Moorhouse, M., Barry, P. The protein databank. Bioinformatics Biocomputing and Perl. , (2005).
- Tria, G., Mertens, H. D. T., Kachala, M., Svergun, D. I. Advanced ensemble modelling of flexible macromolecules using X-ray solution scattering. IUCrJ. 2 (2), 207-217 (2015).
- Holderer, O., Monkenbusch, M., Schätzler, R., Kleines, H., Westerhausen, W., Richter, D. The JCNS neutron spin-echo spectrometer J-NSE at the FRM II. Measurement Science and Technology. 90 (4), 043107 (2008).
- Biehl, R., Monkenbusch, M., Richter, D. Exploring internal protein dynamics by neutron spin echo spectroscopy. Soft Matter. 7 (4), 1299-1307 (2011).
- Stingaciu, L. R., Ivanova, O., Ohl, M., Biehl, R., Richter, D. Fast antibody fragment motion: Flexible linkers act as entropic spring. Scientific Reports. 6, 22148 (2016).
- Stadler, A. M., et al. Internal nanosecond dynamics in the intrinsically disordered myelin basic protein. Journal of the American Chemical Society. 136 (19), 6987-6994 (2014).
- Biehl, R., Richter, D. Slow internal protein dynamics in solution. Journal of Physics: Condensed Matter. 26 (50), 503103 (2014).
- Hinsen, K. The molecular modeling toolkit: A new approach to molecular simulations. Journal of Computational Chemistry. 21 (2), 79-85 (2000).
- Uhlenbeck, G. E., Ornstein, L. S. On the theory of the Brownian motion. Physical Review. 36 (5), 823 (1930).
- Wang, M. C., Uhlenbeck, G. E. On the theory of the Brownian motion II. Reviews of Modern Physics. 17, 323 (1945).
- Callaway, D. J. E., Bu, Z. Nanoscale protein domain motion and long-range allostery in signaling proteins-a view from neutron spin echo spectroscopy. Biophysical Reviews. 7 (2), 165-174 (2015).
- Liu, Y. Intermediate scattering function for macromolecules in solution probed by neutron spin echo. Physical Review E. 95, 020501 (2017).
- Liu, Y. Short-time dynamics of proteins in solutions studied by neutron spin echo. Current Opinion in Colloid & Interface Science. 42, 147-156 (2019).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved