È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Studio della dinamica delle proteine tramite spettroscopia eco-spin neutronica
In questo articolo
Riepilogo
Il presente protocollo descrive i metodi per studiare la struttura e la dinamica di due proteine modello che hanno un ruolo importante nella salute umana. La tecnica combina la caratterizzazione biofisica da banco con la spettroscopia eco-spin neutronica per accedere alla dinamica alle scale di tempo e lunghezza rilevanti per i movimenti interdominio delle proteine.
Abstract
L'attività e la funzionalità della maggior parte delle proteine del corpo umano sono correlate ai cambiamenti configurazionali di interi sottodomini all'interno della struttura cristallina della proteina. Le strutture cristalline costruiscono la base per qualsiasi calcolo che descriva la struttura o la dinamica di una proteina, il più delle volte con forti restrizioni geometriche. Tuttavia, queste restrizioni dalla struttura cristallina non sono presenti nella soluzione. La struttura delle proteine nella soluzione può differire dal cristallo a causa di riarrangiamenti di loop o sottodomini sulla scala temporale da pico a nanosecondi (cioè il regime temporale della dinamica proteica interna). Il presente lavoro descrive come è possibile accedere a slow motion su scale temporali di diverse decine di nanosecondi utilizzando lo scattering neutronico. In particolare, la caratterizzazione dinamica di due principali proteine umane, una proteina intrinsecamente disordinata che manca di una struttura secondaria ben definita e una proteina anticorpale classica, è affrontata mediante spettroscopia eco-spin neutronica (NSE) combinata con una vasta gamma di metodi di caratterizzazione di laboratorio. Ulteriori approfondimenti sulla dinamica del dominio proteico sono stati ottenuti utilizzando la modellazione matematica per descrivere i dati sperimentali dei neutroni e determinare il crossover tra i moti diffusivi combinati e i movimenti proteici interni. L'estrazione del contributo dinamico interno alla funzione di scattering intermedio ottenuto da NSE, compresa la scala temporale dei vari movimenti, consente un'ulteriore visione delle proprietà meccaniche delle singole proteine e della morbidezza delle proteine nel loro ambiente quasi naturale nella soluzione proteica affollata.
Introduzione
Sondare la dinamica della materia soffice con neutroni
Studiare le proprietà dinamiche di proteine e peptidi è una parte importante della ricerca biofisica e oggi esistono molti metodi ben sviluppati per accedere a una vasta gamma di paesaggi energetici1. Mettere in relazione la dinamica sperimentalmente rivelata delle proteine con la loro funzione biologica è un compito molto più difficile, che richiede complessi modelli matematici e simulazioni dinamiche assistite da computer. L'importanza della spettroscopia neutronica per l'analisi dei moti proteici è stata sottolineata in diversi studi ben accolti e ampiamente riconosciuti 1,2,3,4,5. Prima di esplorare il variegato panorama energetico della dinamica proteica interna, è necessaria una breve panoramica dei processi dinamici nella materia soffice e di come i neutroni possono accedervi.
La sensibilità dei neutroni alla configurazione isotopica e il tipo di interazioni che mostrano con la materia soffice rende lo scattering neutronico una delle tecniche di indagine più versatili6. Esiste un ampio spettro di scale di lunghezza di correlazione e tempi di correlazione a cui i neutroni possono accedere, dalle eccitazioni nucleari e dalle vibrazioni atomiche ai moti collettivi e ai processi di rilassamento lento come le rotazioni isotrope e i moti diffusivi. Quando si studiano i neutroni sparsi per il loro trasferimento di energia, si possono distinguere tre interazioni principali: lo scattering elastico, in cui non vi è scambio di energia tra neutrone in arrivo e particella nel campione; lo scattering anelastico, con un ampio scambio di energia quantificabile tra neutrone e particella; e il caso peculiare dello scattering quasi-elastico che designa un trasferimento di energia molto piccolo rispetto all'energia neutronica incidente 1,7. Queste interazioni forniscono informazioni precise sul materiale studiato e costituiscono la base teorica di un'ampia varietà di tecniche di scattering neutronico.
Nello scattering elastico, il rivelatore registra le direzioni dei neutroni come un modello di diffrazione, che mostra la posizione degli atomi campione l'uno rispetto all'altro. Vengono acquisite informazioni sulle correlazioni delle posizioni atomiche (cioè l'intensità integrata S(Q) relativa al trasferimento di quantità di moto Q, che riguarda solo le informazioni strutturali). Questo principio costituisce la base della diffrazione neutronica8.
La complessità sorge quando il trasferimento di energia non è più zero a causa di eccitazioni e fluttuazioni interne nel materiale campione. Questo costituisce la base della spettroscopia neutronica, in cui i neutroni sparsi sono studiati in funzione sia del trasferimento di energia E che del trasferimento di quantità di moto Q. Si ottengono informazioni dinamiche e strutturali. La spettroscopia neutronica misura la stessa intensità integrata S(Q) per il trasferimento di energia (cioè la variazione di velocità dei neutroni dovuta allo scattering del campione, S(Q,ω) = S(Q, E), che è anche indicato come fattore di struttura dinamica)9.
Per calcolare lo scattering da un materiale, è più appropriato utilizzare la funzione di correlazione di coppia 7,10. Nel caso della diffrazione, la funzione di correlazione della coppia statica G(r) dà la probabilità di trovare il centro di una particella ad una data distanza r dal centro di un'altra particella. La spettroscopia generalizza la funzione di correlazione della coppia statica e include energia/frequenza/tempo nell'equazione di scattering. La funzione di correlazione di coppia G(r) diventa una funzione del tempo G(r, t), che può essere scomposta in una distinta funzione di correlazione della coppia atomica GD(r, t), e una funzione di auto-correlazione GS(r, t). Questi descrivono due tipi di correlazioni: i moti correlati alle coppie degli atomi che governano lo scattering coerente e l'auto-correlazione che governa lo scattering incoerente10.
Lo scattering coerente è lo scattering da "la media" e dipende dalla fase relativa delle onde sparse. Nel regime di scattering a piccolo angolo, le onde di neutroni sparse da diversi centri di scattering (atomi diversi) interferiscono in modo costruttivo (hanno fasi simili) e il movimento collettivo degli atomi viene osservato con un forte aumento dell'intensità. Lo scattering coerente descrive essenzialmente lo scattering di un singolo neutrone da tutti i nuclei del campione10.
Quando non si verifica alcuna interferenza costruttiva tra le onde di neutroni sparse da centri diversi, un singolo atomo viene seguito nel tempo e si osserva l'auto-correlazione tra la posizione dell'atomo al tempo t = 0 e lo stesso atomo al tempo t. Pertanto, le informazioni sulle posizioni relative degli atomi vengono perse e l'attenzione si concentra solo sulle fluttuazioni locali. La dispersione da fluttuazioni locali governa lo scattering incoerente. Lo scattering incoerente è isotropo, contribuisce al segnale di fondo e degrada il segnale-rumore10,11.
Combinando tutto quanto sopra, distinguiamo quattro principali processi di scattering neutronico10: (1) coerente elastico (misura le correlazioni delle posizioni atomiche), (2) coerente anelastico (misura i moti collettivi degli atomi), (3) elastico incoerente (contribuisce allo sfondo, riduce l'intensità di scattering mediante il fattore di Debye-Waller (DWF) e misura il fattore di struttura incoerente elastica (EISF), descrivendo la geometria dei moti diffusivi in geometria confinata, e (4) incoerente anelastico (misura la dinamica del singolo atomo e l'auto-correlazione).
I processi dinamici a cui i neutroni possono accedere in biologia vanno dallo smorzamento delle vibrazioni atomiche e molecolari a bassa frequenza, all'interazione delle molecole di solvente con le biosuperfici e ai processi di diffusione nello strato di idratazione delle macromolecole e della geometria confinata, ai movimenti diffusivi traslazionali, rotazionali e di burattatura a corto raggio, ai domini proteici e ai moti allosterici1 . L'ampia diversità dei metodi e degli strumenti di neutroni per misurare la dinamica delle proteine si basa su come si ottiene l'acromatizzazione del fascio di neutroni incidente o in uscita e su come viene eseguita l'analisi energetica dei neutroni sparsi. Dagli spettrometri a triplo asse a time-of-flight, backscattering e spin-echo, si possono esplorare processi dinamici con tempi caratteristici compresi tra 1 x 10-14 s e 1 x 10-6 s (da femtosecondi a microsecondi)12.
L'Oak Ridge National Laboratory, con le sue due rinomate sorgenti di neutroni, la Spallation Neutron Source - SNS13 e l'High Isotope Flux Reactor - HFIR14, ha una delle migliori suite di spettrometri per lo studio della dinamica nei biomateriali. Alcuni degli esempi più eloquenti includono l'uso dello spettrometro chopper a neutroni freddi (CNCS) a SNS15 per studiare la perturbazione dinamica dell'acqua di idratazione intorno alla proteina fluorescente verde nella soluzione16 o le vibrazioni collettive sub-picosecondi di diverse proteine17. Un problema ricorrente delle indagini anelastiche sullo scattering dei neutroni è che alcuni processi biologici sono troppo lenti per essere osservati. Senza configurazioni estreme che portano a un'enorme perdita di intensità neutronica, gli spettrometri a tempo di volo sono limitati a una risoluzione di energia di 10 μeV, corrispondente a una scala temporale massima di ~ 200 ps10,11. Questo non è sufficiente per osservare movimenti su larga scala nelle proteine. Pertanto, sono spesso necessari strumenti con una risoluzione energetica più elevata come gli spettrometri backscattering. La combinazione del tempo di volo e delle tecniche di backscattering si è dimostrata efficace per studiare il cambiamento nella dinamica interna del citocromo P450cam (CYP101), un enzima che catalizza l'idrossilazione della canfora18.
La diffusività microscopica misurata dallo spettrometro retrodiffusante di SNS-BASIS19 era sorprendentemente ben definita e poteva essere separata nella diffusività dell'acqua (idratazione, citoplasmatica e acqua simile alla massa) e nella diffusività dei costituenti cellulari nei platelminti planari, il primo animale vivente ad essere studiato mediante scattering neutronico20 . Il backscattering è una tecnica spettroscopica ad alta risoluzione, ma è anche limitata a diversi μeV = diversi nanosecondi, mentre la dinamica lenta nei biomateriali si manifesta anche come il tempo di sopravvivenza della correlazione tra posizione atomica o orientamenti di spin (ad esempio, processi di rilassamento, che si verificano regolarmente nell'intervallo di tempo da dieci a centinaia di nanosecondi).
La spettroscopia eco di spin neutronico (NSE) è l'unica tecnica di scattering neutronico a raggiungere una risoluzione così elevata. A differenza di altre tecniche neutroniche, NSE non richiede l'acromatizzazione del fascio poiché utilizza la fase meccanica quantistica dei neutroni, che è i loro momenti magnetici. La manipolazione dei momenti magnetici consente l'uso di un'ampia distribuzione della lunghezza d'onda del fascio di neutroni, mentre la tecnica è sensibile a variazioni di velocità di neutroni molto piccole nell'ordine di 1 x 10-4. NSE è stato utilizzato con successo per studiare la lenta dinamica delle proteine in soluzione per molte proteine. Tra questi numerosi studi pionieristici, riconosciamo lo studio della flessibilità segmentale dell'immunoglobulina21 suina; i moti del dominio accoppiato nella Taq polimerasi22; i moti di dominio nel tetramero del lievito alcol deidrogenasi23; il cambiamento di conformazione della fosfoglicerato chinasi in base al legame del substrato3; l'attivazione dei moti di dominio e la propagazione dinamica dei segnali allosterici nella proteina 4,24,25) di regolazione dello scambio Na+/H+(NHERF1); la dinamica di uno stato compatto di ione reduttasi mercurico26; e la diffusione dell'emoglobina nei globuli rossi27. Due studi più recenti sulla dinamica delle proteine hanno esposto la flessibilità dell'anticorpo umano Immunoglobulina G (IgG) come molla entropica28 e le caratteristiche del contributo del solvente alla dinamica della proteina basica della mielina intrinsecamente disordinata (MBP)5.
Il presente articolo spiega i principi di base dell'NSE, i molteplici metodi preparatori raccomandati per un'indagine approfondita della dinamica delle proteine, nonché la metodologia e il protocollo sperimentale per l'acquisizione dei dati NSE presso lo spettrometro NSE di SNS, SNS-NSE. Il protocollo caratterizza due proteine: IgG, una normale proteina anticorpale umana, e la proteina intrinsecamente disordinata MBP. Le implicazioni biofisiche, la rilevanza della ricerca degli esempi e i limiti della tecnica sono discussi brevemente.
Spettroscopia NSE, il metodo per le misure di dinamica lenta
NSE è una tecnica polarizzata che utilizza il tempo di volo dei neutroni per misurare lo scambio di energia (perdita di polarizzazione) dovuto all'interazione quasi elastica tra neutroni e atomi in un campione. Al centro della spettroscopia NSE si trovano due principi di base: (1) la capacità dello spin neutronico di precedere nel campo magnetico con una frequenza proporzionale alla forza 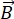 magnetica, vale a dire la frequenza di Larmor29, e (b) l'eco di spin o eco di Hann, che rappresenta la manipolazione e la rifocalizzazione del segnale di polarizzazione quando si applica una serie di impulsi a radiofrequenza30.
magnetica, vale a dire la frequenza di Larmor29, e (b) l'eco di spin o eco di Hann, che rappresenta la manipolazione e la rifocalizzazione del segnale di polarizzazione quando si applica una serie di impulsi a radiofrequenza30.
Le basi del processo NSE possono essere riassunte in pochi semplici passaggi 6,11 utilizzando la Figura 1. (1) Il fascio di neutroni prodotto dalla sorgente (posizione 1) è polarizzato (posizione 2), guidato e trasportato (posizione 3) e arriva all'ingresso dello spettrometro NSE, dove viene ruotato di 90° dal primo pi-mezzo flipper (posizione 4). (2) Il fascio polarizzato (ad esempio, momenti magnetici di neutroni) diventa perpendicolare alle linee del campo magnetico del primo magnete (prima zona di precessione, posizione 5) e inizia a precedere. (3) Alla fine del magnete, gli spin dei neutroni accumulano un certo angolo di precessione proporzionale all'intensità del campo magnetico e al tempo di volo trascorso all'interno (fondamentalmente inversamente proporzionale alla velocità del neutrone). Le singole velocità dei neutroni sono codificate all'interno del loro angolo di precessione alla fine della prima zona di precessione. (4) Vicino alla posizione del campione, il pi-flipper (posizione 6) inverte l'orientamento dello spin di 180°, cambiando il segno dell'angolo di precessione. (5) I neutroni interagiscono con le molecole del campione (posizione 7) e si disperdono. (6) I neutroni sparsi entrano e precedono nella seconda zona di precessione (posizione 8) ma diventano orientati inversamente. (7) Un'altra pinna pi-mezza (posizione 9) viene utilizzata per ruotare l'orientamento dello spin dalla direzione perpendicolare a quella orizzontale. Questo fermerà la precessione, traducendo l'angolo di precessione φ in polarizzazione proporzionale a cos(φ). (8) L'analizzatore (posizione 10) seleziona i neutroni in base a un orientamento. Se l'interazione con il campione è elastica, la velocità del neutrone non cambierà. I neutroni trascorreranno una quantità identica di tempo volando nella prima e nella seconda zona di precessione e gli angoli di precessione accumulati saranno completamente recuperati. La polarizzazione completa viene ripristinata sul rivelatore (posizione 11) come eco della polarizzazione originale (cioè spin-echo). (9) Tuttavia, in NSE, lo scattering è quasi elastico, quindi un piccolo scambio di energia tra neutroni e molecole campione porta a diverse velocità di neutroni dopo la dispersione da parte del campione. A causa delle diverse velocità, i neutroni trascorreranno un ulteriore tempo volando attraverso la seconda zona di precessione e non avranno recuperato correttamente il loro angolo di precessione. Una polarizzazione parziale viene recuperata sul rivelatore e la perdita di polarizzazione dovuta al rilassamento dello spin è proporzionale alla trasformata cos-Fourier della funzione spettrale S(Q, ω), alla funzione di scattering intermedia F(Q, t). (10) Il parametro temporale della funzione F(Q, t) è proporzionale all'intensità del campo magnetico di precessione. La scansione della perdita di polarizzazione in funzione dell'intensità del campo magnetico produce, quindi, una funzione di rilassamento che dipende dai processi dinamici all'interno del campione.

Figura 1: Fotografia dello spettrometro NSE a SNS (SNS-NSE) e schema del percorso di volo dei neutroni con i componenti funzionali più importanti. Da destra a sinistra: 1 = sorgente di neutroni; 2 = choppers-bender-polarizer-sistema otturatore secondario; 3 = guide di trasporto del fascio; 4 = pi/2 flipper per il primo giro di 90°; 5 = prima zona di precessione; 6 = pi flipper per rotazione a 180°; 7 = area del campione e ambiente del campione (qui viene mostrato il crioforno); 8 = seconda zona di precessione; 9 = pi/2 flipper per secondo giro di 90°; 10 = analizzatore; 11 = rilevatore. (Si noti che porzioni di 3, così come 2 e 1, sono situate dietro la parete blu all'interno della schermatura; gli elicotteri sono sostituiti da un selettore di velocità per NSE basato su reattore). Fare clic qui per visualizzare una versione più grande di questa figura.
Protocollo
Il presente lavoro caratterizza due proteine: una proteina anticorpale umana regolare IgG e l'MBP intrinsecamente disordinata. La forma liofilizzata delle proteine è stata ottenuta da fonti commerciali (vedi Tabella dei materiali).
1. Preparazione del campione proteico
- Preparare un tampone da 50 mM di fosfato di sodio + 0,1 M naCl pesando e sciogliendo i rispettivi reagenti solidi in acqua pesante (D2O) (vedi Tabella dei materiali). Questo è il solvente tampone deuterato per IgG.
- Regolare il pH della soluzione tampone a 6,6.
- Preparare un tampone di fosfato di sodio da 20 mM + urea da 6 M pesando e sciogliendo i rispettivi componenti solidi in acqua pesante (D2O). Questo è il solvente tampone deuterato per MBP.
- Regolare il pH della soluzione tampone a 4,7.
- Filtrare i solventi tampone utilizzando filtri di dimensioni dei pori da 0,2 μm (vedere Tabella dei materiali).
- Pesare e sciogliere le polveri liofilizzate proteiche purificate nei solventi deuterati per le rispettive proteine (Fasi 1.1.-1.2.) ad alta concentrazione proteica (~50 mg/ml).
- Caricare la soluzione proteica in cestelli dializzati con membrane di dialisi di 3,5 K MWCO e dializzare contro il tampone filtrato per 24 ore a 10 °C per MBP e 25 °C per IgG (vedi Tabella dei materiali) scuotendo leggermente i tubi per creare un gradiente di diffusione.
- Diluire la soluzione proteica utilizzando il tampone di dialisi in una serie di concentrazioni: 1, 2, 5, 10 e 50 mg/ml.
- Determinare le concentrazioni esatte utilizzando uno spettrofotometro Nanodrop (vedere Tabella dei materiali).
2. Caratterizzazione preliminare del campione mediante diffusione dinamica della luce (DLS)
- Caricare 80 μL di ciascuna soluzione proteica della serie di concentrazioni preparata sopra (Fase 1.6.) nella cella monouso DLS (vedere Tabella dei materiali) e determinare i coefficienti di diffusione, con una media di oltre 10 acquisizioni.
- Tracciare i coefficienti di diffusione traslazionale in funzione della concentrazione proteica ed estrapolare a concentrazione zero.
- Caricare ogni soluzione proteica della serie di concentrazioni nei tubi capillari di un viscosimetro (vedere Tabella dei materiali) e misurare la viscosità dinamica.
- Tracciare le viscosità dinamiche misurate in funzione della concentrazione proteica ed estrapolare a concentrazione zero.
NOTA: L'estrapolazione della diffusione dls a concentrazione zero produce il valore della diffusione traslazionale per una singola proteina. L'estrapolazione della viscosità dinamica a concentrazione zero deve produrre il valore di viscosità dinamica misurato sperimentalmente per la soluzione tampone.
3. Raccolta di scattering a piccolo angolo (neutroni o raggi X)
- Misurare lo scattering neutronico a piccolo angolo (SANS) e/o lo scattering a raggi X a piccolo angolo (SAXS) (vedere Tabella dei materiali) su quattro concentrazioni proteiche, preferibilmente 2 mg/mL, 5 mg/mL, 10 mg/mL e 50 mg/mL.
- Normalizzare gli spettri SANS e SAXS in base alla concentrazione proteica.
- Adatta il fattore di forma proteico P(Q) agli spettri SANS e SAXS utilizzando l'ottimizzazione dell'insieme31 e/o il software SasView32 .
- Calcola il fattore di struttura S(Q, c) dividendo il segnale SANS e SAXS con P(Q) per ogni concentrazione.
NOTA: I lettori interessati a misurare e interpretare i dati di scattering a piccolo angolo come supporto per le misurazioni NSE sono invitati a consultare accuratamente i riferimenti 23,28,31,32,33.
4. Misurazione delle NSE
- Impostare l'esperimento e montare il campione seguendo i passaggi seguenti.
- Selezionare lo spessore della cella per il carico del campione in base alla concentrazione della soluzione proteica, alla temperatura necessaria per la misurazione e alla quantità di soluzione disponibile.
NOTA: Il presente studio ha utilizzato contenitori di quarzo trasparente caricatore dall'alto di 40 mm x 30 mm x 4 mm. - Pulire ripetutamente la cella, alternando detersivo per piatti privo di fosfati (vedi Tabella dei materiali), acqua deionizzata ed etanolo al 70%.
- Asciugare la cella nel forno a convezione; non superare gli 80 °C per le celle al quarzo.
- Caricare 4 ml di soluzione proteica nella cellula e chiudere con tappi. Utilizzare un film di cera o qualsiasi sigillante (vedere Tabella dei materiali) per sigillare le celle del campione.
NOTA: Nel presente studio, sono stati utilizzati 4,8 mL di soluzione a ~50 mg/mL per ottenere un'intensità di dispersione sufficiente. - Caricare 4 mL di tampone per dialisi in un contenitore identico a quello del campione proteico e del sigillo.
- Trasportare i campioni alla beamline, chiudere l'otturatore ed entrare nell'area34 della grotta dell'involucro dello spettrometro.
- Montare la cella campione sul portacampioni in alluminio stringendo le viti e le piastre di tenuta (Figura 2, pannello di sinistra).
NOTA: è possibile montare due celle campione contemporaneamente, dato che è necessario lo stesso protocollo di misurazione per tutti i campioni. - Montare il campione di grafite e/o il campione di polvere di Al2O3 caricati in un contenitore identico al campione proteico. Questi sono gli standard forniti dal supporto della beamline SNS-NSE.
- Posizionare il portacampioni facendolo scorrere delicatamente nella lattina del sistema di forzatura della temperatura (TFS, vedere Tabella dei materiali).
NOTA: TFS è l'ambiente di campionamento più utilizzato presso SNS-NSE e pompa aria secca nel contenitore del campione per raggiungere la temperatura desiderata (Figura 2, pannelli centrale e destro). - Chiudere il coperchio TFS e impostare la temperatura sul valore desiderato accedendo alla schermata interattiva del TFS.
- Montare la telecamera a neutroni (vedi Tabella dei materiali) per l'allineamento dei campioni al fascio.
- Spazzare l'involucro dello strumento, evacuare, chiudere le porte e aprire l'otturatore a fascio.
NOTA: le celle campione sono fornite dal supporto della beamline SNS-NSE. Per le celle campione disponibili e la libreria dell'ambiente di esempio, fare riferimento alla pagina Web della beamline SNS-NSE 7,35.
- Selezionare lo spessore della cella per il carico del campione in base alla concentrazione della soluzione proteica, alla temperatura necessaria per la misurazione e alla quantità di soluzione disponibile.
- Raccogli i dati NSE seguendo i passaggi seguenti.
- Allineare il campione nel fascio di neutroni usando la telecamera a neutroni e le quattro aperture di campionamento indipendenti.
- Aprire il software di raccolta dati SNS-NSE36 e raccogliere statistiche di esempio eseguendo scansioni di diffrazione per gli angoli di diffusione e la lunghezza d'onda desiderati.
- Impostare i parametri di misura in base alle statistiche raccolte per ciascun campione modificando le macro di misura fornite dallo scienziato dello strumento di assistenza.
- Avviare la scansione digitando il nome del protocollo nel prompt dei comandi e acquisire echi per l'esempio.
- Avviare la scansione e acquisire echi anche per il riferimento elastico e il solvente tamponato. Eseguire il funzionamento intermittente dell'otturatore del fascio per il cambio del campione.

Figura 2: Sistema di misurazione NSE. Pannello di sinistra: campioni di soluzione proteica in contenitore di quarzo montato con viti e piastre sul portacampioni in alluminio (Al). Il portacampioni Al offre la possibilità di montare due campioni contemporaneamente all'interno dell'ambiente campione. Pannello centrale: il campione del sistema di forzatura della temperatura (TFS) può essere montato nella fase del campione, la finestra del fascio di neutroni è sulla destra, mentre la telecamera a neutroni utilizzata per l'allineamento è visibile sul lato sinistro. Pannello di destra: posizionare il portacampioni con due campioni nel campione può funzionare con finestre in silicio (Si). Fare clic qui per visualizzare una versione più grande di questa figura.
5. Riduzione dei dati NSE
NOTA: SNS-NSE è dotato di un software dedicato denominato DrSpine (riduzione dei dati per spin-echo)37,38 disponibile sul cluster di analisi remota ORNL Neutron Sciences, una guida rapida per l'utente e supporto di aiuto integrato.
- Accedere a Neutron Sciences Remote Analysis Cluster (vedere Tabella dei materiali) con le credenziali utente ORNL e premere il pulsante Avvia sessione .
- Configurare il software di riduzione dei dati seguendo i passaggi seguenti.
- Nella directory utente, apri una finestra del terminale e digita: source/SNS/software/nse/etc/setup_nse.sh.
- Quindi, digitare: drspine_create_env.sh.
- Creare una cartella per la riduzione dei dati nella home directory e copiare gli script e le macro forniti dalla directory condivisa.
- Modificare, rinominare e salvare la macro di riduzione fornita di conseguenza.
- Digitare drspine al prompt dei comandi e premere invio per avviare l'ambiente di riduzione del software.
- Digitare "il nome della macro di riduzione" modificato al passaggio 5.4. nel prompt dei comandi all'interno dell'ambiente software e premere Invio.
6. Raccordo dati NSE
- Modificare lo script python "stapler-drspine.py", fornito dallo scienziato dello strumento di assistenza, con i nomi dei dati del file ridotti.
NOTA: Lo script python è disponibile gratuitamente per gli utenti dello strumento. - Modificare la funzione per adattarla alla libreria fornita.
- Digitare il nome dello script modificato "stapler-drspine.py" nel prompt dei comandi e premere INVIO per leggere, adattare e tracciare dati NSE ridotti.
NOTA: Lo Scienziato dello Strumento fornirà un modello per la macro di riduzione e lo script python "stapler-drspine.py" in grado di leggere e adattare i dati ridotti NSE. I dati NSE finali ridotti sono in formato ASCII e possono essere letti da vari software preferiti.
Risultati
Le proteine IgG provenienti da proteine MBP sieriche e bovine umane sono state ricostituite ad alte concentrazioni (~50 mg/mL) in tamponi D2O-base. Poiché le proteine sono state disciolte in alte concentrazioni, le soluzioni ottenute sono state soluzioni proteiche affollate. Le dinamiche studiate con NSE soffrono dell'ambiente affollato in cui risiedono le proteine (interazioni fattoriali di struttura ed effetti idrodinamici)5,28,39
Discussione
La spettroscopia NSE offre una visione unica e dettagliata della dinamica delle proteine, che altre tecniche spettroscopiche non possono produrre. Le misurazioni su una scala temporale estesa forniscono osservazioni della diffusione traslazionale e rotazionale delle proteine, come presentato qui. La dinamica segmentale e altre oscillazioni interne si rivelano come un forte decadimento della funzione di scattering coerente S(Q, t) su scala temporale breve e sono ben separate dai processi generali di rilassamento ...
Divulgazioni
L'autore non dichiara interessi finanziari concorrenti e nessun conflitto di interessi. Il contenuto del manoscritto si basa sulla lezione che l'autore ha presentato nella HANDS-Neutron Scattering Applications in Structural Biology School tra il 2019 e il 2021.
Riconoscimenti
Questa ricerca ha utilizzato risorse presso la Spallation Neutron Source (BL-15, BL-6, laboratori di biologia e chimica), un ufficio DOE della Science User Facility gestito dall'Oak Ridge National Laboratory. Questa ricerca ha utilizzato anche risorse del reattore MLZ-FRM2 garching (KWS-2, Phoenix-J-NSE) e del JCNS1 presso Forschungszentrum Jülich GmbH, Germania. L'autore riconosce il Dr. Ralf Biehl e il Dr. Andreas Stadler per il loro aiuto con la modellazione e il loro contributo alla ricerca sulle proteine IgG e MBP, il Dr. Piotr A. Żołnierczuk per il supporto alla riduzione dei dati NSE, il Dr. Changwoo Do per il supporto con le misurazioni SANS e Rhonda Moody e il Dr. Kevin Weiss per il supporto del laboratorio di biochimica SNS.
Materiali
| Name | Company | Catalog Number | Comments |
| Bovine MBP protein solution | Sigma-Aldrich | M1891 | lyophilized powder reconstituted in D2O |
| D2O - heavy water | Sigma-Aldrich | Product No. 151882 | liquid |
| Dionized water | in house | - | for washing / cleanning cells |
| DLS instrument | Zetasizer Nano ZS, FZ-Jülich | - | dynamic light scattering instrument |
| Elastic scattering standards | SNS-NSE, ORNL | - | Al2O3 and Graphite powders |
| Ethanol | Sigma-Aldrich | 65350-M | 70% ethanol for cleaning cells |
| IgG protein solution | Sigma-Aldrich | I4506 | lyophilized powder reconstituted in D2O |
| KWS-2 instrument | JCNS outstation at the MLZ, Garching, Germany | - | small angle neutron instrument |
| Liquinox dish detergent | Alconox | - | Phosphate-free liquid lab glassware cleaner |
| Na2HPO4·7H2O | Sigma-Aldrich | Product No.S9390 | disodium phosphate heptahydrate salt |
| NaCl | Sigma-Aldrich | Product No.S9888 | sodium chloride salt |
| NaH2PO4·H2O | Sigma-Aldrich | Product No. S9638 | monosodium phosphate monohydrate salt |
| Nanodrop spectrophotometer | Thermo Scientific | Catalog number: ND-2000 | NanoDrop 2000/2000c Spectrophotometer |
| Neutron alignment camera | NeutronOptics, Grenoble | NOG210222 | 100 x 100 mm camera with Sony IMX249 CMOS sensor |
| Parafilm M - wax parafilm | Bemis | Parafilm M - 5259-04LC PM996 | all-purpose laboratory film in cardboard dispenser |
| Phoenix-J-NSE Spectrometer | JCNS outstation at the MLZ, Garching, Germany | - | neutron spectrometer |
| SasView | https://www.sasview.org/ | ||
| SAXSpace, Anton Paar instrument | FZ-Jülich | - | small angle x-ray instrument |
| Slide-A-Lyzer dialysis membranes | Thermo Scientific | 88400-88405 | Slide-A-Lyzer mini dialysis devices tubes of 3.5 K MWCO |
| SNS Remote Analysis Cluster | Neutron Science Remote Analysis (sns.gov) | https://analysis.sns.gov | |
| SNS-NSE spectrometer | ORNL, Oak Ridge, TN, USA | - | neutron spectrometer |
| Sterile syringe filters | VWR | N.A. PN:28145-501 | 0.2 µm pore size filters |
| Temperature Forcing System (TFS) | SP Scientific | Part Number 100004055 | sample environment equipment |
| Urea -d4 | Sigma-Aldrich | Product No. 176087 | deuterated Urea salt |
| Viscometer | FZ-Jülich | - | falling ball viscometer |
Riferimenti
- Fitter, J., Gutberlet, T., Katsaras, J. . Neutron Scattering in Biology: Techniques and Applications. , (2006).
- Stadler, A., Monkenbusch, M., Biehl, R., Richter, D., Ollivier, J. Neutron spin-echo and TOF reveals protein dynamics in solution. Journal of the Physical Society of Japan. 82, (2013).
- Inoue, R. Large domain fluctuations on 50-ns timescale enable catalytic activity in phosphoglycerate kinase. Biophysical Journal. 99 (7), 2309-2317 (2010).
- Callaway, D. J. E., et al. Controllable activation of nanoscale dynamics in a disordered protein alters binding kinetics. Journal of Molecular Biology. 429 (7), 987-998 (2017).
- Stingaciu, L. R., Biehl, R., Changwoo, D., Richter, D., Stadler, A. M. Reduced internal friction by osmolyte interaction in intrinsically disordered myelin basic protein. Journal of Physical Chemistry Letters. 11 (1), 292-296 (2020).
- Monkenbusch, M., Richter, D. High resolution neutron spectroscopy-a tool for the investigation of dynamics of polymers and soft matter. Comptes Rendus Physique. , (2007).
- Richter, D., Monkenbusch, M., Schwahn, D. Neutron Scattering. Polymer Science: A Comprehensive Reference, 10 Volume Set. , (2012).
- Wilson, C. C. . Single Crystal Neutron Diffraction From Molecular Materials. , (2000).
- Marshall, W. . Theory of thermal neutron scattering. , (1971).
- . Roger Pynn Introduction & Neutron Scattering "Theory" Available from: https://neutrons.ornl.gov/sites/default/files/intro_to_neutron_scattering.pdf (2004)
- Richter, D. Neutron scattering in polymer physics. Physica B: Condensed Matter. 276-278, 22-29 (2000).
- Harroun, T. A., Wignall, G. D., Katsaras, J. Neutron scattering for biology. Neutron Scattering in Biology. , (2006).
- . SNS Available from: https://neutrons.ornl.gov/sna (2020)
- . HFIR Available from: https://neutrons.ornl.gov/hfir (2020)
- . CNCS Available from: https://neutrons.ornl.gov/cncs (2020)
- Perticaroli, S., et al. Description of hydration water in protein (green fluorescent protein) solution. Journal of the American Chemical Society. 139 (3), 1098-1105 (2017).
- Perticaroli, S., Nickels, J. D., Ehlers, G., Sokolov, A. P. Rigidity, secondary structure, and the universality of the boson peak in proteins. Biophysical Journal. 106 (12), 2667-2674 (2014).
- Miao, Y., et al. Coupled flexibility change in cytochrome p450cam substrate binding determined by neutron scattering, NMR, and molecular dynamics simulation. Biophysical Journal. 103 (10), 2167-2176 (2012).
- Mamontov, E., Zamponi, M., Hammons, S., Keener, W. S., Hagen, M., Herwig, K. W. BASIS: A new backscattering spectrometer at the SNS. Neutron News. 19 (3), 22-24 (2008).
- Mamontov, E. Microscopic diffusion processes measured in living planarians. Scientific Reports. 9, 8708 (2018).
- Alpert, Y., Cser, L., Faragó, B., Franěk, F., Mezei, F., Ostanevich, Y. M. Segmental flexibility in pig immunoglobulin G studied by neutron spin-echo technique. Biopolymers. 24 (9), 1769-1784 (1985).
- Bu, Z., Biehl, R., Monkenbusch, M., Richter, D., Callaway, D. J. E. Coupled protein domain motion in Taq polymerase revealed by neutron spin-echo spectroscopy. Proceedings of the National Academy of Sciences of the United States of America. 102 (49), 17646-17651 (2005).
- Biehl, R., et al. Direct observation of correlated interdomain motion in alcohol dehydrogenase. Physical Review Letters. 101, 138102 (2008).
- Farago, B., Li, J., Cornilescu, G., Callaway, D. J. E., Bu, Z. Activation of nanoscale allosteric protein domain motion revealed by neutron spin echo spectroscopy. Biophysical Journal. 99 (10), 3473-3482 (2010).
- Bu, Z., Callaway, D. J. E. Dynamic propagation of long-range allosteric signals by nanoscale protein domain motion revealed by neutron spin echo spectroscopy. Biophysical Journal. 100 (3), 223 (2011).
- Hong, L., et al. Structure and dynamics of a compact state of a multidomain protein, the mercuric ion reductase. Biophysical Journal. 107 (2), 393-400 (2014).
- Longeville, S., Stingaciu, L. -. R. Hemoglobin diffusion and the dynamics of oxygen capture by red blood cells. Scientific Reports. 7, 10448 (2017).
- Stingaciu, L. R., Ivanova, O., Ohl, M., Biehl, R., Richter, D. Fast antibody fragment motion: Flexible linkers act as entropic spring. Scientific Reports. 6, 22148 (2016).
- Hahn, E. L. Nuclear induction due to free larmor precession. Physical Review. 77, 297 (1950).
- Hahn, E. L. Spin echoes. Physical Review. 80, 580 (1950).
- Svergun, D. I., Koch, M. H. J. Small-angle scattering studies of biological macromolecules in solution. Reports on Progress in Physics. 66 (10), 1735-1782 (2003).
- . Sasview Available from: https://www.sasview.org (2020)
- Zhao, J. K., Gao, C. Y., Liu, D. The extended Q-range small-angle neutron scattering diffractometer at the SNS. Journal of Applied Crystallography. 43, 1068-1077 (2010).
- Ohl, M., et al. The high-resolution neutron spin-echo spectrometer for the SNS with τ ≥ 1 µs. Physica B: Condensed Matter. 350, 147-150 (2004).
- . SNS-NSE web page Available from: https://neutrons.ornl.gov/nse (2022)
- Ohl, M., et al. The spin-echo spectrometer at the Spallation Neutron Source (SNS). Nuclear Instruments and Methods in Physics Research, Section A: Accelerators, Spectrometers, Detectors and Associated Equipment. 696, 85-99 (2012).
- Zolnierczuk, P. A., Holderer, O., Pasini, S., Kozielewski, T., Stingaciu, L. R., Monkenbusch, M. Efficient data extraction from neutron time-of-flight spin-echo raw data. Journal of Applied Crystallography. 52 (5), 1022-1034 (2019).
- . Dr Spine hub Available from: https://jugit.fz-juelich.de/nse/drspine (2022)
- Stadler, A. M., et al. Internal nanosecond dynamics in the intrinsically disordered myelin basic protein. Journal of the American Chemical Society. 136 (19), (2014).
- . FZJ Available from: https://www.fz-juelich.de/portal/EN/AboutUs (2022)
- . KWS2 Available from: https://miz-garching.de/kws-2 (2022)
- Moorhouse, M., Barry, P. The protein databank. Bioinformatics Biocomputing and Perl. , (2005).
- Tria, G., Mertens, H. D. T., Kachala, M., Svergun, D. I. Advanced ensemble modelling of flexible macromolecules using X-ray solution scattering. IUCrJ. 2 (2), 207-217 (2015).
- Holderer, O., Monkenbusch, M., Schätzler, R., Kleines, H., Westerhausen, W., Richter, D. The JCNS neutron spin-echo spectrometer J-NSE at the FRM II. Measurement Science and Technology. 90 (4), 043107 (2008).
- Biehl, R., Monkenbusch, M., Richter, D. Exploring internal protein dynamics by neutron spin echo spectroscopy. Soft Matter. 7 (4), 1299-1307 (2011).
- Stingaciu, L. R., Ivanova, O., Ohl, M., Biehl, R., Richter, D. Fast antibody fragment motion: Flexible linkers act as entropic spring. Scientific Reports. 6, 22148 (2016).
- Stadler, A. M., et al. Internal nanosecond dynamics in the intrinsically disordered myelin basic protein. Journal of the American Chemical Society. 136 (19), 6987-6994 (2014).
- Biehl, R., Richter, D. Slow internal protein dynamics in solution. Journal of Physics: Condensed Matter. 26 (50), 503103 (2014).
- Hinsen, K. The molecular modeling toolkit: A new approach to molecular simulations. Journal of Computational Chemistry. 21 (2), 79-85 (2000).
- Uhlenbeck, G. E., Ornstein, L. S. On the theory of the Brownian motion. Physical Review. 36 (5), 823 (1930).
- Wang, M. C., Uhlenbeck, G. E. On the theory of the Brownian motion II. Reviews of Modern Physics. 17, 323 (1945).
- Callaway, D. J. E., Bu, Z. Nanoscale protein domain motion and long-range allostery in signaling proteins-a view from neutron spin echo spectroscopy. Biophysical Reviews. 7 (2), 165-174 (2015).
- Liu, Y. Intermediate scattering function for macromolecules in solution probed by neutron spin echo. Physical Review E. 95, 020501 (2017).
- Liu, Y. Short-time dynamics of proteins in solutions studied by neutron spin echo. Current Opinion in Colloid & Interface Science. 42, 147-156 (2019).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon