Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
Исследование динамики белка с помощью нейтронной спиновой эхо-спектроскопии
В этой статье
Резюме
Настоящий протокол описывает методы исследования структуры и динамики двух модельных белков, которые играют важную роль в здоровье человека. Метод сочетает в себе настольную биофизическую характеристику с нейтронной спиновой эхо-спектроскопией для доступа к динамике во временных и длинных масштабах, имеющих отношение к междоменным движениям белка.
Аннотация
Активность и функциональность большинства белков человеческого организма связаны с конфигурационными изменениями целых поддоменов в кристаллической структуре белка. Кристаллические структуры создают основу для любого расчета, который описывает структуру или динамику белка, в большинстве случаев с сильными геометрическими ограничениями. Однако эти ограничения от кристаллической структуры отсутствуют в растворе. Структура белков в растворе может отличаться от кристаллической из-за перестановок петель или поддоменов на шкале времени пико к наносекунде (т.е. временному режиму внутренней динамики белка). Настоящая работа описывает, как медленные движения на временных масштабах в несколько десятков наносекунд могут быть доступны с помощью рассеяния нейтронов. В частности, динамическая характеристика двух основных человеческих белков, внутренне неупорядоченного белка, который не имеет четко определенной вторичной структуры и классического белка антитела, решается с помощью нейтронной спиновой эхо-спектроскопии (NSE) в сочетании с широким спектром лабораторных методов характеристики. Дальнейшее понимание динамики белкового домена было достигнуто с использованием математического моделирования для описания экспериментальных данных нейтронов и определения пересечения между комбинированными диффузными и внутренними движениями белка. Извлечение внутреннего динамического вклада в промежуточную функцию рассеяния, полученного из NSE, включая временную шкалу различных движений, позволяет дополнительно увидеть механические свойства отдельных белков и мягкость белков в их почти естественной среде в переполненном белковом растворе.
Введение
Зондирующая динамика мягкого вещества с нейтронами
Исследование динамических свойств белков и пептидов является важной частью биофизических исследований, и сегодня существует множество хорошо разработанных методов для доступа к широкому спектру энергетических ландшафтов1. Соотнесение экспериментально выявленной динамики белков с их биологической функцией является гораздо более сложной задачей, требующей сложных математических моделей и компьютерного моделирования динамики. Важность нейтронной спектроскопии для анализа движений белка была подчеркнута в нескольких хорошо принятых и широко признанных исследованиях 1,2,3,4,5. Прежде чем исследовать разнообразный энергетический ландшафт внутренней динамики белка, требуется краткий обзор динамических процессов в мягкой материи и того, как нейтроны могут получить к ним доступ.
Чувствительность нейтронов к изотопной конфигурации и тип взаимодействий, которые они проявляют с мягким веществом, делает рассеяние нейтронов одним из наиболее универсальных методов исследования6. Существует широкий спектр шкал длин корреляции и времени корреляции, к которым могут получить доступ нейтроны, от ядерных возбуждений и атомных колебаний до коллективных движений и медленных процессов релаксации, таких как изотропные вращения и диффузионные движения. При исследовании рассеянных нейтронов на предмет их переноса энергии можно выделить три основных взаимодействия: упругое рассеяние, при котором отсутствует энергообмен между входящим нейтроном и частицей в образце; неупругое рассеяние с большим, поддающимся количественной оценке энергетическим обменом между нейтроном и частицей; и своеобразный случай квазиупругого рассеяния, обозначающий очень малый перенос энергии по сравнению с падающей энергией нейтронов 1,7. Эти взаимодействия дают точную информацию об исследуемом материале и составляют теоретическую основу широкого спектра методов рассеяния нейтронов.
При упругом рассеянии детектор записывает направления нейтронов как дифракционную картину, которая показывает положение атомов образца относительно друг друга. Получена информация о корреляциях атомных положений (т.е. интегральная интенсивность S(Q) относительно переноса импульса Q, относящаяся только к структурной информации). Этот принцип лежит в основе дифракции нейтронов8.
Сложность возникает, когда передача энергии больше не равна нулю из-за возбуждений и внутренних флуктуаций в материале образца. Это составляет основу нейтронной спектроскопии, в которой рассеянные нейтроны исследуются как функция как переноса энергии E , так и переноса импульса Q. Получена динамическая и структурная информация. Нейтронная спектроскопия измеряет одинаковую интегральную интенсивность S(Q) для передачи энергии (т.е. изменение скорости нейтронов из-за рассеяния образцов, S(Q,ω) = S(Q, E), что также называется коэффициентом динамической структуры)9.
Для расчета рассеяния из материала более адекватно использовать парную корреляционную функцию 7,10. В дифракционном случае статическая парная корреляционная функция G(r) дает вероятность нахождения центра частицы на заданном расстоянии r от центра другой частицы. Спектроскопия обобщает статическую парную корреляционную функцию и включает энергию/частоту/время в уравнение рассеяния. Парная корреляционная функция G(r) становится функцией времени G(r, t), которая может быть разложена на отдельную корреляционную функцию пары атомов GD(r, t) и самокорреляционную функцию GS(r, t). Они описывают два типа корреляций: парно-коррелированные движения атомов, которые управляют когерентным рассеянием, и самокорреляция, которая управляет некогерентным рассеянием10.
Когерентное рассеяние является рассеянием от «среднего» и зависит от относительной фазы рассеянных волн. В малоугловом режиме рассеяния рассеянные нейтронные волны от разных центров рассеяния (разных атомов) конструктивно взаимодействуют (имеют сходные фазы), а коллективное движение атомов наблюдается с сильным усилением интенсивности. Когерентное рассеяние по существу описывает рассеяние одного нейтрона от всех ядер в образце10.
Когда между рассеянными нейтронными волнами из разных центров не возникает конструктивной интерференции, во времени следует один атом, и наблюдается самокорреляция между положением атома в момент времени t = 0 и того же атома в момент времени t. Таким образом, информация об относительных положениях атомов теряется, а фокус делается только на локальных флуктуациях. Рассеяние от локальных флуктуаций управляет некогерентным рассеянием. Некогерентное рассеяние изотропно, способствует фоновому сигналу и ухудшает сигнал-шум10,11.
Объединив все вышеперечисленное, мы выделяем четыре основных процесса рассеяния нейтронов10: (1) упругий когерентный (измеряет корреляции атомных положений), (2) неупругий когерентный (измеряет коллективные движения атомов), (3) упругий некогерентный (способствует фону, уменьшает интенсивность рассеяния на коэффициент Дебая-Уоллера (DWF) и измеряет коэффициент упругой некогерентной структуры (EISF), описывая геометрию диффузионных движений в замкнутой геометрии, и (4) неупругий некогерентный (измеряет динамику одного атома и самокорреляцию).
Динамические процессы, к которым нейтроны могут получить доступ в биологии, варьируются от демпфирования низкочастотных атомных и молекулярных колебаний, взаимодействия молекул растворителей с биоповерхностями и диффузионных процессов в гидратационном слое макромолекул и ограниченной геометрии до прямых поступательных, вращательных и кувыркающихся диффузных движений, белковых доменов и аллостерических движений1 . Широкое разнообразие нейтронных методов и приборов для измерения динамики белка основано на том, как достигается ахроматизация падающего или исходящего пучка нейтронов и как выполняется энергетический анализ рассеянных нейтронов. От трехосевых до спектрометров времени полета, обратного рассеяния и спин-эхо можно исследовать динамические процессы с характерным временем от 1 х 10-14 с до 1 х 10-6 с (фемтосекунды до микросекунд)12.
Национальная лаборатория Оук-Ридж с двумя известными источниками нейтронов, источником нейтронов Spallation - SNS13 и реактором с высоким изотопным потоком - HFIR14, имеет один из лучших наборов спектрометров для исследования динамики в биоматериалах. Некоторые из наиболее красноречивых примеров включают использование спектрометра холодного нейтронного измельчителя (CNCS) в SNS15 для исследования динамического возмущения гидратационной воды вокруг зеленого флуоресцентного белка в растворе16 или субпикосекундных коллективных колебаний нескольких белков17. Повторяющаяся проблема исследований неупругого рассеяния нейтронов заключается в том, что некоторые биологические процессы слишком медленны, чтобы их можно было наблюдать. Без экстремальных установок, которые приводят к огромной потере интенсивности нейтронов, спектрометры времени пролета ограничены разрешением энергии 10 мкЭВ, что соответствует максимальной шкале времени ~ 200 пс10,11. Этого недостаточно для наблюдения крупномасштабных движений в белках. Поэтому часто требуются приборы с более высоким энергетическим разрешением, такие как спектрометры обратного рассеяния. Сочетание методов времени полета и обратного рассеяния оказалось мощным для исследования изменения внутренней динамики цитохрома P450cam (CYP101), фермента, который катализирует гидроксилирование камфоры18.
Микроскопическая диффузия, измеренная спектрометром обратного рассеяния в SNS-BASIS19 , была удивительно хорошо определена и могла быть разделена на диффузионность воды (гидратационная, цитоплазматическая и объемная вода) и диффузионность клеточных компонентов у плоских плоских червей, первого живого животного, изучаемого рассеянием нейтронов20 . Обратное рассеяние является спектроскопическим методом с высоким разрешением, но оно также ограничено несколькими мкэВ = несколькими наносекундами, в то время как медленная динамика в биоматериалах также проявляется как время выживания корреляции между положением атома или ориентацией спина (например, релаксационные процессы, которые регулярно происходят в диапазоне времени от десяти до сотен наносекунд).
Нейтронная спиновая эхо-спектроскопия (NSE) является единственным методом рассеяния нейтронов, достигающим такого высокого разрешения. В отличие от других нейтронных методов, NSE не требует ахроматизации пучка, поскольку использует квантово-механическую фазу нейтронов, которая является их магнитными моментами. Манипулирование магнитными моментами позволяет использовать широкое распределение длин волн пучка нейтронов, при этом методика чувствительна к очень малым изменениям скорости нейтронов порядка 1 х 10-4. NSE был успешно использован для исследования медленной динамики белков в растворе для многих белков. Среди этих многочисленных новаторских исследований мы признаем изучение сегментарной гибкости иммуноглобулина свиней21; связанные доменные движения в Taq полимеразе22; доменные движения в тетрамере дрожжевого спиртдегидрогеназы23; изменение конформации в фосфоглицераткиназе при связывании субстрата3; активация доменных движений и динамическое распространение аллостерических сигналов в белке регуляторного кофактора обмена Na+/H+ 1 (NHERF1) 4,24,25; динамика компактного состояния ртутной ионредуктазы26; и диффузия гемоглобина в эритроцитах27. Два более поздних исследования в области динамики белка выявили гибкость антитела человека Иммуноглобулина G (IgG) в качестве энтропийного источника28 и характеристики вклада растворителя в динамику внутренне неупорядоченного основного белка миелина (MBP)5.
В настоящей статье объясняются основные принципы NSE, множественные подготовительные методы, рекомендуемые для тщательного исследования динамики белка, а также методология и экспериментальный протокол сбора данных NSE на спектрометре NSE в SNS, SNS-NSE. Протокол характеризует два белка: IgG, обычный белок антител человека, и внутренне неупорядоченный белок MBP. Биофизические последствия, исследовательская актуальность примеров и ограничения метода обсуждаются кратко.
NSE спектроскопия, метод измерения медленной динамики
NSE - это поляризованный метод, который использует нейтронное время пролета для измерения обмена энергией (потери поляризации) из-за квазиупругого взаимодействия между нейтронами и атомами в образце. В основе спектроскопии NSE лежат два основных принципа: (1) способность спина нейтрона прецессировать в магнитном поле с частотой, пропорциональной магнитной силе, а именно частоте 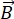 Лармора29, и (б) спин-эхо или эхо Ханна, представляющее собой манипулирование и перефокусировку поляризационного сигнала при подаче серии радиочастотных импульсов30.
Лармора29, и (б) спин-эхо или эхо Ханна, представляющее собой манипулирование и перефокусировку поляризационного сигнала при подаче серии радиочастотных импульсов30.
Основы процесса NSE можно резюмировать в нескольких простых шагах 6,11, используя рисунок 1. (1) Пучок нейтронов, создаваемый источником (положение 1), поляризован (положение 2), направляется и транспортируется (положение 3) и поступает на вход в спектрометр NSE, где он поворачивается на 90° первым пи-полуфлаппером (положение 4). (2) Поляризованный пучок (например, магнитные моменты нейтронов) становится перпендикулярным линиям магнитного поля первого магнита (первая зона прецессии, положение 5) и начинает прецессировать. (3) На конце магнита спины нейтронов накапливают определенный угол прецессии, пропорциональный напряженности магнитного поля и времени полета, проведенному внутри (в основном обратно пропорционален скорости нейтрона). Отдельные скорости нейтронов кодируются в пределах угла прецессии в конце первой зоны прецессии. (4) Вблизи положения образца пи-флиппер (положение 6) меняет ориентацию спина на 180°, изменяя знак угла прецессии. (5) Нейтроны взаимодействуют с молекулами образца (позиция 7) и рассеиваются. (6) Рассеянные нейтроны входят и прецессируют во второй зоне прецессии (позиция 8), но становятся обратно ориентированными. (7) Другой пи-полуплащ (положение 9) используется для поворота ориентации спина от перпендикулярного к горизонтальному направлению. Это остановит прецессию, переводя угол прецессии φ в поляризацию, пропорциональную cos(φ). (8) Анализатор (позиция 10) выбирает нейтроны на основе одной ориентации. Если взаимодействие с образцом упругое, скорость нейтрона не изменится. Нейтроны будут проводить одинаковое количество времени, пролетая в первой и второй зонах прецессии, а накопленные углы прецессии полностью восстанавливаются. Полная поляризация восстанавливается на детекторе (позиция 11) как эхо исходной поляризации (т.е. спин-эхо). (9) Однако в NSE рассеяние является квазиупругим, поэтому небольшой обмен энергией между нейтронами и молекулами образца приводит к различным скоростям нейтронов после рассеяния образцом. Из-за различных скоростей нейтроны проведут дополнительное время, пролетая через вторую зону прецессии, и не смогут должным образом восстановить свой угол прецессии. На детекторе извлекается частичная поляризация, а потеря поляризации из-за спиновой релаксации пропорциональна кос-фурье-преобразованию спектральной функции S(Q, ω), промежуточной функции рассеяния F(Q, t). (10) Параметр времени функции F(Q, t) пропорционален напряженности магнитного поля прецессии. Сканирование потери поляризации в зависимости от напряженности магнитного поля дает, следовательно, релаксационную функцию, которая зависит от динамических процессов внутри образца.

Рисунок 1: Фотография спектрометра NSE в SNS (SNS-NSE) и схемы траектории полета нейтронов с наиболее важными функциональными компонентами. Справа налево: 1 = источник нейтронов; 2 = система измельчителей-бендеров-поляризаторов-вторичных затворов; 3 = направляющие для транспортировки луча; 4 = пи/2 флиппера для первого вращения на 90°; 5 = первая зона прецессии; 6 = пи-флиппер для вращения на 180°; 7 = площадь образца и среда отбора проб (здесь показана криопечь); 8 = вторая зона прецессии; 9 = пи/2 флиппера для второго вращения на 90°; 10 = анализатор; 11 = детектор. (Обратите внимание, что части 3, а также 2 и 1 расположены за синей стеной внутри экранирования; измельчители заменены селектором скорости для NSE на основе реактора). Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
протокол
Настоящая работа характеризует два белка: обычный белок антител человека IgG и внутренне неупорядоченный MBP. Лиофилизированную форму белков получали из коммерческих источников (см. Таблицу материалов).
1. Пробоподготовка белка
- Приготовьте буфер 50 мМ фосфата натрия + 0,1 М NaCl путем взвешивания и растворения соответствующих твердых реагентов в тяжелой воде (D2O) (см. Таблицу материалов). Это дейтерированный буферный растворитель для IgG.
- Отрегулируйте рН буферного раствора до 6,6.
- Приготовьте буфер 20 мМ фосфата натрия + 6 М мочевины путем взвешивания и растворения соответствующих твердых компонентов в тяжелой воде (D2O). Это дейтерированный буферный растворитель для MBP.
- Отрегулируйте рН буферного раствора до 4,7.
- Фильтруйте буферные растворители с помощью фильтров размером пор 0,2 мкм (см. Таблицу материалов).
- Взвешивают и растворяют очищенные белковые лиофилизированные порошки в дейтерированных растворителях для соответствующих белков (этапы 1.1.-1.2.) при высокой концентрации белка (~50 мг/мл).
- Загрузите белковый раствор в диализные корзины с диализными мембранами 3,5 К MWCO и диализуйте против фильтрованного буфера в течение 24 ч при 10 °C для MBP и 25 °C для IgG (см. Таблицу материалов) путем слегкаго встряхивания трубок для создания диффузионного градиента.
- Разбавляют белковый раствор с помощью диализного буфера в ряде концентраций: 1, 2, 5, 10 и 50 мг/мл.
- Определите точные концентрации с помощью спектрофотометра Nanodrop (см. Таблицу материалов).
2. Предварительная характеристика образца динамическим рассеянием света (DLS)
- Загрузите 80 мкл каждого белкового раствора из серии концентраций, приготовленной выше (этап 1.6.), в одноразовую ячейку DLS (см. Таблицу материалов) и определите коэффициенты диффузии, в среднем более 10 приобретений.
- Построить коэффициенты поступательной диффузии как функцию концентрации белка и экстраполировать на нулевую концентрацию.
- Загрузите каждый белковый раствор из серии концентраций в капиллярные трубки вискозиметра (см. Таблицу материалов) и измерьте динамическую вязкость.
- График динамической вязкости, измеренной как функция концентрации белка и экстраполируемой до нулевой концентрации.
ПРИМЕЧАНИЕ: Экстраполяция диффузии DLS до нулевой концентрации дает значение трансляционной диффузии для одного единственного белка. Экстраполяция динамической вязкости до нулевой концентрации должна давать динамическое значение вязкости, измеренное экспериментально для буферного раствора.
3. Коллекция малоуглового рассеяния (нейтронного или рентгеновского)
- Измерьте малоугловое рассеяние нейтронов (SANS) и/или малоугловое рентгеновское рассеяние (SAXS) (см. Таблицу материалов) на четырех концентрациях белка, предпочтительно 2 мг/мл, 5 мг/мл, 10 мг/мл и 50 мг/мл.
- Нормализуют спектры SANS и SAXS по концентрации белка.
- Подгонка форм-фактора белка P(Q) к спектрам SANS и SAXS с помощью оптимизации ансамбля31 и/или программного обеспечения SasView32 .
- Рассчитайте структурный коэффициент S(Q, c), разделив сигнал SANS и SAXS на P(Q) для каждой концентрации.
ПРИМЕЧАНИЕ: Читателям, заинтересованным в том, как измерять и интерпретировать данные малоуглового рассеяния в качестве поддержки измерений NSE, рекомендуется тщательно ознакомиться со ссылками 23,28,31,32,33.
4. Измерение NSE
- Настройте эксперимент и подключите образец, выполнив следующие действия.
- Выберите толщину ячейки для загрузки образца на основе концентрации белкового раствора, температуры, необходимой для измерения, и количества доступного раствора.
ПРИМЕЧАНИЕ: В настоящем исследовании использовались прозрачные кварцевые контейнеры верхнего погрузчика размером 40 мм х 30 мм х 4 мм. - Очищайте ячейку многократно, чередуя безфосфатное средство для мытья посуды (см. Таблицу материалов), деионизированную воду и 70% этанол.
- Высушите ячейку в конвекционной печи; не превышают 80 °C для кварцевых ячеек.
- Загрузите в клетку 4 мл белкового раствора и закройте колпачками. Используйте восковую пленку или любой герметик (см. Таблицу материалов) для герметизации клеток образца.
ПРИМЕЧАНИЕ: В настоящем исследовании для получения достаточной интенсивности рассеяния использовалось 4,8 мл раствора при ~50 мг/мл. - Загрузите 4 мл диализного буфера в тот же контейнер, что и образец белка и уплотнение.
- Транспортируйте образцы к линии луча, закройте затвор и войдите в зону34 пещеры в корпусе спектрометра.
- Установите ячейку образца на алюминиевый держатель образца, затянув винты и удерживающие пластины (рисунок 2, левая панель).
ПРИМЕЧАНИЕ: Можно установить две пробные ячейки одновременно, учитывая, что для всех образцов необходим один и тот же протокол измерения. - Установите образец графита и/или образец порошка Al2O3 , загруженный в контейнер, идентичный образцу белка. Это стандарты, обеспечиваемые поддержкой линии луча SNS-NSE.
- Поместите держатель образца, осторожно вставив его в банку системы температурного воздействия (TFS, см. Таблицу материалов).
ПРИМЕЧАНИЕ: TFS является наиболее используемой средой для отбора проб в SNS-NSE и закачивает сухой воздух в контейнер для образцов для достижения желаемой температуры (рисунок 2, средняя и правая панели). - Закройте крышку TFS и установите температуру на требуемое значение, открыв интерактивный экран TFS.
- Установите нейтронную камеру (см. Таблицу материалов) для выравнивания образцов по пучку.
- Подметите корпус прибора, отсоедините, закройте двери и откройте балочный затвор.
ПРИМЕЧАНИЕ: Образцы ячеек обеспечиваются поддержкой линии луча SNS-NSE. Доступные образцы ячеек и библиотека образцов среды см. на веб-странице 7,35 линии луча SNS-NSE.
- Выберите толщину ячейки для загрузки образца на основе концентрации белкового раствора, температуры, необходимой для измерения, и количества доступного раствора.
- Соберите данные NSE, выполнив следующие действия.
- Выровняйте образец в пучке нейтронов с помощью нейтронной камеры и четырех независимых апертур образца.
- Откройте программное обеспечение для сбора данных SNS-NSE36 и соберите статистику выборки, выполнив дифракционное сканирование для требуемых углов рассеяния и длины волны.
- Настройте параметры измерения на основе статистики, собранной для каждого образца, отредактировав макросы измерения, предоставленные помощником Специалиста по приборам.
- Начните сканирование, введя имя протокола в командной строке и получив эхо для образца.
- Начните сканирование и получите эхо также для упругого эталона и буферизованного растворителя. Выполните прерывистую операцию затвора луча для замены образца.

Рисунок 2: Измерительная система NSE. Левая панель: образцы белкового раствора в кварцевом контейнере, установленном с винтами и пластинами на алюминиевом (Al) держателе образца. Держатель для образцов Al обеспечивает возможность одновременного монтажа двух образцов в среде отбора проб. Средняя панель: Образец системы температурного воздействия (TFS) может быть установлен на стадии образца, окно нейтронного пучка находится справа, в то время как нейтронная камера, используемая для выравнивания, видна с левой стороны. Правая панель: размещение держателя образца с двумя образцами в образце может работать с кремниевыми (Si) окнами. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
5. Сокращение данных NSE
ПРИМЕЧАНИЕ: SNS-NSE оснащен специальным программным обеспечением под названием DrSpine (уменьшение данных для спин-эха)37,38, доступным в кластере удаленного анализа ORNL Neutron Sciences, кратким руководством пользователя и встроенной справочной поддержкой.
- Войдите в кластер удаленного анализа Neutron Sciences (см. Таблицу материалов) с учетными данными пользователя ORNL и нажмите кнопку Запустить сеанс .
- Настройте программное обеспечение для уменьшения объема данных, выполнив следующие действия.
- В каталоге пользователя откройте окно терминала и введите: source/SNS/software/nse/etc/setup_nse.sh.
- Далее введите: drspine_create_env.sh.
- Создайте папку для сокращения данных в домашнем каталоге и скопируйте предоставленные сценарии и макросы из общего каталога.
- Отредактируйте, переименуйте и сохраните макрос сокращения, предоставленный соответствующим образом.
- Введите drspine в командной строке и нажмите клавишу ВВОД , чтобы запустить среду сокращения программного обеспечения.
- Введите "имя макроса сокращения", отредактированное на шаге 5.4. в командной строке в программной среде и нажмите клавишу ВВОД.
6. Подгонка данных NSE
- Отредактируйте скрипт python "stapler-drspine.py", предоставленный помощником Instrument Scientist, с именами уменьшенных данных файла.
ПРИМЕЧАНИЕ: Скрипт python находится в свободном доступе для пользователей инструмента. - Отредактируйте функцию в соответствии с предоставленной библиотекой.
- Введите в командной строке имя редактируемого сценария "stapler-drspine.py" и нажмите клавишу ВВОД , чтобы прочитать, подогнать и отобразить уменьшенные данные NSE.
ПРИМЕЧАНИЕ: Instrument Scientist предоставит шаблон для макроса уменьшения и скрипта python "stapler-drspine.py", который может считывать и подгонять уменьшенные данные NSE. Окончательные сокращенные данные NSE представлены в формате ASCII и могут быть прочитаны различным предпочтительным программным обеспечением.
Результаты
Белок IgG из человеческой сыворотки и бычьих белков MBP был восстановлен в высоких концентрациях (~ 50 мг / мл) в буферахD2O-основания. Поскольку белки растворяли в высоких концентрациях, полученные растворы представляли собой переполненные белковые растворы. Динамика, исследованная с ...
Обсуждение
Спектроскопия NSE дает уникальное и подробное представление о динамике белков, которое другие спектроскопические методы не могут дать. Измерения в расширенном масштабе времени обеспечивают наблюдения как за поступательной, так и за ротационной диффузией белков, как представлено здесь...
Раскрытие информации
Автор заявляет об отсутствии конкурирующих финансовых интересов и отсутствии конфликта интересов. Содержание рукописи основано на лекции автора, представленной в Hands-Neutron Scattering Applications in Structural Biology School between 2019-2021.
Благодарности
В этом исследовании использовались ресурсы в Spallation Neutron Source (BL-15, BL-6, Biology and Chemistry labs), Офисе научного пользовательского центра Министерства энергетики США, управляемом Национальной лабораторией Оук-Ридж. В этом исследовании также использовались ресурсы реактора MLZ-FRM2 Garching (KWS-2, Phoenix-J-NSE) и JCNS1 в Forschungszentrum Jülich GmbH, Германия. Автор благодарит д-ра Ральфа Биля и д-ра Андреаса Штадлера за их помощь в моделировании и их вклад в исследования белков IgG и MBP, д-ра Петра А. Жолнерчука за поддержку сокращения данных NSE, д-ра Чангву До за поддержку измерений SANS и Ронду и д-ра Кевина Вайса за поддержку лаборатории биохимии SNS.
Материалы
| Name | Company | Catalog Number | Comments |
| Bovine MBP protein solution | Sigma-Aldrich | M1891 | lyophilized powder reconstituted in D2O |
| D2O - heavy water | Sigma-Aldrich | Product No. 151882 | liquid |
| Dionized water | in house | - | for washing / cleanning cells |
| DLS instrument | Zetasizer Nano ZS, FZ-Jülich | - | dynamic light scattering instrument |
| Elastic scattering standards | SNS-NSE, ORNL | - | Al2O3 and Graphite powders |
| Ethanol | Sigma-Aldrich | 65350-M | 70% ethanol for cleaning cells |
| IgG protein solution | Sigma-Aldrich | I4506 | lyophilized powder reconstituted in D2O |
| KWS-2 instrument | JCNS outstation at the MLZ, Garching, Germany | - | small angle neutron instrument |
| Liquinox dish detergent | Alconox | - | Phosphate-free liquid lab glassware cleaner |
| Na2HPO4·7H2O | Sigma-Aldrich | Product No.S9390 | disodium phosphate heptahydrate salt |
| NaCl | Sigma-Aldrich | Product No.S9888 | sodium chloride salt |
| NaH2PO4·H2O | Sigma-Aldrich | Product No. S9638 | monosodium phosphate monohydrate salt |
| Nanodrop spectrophotometer | Thermo Scientific | Catalog number: ND-2000 | NanoDrop 2000/2000c Spectrophotometer |
| Neutron alignment camera | NeutronOptics, Grenoble | NOG210222 | 100 x 100 mm camera with Sony IMX249 CMOS sensor |
| Parafilm M - wax parafilm | Bemis | Parafilm M - 5259-04LC PM996 | all-purpose laboratory film in cardboard dispenser |
| Phoenix-J-NSE Spectrometer | JCNS outstation at the MLZ, Garching, Germany | - | neutron spectrometer |
| SasView | https://www.sasview.org/ | ||
| SAXSpace, Anton Paar instrument | FZ-Jülich | - | small angle x-ray instrument |
| Slide-A-Lyzer dialysis membranes | Thermo Scientific | 88400-88405 | Slide-A-Lyzer mini dialysis devices tubes of 3.5 K MWCO |
| SNS Remote Analysis Cluster | Neutron Science Remote Analysis (sns.gov) | https://analysis.sns.gov | |
| SNS-NSE spectrometer | ORNL, Oak Ridge, TN, USA | - | neutron spectrometer |
| Sterile syringe filters | VWR | N.A. PN:28145-501 | 0.2 µm pore size filters |
| Temperature Forcing System (TFS) | SP Scientific | Part Number 100004055 | sample environment equipment |
| Urea -d4 | Sigma-Aldrich | Product No. 176087 | deuterated Urea salt |
| Viscometer | FZ-Jülich | - | falling ball viscometer |
Ссылки
- Fitter, J., Gutberlet, T., Katsaras, J. . Neutron Scattering in Biology: Techniques and Applications. , (2006).
- Stadler, A., Monkenbusch, M., Biehl, R., Richter, D., Ollivier, J. Neutron spin-echo and TOF reveals protein dynamics in solution. Journal of the Physical Society of Japan. 82, (2013).
- Inoue, R. Large domain fluctuations on 50-ns timescale enable catalytic activity in phosphoglycerate kinase. Biophysical Journal. 99 (7), 2309-2317 (2010).
- Callaway, D. J. E., et al. Controllable activation of nanoscale dynamics in a disordered protein alters binding kinetics. Journal of Molecular Biology. 429 (7), 987-998 (2017).
- Stingaciu, L. R., Biehl, R., Changwoo, D., Richter, D., Stadler, A. M. Reduced internal friction by osmolyte interaction in intrinsically disordered myelin basic protein. Journal of Physical Chemistry Letters. 11 (1), 292-296 (2020).
- Monkenbusch, M., Richter, D. High resolution neutron spectroscopy-a tool for the investigation of dynamics of polymers and soft matter. Comptes Rendus Physique. , (2007).
- Richter, D., Monkenbusch, M., Schwahn, D. Neutron Scattering. Polymer Science: A Comprehensive Reference, 10 Volume Set. , (2012).
- Wilson, C. C. . Single Crystal Neutron Diffraction From Molecular Materials. , (2000).
- Marshall, W. . Theory of thermal neutron scattering. , (1971).
- . Roger Pynn Introduction & Neutron Scattering "Theory" Available from: https://neutrons.ornl.gov/sites/default/files/intro_to_neutron_scattering.pdf (2004)
- Richter, D. Neutron scattering in polymer physics. Physica B: Condensed Matter. 276-278, 22-29 (2000).
- Harroun, T. A., Wignall, G. D., Katsaras, J. Neutron scattering for biology. Neutron Scattering in Biology. , (2006).
- . SNS Available from: https://neutrons.ornl.gov/sna (2020)
- . HFIR Available from: https://neutrons.ornl.gov/hfir (2020)
- . CNCS Available from: https://neutrons.ornl.gov/cncs (2020)
- Perticaroli, S., et al. Description of hydration water in protein (green fluorescent protein) solution. Journal of the American Chemical Society. 139 (3), 1098-1105 (2017).
- Perticaroli, S., Nickels, J. D., Ehlers, G., Sokolov, A. P. Rigidity, secondary structure, and the universality of the boson peak in proteins. Biophysical Journal. 106 (12), 2667-2674 (2014).
- Miao, Y., et al. Coupled flexibility change in cytochrome p450cam substrate binding determined by neutron scattering, NMR, and molecular dynamics simulation. Biophysical Journal. 103 (10), 2167-2176 (2012).
- Mamontov, E., Zamponi, M., Hammons, S., Keener, W. S., Hagen, M., Herwig, K. W. BASIS: A new backscattering spectrometer at the SNS. Neutron News. 19 (3), 22-24 (2008).
- Mamontov, E. Microscopic diffusion processes measured in living planarians. Scientific Reports. 9, 8708 (2018).
- Alpert, Y., Cser, L., Faragó, B., Franěk, F., Mezei, F., Ostanevich, Y. M. Segmental flexibility in pig immunoglobulin G studied by neutron spin-echo technique. Biopolymers. 24 (9), 1769-1784 (1985).
- Bu, Z., Biehl, R., Monkenbusch, M., Richter, D., Callaway, D. J. E. Coupled protein domain motion in Taq polymerase revealed by neutron spin-echo spectroscopy. Proceedings of the National Academy of Sciences of the United States of America. 102 (49), 17646-17651 (2005).
- Biehl, R., et al. Direct observation of correlated interdomain motion in alcohol dehydrogenase. Physical Review Letters. 101, 138102 (2008).
- Farago, B., Li, J., Cornilescu, G., Callaway, D. J. E., Bu, Z. Activation of nanoscale allosteric protein domain motion revealed by neutron spin echo spectroscopy. Biophysical Journal. 99 (10), 3473-3482 (2010).
- Bu, Z., Callaway, D. J. E. Dynamic propagation of long-range allosteric signals by nanoscale protein domain motion revealed by neutron spin echo spectroscopy. Biophysical Journal. 100 (3), 223 (2011).
- Hong, L., et al. Structure and dynamics of a compact state of a multidomain protein, the mercuric ion reductase. Biophysical Journal. 107 (2), 393-400 (2014).
- Longeville, S., Stingaciu, L. -. R. Hemoglobin diffusion and the dynamics of oxygen capture by red blood cells. Scientific Reports. 7, 10448 (2017).
- Stingaciu, L. R., Ivanova, O., Ohl, M., Biehl, R., Richter, D. Fast antibody fragment motion: Flexible linkers act as entropic spring. Scientific Reports. 6, 22148 (2016).
- Hahn, E. L. Nuclear induction due to free larmor precession. Physical Review. 77, 297 (1950).
- Hahn, E. L. Spin echoes. Physical Review. 80, 580 (1950).
- Svergun, D. I., Koch, M. H. J. Small-angle scattering studies of biological macromolecules in solution. Reports on Progress in Physics. 66 (10), 1735-1782 (2003).
- . Sasview Available from: https://www.sasview.org (2020)
- Zhao, J. K., Gao, C. Y., Liu, D. The extended Q-range small-angle neutron scattering diffractometer at the SNS. Journal of Applied Crystallography. 43, 1068-1077 (2010).
- Ohl, M., et al. The high-resolution neutron spin-echo spectrometer for the SNS with τ ≥ 1 µs. Physica B: Condensed Matter. 350, 147-150 (2004).
- . SNS-NSE web page Available from: https://neutrons.ornl.gov/nse (2022)
- Ohl, M., et al. The spin-echo spectrometer at the Spallation Neutron Source (SNS). Nuclear Instruments and Methods in Physics Research, Section A: Accelerators, Spectrometers, Detectors and Associated Equipment. 696, 85-99 (2012).
- Zolnierczuk, P. A., Holderer, O., Pasini, S., Kozielewski, T., Stingaciu, L. R., Monkenbusch, M. Efficient data extraction from neutron time-of-flight spin-echo raw data. Journal of Applied Crystallography. 52 (5), 1022-1034 (2019).
- . Dr Spine hub Available from: https://jugit.fz-juelich.de/nse/drspine (2022)
- Stadler, A. M., et al. Internal nanosecond dynamics in the intrinsically disordered myelin basic protein. Journal of the American Chemical Society. 136 (19), (2014).
- . FZJ Available from: https://www.fz-juelich.de/portal/EN/AboutUs (2022)
- . KWS2 Available from: https://miz-garching.de/kws-2 (2022)
- Moorhouse, M., Barry, P. The protein databank. Bioinformatics Biocomputing and Perl. , (2005).
- Tria, G., Mertens, H. D. T., Kachala, M., Svergun, D. I. Advanced ensemble modelling of flexible macromolecules using X-ray solution scattering. IUCrJ. 2 (2), 207-217 (2015).
- Holderer, O., Monkenbusch, M., Schätzler, R., Kleines, H., Westerhausen, W., Richter, D. The JCNS neutron spin-echo spectrometer J-NSE at the FRM II. Measurement Science and Technology. 90 (4), 043107 (2008).
- Biehl, R., Monkenbusch, M., Richter, D. Exploring internal protein dynamics by neutron spin echo spectroscopy. Soft Matter. 7 (4), 1299-1307 (2011).
- Stingaciu, L. R., Ivanova, O., Ohl, M., Biehl, R., Richter, D. Fast antibody fragment motion: Flexible linkers act as entropic spring. Scientific Reports. 6, 22148 (2016).
- Stadler, A. M., et al. Internal nanosecond dynamics in the intrinsically disordered myelin basic protein. Journal of the American Chemical Society. 136 (19), 6987-6994 (2014).
- Biehl, R., Richter, D. Slow internal protein dynamics in solution. Journal of Physics: Condensed Matter. 26 (50), 503103 (2014).
- Hinsen, K. The molecular modeling toolkit: A new approach to molecular simulations. Journal of Computational Chemistry. 21 (2), 79-85 (2000).
- Uhlenbeck, G. E., Ornstein, L. S. On the theory of the Brownian motion. Physical Review. 36 (5), 823 (1930).
- Wang, M. C., Uhlenbeck, G. E. On the theory of the Brownian motion II. Reviews of Modern Physics. 17, 323 (1945).
- Callaway, D. J. E., Bu, Z. Nanoscale protein domain motion and long-range allostery in signaling proteins-a view from neutron spin echo spectroscopy. Biophysical Reviews. 7 (2), 165-174 (2015).
- Liu, Y. Intermediate scattering function for macromolecules in solution probed by neutron spin echo. Physical Review E. 95, 020501 (2017).
- Liu, Y. Short-time dynamics of proteins in solutions studied by neutron spin echo. Current Opinion in Colloid & Interface Science. 42, 147-156 (2019).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены