É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Estudo da Dinâmica da Proteína via Espectroscopia de Eco de Neutron Spin
Neste Artigo
Resumo
O presente protocolo descreve métodos para investigar a estrutura e a dinâmica de duas proteínas modelo que têm um papel importante na saúde humana. A técnica combina caracterização biofísica de bancada com espectroscopia de eco de giro de nêutrons para acessar a dinâmica em escalas de tempo e comprimento relevantes para movimentos interdomínios proteicos.
Resumo
A atividade e funcionalidade da maioria das proteínas do corpo humano estão relacionadas a mudanças configurais de subdomínios inteiros dentro da estrutura cristalina proteica. As estruturas cristalinas constroem a base para qualquer cálculo que descreva a estrutura ou dinâmica de uma proteína, na maioria das vezes com fortes restrições geométricas. No entanto, essas restrições da estrutura cristalina não estão presentes na solução. A estrutura das proteínas na solução pode diferir do cristal devido a rearranjos de laços ou subdomínios no pico para escala de tempo nanossegundo (ou seja, o regime de tempo da dinâmica proteica interna). O presente trabalho descreve como as câmeras lentas em escalas de tempo de várias dezenas de nanossegundos podem ser acessadas usando dispersão de nêutrons. Em particular, a caracterização dinâmica de duas grandes proteínas humanas, uma proteína intrinsecamente desordenada que carece de uma estrutura secundária bem definida e uma proteína de anticorpos clássicos, é abordada pela espectroscopia de eco de spin de nêutrons (NSE) combinada com uma ampla gama de métodos de caracterização laboratorial. Mais insights sobre a dinâmica do domínio proteico foram obtidos usando modelagem matemática para descrever os dados experimentais de nêutrons e determinar o cruzamento entre movimentos difusivos combinados e proteicos internos. A extração da contribuição dinâmica interna para a função de dispersão intermediária obtida da NSE, incluindo a escala de tempo dos diversos movimentos, permite maior visão nas propriedades mecânicas de proteínas únicas e na maciez das proteínas em seu ambiente quase natural na solução de proteínas lotadas.
Introdução
Dinâmica de sondagem de matéria macia com nêutrons
Investigar as propriedades dinâmicas de proteínas e peptídeos é uma parte importante da pesquisa biofísica, e muitos métodos bem desenvolvidos existem hoje para acessar uma ampla gama de paisagens energéticas1. Relacionar a dinâmica experimentalmente revelada das proteínas à sua função biológica é uma tarefa muito mais difícil, exigindo modelos matemáticos complexos e simulações dinâmicas auxiliadas por computador. A importância da espectroscopia de nêutrons para a análise de movimentos proteicos tem sido enfatizada em diversos estudos bem recebidos e amplamente reconhecidos 1,2,3,4,5. Antes de explorar a paisagem energética diversificada da dinâmica interna de proteínas, uma breve visão geral dos processos dinâmicos em matéria suave e como os nêutrons podem acessá-los é necessária.
A sensibilidade dos nêutrons à configuração isotópica e o tipo de interações que exibem com matéria macia faz da dispersão de nêutrons uma das técnicas de investigação mais versáteis6. Há um amplo espectro de escalas de comprimento de correlação e tempos de correlação que os nêutrons podem acessar, desde excitações nucleares e vibrações atômicas até movimentos coletivos e processos de relaxamento lentos, como rotações isotrópicas e movimentos difusivos. Ao investigar os nêutrons dispersos para sua transferência de energia, três interações principais podem ser distinguidas: a dispersão elástica, na qual não há troca de energia entre nêutrons e partículas de entrada na amostra; a dispersão inelástica, com uma grande troca de energia quantificável entre nêutrons e partículas; e o caso peculiar de dispersão quase elástica que designa uma transferência de energia muito pequena em comparação com o incidente de energia de nêutrons 1,7. Essas interações dão informações precisas sobre o material investigado e formam a base teórica de uma ampla variedade de técnicas de dispersão de nêutrons.
Em dispersão elástica, o detector registra as direções dos nêutrons como um padrão de difração, que mostra a posição dos átomos amostrais em relação um ao outro. Informações sobre as correlações das posições atômicas são adquiridas (ou seja, intensidade integrada S(Q) relativa à transferência de impulso Q, que diz respeito apenas à informação estrutural). Este princípio forma a base da difração de nêutrons8.
A complexidade surge quando a transferência de energia não é mais zero devido a excitações e flutuações internas no material amostral. Isso forma a base da espectroscopia de nêutrons, na qual os nêutrons dispersos são investigados em função tanto da transferência de energia E quanto da transferência de impulso Q. Informações dinâmicas e estruturais são obtidas. A espectroscopia de nêutrons mede a mesma intensidade integrada S(Q) para transferência de energia (ou seja, mudança de velocidade dos nêutrons devido à dispersão amostral, S(Q,ω) = S(Q, E), que também é referido como o fator de estrutura dinâmica)9.
Para calcular a dispersão de um material, é mais adequado usar a função de correlação do par 7,10. No caso da difração, a função de correlação do par estático G(r) dá a probabilidade de encontrar o centro de uma partícula a uma determinada distância r do centro de outra partícula. A espectroscopia generaliza a função de correlação do par estático e inclui energia/frequência/tempo na equação de dispersão. A função de correlação de par G(r) torna-se uma função do tempo G(r, t), que pode ser decomposta em uma função de correlação de par de átomos distinta GD(r, t), e uma função de auto-correlação GS(r, t). Estes descrevem dois tipos de correlações: movimentos correlacionados por pares de átomos que regem a dispersão coerente, e a auto-correlação que rege a dispersão incoerente10.
A dispersão coerente é a dispersão da "média" e depende da fase relativa das ondas dispersas. No regime de dispersão de pequenos ângulos, as ondas de nêutrons dispersas de diferentes centros de dispersão (diferentes átomos) interferem construtivamente (têm fases semelhantes), e o movimento coletivo dos átomos é observado com forte aumento de intensidade. A dispersão coerente descreve essencialmente a dispersão de um único nêutron de todos os núcleos da amostra10.
Quando não ocorre interferência construtiva entre as ondas de nêutrons dispersas de diferentes centros, um único átomo é seguido no tempo, e a auto-correlação entre a posição do átomo no momento t = 0 e o mesmo átomo no momento em que t é observado. Assim, as informações sobre as posições relativas dos átomos são perdidas, e o foco está apenas nas flutuações locais. A dispersão das flutuações locais rege a dispersão incoerente. A dispersão incoerente é isotrópica, contribui para o sinal de fundo e degrada o sinal-para-ruído10,11.
Combinando todos os processos acima, distinguimos quatro grandes processos de dispersão de nêutrons10: (1) elástico coerente (mede as correlações das posições atômicas), (2) inelástico coerente (mede movimentos coletivos de átomos), (3) elástico incoerente (contribui para o fundo, reduz a intensidade de dispersão pelo fator Debye-Waller (DWF) e mede o fator de estrutura incoerente elástico (EISF), descrevendo a geometria dos movimentos difusivos na geometria confinada, e (4) incoerentes inelesses (mede dinâmica de átomo único e auto correlação).
Os processos dinâmicos que os nêutrons podem acessar na biologia vão desde o amortecimento de vibrações atômicas e moleculares de baixa frequência, a interação de moléculas de solventes com bio-superfícies e processos de difusão na camada de hidratação de macromoléculas e geometria confinada, até movimentos translacionais, rotacionais e difusivos de curto alcance, e domínios proteicos e movimentos alotéricos1 . A ampla diversidade de métodos e instrumentos de nêutrons para medir a dinâmica proteica baseia-se na forma como a acromatização do feixe de nêutrons incidente ou saída é alcançada e como a análise energética dos nêutrons dispersos é realizada. De eixo triplo a espectrômetros de retorno e spin-echo, pode-se explorar processos dinâmicos com tempos característicos entre 1 x 10-14 s e 1 x 10-6 s (femtosegundos a microsegundos)12.
Oak Ridge National Laboratory, com suas duas renomadas fontes de nêutrons, a Fonte de Nêutrons Spallation - SNS13 e o Reator de Fluxo de Alto Isótopo - HFIR14, tem uma das melhores suítes de espectrômetros para investigar dinâmicas em bioquímicos. Alguns dos exemplos mais eloquentes incluem o uso do espectrômetro de helicóptero de nêutrons frio (CNCS) no SNS15 para investigar a perturbação dinâmica da água de hidratação em torno da proteína fluorescente verde na solução16 ou as vibrações coletivas sub-picosegundos de várias proteínas17. Um problema recorrente das investigações inelásticas de dispersão de nêutrons é que alguns processos biológicos são muito lentos para serem observados. Sem configurações extremas que levam a uma enorme perda de intensidade de nêutrons, os espectrômetros de tempo de voo são limitados à resolução de energia de 10 μeV, correspondendo a uma escala de tempo máxima de ~200 ps10,11. Isso não é suficiente para observar movimentos em larga escala em proteínas. Portanto, instrumentos com maior resolução de energia, como os espectrômetros de backscattering, são frequentemente necessários. A combinação das técnicas de tempo de voo e backscattering tem se mostrado poderosa para investigar a mudança na dinâmica interna do Cytochrome P450cam (CYP101), uma enzima que catalisa a cânfora de hidroxiagem18.
A difusividade microscópica medida pelo espectrômetro de backscattering no SNS-BASE19 foi surpreendentemente bem definida e poderia ser separada na difusividade da água (hidratação, citoplasmático e água a granel) e a difusividade dos constituintes celulares em flatworms planárias, o primeiro animal vivo a ser estudado pela dispersão de nêutrons20 . Backscattering é uma técnica espectroscópica de alta resolução, mas também é limitada a vários μeV = vários nanossegundos, enquanto a dinâmica lenta em biomateriais também se manifesta como o tempo de sobrevivência da correlação entre posição atômica ou orientações de spin (por exemplo, processos de relaxamento, que acontecem regularmente na faixa de tempo de dez a centenas de nanossegundos).
A espectroscopia de eco de giro de nêutrons (NSE) é a única técnica de dispersão de nêutrons a atingir tal alta resolução. Ao contrário de outras técnicas de nêutrons, a NSE não requer acromatização do feixe, pois usa a fase mecânica quântica dos nêutrons, que são seus momentos magnéticos. A manipulação de momentos magnéticos permite o uso de uma ampla distribuição de comprimento de onda de feixe de nêutrons, enquanto a técnica é sensível a pequenas mudanças de velocidade de nêutrons na ordem de 1 x 10-4. A NSE tem sido usada com sucesso para investigar a lenta dinâmica das proteínas em solução para muitas proteínas. Entre esses muitos estudos pioneiros, reconhecemos o estudo da flexibilidade segmental da imunoglobulinasuína 21; os movimentos de domínio acoplado na polimerase Taq22; os movimentos de domínio no tetramer do álcool levedura desidrogenase23; a alteração da conformação na quinase fosfoglicerato sobre a ligação do substrato3; a ativação dos movimentos de domínio e a propagação dinâmica de sinais aluséricos na proteína de cofactor regulatório de câmbio Na+/H+ 1 (NHERF1) 4,24,25; a dinâmica de um estado compacto de redução de íons mercuricos26; e a difusão da hemoglobina em glóbulos vermelhos27. Dois estudos mais recentes em dinâmica proteica expuseram a flexibilidade do anticorpo humano Imunoglobulina G (IgG) como uma mola entropica28 e as características da contribuição do solvente para a dinâmica da proteína básica de mielina intrinsecamente desordenada (MBP)5.
O presente artigo explica os princípios básicos da NSE, os múltiplos métodos preparatórios recomendados para uma investigação minuciosa da dinâmica proteica, bem como a metodologia e o protocolo experimental para aquisição de dados NSE no espectrômetro NSE na SNS, SNS-NSE. O protocolo caracteriza duas proteínas: IgG, uma proteína de anticorpos humanos regular, e a proteína intrinsecamente desordenada MBP. As implicações biofísicas, a relevância da pesquisa dos exemplos e as limitações da técnica são discutidas brevemente.
Espectroscopia NSE, o método para medições dinâmicas lentas
NSE é uma técnica polarizada que usa o tempo de voo de nêutrons para medir a troca de energia (perda de polarização) devido à interação quase elástica entre nêutrons e átomos em uma amostra. No núcleo da espectroscopia NSE estão dois princípios básicos: (1) a capacidade do giro de nêutrons para precess no campo magnético com uma frequência proporcional à força 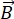 magnética , ou seja, a frequência larmor29, e (b) o spin-echo ou Hann echo, representando a manipulação e refoco do sinal de polarização ao aplicar uma série de pulsos de radiofrequência30.
magnética , ou seja, a frequência larmor29, e (b) o spin-echo ou Hann echo, representando a manipulação e refoco do sinal de polarização ao aplicar uma série de pulsos de radiofrequência30.
O básico do processo NSE pode ser resumido em algumas etapas simples 6,11 usando a Figura 1. (1) O feixe de nêutrons produzido pela fonte (posição 1) é polarizado (posição 2), guiado e transportado (posição 3), e chega à entrada do espectrômetro NSE, onde é girado por 90° pela primeira nadadeira pi-half (posição 4). (2) O feixe polarizado (por exemplo, momentos magnéticos de nêutrons) torna-se perpendicular às linhas de campo magnético do primeiro ímã (primeira zona de pré-cessão, posição 5) e começa a precess. (3) No final do ímã, os giros de nêutrons acumulam um certo ângulo de pré-cessão proporcional à força do campo magnético e ao tempo de voo gasto no interior (basicamente inversamente proporcional à velocidade de nêutrons). As velocidades individuais de nêutrons são codificadas dentro de seu ângulo de precessão no final da primeira zona de precessão. (4) Perto da posição da amostra, o pi-flipper (posição 6) inverte a orientação da rotação em 180°, alterando o sinal do ângulo de precessão. (5) Os nêutrons interagem com as moléculas da amostra (posição 7) e ficam dispersos. (6) Os nêutrons dispersos entram e precess na segunda zona de pré-cessão (posição 8), mas tornam-se orientados para a inversão. (7) Outra nadadeira pi-half (posição 9) é usada para girar a orientação do giro do perpendicular à direção horizontal. Isso impedirá a precessão, traduzindo o ângulo de precessão φ em polarização proporcional à cos(φ). (8) O analisador (posição 10) seleciona os nêutrons com base em uma orientação. Se a interação com a amostra for elástica, a velocidade do nêutron não mudará. Os nêutrons passarão um tempo idêntico voando na primeira e segunda zonas de pré-cessão, e os ângulos acumulados de precessão estão totalmente recuperados. A polarização completa é restaurada no detector (posição 11) como um eco da polarização original (ou seja, spin-echo). (9) No entanto, em NSE, a dispersão é quase elástica, de modo que uma pequena troca de energia entre nêutrons e moléculas de amostras leva a diferentes velocidades de nêutrons após a dispersão pela amostra. Devido às diferentes velocidades, os nêutrons passarão um tempo adicional voando pela segunda zona de pré-cessão e não terão recuperado adequadamente seu ângulo de precessão. Uma polarização parcial é recuperada no detector, e a perda de polarização devido ao relaxamento do giro é proporcional à transformação cos-Fourier da função espectral S(Q, ω), a função de dispersão intermediária F(Q, t). (10) O parâmetro de tempo da função F(Q, t) é proporcional à força do campo magnético de pré-cessão. A varredura da perda de polarização em função da força do campo magnético rende, portanto, uma função de relaxamento que depende dos processos dinâmicos dentro da amostra.

Figura 1: Fotografia do espectrômetro NSE no SNS (SNS-NSE) e esquema de caminho de mosca de nêutrons com os componentes funcionais mais importantes. Da direita para a esquerda: 1 = fonte de nêutrons; 2 = sistema de obturadores-bender-bender-polarizador-secundário; 3 = guias de transporte de vigas; 4 = flipper pi/2 para o primeiro giro de 90°; 5 = primeira zona de pré-cessão; 6 = flipper pi para giro de 180°; 7 = área amostral e ambiente amostral (aqui, mostra-se o crio-forno); 8 = segunda zona de pré-cessão; 9 = pi/2 flipper para o segundo giro de 90°; 10 = analisador; 11 = detector. (Observe que as porções de 3, bem como 2 e 1 , estão situadas atrás da parede azul dentro da blindagem; os helicópteros são substituídos por um seletor de velocidade para NSE baseado em reator). Clique aqui para ver uma versão maior desta figura.
Protocolo
O presente trabalho caracteriza duas proteínas: uma proteína de anticorpos humanos regular IgG, e a MBP intrinsecamente desordenada. A forma liofilizada das proteínas foi obtida a partir de fontes comerciais (ver Tabela de Materiais).
1. Preparação da amostra de proteína
- Prepare um fosfato de sódio de 50 mM + tampão de NaCl de 0,1 M, pesando e dissolvendo os respectivos reagentes sólidos em água pesada (D2O) (ver Tabela de Materiais). Este é o solvente tampão deuterado para IgG.
- Ajuste o pH da solução tampão para 6.6.
- Prepare um fosfato de sódio de 20 mM + tampão de ureia de 6 M pesando e dissolvendo os respectivos componentes sólidos em água pesada (D2O). Este é o solvente tampão deuterado para MBP.
- Ajuste o pH da solução tampão para 4.7.
- Filtre os solventes tampão usando filtros de tamanho de poros de 0,2 μm (ver Tabela de Materiais).
- Pesar e dissolver os pós liofilizados de proteínas purificadas nos solventes deuterados para as respectivas proteínas (Etapas 1.1.-1.2.) em alta concentração proteica (~50 mg/mL).
- Carregue a solução proteica em cestos de diálise com membranas de diálise de 3,5 K MWCO, e dique contra o tampão filtrado por 24h a 10 °C para MBP e 25 °C para IgG (ver Tabela de Materiais) sacudindo ligeiramente os tubos para criar um gradiente de difusão.
- Diluir a solução proteica utilizando o tampão de diálise em uma série de concentrações: 1, 2, 5, 10 e 50 mg/mL.
- Determine as concentrações exatas usando um espectrômetro nanodrop (ver Tabela de Materiais).
2. Caracterização preliminar da amostra por dispersão dinâmica de luz (DLS)
- Carregue 80 μL de cada solução proteica da série de concentração preparada acima (Passo 1.6.) na célula descartável DLS (ver Tabela de Materiais) e determine os coeficientes de difusão, com média superior a 10 aquisições.
- Traçar os coeficientes de difusão translacional em função da concentração proteica e extrapolar para concentração zero.
- Carregue cada solução proteica da série de concentração nos tubos capilares de um viscometro (ver Tabela de Materiais) e meça a viscosidade dinâmica.
- Traçar as viscosidades dinâmicas medidas em função da concentração proteica e extrapolar para concentração zero.
NOTA: A extrapolação da difusão do DLS para concentração zero produz o valor da difusão translacional para uma única proteína. A extrapolação da viscosidade dinâmica para a concentração zero deve produzir o valor dinâmico de viscosidade medido experimentalmente para a solução tampão.
3. Coleta de dispersão de pequenos ângulos (nêutrons ou raio-x)
- Meça a dispersão de nêutrons de pequeno ângulo (SANS) e/ou dispersão de raios-X de pequeno ângulo (SAXS) (ver Tabela de Materiais) em quatro concentrações proteicas, de preferência 2 mg/mL, 5 mg/mL, 10 mg/mL e 50 mg/mL.
- Normalize os espectros SANS e SAXS por concentração de proteínas.
- Ajuste o fator de forma proteica P(Q) no espectro SANS e SAXS usando a otimização do conjunto31 e/ou o software SasView32 .
- Calcule o fator estrutura S(Q, c) dividindo o sinal SANS e SAXS com P(Q) para cada concentração.
NOTA: Os leitores interessados em medir e interpretar dados de dispersão de pequenos ângulos como suporte às medições nse são encorajados a consultar minuciosamente as referências 23,28,31,32,33.
4. Medição de NSE
- Configure-se para o experimento e monte a amostra seguindo os passos abaixo.
- Selecione a espessura da célula para carregamento amostral com base na concentração da solução proteica, na temperatura necessária para a medição e na quantidade de solução disponível.
NOTA: O presente estudo utilizou recipientes de quartzo transparentes de 40 mm x 30 mm x 4 mm. - Limpe a célula repetidamente, alternando entre detergente de prato livre de fosfato (ver Tabela de Materiais), água deionizada e 70% etanol.
- Seque a célula no forno de convecção; não exceda 80 °C para as células de quartzo.
- Carregue 4 mL de solução proteica na célula e feche com tampas. Use filme de cera ou qualquer selante (ver Tabela de Materiais) para selar as células da amostra.
NOTA: No presente estudo, foram utilizados 4,8 mL de solução a ~50 mg/mL para obter intensidade de dispersão suficiente. - Carregue 4 mL de tampão de diálise em um recipiente idêntico à amostra e vedação de proteínas.
- Transporte amostras para a linha de feixe, feche o obturador e entre na área da caverna do gabinete do espectrômetro34.
- Monte a célula amostra no suporte da amostra de alumínio apertando os parafusos e as placas de retenção (Figura 2, painel esquerdo).
NOTA: Pode-se montar duas células amostrais ao mesmo tempo, dado que o mesmo protocolo de medição é necessário para todas as amostras. - Monte a amostra de grafite e/ou Al2O3 amostra de pó carregada em um recipiente idêntico à amostra de proteína. Estes são padrões fornecidos pelo suporte de linha de vigas SNS-NSE.
- Coloque o suporte da amostra deslizando-o suavemente na lata do sistema de força de temperatura (TFS, consulte Tabela de Materiais).
NOTA: O TFS é o ambiente amostral mais utilizado no SNS-NSE e bombeia ar seco no recipiente de amostra para atingir a temperatura desejada (Figura 2, painéis médios e direito). - Feche a tampa TFS e defina a temperatura ao valor desejado acessando a tela interativa do TFS.
- Monte a câmera de nêutrons (ver Tabela de Materiais) para o alinhamento das amostras ao feixe.
- Varrer o compartimento do instrumento, evacuar, fechar as portas e abrir o obturador.
NOTA: As células amostrais são fornecidas pelo suporte de feixe SNS-NSE. Para células de amostra disponíveis e a biblioteca de ambientes de amostra, consulte a página webde feixe SNS-NSE 7,35.
- Selecione a espessura da célula para carregamento amostral com base na concentração da solução proteica, na temperatura necessária para a medição e na quantidade de solução disponível.
- Colete os dados nse seguindo as etapas abaixo.
- Alinhe a amostra no feixe de nêutrons usando a câmera de nêutrons e as quatro aberturas de amostra independentes.
- Abra o software de coleta de dados SNS-NSE36 e colete estatísticas de amostra executando varreduras de difração para os ângulos de dispersão desejados e comprimento de onda.
- Configure os parâmetros de medição com base nas estatísticas coletadas para cada amostra, editando as macros de medição fornecidas pelo Cientista de Instrumentos Auxiliares.
- Comece a digitalizar digitando o nome do protocolo no prompter de comando e adquira ecos para a amostra.
- Comece a digitalizar e adquira ecos também para a referência elástica e o solvente tamponado. Realize a operação do obturador intermitente para a troca da amostra.

Figura 2: Sistema de medição NSE. Painel esquerdo: amostras de solução proteica em recipiente de quartzo montado com parafusos e placas no suporte de amostra de alumínio (Al). O porta-amostras Al oferece a possibilidade de montar duas amostras simultaneamente dentro do ambiente amostral. Painel médio: A amostra do sistema de força de temperatura (TFS) pode ser montada no estágio da amostra a janela do feixe de nêutrons está à direita, enquanto a câmera de nêutrons usada para alinhamento é visível no lado esquerdo. Painel direito: colocar o suporte de amostra com duas amostras na amostra pode funcionar com janelas de silicone (Si). Clique aqui para ver uma versão maior desta figura.
5. Redução de dados da NSE
NOTA: O SNS-NSE está equipado com um software dedicado chamado DrSpine (redução de dados para spin-echo)37,38 disponível no OrNL Neutron Sciences Remote Analysis Cluster, um Guia de Usuário Rápido e suporte de ajuda incorporado.
- Faça login no Cluster de Análise Remota da Neutron Sciences (ver Tabela de Materiais) com as credenciais do usuário ORNL e pressione o botão Sessão de Lançamento .
- Configure o software de redução de dados seguindo as etapas abaixo.
- No diretório do usuário, abra uma janela e tipo de terminal: fonte/SNS/software/nse/etc/setup_nse.sh.
- Em seguida, digite: drspine_create_env.sh.
- Crie uma pasta para a redução de dados no diretório inicial e copie os scripts e macros fornecidos do diretório compartilhado.
- Editar, renomear e salvar a macro de redução fornecida em conformidade.
- Digite o drspine no prompter de comando e pressione enter para iniciar o ambiente de redução de software.
- Digite "o nome da macro de redução" editado no Passo 5.4. no prompter de comando dentro do ambiente de software e pressione Enter.
6. Montagem de dados NSE
- Edite o script python "stapler-drspine.py", fornecido pelo Assistente Cientista de Instrumentos, com os nomes dos dados de arquivo reduzidos.
NOTA: O script python está livremente disponível para os usuários do instrumento. - Edite a função para caber na biblioteca fornecida.
- Digite o nome do script editado "stapler-drspine.py" no prompter de comando e pressione Enter para ler, encaixar e plotar dados NSE reduzidos.
NOTA: O Instrument Scientist fornecerá um modelo para a macro de redução e o script python "stapler-drspine.py" que pode ler e encaixar dados reduzidos da NSE. Os dados nse reduzidos finais estão no formato ASCII e podem ser lidos por vários softwares preferidos.
Resultados
A proteína IgG do soro humano e das proteínas MBP bovinas foram reconstituídas em altas concentrações (~50 mg/mL) em buffers d2base O. Como as proteínas foram dissolvidas em altas concentrações, as soluções obtidas foram soluções de proteínas lotadas. A dinâmica investigada utilizando NSE sofre do ambiente lotado em que as proteínas residem (interações de fatores estruturais e efeitos hidrodinâmicos)5,28,39
Discussão
A espectroscopia NSE oferece uma visão única e detalhada da dinâmica das proteínas, que outras técnicas espectroscópicas não podem produzir. Medições em uma escala de tempo estendida fornecem observações da difusão translacional e rotacional das proteínas, conforme apresentado aqui. A dinâmica segmentar e outras oscilações internas revelam-se como uma forte decadência da função de dispersão coerente S(Q, t) em uma escala de tempo curto e estão bem separados dos processos gerais de relaxament...
Divulgações
O autor declara que não há interesses financeiros concorrentes e nenhum conflito de interesses. O conteúdo do manuscrito é baseado na palestra que o autor apresentou na HANDS-Neutron Scattering Applications in Structural Biology School entre 2019-2021.
Agradecimentos
Esta pesquisa utilizou recursos nos laboratórios de Neutron Da Spallation (BL-15, BL-6, Biologia e Química), um DoE Office of the Science User Facility operado pelo Laboratório Nacional de Oak Ridge. Esta pesquisa também utilizou recursos no reator MLZ-FRM2 Garching (KWS-2, Phoenix-J-NSE) e no JCNS1 em Forschungszentrum Jülich GmbH, Alemanha. O autor reconhece o Dr. Ralf Biehl e o Dr. Andreas Stadler por sua ajuda na modelagem e sua contribuição para a pesquisa de proteínas IgG e MBP, Dr. Piotr A. Żołnierczuk para suporte à redução de dados NSE, Dr. Changwoo Do para apoio com medições sans, e Rhonda Moody e Dr. Kevin Weiss para suporte labry bioquímico SNS.
Materiais
| Name | Company | Catalog Number | Comments |
| Bovine MBP protein solution | Sigma-Aldrich | M1891 | lyophilized powder reconstituted in D2O |
| D2O - heavy water | Sigma-Aldrich | Product No. 151882 | liquid |
| Dionized water | in house | - | for washing / cleanning cells |
| DLS instrument | Zetasizer Nano ZS, FZ-Jülich | - | dynamic light scattering instrument |
| Elastic scattering standards | SNS-NSE, ORNL | - | Al2O3 and Graphite powders |
| Ethanol | Sigma-Aldrich | 65350-M | 70% ethanol for cleaning cells |
| IgG protein solution | Sigma-Aldrich | I4506 | lyophilized powder reconstituted in D2O |
| KWS-2 instrument | JCNS outstation at the MLZ, Garching, Germany | - | small angle neutron instrument |
| Liquinox dish detergent | Alconox | - | Phosphate-free liquid lab glassware cleaner |
| Na2HPO4·7H2O | Sigma-Aldrich | Product No.S9390 | disodium phosphate heptahydrate salt |
| NaCl | Sigma-Aldrich | Product No.S9888 | sodium chloride salt |
| NaH2PO4·H2O | Sigma-Aldrich | Product No. S9638 | monosodium phosphate monohydrate salt |
| Nanodrop spectrophotometer | Thermo Scientific | Catalog number: ND-2000 | NanoDrop 2000/2000c Spectrophotometer |
| Neutron alignment camera | NeutronOptics, Grenoble | NOG210222 | 100 x 100 mm camera with Sony IMX249 CMOS sensor |
| Parafilm M - wax parafilm | Bemis | Parafilm M - 5259-04LC PM996 | all-purpose laboratory film in cardboard dispenser |
| Phoenix-J-NSE Spectrometer | JCNS outstation at the MLZ, Garching, Germany | - | neutron spectrometer |
| SasView | https://www.sasview.org/ | ||
| SAXSpace, Anton Paar instrument | FZ-Jülich | - | small angle x-ray instrument |
| Slide-A-Lyzer dialysis membranes | Thermo Scientific | 88400-88405 | Slide-A-Lyzer mini dialysis devices tubes of 3.5 K MWCO |
| SNS Remote Analysis Cluster | Neutron Science Remote Analysis (sns.gov) | https://analysis.sns.gov | |
| SNS-NSE spectrometer | ORNL, Oak Ridge, TN, USA | - | neutron spectrometer |
| Sterile syringe filters | VWR | N.A. PN:28145-501 | 0.2 µm pore size filters |
| Temperature Forcing System (TFS) | SP Scientific | Part Number 100004055 | sample environment equipment |
| Urea -d4 | Sigma-Aldrich | Product No. 176087 | deuterated Urea salt |
| Viscometer | FZ-Jülich | - | falling ball viscometer |
Referências
- Fitter, J., Gutberlet, T., Katsaras, J. . Neutron Scattering in Biology: Techniques and Applications. , (2006).
- Stadler, A., Monkenbusch, M., Biehl, R., Richter, D., Ollivier, J. Neutron spin-echo and TOF reveals protein dynamics in solution. Journal of the Physical Society of Japan. 82, (2013).
- Inoue, R. Large domain fluctuations on 50-ns timescale enable catalytic activity in phosphoglycerate kinase. Biophysical Journal. 99 (7), 2309-2317 (2010).
- Callaway, D. J. E., et al. Controllable activation of nanoscale dynamics in a disordered protein alters binding kinetics. Journal of Molecular Biology. 429 (7), 987-998 (2017).
- Stingaciu, L. R., Biehl, R., Changwoo, D., Richter, D., Stadler, A. M. Reduced internal friction by osmolyte interaction in intrinsically disordered myelin basic protein. Journal of Physical Chemistry Letters. 11 (1), 292-296 (2020).
- Monkenbusch, M., Richter, D. High resolution neutron spectroscopy-a tool for the investigation of dynamics of polymers and soft matter. Comptes Rendus Physique. , (2007).
- Richter, D., Monkenbusch, M., Schwahn, D. Neutron Scattering. Polymer Science: A Comprehensive Reference, 10 Volume Set. , (2012).
- Wilson, C. C. . Single Crystal Neutron Diffraction From Molecular Materials. , (2000).
- Marshall, W. . Theory of thermal neutron scattering. , (1971).
- . Roger Pynn Introduction & Neutron Scattering "Theory" Available from: https://neutrons.ornl.gov/sites/default/files/intro_to_neutron_scattering.pdf (2004)
- Richter, D. Neutron scattering in polymer physics. Physica B: Condensed Matter. 276-278, 22-29 (2000).
- Harroun, T. A., Wignall, G. D., Katsaras, J. Neutron scattering for biology. Neutron Scattering in Biology. , (2006).
- . SNS Available from: https://neutrons.ornl.gov/sna (2020)
- . HFIR Available from: https://neutrons.ornl.gov/hfir (2020)
- . CNCS Available from: https://neutrons.ornl.gov/cncs (2020)
- Perticaroli, S., et al. Description of hydration water in protein (green fluorescent protein) solution. Journal of the American Chemical Society. 139 (3), 1098-1105 (2017).
- Perticaroli, S., Nickels, J. D., Ehlers, G., Sokolov, A. P. Rigidity, secondary structure, and the universality of the boson peak in proteins. Biophysical Journal. 106 (12), 2667-2674 (2014).
- Miao, Y., et al. Coupled flexibility change in cytochrome p450cam substrate binding determined by neutron scattering, NMR, and molecular dynamics simulation. Biophysical Journal. 103 (10), 2167-2176 (2012).
- Mamontov, E., Zamponi, M., Hammons, S., Keener, W. S., Hagen, M., Herwig, K. W. BASIS: A new backscattering spectrometer at the SNS. Neutron News. 19 (3), 22-24 (2008).
- Mamontov, E. Microscopic diffusion processes measured in living planarians. Scientific Reports. 9, 8708 (2018).
- Alpert, Y., Cser, L., Faragó, B., Franěk, F., Mezei, F., Ostanevich, Y. M. Segmental flexibility in pig immunoglobulin G studied by neutron spin-echo technique. Biopolymers. 24 (9), 1769-1784 (1985).
- Bu, Z., Biehl, R., Monkenbusch, M., Richter, D., Callaway, D. J. E. Coupled protein domain motion in Taq polymerase revealed by neutron spin-echo spectroscopy. Proceedings of the National Academy of Sciences of the United States of America. 102 (49), 17646-17651 (2005).
- Biehl, R., et al. Direct observation of correlated interdomain motion in alcohol dehydrogenase. Physical Review Letters. 101, 138102 (2008).
- Farago, B., Li, J., Cornilescu, G., Callaway, D. J. E., Bu, Z. Activation of nanoscale allosteric protein domain motion revealed by neutron spin echo spectroscopy. Biophysical Journal. 99 (10), 3473-3482 (2010).
- Bu, Z., Callaway, D. J. E. Dynamic propagation of long-range allosteric signals by nanoscale protein domain motion revealed by neutron spin echo spectroscopy. Biophysical Journal. 100 (3), 223 (2011).
- Hong, L., et al. Structure and dynamics of a compact state of a multidomain protein, the mercuric ion reductase. Biophysical Journal. 107 (2), 393-400 (2014).
- Longeville, S., Stingaciu, L. -. R. Hemoglobin diffusion and the dynamics of oxygen capture by red blood cells. Scientific Reports. 7, 10448 (2017).
- Stingaciu, L. R., Ivanova, O., Ohl, M., Biehl, R., Richter, D. Fast antibody fragment motion: Flexible linkers act as entropic spring. Scientific Reports. 6, 22148 (2016).
- Hahn, E. L. Nuclear induction due to free larmor precession. Physical Review. 77, 297 (1950).
- Hahn, E. L. Spin echoes. Physical Review. 80, 580 (1950).
- Svergun, D. I., Koch, M. H. J. Small-angle scattering studies of biological macromolecules in solution. Reports on Progress in Physics. 66 (10), 1735-1782 (2003).
- . Sasview Available from: https://www.sasview.org (2020)
- Zhao, J. K., Gao, C. Y., Liu, D. The extended Q-range small-angle neutron scattering diffractometer at the SNS. Journal of Applied Crystallography. 43, 1068-1077 (2010).
- Ohl, M., et al. The high-resolution neutron spin-echo spectrometer for the SNS with τ ≥ 1 µs. Physica B: Condensed Matter. 350, 147-150 (2004).
- . SNS-NSE web page Available from: https://neutrons.ornl.gov/nse (2022)
- Ohl, M., et al. The spin-echo spectrometer at the Spallation Neutron Source (SNS). Nuclear Instruments and Methods in Physics Research, Section A: Accelerators, Spectrometers, Detectors and Associated Equipment. 696, 85-99 (2012).
- Zolnierczuk, P. A., Holderer, O., Pasini, S., Kozielewski, T., Stingaciu, L. R., Monkenbusch, M. Efficient data extraction from neutron time-of-flight spin-echo raw data. Journal of Applied Crystallography. 52 (5), 1022-1034 (2019).
- . Dr Spine hub Available from: https://jugit.fz-juelich.de/nse/drspine (2022)
- Stadler, A. M., et al. Internal nanosecond dynamics in the intrinsically disordered myelin basic protein. Journal of the American Chemical Society. 136 (19), (2014).
- . FZJ Available from: https://www.fz-juelich.de/portal/EN/AboutUs (2022)
- . KWS2 Available from: https://miz-garching.de/kws-2 (2022)
- Moorhouse, M., Barry, P. The protein databank. Bioinformatics Biocomputing and Perl. , (2005).
- Tria, G., Mertens, H. D. T., Kachala, M., Svergun, D. I. Advanced ensemble modelling of flexible macromolecules using X-ray solution scattering. IUCrJ. 2 (2), 207-217 (2015).
- Holderer, O., Monkenbusch, M., Schätzler, R., Kleines, H., Westerhausen, W., Richter, D. The JCNS neutron spin-echo spectrometer J-NSE at the FRM II. Measurement Science and Technology. 90 (4), 043107 (2008).
- Biehl, R., Monkenbusch, M., Richter, D. Exploring internal protein dynamics by neutron spin echo spectroscopy. Soft Matter. 7 (4), 1299-1307 (2011).
- Stingaciu, L. R., Ivanova, O., Ohl, M., Biehl, R., Richter, D. Fast antibody fragment motion: Flexible linkers act as entropic spring. Scientific Reports. 6, 22148 (2016).
- Stadler, A. M., et al. Internal nanosecond dynamics in the intrinsically disordered myelin basic protein. Journal of the American Chemical Society. 136 (19), 6987-6994 (2014).
- Biehl, R., Richter, D. Slow internal protein dynamics in solution. Journal of Physics: Condensed Matter. 26 (50), 503103 (2014).
- Hinsen, K. The molecular modeling toolkit: A new approach to molecular simulations. Journal of Computational Chemistry. 21 (2), 79-85 (2000).
- Uhlenbeck, G. E., Ornstein, L. S. On the theory of the Brownian motion. Physical Review. 36 (5), 823 (1930).
- Wang, M. C., Uhlenbeck, G. E. On the theory of the Brownian motion II. Reviews of Modern Physics. 17, 323 (1945).
- Callaway, D. J. E., Bu, Z. Nanoscale protein domain motion and long-range allostery in signaling proteins-a view from neutron spin echo spectroscopy. Biophysical Reviews. 7 (2), 165-174 (2015).
- Liu, Y. Intermediate scattering function for macromolecules in solution probed by neutron spin echo. Physical Review E. 95, 020501 (2017).
- Liu, Y. Short-time dynamics of proteins in solutions studied by neutron spin echo. Current Opinion in Colloid & Interface Science. 42, 147-156 (2019).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados