A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
استكشاف التمثيل الغذائي ولزوجة الخلايا السرطانية باستخدام الفحص المجهري للتصوير الفلوري مدى الحياة
In This Article
Summary
هنا ، نوضح استخدام الفحص المجهري للتصوير مدى الحياة الفلورية (FLIM) لتصوير التمثيل الغذائي الخلوي ولزوجة غشاء البلازما بالتتابع في ثقافة الخلايا السرطانية الحية. يتم إجراء تقييمات التمثيل الغذائي عن طريق الكشف عن التألق الداخلي. يتم قياس اللزوجة باستخدام دوار جزيئي فلورسنت.
Abstract
اللزوجة هي خاصية فيزيائية مهمة للغشاء البيولوجي ، لأنها واحدة من المعلمات الرئيسية لتنظيم الحالة المورفولوجية والفسيولوجية للخلايا الحية. من المعروف أن أغشية البلازما للخلايا السرطانية لها تغييرات كبيرة في تكوينها وهيكلها وخصائصها الوظيفية. إلى جانب التمثيل الغذائي غير المنظم للجلوكوز والدهون ، تساعد خصائص الغشاء المحددة هذه الخلايا السرطانية على التكيف مع البيئة المكروية المعادية وتطوير مقاومة للعلاجات الدوائية. هنا ، نوضح استخدام الفحص المجهري للتصوير مدى الحياة الفلورية (FLIM) لتصوير التمثيل الغذائي الخلوي ولزوجة غشاء البلازما بالتتابع في ثقافة الخلايا السرطانية الحية. يتم إجراء تقييمات التمثيل الغذائي عن طريق الكشف عن تألق العوامل المساعدة الأيضية الداخلية ، مثل انخفاض نيكوتيناميد الأدينين ثنائي النوكليوتيد NAD (P) H والفلافينات المؤكسدة. يتم قياس اللزوجة باستخدام دوار جزيئي فلوري ، وهو صبغة اصطناعية حساسة للزوجة ، مع اعتماد قوي على عمر التألق على لزوجة البيئة المباشرة. تمكننا هذه التقنيات مجتمعة من فهم الروابط بين حالة الغشاء والمظهر الأيضي للخلايا السرطانية بشكل أفضل وتصور التغييرات التي يسببها العلاج الكيميائي.
Introduction
يصاحب التحول الخبيث للخلايا تغيرات متعددة في حالتها المورفولوجية والفسيولوجية. يتطلب النمو السريع وغير المنضبط للخلايا السرطانية إعادة تنظيم أساسي للمسارات الكيميائية الحيوية المسؤولة عن إنتاج الطاقة والتخليق الحيوي. السمات المميزة لعملية التمثيل الغذائي للسرطان هي تعزيز معدل تحلل السكر ، حتى تحت تركيزات الأكسجين الطبيعية (تأثير واربورغ) ، واستخدام الأحماض الأمينية والأحماض الدهنية واللاكتات كوقود بديل ، وإنتاج عالي من أنواع الأكسدة التفاعلية في وجود مستويات عالية من مضادات الأكسدة ، وزيادة التخليق الحيوي للأحماض الدهنية1،2. من المقدر الآن أن التمثيل الغذائي للخلايا السرطانية مرن للغاية ، مما يسمح لها بالتكيف مع البيئة غير المواتية وغير المتجانسة ويوفر ميزة إضافية للبقاءعلى قيد الحياة 3.
يدعم التمثيل الغذائي المتغير التنظيم المحدد وتكوين أغشية الخلايا السرطانية. يختلف المظهر الدهني لغشاء البلازما في الخلايا السرطانية كميا عن الخلايا غير السرطانية. التغييرات الرئيسية في القبة الدهنية هي زيادة مستوى الدهون الفوسفورية بما في ذلك الفوسفاتيديلينوسيتول, فوسفاتيديلسيرين, فوسفاتيديل إيلانولامين وفوسفاتيديل كولين, انخفاض مستوى السفينجوميلين, زيادة كمية الكوليسترول, وانخفاض درجة عدم تشبع الأحماض الدهنية, على سبيل المثال لا الحصر 4,5,6. لذلك ، فإن الخصائص الفيزيائية للغشاء ، مثل لزوجة الغشاء ، معكوس السيولة ، تتغير حتما. نظرا لأن اللزوجة تحدد نفاذية الأغشية البيولوجية وتتحكم في نشاط البروتينات المرتبطة بالغشاء (الإنزيمات والناقلات والمستقبلات) ، فإن تنظيمها الاستتبابي أمر حيوي لعمل الخلية. في الوقت نفسه ، يعد تعديل اللزوجة من خلال تعديل ملف تعريف الدهون الغشائية أمرا مهما لهجرة / غزو الخلايا والبقاء على قيد الحياة عند التغييرات الشرطية.
ظهر الفحص المجهري للتصوير مدى الحياة الفلوري (FLIM) كنهج قوي للتقييم غير الجراحي للمعلمات المتعددة في الخلايا الحية ، باستخدام التألق الداخلي أو المجسات الخارجية7. يتم تحقيق FLIM بشكل شائع على مجهر المسح الضوئي بالليزر متعدد الفوتون ، والذي يوفر دقة خلوية (فرعية). نظرا لكونها مجهزة بوحدة عد الفوتون أحادي الارتباط بالوقت (TCSPC) ، فإنها تتيح قياسات مضان محلولة بمرور الوقت بدقة عالية8.
يعتمد فحص التمثيل الغذائي الخلوي بواسطة FLIM على قياس التألق للعوامل المساعدة الذاتية لنازعة الهيدروجين ، وانخفاض نيكوتيناميد الأدينين ثنائي النوكليوتيد (الفوسفات) NAD (P) H والفلافينات المؤكسدة - فلافين الأدينين ثنائي النوكليوتيد FAD والفلافين أحادي النوكليوتيد FMN ، التي تعمل كناقلات إلكترون في عدد من التفاعلات الكيميائية الحيوية7،9،10. التألق المكتشف ل NAD (P) H هو من NADH وشكله الفسفوري ، NADPH ، حيث أنهما متطابقان طيفيا تقريبا. عادة ما يتناسب اضمحلال التألق ل NAD (P) H والفلافينات مع وظيفة ثنائية الأسية. في حالة NAD (P) H ، يعزى المكون الأول (~ 0.3-0.5 نانوثانية ، ~ 70٪ -80٪) إلى حالته الحرة المرتبطة بتحلل السكر ، والمكون الثاني (~ 1.2-2.5 نانوثانية ، ~ 20٪ -30٪) إلى حالته المرتبطة بالبروتين ، المرتبطة بتنفس الميتوكوندريا. في حالة الفلافينات ، يمكن تعيين المكون القصير (~ 0.3-0.4 نانوثانية ، ~ 75٪ -85٪) للحالة المروية ل FAD والمكون الطويل (~ 2.5-2.8 نانوثانية ، ~ 15٪ -25٪) إلى FAD و FMN و riboflavin غير المروي. تؤدي التغيرات في المستويات النسبية لتحلل السكر ، وتحلل الغلوتامينات ، والفسفرة المؤكسدة ، وتخليق الأحماض الدهنية إلى تغيرات في الكسور قصيرة والطويلة العمر للعوامل المساعدة. بالإضافة إلى ذلك ، تعكس نسبة شدة التألق لهذه الفلوروفورات (نسبة الأكسدة والاختزال) حالة الأكسدة والاختزال الخلوية وتستخدم أيضا كمؤشر أيضي. على الرغم من أن نسبة الأكسدة والاختزال تقدم مقياسا أبسط ، مقارنة بعمر التألق ، من حيث الحصول على البيانات ، فإن FLIM مفيد لتقدير NAD (P) H و FAD ، لأن عمر التألق هو سمة جوهرية للفلوروفور ولا يتأثر تقريبا بعوامل مثل قوة الإثارة ، والتبييض الضوئي ، والتركيز ، وتشتت الضوء والامتصاص ، خاصة في الأنسجة ، على عكس شدة الانبعاث.
تعتمد إحدى الطرق الملائمة لرسم خريطة اللزوجة في الخلايا والأنسجة الحية على المستوى المجهري على استخدام الدوارات الجزيئية الفلورية ، والأصباغ الاصطناعية الصغيرة الحساسة للزوجة ، حيث تعتمد معلمات التألق بشدة على اللزوجة المحلية11،12. في الوسط اللزج ، يزداد عمر التألق للدوار بسبب تباطؤ الالتواء أو الدوران داخل الجزيئات. من بين الدوارات الجزيئية ، تعتبر مشتقات ثنائي بيروثيرمين البورون (BODIPY) مناسبة تماما لاستشعار اللزوجة في الأنظمة البيولوجية حيث أن لديها نطاقا ديناميكيا جيدا من عمر التألق في النطاق الفسيولوجي للزوجة ، واستقلال درجة الحرارة ، واضمحلال التألق الأحادي الأسي الذي يسمح بتفسير البيانات مباشرة ، وقابلية الذوبان في الماء الكافية والسمية الخلويةالمنخفضة 13،14. تم إثبات التقييمات الكمية لللزوجة الدقيقة باستخدام الدوارات القائمة على BODIPY و FLIM سابقا على الخلايا السرطانية في المختبر ، وكرويات الورم متعددة الخلايا وورم الفأر في الجسم الحي15،16.
هنا ، نقدم وصفا مفصلا لمنهجيات الفحص المتسلسلة لدراسة التمثيل الغذائي الخلوي ولزوجة غشاء البلازما في الخلايا السرطانية في المختبر بواسطة FLIM. لتجنب تلوث التألق الداخلي الضعيف نسبيا مع مضان الدوار القائم على BODYPY ، يتم إجراء تصوير لنفس الطبقة من الخلايا بالتتابع مع تألق NAD (P) H و FAD المصور أولا. يتم قياس عمر التألق للعوامل المساعدة في السيتوبلازم ، ويتم قياس عمر التألق للدوار في أغشية البلازما للخلايا عن طريق الاختيار اليدوي للمناطق المقابلة كمناطق ذات أهمية. تم تطبيق البروتوكول لربط حالة التمثيل الغذائي واللزوجة لخطوط الخلايا السرطانية المختلفة ولتقييم التغييرات بعد العلاج الكيميائي.
لا يختلف بروتوكول تحضير عينة FLIM عن بروتوكول الفحص المجهري الفلوري متحد البؤر. بمجرد الحصول على البيانات ، تتمثل المهمة الرئيسية في استخراج عمر التألق من البيانات الأولية. يتم إثبات أداء البروتوكول باستخدام HCT116 (سرطان القولون والمستقيم البشري) ، CT26 (سرطان القولون الفئراني) ، HeLa (سرطان عنق الرحم البشري) ، و huFB (الخلايا الليفية لجلد الإنسان).
Protocol
1. وصف الحد الأدنى من الإعداد لأداء FLIM
- لإجراء هذه التجربة ، تأكد من توفر الإعداد المطلوب: مجهر متحد البؤر مقلوب ، ليزر نبضي ، عادة ps أو fs ، مع إشارة التزامن ، كاشف عد الفوتون السريع (استجابة زمنية 150 ps) وإلكترونيات عد الفوتون ، منافذ الإخراج والإدخال المتاحة للكاشف والليزر ، على التوالي ، على المجهر ، نبضات ساعة المسح من وحدة التحكم في المسح المجهري ، رأس المسح الضوئي للمجهر مع مجمعات شعاع الليزر ومقسمات الشعاع ثنائية اللون الرئيسية المناسبة للطول الموجي لليزر المستخدم في FLIM.
- إذا تم استخدام إثارة ثنائية الفوتون ل FLIM ، فتأكد من أن المجهر يحتوي على منفذ NDD.
- بالنسبة لدراسات خلايا الثدييات ، خاصة للتجارب طويلة المدى ، تأكد من الحفاظ على حاضنة ثاني أكسيد الكربون2 في درجة الحرارة المطلوبة.
ملاحظة: للاطلاع على النظام المستخدم في هذه التجربة، راجع جدول المواد.
2. تحضير الخلايا للفحص المجهري
- قم بزراعة الخلايا بشكل روتيني في حاضنة عند 37 درجة مئوية مع 5٪ ثاني أكسيد الكربون2 وأجواء رطبة.
- للفحص المجهري ، قم بإعداد معلق الخلية في وسط ثقافة كامل بتركيز 1 × 106 خلايا / مل.
ملاحظة: تعتمد تركيزات الخلية وظروف الوسائط على الخلية. يجب تكييف عدد الخلايا المستخدمة في البذر ووقت الحضانة للحصول على التقاء 70٪ -80٪ في الطبق المجهري. - قم بزرع الخلايا على أطباق زراعة الخلايا ذات القاع الزجاجي مقاس 35 مم (1 × 105 خلايا في 100 ميكرولتر لكل طبق) باستخدام ماصة أوتوماتيكية سعة 200 ميكرولتر.
ملاحظة: استخدم أطباق ذات قاع زجاجي شبكي للخلايا التي تزرع لمراقبة الخلايا في نفس مجالات الرؤية المجهرية في الديناميكيات.- أثناء البذر اليدوي ، تأكد من أن طرف الماصة لا يخدش قاع الطبق أو جوانبه لتجنب إتلاف القاع.
- ضع الطبق في حاضنة ثاني أكسيد الكربون2 (37 درجة مئوية ، 5٪ ثاني أكسيد الكربون2 ، جو رطب) واحتضن الخلايا لمدة 24 ساعة.
- بعد 24 ساعة ، أخرج الطبق من الحاضنة وتحقق من مورفولوجيا الخلايا والتقائها تحت المجهر الضوئي. إذا لم تصل الخلايا إلى حوالي 80٪ التقاء ، فقم باحتضانها لمدة 24 ساعة إضافية.
- قم بإزالة الوسط القديم برفق من الطبق باستخدام ماصة أوتوماتيكية سعة 1,000 ميكرولتر وأضف 2 مل من وسط DMEM بدون أحمر الفينول (على سبيل المثال، DMEM Life أو FluoroBrite).
ملاحظة: يمكن استخدام وسائط ثقافية مختلفة للتصوير. تجنب الفينول الأحمر في الوسط عند استخدام الخلايا للفحص المجهري. - ضع الطبق في الحاضنة لمدة 60-120 دقيقة للسماح بتكيف الخلايا.
3. FLIM من العوامل المساعدة الأيضية
- ضع الطبق ذو القاع الزجاجي مع الخلايا (من الخطوة 2.7) على مرحلة المجهر.
- انقر فوق علامة التبويب تحديد الموقع في برنامج مجهر المسح الضوئي بالليزر (على سبيل المثال ، ZEN - ZEISS Efficient Navigation) ، ثم انقر فوق ضوء الإرسال (TL) لتشغيل الضوء.
- ابحث عن المستوى البؤري للعينة من خلال الرؤية من خلال العدسة على مستوى الشريحة المركزية للخلايا ، حيث تشغل الخلايا المربع إلى أقصى حد (عند التكبير 40x).
- انقر فوق الزر "إيقاف " لإطفاء الضوء.
- افتح علامة التبويب اكتساب . للحصول على صور شدة الإرسال والتألق الذاتي ل NAD (P) H الداخلي ، أدخل الإعدادات التالية: الطول الموجي للإثارة: وضع الفوتون 750 نانومتر ، نطاق التسجيل: 450-490 نانومتر ، طاقة الليزر: 5٪ (~ 6 ميجاوات) ، حجم الصورة: 1024 × 1024 بكسل.
ملاحظة: يعتمد اختيار الطول الموجي للإثارة ومدى التسجيل على الخصائص الطيفية (الإثارة القصوى والانبعاث الأقصى) ل NAD(P)H17.- استخدم عدسة موضوعية غمر الزيت С Plan-Apochromat 40x / 1.3 NA للحصول على الصورة.
- اضغط على الزر Snap واحفظ الصورة بتنسيق ZEN.
- للحصول على صور شدة الإرسال والتألق الذاتي ل FAD ، قم بتغيير الطول الموجي للإثارة إلى 900 نانومتر. اضبط نطاق التسجيل: 500-550 نانومتر ، وطاقة الليزر: 9٪ (~ 6 ميجاوات) ، وحجم الصورة: 1024 × 1024 بكسل.
ملاحظة: يعتمد اختيار الطول الموجي للإثارة ومدى التسجيل على الخصائص الطيفية (الإثارة القصوى والانبعاث الأقصى) لFAD 18.- استخدم عدسة موضوعية غمر الزيت С Plan-Apochromat 40x / 1.3 NA للحصول على الصور.
- اضغط على الزر Snap واحفظ الصورة بتنسيق ZEN.
- بالنسبة ل NAD (P) H ، قم بتعيين المعلمات كما هو موضح في الخطوة 3.5 في برنامج مجهر المسح بالليزر. قم بتغيير حجم الصورة إلى 256 × 256 بكسل.
- أدخل المعلمات التالية في قائمة SPCM (وحدات عد الفوتون الفردي) برنامج تشغيل وحدة FLIM: وقت التجميع: 60 ثانية ؛ نطاق TAC: 5.00E-8 ؛ أدنى حد للعقود مقابل الفروقات: -29.41; دقة ADC: 256 ، حجم الصورة: 256 × 256 بكسل.
- امسح العينة ضوئيا لمدة 60 ثانية ، وتوقف عن المسح وحفظ صورة FLIM التي تم الحصول عليها من NAD (P) H.
- تحقق من بيانات FLIM التي تم الحصول عليها. لهذا ، افتح البيانات الأولية في برنامج الصورة ، وحدد بكسل في سيتوبلازم الخلية عن طريق وضع المؤشر فوقه وتحليل اضمحلال التألق في هذا البكسل. يجب أن تكون شدة البكسل ≥3,000 فوتون لكل منحنى اضمحلال عند التجميع 1.
ملاحظة: إذا كان عدد الفوتونات أقل من 3,000 ، فقم بزيادة قوة الليزر أو وقت جمع الصور ، مع التحكم في مورفولوجيا الخلايا ومعدل عد الفوتونات. عادة ، إذا تجاوز الانخفاض في معدل العد 10٪ من القيمة الأولية ، يحدث التبييض الضوئي. - بالنسبة إلى FAD ، قم بتعيين المعلمات كما هو موضح في الخطوة 3.7 في برنامج مجهر المسح بالليزر. قم بتغيير حجم الصورة إلى 256 × 256 بكسل.
- أدخل المعلمات التالية في قائمة SPCM (وحدات عد الفوتون الفردي) برنامج تشغيل وحدة FLIM: وقت التجميع: 60 ثانية ؛ نطاق TAC: 5.00E-8 ؛ أدنى حد للعقود مقابل الفروقات: -29.41; دقة ADC: 256 ، حجم الصورة: 256 × 256 بكسل.
- امسح العينة ضوئيا لمدة 60 ثانية. توقف عن المسح وحفظ صورة FLIM التي تم الحصول عليها من FAD.
ملاحظة: المعلمات المشار إليها في الخطوتين 3.10 و 3.15 خاصة بالإلكترونيات والكاشف المستخدم. - تحقق من البيانات التي تم الحصول عليها كما هو موضح في الخطوة 3.12.
- كرر الخطوات 3.5-3.16 لتسجيل صور FLIM من مجالات رؤية مختلفة.
4. تلطيخ الخلايا بالدوار الجزيئي الفلوري
ملاحظة: يتم تصوير الخلايا في محلول الدوار الجزيئي الفلوري دون غسلها في درجة حرارة الغرفة (~ 20 درجة مئوية) لإبطاء استيعاب الدوار. تعتمد لزوجة الغشاء على درجة الحرارة ، كما هو موضح في أعمالنا السابقة19،20. يجب إيقاف تشغيل مرحلة التحكم في درجة حرارة المجهر مسبقا ، أي قبل إضافة الدوار إلى الخلايا. بالنسبة لإعدادنا ، يستغرق تبريد المرحلة حوالي 10 دقائق.
- قم بإعداد محلول مخزون عام للدوار الجزيئي الفلوري BODIPY 2 (المخزون 1 ، 25.7 مليمتر).
- افتح BODIPY 2 في بيئة معقمة ووزن حوالي 2 مجم باستخدام ميزان دقيق. ضعه بعناية في أنبوب الطرد المركزي الدقيق.
- استخدم ماصة أوتوماتيكية سعة 20 ميكرولتر لإضافة 3 ميكرولتر من مذيب مناسب (على سبيل المثال، DMSO).
- بمجرد أن تذوب العينة تماما في DMSO، أضف 297 ميكرولتر من PBS المعقم واخلطها جيدا باستخدام ماصة أوتوماتيكية سعة 200 ميكرولتر.
ملاحظة: قم بتخزين محلول المرق في الثلاجة عند +4 درجة مئوية في عبوة داكنة. بمجرد إعادة تعليقه ، يمكن تخزينه في الثلاجة لعدة أشهر.
- قم بإعداد مخزون 2 (8.9 ملليمتر) بإضافة 25 ميكرولتر من محلول المخزون العام (المخزون 1) إلى أنبوب الطرد المركزي الدقيق ، متبوعا ب 48 ميكرولتر من PBS المعقم. امزج برفق باستخدام ماصة أوتوماتيكية سعة 200 ميكرولتر.
ملاحظة: استخدم المخزون 2 لتحضير مخزون التلوين النهائي ، والذي يتم تطبيقه على تلطيخ الخلايا نظرا لأن تركيز الميكرومولار مطلوب. - استبدل وسط الثقافة في الطبق برفق (من الخطوة 3.1) بمحلول هانك المثلج بدون Ca2 + / Mg2+ واحتضن الخلايا عند +4 درجة مئوية لمدة 3 دقائق.
ملاحظة: يؤدي استخدام محلول مثلج بارد وحضانة عند +4 درجة مئوية إلى إبطاء استيعاب الدوار الجزيئي ، ويستمر التلوين الموضعي للغشاء لمدة 20-30 دقيقة. - قم بإعداد محلول التلوين النهائي الذي يحتوي على 4.5 ميكرومتر من BODIPY 2 عن طريق إضافة 1 ميكرولتر من محلول هانك المثلج البارد أو PBS.
ملاحظة: يمكن زيادة تركيز BODIPY 2 في محلول التلوين النهائي إلى ~ 10 ميكرومتر دون أي تأثيرات سامة على الخلايا ، مما يؤدي إلى تلطيخ أكثر كفاءة وعدد أكبر من الفوتونات المجمعة. عند تركيزات أعلى ، قد يحدث التحميل الزائد لكاشف FLIM. - استنشق محلول هانك من طبق زراعة الخلايا واستبدله بمحلول مثلج 4.5 ميكرومتر من BODIPY 2. يتم تصوير الخلايا في محلول BODIPY 2 دون غسل.
5. FLIM من الدوار الجزيئي الفلوري في الخلايا
ملاحظة: قم دائما بإجراء FLIM للدوار الجزيئي الفلوري بعد FLIM للعوامل المساعدة الأيضية لأن طيف التألق ل BODIPY 2 يتداخل مع انبعاث العوامل المساعدة الداخلية NAD (Р) H و FAD12،17،18.
- انقل الطبق مع الخلايا الملطخة إلى مرحلة المجهر (~ 20 درجة مئوية) للتصوير.
- قم بتعيين المعلمات التالية لوضع الفوتون الواحد في برنامج مجهر المسح بالليزر: الإثارة عند الطول الموجي 488 نانومتر باستخدام ليزر أيون الأرجون ، قوة الليزر 1٪ -2٪ ، نطاق التسجيل 500-550 نانومتر الطول الموجي.
- استخدم عدسة موضوعية غمر الزيت С Plan-Apochromat 40x / 1.3 NA للحصول على الصور.
- اضغط على زر Live . ابدأ المسح الضوئي واستخدام تحديد المواقع XY و Z من خلال مرحلة آلية متكاملة ، واضبط التركيز واحصل على صور شدة الإرسال والفلورة للخلايا في نافذة معاينة. احفظ الصور التي تم الحصول عليها، إذا لزم الأمر.
- تحقق من صورة النقل والفلورة المتداخلة لمعرفة ما إذا كان مضان الدوار قادما من الموقع المتوقع (غشاء البلازما للخلية).
- أدخل المعلمات التالية في قائمة برنامج SPCM لوحدة FLIM: وقت التجميع: 60 ثانية. نطاق TAC: 5.00E-8 ؛ أدنى حد للعقود مقابل الفروقات: -29.41; دقة ADC: 256 ، حجم الصورة: 256 × 256 بكسل.
ملاحظة: اعتمادا على تكوين النظام وأجهزة الكشف المستخدمة في FLIM، قد تختلف معلمات الحصول على الصور. - اضبط ليزر Ti: Sapphire للمجهر على طول موجي 850 نانومتر وطاقة الليزر إلى 1٪ -2٪ ل FLIM ثنائي الفوتون.
- حدد علامة التبويب مستمر في برنامج مجهر المسح الضوئي بالليزر، ثم اضغط على ابدأ في برنامج SPCM. امسح العينة ضوئيا لمدة 60 ثانية ، وتوقف عن المسح الضوئي ، واحفظ صورة FLIM التي تم الحصول عليها.
- تحقق من بيانات FLIM التي تم الحصول عليها. لهذا ، قم بتحميل البيانات الأولية في برنامج SPCImage لتحليل بيانات FLIM ، وحدد بكسل في غشاء الخلية عن طريق وضع المؤشر فوقه وتحليل اضمحلال التألق في هذا البكسل. يجب أن تكون شدة البكسل ≥5,000 لكل تضويق (ربما بما في ذلك التجميع) في وقت تجميع معقول (60-120 ثانية).
- كرر الخطوات 5.4-5.8 لتسجيل صور FLIM للخلايا من مجالات رؤية مختلفة.
ملاحظة: يجب أن تقتصر قياسات FLIM للخلايا الحية الملطخة ب BODIPY 2 على ~ 30 دقيقة بعد إضافة BODIPY 2.
6. تحليل البيانات
- تحليل شدة الفلورة: نسبة الأكسدة والاختزال
- افتح الصور لشدة التألق ل NAD (P) H و FAD باستخدام ImageJ.
- قم بتمييز منطقة خالية من الخلايا في صورة NAD(P)H باستخدام دائرة أو خيار مربع. انقر فوق قياس ، ثم انقر فوق طرح (حدد عملية في اللوحة الرئيسية ، ثم الرياضيات والطرح) لطرح القيمة التي تم الحصول عليها لإشارة الخلفية.
- كرر الخطوة 6.1.2 لصورة FAD.
- احصل على صورة نسبة الأكسدة والاختزال بقسمة شدة مضان FAD على شدة مضان NAD (P) H. قم بذلك عن طريق تحديد عملية على اللوحة الرئيسية ، ثم حدد حاسبة الصورة وتقسيم. حدد المربع إنشاء نافذة جديدة، ثم اضغط على موافق.
- احفظ الصورة بتنسيق TIFF.
- لحساب نسبة الأكسدة والاختزال ، حدد منطقة السيتوبلازم في الخلية المحددة على صورة TIFF واضغط على المفتاح M . كرر ذلك لجميع الخلايا ذات الأهمية.
- استيراد القياس إلى مستند جدول بيانات.
ملاحظة: بدلا من ذلك ، يمكن قياس شدة التألق ل NAD (P) H و FAD في الخلايا باستخدام البرنامج القياسي للمجهر ويمكن الحصول على نسبة الأكسدة والاختزال بقسمة هذه القيم في برنامج جدول البيانات.
- تحليل بيانات FLIM: التمثيل الغذائي
- استيراد صورة FLIM ل NAD (P) H إلى برنامج SPCImage.
- قم بتطبيق ملاءمة تضموس ثنائي الأس على الصورة عن طريق وضع 2 في قسم المكونات .
- قم بإصلاح معلمة الإزاحة عن طريق تحديد المربع المقابل في برنامج SPCImage.
- انتقل إلى الخيارات وحدد نموذج. استخدم نموذج التركيب متعدد الأس غير المكتمل وطريقة الملاءمة MLE.
- اضبط التجميع لتحقيق شدة بكسل تبلغ ≥5000 فوتون لكل منحنى اضمحلال.
- تحقق من قيمة χ2. يشير χ2≤ 1.20 إلى أن النموذج المستخدم يوفر ملاءمة معقولة.
- احسب الرسم البياني لعمر الفلورسنت في كل صورة بالنقر فوق القائمة العلوية حساب ، ثم على مصفوفة الاضمحلال.
- حدد المنطقة الموجودة في السيتوبلازم للخلية المحددة كمنطقة اهتمام.
- قم بتحليل مكونات العمر القصير والطويل (τ1 و τ2 ، على التوالي) والسعة النسبية لمكونات العمر (a1 وa 2 ،حيث 1 + a2 = 100٪) باستخدام خيار اللون .
- تصدير القياسات إلى برنامج جداول بيانات.
- كرر الخطوات 6.2.8-6.2.10 لكل خلية ذات أهمية.
- كرر الخطوات 6.2.1-6.2.11 لصورة FAD.
- تحليل بيانات FLIM: اللزوجة
- استيراد صورة FLIM إلى برنامج SPCImage لتحليل بيانات FLIM.
- قم بإزالة العلامة الموجودة في مربع Scatter .
- ضع 1 في قسم المكونات ، حيث يجب أن يتناسب اضمحلال مضان الدوار مع نموذج أحادي الأس.
- اضبط التجميع لتحقيق شدة بكسل تبلغ ≥5000 فوتون لكل منحنى اضمحلال.
- تحقق من قيمة χ2 في غشاء البلازما. تشير قيمة χ2≤ 1.20 إلى أن النموذج المستخدم يوفر ملاءمة معقولة. في حالة χ2≥ 1.20 ، لا ينطبق التقريب الأسي الأحادي ، ويمكن أن تشير هذه البيانات إلى تراكم الصبغة ويجب التخلص منها. يجعل التجميع من المستحيل استخدام منحنيات المعايرة والخيوط في تقديرات اللزوجة غير الصحيحة.
ملاحظة: قد يكون الاضمحلال الأسي ثنائي الأس مؤشرا على التجميع. على المجهر مع وحدة FLIM مع مرشحات متغيرة ، يمكن اكتشاف ذلك عن طريق اختبار نطاقات الطول الموجي للبث المونومر ونوعي الركام ، 500-550 نانومتر و 580-650 نانومتر ، كما هو موضح في المرجع21. - قم بإنشاء الرسم البياني لعمر التألق τ لكل صورة بالنقر فوق القائمة العلوية حساب ، ثم على مصفوفة الاضمحلال.
- حدد منطقة غشاء البلازما للخلية الفردية ذات الاضمحلال الأحادي الأسي ، χ2≤ 1.20 ، باستخدام خيار عائد الاستثمار.
- تصدير قيمة عمر التألق إلى برنامج جداول بيانات.
- كرر الخطوات 6.3.7-6.3.8 لكل خلية ذات أهمية.
- قم بتحويل الأعمار المقاسة تجريبيا ل BODIPY 2 (بالنانوثانية) إلى قيم اللزوجة (بالقسط الكتلي) باستخدام المعادلة التالية (التي تم الحصول عليها سابقا على أساس مخطط معايرة BODIPY في مخاليط الميثانول / الجلسرين):
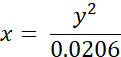
حيث x - اللزوجة (في cP) ، y - عمر التألق τ (في ns).
ملاحظة: يعد IRF (وظيفة استجابة الأداة) جزءا مهما من تركيب FLIM. في SPCImage ، يتم حساب IRF تلقائيا من الحافة الصاعدة لمنحنيات اضمحلال التألق. وفي الوقت نفسه ، يمكن تسجيل IRF باستخدام عينة غير فلورية ، على سبيل المثال ، السيراميك ، أو عينة تنتج إشارة SHG (الجيل التوافقي الثاني) ، على سبيل المثال ، الكولاجين أو بلورات اليوريا أو السكر. لا يوصى باستخدام IRFs المسجلة إذا كان هناك خيار لحسابها في البرنامج.
النتائج
باستخدام البروتوكول الموضح هنا ، قمنا بتصور العوامل المساعدة الأيضية ولزوجة الغشاء المجهري في الخلايا المزروعة الحية باستخدام FLIM. تم إجراء القياسات في خطوط خلايا سرطانية مختلفة - سرطان القولون والمستقيم البشري HCT116 ، وسرطان القولون الفئراني CT26 ، وسرطا...
Discussion
يوضح هذا البروتوكول إمكانيات FLIM للتحليل متعدد المعلمات والوظيفية والفيزيائية الحيوية للخلايا السرطانية. يتيح لنا الجمع بين التصوير الأيضي البصري القائم على التألق الداخلي وقياسات لزوجة غشاء البلازما باستخدام الملصقات الخارجية باستخدام الدوار الجزيئي الفلوري توصيف ?...
Disclosures
المؤلفون ليس لديهم ما يكشفون عنه.
Acknowledgements
تم دعم تطوير بروتوكول التصوير الأيضي من قبل وزارة الصحة في الاتحاد الروسي (مهمة حكومية ، رقم التسجيل АААА-А20-120022590098-0). تم دعم دراسة اللزوجة من قبل مؤسسة العلوم الروسية (المشروع رقم 20-14-00111). يعرب المؤلفون عن امتنانهم لأنطون بليخانوف (PRMU) لمساعدته في إنتاج الفيديو.
Materials
| Name | Company | Catalog Number | Comments |
| Item/Device | |||
| Cell culture incubator | Sanyo | 37°C, 5% CO2, humidified atmosphere | |
| Centrifuge 5702 R | Eppendorf | 5703000010 | |
| imageJ 1.53c | Wayne Rasband (NIH) | ||
| FLIM module Simple Tau 152 TCSPC (in LSM 880) | Becker & Hickl GmbH | ||
| Laminar flow hood | ThermoFisher Scientific | ||
| Leica microscope DFC290 | Leica Microsystems | ||
| LSM 880 confocal microscope | Carl Zeiss | ||
| Ti:Sapphire femtosecond laser Mai Tai | Spectra Physics | ||
| Microscope incubator XLmulti S DARK LS | PeCon GmbH | 273-800 050 | |
| Mechanical pipettor | Sartorius mLINE | volume 0.5-10 μL; 20-200 μL; 100-1000 μL | |
| Oil-immersion objective C-Apochromat 40×/1.2 NA W Korr (in LSM 880) | Carl Zeiss | 421767-9971-790 | |
| Power-Tau 152 module with the detector HPM-100-40 | Becker&Hickl GmbH | ||
| SPCImage software | Becker & Hickl GmbH | SPC 9.8; SPCImage 8.3 | |
| ZEN software | Carl Zeiss | ZEN 2.1 SP3 (black), Version 14.0.0.201 | |
| Reagent/Material | |||
| 5-fluorouracil | Medac GmbH | 3728044 | |
| DMEM | Gibco, Life Technologies | 31885023 | |
| DMSO | PanEco | F135 | |
| FBS | Hyclone | A3160801 | |
| FluoroBright DMEM | Gibco, Life Technologies | A1896701 | |
| Hank’s solution without Ca2+/Mg2+ | Gibco, Life Technologies | 14175 | |
| l-Glutamine | PanEco | F032 | |
| Mammalian cells | HCT116, CT26, HeLa Kyoto, huFB | ||
| Molecular rotor BODIPY 2 | Synthesized and Supplied by Marina Kuimova Group, Imperial College London | ||
| Penicillin/streptomycin | PanEco | A065 | |
| Tissue culture dish with cover glass-bottom FluoroDishes | World Precision Instruments, Inc | ||
| Trypsin- EDTA 0.25% | PanEco | P034 | |
| Versen buffer | PanEco | R080p |
References
- Vazquez, A., et al. Cancer metabolism at a glance. Journal of Cell Science. 129 (18), 3367-3373 (2016).
- Li, Z., Zhang, H. Reprogramming of glucose, fatty acid, and amino acid metabolism for cancer progression. Cellular and Molecular Life Sciences. 73 (2), 377-392 (2015).
- Strickaert, A., et al. Cancer heterogeneity is not compatible with one unique cancer cell metabolic map. Oncogene. 36 (19), 2637-2642 (2016).
- Szlasa, W., Zendran, I., Zalesińska, A., Tarek, M., Kulbacka, J. Lipid composition of the cancer cell membrane. Journal of Bioenergetics and Biomembranes. 52, 321-342 (2020).
- Preta, G. New insights into targeting membrane lipids for cancer therapy. Frontiers in Cell and Developmental Biology. 8, 571237 (2020).
- Kojima, K. Molecular aspects of the plasma membrane in tumor cells. Nagoya Journal of Medical Science. 56, 1-18 (1993).
- Datta, R., Heaster, T. M., Sharick, J. T., Gillette, A. A., Skala, M. C. Fluorescence lifetime imaging microscopy: fundamentals and advances in instrumentation, analysis, and applications. Journal of Biomedical Optics. 25 (7), 071203 (2020).
- Becker, W. Advanced time-correlated single photon counting applications. Springer Series in Chemical Physics. , (2015).
- Shirmanova, M. V., Shcheslavskiy, V. I., Lukina, M. M., Becker, W., Zagaynova, E. V. Exploring tumor metabolism with time-resolved fluorescence methods: From single cells to a whole tumor. Multimodal Optical Diagnostics of Cancer. 3, 133-155 (2020).
- Kalinina, S., Rück, A. FLIM and PLIM in biomedical research. An innovative way to combine autofluorescence and oxygen measurements. Photonics & Lasers in Medicine. 5 (4), 257-266 (2016).
- Kuimova, M. K. Molecular rotors image intracellular viscosity. Chimia. 66 (4), 159-165 (2012).
- Kuimova, M. K. Mapping viscosity in cells using molecular rotors. Physical Chemistry Chemical Physics. 14 (37), 12671 (2012).
- Haidekker, M. A., Theodorakis, E. A. Molecular rotors-fluorescent biosensors for viscosity and flow. Organic & Biomolecular Chemistry. 5, 1669-1678 (2007).
- Liu, X., et al. Molecular mechanism of viscosity sensitivity in BODIPY rotors and application to motion-based fluorescent sensors. ACS Sensors. 5 (3), 731-739 (2020).
- Shirmanova, M. V., Shimolina, L. E., Lukina, M. M., Zagaynova, E. V., Kuimova, M. K., Dmitriev, R. Live cell imaging of viscosity in 3D tumour cell models. Multi-Parametric Live Cell Microscopy of 3D Tissue Models. Advances in Experimental Medicine and Biology. 1035, 143-153 (2017).
- Shimolina, L. E., et al. Imaging tumor microscopic viscosity in vivo using molecular rotors. Scientific Reports. 7, 41097 (2017).
- Scott, T. G., Spencer, R. D., Leonard, N. J., Weber, G. Synthetic spectroscopic models related to coenzymes and base pairs. V. Emission properties of NADH. Studies of fluorescence lifetimes and quantum efficiencies of NADH, AcPyADH, [reduced acetylpyridineadenine dinucleotide] and simplified synthetic models. Journal of the American Chemical Society. 92 (3), 687-695 (1970).
- Harvey, R. A., Damle, S. A fluorescent modification of flavin adenine dinucleotide. FEBS Letters. 26 (1-2), 341-343 (1972).
- Kubánková, M., Summers, P., López-Duarte, I., Kiryushko, D., Kuimova, M. K. Microscopic viscosity of neuronal plasma membranes measured using fluorescent molecular rotors: Effects of oxidative stress and neuroprotection. ACS Applied Materials and Interfaces. 11, 36307-36315 (2019).
- Kubánková, M., López-Duarte, I., Kiryushko, D., Kuimova, M. K. Molecular rotors report on changes in live cell plasma membrane microviscosity upon interaction with beta-amyloid aggregates. Soft Matter. 14, 9466-9474 (2018).
- Wu, Y., et al. Molecular rheometry: Direct determination of viscosity in Lo and Ld lipid phases via fluorescence lifetime imaging. Physical Chemistry Chemical Physics. 15 (36), 14986 (2013).
- Shimolina, L. E., et al. Mapping cisplatin-induced viscosity alterations in cancer cells using molecular rotor and fluorescence lifetime imaging microscopy. Journal of Biomedical Optics. 25 (12), 126004 (2020).
- Berezin, M. Y., Achilefu, S. Fluorescence lifetime measurements and biological imaging. Chemical Reviews. 110 (5), 2641-2684 (2010).
- Skala, M. C., et al. In vivo multiphoton fluorescence lifetime imaging of protein-bound and free nicotinamide adenine dinucleotide in normal and precancerous epithelia. Journal of Biomedical Optics. 12 (2), 024014 (2007).
- Rück, A., Hauser, C., Mosch, S., Kalinina, S. Spectrally resolved fluorescence lifetime imaging to investigate cell metabolism in malignant and nonmalignant oral mucosa cells. Journal of Biomedical Optics. 19 (9), 096005 (2014).
- Lee, D. -. H., Li, X., Ma, N., Digman, M. A., Lee, A. P. Rapid and label-free identification of single leukemia cells from blood in a high-density microfluidic trapping array by fluorescence lifetime imaging microscopy. Lab on a Chip. 18 (9), 1349-1358 (2018).
- Lukina, M. M., et al. Interrogation of tumor metabolism in tissue samples ex vivo using fluorescence lifetime imaging of NAD(P)H. Methods and Applications in Fluorescence. 8 (1), 014002 (2019).
- Alam, S. R., et al. Investigation of mitochondrial metabolic response to doxorubicin in prostate cancer cells: An NADH, FAD and Tryptophan FLIM assay. Scientific Reports. 7 (1), 10451 (2017).
- Walsh, A. J., et al. Quantitative optical imaging of primary tumor organoid metabolism predicts drug response in breast cancer. Cancer Research. 74 (18), 5184-5194 (2014).
- Lukina, M. M., et al. In vivo metabolic and SHG imaging for monitoring of tumor response to chemotherapy. Cytometry Part A. 95 (1), 47-55 (2019).
- Shah, A. T., et al. Optical metabolic imaging of treatment response in human head and neck squamous cell carcinoma. PLoS One. 9 (3), 90746 (2014).
- Sergeeva, T. F., et al. Relationship between intracellular pH, metabolic co-factors, and caspase-3 activation in cancer cells during apoptosis. Biochimica et Biophysica Acta (BBA) - Molecular Cell Research. 1864 (3), 604-611 (2017).
- Shirmanova, M. V., et al. Chemotherapy with cisplatin: insights into intracellular pH and metabolic landscape of cancer cells in vitro and in vivo. Scientific Reports. 7, 8911 (2017).
- Lukina, M. M., et al. Metabolic cofactors NAD(P)H and FAD as potential indicators of cancer cell response to chemotherapy with paclitaxel. Biochimica et Biophysica Acta (BBA) - General Subjects. 1862 (8), 1693-1700 (2018).
- Druzhkova, I. N., et al. The metabolic interaction of cancer cells and fibroblasts - coupling between NAD(P)H and FAD, intracellular pH and hydrogen peroxide. Cell Cycle. 15 (9), 1257-1266 (2016).
- Lakowicz, J., Szmacinski, H., Nowaczyk, K., Johnson, M. Fluorescence lifetime imaging of free and protein bound NADH. Proceedings of the National Academy of Sciences of the United States of America. 89 (4), 1271-1275 (1992).
- Vanden Berg, P. A. W., Feenstra, K. A., Mark, A. E., Berendsen, H. J. C., Visser, A. J. W. G. Dynamic conformations of flavin adenine dinucleotide: Simulated molecular dynamics of the flavin cofactor related to the time-resolved fluorescence characteristics. The Journal of Physical Chemistry B. 106 (34), 8858-8869 (2002).
- Liu, Z., et al. Mapping metabolic changes by noninvasive, multiparametric, high-resolution imaging using endogenous contrast. Science Advances. 4 (3), (2018).
- Blacker, T. S., et al. Separating NADH and NADPH fluorescence in live cells and tissues using FLIM. Nature Communications. 5, 3936 (2014).
- Lu, W., Wang, L., Chen, L., Hui, S., Rabinowitz, J. D. Extraction and quantitation of nicotinamide adenine dinucleotide redox cofactors. Antioxidants and Redox Signaling. 28 (3), 167-179 (2018).
- Ranjit, S., Malacrida, L., Jameson, D. M., Gratton, E. Fit-free analysis of fluorescence lifetime imaging data using the phasor approach. Nature Protocols. 13 (9), 1979-2004 (2018).
- Stringari, C., et al. Phasor approach to fluorescence lifetime microscopy distinguishes different metabolic states of germ cells in a live tissue. Proceedings of the National Academy of Sciences of the United States of America. 108 (33), 13582-13587 (2011).
- Smith, J. T., et al. Fast fit-free analysis of fluorescence lifetime imaging via deep learning. Proceedings of the National Academy of Sciences of the United States of America. 116 (48), 24019-24030 (2019).
- Wang, S., Chacko, J. V., Sagar, A. K., Eliceiri, K. W., Yuan, M. Nonparametric empirical Bayesian framework for fluorescence-lifetime imaging microscopy. Biomedical Optics Express. 10 (11), 5497-5517 (2019).
- Quinn, K. P., et al. Quantitative metabolic imaging using endogenous fluorescence to detect stem cell differentiation. Scientific Reports. 3 (1), 3432 (2013).
- Hou, J., et al. Correlating two-photon excited fluorescence imaging of breast cancer cellular redox state with seahorse flux analysis of normalized cellular oxygen consumption. Journal of Biomedical Optics. 21 (6), 060503 (2016).
- Wang, H., et al. Red-emitting fluorescence probe for sensing viscosity in living cells. Chemical Papers. 74, 1071-1078 (2020).
- Chen, B., et al. Sensing and imaging of mitochondrial viscosity in living cells by a red photoluminescent probe with long lifetime. Chemical Communications. 55, 7410 (2019).
- Shen, B., Wang, L. F., Zhi, X., Qian, Y. Construction of a red emission BODIPY-based probe for tracing lysosomal viscosity changes in culture cells. Sensors and Actuators B: Chemical. 304, 127271 (2019).
- Chen, T., Chen, Z., Liu, R., Zheng, S. NIR fluorescent probe for detection of viscosity and lysosome imaging in live cells. Organic and Biomolecular Chemistry. 17, 6398 (2019).
- Angelucci, C., et al. Epithelial-stromal interactions in human breast cancer: effects on adhesion, plasma membrane fluidity and migration speed and directness. PLoS One. 7 (12), 50804 (2012).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved