Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
Исследование метаболизма и вязкости раковых клеток с помощью флуоресцентной микроскопии с визуализацией в течение жизни
В этой статье
Резюме
В данной работе мы демонстрируем использование флуоресцентной микроскопии с визуализацией времени жизни (FLIM) для последовательного отображения клеточного метаболизма и вязкости плазматической мембраны в культуре живых раковых клеток. Метаболическая оценка проводится путем обнаружения эндогенной флуоресценции. Вязкость измеряется с помощью флуоресцентного молекулярного ротора.
Аннотация
Вязкость является важным физическим свойством биологической мембраны, так как является одним из ключевых параметров для регуляции морфологического и физиологического состояния живых клеток. Известно, что плазматические мембраны опухолевых клеток имеют значительные изменения в своем составе, структуре и функциональных характеристиках. Наряду с нарушением регуляции метаболизма глюкозы и липидов, эти специфические свойства мембран помогают опухолевым клеткам адаптироваться к враждебному микроокружению и развивать устойчивость к медикаментозной терапии. В данной работе мы демонстрируем использование флуоресцентной микроскопии с визуализацией времени жизни (FLIM) для последовательного отображения клеточного метаболизма и вязкости плазматической мембраны в культуре живых раковых клеток. Метаболические оценки проводятся путем обнаружения флуоресценции эндогенных метаболических кофакторов, таких как восстановленный никотинамидадениндинуклеотид NAD(P)H и окисленные флавины. Вязкость измеряется с помощью флуоресцентного молекулярного ротора, синтетического вязкочувствительного красителя, с сильной зависимостью времени жизни флуоресценции от вязкости ближайшего окружения. В сочетании эти методы позволяют нам лучше понять связь между состоянием мембраны и метаболическим профилем раковых клеток, а также визуализировать изменения, вызванные химиотерапией.
Введение
Злокачественная трансформация клеток сопровождается множественными изменениями их морфологического и физиологического состояния. Быстрый и неконтролируемый рост раковых клеток требует фундаментальной реорганизации биохимических путей, ответственных за производство энергии и биосинтез. Характерными признаками метаболизма рака являются повышенная скорость гликолиза даже при нормальной концентрации кислорода (эффект Варбурга), использование аминокислот, жирных кислот и лактата в качестве альтернативных видов топлива, высокая продукция АФК в присутствии высоких уровней антиоксидантов и повышенный биосинтез жирных кислот 1,2. В настоящее время признано, что метаболизм раковых клеток обладает высокой гибкостью, что позволяет им адаптироваться к неблагоприятной и неоднородной среде и обеспечиваетдополнительное преимущество в выживаемости.
Измененный метаболизм поддерживает специфическую организацию и состав мембран опухолевых клеток. Липидный профиль плазматической мембраны в раковых клетках количественно отличается от нераковых клеток. Основными изменениями в липидоме являются повышенный уровень фосфолипидов, включая фосфатидилинозитол, фосфатидилсерин, фосфатидилэтаноламин и фосфатидилхолин, снижение уровня сфингомиелина, увеличение количества холестерина и снижение степени ненасыщенности жирных кислот, иэто лишь некоторые из 4,5,6. Поэтому физические свойства мембраны, такие как вязкость мембраны, обратная текучести, неизбежно изменяются. Поскольку вязкость определяет проницаемость биологических мембран и контролирует активность мембраноассоциированных белков (ферментов, транспортеров, рецепторов), ее гомеостатическая регуляция жизненно важна для функционирования клеток. В то же время изменение вязкости путем корректировки липидного профиля мембраны важно для миграции/инвазии клеток и выживания при условных изменениях.
Флуоресцентная микроскопия с визуализацией в течение жизни (FLIM) стала мощным подходом к неинвазивной оценке множественных параметров в живых клетках с использованием эндогенной флуоресценции или экзогенных зондов7. FLIM обычно реализуется на многофотонных лазерных сканирующих микроскопах, которые обеспечивают (суб)клеточное разрешение. Будучи оснащенным модулем коррелированного по времени однофотонного счета (TCSPC), он позволяет измерять флуоресценцию с временным разрешением с высокой точностью8.
Зондирование клеточного метаболизма методом FLIM основано на флуоресцентном измерении эндогенных кофакторов дегидрогеназ, восстановленного никотинамидадениндинуклеотида (фосфата) NAD(P)H и окисленных флавинов - флавинадениндинуклеотида FAD и флавинмононуклеотида FMN, которые выступают в качестве переносчиков электронов в ряде биохимических реакций 7,9,10. Обнаруженная флуоресценция NAD(P)H происходит от NADH и его фосфорилированной формы, NADPH, поскольку они спектрально почти идентичны. Как правило, флуоресцентные распады NAD(P)H и флавинов укладываются в биэкспоненциальную функцию. В случае NAD(P)H первый компонент (~0,3-0,5 нс, ~70%-80%) относится к его свободному состоянию, связанному с гликолизом, а второй компонент (~1,2-2,5 нс, ~20%-30%) — к его связанному с белками состоянию, связанному с митохондриальным дыханием. В случае флавинов короткая составляющая (~0,3-0,4 нс, ~75%-85%) может быть отнесена к закаленному состоянию FAD, а длинная составляющая (~2,5-2,8 нс, ~15%-25%) - к непогашенной FAD, FMN и рибофлавину. Изменения в относительных уровнях гликолиза, глутаминолиза, окислительного фосфорилирования и синтеза жирных кислот приводят к изменениям коротко- и долгоживущих фракций кофакторов. Кроме того, отношение интенсивности флуоресценции этих флуорофоров (окислительно-восстановительное отношение) отражает клеточный окислительно-восстановительный статус и также используется в качестве метаболического индикатора. Несмотря на то, что окислительно-восстановительный коэффициент представляет собой более простую метрику по сравнению со временем жизни флуоресценции, с точки зрения сбора данных, FLIM выгоден для оценки NAD(P)H и FAD, потому что время жизни флуоресценции является внутренней характеристикой флуорофора и почти не зависит от таких факторов, как мощность возбуждения, фотообесцвечивание, фокусировка, рассеяние и поглощение света, особенно в тканях. в отличие от интенсивности излучения.
Один из удобных способов картирования вязкости в живых клетках и тканях на микроскопическом уровне основан на использовании флуоресцентных молекулярных роторов, небольших синтетических вязкочувствительных красителей, в которых параметры флуоресценции сильно зависят от локальной вязкости11,12. В вязкой среде время жизни ротора флуоресценции увеличивается из-за замедления внутримолекулярного скручивания или вращения. Среди молекулярных роторов производные бор-дипиррометена (BODIPY) хорошо подходят для определения вязкости в биологических системах, поскольку они обладают хорошим динамическим диапазоном времени жизни флуоресценции в физиологическом диапазоне вязкости, температурной независимостью, моноэкспоненциальными затуханиями флуоресценции, которые позволяют легко интерпретировать данные, достаточной растворимостью в воде и низкой цитотоксичностью13,14. Количественная оценка микровязкости с использованием роторов на основе BODIPY и FLIM ранее была продемонстрирована на раковых клетках in vitro, сфероидах многоклеточных опухолей и опухолях мышей in vivo15,16.
В данной статье мы представляем подробное описание методик последовательного зондирования для изучения клеточного метаболизма и вязкости плазматических мембран в раковых клетках in vitro с помощью FLIM. Чтобы избежать контаминации относительно слабой эндогенной флуоресценции флуоресценцией ротора на основе BODIPY, визуализация одного и того же слоя клеток выполняется последовательно с визуализацией флуоресценции NAD(P)H и FAD. Время жизни флуоресценции кофакторов измеряется в цитоплазме, а время жизни флуоресценции ротора измеряется в плазматических мембранах клеток путем ручного выбора соответствующих зон в качестве областей интереса. Протокол был применен для корреляции метаболического состояния и вязкости различных линий раковых клеток, а также для оценки изменений после химиотерапии.
Протокол пробоподготовки методом FLIM не отличается от протокола конфокальной флуоресцентной микроскопии. После того, как данные получены, основной задачей является извлечение времени жизни флуоресценции из необработанных данных. Эффективность протокола продемонстрирована с использованием клеток HCT116 (колоректальная карцинома человека), CT26 (карцинома толстой кишки мыши), HeLa (карцинома шейки матки человека) и huFB (фибробласты кожи человека).
протокол
1. Описание минимальной настройки для выполнения FLIM
- Для проведения этого эксперимента убедитесь, что имеется необходимая установка: инвертированный конфокальный микроскоп, импульсный лазер, обычно ps или fs, с сигналом синхронизации, детектор быстрого подсчета фотонов (временная характеристика 150 пс) и электроника для подсчета фотонов, доступные выходные и входные порты для детектора и лазера, соответственно, на микроскопе, импульсы сканирования часов от контроллера сканирования микроскопа, сканирующая головка микроскопа с сумматорами лазерного луча и основными дихроичными светоделителями, подходящими для длины волны лазера, используемого для FLIM.
- Если для FLIM используется двухфотонное возбуждение, убедитесь, что микроскоп оснащен портом NDD.
- Для исследований клеток млекопитающих, особенно для долгосрочных экспериментов, убедитесь, что в инкубаторе сCO2 поддерживается желаемая температура.
ПРИМЕЧАНИЕ: О системе, использованной в этом эксперименте, см. Таблицу материалов.
2. Подготовка клеток к микроскопии
- Регулярно выращивайте клетки в инкубаторе при температуре 37 °C с 5%CO2 и влажной атмосфере.
- Для микроскопии клеточную суспензию готовят в полной питательной среде в концентрации 1 х 106 клеток/мл.
ПРИМЕЧАНИЕ: Концентрация клеток и условия среды зависят от клеток. Количество клеток, используемых для посева, и время инкубации должны быть адаптированы таким образом, чтобы получить 70%-80% конфлюенции в микроскопической чашке. - Засейте клетки на стеклянные 35-миллиметровые чашки для клеточных культур (1 x 10,5 клеток в 100 μл на чашку) с помощью автоматической пипетки объемом 200 μл.
ПРИМЕЧАНИЕ: Используйте решетчатые стеклянные донные чашки для засева клеток, чтобы контролировать клетки в одних и тех же микроскопических полях зрения в динамике.- Во время ручного посева следите за тем, чтобы наконечник пипетки не царапал дно или стенки чашки, чтобы не повредить дно.
- Поместите чашку в инкубатор сCO2 (37 °C, 5%CO2, влажная атмосфера) и инкубируйте клетки в течение 24 часов.
- Через 24 ч достаньте чашку из инкубатора и проверьте морфологию и слияние клеток под световым микроскопом. Если клетки не достигли примерно 80% конфлюенции, инкубируйте еще 24 ч.
- Аккуратно удалите старую среду из чашки с помощью автоматической пипетки объемом 1 000 мкл и добавьте 2 мл среды DMEM без фенолового красного (например, DMEM Life или FluoroBrite).
ПРИМЕЧАНИЕ: Для визуализации можно использовать различные питательные среды. Избегайте фенолового красного в среде при использовании клеток для микроскопии. - Поместите чашку в инкубатор на 60-120 минут, чтобы дать возможность адаптироваться клеткам.
3. ФЛИМ метаболических кофакторов
- Поместите чашку со стеклянным дном с ячейками (из шага 2.7) на предметный столик микроскопа.
- Перейдите на вкладку «Найти» в программном обеспечении лазерного сканирующего микроскопа (например, ZEN - ZEISS Efficient Navigation), а затем нажмите « Свет пропускания» (TL), чтобы включить свет.
- Найдите фокальную плоскость образца путем просмотра через окуляр на центральном уровне среза ячеек, где квадрат максимально занят клетками (при увеличении 40х).
- Нажмите на кнопку ВЫКЛ, чтобы выключить свет.
- Откройте вкладку Приобретение . Для получения изображений интенсивности пропускания и автофлуоресценции эндогенного NAD(P)H введите следующие настройки: Длина волны возбуждения: двухфотонная мода 750 нм, Диапазон регистрации: 450-490 нм, Мощность лазера: 5% (~6 мВт), Размер изображения: 1024 x 1024 пикселей.
ПРИМЕЧАНИЕ: Выбор длины волны возбуждения и диапазона регистрации основан на спектральных характеристиках (максимальное возбуждение и максимальное излучение) NAD(P)H17.- Для получения изображения используйте масляный иммерсионный объектив СPlan-Apochromat 40x/1.3 NA.
- Нажмите кнопку Snap и сохраните изображение в формате ZEN.
- Чтобы получить изображения интенсивности пропускания и автофлуоресценции FAD, измените длину волны возбуждения на 900 нм. Установите диапазон приводки: 500-550 нм, мощность лазера: 9% (~6 мВт) и размер изображения: 1024 x 1024 пикселей.
ПРИМЕЧАНИЕ: Выбор длины волны возбуждения и диапазона регистрации основан на спектральных характеристиках (максимальное возбуждение и максимальное излучение) FAD18.- Для получения изображения используйте масляный иммерсионный объектив СPlan-Apochromat 40x/1.3 NA.
- Нажмите кнопку Snap и сохраните изображение в формате ZEN.
- Для параметра NAD(P)H установите параметры, описанные на шаге 3.5, в программном обеспечении лазерного сканирующего микроскопа. Поменяйте Размер изображения на 256 x 256 пикселей.
- Введите следующие параметры в меню SPCM (Single Photon Counting Modules) Операционное программное обеспечение модуля FLIM: Время сбора: 60 с; Диапазон TAC: 5.00E-8; Минимальный лимит CFD: -29.41; Разрешение АЦП: 256, размер изображения: 256 x 256 пикселей.
- Отсканируйте образец в течение 60 с, остановите сканирование и сохраните полученное FLIM-изображение NAD(P)H.
- Проверьте полученные данные FLIM. Для этого откройте исходные данные в программе для обработки изображений, выберите пиксель в цитоплазме клетки, наведя на него курсор, и проанализируйте распад флуоресценции в этом пикселе. Интенсивность пикселей должна составлять ≥3000 фотонов на кривую распада при биннинге 1.
Примечание: Если количество фотонов меньше 3000, увеличьте мощность лазера или время сбора изображений, контролируя при этом морфологию клеток и скорость подсчета фотонов. Как правило, если падение скорости подсчета превышает 10% от исходного значения, происходит фотообесцвечивание. - Для FAD задайте параметры, описанные в шаге 3.7, в программном обеспечении лазерного сканирующего микроскопа. Измените размер изображения на 256 x 256 пикселей.
- Введите следующие параметры в меню SPCM (Single Photon Counting Modules) Операционное программное обеспечение модуля FLIM: Время сбора: 60 с; Диапазон TAC: 5.00E-8; Минимальный лимит CFD: -29.41; Разрешение АЦП: 256, размер изображения: 256 x 256 пикселей.
- Сканируйте образец в течение 60 с. Остановите сканирование и сохраните полученное FLIM изображение FAD.
ПРИМЕЧАНИЕ: Параметры, указанные в шагах 3.10 и 3.15, относятся к используемой электронике и детектору. - Проверьте полученные данные, как описано в шаге 3.12.
- Повторите шаги 3.5-3.16 для записи изображений FLIM с разных полей зрения.
4. Окрашивание клеток флуоресцентным молекулярным ротором
ПРИМЕЧАНИЕ: Клетки визуализируются в растворе флуоресцентного молекулярного ротора без промывки при комнатной температуре (~20 °C) для замедления интернализации ротора. Вязкость мембраны зависит от температуры, как показано в наших предыдущих работах19,20. Температурный столик микроскопа должен быть выключен заранее, т.е. до того, как ротор будет добавлен к элементам. Для нашей установки охлаждение сцены занимает около 10 минут.
- Приготовьте общий стоковый раствор флуоресцентного молекулярного ротора BODIPY 2 (Stock 1, 25,7 мМ).
- Откройте BODIPY 2 в стерильной среде и весьте примерно 2 мг, используя точные весы. Аккуратно поместите его в микроцентрифужную пробирку.
- С помощью автоматической дозатора объемом 20 мкл добавьте 3 мкл подходящего растворителя (например, ДМСО).
- После того, как образец полностью растворится в ДМСО, добавьте 297 мкл стерильного PBS и тщательно перемешайте с помощью автоматической пипетки объемом 200 мкл.
ПРИМЕЧАНИЕ: Хранить исходный раствор в холодильнике при температуре +4 °C в темной упаковке. После повторного взвешивания он может храниться в холодильнике в течение нескольких месяцев.
- Приготовьте бульон 2 (8,9 мМ), добавив 25 мкл общего исходного раствора (бульон 1) в микроцентрифужную пробирку, а затем 48 мкл стерильного PBS. Аккуратно перемешайте с помощью автоматической пипетки объемом 200 μл.
ПРИМЕЧАНИЕ: Используйте материал 2 для приготовления окончательного окрашивающего материала, который применяется для окрашивания клеток, поскольку требуется микромолярная концентрация. - Аккуратно замените питательную среду в чашке (начиная с шага 3.1) ледяным раствором Ганка без Ca2+/Mg2+ и инкубируйте клетки при температуре +4 °C в течение 3 минут.
ПРИМЕЧАНИЕ: Использование ледяного раствора и инкубация при +4 °C замедляет интернализацию молекулярного ротора, а локальное окрашивание мембраны сохраняется в течение 20-30 минут. - Приготовьте окончательный окрашивающий раствор, содержащий 4,5 мкМ BODIPY 2, добавив 1 мкл Stock 2 к 999 мкл ледяного раствора Ганка или PBS.
ПРИМЕЧАНИЕ: Концентрация BODIPY 2 в окончательном окрашивающем растворе может быть увеличена до ~10 мкМ без какого-либо токсического воздействия на клетки, что приводит к более эффективному окрашиванию и большему количеству собранных фотонов. При более высоких концентрациях может произойти перегрузка детектора FLIM. - Отсадите раствор Ханка из чашки для культивирования клеток и замените его ледяным 4,5 μМ раствором BODIPY 2. Клетки визуализируются в растворе BODIPY 2 без промывки.
5. FLIM флуоресцентного молекулярного ротора в клетках
ПРИМЕЧАНИЕ: Всегда проводите FLIM флуоресцентного молекулярного ротора после FLIM метаболических кофакторов, поскольку спектр флуоресценции BODIPY 2 перекрывается с излучением эндогенных кофакторов NAD(Р)H и FAD 12,17,18.
- Перенесите чашку с окрашенными клетками на предметный столик микроскопа (~20 °C) для визуализации.
- В программном обеспечении лазерного сканирующего микроскопа для однофотонной моды задайте следующие параметры: возбуждение на длине волны 488 нм аргоновым ионным лазером, мощность лазера 1%-2%, регистрационный диапазон 500-550 нм.
- Для получения изображения используйте масляный иммерсионный объектив СPlan-Apochromat 40x/1.3 NA.
- Нажмите кнопку «Трансляция ». Начните сканирование и используйте позиционирование XY и Z с помощью встроенного моторизованного столика, отрегулируйте фокус и получите изображения пропускания и интенсивности флуоресценции клеток в окне предварительного просмотра. Сохраните полученные изображения, если это необходимо.
- Проверьте перекрывающееся изображение пропускания и флуоресценции, чтобы увидеть, исходит ли флуоресценция ротора из ожидаемого места (плазматической мембраны клетки).
- Введите в меню программного обеспечения SPCM модуля FLIM следующие параметры: Время сбора: 60 с; Диапазон TAC: 5.00E-8; Минимальный лимит CFD: -29.41; Разрешение АЦП: 256, размер изображения: 256 x 256 пикселей.
ПРИМЕЧАНИЕ: В зависимости от конфигурации системы и детекторов, используемых для FLIM, параметры получения изображения могут отличаться. - Настройте титановый сапфировый лазер микроскопа на длину волны 850 нм, а мощность лазера на 1%-2% для двухфотонного FLIM.
- Выберите вкладку «Непрерывно» в программном обеспечении лазерного сканирующего микроскопа, а затем нажмите «Пуск » в программном обеспечении SPCM. Отсканируйте образец в течение 60 с, остановите сканирование и сохраните полученное изображение FLIM.
- Проверьте полученные данные FLIM. Для этого загрузите исходные данные в программу для анализа данных FLIM SPCImage, выберите пиксель в мембране клетки, наведя на него курсор, и проанализируйте затухание флуоресценции в этом пикселе. Интенсивность пикселей должна составлять ≥5 000 за один распад (возможно, включая группирование) при разумном времени сбора (60-120 с).
- Повторите шаги 5.4-5.8 для записи FLIM-изображений ячеек из разных полей зрения.
ПРИМЕЧАНИЕ: Флим-измерения живых клеток, окрашенных BODIPY 2, должны быть ограничены ~30 мин после добавления BODIPY 2.
6. Анализ данных
- Анализ интенсивности флуоресценции: окислительно-восстановительное отношение
- Откройте изображения интенсивности флуоресценции NAD(P)H и FAD с помощью ImageJ.
- Выделите свободную от ячеек область на изображении NAD(P)H с помощью круга или квадрата. Нажмите на Измерить, а затем нажать на Вычесть (выберите Процесс на главной панели, а затем Математика и Вычитание), чтобы вычесть полученное значение фонового сигнала.
- Повторите шаг 6.1.2 для изображения FAD.
- Получите изображение окислительно-восстановительного отношения путем деления интенсивности флуоресценции FAD на интенсивность флуоресценции NAD(P)H. Для этого выберите «Процесс» на главной панели, а затем выберите «Калькулятор изображений» и «Разделить»; поставьте галочку в поле Создать новое окно, а затем нажмите OK.
- Сохраните изображение в формате TIFF.
- Чтобы рассчитать окислительно-восстановительное отношение, выберите область цитоплазмы в конкретной клетке на изображении TIFF и нажмите клавишу M . Повторите для всех интересующих ячеек.
- Импортируйте данные измерений в документ электронной таблицы.
ПРИМЕЧАНИЕ: В качестве альтернативы, интенсивность флуоресценции NAD(P)H и FAD в клетках может быть измерена с помощью стандартного программного обеспечения микроскопа, а окислительно-восстановительный коэффициент может быть получен путем деления этих значений в программном обеспечении для работы с электронными таблицами.
- Анализ данных FLIM: метаболизм
- Импортируйте FLIM-образ NAD(P)H в программное обеспечение SPCImage.
- Примените к изображению подгонку по биэкспоненциальному затуханию, поставив 2 в разделе Компоненты .
- Исправьте параметр Смещение , установив соответствующий флажок в программе SPCImage.
- Перейдите в раздел «Параметры» и выберите «Модель». Используйте модель неполной мультиэкспоненциальной аппроксимации и метод подгонки MLE.
- Настройте биннинг для достижения интенсивности пикселей в ≥5000 фотонов на кривую распада.
- Проверьте значение χ2. Значение χ2≤ 1,20 указывает на то, что используемая модель обеспечивает разумное соответствие.
- Рассчитайте гистограмму времени жизни флуоресценции на каждом изображении, нажав на верхнее меню Рассчитать, а затем на Decay Matrix.
- Выберите область в цитоплазме конкретной клетки в качестве области интереса.
- Проанализируйте короткую и длинную составляющие жизни (τ1 и τ2 соответственно) и относительные амплитуды составляющих жизни (a1 иa 2, гдеa 1 +a 2 = 100 %) с помощью опции «Цвет ».
- Экспортируйте измерения в программное обеспечение для работы с электронными таблицами.
- Повторите шаги 6.2.8-6.2.10 для каждой интересующей клетки.
- Повторите шаги 6.2.1-6.2.11 для FAD изображения.
- Анализ данных FLIM: вязкость
- Импортируйте изображение FLIM в программное обеспечение для анализа данных FLIM SPCImage.
- Снимите отметку в поле Scatter .
- Поставьте 1 в разделе Компоненты , так как затухание флуоресценции ротора должно соответствовать моноэкспоненциальной модели.
- Настройте группирование, чтобы достичь интенсивности пикселей в ≥5000 фотонов на кривую распада.
- Проверьте значение χ2 в плазматической мембране. Значение χ2≤ 1,20 указывает на то, что используемая модель обеспечивает разумное соответствие. В случае χ2≥ 1,20 моноэкспоненциальная аппроксимация неприменима, такие данные могут указывать на агрегацию красителя и должны быть отброшены. Агрегация делает невозможным использование калибровочных кривых и приводит к неправильным оценкам вязкости.
Примечание: Биэкспоненциальные распады могут указывать на агрегацию. На микроскопе с модулем FLIM с переменными фильтрами это можно обнаружить, испытав диапазоны длин волн мономера и агрегатного излучения 500–550 нм и 580–650 нм, как описано в ссылке21. - Сгенерируйте гистограмму времени жизни флуоресценции τ для каждого изображения, нажав на верхнее меню Calculate, а затем на Decay Matrix.
- Выберите область плазматической мембраны отдельной клетки с моноэкспоненциальным распадом, χ2≤ 1.20, используя опцию ROI.
- Экспортируйте значение времени жизни флуоресценции в программное обеспечение для работы с электронными таблицами.
- Повторите шаги 6.3.7-6.3.8 для каждой интересующей вас ячейки.
- Преобразуем экспериментально измеренные времена жизни BODIPY 2 (в нс) в значения вязкости (в сП) с помощью следующего уравнения (ранее полученного на основе графика калибровки BODIPY в смесях метанола/глицерина):
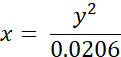
где x - вязкость (в сП), y - время жизни флуоресценции τ (в нс).
ПРИМЕЧАНИЕ: IRF (функция отклика прибора) является важной частью фитинга FLIM. В SPCImage IRF автоматически рассчитывается по переднему краю кривых затухания флуоресценции. Между тем, IRF может быть записан с использованием нефлуоресцентного образца, например, керамики, или образца, который производит сигнал ГВГ (генерация второй гармоники), например, коллагена, кристаллов мочевины или сахара. Использование записанных IRF не рекомендуется, если в программном обеспечении есть возможность их расчета.
Результаты
Используя описанный здесь протокол, мы визуализировали метаболические кофакторы и микроскопическую вязкость мембран в живых культивируемых клетках с помощью FLIM. Измерения проводились на различных линиях раковых клеток - колоректальной карциноме ?...
Обсуждение
Этот протокол иллюстрирует возможности FLIM для многопараметрического, функционального и биофизического анализа раковых клеток. Сочетание оптической метаболической визуализации, основанной на эндогенной флуоресценции, и измерения вязкости плазматической мембраны ...
Раскрытие информации
Авторам нечего раскрывать.
Благодарности
Разработка протокола метаболической визуализации выполнена при поддержке Министерства здравоохранения Российской Федерации (Государственное задание, регистрационный No АААА-А20-120022590098-0). Исследование вязкости выполнено при поддержке Российского научного фонда (проект No 20-14-00111). Авторы выражают благодарность Антону Плеханову (ПРМУ) за помощь в создании видео.
Материалы
| Name | Company | Catalog Number | Comments |
| Item/Device | |||
| Cell culture incubator | Sanyo | 37°C, 5% CO2, humidified atmosphere | |
| Centrifuge 5702 R | Eppendorf | 5703000010 | |
| imageJ 1.53c | Wayne Rasband (NIH) | ||
| FLIM module Simple Tau 152 TCSPC (in LSM 880) | Becker & Hickl GmbH | ||
| Laminar flow hood | ThermoFisher Scientific | ||
| Leica microscope DFC290 | Leica Microsystems | ||
| LSM 880 confocal microscope | Carl Zeiss | ||
| Ti:Sapphire femtosecond laser Mai Tai | Spectra Physics | ||
| Microscope incubator XLmulti S DARK LS | PeCon GmbH | 273-800 050 | |
| Mechanical pipettor | Sartorius mLINE | volume 0.5-10 μL; 20-200 μL; 100-1000 μL | |
| Oil-immersion objective C-Apochromat 40×/1.2 NA W Korr (in LSM 880) | Carl Zeiss | 421767-9971-790 | |
| Power-Tau 152 module with the detector HPM-100-40 | Becker&Hickl GmbH | ||
| SPCImage software | Becker & Hickl GmbH | SPC 9.8; SPCImage 8.3 | |
| ZEN software | Carl Zeiss | ZEN 2.1 SP3 (black), Version 14.0.0.201 | |
| Reagent/Material | |||
| 5-fluorouracil | Medac GmbH | 3728044 | |
| DMEM | Gibco, Life Technologies | 31885023 | |
| DMSO | PanEco | F135 | |
| FBS | Hyclone | A3160801 | |
| FluoroBright DMEM | Gibco, Life Technologies | A1896701 | |
| Hank’s solution without Ca2+/Mg2+ | Gibco, Life Technologies | 14175 | |
| l-Glutamine | PanEco | F032 | |
| Mammalian cells | HCT116, CT26, HeLa Kyoto, huFB | ||
| Molecular rotor BODIPY 2 | Synthesized and Supplied by Marina Kuimova Group, Imperial College London | ||
| Penicillin/streptomycin | PanEco | A065 | |
| Tissue culture dish with cover glass-bottom FluoroDishes | World Precision Instruments, Inc | ||
| Trypsin- EDTA 0.25% | PanEco | P034 | |
| Versen buffer | PanEco | R080p |
Ссылки
- Vazquez, A., et al. Cancer metabolism at a glance. Journal of Cell Science. 129 (18), 3367-3373 (2016).
- Li, Z., Zhang, H. Reprogramming of glucose, fatty acid, and amino acid metabolism for cancer progression. Cellular and Molecular Life Sciences. 73 (2), 377-392 (2015).
- Strickaert, A., et al. Cancer heterogeneity is not compatible with one unique cancer cell metabolic map. Oncogene. 36 (19), 2637-2642 (2016).
- Szlasa, W., Zendran, I., Zalesińska, A., Tarek, M., Kulbacka, J. Lipid composition of the cancer cell membrane. Journal of Bioenergetics and Biomembranes. 52, 321-342 (2020).
- Preta, G. New insights into targeting membrane lipids for cancer therapy. Frontiers in Cell and Developmental Biology. 8, 571237 (2020).
- Kojima, K. Molecular aspects of the plasma membrane in tumor cells. Nagoya Journal of Medical Science. 56, 1-18 (1993).
- Datta, R., Heaster, T. M., Sharick, J. T., Gillette, A. A., Skala, M. C. Fluorescence lifetime imaging microscopy: fundamentals and advances in instrumentation, analysis, and applications. Journal of Biomedical Optics. 25 (7), 071203 (2020).
- Becker, W. Advanced time-correlated single photon counting applications. Springer Series in Chemical Physics. , (2015).
- Shirmanova, M. V., Shcheslavskiy, V. I., Lukina, M. M., Becker, W., Zagaynova, E. V. Exploring tumor metabolism with time-resolved fluorescence methods: From single cells to a whole tumor. Multimodal Optical Diagnostics of Cancer. 3, 133-155 (2020).
- Kalinina, S., Rück, A. FLIM and PLIM in biomedical research. An innovative way to combine autofluorescence and oxygen measurements. Photonics & Lasers in Medicine. 5 (4), 257-266 (2016).
- Kuimova, M. K. Molecular rotors image intracellular viscosity. Chimia. 66 (4), 159-165 (2012).
- Kuimova, M. K. Mapping viscosity in cells using molecular rotors. Physical Chemistry Chemical Physics. 14 (37), 12671 (2012).
- Haidekker, M. A., Theodorakis, E. A. Molecular rotors-fluorescent biosensors for viscosity and flow. Organic & Biomolecular Chemistry. 5, 1669-1678 (2007).
- Liu, X., et al. Molecular mechanism of viscosity sensitivity in BODIPY rotors and application to motion-based fluorescent sensors. ACS Sensors. 5 (3), 731-739 (2020).
- Shirmanova, M. V., Shimolina, L. E., Lukina, M. M., Zagaynova, E. V., Kuimova, M. K., Dmitriev, R. Live cell imaging of viscosity in 3D tumour cell models. Multi-Parametric Live Cell Microscopy of 3D Tissue Models. Advances in Experimental Medicine and Biology. 1035, 143-153 (2017).
- Shimolina, L. E., et al. Imaging tumor microscopic viscosity in vivo using molecular rotors. Scientific Reports. 7, 41097 (2017).
- Scott, T. G., Spencer, R. D., Leonard, N. J., Weber, G. Synthetic spectroscopic models related to coenzymes and base pairs. V. Emission properties of NADH. Studies of fluorescence lifetimes and quantum efficiencies of NADH, AcPyADH, [reduced acetylpyridineadenine dinucleotide] and simplified synthetic models. Journal of the American Chemical Society. 92 (3), 687-695 (1970).
- Harvey, R. A., Damle, S. A fluorescent modification of flavin adenine dinucleotide. FEBS Letters. 26 (1-2), 341-343 (1972).
- Kubánková, M., Summers, P., López-Duarte, I., Kiryushko, D., Kuimova, M. K. Microscopic viscosity of neuronal plasma membranes measured using fluorescent molecular rotors: Effects of oxidative stress and neuroprotection. ACS Applied Materials and Interfaces. 11, 36307-36315 (2019).
- Kubánková, M., López-Duarte, I., Kiryushko, D., Kuimova, M. K. Molecular rotors report on changes in live cell plasma membrane microviscosity upon interaction with beta-amyloid aggregates. Soft Matter. 14, 9466-9474 (2018).
- Wu, Y., et al. Molecular rheometry: Direct determination of viscosity in Lo and Ld lipid phases via fluorescence lifetime imaging. Physical Chemistry Chemical Physics. 15 (36), 14986 (2013).
- Shimolina, L. E., et al. Mapping cisplatin-induced viscosity alterations in cancer cells using molecular rotor and fluorescence lifetime imaging microscopy. Journal of Biomedical Optics. 25 (12), 126004 (2020).
- Berezin, M. Y., Achilefu, S. Fluorescence lifetime measurements and biological imaging. Chemical Reviews. 110 (5), 2641-2684 (2010).
- Skala, M. C., et al. In vivo multiphoton fluorescence lifetime imaging of protein-bound and free nicotinamide adenine dinucleotide in normal and precancerous epithelia. Journal of Biomedical Optics. 12 (2), 024014 (2007).
- Rück, A., Hauser, C., Mosch, S., Kalinina, S. Spectrally resolved fluorescence lifetime imaging to investigate cell metabolism in malignant and nonmalignant oral mucosa cells. Journal of Biomedical Optics. 19 (9), 096005 (2014).
- Lee, D. -. H., Li, X., Ma, N., Digman, M. A., Lee, A. P. Rapid and label-free identification of single leukemia cells from blood in a high-density microfluidic trapping array by fluorescence lifetime imaging microscopy. Lab on a Chip. 18 (9), 1349-1358 (2018).
- Lukina, M. M., et al. Interrogation of tumor metabolism in tissue samples ex vivo using fluorescence lifetime imaging of NAD(P)H. Methods and Applications in Fluorescence. 8 (1), 014002 (2019).
- Alam, S. R., et al. Investigation of mitochondrial metabolic response to doxorubicin in prostate cancer cells: An NADH, FAD and Tryptophan FLIM assay. Scientific Reports. 7 (1), 10451 (2017).
- Walsh, A. J., et al. Quantitative optical imaging of primary tumor organoid metabolism predicts drug response in breast cancer. Cancer Research. 74 (18), 5184-5194 (2014).
- Lukina, M. M., et al. In vivo metabolic and SHG imaging for monitoring of tumor response to chemotherapy. Cytometry Part A. 95 (1), 47-55 (2019).
- Shah, A. T., et al. Optical metabolic imaging of treatment response in human head and neck squamous cell carcinoma. PLoS One. 9 (3), 90746 (2014).
- Sergeeva, T. F., et al. Relationship between intracellular pH, metabolic co-factors, and caspase-3 activation in cancer cells during apoptosis. Biochimica et Biophysica Acta (BBA) - Molecular Cell Research. 1864 (3), 604-611 (2017).
- Shirmanova, M. V., et al. Chemotherapy with cisplatin: insights into intracellular pH and metabolic landscape of cancer cells in vitro and in vivo. Scientific Reports. 7, 8911 (2017).
- Lukina, M. M., et al. Metabolic cofactors NAD(P)H and FAD as potential indicators of cancer cell response to chemotherapy with paclitaxel. Biochimica et Biophysica Acta (BBA) - General Subjects. 1862 (8), 1693-1700 (2018).
- Druzhkova, I. N., et al. The metabolic interaction of cancer cells and fibroblasts - coupling between NAD(P)H and FAD, intracellular pH and hydrogen peroxide. Cell Cycle. 15 (9), 1257-1266 (2016).
- Lakowicz, J., Szmacinski, H., Nowaczyk, K., Johnson, M. Fluorescence lifetime imaging of free and protein bound NADH. Proceedings of the National Academy of Sciences of the United States of America. 89 (4), 1271-1275 (1992).
- Vanden Berg, P. A. W., Feenstra, K. A., Mark, A. E., Berendsen, H. J. C., Visser, A. J. W. G. Dynamic conformations of flavin adenine dinucleotide: Simulated molecular dynamics of the flavin cofactor related to the time-resolved fluorescence characteristics. The Journal of Physical Chemistry B. 106 (34), 8858-8869 (2002).
- Liu, Z., et al. Mapping metabolic changes by noninvasive, multiparametric, high-resolution imaging using endogenous contrast. Science Advances. 4 (3), (2018).
- Blacker, T. S., et al. Separating NADH and NADPH fluorescence in live cells and tissues using FLIM. Nature Communications. 5, 3936 (2014).
- Lu, W., Wang, L., Chen, L., Hui, S., Rabinowitz, J. D. Extraction and quantitation of nicotinamide adenine dinucleotide redox cofactors. Antioxidants and Redox Signaling. 28 (3), 167-179 (2018).
- Ranjit, S., Malacrida, L., Jameson, D. M., Gratton, E. Fit-free analysis of fluorescence lifetime imaging data using the phasor approach. Nature Protocols. 13 (9), 1979-2004 (2018).
- Stringari, C., et al. Phasor approach to fluorescence lifetime microscopy distinguishes different metabolic states of germ cells in a live tissue. Proceedings of the National Academy of Sciences of the United States of America. 108 (33), 13582-13587 (2011).
- Smith, J. T., et al. Fast fit-free analysis of fluorescence lifetime imaging via deep learning. Proceedings of the National Academy of Sciences of the United States of America. 116 (48), 24019-24030 (2019).
- Wang, S., Chacko, J. V., Sagar, A. K., Eliceiri, K. W., Yuan, M. Nonparametric empirical Bayesian framework for fluorescence-lifetime imaging microscopy. Biomedical Optics Express. 10 (11), 5497-5517 (2019).
- Quinn, K. P., et al. Quantitative metabolic imaging using endogenous fluorescence to detect stem cell differentiation. Scientific Reports. 3 (1), 3432 (2013).
- Hou, J., et al. Correlating two-photon excited fluorescence imaging of breast cancer cellular redox state with seahorse flux analysis of normalized cellular oxygen consumption. Journal of Biomedical Optics. 21 (6), 060503 (2016).
- Wang, H., et al. Red-emitting fluorescence probe for sensing viscosity in living cells. Chemical Papers. 74, 1071-1078 (2020).
- Chen, B., et al. Sensing and imaging of mitochondrial viscosity in living cells by a red photoluminescent probe with long lifetime. Chemical Communications. 55, 7410 (2019).
- Shen, B., Wang, L. F., Zhi, X., Qian, Y. Construction of a red emission BODIPY-based probe for tracing lysosomal viscosity changes in culture cells. Sensors and Actuators B: Chemical. 304, 127271 (2019).
- Chen, T., Chen, Z., Liu, R., Zheng, S. NIR fluorescent probe for detection of viscosity and lysosome imaging in live cells. Organic and Biomolecular Chemistry. 17, 6398 (2019).
- Angelucci, C., et al. Epithelial-stromal interactions in human breast cancer: effects on adhesion, plasma membrane fluidity and migration speed and directness. PLoS One. 7 (12), 50804 (2012).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены