需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
使用荧光寿命成像显微镜探测癌细胞的代谢和粘度
摘要
在这里,我们展示了使用荧光寿命成像显微镜 (FLIM) 对活癌细胞培养物中的细胞代谢和质膜粘度进行序贯成像。通过检测内源性荧光进行代谢评估。使用荧光分子转子测量粘度。
摘要
粘度是生物膜的重要物理性质,因为它是调节活细胞形态和生理状态的关键参数之一。已知肿瘤细胞的质膜在其组成、结构和功能特性方面发生显着变化。除了葡萄糖和脂质代谢失调外,这些特定的膜特性有助于肿瘤细胞适应恶劣的微环境并对药物治疗产生耐药性。在这里,我们展示了使用荧光寿命成像显微镜 (FLIM) 对活癌细胞培养物中的细胞代谢和质膜粘度进行序贯成像。通过检测内源性代谢辅助因子的荧光进行代谢评估,例如还原的烟酰胺腺嘌呤二核苷酸 NAD(P)H 和氧化黄素。使用荧光分子转子(一种合成粘度敏感染料)测量粘度,其荧光寿命对周围环境的粘度有很强的依赖性。结合这些技术,使我们能够更好地了解癌细胞的膜状态和代谢特征之间的联系,并可视化化疗诱导的变化。
引言
细胞的恶性转化伴随着其形态和生理状态的多种改变。癌细胞的快速和不受控制的生长需要对负责能量产生和生物合成的生化途径进行根本性的重组。癌症代谢的特征标志是糖酵解速率提高,即使在正常氧浓度(Warburg 效应)下,使用氨基酸、脂肪酸和乳酸作为替代燃料,在高抗氧化剂水平存在下产生高 ROS,以及增加脂肪酸的生物合成 1,2.现在人们认识到,癌细胞代谢具有高度的灵活性,这使它们能够适应不利和异质的环境,并提供额外的生存优势3。
代谢改变支持肿瘤细胞膜的特异性组织和组成。癌细胞质膜的脂质谱在数量上与非癌细胞不同。脂质组的主要变化是磷脂水平升高,包括磷脂酰肌醇、磷脂酰丝氨酸、磷脂酰乙醇胺和磷脂酰胆碱,鞘磷脂水平降低,胆固醇含量增加,脂肪酸不饱和度降低,仅举几例 4,5,6.因此,膜的物理性质,如膜粘度,即流动性的倒数,不可避免地会发生变化。由于粘度决定了生物膜的通透性并控制膜相关蛋白(酶、转运蛋白、受体)的活性,因此其稳态调节对细胞功能至关重要。同时,通过调整膜脂质谱来改变粘度对于细胞迁移/侵袭和条件变化后的存活很重要。
荧光寿命成像显微镜 (FLIM) 已成为一种使用内源性荧光或外源性探针对活细胞中多个参数进行无创评估的强大方法7。FLIM 通常在多光子激光扫描显微镜上实现,该显微镜提供(亚)细胞分辨率。它配备了时间相关单光子计数 (TCSPC) 模块,能够以高精度进行时间分辨荧光测量8。
FLIM 对细胞代谢的探测基于脱氢酶内源性辅因子的荧光测量,即还原的烟酰胺腺嘌呤二核苷酸(磷酸盐)NAD(P)H 和氧化黄素 - 黄素腺嘌呤二核苷酸 FAD 和黄素单核苷酸 FMN,它们在许多生化反应中充当电子载体 7,9,10.检测到的 NAD(P)H 荧光来自 NADH 及其磷酸化形式 NADPH,因为它们在光谱上几乎相同。通常,NAD(P)H 和黄素的荧光衰减适合双指数函数。在 NAD(P)H 的情况下,第一组分 (~0.3-0.5 ns, ~70%-80%) 归因于其游离状态,与糖酵解有关,第二组分 (~1.2-2.5 ns, ~20%-30%) 归因于其蛋白质结合状态,与线粒体呼吸有关。在黄素的情况下,短组分 (~0.3-0.4 ns, ~75%-85%) 可以分配给 FAD 的淬灭状态,而长组分 (~2.5-2.8 ns, ~15%-25%) 可以分配给未淬灭的 FAD、FMN 和核黄素。糖酵解、谷氨酰胺分解、氧化磷酸化和脂肪酸合成相对水平的改变导致辅因子的短寿命和长寿命分数发生变化。此外,这些荧光团的荧光强度比值(氧化还原比值)反映了细胞氧化还原状态,也用作代谢指标。虽然氧化还原比是一个更简单的指标,但与荧光寿命相比,就数据采集而言,FLIM 有利于估计 NAD(P)H 和 FAD,因为荧光寿命是荧光团的固有特性,几乎不受激发功率、光漂白、聚焦、光散射和吸收等因素的影响,尤其是在组织中, 与发射强度不同。
在微观水平上绘制活细胞和组织粘度的便捷方法之一是使用荧光分子转子,这是一种小型合成粘度敏感染料,其中荧光参数强烈取决于局部粘度11,12。在粘性介质中,由于分子内扭曲或旋转的减慢,转子的荧光寿命增加。在分子转子中,硼二吡咯甲烯 (BODIPY) 的衍生物非常适合检测生物系统中的粘度,因为它们在生理粘度范围内具有良好的荧光寿命动态范围、温度独立性、允许直接数据解释的单指数荧光衰减、足够的水溶性和低细胞毒性13,14.先前已在体外癌细胞、多细胞肿瘤球体和小鼠肿瘤上使用基于 BODIPY 的转子和 FLIM 对微粘度进行定量评估 15,16。
在这里,我们详细描述了通过 FLIM 在体外 研究癌细胞中细胞代谢和质膜粘度的顺序探测方法。为了避免相对较弱的内源性荧光被基于 BODIPY 的转子的荧光污染,依次进行同一层细胞的成像,首先对 NAD(P)H 和 FAD 的荧光进行成像。在细胞质中测量辅因子的荧光寿命,通过手动选择相应的区域作为感兴趣区域,在细胞质膜中测量转子的荧光寿命。该方案用于关联不同癌细胞系的代谢状态和粘度,并评估化疗后的变化。
FLIM 样品制备的方案与共聚焦荧光显微镜的方案没有区别。采集数据后,主要任务是从原始数据中提取荧光寿命。使用 HCT116 (人结直肠癌)、CT26 (小鼠结肠癌)、HeLa (人宫颈癌)和 huFB (人皮肤成纤维细胞) 细胞证明了该方案的性能。
Access restricted. Please log in or start a trial to view this content.
研究方案
1. 执行 FLIM 的最小设置说明
- 要进行此实验,请确保所需的设置可用:倒置共聚焦显微镜、脉冲激光器(通常为 ps 或 fs),带有同步信号、快速光子计数检测器(时间响应 150 ps)和光子计数电子设备、检测器和激光器的可用输出和输入端口,分别在显微镜上,来自显微镜扫描控制器的扫描时钟脉冲, 显微镜的扫描头带有激光合束器和适用于用于 FLIM 的激光波长的主二向色分束器。
- 如果双光子激发用于 FLIM,请确保显微镜包含 NDD 端口。
- 对于哺乳动物细胞研究,尤其是长期实验,请确保将 CO2 培养箱保持在所需温度。
注意:有关本实验中使用的系统,请参阅 材料表。
2. 制备用于显微镜的细胞
- 在 37 °C、5% CO2 和潮湿气氛的培养箱中常规培养细胞。
- 对于显微镜检查,在完全培养基中制备浓度为 1 x 106 个细胞/mL 的细胞悬液。
注:细胞浓度和培养基条件取决于细胞。应调整用于接种的细胞数和孵育时间,以便在显微培养皿中获得 70%-80% 的汇合度。 - 使用 200 μL 自动移液器将细胞接种在玻璃底 35 mm 细胞培养皿(每皿 100 μL 中 1 x 105 个细胞)。
注:使用网格玻璃底培养皿进行细胞接种,以在动态中监测相同微观视野中的细胞。- 在手动接种过程中,确保移液器吸头不会刮伤培养皿的底部或侧面,以免损坏底部。
- 将培养皿置于 CO2 培养箱(37 °C,5% CO2,潮湿气氛)中,并将细胞孵育 24 小时。
- 24 小时后,从培养箱中取出培养皿,并在光学显微镜下检查细胞的形态和汇合度。如果细胞未达到约 80% 汇合度,则再孵育 24 小时。
- 使用 1,000 μL 自动移液器从培养皿中轻轻去除旧培养基,并加入 2 mL 不含酚红的 DMEM 培养基(例如 DMEM Life 或 FluoroBrite)。
注:不同的培养基可用于成像。使用细胞进行显微镜检查时,避免培养基中的酚红。 - 将培养皿放入培养箱中 60-120 分钟,以使细胞适应。
3. 代谢辅助因子的 FLIM
- 将装有细胞的玻璃底培养皿(来自步骤 2.7)放在显微镜载物台上。
- 单击激光扫描显微镜软件(例如 ZEN - ZEISS Efficient Navigation)中的 “定位 ”选项卡,然后单击 “透射光 (TL) ”以打开灯。
- 通过目镜观察细胞的中心切片水平,找到样品的焦平面,其中正方形最多被细胞占据(放大 40 倍)。
- 单击 OFF 按钮关闭灯。
- 打开 Acquisition 选项卡。要获得内源性 NAD(P)H 的透射和自发荧光强度图像,请输入以下设置: 激发波长:双光子模式 750 nm, 配准范围:450-490 nm, 激光功率:5% (~6 mW), 图像尺寸:1024 x 1024 像素。
注意:激发波长和配准范围的选择基于 NAD(P)H17 的光谱特性(最大激发和最大发射)。- 使用油浸物镜 С Plan-Apochromat 40x/1.3 NA 进行图像采集。
- 按 Snap 按钮并以 ZEN 格式保存图像。
- 要获得 FAD 的透射和自发荧光强度图像,请将 激发波长 更改为 900 nm。设置 套准范围:500-550 nm, 激光功率:9% (~6 mW), 图像大小:1024 x 1024 像素。
注:激发波长和配准范围的选择基于 FAD18 的光谱特性(最大激发和最大发射)。- 使用油浸物镜 С Plan-Apochromat 40x/1.3 NA 进行图像采集。
- 按 Snap 按钮并以 ZEN 格式保存图像。
- 对于 NAD(P)H,请按照激光扫描显微镜软件中步骤 3.5 中的说明设置参数。更改 图像大小 至 256 x 256 像素。
- 在 SPCM(单光子计数模块)菜单下输入以下参数 FLIM 模块作软件: 采集时间:60 s; TAC 范围:5.00E-8; 差价合约限价低点:-29.41; ADC 分辨率:256, 图像尺寸:256 x 256 像素。
- 扫描样品 60 秒,停止扫描并保存获得的 NAD(P)H FLIM 图像。
- 检查获取的 FLIM 数据。为此,在图像软件 中打开 原始数据,通过将光标放在细胞质上来选择细胞细胞质中的一个像素,并分析该像素中的荧光衰减。像素强度应为像素合并 1 时每条衰变曲线 ≥3,000 个光子。
注意:如果光子数低于 3,000,则增加激光功率或图像收集时间,同时控制细胞的形态和光子计数速率。通常,如果计数率下降超过初始值的 10%,则会发生光漂白。 - 对于 FAD,请按照激光扫描显微镜软件中步骤 3.7 中的说明设置参数。将图像大小更改为 256 x 256 像素。
- 在 SPCM(单光子计数模块)菜单下输入以下参数 FLIM 模块作软件: 采集时间:60 s; TAC 范围:5.00E-8; 差价合约限价低点:-29.41; ADC 分辨率:256, 图像尺寸:256 x 256 像素。
- 扫描样品 60 秒。停止扫描并保存获取的 FAD 的 FLIM 镜像。
注意:步骤 3.10 和 3.15 中指示的参数特定于所使用的电子设备和探测器。 - 按照步骤 3.12 中的说明检查获得的数据。
- 重复步骤 3.5-3.16 以记录来自不同视野的 FLIM 图像。
4. 用荧光分子转子对细胞进行染色
注:在荧光分子转子溶液中对细胞进行成像,无需在室温 (~20 °C) 下洗涤,以减慢转子的内化。膜粘度与温度有关,正如我们之前的工作19,20 所示。显微镜的温控载物台应提前关闭,即在将转子添加到细胞之前。对于我们的设置,舞台的冷却大约需要 10 分钟。
- 制备荧光分子转子 BODIPY 2 的一般储备溶液(储备液 1,25.7 mM)。
- 在无菌环境中打开 BODIPY 2,使用准确天平称量约 2 mg。小心地将其放入微量离心管中。
- 使用 20 μL 自动移液器添加 3 μL 合适的溶剂(例如 DMSO)。
- 一旦样品完全溶解在 DMSO 中,加入 297 μL 无菌 PBS,并使用 200 μL 自动移液器充分混合。
注:将储备溶液存放在 +4 °C 的冰箱中,采用深色包装。一旦重悬,它可以在冰箱中存放几个月。
- 向微量离心管中加入 25 μL 通用储备液(储备液 1),然后加入 48 μL 无菌 PBS,制备储备液 2 (8.9 mM)。使用 200 μL 自动移液器轻轻混匀。
注:使用原液 2 制备最终染色原液,由于需要微摩尔浓度,因此用于细胞染色。 - 用不含 Ca2+/Mg2+ 的冰冷 Hank 溶液轻轻更换培养皿中的培养基(来自步骤 3.1),并在 +4 °C 下孵育细胞 3 分钟。
注意:使用冰冷溶液并在 +4 °C 下孵育会减慢分子转子的内化,并且膜的局部染色持续 20-30 分钟。 - 通过将 1 μL 原液 2 添加到 999 μL 冰冷的 Hank 溶液或 PBS 中,制备含有 4.5 μM BODIPY 2 的最终染色溶液。
注:最终染色溶液中 BODIPY 2 的浓度可增加至 ~10 μM,对细胞无任何毒性影响,从而导致更有效的染色和更多的光子收集。在较高浓度下,FLIM 检测器可能会过载。 - 从细胞培养皿中吸出 Hank's 溶液,并用冰冷的 4.5 μM BODIPY 2 溶液代替。将细胞在 BODIPY 2 溶液中成像而不洗涤。
5. 细胞中荧光分子转子的 FLIM
注意:在代谢辅因子的 FLIM 之后,始终对荧光分子转子进行 FLIM,因为 BODIPY 2 的荧光光谱与内源性辅因子 NAD(Р)H 和 FAD 12,17,18 的发射重叠。
- 将带有染色细胞的培养皿转移到显微镜载物台 (~20 °C) 进行成像。
- 在激光扫描显微镜的软件中为单光子模式设置以下参数:用氩离子激光器在 488 nm 波长处 激发 , 激光功率 1%-2%, 配准范围 500-550 nm 波长。
- 使用油浸物镜 С Plan-Apochromat 40x/1.3 NA 进行图像采集。
- 按下 Live 按钮。通过集成电动载物台开始扫描并使用 XY 和 Z 定位,调整焦点并在预览窗口中获得细胞的透射和荧光强度图像。如果需要, 保存 获取的图像。
- 检查重叠的透射和荧光图像,查看转子的荧光是否来自预期位置(细胞质膜)。
- 在 FLIM 模块的 SPCM 软件菜单中输入以下参数: 采集时间:60 s; TAC 范围:5.00E-8; 差价合约限价低点:-29.41; ADC 分辨率:256, 图像尺寸:256 x 256 像素。
注意:根据用于 FLIM 的系统配置和探测器,图像采集的参数可能会有所不同。 - 将显微镜的Ti:Sapphire激光器调整为850 nm的 波长 ,将双光子FLIM的 激光功率 调整为1%-2%。
- 在激光扫描显微镜软件中选择 Continuous 选项卡,然后在 SPCM 软件中按 Start 。扫描样品 60 秒,停止扫描,并保存获取的 FLIM 图像。
- 检查获取的 FLIM 数据。为此,将原始数据加载到 FLIM 数据分析 SPCImage 软件中,通过将光标放在细胞膜上来选择细胞膜中的一个像素,然后分析该像素中的荧光衰减。在合理的收集时间 (60-120 s) 下,每次衰减(可能包括合并)的像素强度应为 ≥5,000。
- 重复步骤 5.4-5.8 以记录来自不同视野的细胞的 FLIM 图像。
注:用 BODIPY 2 染色的活细胞的 FLIM 测量应限制在添加 BODIPY 2 后 ~30 分钟。
6. 数据分析
- 荧光强度分析:氧化还原比
- 使用 ImageJ 打开 NAD(P)H 和 FAD 荧光强度的图像。
- 使用圆形或方形选项突出显示 NAD(P)H 图像中的无单元格区域。单击 Measure,然后单击 Subtract(在主面板上选择 Process ,然后选择 Math 和 Subtract)以减去获得的背景信号值。
- 对 FAD 映像重复步骤 6.1.2。
- 通过将 FAD 荧光强度除以 NAD(P)H 荧光强度来获得氧化还原比率的图像。通过选择 处理 在主面板上,然后选择 图像计算器 和 除法;选中 创建新窗口 框,然后按 确定.
- 以 TIFF 格式存储图像。
- 要计算氧化还原比率,请在 TIFF 图像上选择特定细胞中的细胞质区域,然后按 M 键。对所有感兴趣的细胞重复此作。
- 将测量导入电子表格文档。
注意:或者,可以使用显微镜的标准软件测量细胞中 NAD(P)H 和 FAD 的荧光强度,并且可以通过在电子表格软件中除以这些值来获得氧化还原比率。
- FLIM 数据分析:代谢
- 将 NAD(P)H 的 FLIM 图像导入 SPCImage 软件。
- 通过在 Components (组件) 部分中放置 2 来对图像应用双指数衰减拟合。
- 通过选中 SPCImage 软件中的相应框来修复 Offset 参数。
- 转到 选项 并选择 模型。使用 不完全多指数 拟合模型和 Fit 方法 MLE。
- 调整像素合并以实现每条衰减曲线 ≥5000 个光子的像素强度。
- 检查 χ2 值。χ2≤ 1.20 表示所使用的模型提供了合理的拟合。
- 通过单击顶部菜单 Calculate 和 Decay Matrix 来计算每个图像中荧光寿命的直方图。
- 选择特定细胞的细胞质中的区域作为感兴趣的区域。
- 使用 Color 选项分析短寿命分量和长寿命分量(分别为 τ1 和 τ2)以及寿命分量的相对振幅(a1 和a 2,其中a 1 +a 2 = 100%)。
- 将测量值导出到电子表格软件。
- 对每个感兴趣的细胞重复步骤 6.2.8-6.2.10。
- 对 FAD 图像重复步骤 6.2.1-6.2.11。
- FLIM 数据分析:粘度
- 将 FLIM 图像导入 FLIM 数据分析 SPCImage 软件。
- 删除 Scatter 框中的标记。
- 在 Components 部分输入 1,因为转子荧光衰减应该适合单指数模型。
- 调整像素合并以实现每个衰减曲线 ≥5000 个光子的像素强度。
- 检查质膜中的 χ2 值。值 χ2≤ 1.20 表示所使用的模型提供了合理的拟合。在 χ2 的情况下≥ 1.20,单指数近似不适用,此类数据可以指示染料聚集,应丢弃。聚合使得无法在不正确的粘度估计中使用校准曲线和导联。
注意:双指数衰减可能表示聚集。在带有可变滤光片的 FLIM 模块的显微镜上,可以通过测试单体和聚集体特异性发射波长范围 500-550 nm 和 580-650 nm 来检测,如参考文献21 所述。 - 通过单击顶部菜单 Calculate 生成每个图像的荧光寿命 τ 的直方图,然后单击 Decay Matrix。
- 使用 ROI 选项选择具有单指数衰变的单个细胞的质膜区域 χ2≤ 1.20。
- 将荧光寿命的值导出到电子表格软件中。
- 对每个感兴趣的细胞重复步骤 6.3.7-6.3.8。
- 使用以下公式(先前根据 BODIPY 在甲醇/甘油混合物中的校准图获得)将 BODIPY 2 的实验测量寿命(以 ns 为单位)转换为粘度值(以 cP 为单位):
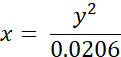
其中 x - 粘度(以 cP 为单位),y - 荧光寿命 τ(以 ns 为单位)。
注:IRF(仪器响应函数)是 FLIM 验配的重要组成部分。在 SPCImage 中,IRF 是根据荧光衰减曲线的上升沿自动计算的。同时,可以使用非荧光样品(例如陶瓷)或产生 SHG(二次谐波产生)信号的样品(例如胶原蛋白、尿素晶体或糖)来记录 IRF。如果软件中有计算记录的 IRF 的选项,则不建议使用记录的 IRF。
Access restricted. Please log in or start a trial to view this content.
结果
使用此处描述的方案,我们使用 FLIM 可视化了活培养细胞中的代谢辅因子和微观膜粘度。已在不同的癌细胞系中进行了测量 - 人结直肠癌 HCT116、小鼠结肠癌 CT26、人宫颈癌 HeLa Kyoto 和人皮肤成纤维细胞 huFB。
基于荧光强度的氧化还原比值 FAD/NAD(P)H 以及 NAD(P)H 和 FAD 的荧光寿命允许评估细胞代谢状...
Access restricted. Please log in or start a trial to view this content.
讨论
该方案说明了 FLIM 用于癌细胞多参数、功能和生物物理分析的可能性。基于内源性荧光的光学代谢成像与使用荧光分子转子的外源性标记测量质膜粘度的结合使我们能够表征细胞培养物中活癌细胞中这两个参数之间的相互联系,并跟踪响应化疗的变化。
双光子激发荧光寿命成像显微镜 (FLIM) 是一种很有前途的技术,用于对活细胞和组织的功能状态?...
Access restricted. Please log in or start a trial to view this content.
披露声明
作者没有什么可披露的。
致谢
代谢成像方案的开发得到了俄罗斯联邦卫生部的支持(政府分配,注册号:АААА-А20-120022590098-0)。粘度的研究得到了俄罗斯科学基金会的支持(项目编号 20-14-00111)。作者感谢 Anton Plekhanov (PRMU) 在视频制作方面的帮助。
Access restricted. Please log in or start a trial to view this content.
材料
| Name | Company | Catalog Number | Comments |
| Item/Device | |||
| Cell culture incubator | Sanyo | 37°C, 5% CO2, humidified atmosphere | |
| Centrifuge 5702 R | Eppendorf | 5703000010 | |
| imageJ 1.53c | Wayne Rasband (NIH) | ||
| FLIM module Simple Tau 152 TCSPC (in LSM 880) | Becker & Hickl GmbH | ||
| Laminar flow hood | ThermoFisher Scientific | ||
| Leica microscope DFC290 | Leica Microsystems | ||
| LSM 880 confocal microscope | Carl Zeiss | ||
| Ti:Sapphire femtosecond laser Mai Tai | Spectra Physics | ||
| Microscope incubator XLmulti S DARK LS | PeCon GmbH | 273-800 050 | |
| Mechanical pipettor | Sartorius mLINE | volume 0.5-10 μL; 20-200 μL; 100-1000 μL | |
| Oil-immersion objective C-Apochromat 40×/1.2 NA W Korr (in LSM 880) | Carl Zeiss | 421767-9971-790 | |
| Power-Tau 152 module with the detector HPM-100-40 | Becker&Hickl GmbH | ||
| SPCImage software | Becker & Hickl GmbH | SPC 9.8; SPCImage 8.3 | |
| ZEN software | Carl Zeiss | ZEN 2.1 SP3 (black), Version 14.0.0.201 | |
| Reagent/Material | |||
| 5-fluorouracil | Medac GmbH | 3728044 | |
| DMEM | Gibco, Life Technologies | 31885023 | |
| DMSO | PanEco | F135 | |
| FBS | Hyclone | A3160801 | |
| FluoroBright DMEM | Gibco, Life Technologies | A1896701 | |
| Hank’s solution without Ca2+/Mg2+ | Gibco, Life Technologies | 14175 | |
| l-Glutamine | PanEco | F032 | |
| Mammalian cells | HCT116, CT26, HeLa Kyoto, huFB | ||
| Molecular rotor BODIPY 2 | Synthesized and Supplied by Marina Kuimova Group, Imperial College London | ||
| Penicillin/streptomycin | PanEco | A065 | |
| Tissue culture dish with cover glass-bottom FluoroDishes | World Precision Instruments, Inc | ||
| Trypsin- EDTA 0.25% | PanEco | P034 | |
| Versen buffer | PanEco | R080p |
参考文献
- Vazquez, A., et al. Cancer metabolism at a glance. Journal of Cell Science. 129 (18), 3367-3373 (2016).
- Li, Z., Zhang, H. Reprogramming of glucose, fatty acid, and amino acid metabolism for cancer progression. Cellular and Molecular Life Sciences. 73 (2), 377-392 (2015).
- Strickaert, A., et al. Cancer heterogeneity is not compatible with one unique cancer cell metabolic map. Oncogene. 36 (19), 2637-2642 (2016).
- Szlasa, W., Zendran, I., Zalesińska, A., Tarek, M., Kulbacka, J. Lipid composition of the cancer cell membrane. Journal of Bioenergetics and Biomembranes. 52, 321-342 (2020).
- Preta, G. New insights into targeting membrane lipids for cancer therapy. Frontiers in Cell and Developmental Biology. 8, 571237(2020).
- Kojima, K. Molecular aspects of the plasma membrane in tumor cells. Nagoya Journal of Medical Science. 56, 1-18 (1993).
- Datta, R., Heaster, T. M., Sharick, J. T., Gillette, A. A., Skala, M. C. Fluorescence lifetime imaging microscopy: fundamentals and advances in instrumentation, analysis, and applications. Journal of Biomedical Optics. 25 (7), 071203(2020).
- Becker, W. Advanced time-correlated single photon counting applications. Springer Series in Chemical Physics. , Springer. (2015).
- Shirmanova, M. V., Shcheslavskiy, V. I., Lukina, M. M., Becker, W., Zagaynova, E. V. Exploring tumor metabolism with time-resolved fluorescence methods: From single cells to a whole tumor. Multimodal Optical Diagnostics of Cancer. 3, 133-155 (2020).
- Kalinina, S., Rück, A. FLIM and PLIM in biomedical research. An innovative way to combine autofluorescence and oxygen measurements. Photonics & Lasers in Medicine. 5 (4), 257-266 (2016).
- Kuimova, M. K. Molecular rotors image intracellular viscosity. Chimia. 66 (4), 159-165 (2012).
- Kuimova, M. K. Mapping viscosity in cells using molecular rotors. Physical Chemistry Chemical Physics. 14 (37), 12671(2012).
- Haidekker, M. A., Theodorakis, E. A. Molecular rotors-fluorescent biosensors for viscosity and flow. Organic & Biomolecular Chemistry. 5, 1669-1678 (2007).
- Liu, X., et al. Molecular mechanism of viscosity sensitivity in BODIPY rotors and application to motion-based fluorescent sensors. ACS Sensors. 5 (3), 731-739 (2020).
- Shirmanova, M. V., Shimolina, L. E., Lukina, M. M., Zagaynova, E. V., Kuimova, M. K. Live cell imaging of viscosity in 3D tumour cell models. Multi-Parametric Live Cell Microscopy of 3D Tissue Models. Advances in Experimental Medicine and Biology. Dmitriev, R. 1035, 143-153 (2017).
- Shimolina, L. E., et al. Imaging tumor microscopic viscosity in vivo using molecular rotors. Scientific Reports. 7, 41097(2017).
- Scott, T. G., Spencer, R. D., Leonard, N. J., Weber, G. Synthetic spectroscopic models related to coenzymes and base pairs. V. Emission properties of NADH. Studies of fluorescence lifetimes and quantum efficiencies of NADH, AcPyADH, [reduced acetylpyridineadenine dinucleotide] and simplified synthetic models. Journal of the American Chemical Society. 92 (3), 687-695 (1970).
- Harvey, R. A., Damle, S. A fluorescent modification of flavin adenine dinucleotide. FEBS Letters. 26 (1-2), 341-343 (1972).
- Kubánková, M., Summers, P., López-Duarte, I., Kiryushko, D., Kuimova, M. K. Microscopic viscosity of neuronal plasma membranes measured using fluorescent molecular rotors: Effects of oxidative stress and neuroprotection. ACS Applied Materials and Interfaces. 11, 36307-36315 (2019).
- Kubánková, M., López-Duarte, I., Kiryushko, D., Kuimova, M. K. Molecular rotors report on changes in live cell plasma membrane microviscosity upon interaction with beta-amyloid aggregates. Soft Matter. 14, 9466-9474 (2018).
- Wu, Y., et al. Molecular rheometry: Direct determination of viscosity in Lo and Ld lipid phases via fluorescence lifetime imaging. Physical Chemistry Chemical Physics. 15 (36), 14986(2013).
- Shimolina, L. E., et al. Mapping cisplatin-induced viscosity alterations in cancer cells using molecular rotor and fluorescence lifetime imaging microscopy. Journal of Biomedical Optics. 25 (12), 126004(2020).
- Berezin, M. Y., Achilefu, S. Fluorescence lifetime measurements and biological imaging. Chemical Reviews. 110 (5), 2641-2684 (2010).
- Skala, M. C., et al. In vivo multiphoton fluorescence lifetime imaging of protein-bound and free nicotinamide adenine dinucleotide in normal and precancerous epithelia. Journal of Biomedical Optics. 12 (2), 024014(2007).
- Rück, A., Hauser, C., Mosch, S., Kalinina, S. Spectrally resolved fluorescence lifetime imaging to investigate cell metabolism in malignant and nonmalignant oral mucosa cells. Journal of Biomedical Optics. 19 (9), 096005(2014).
- Lee, D. -H., Li, X., Ma, N., Digman, M. A., Lee, A. P. Rapid and label-free identification of single leukemia cells from blood in a high-density microfluidic trapping array by fluorescence lifetime imaging microscopy. Lab on a Chip. 18 (9), 1349-1358 (2018).
- Lukina, M. M., et al. Interrogation of tumor metabolism in tissue samples ex vivo using fluorescence lifetime imaging of NAD(P)H. Methods and Applications in Fluorescence. 8 (1), 014002(2019).
- Alam, S. R., et al. Investigation of mitochondrial metabolic response to doxorubicin in prostate cancer cells: An NADH, FAD and Tryptophan FLIM assay. Scientific Reports. 7 (1), 10451(2017).
- Walsh, A. J., et al. Quantitative optical imaging of primary tumor organoid metabolism predicts drug response in breast cancer. Cancer Research. 74 (18), 5184-5194 (2014).
- Lukina, M. M., et al. In vivo metabolic and SHG imaging for monitoring of tumor response to chemotherapy. Cytometry Part A. 95 (1), 47-55 (2019).
- Shah, A. T., et al. Optical metabolic imaging of treatment response in human head and neck squamous cell carcinoma. PLoS One. 9 (3), 90746(2014).
- Sergeeva, T. F., et al. Relationship between intracellular pH, metabolic co-factors, and caspase-3 activation in cancer cells during apoptosis. Biochimica et Biophysica Acta (BBA) - Molecular Cell Research. 1864 (3), 604-611 (2017).
- Shirmanova, M. V., et al. Chemotherapy with cisplatin: insights into intracellular pH and metabolic landscape of cancer cells in vitro and in vivo. Scientific Reports. 7, 8911(2017).
- Lukina, M. M., et al. Metabolic cofactors NAD(P)H and FAD as potential indicators of cancer cell response to chemotherapy with paclitaxel. Biochimica et Biophysica Acta (BBA) - General Subjects. 1862 (8), 1693-1700 (2018).
- Druzhkova, I. N., et al. The metabolic interaction of cancer cells and fibroblasts - coupling between NAD(P)H and FAD, intracellular pH and hydrogen peroxide. Cell Cycle. 15 (9), 1257-1266 (2016).
- Lakowicz, J., Szmacinski, H., Nowaczyk, K., Johnson, M. Fluorescence lifetime imaging of free and protein bound NADH. Proceedings of the National Academy of Sciences of the United States of America. 89 (4), 1271-1275 (1992).
- Vanden Berg, P. A. W., Feenstra, K. A., Mark, A. E., Berendsen, H. J. C., Visser, A. J. W. G. Dynamic conformations of flavin adenine dinucleotide: Simulated molecular dynamics of the flavin cofactor related to the time-resolved fluorescence characteristics. The Journal of Physical Chemistry B. 106 (34), 8858-8869 (2002).
- Liu, Z., et al. Mapping metabolic changes by noninvasive, multiparametric, high-resolution imaging using endogenous contrast. Science Advances. 4 (3), (2018).
- Blacker, T. S., et al. Separating NADH and NADPH fluorescence in live cells and tissues using FLIM. Nature Communications. 5, 3936(2014).
- Lu, W., Wang, L., Chen, L., Hui, S., Rabinowitz, J. D. Extraction and quantitation of nicotinamide adenine dinucleotide redox cofactors. Antioxidants and Redox Signaling. 28 (3), 167-179 (2018).
- Ranjit, S., Malacrida, L., Jameson, D. M., Gratton, E. Fit-free analysis of fluorescence lifetime imaging data using the phasor approach. Nature Protocols. 13 (9), 1979-2004 (2018).
- Stringari, C., et al. Phasor approach to fluorescence lifetime microscopy distinguishes different metabolic states of germ cells in a live tissue. Proceedings of the National Academy of Sciences of the United States of America. 108 (33), 13582-13587 (2011).
- Smith, J. T., et al. Fast fit-free analysis of fluorescence lifetime imaging via deep learning. Proceedings of the National Academy of Sciences of the United States of America. 116 (48), 24019-24030 (2019).
- Wang, S., Chacko, J. V., Sagar, A. K., Eliceiri, K. W., Yuan, M. Nonparametric empirical Bayesian framework for fluorescence-lifetime imaging microscopy. Biomedical Optics Express. 10 (11), 5497-5517 (2019).
- Quinn, K. P., et al. Quantitative metabolic imaging using endogenous fluorescence to detect stem cell differentiation. Scientific Reports. 3 (1), 3432(2013).
- Hou, J., et al. Correlating two-photon excited fluorescence imaging of breast cancer cellular redox state with seahorse flux analysis of normalized cellular oxygen consumption. Journal of Biomedical Optics. 21 (6), 060503(2016).
- Wang, H., et al. Red-emitting fluorescence probe for sensing viscosity in living cells. Chemical Papers. 74, 1071-1078 (2020).
- Chen, B., et al. Sensing and imaging of mitochondrial viscosity in living cells by a red photoluminescent probe with long lifetime. Chemical Communications. 55, 7410(2019).
- Shen, B., Wang, L. F., Zhi, X., Qian, Y. Construction of a red emission BODIPY-based probe for tracing lysosomal viscosity changes in culture cells. Sensors and Actuators B: Chemical. 304, 127271(2019).
- Chen, T., Chen, Z., Liu, R., Zheng, S. NIR fluorescent probe for detection of viscosity and lysosome imaging in live cells. Organic and Biomolecular Chemistry. 17, 6398(2019).
- Angelucci, C., et al. Epithelial-stromal interactions in human breast cancer: effects on adhesion, plasma membrane fluidity and migration speed and directness. PLoS One. 7 (12), 50804(2012).
Access restricted. Please log in or start a trial to view this content.
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。