Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Sondeo del metabolismo y la viscosidad de las células cancerosas mediante microscopía de imágenes de fluorescencia de por vida
En este artículo
Resumen
Aquí, demostramos el uso de la microscopía de fluorescencia para obtener imágenes secuenciales del metabolismo celular y la viscosidad de la membrana plasmática en cultivos de células cancerosas vivas. Las evaluaciones metabólicas se realizan mediante la detección de fluorescencia endógena. La viscosidad se mide utilizando un rotor molecular fluorescente.
Resumen
La viscosidad es una propiedad física importante de una membrana biológica, ya que es uno de los parámetros clave para la regulación del estado morfológico y fisiológico de las células vivas. Se sabe que las membranas plasmáticas de las células tumorales tienen alteraciones significativas en su composición, estructura y características funcionales. Junto con el metabolismo desregulado de la glucosa y los lípidos, estas propiedades específicas de la membrana ayudan a las células tumorales a adaptarse al microambiente hostil y a desarrollar resistencia a las terapias farmacológicas. Aquí, demostramos el uso de la microscopía de fluorescencia para obtener imágenes secuenciales del metabolismo celular y la viscosidad de la membrana plasmática en cultivos de células cancerosas vivas. Las evaluaciones metabólicas se realizan mediante la detección de fluorescencia de cofactores metabólicos endógenos, como la nicotinamida, adenina, dinucleótido NAD(P)H reducidos y flavinas oxidadas. La viscosidad se mide utilizando un rotor molecular fluorescente, un colorante sintético sensible a la viscosidad, con una fuerte fluorescencia que depende de la viscosidad del entorno inmediato. En combinación, estas técnicas nos permiten comprender mejor los vínculos entre el estado de la membrana y el perfil metabólico de las células cancerosas y visualizar los cambios inducidos por la quimioterapia.
Introducción
La transformación maligna de las células se acompaña de múltiples alteraciones en su estado morfológico y fisiológico. El crecimiento rápido e incontrolado de las células cancerosas requiere una reorganización fundamental de las vías bioquímicas responsables de la producción de energía y la biosíntesis. Las características distintivas del metabolismo del cáncer son el aumento de la tasa de glucólisis, incluso bajo las concentraciones normales de oxígeno (el efecto Warburg), el uso de aminoácidos, ácidos grasos y lactato como combustibles alternativos, la alta producción de ROS en presencia de altos niveles de antioxidantes y el aumento de la biosíntesis de ácidos grasos 1,2. En la actualidad se aprecia que el metabolismo de las células cancerosas es muy flexible, lo que les permite adaptarse a un entorno desfavorable y heterogéneo y proporciona una ventaja adicional de supervivencia3.
La alteración del metabolismo favorece la organización y composición específicas de las membranas de las células tumorales. El perfil lipídico de la membrana plasmática en las células cancerosas difiere cuantitativamente de las células no cancerosas. Los principales cambios en el lipidoma son el aumento del nivel de fosfolípidos, incluidos el fosfatidilinositol, la fosfatidilserina, la fosfatidiletanolamina y la fosfatidilcolina, la disminución del nivel de esfingomielina, el aumento de la cantidad de colesterol y un menor grado de insaturación de ácidos grasos, por nombrar algunos 4,5,6. Por lo tanto, las propiedades físicas de la membrana, como la viscosidad de la membrana, la inversa de la fluidez, cambian inevitablemente. Dado que la viscosidad determina la permeabilidad de las membranas biológicas y controla la actividad de las proteínas asociadas a la membrana (enzimas, transportadores, receptores), su regulación homeostática es vital para el funcionamiento celular. Al mismo tiempo, la modificación de la viscosidad a través del ajuste del perfil lipídico de la membrana es importante para la migración/invasión celular y la supervivencia ante cambios condicionales.
La microscopía de fluorescencia de por vida (FLIM) se ha convertido en un enfoque potente para la evaluación no invasiva de múltiples parámetros en células vivas, utilizando fluorescencia endógena o sondas exógenas7. FLIM se realiza comúnmente en un microscopio de barrido láser multifotónico, que proporciona una resolución (sub)celular. Al estar equipado con el módulo de conteo de fotón único correlacionado en el tiempo (TCSPC), permite mediciones de fluorescencia resueltas en el tiempo con alta precisión8.
El sondeo del metabolismo celular por FLIM se basa en la medición de la fluorescencia de los cofactores endógenos de las deshidrogenasas, el dinucleótido (fosfato) de nicotinamida adenina (fosfato) NAD(P)H reducido y las flavinas oxidadas: flavina adenina dinucleótido FAD y flavina mononucleótido FMN, que actúan como transportadores de electrones en una serie de reacciones bioquímicas 7,9,10. La fluorescencia detectada de NAD(P)H proviene de NADH y su forma fosforilada, NADPH, ya que son espectralmente casi idénticos. Típicamente, las desintegraciones de fluorescencia de NAD(P)H y flavinas se ajustan a una función biexponencial. En el caso de NAD(P)H, el primer componente (~0,3-0,5 ns, ~70%-80%) se atribuye a su estado libre, asociado a la glucólisis, y el segundo componente (~1,2-2,5 ns, ~20%-30%) a su estado unido a proteínas, asociado a la respiración mitocondrial. En el caso de las flavinas, el componente corto (~0,3-0,4 ns, ~75%-85%) se puede asignar al estado templado de FAD y el componente largo (~2,5-2,8 ns, ~15%-25%) a FAD, FMN y riboflavina no templados. Las alteraciones en los niveles relativos de glucólisis, glutaminólisis, fosforilación oxidativa y síntesis de ácidos grasos dan lugar a cambios en las fracciones de vida corta y larga de los cofactores. Además, la relación de intensidad de fluorescencia de estos fluoróforos (la relación redox) refleja el estado redox celular y también se utiliza como indicador metabólico. Aunque la relación redox presenta una métrica más simple, en comparación con la vida útil de la fluorescencia, en términos de adquisición de datos, FLIM es ventajosa para estimar NAD(P)H y FAD, porque la vida útil de la fluorescencia es una característica intrínseca del fluoróforo y casi no está influenciada por factores tales como el poder de excitación, el fotoblanqueo, el enfoque, la dispersión y absorción de luz, especialmente en los tejidos. a diferencia de la intensidad de emisión.
Una de las formas convenientes de mapear la viscosidad en células y tejidos vivos a nivel microscópico se basa en el uso de rotores moleculares fluorescentes, pequeños tintes sintéticos sensibles a la viscosidad, en los que los parámetros de fluorescencia dependen en gran medida de la viscosidad local11,12. En un medio viscoso, la vida útil de fluorescencia del rotor aumenta debido a la desaceleración de la torsión o rotación intramolecular. Entre los rotores moleculares, los derivados del boro dipirrometeno (BODIPY) son muy adecuados para la detección de viscosidad en sistemas biológicos, ya que tienen un buen rango dinámico de tiempos de vida de fluorescencia en el rango fisiológico de viscosidades, independencia de temperatura, desintegraciones de fluorescencia monoexponenciales que permiten una interpretación sencilla de los datos, suficiente solubilidad en agua y baja citotoxicidad13,14. Las evaluaciones cuantitativas de la microviscosidad utilizando rotores basados en BODIPY y FLIM se han demostrado previamente en células cancerosas in vitro, esferoides tumorales multicelulares y tumores de ratón in vivo15,16.
Aquí, presentamos una descripción detallada de las metodologías de sondeo secuencial para estudiar el metabolismo celular y la viscosidad de la membrana plasmática en células cancerosas in vitro mediante FLIM. Para evitar la contaminación de la fluorescencia endógena relativamente débil con la fluorescencia del rotor basado en BODIPY, las imágenes de la misma capa de células se realizan secuencialmente con la fluorescencia de NAD(P)H y FAD fotografiadas primero. Los tiempos de vida de fluorescencia de los cofactores se miden en el citoplasma, y el tiempo de vida de fluorescencia del rotor se mide en las membranas plasmáticas de las células mediante la selección manual de las zonas correspondientes como regiones de interés. El protocolo se aplicó para correlacionar el estado metabólico y la viscosidad de diferentes líneas celulares cancerosas y para evaluar los cambios después de la quimioterapia.
El protocolo para la preparación de muestras FLIM no difiere del de la microscopía de fluorescencia confocal. Una vez que se han adquirido los datos, la tarea principal es extraer la vida útil de la fluorescencia de los datos sin procesar. El rendimiento del protocolo se demuestra utilizando células HCT116 (carcinoma colorrectal humano), CT26 (carcinoma de colon murino), HeLa (carcinoma cervical humano) y huFB (fibroblastos de piel humana).
Protocolo
1. Descripción de la configuración mínima para realizar FLIM
- Para realizar este experimento, asegúrese de que la configuración requerida esté disponible: un microscopio confocal invertido, un láser pulsado, generalmente un ps o fs, con la señal de sincronización, un detector de conteo rápido de fotones (respuesta de tiempo de 150 ps) y electrónica de conteo de fotones, puertos de salida y entrada disponibles para el detector y el láser, respectivamente, en el microscopio, los pulsos del reloj de escaneo del controlador de escaneo del microscopio, el cabezal de escaneo del microscopio con los combinadores de haz láser y los divisores de haz dicroicos principales adecuados para la longitud de onda del láser utilizado para FLIM.
- Si se utiliza la excitación de dos fotones para FLIM, asegúrese de que el microscopio contenga el puerto NDD.
- Para estudios con células de mamíferos, especialmente para experimentos a largo plazo, asegúrese de tener una incubadora de CO2 mantenida a la temperatura deseada.
NOTA: Para el sistema utilizado en este experimento, consulte la Tabla de Materiales.
2. Preparación de células para microscopía
- Cultive las células de forma rutinaria en una incubadora a 37 °C con 5% de CO2 y en un ambiente húmedo.
- Para la microscopía, prepare la suspensión celular en un medio de cultivo completo a una concentración de 1 x 106 células/mL.
NOTA: Las concentraciones de células y las condiciones de los medios dependen de las células. El número de células utilizadas para la siembra y el tiempo de incubación deben adaptarse para obtener una confluencia del 70%-80% en la placa microscópica. - Siembre las células en placas de cultivo celular de 35 mm con fondo de cristal (1 x 10,5 células en 100 μL por placa) utilizando una pipeta automática de 200 μL.
NOTA: Utilice placas cuadriculadas con fondo de vidrio para la siembra de células para monitorear las células en los mismos campos de visión microscópicos en dinámica.- Durante la siembra manual, asegúrese de que la punta de la pipeta no raye el fondo o los lados del plato para evitar dañar el fondo.
- Coloque la placa en la incubadora de CO2 (37 °C, 5% de CO2, atmósfera húmeda) e incube las celdas durante 24 h.
- Pasadas las 24 horas, retire la placa de la incubadora y compruebe la morfología y la confluencia de las células bajo el microscopio óptico. Si las células no alcanzaron aproximadamente el 80% de confluencia, incube durante 24 horas adicionales.
- Retire suavemente el medio viejo de la placa con una pipeta automática de 1.000 μl y añada 2 ml de medio DMEM sin rojo de fenol (por ejemplo, DMEM Life o FluoroBrite).
NOTA: Se pueden utilizar diferentes medios de cultivo para la obtención de imágenes. Evite el rojo de fenol en el medio cuando utilice células para microscopía. - Coloque el plato en la incubadora durante 60-120 minutos para permitir la adaptación de las células.
3. FLIM de los cofactores metabólicos
- Coloque la placa con fondo de vidrio con las células (del paso 2.7) en la platina del microscopio.
- Haga clic en la pestaña Localizar del software del microscopio de escaneo láser (p. ej., ZEN - ZEISS Efficient Navigation) y, a continuación, haga clic en Transmission Light (TL) para encender la luz.
- Encuentre el plano focal de la muestra mirando a través del ocular en el nivel de corte central de las celdas, donde un cuadrado está ocupado al máximo por celdas (con un aumento de 40x).
- Haga clic en el botón APAGADO para apagar la luz.
- Abra la pestaña Adquisición . Para obtener imágenes de transmisión e intensidad de autofluorescencia de NAD(P)H endógeno, introduzca los siguientes ajustes: Longitud de onda de excitación: modo de dos fotones 750 nm, Rango de registro: 450-490 nm, Potencia del láser: 5% (~6 mW), Tamaño de la imagen: 1024 x 1024 píxeles.
NOTA: La elección de la longitud de onda de excitación y el rango de registro se basa en las características espectrales (máxima excitación y máxima emisión) de NAD(P)H17.- Utilice una lente de objetivo de inmersión en aceite С Plan-Apochromat 40x/1.3 NA para la adquisición de imágenes.
- Presione el botón Snap y guarde la imagen en formato ZEN.
- Para obtener las imágenes de transmisión e intensidad de autofluorescencia de FAD, cambie la longitud de onda de excitación a 900 nm. Establezca el rango de registro: 500-550 nm, la potencia del láser: 9% (~ 6 mW) y el tamaño de la imagen: 1024 x 1024 píxeles.
NOTA: La elección de la longitud de onda de excitación y el rango de registro se basa en las características espectrales (máxima excitación y máxima emisión) de FAD18.- Utilice una lente de objetivo de inmersión en aceite С Plan-Apochromat 40x/1.3 NA para la adquisición de imágenes.
- Presione el botón Snap y guarde la imagen en formato ZEN.
- Para NAD(P)H, configure los parámetros como se describe en el paso 3.5 en el software del microscopio de escaneo láser. Cambia el tamaño de la imagen a 256 x 256 píxeles.
- Introduzca los siguientes parámetros en el menú de SPCM (Single Photon Counting Modules) Software operativo del módulo FLIM: Tiempo de recolección: 60 s; Rango TAC: 5.00E-8; Límite mínimo de CFD: -29.41; Resolución ADC: 256, tamaño de imagen: 256 x 256 píxeles.
- Escanee la muestra durante 60 s, detenga la exploración y guarde la imagen FLIM obtenida de NAD(P)H.
- Compruebe los datos FLIM obtenidos. Para esto, abra los datos sin procesar en el software de imagen, seleccione un píxel en el citoplasma de la celda colocando el cursor sobre él y analice el decaimiento de fluorescencia en este píxel. Las intensidades de píxeles deben ser de ≥3.000 fotones por curva de decaimiento en el binning 1.
NOTA: Si el número de fotones es inferior a 3.000, aumente la potencia del láser o el tiempo de recopilación de imágenes, mientras controla la morfología de las células y la tasa de recuento de fotones. Normalmente, si la caída de la tasa de recuento supera el 10% del valor inicial, se produce el fotoblanqueo. - Para FAD, configure los parámetros como se describe en el paso 3.7 en el software del microscopio de escaneo láser. Cambia el tamaño de la imagen a 256 x 256 píxeles.
- Introduzca los siguientes parámetros en el menú de SPCM (Single Photon Counting Modules) Software operativo del módulo FLIM: Tiempo de recolección: 60 s; Rango TAC: 5.00E-8; Límite mínimo de CFD: -29.41; Resolución ADC: 256, tamaño de imagen: 256 x 256 píxeles.
- Escanee la muestra durante 60 s. Detenga el escaneo y guarde la imagen FLIM obtenida de FAD.
NOTA: Los parámetros indicados en los pasos 3.10 y 3.15 son específicos para la electrónica y el detector utilizados. - Verifique los datos obtenidos como se describe en el paso 3.12.
- Repita los pasos 3.5-3.16 para grabar imágenes FLIM desde diferentes campos de visión.
4. Tinción de células con el rotor molecular fluorescente
NOTA: Las células se visualizan en la solución del rotor molecular fluorescente sin lavar a temperatura ambiente (~20 °C) para ralentizar la internalización del rotor. La viscosidad de la membrana depende de la temperatura, como se ha demostrado en nuestros trabajos anteriores19,20. La etapa de temperatura controlada del microscopio debe apagarse antes, es decir, antes de agregar el rotor a las celdas. Para nuestra configuración, el enfriamiento del escenario tarda unos 10 minutos.
- Prepare una solución madre general del rotor molecular fluorescente BODIPY 2 (Stock 1, 25,7 mM).
- Abra BODIPY 2 en un ambiente estéril y pese aproximadamente 2 mg, utilizando una balanza precisa. Colóquelo con cuidado en un tubo de microcentrífuga.
- Utilice una pipeta automática de 20 μL para añadir 3 μL de un disolvente adecuado (por ejemplo, DMSO).
- Una vez que la muestra se disuelva completamente en DMSO, agregue 297 μL de PBS estéril y mezcle bien con una pipeta automática de 200 μL.
NOTA: Guarde la solución madre en el refrigerador a +4 °C en un envase oscuro. Una vez resuspendido, se puede almacenar en el refrigerador durante varios meses.
- Prepare un Stock 2 (8,9 mM) añadiendo 25 μL de la solución madre general (Stock 1) a un tubo de microcentrífuga, seguido de 48 μL de PBS estéril. Mezclar suavemente con una pipeta automática de 200 μL.
NOTA: Utilice el material 2 para preparar el material de tinción final, que se aplica para la tinción celular ya que se requiere una concentración micromolar. - Sustituya suavemente los medios de cultivo de la placa (a partir del paso 3.1) por una solución de Hank helada sin Ca2+/Mg2+ e incube las células a +4 °C durante 3 min.
NOTA: El uso de solución helada y la incubación a +4 °C ralentiza la internalización del rotor molecular, y la tinción local de la membrana persiste durante 20-30 min. - Prepare la solución de tinción final que contenga 4,5 μM de BODIPY 2 añadiendo 1 μL de Stock 2 a 999 μL de solución de Hank's helada o PBS.
NOTA: La concentración de BODIPY 2 en la solución de tinción final se puede aumentar a ~ 10 μM sin ningún efecto tóxico en las células, lo que resulta en una tinción más eficiente y un mayor número de fotones recolectados. A concentraciones más altas, puede producirse una sobrecarga del detector FLIM. - Aspire la solución de Hank's de la placa de cultivo celular y reemplácela con una solución helada de 4,5 μM de BODIPY 2. Las células se visualizan en la solución BODIPY 2 sin lavar.
5. FLIM del rotor molecular fluorescente en las células
NOTA: Siempre realice FLIM del rotor molecular fluorescente después de FLIM de cofactores metabólicos porque el espectro de fluorescencia de BODIPY 2 se superpone con la emisión de cofactores endógenos NAD(Р)H y FAD 12,17,18.
- Transfiera la placa con las células teñidas a la platina del microscopio (~20 °C) para la obtención de imágenes.
- Configure los siguientes parámetros para el modo de un fotón en el software del microscopio de escaneo láser: Excitación a la longitud de onda de 488 nm con un láser de iones de argón, potencia del láser 1% -2%, rango de registro 500-550 nm de longitud de onda.
- Utilice una lente de objetivo de inmersión en aceite С Plan-Apochromat 40x/1.3 NA para la adquisición de imágenes.
- Presione el botón Live . Comience a escanear y utilice el posicionamiento XY y Z mediante una platina motorizada integrada, ajuste el enfoque y obtenga imágenes de transmisión e intensidad de fluorescencia de las células en una ventana de vista previa. Guarde las imágenes obtenidas, si es necesario.
- Verifique la imagen de transmisión y fluorescencia superpuesta para ver si la fluorescencia del rotor proviene de la ubicación esperada (membrana plasmática de la célula).
- Introduzca los siguientes parámetros en el menú del software SPCM del módulo FLIM: Tiempo de recogida: 60 s; Rango TAC: 5.00E-8; Límite mínimo de CFD: -29.41; Resolución ADC: 256, tamaño de imagen: 256 x 256 píxeles.
NOTA: Dependiendo de la configuración del sistema y de los detectores utilizados para FLIM, los parámetros de adquisición de imágenes pueden variar. - Ajuste el láser Ti:Sapphire del microscopio a una longitud de onda de 850 nm y la potencia del láser a 1%-2% para FLIM de dos fotones.
- Seleccione la pestaña Continuo en el software del microscopio de escaneo láser y, a continuación, pulse Iniciar en el software SPCM. Escanee la muestra durante 60 s, detenga la exploración y guarde la imagen FLIM obtenida.
- Compruebe los datos FLIM obtenidos. Para ello, cargue los datos en bruto en el software de análisis de datos FLIM SPCImage, seleccione un píxel en la membrana de la célula colocando el cursor sobre él y analice la decaimiento de la fluorescencia en este píxel. Las intensidades de píxeles deben ser de ≥5.000 por decaimiento (posiblemente incluyendo el agrupamiento) en un tiempo de recopilación razonable (60-120 s).
- Repita los pasos 5.4-5.8 para grabar imágenes FLIM de células desde diferentes campos de visión.
NOTA: Las mediciones de FLIM de células vivas teñidas con BODIPY 2 deben limitarse a ~30 minutos después de agregar BODIPY 2.
6. Análisis de datos
- Análisis de intensidad de fluorescencia: relación redox
- Abra imágenes de la intensidad de fluorescencia de NAD(P)H y FAD utilizando ImageJ.
- Resalte un área sin celdas en la imagen NAD(P)H con una opción circular o cuadrada. Haga clic en Medir, y luego haga clic en Restar (seleccione Proceso en el panel principal, y luego Matemáticas y Restar) para restar el valor obtenido de la señal de fondo.
- Repita el paso 6.1.2 para la imagen FAD.
- Obtenga la imagen de la relación redox dividiendo la intensidad de fluorescencia de FAD por la intensidad de fluorescencia de NAD(P)H. Para ello, seleccione Proceso en el panel principal y, a continuación, seleccione Calculadora de imágenes y Dividir; marque la casilla Crear nueva ventana y, a continuación, presione Aceptar.
- Guarde la imagen en formato TIFF.
- Para calcular la relación redox, seleccione la región del citoplasma en la celda específica en la imagen TIFF y presione la tecla M . Repita el procedimiento para todas las celdas de interés.
- Importe la medición a un documento de hoja de cálculo.
NOTA: Alternativamente, las intensidades de fluorescencia de NAD(P)H y FAD en las células se pueden medir utilizando el software estándar del microscopio y la relación redox se puede obtener dividiendo estos valores en el software de la hoja de cálculo.
- Análisis de datos FLIM: metabolismo
- Importe la imagen FLIM de NAD(P)H en el software SPCImage.
- Aplique un ajuste de decaimiento biexponencial a la imagen colocando 2 en la sección Componentes .
- Corrija el parámetro Offset marcando la casilla correspondiente en el software SPCImage.
- Vaya a Opciones y seleccione Modelo. Utilice el modelo de ajuste multiexponencial incompleto y el método de ajuste MLE.
- Ajuste la agrupación para lograr intensidades de píxeles de ≥5000 fotones por curva de decaimiento.
- Compruebe el valor χ2. El χ2≤ 1.20 indica que el modelo utilizado proporciona un ajuste razonable.
- Calcule el histograma de la vida útil de los fluorescentes en cada imagen haciendo clic en el menú superior Calcular, y luego en Matriz de desintegración.
- Seleccione el área en el citoplasma de la célula específica como región de interés.
- Analice los componentes de vida útil corta y larga (τ1 y τ2, respectivamente) y las amplitudes relativas de los componentes de vida útil (a1 y a2, donde a1 + a2 = 100%) utilizando la opción Color .
- Exporte las mediciones a un software de hoja de cálculo.
- Repita los pasos 6.2.8-6.2.10 para cada celda de interés.
- Repita los pasos 6.2.1-6.2.11 para la imagen FAD.
- Análisis de datos FLIM: viscosidad
- Importe la imagen FLIM en el software SPCImage de análisis de datos FLIM.
- Elimine la marca en el cuadro de dispersión .
- Coloque 1 en la sección Componentes , ya que el decaimiento de la fluorescencia del rotor debe ajustarse a un modelo monoexponencial.
- Ajuste la agrupación para lograr una intensidad de píxeles de ≥5000 fotones por curva de desintegración.
- Compruebe el valor de χ2 en la membrana plasmática. Un valor de χ2≤ 1,20 indica que el modelo utilizado proporciona un ajuste razonable. En el caso de χ2≥ 1.20, la aproximación monoexponencial no es aplicable, tales datos pueden indicar la agregación del colorante y deben descartarse. La agregación hace que sea imposible utilizar las curvas de calibración y conduce a estimaciones de viscosidad incorrectas.
NOTA: Las decaciones biexponenciales pueden ser indicativas de agregación. En un microscopio con módulo FLIM con filtros variables, esto se puede detectar probando rangos de longitudes de onda de emisión específicos de monómeros y agregados, 500-550 nm y 580-650 nm, como se describe en la referencia21. - Genere el histograma de la vida útil de la fluorescencia τ para cada imagen haciendo clic en el menú superior Calcular, y luego en Matriz de desintegración.
- Seleccione la región de la membrana plasmática de la célula individual con decaimiento monoexponencial, χ2≤ 1.20, utilizando la opción ROI.
- Exporte el valor de la vida útil de la fluorescencia a un software de hoja de cálculo.
- Repita los pasos 6.3.7-6.3.8 para cada celda de interés.
- Convierta las vidas útiles medidas experimentalmente de BODIPY 2 (en ns) a valores de viscosidad (en cP) utilizando la siguiente ecuación (obtenida previamente sobre la base del gráfico de calibración de BODIPY en mezclas de metanol/glicerol):
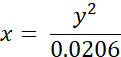
donde x - viscosidad (en cP), y - tiempo de vida de fluorescencia τ (en ns).
NOTA: IRF (Función de Respuesta de Instrumento) es una parte importante de la adaptación FLIM. En SPCImage, IRF se calcula automáticamente a partir del flanco ascendente de las curvas de decaimiento de fluorescencia. Mientras tanto, IRF se puede registrar utilizando una muestra no fluorescente, por ejemplo, cerámica, o una muestra que produce una señal SHG (segunda generación armónica), por ejemplo, colágeno, cristales de urea o azúcar. No se recomienda el uso de los IRF registrados si existe la opción de calcularlos en el software.
Resultados
Utilizando el protocolo descrito aquí, hemos visualizado los cofactores metabólicos y la viscosidad microscópica de la membrana en células vivas cultivadas utilizando FLIM. Las mediciones se han realizado en diferentes líneas celulares de cáncer: carcinoma colorrectal humano HCT116, carcinoma de colon murino CT26, cáncer de cuello uterino humano HeLa Kyoto y fibroblastos de piel humana huFB.
Discusión
Este protocolo ilustra las posibilidades de FLIM para el análisis multiparamétrico, funcional y biofísico de células cancerosas. La combinación de la imagen metabólica óptica basada en fluorescencia endógena y las medidas de la viscosidad de la membrana plasmática mediante marcaje exógeno con rotor molecular fluorescente nos permite caracterizar las interconexiones entre estos dos parámetros en células cancerosas vivas en un cultivo celular y seguir los cambios en respuesta a...
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
El desarrollo del protocolo de imágenes metabólicas fue apoyado por el Ministerio de Salud de la Federación de Rusia (Asignación Gubernamental, número de registro АААА-А20-120022590098-0). El estudio de la viscosidad fue apoyado por la Fundación Rusa de Ciencias (Proyecto No. 20-14-00111). Los autores están agradecidos a Anton Plekhanov (PRMU) por su ayuda con la producción de video.
Materiales
| Name | Company | Catalog Number | Comments |
| Item/Device | |||
| Cell culture incubator | Sanyo | 37°C, 5% CO2, humidified atmosphere | |
| Centrifuge 5702 R | Eppendorf | 5703000010 | |
| imageJ 1.53c | Wayne Rasband (NIH) | ||
| FLIM module Simple Tau 152 TCSPC (in LSM 880) | Becker & Hickl GmbH | ||
| Laminar flow hood | ThermoFisher Scientific | ||
| Leica microscope DFC290 | Leica Microsystems | ||
| LSM 880 confocal microscope | Carl Zeiss | ||
| Ti:Sapphire femtosecond laser Mai Tai | Spectra Physics | ||
| Microscope incubator XLmulti S DARK LS | PeCon GmbH | 273-800 050 | |
| Mechanical pipettor | Sartorius mLINE | volume 0.5-10 μL; 20-200 μL; 100-1000 μL | |
| Oil-immersion objective C-Apochromat 40×/1.2 NA W Korr (in LSM 880) | Carl Zeiss | 421767-9971-790 | |
| Power-Tau 152 module with the detector HPM-100-40 | Becker&Hickl GmbH | ||
| SPCImage software | Becker & Hickl GmbH | SPC 9.8; SPCImage 8.3 | |
| ZEN software | Carl Zeiss | ZEN 2.1 SP3 (black), Version 14.0.0.201 | |
| Reagent/Material | |||
| 5-fluorouracil | Medac GmbH | 3728044 | |
| DMEM | Gibco, Life Technologies | 31885023 | |
| DMSO | PanEco | F135 | |
| FBS | Hyclone | A3160801 | |
| FluoroBright DMEM | Gibco, Life Technologies | A1896701 | |
| Hank’s solution without Ca2+/Mg2+ | Gibco, Life Technologies | 14175 | |
| l-Glutamine | PanEco | F032 | |
| Mammalian cells | HCT116, CT26, HeLa Kyoto, huFB | ||
| Molecular rotor BODIPY 2 | Synthesized and Supplied by Marina Kuimova Group, Imperial College London | ||
| Penicillin/streptomycin | PanEco | A065 | |
| Tissue culture dish with cover glass-bottom FluoroDishes | World Precision Instruments, Inc | ||
| Trypsin- EDTA 0.25% | PanEco | P034 | |
| Versen buffer | PanEco | R080p |
Referencias
- Vazquez, A., et al. Cancer metabolism at a glance. Journal of Cell Science. 129 (18), 3367-3373 (2016).
- Li, Z., Zhang, H. Reprogramming of glucose, fatty acid, and amino acid metabolism for cancer progression. Cellular and Molecular Life Sciences. 73 (2), 377-392 (2015).
- Strickaert, A., et al. Cancer heterogeneity is not compatible with one unique cancer cell metabolic map. Oncogene. 36 (19), 2637-2642 (2016).
- Szlasa, W., Zendran, I., Zalesińska, A., Tarek, M., Kulbacka, J. Lipid composition of the cancer cell membrane. Journal of Bioenergetics and Biomembranes. 52, 321-342 (2020).
- Preta, G. New insights into targeting membrane lipids for cancer therapy. Frontiers in Cell and Developmental Biology. 8, 571237 (2020).
- Kojima, K. Molecular aspects of the plasma membrane in tumor cells. Nagoya Journal of Medical Science. 56, 1-18 (1993).
- Datta, R., Heaster, T. M., Sharick, J. T., Gillette, A. A., Skala, M. C. Fluorescence lifetime imaging microscopy: fundamentals and advances in instrumentation, analysis, and applications. Journal of Biomedical Optics. 25 (7), 071203 (2020).
- Becker, W. Advanced time-correlated single photon counting applications. Springer Series in Chemical Physics. , (2015).
- Shirmanova, M. V., Shcheslavskiy, V. I., Lukina, M. M., Becker, W., Zagaynova, E. V. Exploring tumor metabolism with time-resolved fluorescence methods: From single cells to a whole tumor. Multimodal Optical Diagnostics of Cancer. 3, 133-155 (2020).
- Kalinina, S., Rück, A. FLIM and PLIM in biomedical research. An innovative way to combine autofluorescence and oxygen measurements. Photonics & Lasers in Medicine. 5 (4), 257-266 (2016).
- Kuimova, M. K. Molecular rotors image intracellular viscosity. Chimia. 66 (4), 159-165 (2012).
- Kuimova, M. K. Mapping viscosity in cells using molecular rotors. Physical Chemistry Chemical Physics. 14 (37), 12671 (2012).
- Haidekker, M. A., Theodorakis, E. A. Molecular rotors-fluorescent biosensors for viscosity and flow. Organic & Biomolecular Chemistry. 5, 1669-1678 (2007).
- Liu, X., et al. Molecular mechanism of viscosity sensitivity in BODIPY rotors and application to motion-based fluorescent sensors. ACS Sensors. 5 (3), 731-739 (2020).
- Shirmanova, M. V., Shimolina, L. E., Lukina, M. M., Zagaynova, E. V., Kuimova, M. K., Dmitriev, R. Live cell imaging of viscosity in 3D tumour cell models. Multi-Parametric Live Cell Microscopy of 3D Tissue Models. Advances in Experimental Medicine and Biology. 1035, 143-153 (2017).
- Shimolina, L. E., et al. Imaging tumor microscopic viscosity in vivo using molecular rotors. Scientific Reports. 7, 41097 (2017).
- Scott, T. G., Spencer, R. D., Leonard, N. J., Weber, G. Synthetic spectroscopic models related to coenzymes and base pairs. V. Emission properties of NADH. Studies of fluorescence lifetimes and quantum efficiencies of NADH, AcPyADH, [reduced acetylpyridineadenine dinucleotide] and simplified synthetic models. Journal of the American Chemical Society. 92 (3), 687-695 (1970).
- Harvey, R. A., Damle, S. A fluorescent modification of flavin adenine dinucleotide. FEBS Letters. 26 (1-2), 341-343 (1972).
- Kubánková, M., Summers, P., López-Duarte, I., Kiryushko, D., Kuimova, M. K. Microscopic viscosity of neuronal plasma membranes measured using fluorescent molecular rotors: Effects of oxidative stress and neuroprotection. ACS Applied Materials and Interfaces. 11, 36307-36315 (2019).
- Kubánková, M., López-Duarte, I., Kiryushko, D., Kuimova, M. K. Molecular rotors report on changes in live cell plasma membrane microviscosity upon interaction with beta-amyloid aggregates. Soft Matter. 14, 9466-9474 (2018).
- Wu, Y., et al. Molecular rheometry: Direct determination of viscosity in Lo and Ld lipid phases via fluorescence lifetime imaging. Physical Chemistry Chemical Physics. 15 (36), 14986 (2013).
- Shimolina, L. E., et al. Mapping cisplatin-induced viscosity alterations in cancer cells using molecular rotor and fluorescence lifetime imaging microscopy. Journal of Biomedical Optics. 25 (12), 126004 (2020).
- Berezin, M. Y., Achilefu, S. Fluorescence lifetime measurements and biological imaging. Chemical Reviews. 110 (5), 2641-2684 (2010).
- Skala, M. C., et al. In vivo multiphoton fluorescence lifetime imaging of protein-bound and free nicotinamide adenine dinucleotide in normal and precancerous epithelia. Journal of Biomedical Optics. 12 (2), 024014 (2007).
- Rück, A., Hauser, C., Mosch, S., Kalinina, S. Spectrally resolved fluorescence lifetime imaging to investigate cell metabolism in malignant and nonmalignant oral mucosa cells. Journal of Biomedical Optics. 19 (9), 096005 (2014).
- Lee, D. -. H., Li, X., Ma, N., Digman, M. A., Lee, A. P. Rapid and label-free identification of single leukemia cells from blood in a high-density microfluidic trapping array by fluorescence lifetime imaging microscopy. Lab on a Chip. 18 (9), 1349-1358 (2018).
- Lukina, M. M., et al. Interrogation of tumor metabolism in tissue samples ex vivo using fluorescence lifetime imaging of NAD(P)H. Methods and Applications in Fluorescence. 8 (1), 014002 (2019).
- Alam, S. R., et al. Investigation of mitochondrial metabolic response to doxorubicin in prostate cancer cells: An NADH, FAD and Tryptophan FLIM assay. Scientific Reports. 7 (1), 10451 (2017).
- Walsh, A. J., et al. Quantitative optical imaging of primary tumor organoid metabolism predicts drug response in breast cancer. Cancer Research. 74 (18), 5184-5194 (2014).
- Lukina, M. M., et al. In vivo metabolic and SHG imaging for monitoring of tumor response to chemotherapy. Cytometry Part A. 95 (1), 47-55 (2019).
- Shah, A. T., et al. Optical metabolic imaging of treatment response in human head and neck squamous cell carcinoma. PLoS One. 9 (3), 90746 (2014).
- Sergeeva, T. F., et al. Relationship between intracellular pH, metabolic co-factors, and caspase-3 activation in cancer cells during apoptosis. Biochimica et Biophysica Acta (BBA) - Molecular Cell Research. 1864 (3), 604-611 (2017).
- Shirmanova, M. V., et al. Chemotherapy with cisplatin: insights into intracellular pH and metabolic landscape of cancer cells in vitro and in vivo. Scientific Reports. 7, 8911 (2017).
- Lukina, M. M., et al. Metabolic cofactors NAD(P)H and FAD as potential indicators of cancer cell response to chemotherapy with paclitaxel. Biochimica et Biophysica Acta (BBA) - General Subjects. 1862 (8), 1693-1700 (2018).
- Druzhkova, I. N., et al. The metabolic interaction of cancer cells and fibroblasts - coupling between NAD(P)H and FAD, intracellular pH and hydrogen peroxide. Cell Cycle. 15 (9), 1257-1266 (2016).
- Lakowicz, J., Szmacinski, H., Nowaczyk, K., Johnson, M. Fluorescence lifetime imaging of free and protein bound NADH. Proceedings of the National Academy of Sciences of the United States of America. 89 (4), 1271-1275 (1992).
- Vanden Berg, P. A. W., Feenstra, K. A., Mark, A. E., Berendsen, H. J. C., Visser, A. J. W. G. Dynamic conformations of flavin adenine dinucleotide: Simulated molecular dynamics of the flavin cofactor related to the time-resolved fluorescence characteristics. The Journal of Physical Chemistry B. 106 (34), 8858-8869 (2002).
- Liu, Z., et al. Mapping metabolic changes by noninvasive, multiparametric, high-resolution imaging using endogenous contrast. Science Advances. 4 (3), (2018).
- Blacker, T. S., et al. Separating NADH and NADPH fluorescence in live cells and tissues using FLIM. Nature Communications. 5, 3936 (2014).
- Lu, W., Wang, L., Chen, L., Hui, S., Rabinowitz, J. D. Extraction and quantitation of nicotinamide adenine dinucleotide redox cofactors. Antioxidants and Redox Signaling. 28 (3), 167-179 (2018).
- Ranjit, S., Malacrida, L., Jameson, D. M., Gratton, E. Fit-free analysis of fluorescence lifetime imaging data using the phasor approach. Nature Protocols. 13 (9), 1979-2004 (2018).
- Stringari, C., et al. Phasor approach to fluorescence lifetime microscopy distinguishes different metabolic states of germ cells in a live tissue. Proceedings of the National Academy of Sciences of the United States of America. 108 (33), 13582-13587 (2011).
- Smith, J. T., et al. Fast fit-free analysis of fluorescence lifetime imaging via deep learning. Proceedings of the National Academy of Sciences of the United States of America. 116 (48), 24019-24030 (2019).
- Wang, S., Chacko, J. V., Sagar, A. K., Eliceiri, K. W., Yuan, M. Nonparametric empirical Bayesian framework for fluorescence-lifetime imaging microscopy. Biomedical Optics Express. 10 (11), 5497-5517 (2019).
- Quinn, K. P., et al. Quantitative metabolic imaging using endogenous fluorescence to detect stem cell differentiation. Scientific Reports. 3 (1), 3432 (2013).
- Hou, J., et al. Correlating two-photon excited fluorescence imaging of breast cancer cellular redox state with seahorse flux analysis of normalized cellular oxygen consumption. Journal of Biomedical Optics. 21 (6), 060503 (2016).
- Wang, H., et al. Red-emitting fluorescence probe for sensing viscosity in living cells. Chemical Papers. 74, 1071-1078 (2020).
- Chen, B., et al. Sensing and imaging of mitochondrial viscosity in living cells by a red photoluminescent probe with long lifetime. Chemical Communications. 55, 7410 (2019).
- Shen, B., Wang, L. F., Zhi, X., Qian, Y. Construction of a red emission BODIPY-based probe for tracing lysosomal viscosity changes in culture cells. Sensors and Actuators B: Chemical. 304, 127271 (2019).
- Chen, T., Chen, Z., Liu, R., Zheng, S. NIR fluorescent probe for detection of viscosity and lysosome imaging in live cells. Organic and Biomolecular Chemistry. 17, 6398 (2019).
- Angelucci, C., et al. Epithelial-stromal interactions in human breast cancer: effects on adhesion, plasma membrane fluidity and migration speed and directness. PLoS One. 7 (12), 50804 (2012).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados