A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
בדיקת חילוף החומרים והצמיגות של תאים סרטניים באמצעות מיקרוסקופ הדמיה פלואורסצנטי לכל החיים
In This Article
Summary
כאן, אנו מדגימים את השימוש במיקרוסקופ הדמיה לכל החיים פלואורסצנטי (FLIM) כדי לדמות ברצף את חילוף החומרים התאי ואת צמיגות קרום הפלזמה בתרבית תאי סרטן חיים. הערכות מטבוליות מבוצעות על ידי זיהוי פלואורסצנטיות אנדוגנית. הצמיגות נמדדת באמצעות רוטור מולקולרי פלואורסצנטי.
Abstract
צמיגות היא תכונה פיזיקלית חשובה של קרום ביולוגי, מכיוון שהיא אחד הפרמטרים המרכזיים לוויסות המצב המורפולוגי והפיזיולוגי של תאים חיים. ידוע כי לממברנות הפלזמה של תאי הגידול יש שינויים משמעותיים בהרכבם, במבנה ובמאפיינים התפקודיים שלהם. יחד עם חילוף חומרים לא מווסת של גלוקוז ושומנים, תכונות הממברנה הספציפיות הללו עוזרות לתאי הגידול להסתגל למיקרו-סביבה העוינת ולפתח עמידות לטיפולים תרופתיים. כאן, אנו מדגימים את השימוש במיקרוסקופ הדמיה לכל החיים פלואורסצנטי (FLIM) כדי לדמות ברצף את חילוף החומרים התאי ואת צמיגות קרום הפלזמה בתרבית תאי סרטן חיים. הערכות מטבוליות מבוצעות על ידי זיהוי פלואורסצנטיות של גורמים מטבוליים אנדוגניים, כגון ניקוטינמיד אדנין דינוקלאוטיד מופחת NAD(P)H ופלבינות מחומצנות. הצמיגות נמדדת באמצעות רוטור מולקולרי פלואורסצנטי, צבע סינתטי רגיש לצמיגות, עם תלות חזקה לאורך חיי הקרינה בצמיגות הסביבה הקרובה. בשילוב, טכניקות אלה מאפשרות לנו להבין טוב יותר את הקשרים בין מצב הממברנה לפרופיל המטבולי של תאים סרטניים ולדמיין את השינויים הנגרמים על ידי כימותרפיה.
Introduction
טרנספורמציה ממאירה של תאים מלווה בשינויים מרובים במצבם המורפולוגי והפיזיולוגי. צמיחה מהירה ובלתי מבוקרת של תאים סרטניים דורשת ארגון מחדש של מסלולים ביוכימיים האחראים על ייצור אנרגיה וביוסינתזה. סימני ההיכר האופייניים למטבוליזם של סרטן הם קצב גליקוליזה מוגבר, אפילו בריכוזי החמצן הרגילים (אפקט ורבורג), שימוש בחומצות אמינו, חומצות שומן ולקטט כדלקים חלופיים, ייצור ROS גבוה בנוכחות רמות גבוהות של נוגדי חמצון וביוסינתזה מוגברת של חומצות שומן 1,2. כיום מעריכים כי חילוף החומרים של תאי הסרטן גמיש ביותר, מה שמאפשר להם להסתגל לסביבה השלילית וההטרוגנית ומספק יתרון הישרדות נוסף3.
חילוף חומרים שונה תומך בארגון ובהרכב הספציפי של ממברנות תאי הגידול. פרופיל השומנים של קרום הפלזמה בתאי סרטן שונה כמותית מהתאים הלא סרטניים. השינויים העיקריים בליפידום הם רמה מוגברת של פוספוליפידים כולל פוספטידילינוזיטול, פוספטידילסרין, פוספטידיל-אתנולמין ופוספטידילכולין, ירידה ברמת ספינגומיאלין, עלייה בכמות הכולסטרול ודרגה נמוכה יותר של אי-רוויה של חומצות שומן, אם להזכיר כמה 4,5,6. לכן, התכונות הפיזיקליות של הממברנה, כמו צמיגות הממברנה, היפוך הנזילות, משתנות בהכרח. מכיוון שהצמיגות קובעת את החדירות של ממברנות ביולוגיות ושולטת בפעילותם של חלבונים הקשורים לממברנה (אנזימים, טרנספורטרים, קולטנים), הוויסות ההומאוסטטי שלה חיוני לתפקוד התא. יחד עם זאת, שינוי הצמיגות באמצעות התאמת פרופיל השומנים של הממברנה חשוב לנדידה/פלישה של תאים והישרדות עם שינויים מותנים.
מיקרוסקופ הדמיה לכל החיים פלואורסצנטי (FLIM) התגלה כגישה רבת עוצמה להערכה לא פולשנית של פרמטרים מרובים בתאים חיים, תוך שימוש בפלואורסצנציה אנדוגנית או בדיקות אקסוגניות7. FLIM מתממש בדרך כלל במיקרוסקופ סריקת לייזר רב-פוטוני, המספק רזולוציה (תת-תאית). בהיותו מצויד במודול ספירת פוטונים בודדים בקורלציה בזמן (TCSPC), הוא מאפשר מדידות ברזולוציה בזמן של פלואורסצנטיות בדיוק גבוה8.
בדיקת חילוף החומרים התאי על ידי FLIM מבוססת על מדידת הקרינה של קופקטורים אנדוגניים של דהידרוגנאזות, הניקוטינמיד אדנין דינוקלאוטיד (פוספט) המופחת NAD(P)H ופלבינות מחומצנות - פלבין אדנין דינוקלאוטיד FAD ופלווין מונונוקלאוטיד FMN, הפועלים כנשאי אלקטרונים במספר תגובות ביוכימיות 7,9,10. הקרינה שזוהתה של NAD(P)H היא מ-NADH וצורתו הזרחנית, NADPH, מכיוון שהם כמעט זהים מבחינה ספקטרלית. בדרך כלל, דעיכות פלואורסצנטיות של NAD(P)H ופלבינס מתאימות לפונקציה דו-מעריכית. במקרה של NAD(P)H, המרכיב הראשון (~0.3-0.5 ns, ~70%-80%) מיוחס למצבו החופשי, הקשור לגליקוליזה, והמרכיב השני (~1.2-2.5 ns, ~20%-30%) למצבו הקשור לחלבון, הקשור לנשימה מיטוכונדריאלית. במקרה של פלבינים, ניתן להקצות את הרכיב הקצר (~0.3-0.4 ns, ~75%-85%) למצב המרווה של FAD ואת הרכיב הארוך (~2.5-2.8 ns, ~15%-25%) ל-FAD, FMN וריבופלבין לא מרווים. שינויים ברמות היחסיות של גליקוליזה, גלוטמינוליזה, זרחון חמצוני וסינתזת חומצות שומן גורמים לשינויים בשברי החיים הקצרים והארוכים של הקו-פקטורים. בנוסף, יחס עוצמת הקרינה של פלואורופורים אלה (יחס חמצון החיזור) משקף את מצב החמצון החיזור הסלולרי ומשמש גם כאינדיקטור מטבולי. למרות שיחס החיזור מציג מדד פשוט יותר, בהשוואה לאורך חיי הקרינה, מבחינת רכישת נתונים, FLIM הוא יתרון להערכת NAD(P)H ו-FAD, מכיוון שאורך חיי הקרינה הוא מאפיין מהותי של הפלואורופור וכמעט אינו מושפע מגורמים כמו כוח עירור, פוטו-הלבנה, מיקוד, פיזור אור וספיגה, במיוחד ברקמות, בניגוד לעוצמת הפליטה.
אחת הדרכים הנוחות למיפוי צמיגות בתאים חיים ורקמות ברמה המיקרוסקופית מבוססת על שימוש ברוטורים מולקולריים פלואורסצנטיים, צבעים סינתטיים קטנים רגישים לצמיגות, שבהם פרמטרי הקרינה תלויים מאוד בצמיגות המקומית11,12. בתווך צמיג, אורך החיים הקרינה של הרוטור גדל עקב האטת הפיתול או הסיבוב התוך מולקולרי. מבין הרוטורים המולקולריים, הנגזרות של בורון דיפירומתן (BODIPY) מתאימות היטב לחישת צמיגות במערכות ביולוגיות מכיוון שיש להן טווח דינמי טוב של אורך חיים פלואורסצנטי בטווח פיזיולוגי של צמיגות, עצמאות טמפרטורה, דעיכות פלואורסצנטיות חד-מעריכיות המאפשרות פירוש נתונים פשוט, מסיסות מספקת במים וציטוטוקסיות נמוכה13,14. הערכות כמותיות של מיקרו-צמיגות באמצעות רוטורים מבוססי BODIPY ו-FLIM הודגמו בעבר על תאים סרטניים במבחנה, ספרואידים של גידול רב-תאי וגידול עכבר in vivo15,16.
כאן, אנו מציגים תיאור מפורט של מתודולוגיות בדיקה עוקבות לחקר חילוף החומרים התאי וצמיגות קרום הפלזמה בתאי סרטן במבחנה על ידי FLIM. כדי למנוע זיהום של הקרינה האנדוגנית החלשה יחסית עם הקרינה של הרוטור מבוסס BODIPY, הדמיה של אותה שכבת תאים מתבצעת ברצף עם הקרינה של NAD(P)H ו-FAD שצולמה תחילה. אורך החיים הקרינה של הקו-פקטורים נמדד בציטופלזמה, ואורך החיים הקרינה של הרוטור נמדד בממברנות הפלזמה של התאים על ידי בחירה ידנית של אזורים מתאימים כאזורי עניין. הפרוטוקול יושם כדי לתאם את המצב המטבולי והצמיגות עבור קווי תאים סרטניים שונים ולהעריך את השינויים לאחר כימותרפיה.
הפרוטוקול להכנת דגימת FLIM אינו שונה מזה של מיקרוסקופיה פלואורסצנטית קונפוקלית. לאחר רכישת הנתונים, המשימה העיקרית היא לחלץ את אורך חיי הקרינה מהנתונים הגולמיים. ביצועי הפרוטוקול מודגמים באמצעות תאי HCT116 (קרצינומה של המעי הגס האנושי), CT26 (קרצינומה של המעי הגס של העכברים), HeLa (קרצינומה של צוואר הרחם האנושי) ותאי huFB (פיברובלסטים של עור אנושי).
Access restricted. Please log in or start a trial to view this content.
Protocol
1. תיאור ההגדרה המינימלית לביצוע FLIM
- כדי לבצע ניסוי זה, ודא שההגדרה הנדרשת זמינה: מיקרוסקופ קונפוקלי הפוך, לייזר פועם, בדרך כלל ps או fs, עם אות הסנכרון, גלאי ספירת פוטונים מהיר (תגובת זמן 150 ps) ואלקטרוניקה לספירת פוטונים, יציאות פלט וקלט זמינות עבור הגלאי והלייזר, בהתאמה, במיקרוסקופ, פעימות שעון הסריקה מבקר סריקת המיקרוסקופ, ראש הסריקה של המיקרוסקופ עם משלבי קרן הלייזר ומפצלי הקרן הדיכרואיים הראשיים המתאימים לאורך הגל של הלייזר המשמש ל-FLIM.
- אם נעשה שימוש בעירור דו-פוטוני עבור FLIM, ודא שהמיקרוסקופ מכיל את יציאת ה-NDD.
- עבור מחקרי תאי יונקים, במיוחד עבור ניסויים ארוכי טווח, ודא שיש חממת CO2 נשמרת בטמפרטורה הרצויה.
הערה: עבור המערכת המשמשת בניסוי זה, ראה טבלת חומרים.
2. הכנת תאים למיקרוסקופיה
- גדל את התאים באופן שגרתי באינקובטור בטמפרטורה של 37 מעלות צלזיוס עם 5% CO2 ואווירה לחה.
- למיקרוסקופיה, הכינו את תרחיף התאים במדיום תרבית שלם בריכוז של 1 x 106 תאים/מ"ל.
הערה: ריכוזי התאים ותנאי המדיה תלויים בתא. יש להתאים את מספר התאים המשמשים לזריעה ואת זמן הדגירה כדי להשיג מפגש של 70%-80% בצלחת המיקרוסקופית. - זרעו את התאים על צלחות תרבית תאים 35 מ"מ עם תחתית זכוכית (1 x 105 תאים ב-100 מיקרוליטר למנה) באמצעות פיפטה אוטומטית של 200 מיקרוליטר.
הערה: השתמש בכלי זכוכית מרושתים עבור זריעת התאים כדי לנטר תאים באותם שדות ראייה מיקרוסקופיים בדינמיקה.- במהלך הזריעה הידנית, ודא שקצה הפיפטה אינו מגרד את תחתית המנה או את דפנות המנה כדי למנוע פגיעה בתחתית.
- מניחים את המנה בחממת CO2 (37 מעלות צלזיוס, 5% CO2, אווירה לחה) ודוגרים על התאים למשך 24 שעות.
- לאחר 24 שעות, הוציאו את הצלחת מהאינקובטור ובדקו את המורפולוגיה והמפגש של התאים תחת מיקרוסקופ האור. אם התאים לא הגיעו לכ-80% מפגש, דגרו למשך 24 שעות נוספות.
- מוציאים בעדינות את המדיום הישן מהצלחת באמצעות פיפטה אוטומטית של 1,000 מיקרוליטר ומוסיפים 2 מ"ל של מדיום DMEM ללא פנול אדום (למשל, DMEM Life או FluoroBrite).
הערה: ניתן להשתמש במדיות תרבות שונות להדמיה. הימנע מפנול אדום במדיום בעת שימוש בתאים למיקרוסקופיה. - מניחים את המנה בחממה למשך 60-120 דקות כדי לאפשר הסתגלות של תאים.
3. FLIM של קופקטורים מטבוליים
- מניחים את צלחת הזכוכית עם התאים (משלב 2.7) על במת המיקרוסקופ.
- לחץ על הכרטיסייה איתור בתוכנת מיקרוסקופ סריקת הלייזר (למשל, ZEN - ZEISS Efficient Navigation), ולאחר מכן לחץ על נורית שידור (TL) כדי להדליק את האור.
- מצא את מישור המוקד של הדגימה על ידי צפייה דרך העינית ברמת הפרוסה המרכזית של התאים, שם ריבוע תפוס באופן מקסימלי על ידי תאים (בהגדלה פי 40).
- לחץ על כפתור OFF כדי לכבות את האור.
- פתח את הכרטיסייה רכישה . כדי לקבל תמונות עוצמת שידור ואוטופלואורסצנטיות של NAD(P)H אנדוגני, הזן את ההגדרות הבאות: אורך גל עירור: מצב שני פוטונים 750 ננומטר, טווח רישום: 450-490 ננומטר, עוצמת לייזר: 5% (~6 mW), גודל תמונה: 1024 x 1024 פיקסלים.
הערה: הבחירה באורך גל העירור וטווח הרישום מבוססת על המאפיינים הספקטרליים (עירור מקסימלי ופליטה מקסימלית) של NAD(P)H17.- השתמש בעדשת אובייקטיבית לטבילה בשמן С Plan-Apochromat 40x/1.3 NA לרכישת התמונה.
- לחץ על כפתור הצמד ושמור את התמונה בפורמט ZEN.
- כדי לקבל את תמונות עוצמת השידור והאוטופלואורסצנטיות של FAD, שנה את אורך גל העירור ל-900 ננומטר. הגדר את טווח הרישום: 500-550 ננומטר, עוצמת לייזר: 9% (~6 mW) וגודל תמונה: 1024 x 1024 פיקסלים.
הערה: הבחירה באורך גל העירור וטווח הרישום מבוססת על המאפיינים הספקטרליים (עירור מקסימלי ופליטה מקסימלית) של FAD18.- השתמש בעדשת אובייקטיבית לטבילה בשמן С Plan-Apochromat 40x/1.3 NA לרכישת תמונה.
- לחץ על כפתור הצמד ושמור את התמונה בפורמט ZEN.
- עבור NAD(P)H, הגדר את הפרמטרים כמתואר בשלב 3.5 בתוכנת מיקרוסקופ סריקת הלייזר. שנה את גודל התמונה ל- 256 x 256 פיקסלים.
- הזן את הפרמטרים הבאים בתפריט של SPCM (מודולי ספירת פוטונים בודדים) תוכנת הפעלה של מודול FLIM: זמן איסוף: 60 שניות; טווח TAC: 5.00E-8; מגבלת CFD נמוכה: -29.41; רזולוציית ADC: 256, גודל תמונה: 256 x 256 פיקסלים.
- סרוק את הדגימה למשך 60 שניות, הפסק את הסריקה ושמור את תמונת ה-FLIM שהתקבלה של NAD(P)H.
- בדוק את נתוני ה-FLIM שהתקבלו. לשם כך, פתח את הנתונים הגולמיים בתוכנת התמונה, בחר פיקסל בציטופלזמה של התא על ידי הצבת הסמן מעליו ונתח את דעיכת הקרינה בפיקסל זה. עוצמות הפיקסלים צריכות להיות ≥3,000 פוטונים לכל עקומת דעיכה ב-binning 1.
הערה: אם מספר הפוטונים נמוך מ-3,000, הגדל את עוצמת הלייזר או את זמן איסוף התמונה, תוך שליטה במורפולוגיה של התאים ובקצב ספירת הפוטונים. בדרך כלל, אם הירידה בשיעור הספירה עולה על 10% מהערך ההתחלתי, מתרחשת הלבנת פוטו. - עבור FAD, הגדר את הפרמטרים כמתואר בשלב 3.7 בתוכנת מיקרוסקופ סריקת הלייזר. שנה את גודל התמונה ל- 256 x 256 פיקסלים.
- הזן את הפרמטרים הבאים בתפריט של SPCM (מודולי ספירת פוטונים בודדים) תוכנת הפעלה של מודול FLIM: זמן איסוף: 60 שניות; טווח TAC: 5.00E-8; מגבלת CFD נמוכה: -29.41; רזולוציית ADC: 256, גודל תמונה: 256 x 256 פיקסלים.
- סרוק את הדגימה למשך 60 שניות. הפסק את הסריקה ושמור את תמונת ה-FLIM שהתקבלה של FAD.
הערה: הפרמטרים המצוינים בשלבים 3.10 ו-3.15 הם ספציפיים לאלקטרוניקה ולגלאי המשמש. - בדוק את הנתונים שהתקבלו כמתואר בשלב 3.12.
- חזור על שלבים 3.5-3.16 כדי להקליט תמונות FLIM משדות ראייה שונים.
4. צביעת תאים עם הרוטור המולקולרי הפלואורסצנטי
הערה: התאים מצולמים בתמיסת הרוטור המולקולרית הפלואורסצנטית ללא שטיפה בטמפרטורת החדר (~20 מעלות צלזיוס) כדי להאט את הפנמת הרוטור. צמיגות הממברנה תלויה בטמפרטורה, כפי שהודגם בעבודותינו הקודמות19,20. יש לכבות מראש את השלב מבוקר הטמפרטורה של המיקרוסקופ, כלומר לפני הוספת הרוטור לתאים. עבור ההתקנה שלנו, קירור השלב לוקח כ -10 דקות.
- הכן תמיסת מלאי כללית של הרוטור המולקולרי הפלואורסצנטי BODIPY 2 (מלאי 1, 25.7 מ"מ).
- יש לפתוח את BODIPY 2 בסביבה סטרילית ולשקול כ-2 מ"ג, תוך שימוש באיזון מדויק. הניחו אותו בזהירות בצינור מיקרו-צנטריפוגה.
- השתמש בפיפטה אוטומטית של 20 מיקרוליטר כדי להוסיף 3 מיקרוליטר של ממס מתאים (למשל, DMSO).
- לאחר שהדגימה מתמוססת לחלוטין ב-DMSO, הוסיפו 297 מיקרוליטר של PBS סטרילי וערבבו היטב באמצעות פיפטה אוטומטית של 200 מיקרוליטר.
הערה: אחסן את תמיסת המלאי במקרר בטמפרטורה של +4 מעלות צלזיוס באריזה כהה. לאחר ההשעיה, ניתן לאחסן אותו במקרר למשך מספר חודשים.
- הכן מלאי 2 (8.9 מ"מ) על ידי הוספת 25 מיקרוליטר של תמיסת המלאי הכללי (מלאי 1) לצינור מיקרוצנטריפוגה, ואחריו 48 מיקרוליטר של PBS סטרילי. מערבבים בעדינות בעזרת פיפטה אוטומטית של 200 מיקרוליטר.
הערה: השתמש במלאי 2 להכנת מלאי הצביעה הסופי, המיושם לצביעה של תאים מכיוון שנדרש ריכוז מיקרו-מולרי. - החלף בעדינות את אמצעי התרבית בצלחת (משלב 3.1) בתמיסת האנק הקרה כקרח ללא Ca2+/Mg2+ ודגר תאים בטמפרטורה של +4 מעלות צלזיוס למשך 3 דקות.
הערה: השימוש בתמיסה קרה כקרח ודגירה בטמפרטורה של +4 מעלות צלזיוס מאט את הפנמת הרוטור המולקולרי, והצביעה המקומית של הממברנה נמשכת 20-30 דקות. - הכן את תמיסת הצביעה הסופית המכילה 4.5 מיקרומטר של BODIPY 2 על ידי הוספת 1 מיקרוליטר של תמיסת האנק הקרה כקרח או PBS של 999 מיקרוליטר.
הערה: ניתן להגדיל את הריכוז של BODIPY 2 בתמיסת הצביעה הסופית ל-~10 מיקרומטר ללא כל השפעות רעילות על התאים, מה שמביא לצביעה יעילה יותר ולמספר גדול יותר של פוטונים שנאספו. בריכוזים גבוהים יותר, עלולה להתרחש עומס יתר של גלאי FLIM. - שאפו את תמיסת האנק מצלחת תרבית התאים והחליפו בתמיסה קרה כקרח של 4.5 מיקרומטר של BODIPY 2. התאים מצולמים בתמיסת BODIPY 2 ללא כביסה.
5. FLIM של הרוטור המולקולרי הפלואורסצנטי בתאים
הערה: בצע תמיד FLIM של הרוטור המולקולרי הפלואורסצנטי לאחר FLIM של קופקטורים מטבוליים מכיוון שספקטרום הקרינה של BODIPY 2 חופף לפליטה של קופקטורים אנדוגניים NAD(Р)H ו-FAD 12,17,18.
- מעבירים את הכלי עם התאים המוכתמים לשלב המיקרוסקופ (~20 מעלות צלזיוס) להדמיה.
- הגדר את הפרמטרים הבאים למצב פוטון אחד בתוכנת מיקרוסקופ סריקת הלייזר: עירור באורך גל של 488 ננומטר עם לייזר יון ארגון, הספק לייזר 1%-2%, טווח רישום אורך גל 500-550 ננומטר.
- השתמש בעדשת אובייקטיבית לטבילה בשמן С Plan-Apochromat 40x/1.3 NA לרכישת תמונה.
- לחץ על כפתור Live . התחל לסרוק ולהשתמש במיקום XY ו-Z על ידי שלב ממונע משולב, כוונן את המיקוד וקבל תמונות עוצמת שידור ופלורסנט של תאים בחלון תצוגה מקדימה. שמור את התמונות שהתקבלו, במידת הצורך.
- בדוק את תמונת השידור והפלואורסצנציה החופפת כדי לראות אם הקרינה של הרוטור מגיעה מהמיקום הצפוי (קרום הפלזמה של התא).
- הזן את הפרמטרים הבאים בתפריט של תוכנת SPCM של מודול FLIM: זמן איסוף: 60 שניות; טווח TAC: 5.00E-8; מגבלת CFD נמוכה: -29.41; רזולוציית ADC: 256, גודל תמונה: 256 x 256 פיקסלים.
הערה: בהתאם לתצורת המערכת ולגלאים המשמשים ל-FLIM, הפרמטרים של רכישת התמונה עשויים להשתנות. - התאם את לייזר Ti:Sapphire של המיקרוסקופ לאורך גל של 850 ננומטר ואת עוצמת הלייזר ל-1%-2% עבור FLIM דו-פוטוני.
- בחר בכרטיסייה רציף בתוכנת מיקרוסקופ סריקת הלייזר ולאחר מכן לחץ על התחל בתוכנת SPCM. סרוק את הדגימה למשך 60 שניות, הפסק את הסריקה ושמור את תמונת ה-FLIM שהתקבלה.
- בדוק את נתוני ה-FLIM שהתקבלו. לשם כך, טען את הנתונים הגולמיים בתוכנת SPCImage לניתוח נתוני FLIM, בחר פיקסל בקרום התא על ידי הצבת הסמן מעליו ונתח את דעיכת הקרינה בפיקסל זה. עוצמות הפיקסלים צריכות להיות ≥5,000 לכל דעיכה (אולי כולל binning) בזמן איסוף סביר (60-120 שניות).
- חזור על שלבים 5.4-5.8 כדי להקליט תמונות FLIM של תאים משדות ראייה שונים.
הערה: יש להגביל את מדידות ה-FLIM של תאים חיים המוכתמים ב-BODIPY 2 ל-~30 דקות לאחר הוספת BODIPY 2.
6. ניתוח נתונים
- ניתוח עוצמת הקרינה: יחס חמצון חיזור
- תמונות פתוחות של עוצמת הקרינה של NAD(P)H ו-FAD באמצעות ImageJ.
- סמנו אזור נטול תאים בתמונת NAD(P)H באמצעות אפשרות עיגול או ריבוע. לחץ על מדידה ולאחר מכן לחץ על חיסור (בחר תהליך בלוח הראשי ולאחר מכן מתמטיקה וחיסור) כדי להחסיר את הערך שהתקבל של אות הרקע.
- חזור על שלב 6.1.2 עבור תמונת FAD.
- השג את התמונה של יחס החיזור על ידי חלוקת עוצמת הקרינה של FAD בעוצמת הקרינה NAD(P)H. עשה זאת על-ידי בחירה באפשרות תהליך בלוח הראשי ולאחר מכן בחר מחשבון תמונות וחלקו; סמן את התיבה צור חלון חדש ולאחר מכן לחץ על אישור.
- שמור את התמונה בתבנית TIFF.
- כדי לחשב את יחס החיזור, בחר את אזור הציטופלזמה בתא הספציפי בתמונת ה-TIFF ולחץ על מקש M . חזור על הפעולה עבור כל התאים המעניינים.
- ייבא את המדידה למסמך גיליון אלקטרוני.
הערה: לחלופין, ניתן למדוד את עוצמות הקרינה של NAD(P)H ו-FAD בתאים באמצעות תוכנה סטנדרטית של המיקרוסקופ וניתן לקבל את יחס החיזור על ידי חלוקת ערכים אלה בתוכנת הגיליון האלקטרוני.
- ניתוח נתוני FLIM: מטבוליזם
- ייבא תמונת FLIM של NAD(P)H לתוכנת SPCImage.
- החל התאמה דו-מעריכית על התמונה על ידי הכנסת 2 לקטע רכיבים .
- תקן את הפרמטר Offset על ידי סימון התיבה המתאימה בתוכנת SPCImage.
- עבור אל אפשרויות ובחר דגם. השתמש במודל התאמה רב-מעריכי לא שלם ובשיטת התאמה MLE.
- התאם את binning כדי להשיג עוצמות פיקסלים של ≥5000 פוטונים לכל עקומת דעיכה.
- בדוק את הערך χ2. ה-χ2≤ 1.20 מציין שהמודל המשמש מספק התאמה סבירה.
- חשב את ההיסטוגרמה של אורך החיים הפלואורסצנטי בכל תמונה על ידי לחיצה על התפריט העליון חישוב ולאחר מכן על מטריצת דעיכה.
- בחר את האזור בציטופלזמה של התא הספציפי כאזור עניין.
- נתח את רכיבי אורך החיים הקצר והארוך (τ1 ו- τ2, בהתאמה) ואת המשרעות היחסיות של רכיבי אורך החיים (a1 ו-a 2, כאשרa 1 +a 2 = 100%) באמצעות האפשרות צבע .
- ייצא את המדידות לתוכנת גיליונות אלקטרוניים.
- חזור על שלבים 6.2.8-6.2.10 עבור כל תא מעניין.
- חזור על שלבים 6.2.1-6.2.11 עבור תמונת FAD.
- ניתוח נתוני FLIM: צמיגות
- ייבא תמונת FLIM לתוכנת SPCImage לניתוח נתונים של FLIM.
- הסר את הסימן בתיבה פיזור .
- שים 1 בסעיף רכיבים , מכיוון שדעיכת הקרינה של הרוטור צריכה להתאים למודל חד-מעריכי.
- התאם את binning כדי להשיג עוצמת פיקסלים של ≥5000 פוטונים לכל עקומת דעיכה.
- בדוק את ערך χ2 בקרום הפלזמה. ערך של χ2≤ 1.20 מצביע על כך שהמודל המשמש מספק התאמה סבירה. במקרה של χ2≥ 1.20, קירוב חד-מעריכי אינו ישים, נתונים כאלה יכולים להצביע על צבירת הצבע ויש להשליך אותם. צבירה אינה מאפשרת להשתמש בעקומות הכיול ומובילה בהערכות צמיגות שגויות.
הערה: דעיכות דו-מעריכיות עשויות להעיד על צבירה. במיקרוסקופ עם מודול FLIM עם מסננים משתנים, ניתן לזהות זאת על ידי בדיקת טווחי אורכי גל פליטה ספציפיים למונומר ולמצטבר, 500-550 ננומטר ו-580-650 ננומטר, כמתואר בהתייחסות21. - צור את ההיסטוגרמה של אורך חיי הקרינה τ עבור כל תמונה על ידי לחיצה על התפריט העליון חישוב, ולאחר מכן על מטריצת דעיכה.
- בחר את אזור קרום הפלזמה של תא בודד עם דעיכה חד-מעריכית, χ2≤ 1.20, באמצעות אפשרות החזר ROI.
- ייצא את הערך של אורך החיים של הקרינה לתוכנת גיליון אלקטרוני.
- חזור על שלבים 6.3.7-6.3.8 עבור כל תא מעניין.
- המר את משך החיים שנמדד בניסוי של BODIPY 2 (ב-ns) לערכי צמיגות (ב-cP) באמצעות המשוואה הבאה (שהושגה בעבר על בסיס עלילת הכיול של BODIPY בתערובות מתנול/גליצרול):
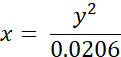
כאשר x - צמיגות (ב- cP), y - אורך חיים פלואורסצנטי τ (ב- ns).
הערה: IRF (פונקציית תגובת מכשיר) היא חלק חשוב בהתאמת FLIM. ב- SPCImage IRF מחושב אוטומטית מהקצה העולה של עקומות הדעיכה הקרינה. בינתיים, ניתן לרשום IRF באמצעות דגימה שאינה פלואורסצנטית, למשל, קרמיקה, או דגימה המייצרת אות SHG (Second Harmonic Generation), למשל, קולגן, גבישי אוריאה או סוכר. השימוש ב- IRF המוקלט אינו מומלץ אם יש אפשרות לחשב אותו בתוכנה.
Access restricted. Please log in or start a trial to view this content.
תוצאות
באמצעות הפרוטוקול המתואר כאן, דמיינו את הקו-פקטורים המטבוליים ואת צמיגות הממברנה המיקרוסקופית בתאים מתורבתים חיים באמצעות FLIM. המדידות נעשו בקווי תאים סרטניים שונים - קרצינומה של המעי הגס האנושי HCT116, קרצינומה של המעי הגס של העכברים CT26, סרטן צוואר הרחם ה?...
Access restricted. Please log in or start a trial to view this content.
Discussion
פרוטוקול זה ממחיש את האפשרויות של FLIM לניתוח רב-פרמטרי, תפקודי וביופיזיקלי של תאים סרטניים. השילוב של הדמיה מטבולית אופטית המבוססת על פלואורסצנטיות אנדוגנית ומדידות צמיגות קרום הפלזמה באמצעות תיוג אקסוגני עם רוטור מולקולרי פלואורסצנטי מאפשר לנו לאפיין את קשרי הגומלין ?...
Access restricted. Please log in or start a trial to view this content.
Disclosures
למחברים אין מה לחשוף.
Acknowledgements
פיתוח פרוטוקול הדמיה מטבולית נתמך על ידי משרד הבריאות של הפדרציה הרוסית (משימה ממשלתית, מס' רישום АААА-А20-120022590098-0). חקר הצמיגות נתמך על ידי הקרן הרוסית למדע (פרויקט מס' 20-14-00111). המחברים מודים לאנטון פלחאנוב (PRMU) על עזרתו בהפקת וידאו.
Access restricted. Please log in or start a trial to view this content.
Materials
| Name | Company | Catalog Number | Comments |
| Item/Device | |||
| Cell culture incubator | Sanyo | 37°C, 5% CO2, humidified atmosphere | |
| Centrifuge 5702 R | Eppendorf | 5703000010 | |
| imageJ 1.53c | Wayne Rasband (NIH) | ||
| FLIM module Simple Tau 152 TCSPC (in LSM 880) | Becker & Hickl GmbH | ||
| Laminar flow hood | ThermoFisher Scientific | ||
| Leica microscope DFC290 | Leica Microsystems | ||
| LSM 880 confocal microscope | Carl Zeiss | ||
| Ti:Sapphire femtosecond laser Mai Tai | Spectra Physics | ||
| Microscope incubator XLmulti S DARK LS | PeCon GmbH | 273-800 050 | |
| Mechanical pipettor | Sartorius mLINE | volume 0.5-10 μL; 20-200 μL; 100-1000 μL | |
| Oil-immersion objective C-Apochromat 40×/1.2 NA W Korr (in LSM 880) | Carl Zeiss | 421767-9971-790 | |
| Power-Tau 152 module with the detector HPM-100-40 | Becker&Hickl GmbH | ||
| SPCImage software | Becker & Hickl GmbH | SPC 9.8; SPCImage 8.3 | |
| ZEN software | Carl Zeiss | ZEN 2.1 SP3 (black), Version 14.0.0.201 | |
| Reagent/Material | |||
| 5-fluorouracil | Medac GmbH | 3728044 | |
| DMEM | Gibco, Life Technologies | 31885023 | |
| DMSO | PanEco | F135 | |
| FBS | Hyclone | A3160801 | |
| FluoroBright DMEM | Gibco, Life Technologies | A1896701 | |
| Hank’s solution without Ca2+/Mg2+ | Gibco, Life Technologies | 14175 | |
| l-Glutamine | PanEco | F032 | |
| Mammalian cells | HCT116, CT26, HeLa Kyoto, huFB | ||
| Molecular rotor BODIPY 2 | Synthesized and Supplied by Marina Kuimova Group, Imperial College London | ||
| Penicillin/streptomycin | PanEco | A065 | |
| Tissue culture dish with cover glass-bottom FluoroDishes | World Precision Instruments, Inc | ||
| Trypsin- EDTA 0.25% | PanEco | P034 | |
| Versen buffer | PanEco | R080p |
References
- Vazquez, A., et al. Cancer metabolism at a glance. Journal of Cell Science. 129 (18), 3367-3373 (2016).
- Li, Z., Zhang, H. Reprogramming of glucose, fatty acid, and amino acid metabolism for cancer progression. Cellular and Molecular Life Sciences. 73 (2), 377-392 (2015).
- Strickaert, A., et al. Cancer heterogeneity is not compatible with one unique cancer cell metabolic map. Oncogene. 36 (19), 2637-2642 (2016).
- Szlasa, W., Zendran, I., Zalesińska, A., Tarek, M., Kulbacka, J. Lipid composition of the cancer cell membrane. Journal of Bioenergetics and Biomembranes. 52, 321-342 (2020).
- Preta, G. New insights into targeting membrane lipids for cancer therapy. Frontiers in Cell and Developmental Biology. 8, 571237(2020).
- Kojima, K. Molecular aspects of the plasma membrane in tumor cells. Nagoya Journal of Medical Science. 56, 1-18 (1993).
- Datta, R., Heaster, T. M., Sharick, J. T., Gillette, A. A., Skala, M. C. Fluorescence lifetime imaging microscopy: fundamentals and advances in instrumentation, analysis, and applications. Journal of Biomedical Optics. 25 (7), 071203(2020).
- Becker, W. Advanced time-correlated single photon counting applications. Springer Series in Chemical Physics. , Springer. (2015).
- Shirmanova, M. V., Shcheslavskiy, V. I., Lukina, M. M., Becker, W., Zagaynova, E. V. Exploring tumor metabolism with time-resolved fluorescence methods: From single cells to a whole tumor. Multimodal Optical Diagnostics of Cancer. 3, 133-155 (2020).
- Kalinina, S., Rück, A. FLIM and PLIM in biomedical research. An innovative way to combine autofluorescence and oxygen measurements. Photonics & Lasers in Medicine. 5 (4), 257-266 (2016).
- Kuimova, M. K. Molecular rotors image intracellular viscosity. Chimia. 66 (4), 159-165 (2012).
- Kuimova, M. K. Mapping viscosity in cells using molecular rotors. Physical Chemistry Chemical Physics. 14 (37), 12671(2012).
- Haidekker, M. A., Theodorakis, E. A. Molecular rotors-fluorescent biosensors for viscosity and flow. Organic & Biomolecular Chemistry. 5, 1669-1678 (2007).
- Liu, X., et al. Molecular mechanism of viscosity sensitivity in BODIPY rotors and application to motion-based fluorescent sensors. ACS Sensors. 5 (3), 731-739 (2020).
- Shirmanova, M. V., Shimolina, L. E., Lukina, M. M., Zagaynova, E. V., Kuimova, M. K. Live cell imaging of viscosity in 3D tumour cell models. Multi-Parametric Live Cell Microscopy of 3D Tissue Models. Advances in Experimental Medicine and Biology. Dmitriev, R. 1035, 143-153 (2017).
- Shimolina, L. E., et al. Imaging tumor microscopic viscosity in vivo using molecular rotors. Scientific Reports. 7, 41097(2017).
- Scott, T. G., Spencer, R. D., Leonard, N. J., Weber, G. Synthetic spectroscopic models related to coenzymes and base pairs. V. Emission properties of NADH. Studies of fluorescence lifetimes and quantum efficiencies of NADH, AcPyADH, [reduced acetylpyridineadenine dinucleotide] and simplified synthetic models. Journal of the American Chemical Society. 92 (3), 687-695 (1970).
- Harvey, R. A., Damle, S. A fluorescent modification of flavin adenine dinucleotide. FEBS Letters. 26 (1-2), 341-343 (1972).
- Kubánková, M., Summers, P., López-Duarte, I., Kiryushko, D., Kuimova, M. K. Microscopic viscosity of neuronal plasma membranes measured using fluorescent molecular rotors: Effects of oxidative stress and neuroprotection. ACS Applied Materials and Interfaces. 11, 36307-36315 (2019).
- Kubánková, M., López-Duarte, I., Kiryushko, D., Kuimova, M. K. Molecular rotors report on changes in live cell plasma membrane microviscosity upon interaction with beta-amyloid aggregates. Soft Matter. 14, 9466-9474 (2018).
- Wu, Y., et al. Molecular rheometry: Direct determination of viscosity in Lo and Ld lipid phases via fluorescence lifetime imaging. Physical Chemistry Chemical Physics. 15 (36), 14986(2013).
- Shimolina, L. E., et al. Mapping cisplatin-induced viscosity alterations in cancer cells using molecular rotor and fluorescence lifetime imaging microscopy. Journal of Biomedical Optics. 25 (12), 126004(2020).
- Berezin, M. Y., Achilefu, S. Fluorescence lifetime measurements and biological imaging. Chemical Reviews. 110 (5), 2641-2684 (2010).
- Skala, M. C., et al. In vivo multiphoton fluorescence lifetime imaging of protein-bound and free nicotinamide adenine dinucleotide in normal and precancerous epithelia. Journal of Biomedical Optics. 12 (2), 024014(2007).
- Rück, A., Hauser, C., Mosch, S., Kalinina, S. Spectrally resolved fluorescence lifetime imaging to investigate cell metabolism in malignant and nonmalignant oral mucosa cells. Journal of Biomedical Optics. 19 (9), 096005(2014).
- Lee, D. -H., Li, X., Ma, N., Digman, M. A., Lee, A. P. Rapid and label-free identification of single leukemia cells from blood in a high-density microfluidic trapping array by fluorescence lifetime imaging microscopy. Lab on a Chip. 18 (9), 1349-1358 (2018).
- Lukina, M. M., et al. Interrogation of tumor metabolism in tissue samples ex vivo using fluorescence lifetime imaging of NAD(P)H. Methods and Applications in Fluorescence. 8 (1), 014002(2019).
- Alam, S. R., et al. Investigation of mitochondrial metabolic response to doxorubicin in prostate cancer cells: An NADH, FAD and Tryptophan FLIM assay. Scientific Reports. 7 (1), 10451(2017).
- Walsh, A. J., et al. Quantitative optical imaging of primary tumor organoid metabolism predicts drug response in breast cancer. Cancer Research. 74 (18), 5184-5194 (2014).
- Lukina, M. M., et al. In vivo metabolic and SHG imaging for monitoring of tumor response to chemotherapy. Cytometry Part A. 95 (1), 47-55 (2019).
- Shah, A. T., et al. Optical metabolic imaging of treatment response in human head and neck squamous cell carcinoma. PLoS One. 9 (3), 90746(2014).
- Sergeeva, T. F., et al. Relationship between intracellular pH, metabolic co-factors, and caspase-3 activation in cancer cells during apoptosis. Biochimica et Biophysica Acta (BBA) - Molecular Cell Research. 1864 (3), 604-611 (2017).
- Shirmanova, M. V., et al. Chemotherapy with cisplatin: insights into intracellular pH and metabolic landscape of cancer cells in vitro and in vivo. Scientific Reports. 7, 8911(2017).
- Lukina, M. M., et al. Metabolic cofactors NAD(P)H and FAD as potential indicators of cancer cell response to chemotherapy with paclitaxel. Biochimica et Biophysica Acta (BBA) - General Subjects. 1862 (8), 1693-1700 (2018).
- Druzhkova, I. N., et al. The metabolic interaction of cancer cells and fibroblasts - coupling between NAD(P)H and FAD, intracellular pH and hydrogen peroxide. Cell Cycle. 15 (9), 1257-1266 (2016).
- Lakowicz, J., Szmacinski, H., Nowaczyk, K., Johnson, M. Fluorescence lifetime imaging of free and protein bound NADH. Proceedings of the National Academy of Sciences of the United States of America. 89 (4), 1271-1275 (1992).
- Vanden Berg, P. A. W., Feenstra, K. A., Mark, A. E., Berendsen, H. J. C., Visser, A. J. W. G. Dynamic conformations of flavin adenine dinucleotide: Simulated molecular dynamics of the flavin cofactor related to the time-resolved fluorescence characteristics. The Journal of Physical Chemistry B. 106 (34), 8858-8869 (2002).
- Liu, Z., et al. Mapping metabolic changes by noninvasive, multiparametric, high-resolution imaging using endogenous contrast. Science Advances. 4 (3), (2018).
- Blacker, T. S., et al. Separating NADH and NADPH fluorescence in live cells and tissues using FLIM. Nature Communications. 5, 3936(2014).
- Lu, W., Wang, L., Chen, L., Hui, S., Rabinowitz, J. D. Extraction and quantitation of nicotinamide adenine dinucleotide redox cofactors. Antioxidants and Redox Signaling. 28 (3), 167-179 (2018).
- Ranjit, S., Malacrida, L., Jameson, D. M., Gratton, E. Fit-free analysis of fluorescence lifetime imaging data using the phasor approach. Nature Protocols. 13 (9), 1979-2004 (2018).
- Stringari, C., et al. Phasor approach to fluorescence lifetime microscopy distinguishes different metabolic states of germ cells in a live tissue. Proceedings of the National Academy of Sciences of the United States of America. 108 (33), 13582-13587 (2011).
- Smith, J. T., et al. Fast fit-free analysis of fluorescence lifetime imaging via deep learning. Proceedings of the National Academy of Sciences of the United States of America. 116 (48), 24019-24030 (2019).
- Wang, S., Chacko, J. V., Sagar, A. K., Eliceiri, K. W., Yuan, M. Nonparametric empirical Bayesian framework for fluorescence-lifetime imaging microscopy. Biomedical Optics Express. 10 (11), 5497-5517 (2019).
- Quinn, K. P., et al. Quantitative metabolic imaging using endogenous fluorescence to detect stem cell differentiation. Scientific Reports. 3 (1), 3432(2013).
- Hou, J., et al. Correlating two-photon excited fluorescence imaging of breast cancer cellular redox state with seahorse flux analysis of normalized cellular oxygen consumption. Journal of Biomedical Optics. 21 (6), 060503(2016).
- Wang, H., et al. Red-emitting fluorescence probe for sensing viscosity in living cells. Chemical Papers. 74, 1071-1078 (2020).
- Chen, B., et al. Sensing and imaging of mitochondrial viscosity in living cells by a red photoluminescent probe with long lifetime. Chemical Communications. 55, 7410(2019).
- Shen, B., Wang, L. F., Zhi, X., Qian, Y. Construction of a red emission BODIPY-based probe for tracing lysosomal viscosity changes in culture cells. Sensors and Actuators B: Chemical. 304, 127271(2019).
- Chen, T., Chen, Z., Liu, R., Zheng, S. NIR fluorescent probe for detection of viscosity and lysosome imaging in live cells. Organic and Biomolecular Chemistry. 17, 6398(2019).
- Angelucci, C., et al. Epithelial-stromal interactions in human breast cancer: effects on adhesion, plasma membrane fluidity and migration speed and directness. PLoS One. 7 (12), 50804(2012).
Access restricted. Please log in or start a trial to view this content.
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved