JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
Fluorescence Lifetime Imaging Microscopy를 사용한 암세포의 대사 및 점도 조사
요약
여기에서는 살아있는 암세포 배양에서 세포 대사 및 원형질막 점도를 순차적으로 이미지화하기 위해 형광 수명 이미징 현미경(FLIM)을 사용하는 방법을 보여줍니다. 대사 평가는 내인성 형광을 검출하여 수행됩니다. 점도는 형광 분자 로터를 사용하여 측정됩니다.
초록
점도는 살아있는 세포의 형태학적, 생리학적 상태를 조절하는 핵심 매개변수 중 하나이기 때문에 생물학적 막의 중요한 물리적 특성입니다. 종양 세포의 원형질막은 구성, 구조 및 기능적 특성에 상당한 변화가 있는 것으로 알려져 있습니다. 포도당과 지질의 조절되지 않는 대사와 함께 이러한 특정 멤브레인 특성은 종양 세포가 적대적인 미세 환경에 적응하고 약물 요법에 대한 내성을 발달시키는 데 도움이 됩니다. 여기에서는 살아있는 암세포 배양에서 세포 대사 및 원형질막 점도를 순차적으로 이미지화하기 위해 형광 수명 이미징 현미경(FLIM)을 사용하는 방법을 보여줍니다. 대사 평가는 환원된 니코틴아미드 아데닌 디뉴클레오티드 NAD(P)H 및 산화된 플라빈과 같은 내인성 대사 보조 인자의 형광을 검출하여 수행됩니다. 점도는 합성 점도에 민감한 염료인 형광 분자 로터를 사용하여 측정되며, 형광 수명은 즉각적인 환경의 점도에 크게 의존합니다. 이러한 기술을 함께 사용하면 암 세포의 막 상태와 대사 프로필 사이의 연결을 더 잘 이해하고 화학 요법으로 유도된 변화를 시각화할 수 있습니다.
서문
세포의 악성 변형은 세포의 형태학적, 생리학적 상태의 여러 변화를 동반합니다. 암세포의 빠르고 통제되지 않은 성장은 에너지 생산과 생합성을 담당하는 생화학적 경로의 근본적인 재조직을 필요로 합니다. 암 대사의 특징은 정상적인 산소 농도(바르부르크 효과)에서도 해당과정 속도가 빨라지고, 아미노산, 지방산 및 젖산염을 대체 연료로 사용하며, 높은 항산화 수준이 있는 상태에서 높은 ROS 생산, 지방산의 생합성 증가를 들 수 있습니다 1,2. 암세포의 대사는 매우 유연하여 불리하고 이질적인 환경에 적응할 수 있게 해주고 추가적인 생존 이점을 제공한다는 것이 이제 인정되고 있다3.
변화된 신진대사는 종양 세포막의 특정 조직과 구성을 지원합니다. 암세포에서 원형질막의 지질 프로필은 비암성 세포와 양적으로 다릅니다. 지질체의 주요 변화는 포스파티딜이노시톨, 포스파티딜세린, 포스파티딜에탄올아민 및 포스파티딜콜린을 포함한 인지질의 수치 증가, 스핑고미엘린 수치 감소, 콜레스테롤 양 증가, 지방산의 불포화도 감소 등입니다 4,5,6. 따라서 멤브레인 점도와 같은 멤브레인의 물리적 특성, 유동성의 반대는 필연적으로 변합니다. 점도는 생체막의 투과성을 결정하고 막 관련 단백질(효소, 수송체, 수용체)의 활동을 제어하기 때문에 항상성 조절은 세포 기능에 필수적입니다. 동시에 막 지질 프로파일의 조정을 통한 점도 변형은 세포 이동/침입 및 조건부 변화에 대한 생존에 중요합니다.
FLIM(Fluorescence Lifetime imaging microscopy)은 내인성 형광 또는 외인성 프로브를 사용하여 살아있는 세포의 여러 매개변수를 비침습적으로 평가하기 위한 강력한 접근 방식으로 부상했습니다7. FLIM은 일반적으로 (sub)cellular resolution을 제공하는 다광자 레이저 스캐닝 현미경에서 구현됩니다. TCSPC(Time-Correlated Single-Photon Counting (Time-Correlated Single-Photon Counting ) 모듈이 장착되어 있어 높은 정확도로 형광을 시간 분해 측정할 수 있습니다8.
FLIM에 의한 세포 대사의 조사는 탈수소효소, 환원된 니코틴아미드 아데닌 디뉴클레오티드(인산염) NAD(P)H 및 산화된 플라빈(플라빈 아데닌 디뉴클레오티드 FAD 및 플라빈 모노뉴클레오티드 FMN)의 내인성 보조 인자의 형광 측정을 기반으로 하며, 이는 여러 생화학 반응에서 전자 운반체 역할을 합니다 7,9,10. NAD(P)H의 검출된 형광은 NADH와 인산화 형태인 NADPH에서 유래한 것으로, 스펙트럼이 거의 동일하기 때문입니다. 일반적으로 NAD(P)H 및 flavin의 형광 감쇠는 bi-exponential 함수에 적합합니다. NAD(P)H의 경우, 첫 번째 성분(~0.3-0.5 ns, ~70%-80%)은 해당과정과 관련된 자유 상태에 기인하고, 두 번째 성분(~1.2-2.5 ns, ~20%-30%)은 미토콘드리아 호흡과 관련된 단백질 결합 상태에 기인합니다. 플라빈의 경우, 짧은 성분(~0.3-0.4 ns, ~75%-85%)은 FAD의 담금질 상태에 할당될 수 있고 긴 성분(~2.5-2.8 ns, ~15%-25%)은 담금질되지 않은 FAD, FMN 및 리보플라빈에 할당될 수 있습니다. 해당과정(glycolysis), 글루타미놀용해(glutaminolysis), 산화적 인산화(oxidative phosphorylation) 및 지방산 합성(fatty acid synthesis)의 상대적 수준의 변화는 보조인자의 단수명 및 장수명 분획의 변화를 초래합니다. 또한 이러한 형광단의 형광 강도 비율(산화 환원 비율)은 세포 산화 환원 상태를 반영하며 대사 지표로도 사용됩니다. 산화 환원 비율은 형광 수명에 비해 더 간단한 메트릭을 제시하지만, 데이터 수집 측면에서 FLIM은 형광단의 고유한 특성이며 특히 조직에서 여기력, 광표백, 초점, 광 산란 및 흡수와 같은 요인의 영향을 거의 받지 않기 때문에 NAD(P)H 및 FAD를 추정하는 데 유리합니다. 방출 강도와 다릅니다.
미세한 수준에서 살아있는 세포와 조직의 점도를 매핑하는 편리한 방법 중 하나는 형광 매개 변수가 국소 점도11,12에 크게 의존하는 작은 합성 점도에 민감한 염료 인 형광 분자 로터의 사용을 기반으로합니다. 점성 매질에서 로터의 형광 수명은 분자 내 비틀림 또는 회전의 속도가 느려지기 때문에 증가합니다. 분자 로터 중 붕소 디피로메텐(BODIPY)의 유도체는 생리학적 점도 범위에서 형광 수명의 우수한 동적 범위, 온도 독립성, 간단한 데이터 해석을 가능하게 하는 단지수 형광 감쇠, 충분한 수용성 및 낮은 세포 독성13,14. BODIPY 기반 로터 및 FLIM을 사용한 미세점도의 정량적 평가는 이전에 암세포 체외, 다세포 종양 스페로이드 및 생체 내 마우스 종양에 대해 입증되었습니다 15,16.
여기에서는 FLIM에 의한 in vitro 암세포의 세포 대사 및 원형질막 점도를 연구하기 위한 순차적 프로빙 방법론에 대한 자세한 설명을 제시합니다. BODIPY 기반 로터의 형광으로 인해 상대적으로 약한 내인성 형광이 오염되는 것을 방지하기 위해, NAD(P)H 및 FAD의 형광을 먼저 이미징하여 동일한 세포층의 이미징을 순차적으로 수행합니다. 보조 인자의 형광 수명은 세포질에서 측정되며, 로터의 형광 수명은 관심 영역으로 해당 영역을 수동으로 선택하여 세포의 원형질막에서 측정됩니다. 이 프로토콜은 서로 다른 암 세포주에 대한 대사 상태와 점도를 상관시키고 화학 요법 후 변화를 평가하기 위해 적용되었습니다.
FLIM 시료 전처리를 위한 프로토콜은 컨포칼 형광 현미경 검사를 위한 프로토콜과 다르지 않습니다. 데이터를 수집하면 주요 작업은 원시 데이터에서 형광 수명을 추출하는 것입니다. 프로토콜의 성능은 HCT116(인간 결장직장암), CT26(쥐 결장암), HeLa(인간 자궁경부 암종) 및 huFB(인간 피부 섬유아세포) 세포를 사용하여 입증되었습니다.
프로토콜
1. FLIM을 수행하기 위한 최소 설정에 대한 설명
- 이 실험을 수행하려면 필요한 설정을 사용할 수 있는지 확인하십시오: 도립 컨포칼 현미경, 펄스 레이저(일반적으로 ps 또는 fs), 동기화 신호, 고속 광자 계수 검출기(시간 응답 150ps) 및 광자 계수 전자 장치, 검출기 및 레이저에 각각 사용 가능한 출력 및 입력 포트, 현미경에서 현미경 스캔 컨트롤러의 스캔 클럭 펄스, 레이저 빔 결합기와 FLIM에 사용되는 레이저의 파장에 적합한 메인 이색성 빔 스플리터가 있는 현미경의 스캔 헤드.
- FLIM에 이광자 여기를 사용하는 경우 현미경에 NDD 포트가 포함되어 있는지 확인하십시오.
- 포유류 세포 연구, 특히 장기 실험의 경우 CO2 인큐베이터를 원하는 온도로 유지해야 합니다.
참고: 이 실험에 사용된 시스템은 재료 표를 참조하십시오.
2. 현미경 검사를위한 세포 준비
- 37°C의 인큐베이터에서 5% CO2 및 습한 대기에서 세포를 정기적으로 성장시킵니다.
- 현미경 검사의 경우, 1 x 106 cells/mL 농도의 완전한 배양 배지에서 세포 현탁액을 준비합니다.
참고: 세포 농도 및 매체 조건은 세포에 따라 다릅니다. 파종에 사용되는 세포의 수와 배양 시간은 현미경 접시에서 70%-80% 합류를 얻도록 조정해야 합니다. - 200 μL 자동 피펫을 사용하여 바닥이 유리로 된 35mm 세포 배양 접시(접시당 100 μL에5 개의 세포)에 세포를 파딩합니다.
참고: 파종하는 세포에 격자가 있는 유리 바닥 접시를 사용하여 역학에서 동일한 현미경 시야의 세포를 모니터링합니다.- 수동으로 파종하는 동안 바닥이 손상되지 않도록 피펫 팁이 접시의 바닥이나 측면을 긁지 않는지 확인하십시오.
- 접시를 CO2 인큐베이터(37°C, 5% CO2, 습한 분위기)에 놓고 24시간 동안 세포를 배양합니다.
- 24시간 후 인큐베이터에서 접시를 꺼내 광학 현미경으로 세포의 형태와 합류를 확인합니다. 세포가 약 80% 포화도에 도달하지 못하면 추가로 24시간 동안 배양합니다.
- 1,000μL 자동 피펫을 사용하여 접시에서 오래된 배지를 부드럽게 제거하고 페놀 레드가 없는 DMEM 배지 2mL(예: DMEM Life 또는 FluoroBrite)를 추가합니다.
참고: 이미징에 다양한 배양 배지를 사용할 수 있습니다. 현미경 검사를 위해 세포를 사용할 때 배지에서 페놀 레드를 피하십시오. - 세포가 적응할 수 있도록 접시를 인큐베이터에 60-120분 동안 놓습니다.
3. 대사 보조 인자의 FLIM
- 세포가 있는 유리 바닥 접시를 현미경 스테이지에 놓습니다(2.7단계부터).
- 레이저 스캐닝 현미경 소프트웨어(예: ZEN - ZEISS Efficient Navigation)에서 위치 탭을 클릭한 다음 투과광(TL) 을 클릭하여 조명을 켭니다.
- 세포의 중앙 슬라이스 수준에서 접안렌즈를 통해 보면서 샘플의 초점면을 찾을 수 있으며, 여기서 사각형은 세포가 최대로 차지합니다(배율 40배에서).
- OFF 버튼을 클릭하여 조명을 끕니다.
- 획득 탭을 엽니다. 내인성 NAD(P)H의 투과 및 자가형광 강도 이미지를 얻으려면 여기 파장: 이광자 모드 750nm, 등록 범위: 450-490nm, 레이저 출력: 5%(~6mW), 이미지 크기: 1024 x 1024 픽셀.
참고: 여기 파장 및 등록 범위의 선택은 NAD(P)H17의 스펙트럼 특성(최대 여기 및 최대 방출)을 기반으로 합니다.- 이미지 획득을 위해 오일 이멀젼 대물 렌즈 С Plan-Apochromat 40x/1.3 NA를 사용합니다.
- 스냅 버튼을 누르고 이미지를 ZEN 형식으로 저장합니다.
- FAD의 투과 및 자가형광 강도 이미지를 얻으려면 여기 파장 을 900nm로 변경합니다. 등록 범위: 500-550nm, 레이저 출력: 9%(~6mW) 및 이미지 크기: 1024 x 1024 픽셀을 설정합니다.
참고: 여기 파장 및 등록 범위의 선택은 FAD18의 스펙트럼 특성(최대 여기 및 최대 방출)을 기반으로 합니다.- 이미지 획득을 위해 오일 이멀젼 대물 렌즈 С Plan-Apochromat 40x/1.3 NA를 사용하십시오.
- 스냅 버튼을 누르고 이미지를 ZEN 형식으로 저장합니다.
- NAD(P)H의 경우 레이저 스캐닝 현미경 소프트웨어의 3.5단계에서 설명한 대로 매개변수를 설정합니다. 이미지 크기를 256 x 256 픽셀로 변경합니다.
- FLIM 모듈의 SPCM(Single Photon Counting Modules) 운영 소프트웨어 메뉴에 다음 매개변수를 입력합니다. 수집 시간: 60초; TAC 범위: 5.00E-8; CFD 한도 낮음: -29.41; ADC 해상도: 256, 이미지 크기: 256 x 256 픽셀.
- 샘플을 60초 동안 스캔하고 스캔을 중지하고 얻은 NAD(P)H의 FLIM 이미지를 저장합니다.
- 획득한 FLIM 데이터를 확인합니다. 이를 위해 이미지 소프트웨어에서 원시 데이터를 열고 커서를 세포의 세포질에 있는 픽셀을 선택하고 이 픽셀의 형광 붕괴를 분석합니다. 픽셀 강도는 비닝 1에서 감쇠 곡선당 ≥3,000 광자여야 합니다.
참고: 광자 수가 3,000개 미만인 경우 레이저 출력 또는 이미지 수집 시간을 늘리는 동시에 세포의 형태와 광자 계수 속도를 제어합니다. 일반적으로 계수율의 감소가 초기 값의 10%를 초과하면 광표백이 발생합니다. - FAD의 경우 레이저 스캐닝 현미경 소프트웨어의 3.7단계에서 설명한 대로 매개변수를 설정합니다. 이미지 크기를 256 x 256 픽셀로 변경합니다.
- FLIM 모듈의 SPCM(Single Photon Counting Modules) 운영 소프트웨어 메뉴에 다음 매개변수를 입력합니다. 수집 시간: 60초; TAC 범위: 5.00E-8; CFD 한도 낮음: -29.41; ADC 해상도: 256, 이미지 크기: 256 x 256 픽셀.
- 샘플을 60초 동안 스캔합니다. 스캔을 중지하고 얻은 FAD의 FLIM 이미지를 저장합니다.
알림: 3.10 및 3.15 단계에 표시된 매개변수는 사용된 전자 장치 및 감지기에 따라 다릅니다. - 3.12단계에 설명된 대로 얻은 데이터를 확인합니다.
- 3.5-3.16단계를 반복하여 다른 시야에서 FLIM 이미지를 기록합니다.
4. 형광 분자 로터를 사용한 세포 염색
참고: 세포는 로터의 내부화를 늦추기 위해 실온(~20°C)에서 세척하지 않고 형광 분자 로터 용액에서 이미징됩니다. 멤브레인 점도는 이전 작업19,20에서 입증된 바와 같이 온도에 따라 달라집니다. 현미경의 온도 제어 스테이지는 미리, 즉 로터를 셀에 추가하기 전에 꺼야 합니다. 이 설정의 경우 스테이지 냉각에는 약 10분이 걸립니다.
- 형광 분자 로터 BODIPY 2(스톡 1, 25.7 mM)의 일반 스톡 용액을 준비합니다.
- 멸균 환경에서 BODIPY 2를 열고 정확한 저울을 사용하여 무게를 약 2mg으로 합니다. 마이크로 원심분리기 튜브에 조심스럽게 넣으십시오.
- 자동 20 μL 피펫을 사용하여 3 μL의 적절한 용매(예: DMSO)를 추가합니다.
- 샘플이 DMSO에서 완전히 용해되면 297μL의 멸균 PBS를 추가하고 자동 200μL 파이펫을 사용하여 완전히 혼합합니다.
알림: 원액을 어두운 포장에 넣어 +4 °C의 냉장고에 보관하십시오. 일단 다시 부유하면 몇 달 동안 냉장고에 보관할 수 있습니다.
- 마이크로 원심분리 튜브에 일반 원액 용액(Stock 1) 25μL를 첨가한 다음 멸균 PBS 48μL를 첨가하여 스톡 2(8.9mM)를 준비합니다. 자동 200 μL 피펫을 사용하여 부드럽게 혼합합니다.
참고: 스톡 2를 사용하여 미세몰 농도가 필요하기 때문에 세포 염색에 적용되는 최종 염색 스톡을 준비합니다. - 접시의 배양 배지(3.1단계)를 Ca2+/Mg2+ 가 없는 얼음처럼 차가운 행크 용액으로 부드럽게 교체하고 +4°C에서 3분 동안 세포를 배양합니다.
참고: 얼음처럼 차가운 용액을 사용하고 +4°C에서 배양하면 분자 회전자의 내부화가 느려지고 멤브레인의 국소 염색이 20-30분 동안 지속됩니다. - 999 μL의 얼음처럼 차가운 행크 용액 또는 PBS에 1 μL의 Stock 2를 첨가하여 4.5 μM의 BODIPY 2를 함유하는 최종 염색 용액을 준비합니다.
참고: 최종 염색 용액에서 BODIPY 2의 농도는 세포에 대한 독성 영향 없이 ~10μM까지 증가할 수 있으며, 이로 인해 보다 효율적인 염색과 더 많은 수의 수집된 광자가 생성됩니다. 더 높은 농도에서는 FLIM 검출기의 과부하가 발생할 수 있습니다. - 세포 배양 접시에서 Hank's 용액을 흡입하고 BODIPY 2의 얼음처럼 차가운 4.5μM 용액으로 교체합니다. 세포는 세척 없이 BODIPY 2 용액에서 이미지화됩니다.
5. 세포에 있는 형광성 분자 회전자의 FLIM
참고: BODIPY 2의 형광 스펙트럼이 내인성 보조 인자 NAD(Р)H 및 FAD 12,17,18의 방출과 겹치기 때문에 대사 보조 인자의 FLIM 후에 항상 형광 분자 로터의 FLIM을 수행하십시오.
- 이미징을 위해 염색된 세포가 있는 접시를 현미경 스테이지(~20°C)로 옮깁니다.
- 레이저 스캐닝 현미경의 소프트웨어에서 단광자 모드에 대한 다음 매개변수를 설정합니다: 아르곤 이온 레이저를 사용한 488nm 파장에서의 여기 , 레이저 출력 1%-2%, 등록 범위 500-550nm 파장.
- 이미지 획득을 위해 오일 이멀젼 대물 렌즈 С Plan-Apochromat 40x/1.3 NA를 사용하십시오.
- 라이브 버튼을 누릅니다. 통합 전동 스테이지를 통해 XY 및 Z 포지셔닝을 스캔 및 사용하고, 초점을 조정하고, 미리보기 창에서 세포의 투과 및 형광 강도 이미지를 얻을 수 있습니다. 필요한 경우 얻은 이미지를 저장합니다.
- 겹쳐진 투과 및 형광 이미지를 확인하여 로터의 형광이 예상 위치(세포의 원형질막)에서 나오는지 확인합니다.
- FLIM 모듈의 SPCM 소프트웨어 메뉴에 다음 매개변수를 입력합니다. 수집 시간: 60초; TAC 범위: 5.00E-8; CFD 한도 낮음: -29.41; ADC 해상도: 256, 이미지 크기: 256 x 256 픽셀.
참고: FLIM에 사용되는 시스템 구성 및 감지기에 따라 이미지 획득 매개변수가 달라질 수 있습니다. - 이광자 FLIM에 대해 현미경의 Ti:Sapphire 레이저를 850nm의 파장 으로 조정하고 레이저 출력을 1%-2%로 조정합니다.
- 레이저 스캐닝 현미경 소프트웨어에서 연속(Continuous ) 탭을 선택한 다음 SPCM 소프트웨어에서 시작을 누릅니다. 샘플을 60초 동안 스캔하고 스캔을 중지한 다음 얻은 FLIM 이미지를 저장합니다.
- 획득한 FLIM 데이터를 확인합니다. 이를 위해 FLIM 데이터 분석 SPCImage 소프트웨어에서 원시 데이터를 로드하고, 세포 멤브레인에 있는 픽셀을 마우스 위에 올려 놓아 픽셀을 선택하고, 이 픽셀의 형광 감소를 분석합니다. 픽셀 강도는 합리적인 수집 시간(60-120초)에 감쇠당 ≥5,000(비닝 포함 가능)이어야 합니다.
- 5.4-5.8단계를 반복하여 다른 시야에서 세포의 FLIM 이미지를 기록합니다.
참고: BODIPY 2로 염색된 살아있는 세포의 FLIM 측정은 BODIPY 2를 첨가한 후 ~30분으로 제한되어야 합니다.
6. 데이터 분석
- 형광 강도 분석: 산화 환원 비율
- ImageJ를 사용하여 NAD(P)H 및 FAD의 형광 강도 이미지를 엽니다.
- NAD(P)H 이미지에서 cell-free 영역을 원 또는 정사각형 옵션을 사용하여 강조 표시합니다. Measure( 측정)를 클릭한 다음 Subtract(빼 기)를 클릭하여(메인 패널에서 Process(프로세스 ) 선택, Math and Subtract(수학 및 빼기)를 클릭하여 배경 신호의 얻은 값을 뺍니다.
- FAD 이미지에 대해 6.1.2단계를 반복합니다.
- FAD 형광 강도를 NAD(P)H 형광 강도로 나누어 산화 환원 비율의 이미지를 얻습니다. 기본 패널에서 Process 를 선택한 다음 Image Calculator 및 Divide를 선택하여 이 작업을 수행합니다. 새 창 만들기 확인란을 선택한 다음 확인을 누릅니다.
- 이미지를 TIFF 형식으로 저장합니다.
- 산화 환원 비율을 계산하려면 TIFF 이미지의 특정 셀에서 세포질 영역을 선택하고 M 키를 누릅니다. 관심 있는 모든 셀에 대해 반복합니다.
- 측정값을 스프레드시트 문서로 가져옵니다.
참고: 또는 현미경의 표준 소프트웨어를 사용하여 세포 내 NAD(P)H 및 FAD의 형광 강도를 측정할 수 있으며 스프레드시트 소프트웨어에서 이러한 값을 나누어 산화 환원 비율을 얻을 수 있습니다.
- FLIM 데이터 분석: 신진대사
- NAD(P)H의 FLIM 이미지를 SPCImage 소프트웨어로 가져옵니다.
- Components 섹션에 2를 입력하여 이미지에 bi-exponential decay fit을 적용합니다.
- SPCImage 소프트웨어에서 해당 상자를 선택하여 오프셋 매개변수를 수정합니다.
- 옵션으로 이동하여 모델을 선택합니다. 불완전 다지수 피팅 모델과 MLE 피팅 방법을 사용합니다.
- 감쇠 곡선당 ≥5000 광자의 픽셀 강도를 달성하도록 비닝을 조정합니다.
- χ2 값을 확인합니다. χ2≤ 1.20은 사용된 모형이 합리적인 적합도를 제공한다는 것을 나타냅니다.
- 각 이미지의 형광 수명에 대한 히스토그램을 계산하려면 상단 메뉴인 Calculate를 클릭한 다음 Decay Matrix를 클릭합니다.
- 특정 세포의 세포질에서 관심 영역으로 영역을 선택합니다.
- 색상 옵션을 사용하여 짧은 수명 구성 요소와 긴 수명 구성 요소(각각 τ1 및 τ2)와 수명 구성 요소의 상대 진폭(a1 및 a2, 여기서 a1 + a2 = 100%)을 분석합니다.
- 측정값을 스프레드시트 소프트웨어로 내보냅니다.
- 관심 있는 각 셀에 대해 6.2.8-6.2.10단계를 반복합니다.
- FAD 이미지에 대해 6.2.1-6.2.11단계를 반복합니다.
- FLIM 데이터 분석: 점도
- FLIM 이미지를 FLIM 데이터 분석 SPCImage 소프트웨어로 가져옵니다.
- 스캐터 상자에서 표시를 제거합니다.
- Components 섹션에 1을 넣으면 로터 형광 감쇠가 단일 지수 모델에 맞아야 하므로 됩니다.
- 비닝을 조정하여 감쇠 곡선당 ≥5000 광자의 픽셀 강도를 달성합니다.
- 원형질막의 χ2 값을 확인합니다. χ2≤ 1.20의 값은 사용된 모형이 합리적인 적합치를 제공한다는 것을 나타냅니다. χ2≥ 1.20의 경우 단일 지수 근사치가 적용되지 않으며 이러한 데이터는 염료 응집을 나타낼 수 있으므로 폐기해야 합니다. 응집은 검량선과 리드를 잘못된 점도 추정치에서 사용할 수 없게 만듭니다.
참고: Bi-exponential decays는 응집을 나타낼 수 있습니다. 가변 필터가 있는 FLIM 모듈이 있는 현미경에서 참고 문헌21에 설명된 대로 단량체 및 응집체별 방출 파장 범위(500-550nm 및 580-650nm)를 테스트하여 이를 감지할 수 있습니다. - 상단 메뉴인 Calculate를 클릭한 다음 Decay Matrix를 클릭하여 각 이미지에 대한 형광 수명 τ의 히스토그램을 생성합니다.
- ROI 옵션을 사용하여 단일 지수 붕괴가 있는 개별 세포의 원형질막 영역(χ2≤ 1.20)을 선택합니다.
- fluorescence lifetime의 가치를 스프레드시트 소프트웨어로 내보내십시오.
- 관심 있는 각 셀에 대해 6.3.7-6.3.8 단계를 반복합니다.
- 다음 방정식을 사용하여 실험적으로 측정된 BODIPY 2의 수명(ns 단위)을 점도 값(cP)으로 변환합니다(이전에는 메탄올/글리세롤 혼합물에서 BODIPY의 보정 플롯을 기반으로 획득).
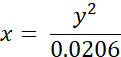
여기서 x - 점도(cP), y - 형광 수명 τ(ns).
참고: IRF(Instrument Response Function)는 FLIM 피팅의 중요한 부분입니다. SPCImage에서 IRF는 형광 감쇠 곡선의 상승 가장자리에서 자동으로 계산됩니다. 한편, IRF는 비형광 샘플(예: 세라믹) 또는 SHG(Second Harmonic Generation) 신호를 생성하는 샘플(예: 콜라겐, 요소 결정 또는 당)을 사용하여 기록할 수 있습니다. 소프트웨어에서 IRF를 계산할 수 있는 옵션이 있는 경우 기록된 IRF를 사용하지 않는 것이 좋습니다.
결과
여기에 설명된 프로토콜을 사용하여 FLIM을 사용하여 살아있는 배양 세포의 대사 보조 인자와 미세한 막 점도를 시각화했습니다. 측정은 인간 결장직장 암종 HCT116, 쥐 결장 암종 CT26, 인간 자궁경부암 HeLa Kyoto 및 인간 피부 섬유아세포 huFB와 같은 다양한 암 세포주에서 수행되었습니다.
형광 강도 기반 ?...
토론
이 프로토콜은 암세포의 다중 파라미터, 기능 및 생물물리학적 분석을 위한 FLIM의 가능성을 보여줍니다. 내인성 형광을 기반으로 한 광학 대사 이미징과 형광 분자 로터를 사용한 외인성 표지를 사용한 원형질막 점도 측정의 결합을 통해 세포 배양에서 살아있는 암세포에서 이 두 매개 변수 간의 상호 연결을 특성화하고 화학 요법에 대한 반응 변화를 추적할 수 있습니다...
공개
저자는 공개할 내용이 없습니다.
감사의 말
대사 영상 프로토콜의 개발은 러시아 연방 보건부(정부 할당, 등록 번호 ААААА-А20-120022590098-0)의 지원을 받았습니다. 점도 연구는 러시아 과학 재단(프로젝트 번호 20-14-00111)의 지원을 받았습니다. 저자는 비디오 제작에 도움을 준 Anton Plekhanov(PRMU)에게 감사드립니다.
자료
| Name | Company | Catalog Number | Comments |
| Item/Device | |||
| Cell culture incubator | Sanyo | 37°C, 5% CO2, humidified atmosphere | |
| Centrifuge 5702 R | Eppendorf | 5703000010 | |
| imageJ 1.53c | Wayne Rasband (NIH) | ||
| FLIM module Simple Tau 152 TCSPC (in LSM 880) | Becker & Hickl GmbH | ||
| Laminar flow hood | ThermoFisher Scientific | ||
| Leica microscope DFC290 | Leica Microsystems | ||
| LSM 880 confocal microscope | Carl Zeiss | ||
| Ti:Sapphire femtosecond laser Mai Tai | Spectra Physics | ||
| Microscope incubator XLmulti S DARK LS | PeCon GmbH | 273-800 050 | |
| Mechanical pipettor | Sartorius mLINE | volume 0.5-10 μL; 20-200 μL; 100-1000 μL | |
| Oil-immersion objective C-Apochromat 40×/1.2 NA W Korr (in LSM 880) | Carl Zeiss | 421767-9971-790 | |
| Power-Tau 152 module with the detector HPM-100-40 | Becker&Hickl GmbH | ||
| SPCImage software | Becker & Hickl GmbH | SPC 9.8; SPCImage 8.3 | |
| ZEN software | Carl Zeiss | ZEN 2.1 SP3 (black), Version 14.0.0.201 | |
| Reagent/Material | |||
| 5-fluorouracil | Medac GmbH | 3728044 | |
| DMEM | Gibco, Life Technologies | 31885023 | |
| DMSO | PanEco | F135 | |
| FBS | Hyclone | A3160801 | |
| FluoroBright DMEM | Gibco, Life Technologies | A1896701 | |
| Hank’s solution without Ca2+/Mg2+ | Gibco, Life Technologies | 14175 | |
| l-Glutamine | PanEco | F032 | |
| Mammalian cells | HCT116, CT26, HeLa Kyoto, huFB | ||
| Molecular rotor BODIPY 2 | Synthesized and Supplied by Marina Kuimova Group, Imperial College London | ||
| Penicillin/streptomycin | PanEco | A065 | |
| Tissue culture dish with cover glass-bottom FluoroDishes | World Precision Instruments, Inc | ||
| Trypsin- EDTA 0.25% | PanEco | P034 | |
| Versen buffer | PanEco | R080p |
참고문헌
- Vazquez, A., et al. Cancer metabolism at a glance. Journal of Cell Science. 129 (18), 3367-3373 (2016).
- Li, Z., Zhang, H. Reprogramming of glucose, fatty acid, and amino acid metabolism for cancer progression. Cellular and Molecular Life Sciences. 73 (2), 377-392 (2015).
- Strickaert, A., et al. Cancer heterogeneity is not compatible with one unique cancer cell metabolic map. Oncogene. 36 (19), 2637-2642 (2016).
- Szlasa, W., Zendran, I., Zalesińska, A., Tarek, M., Kulbacka, J. Lipid composition of the cancer cell membrane. Journal of Bioenergetics and Biomembranes. 52, 321-342 (2020).
- Preta, G. New insights into targeting membrane lipids for cancer therapy. Frontiers in Cell and Developmental Biology. 8, 571237 (2020).
- Kojima, K. Molecular aspects of the plasma membrane in tumor cells. Nagoya Journal of Medical Science. 56, 1-18 (1993).
- Datta, R., Heaster, T. M., Sharick, J. T., Gillette, A. A., Skala, M. C. Fluorescence lifetime imaging microscopy: fundamentals and advances in instrumentation, analysis, and applications. Journal of Biomedical Optics. 25 (7), 071203 (2020).
- Becker, W. Advanced time-correlated single photon counting applications. Springer Series in Chemical Physics. , (2015).
- Shirmanova, M. V., Shcheslavskiy, V. I., Lukina, M. M., Becker, W., Zagaynova, E. V. Exploring tumor metabolism with time-resolved fluorescence methods: From single cells to a whole tumor. Multimodal Optical Diagnostics of Cancer. 3, 133-155 (2020).
- Kalinina, S., Rück, A. FLIM and PLIM in biomedical research. An innovative way to combine autofluorescence and oxygen measurements. Photonics & Lasers in Medicine. 5 (4), 257-266 (2016).
- Kuimova, M. K. Molecular rotors image intracellular viscosity. Chimia. 66 (4), 159-165 (2012).
- Kuimova, M. K. Mapping viscosity in cells using molecular rotors. Physical Chemistry Chemical Physics. 14 (37), 12671 (2012).
- Haidekker, M. A., Theodorakis, E. A. Molecular rotors-fluorescent biosensors for viscosity and flow. Organic & Biomolecular Chemistry. 5, 1669-1678 (2007).
- Liu, X., et al. Molecular mechanism of viscosity sensitivity in BODIPY rotors and application to motion-based fluorescent sensors. ACS Sensors. 5 (3), 731-739 (2020).
- Shirmanova, M. V., Shimolina, L. E., Lukina, M. M., Zagaynova, E. V., Kuimova, M. K., Dmitriev, R. Live cell imaging of viscosity in 3D tumour cell models. Multi-Parametric Live Cell Microscopy of 3D Tissue Models. Advances in Experimental Medicine and Biology. 1035, 143-153 (2017).
- Shimolina, L. E., et al. Imaging tumor microscopic viscosity in vivo using molecular rotors. Scientific Reports. 7, 41097 (2017).
- Scott, T. G., Spencer, R. D., Leonard, N. J., Weber, G. Synthetic spectroscopic models related to coenzymes and base pairs. V. Emission properties of NADH. Studies of fluorescence lifetimes and quantum efficiencies of NADH, AcPyADH, [reduced acetylpyridineadenine dinucleotide] and simplified synthetic models. Journal of the American Chemical Society. 92 (3), 687-695 (1970).
- Harvey, R. A., Damle, S. A fluorescent modification of flavin adenine dinucleotide. FEBS Letters. 26 (1-2), 341-343 (1972).
- Kubánková, M., Summers, P., López-Duarte, I., Kiryushko, D., Kuimova, M. K. Microscopic viscosity of neuronal plasma membranes measured using fluorescent molecular rotors: Effects of oxidative stress and neuroprotection. ACS Applied Materials and Interfaces. 11, 36307-36315 (2019).
- Kubánková, M., López-Duarte, I., Kiryushko, D., Kuimova, M. K. Molecular rotors report on changes in live cell plasma membrane microviscosity upon interaction with beta-amyloid aggregates. Soft Matter. 14, 9466-9474 (2018).
- Wu, Y., et al. Molecular rheometry: Direct determination of viscosity in Lo and Ld lipid phases via fluorescence lifetime imaging. Physical Chemistry Chemical Physics. 15 (36), 14986 (2013).
- Shimolina, L. E., et al. Mapping cisplatin-induced viscosity alterations in cancer cells using molecular rotor and fluorescence lifetime imaging microscopy. Journal of Biomedical Optics. 25 (12), 126004 (2020).
- Berezin, M. Y., Achilefu, S. Fluorescence lifetime measurements and biological imaging. Chemical Reviews. 110 (5), 2641-2684 (2010).
- Skala, M. C., et al. In vivo multiphoton fluorescence lifetime imaging of protein-bound and free nicotinamide adenine dinucleotide in normal and precancerous epithelia. Journal of Biomedical Optics. 12 (2), 024014 (2007).
- Rück, A., Hauser, C., Mosch, S., Kalinina, S. Spectrally resolved fluorescence lifetime imaging to investigate cell metabolism in malignant and nonmalignant oral mucosa cells. Journal of Biomedical Optics. 19 (9), 096005 (2014).
- Lee, D. -. H., Li, X., Ma, N., Digman, M. A., Lee, A. P. Rapid and label-free identification of single leukemia cells from blood in a high-density microfluidic trapping array by fluorescence lifetime imaging microscopy. Lab on a Chip. 18 (9), 1349-1358 (2018).
- Lukina, M. M., et al. Interrogation of tumor metabolism in tissue samples ex vivo using fluorescence lifetime imaging of NAD(P)H. Methods and Applications in Fluorescence. 8 (1), 014002 (2019).
- Alam, S. R., et al. Investigation of mitochondrial metabolic response to doxorubicin in prostate cancer cells: An NADH, FAD and Tryptophan FLIM assay. Scientific Reports. 7 (1), 10451 (2017).
- Walsh, A. J., et al. Quantitative optical imaging of primary tumor organoid metabolism predicts drug response in breast cancer. Cancer Research. 74 (18), 5184-5194 (2014).
- Lukina, M. M., et al. In vivo metabolic and SHG imaging for monitoring of tumor response to chemotherapy. Cytometry Part A. 95 (1), 47-55 (2019).
- Shah, A. T., et al. Optical metabolic imaging of treatment response in human head and neck squamous cell carcinoma. PLoS One. 9 (3), 90746 (2014).
- Sergeeva, T. F., et al. Relationship between intracellular pH, metabolic co-factors, and caspase-3 activation in cancer cells during apoptosis. Biochimica et Biophysica Acta (BBA) - Molecular Cell Research. 1864 (3), 604-611 (2017).
- Shirmanova, M. V., et al. Chemotherapy with cisplatin: insights into intracellular pH and metabolic landscape of cancer cells in vitro and in vivo. Scientific Reports. 7, 8911 (2017).
- Lukina, M. M., et al. Metabolic cofactors NAD(P)H and FAD as potential indicators of cancer cell response to chemotherapy with paclitaxel. Biochimica et Biophysica Acta (BBA) - General Subjects. 1862 (8), 1693-1700 (2018).
- Druzhkova, I. N., et al. The metabolic interaction of cancer cells and fibroblasts - coupling between NAD(P)H and FAD, intracellular pH and hydrogen peroxide. Cell Cycle. 15 (9), 1257-1266 (2016).
- Lakowicz, J., Szmacinski, H., Nowaczyk, K., Johnson, M. Fluorescence lifetime imaging of free and protein bound NADH. Proceedings of the National Academy of Sciences of the United States of America. 89 (4), 1271-1275 (1992).
- Vanden Berg, P. A. W., Feenstra, K. A., Mark, A. E., Berendsen, H. J. C., Visser, A. J. W. G. Dynamic conformations of flavin adenine dinucleotide: Simulated molecular dynamics of the flavin cofactor related to the time-resolved fluorescence characteristics. The Journal of Physical Chemistry B. 106 (34), 8858-8869 (2002).
- Liu, Z., et al. Mapping metabolic changes by noninvasive, multiparametric, high-resolution imaging using endogenous contrast. Science Advances. 4 (3), (2018).
- Blacker, T. S., et al. Separating NADH and NADPH fluorescence in live cells and tissues using FLIM. Nature Communications. 5, 3936 (2014).
- Lu, W., Wang, L., Chen, L., Hui, S., Rabinowitz, J. D. Extraction and quantitation of nicotinamide adenine dinucleotide redox cofactors. Antioxidants and Redox Signaling. 28 (3), 167-179 (2018).
- Ranjit, S., Malacrida, L., Jameson, D. M., Gratton, E. Fit-free analysis of fluorescence lifetime imaging data using the phasor approach. Nature Protocols. 13 (9), 1979-2004 (2018).
- Stringari, C., et al. Phasor approach to fluorescence lifetime microscopy distinguishes different metabolic states of germ cells in a live tissue. Proceedings of the National Academy of Sciences of the United States of America. 108 (33), 13582-13587 (2011).
- Smith, J. T., et al. Fast fit-free analysis of fluorescence lifetime imaging via deep learning. Proceedings of the National Academy of Sciences of the United States of America. 116 (48), 24019-24030 (2019).
- Wang, S., Chacko, J. V., Sagar, A. K., Eliceiri, K. W., Yuan, M. Nonparametric empirical Bayesian framework for fluorescence-lifetime imaging microscopy. Biomedical Optics Express. 10 (11), 5497-5517 (2019).
- Quinn, K. P., et al. Quantitative metabolic imaging using endogenous fluorescence to detect stem cell differentiation. Scientific Reports. 3 (1), 3432 (2013).
- Hou, J., et al. Correlating two-photon excited fluorescence imaging of breast cancer cellular redox state with seahorse flux analysis of normalized cellular oxygen consumption. Journal of Biomedical Optics. 21 (6), 060503 (2016).
- Wang, H., et al. Red-emitting fluorescence probe for sensing viscosity in living cells. Chemical Papers. 74, 1071-1078 (2020).
- Chen, B., et al. Sensing and imaging of mitochondrial viscosity in living cells by a red photoluminescent probe with long lifetime. Chemical Communications. 55, 7410 (2019).
- Shen, B., Wang, L. F., Zhi, X., Qian, Y. Construction of a red emission BODIPY-based probe for tracing lysosomal viscosity changes in culture cells. Sensors and Actuators B: Chemical. 304, 127271 (2019).
- Chen, T., Chen, Z., Liu, R., Zheng, S. NIR fluorescent probe for detection of viscosity and lysosome imaging in live cells. Organic and Biomolecular Chemistry. 17, 6398 (2019).
- Angelucci, C., et al. Epithelial-stromal interactions in human breast cancer: effects on adhesion, plasma membrane fluidity and migration speed and directness. PLoS One. 7 (12), 50804 (2012).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유