A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
تحديد التفاعل الثلاثي بين اثنين من مونومرات من عامل النسخ MADS مربع وبروتين استشعار الكالسيوم من قبل BiFC-FRET-FLIM المقايسة
* These authors contributed equally
In This Article
Summary
هنا نقدم، طريقة لتصور تشكيل معقدة الترنية بين ثلاثة شركاء البروتين باستخدام البروتينات الموسومة الفلورسنت من قبل BiFC مقرها FRET-FLIM المقايسة. هذه الطريقة قيمة لدراسة مجمعات التفاعل البروتين البروتين في الجسم الحي.
Abstract
التفاعلات البروتين البروتين هي جزء لا يتجزأ من جميع العمليات البيولوجية في الخلايا لأنها تلعب دورا حاسما في تنظيم وصيانة وتعديل الوظائف الخلوية. وتشارك هذه التفاعلات في مجموعة واسعة من الظواهر مثل نقل الإشارات، والاستجابة لمسببات الأمراض، والتفاعلات بين الخلايا الخلوية، والعمليات الأيضية والتنموية. وفي حالة عوامل النسخ، قد تؤدي هذه التفاعلات إلى احتكار الوحدات الفرعية، وعزلها في سياقات فرعية محددة مثل النواة والسيتوبلازم وما إلى ذلك، والتي بدورها قد يكون لها تأثير أكثر عمقا على التعبير عن جينات المصب. هنا، ونحن نظهر منهجية لتصور في التفاعل الثلاثي في الجسم الحي باستخدام استكمال الفلورية البيموليكية (BiFC) مقرها Förster الرنين نقل الطاقة (FRET) التي تنطوي على التصوير مدى الحياة الفلورية (FLIM). يتفاعل اثنان من البروتينات المختارة لهذه المظاهرة كشركاء BiFC ، ويستخدم نشاط الفلورسينس المعاد تشكيله لإجراء فحص FRET-FLIM مع الشريك الثالث. وقد استخدمت أربعة إلى خمسة أسابيع من العمر نمو غرفة نمت مصانع نيكوتيانا بنثاميانا كنظام مصنع نموذجي لهذا العرض التوضيحي.
Introduction
تشكل التفاعلات البروتينية البروتينية (PPIs) أساس الأداء السليم للخلايا النواة من خلال تنظيم مختلف العمليات الأيضية والتنموية. بعض PPIs مستقرة، في حين أن البعض الآخر عابر في طبيعته. يمكن تصنيف التفاعلات بناء على عدد الأعضاء ونوعهم في التفاعل مثل ثنائية، ثلاثية، ثلاثية الأرقام، رباعية الأرقام، وهيتيروميك1. قد يؤدي تحديد وتوصيف التفاعلات البروتينية إلى فهم أفضل لوظائف البروتين والشبكات التنظيمية.
عوامل النسخ هي البروتينات التي تشارك في الوظائف التنظيمية. وهي تنظم معدل نسخ جيناتها المصب عن طريق الربط بالحمض النووي. في بعض الأحيان oligomerization أو تشكيل مجمعات من الدرجة العالية عن طريق البروتينات هو شرط أساسي لتنفيذ وظائفهم2. عوامل النسخ مربع MADS النباتية هي الجينات المنزلية التي تنظم عمليات مختلفة مثل انتقال الأزهار، وتطوير الأعضاء الأزهار، والإخصاب، وتطوير البذور، والتألق، والتنمية النباتية. ومن المعروف أنها تشكل مجمعات أعلى ترتيبا التي ترتبط الحمض النووي3،4. دراسة شبكات PPI بين عوامل النسخ والمتفاعلين معها يوفر رؤى حول التعقيد الكامن وراء التنظيم النسخي.
تعبير البروتين العابر في نيكوتيانا بنثاميانا كان نهجا شائعا لدراسة توطين البروتين أو التفاعلات البروتين البروتين في الجسم الحي5. BiFC و FRET هي طرق لدراسة التفاعلات البروتين البروتين في الجسم الحي باستخدام أنظمة المراسل الفلورسنت6. وقد ثبت أن مزيجا من هاتين التقنيتين للكشف عن التفاعل بين ثلاثةبروتينات 7. يتم قياس FRET باستخدام تقنية acceptor photobleaching والانبعاثات الحساسة والتصوير مدى الحياة الفلوري (FLIM). وقد برز الفحص FRET القائم على FLIM كأداة توفر التحديد الكمي الدقيق وخصوصية الصدغية ملعقة لقياسات نقل الطاقة بين اثنين من الجزيئات على أساس العمر الفلوري8. يقيس FLIM الوقت الذي يبقى فيه الفلوروفور في حالة متحمسة قبل انبعاث الفوتون وهو أفضل من التقنيات التي تستخدمقياساتالكثافة وحدها 9و10. وإلى جانب أنظمة heterologous مثل نيكوتيانا بنثاميانا وقشور البشرة البصل، أظهرت تقارير أكثر حداثة استخدام جذور Arabidopsis والشتلات الأرز الشباب، وما إلى ذلك، لتحليل في الجسم الحي من التفاعلات البروتين البروتين في ظل الظروفالأصلية 11،12.
وبخلاف نظام التعبير المناسب، فإن اختيار الشركاء المتفاعلين في اختبارات BiFC و FRET أمر بالغ الأهمية لنجاح هذه التجربة. يجب التحقق من صحة مؤشر أسعار المنتجين بين الشركاء المستخدمين في تكوين BiFC باستخدام عناصر التحكم المناسبة قبل استخدامها كشريك مترافق في تجربة FRET13. يستخدم BiFC الاستكمال الهيكلي للأجزاء N-و C-terminal من البروتين الفلوري. كان القيد الشائع في معظم إن لم يكن كل البروتينات الفلورية المستخدمة في مقايسات BiFC هو التجميع الذاتي بين الشظايا المشتقة غير الفلورية ، مما يساهم في الفلورسنت الإيجابي الزائف ويقلل من نسبة الإشارة إلى الضوضاء (S / N)14. التطورات الأخيرة، بما في ذلك الطفرات نقطة أو موقف تقسيم البروتين الفلورسنت، أدت إلى أزواج BiFC مع زيادة كثافة، وخصوصية أعلى، وارتفاع نسبة S / N15،16. ويمكن أيضا استخدام هذه البروتينات الفلورية لتنفيذ BiFC اعتمادا على مدى ملاءمة التجربة.
تقليديا، وقد استخدمت CFP وYFP كما المانحة والزوج المقبول في التجارب FRET17. ومع ذلك، تم العثور على YFP أو M-Citrine لتكون أفضل المتبرعين FRET (عند استخدامها مع RFP كمقبول) بسبب غلة الكم عالية (QY) خلال التعبير الأصلي من البروتينات المستهدفة في نظام الجذر Arabidopsis. كما يلعب اختيار المروجين (التأسيسيين مقابل الأصليين/المحليين) والفلوروفور دورا حاسما في تصميم تجربة ناجحة بين BiFC-FRET-FLIM. ومن الضروري ملاحظة أن كفاءة الجهات المانحة FRET ومدى ملاءمة أزواج FRET تميل إلى التغير مع التغيير في المروج والنظام البيولوجي المستخدم للتعبير. يعتمد QY للفلوروفور ، الذي يتعلق بسطوعه ، على درجة الحموضة ودرجة الحرارة والنظام البيولوجي قيد الاستخدام. نقترح أن يتم النظر في هذه المعايير بدقة قبل اختيار زوج الفلوروفور لتجربة FRET. النظام البيولوجي، والمروجين، والبروتينات المستخدمة لهذا البروتوكول عملت بشكل جيد مع الفلوروفوريس CFP-YFP لتجربة BIFC FRET-FLIM.
في هذه الدراسة، ونحن دمج ميزة FLIM لتصور التفاعل بين ثلاثة جزيئات البروتين باستخدام BIFC FRET مقرها. في هذه التقنية، يتم وضع علامة اثنين من البروتينات مع البروتين YFP الانقسام والبروتين الثالث مع CFP. منذ كنا مهتمين في دراسة التفاعل بين البروتين MADS مربع (M) homodimer مع بروتين استشعار الكالسيوم (C)، تم وضع علامات هذه البروتينات مع البروتينات الفلورية في pSITE-1CA وpSITE-3CA ناقلات18. اثنين من الشركاء التفاعل، في هذا المقايسة، تم وضع علامات مع N- و C-المحطة أجزاء من YFP في pSPYNE-35S وpSPYCE-35S ناقلات19،ونتائج تفاعلها في إعادة تشكيل YFP الوظيفية التي تعمل كمقبل FRET إلى الشريك التفاعل الثالث، الذي هو الموسومة مع CFP (بوصفها المانحة FRET) (الشكل 1 ). وفي هذه الحالة بالذات، تم التحقق من صحة مؤشر أسعار المنتجين بين اثنين من مونومرات M وبين M و C من خلال أداء BiFC في ثلاثة أنظمة مختلفة جنبا إلى جنب مع نظام الخميرة اثنين الهجين. وقد تم تعبئة هذه النواقل في سلالة Agrobacterium tumifaciens GV3101 عن طريق الكهرومporation. سلالة GV3101 لديه نزع سلاح تي بلازميد pMP90 (pTiC58DT-DNA) مع مقاومة جنتاميسين20. تمت إضافة سلالة البكتيريا الزراعية P19 جنبا إلى جنب مع جميع عمليات التسلل لمنع إسكات المتحولينجنسيا 21. نوصي بأن البروتينات الثلاثة يجب أن تستخدم أيضا في تشكيلات معاكسة للتحقق من صحة التفاعلات الثلاثية.
في هذه التقنية، قمنا بتوظيف FLIM، حيث أولا، يتم قياس عمر المضان للمتبرع (عمر المتبرع غير المروي) في غياب مقبول. بعد ذلك ، يتم قياس عمرها في وجود المقبول (عمر المتبرع المروي). يستخدم هذا الاختلاف في عمر الفلورسينس المانح لحساب كفاءة FRET ، والتي تعتمد على عدد الفوتونات التي تظهر انخفاضا في عمر الفلورية. المذكورة أدناه هو بروتوكول مفصل لتحديد تشكيل مجمع الترنية بين أي ثلاثة بروتينات من خلال التعبير عن عابر البروتينات الموسومة الفلورسنت في نيكوتيانا بنثاميانا وقول تفاعلها من قبل BiFC-FRET-FLIM.
Access restricted. Please log in or start a trial to view this content.
Protocol
1. استنساخ الجينات في ناقلات الدخول والوجهة (الشكل 2)
- تضخيم تسلسل الترميز (CDS) لجينات الفائدة (جينات M و C في حالتنا) بواسطة PCR واستنساخها في ناقلات الإدخال المناسبة (على سبيل المثال، pENTR/D-TOPO vector؛ انظر الجدول 1 للنواقل المستخدمة في هذه التجربة).
- زراعة المستنسخين على لوحات تحتوي على المضادات الحيوية. التحقق من صحة المستنسخات التي يتم تحديدها على المضادات الحيوية عن طريق تقييد الهضم وتسلسل الحمض النووي22،23.
- تعبئة CDSs المعاد تشكيلها من استنساخ الدخول إلى ناقلات الوجهة (pSPYNE-35S، pSPYCE-35S، pSITE-1CA و pSITE-3CA) وتأكيد نقل التسلسلات من الدخول إلى ناقلات الوجهة عن طريق تقييد هضم الإنزيم.
ملاحظة: يتم سرد كافة المتجهات المستخدمة في هذه التجربة في الجدول 1. - وأخيرا، تحويل Agrobacterium GV3101 (pMP90 (جنتR))الخلايا مع ناقلات الوجهة عن طريق الكهرومبوراسيون (الشكل 3)24.
2. ظروف النمو لنباتات نيكوتيانا بنثاميانا
ملاحظة: تنمو النباتات Nicotiana حتى 4-6 مرحلة ورقة في ظروف السيطرة.
- لزراعة نباتات نيكوتيانا، قم بإعداد مزيج التربة عن طريق خلط التربة المتاحة تجاريا مع cocopeat والسماد بنسبة 2:1:1.
- نشر طبقة سمكها بوصة واحدة من خليط التربة هذا في صينية بلاستيكية لجعل سرير التربة وتشبعه بالماء المتأين. رش حوالي 200 بذور في هذا السرير التربة.
- نقلها إلى صينية أكبر تحتوي على 1 سم من الماء الراكد. تغطية هذه الدرج مع غلاف بلاستيكي لإنشاء غرفة الرطوبة.
- نقل هذا الإعداد إلى غرفة النمو المحددة في 23 درجة مئوية مع 16 ساعة ضوء و 8 ساعة دورة مظلمة مع 150-170 μmol/m2ق كثافة الضوء.
- بعد أسبوعين، نقل الشتلات الشباب إلى الأواني الصغيرة، 3-4 بوصة تحتوي على مزيج التربة المشبعة بالمياه.
- ضع هذه الأواني في صواني بلاستيكية ونقلها إلى غرفة النمو لمدة أربعة أسابيع أخرى.
3. إعداد سلالات بكتيرية للتسلل الزراعي
ملاحظة: بالنسبة للتسلل الزراعي، تحتاج السلالات البكتيرية إلى أن تكون طازجة ومختلطة جنبا إلى جنب مع سلالة P19 من Agrobacterium بنسب مناسبة.
- إعداد لوحات أجار 2xYT التي تحتوي على ريفامبيسين (100 ميكروغرام / مل) ، جنتاميسين (25 ميكروغرام / مل) وكاناميسين (50 ميكروغرام / مل) ل Agrobacterium إيواء pSPYNE - 35S وpSPYCE - 35S ناقلات. بالنسبة لنواقل pSITE الذي يحتوي على سلالة، استخدم ريفامبيسين (100 ميكروغرام/مل)، والجنتاميسين (25 ميكروغرام/مل) وspectinomycin (50 ميكروغرام/مل).
- قم بشد سلالات Agrobacterium التي تحتوي على البلازميدات على هذه اللوحات باستخدام حلقات التطعيم العقيمة في غطاء تدفق صفح.
- احتضان هذه في 28 درجة مئوية لمدة 48 ساعة في الظلام.
- بدء هذا الإجراء عن طريق تلقيح Agrobacterium GV3101 سلالة إيواء BiFC و FRET يبني (أعدت في pSPYNE-35S، pSPYCE-35S وناقلات pSITE) من لوحات متتالية في 10 مل من مرق 2xYT تحتوي على المضادات الحيوية المناسبة (ريفامبيسين (100 ميكروغرام / مل)، جنتاميسين (25 ميكروغرام / مل)، الكاناميسين (50 ميكروغرام / مل) أو سبيكتينوميسين (50 ميكروغرام / مل)).
- بالإضافة إلى ذلك، بدء ثقافة سلالة P19 من Agrobacteria عن طريق تلقيح 10 مل من مرق 2xYT التي تحتوي على ريفامبيسين (100 ميكروغرام / مل) وكاناميسين (50 ميكروغرام / مل).
ملاحظة: يتم إضافة سلالة p19 لمنع إسكات المتحولين جنسيا. - تغطية قارورة مع رقائق الألومنيوم والاحتفاظ بها في شاكر حاضنة تعيين في 28 درجة مئوية و 170 دورة في الدقيقة لمدة 16 ساعة في الظلام.
- بعد النمو بين عشية وضحاها، نقل 1 مل من هذه الثقافة إلى cuvette المتاح لقياس الكثافة البصرية (O.D.) من الثقافات في 600 نانومتر باستخدام مطياف.
- امزج بين ثقافات الشريك المناسب BiFC و FRET الذي يحتوي على سلالات بحيث يكون O.D. النهائي لكل ثقافة هو 0.5 و أن p19 هو 0.3 في حجم إجمالي قدره 2 مل.
- لتحقيق هذه النسب، استخدم الصيغة المذكورة أدناه:
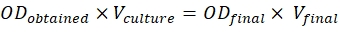
ODالتي تم الحصول عليها = O.D. للثقافة قياسها في 600 نانومتر
V الثقافة = حجم الثقافة المطلوبة
OD النهائي = 0.5 للبناءات و 0.3 لp19
Vالنهائي = الحجم النهائي للتسلل، وهو 2 مل
ملاحظة: يتم تحديد تركيبات البناء المستخدمة في هذه الدراسة في الجدول 2. - الطرد المركزي الثقافات Agrobacterium مختلطة في 3000 × ز لمدة 5 دقائق في درجة حرارة الغرفة وتجاهل بعناية supernatant. Resuspend بيليه في 2 مل من العازلة تسلل أعدت حديثا (10 MM MES، 100 ميكرومتر من Acetosyringone، و 10 mM MgCl2). استخدام خلاط دوامة لجعل تعليق الخلية متجانسة.
- احتضان الأنابيب التي تحتوي على خلايا إعادة الإنفاق في الظلام في درجة حرارة الغرفة لمدة 3 ساعة.
- وفي الوقت نفسه، تسمية كل وعاء النبات مع خليط بناء أنها ستكون تسللت مع. استخدام اثنين من النباتات لكل خليط التسلل.
- ملء حقنة 1 مل إبرة مع مزيج البكتيريا الزراعية. بلطف ولكن بحزم اضغط على حقنة على الجانب abaxial من ورقة موسعة تماما في حين دعم ورقة من الجانب الآخر. ادفع المكبس برفق حتى تمتلئ الحلول في منطقة الورقة بما يعادل 2-3 أضعاف طرف الحقنة.
- التسلل إلى ما يصل إلى أربعة بقع على ورقة و 3-4 أوراق لكل نبات، كما هو مبين في الشكل 4.
ملاحظة: تغيير القفازات أو مسح القفازات مع الكحول 70٪ بين العينات لمنع التلوث المتبادل. - نقل جميع الأواني إلى صينية واحتضان في غرفة النمو في ظل نفس الشروط المذكورة في الخطوة 2.
- تحقق من جزء صغير من ورقة agroinfiltrated في نقاط زمنية مختلفة باستخدام المجهر مضان. عندما تكون الفلورية من كل من YFP وCFP قابلة للكشف في الخلايا ، انتقل إلى المجهر الكونفوكوكال لإجراء فحص BiFC-FRET FLIM. في هذه التجربة، تم إجراء التحليل بعد 3 أيام من التسلل الزراعي.
ملاحظة: تعيين فترة ما بعد الترشيح الزراعي من الحضانة بشكل فردي لكل المروج والجمع بين الجينات لتجنب الإفراط في التعبير عن البروتينات الشيمية المستخدمة في المقايسة BiFC-FRET FLIM. قد يؤدي التعبير المفرط عن البروتينات الشريكة إلى تفاعلات إيجابية زائفة.
4. إعداد الشرائح لتصور مضان
- عندما تكون النباتات جاهزة للتصور، وقطع عينات ورقة مربعة، 5-8 ملم بعيدا عن الجرح التسلل، وجبل لهم في الماء المقطر على الشرائح النظيفة.
ملاحظة: لتقليل الإضاءة الخلفية، قم بتنظيف الشرائح باستخدام الإيثانول بنسبة 80٪ متبوعا بالماء المقطر 3-4 مرات، وتجفيف الهواء، والاحتفاظ بها على ورقة ماصة. - قم بتغطية عينة الورق بغطاء نظيف وختم باستخدام مينا الأظافر.
- تصور هذه العينات تحت المجهر الليزر confocal المسح الضوئي.
5. FRET - FLIM تحليل باستخدام المجهر المسح بالليزر confocal
ملاحظة: في هذا الإجراء، يكون أساس تحديد وقياس التفاعل بين بروتينين هو تقليل عمر الفلورية للشريك المانح FRET عند تفاعله مع المقبول، والذي يستخدم لحساب كفاءة FRET. يزداد التعقيد في حالة التفاعل الثلاثي أكثر لأن FRET-acceptor ، في هذه الحالة ، ليس جزيءا واحدا ولكنه زوج YFP-BiFC منقسم ، والذي يجب أولا إعادة تشكيله في الجسم الحي ليصبح فلوريا وظيفيا من FRET-acceptor. لتنفيذ FRET-FLIM، يحتاج المرء إلى تحديد مضان جزيء المانحة مدى الحياة أولا وحدها ومن ثم في وجود شريك FRET.
- افتح تطبيق FLIM في مجهر المسح بالليزر confocal ، وابدأ وحدة التحكم واستخدم العد الضوئي للتعرف على الأنماط لقياس عمر الفلورسينس. حدد وضع قياس "عد جميع فوتونات" القياسي.
- تحليل عينات من نوعين من النباتات الزراعية المتسللة: واحد مع المانح فقط (C-CFP) والآخر مع المانح والمقبل (C-CFP، جنبا إلى جنب مع M-YFP) على حد سواء.
ملاحظة: نظرا لأن تفاعل بروتين M قد تم التحقق من صحته بالفعل باستخدام بروتين C باستخدام BiFC وY2H ، فمن المتوقع كفاءة FRET جيدة مع هذا الزوج المتفاعل. - بعد ذلك ، قم بمسح ورقة C-CFP الزراعية المتسللة والتركيز على خلية تظهر فلورية CFP جيدة. بدء وضع المسح بالليزر وتعيين نظام التصور CFP وقياسات FLIM(λ ex 440 نانومتر نبض الليزر, λم 480-520 نانومتر بواسطة أجهزة الكشف الهجين, مسح سرعة 512 × 512 بكسل في 400 هرتز).
- ضبط التركيز والتكبير والربح الذكي للتركيز على المنطقة التي تحتاج إلى التقاط.
- قم بإضاءة العينة بقوة ليزر كافية لتحقيق التقاط ما يقرب من 0.1 فوتون لكل نبضة. بالنسبة للعينات ذات كثافة الفلورية المتغيرة، التقط 50 إطارا لجمع الفوتونات الكافية اللازمة لقياس مدى الحياة. معرض CFP اثنين من العمر مضان بسبب التكيف معها تشكيلية; لذلك، احتواء البيانات باستخدام نموذج إعادة تعقيدا n أسي مع الاحتفاظ قيمة n يساوي 2.
- في هذه الإعدادات، يظهر CFP عمرين من 1.0 و 3.2 ns. هنا أعلى، 3.2 ns، يستخدم مدى الحياة لجميع الحسابات اللاحقة25،26.
- لحساب كفاءة FRET باستخدام FLIM ، وهو مقياس لدرجة التفاعل بين اثنين من البروتينات ، خذ عينة ورقة تم اختراقها مع C-CFP و M-YFP. ابحث عن الخلية التي تعبر عن كل من C-CFP و M-YFP وتأكيد أنماط الانبعاثات الخاصة بهم من خلال إثارة لهم باستخدام λex 440 نانومتر نبض الليزر, λem 480-520 نانومتر وλex 514 nm ليزر الضوء الأبيض مع λem 526-550 نانومتر. مسح وتحديد خلية تظهر كل من CFP وYFP مضان.
- بعد تأكيد الفلورسينس من كل من البروتينات، انتقل إلى وحدة التحكم FLIM لقياس عمر CFP باستخدام نفس الإعدادات التي استخدمناها في وقت سابق لقياس عمر C-CFP (الخطوة 5.5).
ملاحظة: هذه الخلية أيضا التعبير عن M-YFP التي يمكن أن تتفاعل مع C-CFP وتسبب انخفاضا في عمر C-CFP. - احتواء الرسم البياني الذي تم الحصول عليه باستخدام نموذج إعادة تشكيل n أسي، مع n = 2. لوحظ انخفاض في عمر CFP من 3.2 إلى 2.6 ns ، مما يشير إلى نقل الطاقة بالرنين Förster بين CFP و YFP (الشكل 5A).
- الآن بدء وحدة التحكم FRET في البرنامج وحساب كفاءة FRET عن طريق إدخال يدويا عمر المانحة غير مروي في المعادلة المقدمة في البرنامج. وكفاءة FRET الملاحظة هي: 56٪.
- التفاعل الثلاثي
- وأخيرا، لتصور التفاعلات بين ثلاثة شركاء، خذ عينة ورقة من النبات الذي تم اختراقه مع C-CFP، M-YFPn، و M-YFPc.
- مسح explant ورقة لخلية التي تظهر كل من CFP وإعادة تشكيلها YFP مضان المنبثقة من التفاعل BiFC بين اثنين من البروتينات M. استخدم نفس أطوال الليزر والانبعاثات كما استخدمت سابقا.
- بعد ذلك، قم بإيقاف تشغيل الليزر 514 نانومتر والانتقال إلى وحدة التحكم FLIM.
ملاحظة: إذا كان Dimer M-YFP يتفاعل مع C-CFP، يجب أن يرى المرء انخفاضا في عمر C-CFP كما لوحظ أثناء تفاعله مع M-YFP. ومع ذلك ، إذا فشل C-CFP في التفاعل مع جهاز M-YFP dimer ، فيجب أن يبقى عمر الفلورسينس عند 3.2 ns. - باستخدام إعدادات مماثلة كما ذكر أعلاه، قياس عمر CFP في وجود YFP المعاد تشكيلها. احتواء الرسم البياني الذي تم الحصول عليه باستخدام نموذج إعادة تعقيدا n-الأسي مع n = 2 ثم الانتقال إلى وحدة التحكم FRET.
ملاحظة: هناك انخفاض في عمر CFP من 3.2 إلى 2.3 ns. حساب كفاءة FRET كما هو موضح أعلاه. كفاءة FRET المحسوبة هي 55٪. ويؤكد انخفاض عمر المتبرع وكفاءة FRET الجيدة بنسبة 55٪ التفاعل الثلاثي بين اثنين من البروتينات M وبروتين C في الجسم الحي (انظر الشكل 5B).
Access restricted. Please log in or start a trial to view this content.
النتائج
يمثل هذا البروتوكول طريقة محسنة للدراسة في التفاعلات الثلاثية للبروتين والبروتين في النباتات. المبدأ الأساسي للبروتوكول هو الجمع بين اثنين من تقنيات التفاعل البروتين الموسومة بالفلورسينس، أي BiFC و FRET، لإنشاء مقايسة لقياس التكوين المعقد الترني بين ثلاثة شركاء البروتين. هنا، استخدمن...
Access restricted. Please log in or start a trial to view this content.
Discussion
ويبين هذا البروتوكول استخدام المقايسة FRET-FLIM التي تستند إلى BIFC للتأكد من تكوين مجمع ترنيري بين مونومرين من بروتين MADS-box وبروتين مستشعر الكالسيوم. تم تكييف البروتوكول من تقرير من قبل Y. جون شيو وآخرون حيث طوروا طريقة FRET المستندة إلى BiFC لتصور المجمع الترنيمي الذي تم تشكيله بين heterodimers Fos-Jun و NFAT ?...
Access restricted. Please log in or start a trial to view this content.
Disclosures
ولا يعلن صاحبا البلاغ عن وجود تضارب في المصالح.
Acknowledgements
NB، GG، SB، KC أشكر بإخلاص لجنة المنح الجامعية (UGC)، UGC-BSR، DBT-INSPIRE، ومجلس البحوث العلمية والصناعية (CSIR) لزمالاتهم البحثية. ولحسن الحظ، نعترف بإدارة التكنولوجيا الأحيائية، وحكومة الهند، وإدارة العلوم والتكنولوجيا، وحكومة الهند، للحصول على الدعم المالي.
Access restricted. Please log in or start a trial to view this content.
Materials
| Name | Company | Catalog Number | Comments |
| 1 ml Syringes without needles | Dispovan | - | |
| Acetosyringone | Sigma-Aldrich | D134406 | |
| Gateway LR Clonase II Enzyme mix | Thermo Fischer Scientific | 11791020 | The vectors used in the study are Gateway based |
| Gentamycin Sulphate | Himedia | CMS461 | |
| Kanamycin Sulphate | Himedia | MB105 | |
| MES hydrate | Sigma-Aldrich | M2933 | |
| MgCl2 | Sigma-Aldrich | M2670 | |
| pENTR/D-TOPO Cloning Kit | Thermo Fischer Scientific | K240020 | The vectors used in the study are Gateway based |
| Phusion high fidelity Taq DNA polymerase | Thermo Fischer Scientific | F530-S | Any High fidelity Polymerase can work |
| Rifampicin | Himedia | CMS1889 | |
| SP8 FALCON Confocal laser scanning microscope | Leica | SP8 FALCON | Any CLSM with FLIM capabilities can be used for this analysis |
| Spectinomycin dihydrochloride pentahydrate | Himedia | TC034 |
References
- Grove, C. A., Walhout, A. J. M. Transcription factor functionality and transcription regulatory networks. Molecular Biosystem. (4), 309-314 (2008).
- Amoutzias, G. D., Robertson, D. L., Van de Peer, Y., Oliver, S. G. Choose your partners: dimerization in eukaryotic transcription factors. Trends in Biochemical Sciences. 33 (5), 220-229 (2008).
- Arora, R., et al. MADS-box gene family in rice: genome-wide identification, organization and expression profiling during reproductive development and stress. BMC Genomics. 8 (242), (2007).
- Theißen, G., Gramzow, L. Structure and evolution of plant MADS domain transcription factors. Plant Transcription Factors: Evolutionary, Structural and Functional Aspects. , 127-138 (2016).
- Schweiger, R., Schwenkert, S. Protein-protein interactions visualized by bimolecular fluorescence complementation in tobacco protoplasts and leaves. Journal of Visualized Experiments: JoVE. (85), (2014).
- Bracha-Drori, K., et al. Detection of protein-protein interactions in plants using bimolecular fluorescence complementation. Plant Journal. 40 (3), 419-427 (2004).
- Shyu, Y. J., Suarez, C. D., Hu, C. Visualization of ternary complexes in living cells by using a BiFC-based FRET assay. Nature Protocols. 3 (11), 1693-1702 (2008).
- Kwaaitaal, M., Keinath, N. F., Pajonk, S., Biskup, C., Panstruga, R. Combined bimolecular fluorescence complementation and förster resonance energy transfer reveals ternary SNARE complex formation in living plant cells. Plant Physiology. 152 (3), 1135-1147 (2010).
- Margineanu, A., et al. Screening for protein-protein interactions using Förster resonance energy transfer (FRET) and fluorescence lifetime imaging microscopy (FLIM). Scientific Reports. 6, (2016).
- Datta, R., Heaster, T. M., Sharick, J. T., Gillette, A. A., Skala, M. C. Fluorescence lifetime imaging microscopy: fundamentals and advances in instrumentation, analysis, and applications. Journal of Biomedical Optics. 25 (07), 1(2020).
- Long, Y., et al. Optimizing FRET-FLIM labeling conditions to detect nuclear protein interactions at native expression levels in living Arabidopsis roots. Frontiers in Plant Science. 9, 1-13 (2018).
- Burman, N., Chandran, D., Khurana, J. P. A Rapid and highly efficient method for transient gene expression in rice plants. Frontiers in Plant Science. , 11(2020).
- Kudla, J., Bock, R. Lighting the way to protein-protein interactions: Recommendations on best practices for bimolecular fluorescence complementation analyses. Plant Cell. 28 (5), 1002-1008 (2016).
- Shyu, Y. J., Hu, C. D. Fluorescence complementation: an emerging tool for biological research. Trends in Biotechnology. 26 (11), 622-630 (2008).
- Kodama, Y., Hu, C. D. An improved bimolecular fluorescence complementation assay with a high signal-to-noise ratio. BioTechniques. 49 (5), 793-803 (2010).
- Kodama, Y., Hu, C. D. Bimolecular fluorescence complementation (BiFC): A 5-year update and future perspectives. BioTechniques. 53 (5), 285-298 (2012).
- Piston, D. W., Kremers, G. J. Fluorescent protein FRET: the good, the bad and the ugly. Trends in Biochemical Sciences. 32 (9), 407-414 (2007).
- Chakrabarty, R., et al. pSITE vectors for stable integration or transient expression of autofluorescent protein fusions in plants: Probing Nicotiana benthamiana- Virus Interactions. Molecular Plant-Microbe Interactions. 20 (7), 740-750 (2007).
- Walter, M., et al. Visualization of protein interactions in living plant cells using bimolecular fluorescence complementation. Plant Journal. 40 (3), 428-438 (2004).
- Hellens, R., Mullineaux, P., Klee, H. A guide to Agrobacterium binary Ti vectors. Trends in Plant Science. 5 (10), 446-451 (2000).
- Van Der Hoorn, R. A. L., Rivas, S., Wulff, B. B. H., Jones, J. D. G., Joosten, M. H. A. J. Rapid migration in gel filtration of the Cf-4 and Cf-9 resistance proteins is an intrinsic property of Cf proteins and not because of their association with high-molecular-weight proteins. Plant Journal. 35 (3), 305-315 (2003).
- Xie, X., et al. Engineering SARS-CoV-2 using a reverse genetic system. Nature Protocols. 16, (2021).
- Xu, J., et al. Optimized plasmid construction strategy for Cas9. Cellular Physiology and Biochemistry. 48, 131-137 (2018).
- Mattanovich, D., et al. Efficient transformation of Agrobacterium spp. by electroporation. Nucleic Acids Research. 17 (16), 6747(1989).
- Rizzo, M. A., Springer, G. H., Granada, B., Piston, D. W. An improved cyan fluorescent protein variant useful for FRET. Nature Biotechnology. 22 (4), 445-449 (2004).
- Tramier, M., et al. Picosecond-hetero-FRET microscopy to probe protein-protein interactions in live cells. Biophysical Journal. 83 (6), 3570-3577 (2002).
- Alvarez, L. A. J., et al. SP8 FALCON: a novel concept in fluorescence lifetime imaging enabling video-rate confocal FLIM. Nature Methods. 20, 2-4 (2019).
- Postma, M., Goedhart, J. Plotsofdata-a web app for visualizing data together with their summaries. PLoS Biology. 17 (3), 1-8 (2019).
- Galperin, E., Verkhusha, V. V., Sorkin, A. Three-chromophore fret microscopy to analyze multiprotein interactions in living cells. Nature Methods. 1 (3), 209-217 (2004).
- Waadt, R., Kudla, J. In plant visualization of protein interactions using bimolecular fluorescence complementation (BiFC). Cold Spring Harbor Protocols. 3 (4), (2008).
Access restricted. Please log in or start a trial to view this content.
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved