A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
קביעת אינטראקציה משולשת בין שני מונומרים של גורם שעתוק של קופסת MADS וחלבון חיישן סידן על ידי BiFC-FRET-FLIM Assay
* These authors contributed equally
In This Article
Summary
כאן אנו מציגים, שיטה לדמיין היווצרות מורכבת טרנרית בין שלושה שותפי חלבון באמצעות חלבונים מתויגים פלואורסצנטית על ידי BiFC מבוסס FRET-FLIM assay. שיטה זו היא בעלת ערך לחקר מתחמי אינטראקציה בין חלבון חלבון ב- vivo.
Abstract
אינטראקציות בין חלבונים לחלבון הן חלק בלתי נפרד מכל התהליכים הביולוגיים בתאים, שכן הן ממלאות תפקיד מכריע בוויסות, תחזוקה ותיקון של תפקודי התא. אינטראקציות אלה מעורבות במגוון רחב של תופעות כגון העברת אותות, תגובת פתוגן, אינטראקציות בין תאים, תהליכים מטבוליים והתפתחותיים. במקרה של גורמי שעתוק, אינטראקציות אלה עשויות להוביל אוליגומריזציה של subunits, הפרדה בהקשרים תת-תאיים ספציפיים כגון הגרעין, ציטופלסמה, וכו ', אשר, בתורו, עשוי להיות השפעה עמוקה יותר על הביטוי של הגנים במורד הזרם. כאן, אנו מדגימים מתודולוגיה כדי לדמיין אינטראקציה משולשת vivo באמצעות השלמת פלואורסצנטיות דו-מולקולרית (BiFC) מבוסס Förster העברת אנרגיה תהודה (FRET) מעורבים הדמיה לכל החיים פלואורסצנטי (FLIM). שניים מהחלבונים שנבחרו להדגמה זו מתקשרים כשותפים של BiFC, ופעילות הפלואורסצנטיות המחודשת שלהם משמשת לבדיקת FRET-FLIM עם השותף השלישי. צמחים בני ארבעה עד חמישה שבועות בני 4 עד 5 שבועות של צמחים מגידול תאית ניקוטיאנה בנתמיאנה שימשו כמערכת הצמחים לדוגמה להדגמה זו.
Introduction
אינטראקציות חלבון-חלבון (PPIs) מהווים את הבסיס לתפקוד תקין של התאים האוקריוטים על ידי ויסות תהליכים מטבוליים והתפתחותיים שונים. חלק מה- PPIs יציבים, בעוד שאחרים ארעיים בטבע. האינטראקציות עשויות להיות מסווגות בהתבסס על מספר וסוג החברים באינטראקציה כגון דימריק, טרימרי, הומומרי טטרמרי והטרומרי1. זיהוי ואפיון של אינטראקציות חלבון עשוי להוביל להבנה טובה יותר של פונקציות חלבון ורשתות רגולטוריות.
גורמי שעתוק הם חלבונים המעורבים בתפקודים רגולטוריים. הם מווסתים את קצב התמלול של הגנים במורד הזרם שלהם על ידי קשירה לדנ"א. לפעמים אוליגומריזציה או היווצרות של מתחמים מסדר גבוה יותר על ידי חלבונים הוא תנאי מוקדם לביצוע הפונקציות שלהם2. גורמי שעתוק של קופסת MADS הם גנים הומיאוטיים המווסתים תהליכים שונים כגון מעבר פרחוני, פיתוח איברים פרחוניים, הפריה, התפתחות זרעים, סנסנס והתפתחות וגטטיבית. הם ידועים ליצור מתחמים מסדר גבוה יותר אשר נקשרים ל- DNA3,4. לימוד רשתות PPI בין גורמי תמלול והאינטראקטיביות שלהם מספק תובנות לגבי המורכבות שבבסיס רגולציית התמלול.
ביטוי חלבון חולף בניקוטיאנה בנתמיאנה היה גישה פופולרית לחקר לוקליזציה של חלבונים או אינטראקציות חלבון-חלבון ב- vivo5. BiFC ו- FRET הן שיטות לחקר אינטראקציות חלבון-חלבון ב- vivo באמצעות מערכות עיתונאי פלואורסצנטיות6. שילוב של שתי טכניקות אלה הוכח לחשוף את האינטראקציה בין שלושה חלבונים7. FRET נמדד באמצעות הדמיה מקבלת, פליטה רגישה וטכניקת הדמיה לכל החיים של פלואורסצנטיות (FLIM). בדיקת FRET מבוססת FLIM התפתחה ככלי המספק כימות מדויק וספציפיות מרחבית למדידות העברת אנרגיה בין שתי מולקולות המבוססות על אורך החיים הפלואורסצנטי שלהם8. FLIM מודד את הזמן פלואורופור נשאר במצב נרגש לפני פולט פוטון והוא טוב יותר מאשר טכניקות המשתמשות מדידות אינטנסיביות לבד9,10. מלבד מערכות הטרולוגיות כגון Nicotiana benthamiana וקליפות אפידרמיס בצל, דיווחים אחרונים הראו את השימוש בשורשים Arabidopsis ושתילי אורז צעירים, וכו ', לניתוח ויוו של אינטראקציות חלבון חלבון בתנאים מקומיים11,12.
מלבד מערכת ביטוי מתאימה, הבחירה של שותפים אינטראקציה עבור BiFC ו FRET גיוסים הוא גם חיוני להצלחת הניסוי הזה. יש לאמת את ה- PPI בין השותפים המשמשים בתצורת BiFC באמצעות פקדים מתאימים לפני השימוש בהם כשותף מצומד בניסוי FRET13. BiFC מנצל את ההשלמה המבנית של חלקים N-ו- C-מסוף של חלבון פלורסנט. מגבלה נפוצה ברוב החלבונים הפלואורסצנטיים המשמשים בבוחות BiFC הייתה ההרכבה העצמית בין שני שברים נגזרים שאינם פלואורסצנטיים, מה שתרם לפלורסצנטיות חיובית כוזבת ומקטין את יחס האות לרעש (S/N)14. התפתחויות אחרונות, כולל מוטציות נקודתיות או מיקום של פיצול חלבון פלואורסצנטי, הולידו זוגות BiFC עם עוצמה מוגברת, ספציפיות גבוהה יותר, יחס S / N גבוה15,16. חלבונים פלואורסצנטיים אלה יכולים לשמש גם לביצוע BiFC בהתאם להתאמת הניסוי.
באופן מסורתי, CFP ו- YFP שימשו כזוג התורם והמקבל בניסויי FRET17. עם זאת, YFP או m-Citrine נמצאו תורמי FRET טובים יותר (כאשר נעשה שימוש עם RFP כמקובל) בגלל התשואה הקוונטית הגבוהה (QY) במהלך הביטוי הטבעי של חלבוני היעד במערכת השורש Arabidopsis. הבחירה של מקדמים (מרכיב לעומת יליד / אנדוגני) פלואורופור גם לשחק תפקיד מכריע בעיצוב ניסוי BiFC-FRET-FLIM מוצלח. חשוב לציין כי היעילות של תורמי FRET והתאמתם של זוגות FRET נוטים להשתנות עם השינוי במקדם ובמערכת הביולוגית המשמשת לביטוי. ה- QY של הפלורופור, המתייחס לבהירותו, תלוי ב- pH, בטמפרטורה ובמערכת הביולוגית הנמצאת בשימוש. אנו מציעים כי קריטריונים אלה ישקלו ביסודיות לפני בחירת זוג פלואורופור לניסוי FRET. המערכת הביולוגית, היזמים והחלבונים המשמשים לפרוטוקול זה עבדו היטב עם פלואורופורים CFP-YFP לניסוי BiFC FRET-FLIM.
במחקר הנוכחי, אנו משלבים את התכונה של FLIM כדי לדמיין את האינטראקציה בין שלוש מולקולות חלבון באמצעות FRET מבוסס BiFC. בטכניקה זו, שני חלבונים מתויגים עם חלבון YFP מפוצל ואת החלבון השלישי עם CFP. מכיוון שהתעניינו בחקר האינטראקציה של הומודימר חלבון תיבת MADS (M) עם חלבון חיישן סידן (C), חלבונים אלה תויגו עם חלבונים פלואורסצנטיים בווקטורים pSITE-1CA ו- pSITE-3CA18. שניים מהשותפים לאינטראקציה, ב-assay זה, תויגו עם חלקים N-ו- C-terminal של YFP ב pSPYNE-35S ו- pSPYCE-35S וקטורים19, והאינטראקציה שלהם גורמת לחזרה של YFP פונקציונלי הפועל כקבל FRET לשותף האינטראקציה השלישי, אשר מתויג עם CFP (מתנהג כתורם FRET) (איור 1 ). במקרה מסוים זה, ה- PPI בין שני מונומרים M ובין M ו- C אומת על ידי ביצוע BiFC בשלוש מערכות שונות יחד עם מערכת שמרים-שתיים-היברידית. וקטורים אלה גויסו לתוך זן אגרובקטריום tumifaciens GV3101 על ידי אלקטרופורציה. זן GV3101 יש pMP90 טי מנוטרל (pTiC58DT-DNA) עם התנגדות גנטמיצין20. זן אגרובקטריום p19 נוסף יחד עם כל ההסתננויות כדי למנוע השתקת transgene21. אנו ממליצים כי שלושת החלבונים ישמשו גם בקונפורמציות הפוכות כדי לאמת את האינטראקציות המשולשות.
בטכניקה זו, השתמשנו FLIM, שבו הראשון, חיי הפלואורסצנטיות של התורם (חיים תורם ללא יראתי) נמדד בהיעדר מקבל. לאחר מכן, חייו נמדדים בנוכחות המקבל (אורך חיים של תורם מרווה). הבדל זה במאסרי החיים של פלואורסצנטיות התורם משמש לחישוב יעילות FRET, אשר תלוי במספר הפוטונים המציגים ירידה בחיי הפלואורסצנטיות. להלן פרוטוקול מפורט כדי לקבוע את היווצרותו של קומפלקס טרנרי בין כל שלושה חלבונים על ידי ביטוי חולף של חלבונים מתויגים פלואורסצנטית ב- Nicotiana benthamiana ו- assaying האינטראקציה שלהם על ידי BiFC-FRET-FLIM.
Protocol
1. שיבוט גנים בווקטורים של כניסה ויעד (איור 2)
- להגביר את רצף הקידוד (CDS) של הגנים מעניינים (גנים M ו- C במקרה שלנו) על ידי PCR ולשכפל אותם בווקטורים כניסה מתאימים (למשל, וקטור pENTR / D-TOPO; ראה טבלה 1 עבור וקטורים המשמשים בניסוי זה).
- לגדל את השיבוטים על צלחות המכילות אנטיביוטיקה. לאמת שיבוטים שנבחרו על אנטיביוטיקה על ידי הגבלת העיכול ורצף DNA22,23.
- גייסו את התקליטורים המשוחזרים משיבוטי כניסה לווקטורים המשמשים כיעד (pSPYNE-35S, pSPYCE-35S, pSITE-1CA ו-pSITE-3CA) ואשרו את העברת הרצפים מהכניסה לווקטורים היעד על ידי הגבלת עיכול אנזימים.
הערה: כל הווקטורים המשמשים בניסוי זה מפורטים בטבלה 1. - לבסוף, להפוך את התאים אגרובקטריום GV3101 (pMP90 (GentR))עם וקטור היעד על ידי אלקטרופורציה (איור 3)24.
2. תנאי צמיחה לצמחים של ניקוטיאנה בנתמיאנה
הערה: לגדל צמחי Nicotiana עד שלב 4-6 עלים בתנאי בקרה.
- כדי לגדל צמחי Nicotiana, להכין את תערובת הקרקע על ידי ערבוב אדמה זמין מסחרית מתערבב עם cocopeat וקומפוסט ביחס של 2:1:1.
- מורחים שכבה בעובי 1 אינץ' של תערובת אדמה זו במגש פלסטיק כדי להפוך את מיטת הקרקע ולהרוות אותה במים דה-יוניים. מפזרים כ -200 זרעים במיטת האדמה הזו.
- מעבירים אותו למגש גדול יותר המכיל 1 ס"מ של מים עומדים. מכסים מגש זה בניילון נצמד ליצירת תא לחות.
- העבר את זה להגדיר לתא צמיחה להגדיר ב 23 °C (50 °F) עם 16 שעות אור 8 שעות מחזור כהה עם 150-170 מיקרומול / מ'2עוצמת האור.
- לאחר שבועיים, מעבירים שתילים צעירים לסירים קטנים בגודל 3-4 אינץ' המכילים תערובת אדמה רוויה במים.
- מניחים את הסירים האלה במגשי פלסטיק ומעבירים אותם לתא הצמיחה לארבעה שבועות נוספים.
3. הכן זני חיידקים לחדירת אגרו
הערה: עבור חדירות חקלאית, זנים חיידקיים צריכים להיות טריים תת תרבותית מעורבב יחד עם זן p19 של אגרובקטריום ביחסים מתאימים.
- הכן 2xYT צלחות אגר המכילות ריפים (100 מיקרוגרם / מ"ל), גנטמיצין (25 מיקרוגרם / מ"ל) וקנמיצין (50 מיקרוגרם / מ"ל) עבור אגרובקטריום מחסה pSPYNE-35S ו וקטור pSPYCE-35S. עבור וקטור pSITE המכיל זן, השתמש רימפיצין (100 מיקרוגרם / מ"ל), גנטמיצין (25 מיקרוגרם / מ"ל) ו spectinomycin (50 מיקרוגרם / מ"ל).
- פס זני אגרובקטריום המכילים את plasmids על לוחות אלה באמצעות לולאות חיסון סטריליות במכסה המנוע זרימה למינאר.
- לדגור אלה ב 28 °C (55 °F) עבור 48 שעות בחושך.
- התחל הליך זה על ידי חיסון אגרובקטריום GV3101 זן מחסה BiFC ו- FRET מבנים (מוכן pSPYNE-35S, pSPYCE-35S ו ו- pSITE וקטורים) מלוחות מפוספסים ב 10 מ"ל של מרק 2xYT המכיל אנטיביוטיקה מתאימה (רימפיצין (100 מיקרוגרם / מ"ל), גנטמיצין (25 מיקרוגרם / מ"ל), kanamycin (50 מיקרוגרם / מ"ל) או spectinomyצין (50 מיקרוגרם / מ"ל)).
- בנוסף, ליזום תרבות של זן p19 של אגרובקטריה על ידי חיסון 10 מ"ל של מרק 2xYT המכיל ריפהמפיצין (100 מיקרוגרם / מ"ל) וקנמיצין (50 מיקרוגרם / מ"ל).
הערה: זן p19 מתווסף כדי למנוע השתקה transgene. - מכסים את הבקבוקון בנייר אלומיניום ושומרים אותם בשייקר האינקובטור שנקבע ב-28 מעלות צלזיוס ו-170 סל"ד ל-16 שעות בחושך.
- לאחר הצמיחה הלילית, העבר 1 מ"ל של תרבות זו ל cuvette חד פעמי כדי למדוד את הצפיפות האופטית (O.D.) של התרבויות ב 600 ננומטר באמצעות ספקטרופוטומטר.
- ערבבו את התרבויות של שותף BiFC ו- FRET מתאים המכיל זנים כך שה- O.D. הסופי של כל תרבות הוא 0.5 וזה של p19 הוא 0.3 בנפח כולל של 2 מ"ל.
- כדי להשיג יחסים אלה, השתמש בנוסחה המוזכרת להלן:
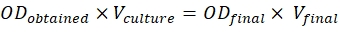
ODשהושג = מנת יתר של התרבות נמדדת ב 600 ננומטר
תרבות V = נפח התרבות הנדרשת
ODסופי = 0.5 למבנים ו- 0.3 עבור עמ ' 19
V סופי = נפח סופי עבור החדירה, שהוא 2 מ"ל
הערה: שילובי הבנייה המשמשים במחקר זה מצוינים בטבלה 2. - צנטריפוגות תרביות אגרובקטריום מעורבות ב 3,000 x ז במשך 5 דקות בטמפרטורת החדר בזהירות להשליך את supernatant. resuspend הכדור ב 2 מ"ל של חיץ חדירה טרי מוכן (10 mM MES, 100 מיקרומטר של Acetosyringone, ו 10 מ"מ MgCl2). השתמש מערבל מערבולת כדי להפוך השעיית תא הומוגני.
- לדגור על הצינורות המכילים תאים resuspended בחושך בטמפרטורת החדר במשך 3 שעות.
- בינתיים, לתייג כל סיר צמח עם תערובת המבנה זה הולך להיות הסתנן עם. השתמש בשני צמחים עבור כל תערובת חדירה.
- מלא מזרק 1 מ"ל ללא מחט עם תערובת אגרובקטריאלית. לחץ בעדינות אך בחוזקה את המזרק על הצד האבקסיאלי של העלה המורחב במלואו תוך תמיכה בעליה מהצד השני. דוחפים בעדינות את הבוכנה עד שהפתרונות מתמלאים באזור העלה שווה ערך לפי 2-3 מקצה המזרק.
- הסתננו עד ארבעה כתמים על עלה ו-3-4 עלים לצמח, כפי שמוצג באיור 4.
הערה: החליפו כפפות או ניגנו כפפות עם 70% אלכוהול בין דגימות כדי למנוע זיהום צולב. - מעבירים את כל הסירים למגש ודגרה בתא צמיחה באותם תנאים שהוזכרו בשלב 2.
- בדוק חלק קטן של העלה agroinfiltrated בנקודות זמן שונות באמצעות מיקרוסקופ פלואורסצנטי. כאשר הפלואורסצנטיות הן מ- YFP והן מ- CFP ניתנות לזיהוי בתאים, המשך למיקרוסקופ הקונפוקליבי לבדיקת BiFC-FRET FLIM. בניסוי זה, הניתוח בוצע 3 ימים לאחר הסתננות אגרו.
הערה: הגדר את תקופת ההדירה שלאחר אגרואין בנפרד עבור כל מקדם ושילוב גנים כדי למנוע ביטוי יתר של חלבונים כימריים המשמשים ב- BiFC-FRET FLIM. ביטוי יתר של חלבוני השותף עלול להוביל לאינטראקציות שווא-חיוביות.
4. הכן שקופיות להדמיה פלואורסצנטית
- כאשר הצמחים מוכנים להדמיה, לחתוך דגימות עלים מרובעים, 5-8 מ"מ מן פצע החדירה, ולהרכיב אותם במים מזוקקים על מגלשות נקיות.
הערה: כדי למזער פלואורסצנטיות ברקע, לנקות את השקופיות עם 80% אתנול ואחריו מים מזוקקים 3-4 פעמים, אוויר לייבש אותם, ולשמור אותם על סדין סופג. - מכסים את דגימת העלים בכיסוי נקי וחותמים באמצעות אמייל ציפורניים.
- דמיינו את הדגימות האלה תחת מיקרוסקופ סריקת לייזר קונפוקלי.
5. ניתוח FRET-FLIM באמצעות מיקרוסקופ סריקת לייזר קונפוקלי
הערה: בהליך זה, הבסיס של קביעת וכימות אינטראקציה בין שני חלבונים הוא הירידה אורך החיים פלואורסצנטי של השותף תורם FRET על האינטראקציה שלה עם הקבלה, אשר משמש לחישוב היעילות של FRET. המורכבות במקרה של אינטראקציה משולשת גדלה עוד יותר מכיוון ש- FRET-acceptor, במקרה זה, אינו מולקולה אחת אלא זוג YFP-BiFC מפוצל, אשר צריך תחילה לקבל מחדש ב vivo כדי להפוך פלואורופור FRET-acceptor פונקציונלי. כדי לבצע FRET-FLIM, יש לקבוע את הפלואורסצנטיות של מולקולת התורם לכל החיים לבדה ולאחר מכן בנוכחות שותף FRET.
- פתח את יישום FLIM במיקרוסקופ סריקת הלייזר הקונפוקלי, הפעל את הקונסולה והשתמש בספירת פוטונים לזיהוי דפוסים כדי למדוד את משך החיים של פלואורסצנטיות. בחרו במצב המדידה הסטנדרטי 'כל ספירת הפוטים'.
- לנתח דגימות משני סוגים של צמחים אגרו-הסתננו: אחד עם התורם בלבד (C-CFP) והשני עם התורם ואת הקבלה (C-CFP, יחד עם M-YFP) שניהם.
הערה: מכיוון שהאינטראקציה של חלבון M כבר אומתה עם חלבון C באמצעות BiFC ו- Y2H, צפויה יעילות FRET טובה עם זוג אינטראקציה זה. - לאחר מכן, סרוק את העלה החקלאי C-CFP הסתנן ולהתמקד בתא מראה פלואורסצנטיות CFP טובה. הפעל את מצב סריקת הלייזר והגדר את המערכת להדמיה של CFP ומדידות FLIM (לייזר דופק λex 440 ננומטר, λem 480-520 ננומטר על ידי גלאים היברידיים, מהירות סריקה 512 x 512 פיקסלים ב 400 הרץ).
- התאם את המיקוד, הזום והרווח החכם כדי להתמקד באזור שיש ללכוד.
- האר את הדגימה בעוצמה לייזר מספקת כדי להשיג לכידה של כ 0.1 פוטונים לכל פעימה. עבור דגימות עם עוצמת פלואורסצנטיות משתנה, לכוד 50 מסגרות כדי לאסוף פוטונים נאותים הנדרשים למדידה לכל החיים. CFP מציגה שתי תקופות חיים פלואורסצנטיות בשל ההסתגלות הקונפורמציה שלה; לכן, התאם את הנתונים באמצעות מודל reconvolution n-מעריכי תוך שמירה על הערך של n שווה ל- 2.
- בהגדרות אלה, CFP מציג שתי תקופות חיים של 1.0 ו- 3.2 ns. כאן גבוה יותר, 3.2 ns, משך חיים משמש עבור כל החישובים הבאים25,26.
- כדי לחשב את יעילות FRET באמצעות FLIM, שהוא המדד של מידת האינטראקציה בין שני חלבונים, לקחת דגימת עלים כי כבר בשיתוף עם C-CFP ו- M-YFP. חפש תא שמבטא הן C-CFP והן M-YFP ולאשר את דפוסי הפליטה שלהם בהתאמה על ידי מרגש אותם באמצעות לייזר דופק λex 440 ננומטר, λem 480-520 ננומטר ו λex 514 ננומטר לייזר אור לבן עם λem 526-550 ננומטר. סרוק וזיהה ברצף תא המציג הן פלואורסצנטיות של CFP והן פלואורסצנטיות YFP.
- לאחר אישור הפלואורסצנטיות משני החלבונים, עבור לקונסולת FLIM כדי למדוד את משך החיים של CFP באמצעות אותן הגדרות בהן השתמשנו קודם לכן למדידת משך החיים של C-CFP (שלב 5.5).
הערה: תא זה מבטא גם M-YFP שיכול לקיים אינטראקציה פוטנציאלית עם C-CFP ולגרום לירידה לאורך החיים של C-CFP. - התאם את הגרף המתקבל באמצעות מודל ההתיישבות המעריכית n, עם n = 2. נצפתה ירידה בתוחלת החיים של CFP מ- 3.2 ל- 2.6 ns, המציינת העברת אנרגיית תהודה של Förster בין CFP ל- YFP (איור 5A).
- כעת הפעל את מסוף FRET בתוכנה וחשב את יעילות FRET על-ידי הזנת משך חיים של תורם ללא רצף באופן ידני במשוואה המסופקת בתוכנה. ויעילות FRET הנצפית היא: 56%.
- אינטראקציה משולשת
- לבסוף, כדי לדמיין את האינטראקציות בין שלושה שותפים, לקחת את דגימת העלה מצמח כי היה בשיתוף עם C-CFP, M-YFPn, ו- M-YFPc.
- סרוק את מסלק העלים עבור תא המציג הן CFP והן פלואורסצנטיות YFP משוחזרת הנובעת מאינטראקציה של BiFC בין שני חלבוני M. השתמש באותם אורכי גל לייזר ופליטה כפי שהיה בשימוש קודם לכן.
- לאחר מכן, כבה את הלייזר 514 ננומטר ועבר לקונסולת FLIM.
הערה: אם עמעום M-YFP אינטראקציה עם C-CFP, יש לראות ירידה לאורך החיים של C-CFP כפי שנצפה במהלך האינטראקציה שלה עם M-YFP. עם זאת, אם C-CFP נכשל אינטראקציה עם M-YFP dimer, אורך החיים פלואורסצנטי שלה צריך להישאר ב 3.2 ns. - באמצעות הגדרות דומות כאמור לעיל, למדוד את משך החיים CFP בנוכחות YFP מחדש. התאם את הגרף המתקבל באמצעות מודל ההתיישבות המעריכית n, עם n = 2, ועבר למסוף FRET.
הערה: יש ירידה לאורך החיים של CFP מ 3.2 ל 2.3 ns. חשב את יעילות FRET כמתואר לעיל. יעילות FRET המחושבת היא 55%. הירידה בחיי התורם ויעילות FRET טובה של 55% מאשרת אינטראקציה משולשת בין שני חלבוני M לבין חלבון C ב-vivo (ראו איור 5B).
תוצאות
פרוטוקול זה מייצג שיטה ממוטבת לחקר אינטראקציות חלבון-חלבון משולש vivo בצמחים. העיקרון הבסיסי של הפרוטוקול הוא לשלב שתי טכניקות אינטראקציה חלבון מתויג פלואורסצנטית, כלומר, BiFC ו FRET, כדי ליצור בדיקת כדי למדוד היווצרות מורכבת טרנרית בין שלושה שותפי חלבון. כאן, השתמשנו FLIM כדי למדוד את חיי הפ...
Discussion
הפרוטוקול הנוכחי מדגים את השימוש בבדיקת FRET-FLIM מבוססת BiFC כדי לברר את היווצרותו של קומפלקס טרנרי בין שני מונומרים של חלבון תיבת MADS וחלבון חיישן סידן. הפרוטוקול מותאם מדו"ח של Y. John Shyu et al. שבו הם פיתחו שיטת FRET מבוססת BiFC כדי לדמיין קומפלקס טרנרי שנוצר בין הטרודימרים פוס-יוני ו- NFAT או p65 בשיטת הפלי...
Disclosures
המחברים אינם מצהירים על ניגודי עניינים.
Acknowledgements
NB, GG, SB, KC מודים בכנות לוועדת המענקים של האוניברסיטה (UGC), UGC-BSR, DBT-INSPIRE והמועצה למחקר מדעי ותעשייתי (CSIR) על מלגות המחקר שלהם. למרבה המזל אנו מכירים במחלקה לביוטכנולוגיה (DBT), ממשלת הודו, המחלקה למדע וטכנולוגיה (DST-FIST), ממשלת הודו לתמיכה כספית.
Materials
| Name | Company | Catalog Number | Comments |
| 1 ml Syringes without needles | Dispovan | - | |
| Acetosyringone | Sigma-Aldrich | D134406 | |
| Gateway LR Clonase II Enzyme mix | Thermo Fischer Scientific | 11791020 | The vectors used in the study are Gateway based |
| Gentamycin Sulphate | Himedia | CMS461 | |
| Kanamycin Sulphate | Himedia | MB105 | |
| MES hydrate | Sigma-Aldrich | M2933 | |
| MgCl2 | Sigma-Aldrich | M2670 | |
| pENTR/D-TOPO Cloning Kit | Thermo Fischer Scientific | K240020 | The vectors used in the study are Gateway based |
| Phusion high fidelity Taq DNA polymerase | Thermo Fischer Scientific | F530-S | Any High fidelity Polymerase can work |
| Rifampicin | Himedia | CMS1889 | |
| SP8 FALCON Confocal laser scanning microscope | Leica | SP8 FALCON | Any CLSM with FLIM capabilities can be used for this analysis |
| Spectinomycin dihydrochloride pentahydrate | Himedia | TC034 |
References
- Grove, C. A., Walhout, A. J. M. Transcription factor functionality and transcription regulatory networks. Molecular Biosystem. (4), 309-314 (2008).
- Amoutzias, G. D., Robertson, D. L., Van de Peer, Y., Oliver, S. G. Choose your partners: dimerization in eukaryotic transcription factors. Trends in Biochemical Sciences. 33 (5), 220-229 (2008).
- Arora, R., et al. MADS-box gene family in rice: genome-wide identification, organization and expression profiling during reproductive development and stress. BMC Genomics. 8 (242), (2007).
- Theißen, G., Gramzow, L. Structure and evolution of plant MADS domain transcription factors. Plant Transcription Factors: Evolutionary, Structural and Functional Aspects. , 127-138 (2016).
- Schweiger, R., Schwenkert, S. Protein-protein interactions visualized by bimolecular fluorescence complementation in tobacco protoplasts and leaves. Journal of Visualized Experiments: JoVE. (85), (2014).
- Bracha-Drori, K., et al. Detection of protein-protein interactions in plants using bimolecular fluorescence complementation. Plant Journal. 40 (3), 419-427 (2004).
- Shyu, Y. J., Suarez, C. D., Hu, C. Visualization of ternary complexes in living cells by using a BiFC-based FRET assay. Nature Protocols. 3 (11), 1693-1702 (2008).
- Kwaaitaal, M., Keinath, N. F., Pajonk, S., Biskup, C., Panstruga, R. Combined bimolecular fluorescence complementation and förster resonance energy transfer reveals ternary SNARE complex formation in living plant cells. Plant Physiology. 152 (3), 1135-1147 (2010).
- Margineanu, A., et al. Screening for protein-protein interactions using Förster resonance energy transfer (FRET) and fluorescence lifetime imaging microscopy (FLIM). Scientific Reports. 6, (2016).
- Datta, R., Heaster, T. M., Sharick, J. T., Gillette, A. A., Skala, M. C. Fluorescence lifetime imaging microscopy: fundamentals and advances in instrumentation, analysis, and applications. Journal of Biomedical Optics. 25 (07), 1 (2020).
- Long, Y., et al. Optimizing FRET-FLIM labeling conditions to detect nuclear protein interactions at native expression levels in living Arabidopsis roots. Frontiers in Plant Science. 9, 1-13 (2018).
- Burman, N., Chandran, D., Khurana, J. P. A Rapid and highly efficient method for transient gene expression in rice plants. Frontiers in Plant Science. , 11 (2020).
- Kudla, J., Bock, R. Lighting the way to protein-protein interactions: Recommendations on best practices for bimolecular fluorescence complementation analyses. Plant Cell. 28 (5), 1002-1008 (2016).
- Shyu, Y. J., Hu, C. D. Fluorescence complementation: an emerging tool for biological research. Trends in Biotechnology. 26 (11), 622-630 (2008).
- Kodama, Y., Hu, C. D. An improved bimolecular fluorescence complementation assay with a high signal-to-noise ratio. BioTechniques. 49 (5), 793-803 (2010).
- Kodama, Y., Hu, C. D. Bimolecular fluorescence complementation (BiFC): A 5-year update and future perspectives. BioTechniques. 53 (5), 285-298 (2012).
- Piston, D. W., Kremers, G. J. Fluorescent protein FRET: the good, the bad and the ugly. Trends in Biochemical Sciences. 32 (9), 407-414 (2007).
- Chakrabarty, R., et al. pSITE vectors for stable integration or transient expression of autofluorescent protein fusions in plants: Probing Nicotiana benthamiana- Virus Interactions. Molecular Plant-Microbe Interactions. 20 (7), 740-750 (2007).
- Walter, M., et al. Visualization of protein interactions in living plant cells using bimolecular fluorescence complementation. Plant Journal. 40 (3), 428-438 (2004).
- Hellens, R., Mullineaux, P., Klee, H. A guide to Agrobacterium binary Ti vectors. Trends in Plant Science. 5 (10), 446-451 (2000).
- Van Der Hoorn, R. A. L., Rivas, S., Wulff, B. B. H., Jones, J. D. G., Joosten, M. H. A. J. Rapid migration in gel filtration of the Cf-4 and Cf-9 resistance proteins is an intrinsic property of Cf proteins and not because of their association with high-molecular-weight proteins. Plant Journal. 35 (3), 305-315 (2003).
- Xie, X., et al. Engineering SARS-CoV-2 using a reverse genetic system. Nature Protocols. 16, (2021).
- Xu, J., et al. Optimized plasmid construction strategy for Cas9. Cellular Physiology and Biochemistry. 48, 131-137 (2018).
- Mattanovich, D., et al. Efficient transformation of Agrobacterium spp. by electroporation. Nucleic Acids Research. 17 (16), 6747 (1989).
- Rizzo, M. A., Springer, G. H., Granada, B., Piston, D. W. An improved cyan fluorescent protein variant useful for FRET. Nature Biotechnology. 22 (4), 445-449 (2004).
- Tramier, M., et al. Picosecond-hetero-FRET microscopy to probe protein-protein interactions in live cells. Biophysical Journal. 83 (6), 3570-3577 (2002).
- Alvarez, L. A. J., et al. SP8 FALCON: a novel concept in fluorescence lifetime imaging enabling video-rate confocal FLIM. Nature Methods. 20, 2-4 (2019).
- Postma, M., Goedhart, J. Plotsofdata-a web app for visualizing data together with their summaries. PLoS Biology. 17 (3), 1-8 (2019).
- Galperin, E., Verkhusha, V. V., Sorkin, A. Three-chromophore fret microscopy to analyze multiprotein interactions in living cells. Nature Methods. 1 (3), 209-217 (2004).
- Waadt, R., Kudla, J. In plant visualization of protein interactions using bimolecular fluorescence complementation (BiFC). Cold Spring Harbor Protocols. 3 (4), (2008).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved