É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Determinação da interação tripartite entre dois monômeros de um fator de transcrição da caixa MADS e uma proteína de sensor de cálcio por Ensaio BiFC-FRET-FLIM
* Estes autores contribuíram igualmente
Neste Artigo
Resumo
Aqui apresentamos, um método para visualizar a formação do complexo ternário entre três parceiros proteicos usando proteínas com marca fluorescente pelo ensaio FRET-FLIM baseado em BiFC. Este método é valioso para estudar complexos de interação proteína-proteína in vivo.
Resumo
As interações proteína-proteína são parte integrante de todos os processos biológicos das células, pois desempenham um papel crucial na regulação, manutenção e alteração das funções celulares. Essas interações estão envolvidas em uma ampla gama de fenômenos, como transdução de sinais, resposta de patógenos, interações célula-células, processos metabólicos e de desenvolvimento. No caso dos fatores de transcrição, essas interações podem levar à oligomerização de subunidades, sequestre em contextos subcelulares específicos como o núcleo, citoplasma, etc., que, por sua vez, podem ter um efeito mais profundo na expressão dos genes a jusante. Aqui, demonstramos uma metodologia para visualizar a interação tripartite in vivo usando a Complementação de Fluorescência Bimolecular (BiFC) baseada em Förster Resonance Energy Transfer (FRET) envolvendo Fluorescence Lifetime Imaging (FLIM). Duas das proteínas selecionadas para esta demonstração interagem como parceiros do BiFC, e sua atividade de fluorescência reconstituída é usada para avaliar o FRET-FLIM com o terceiro parceiro. As plantas nicotiana benthamiana, cultivadas em quatro a cinco semanas, foram usadas como o sistema de plantas modelo para esta demonstração.
Introdução
As interações proteína-proteína (PPIs) formam a base do bom funcionamento das células eucarióticas regulando vários processos metabólicos e de desenvolvimento. Alguns PPIs são estáveis, enquanto outros são transitórios na natureza. As interações podem ser categorizadas com base no número e tipo de membros na interação, como dimeric, trimeric, tetramérrico homomédico e heteromérico1. A identificação e caracterização das interações proteicas pode levar a uma melhor compreensão das funções proteicas e das redes regulatórias.
Os fatores de transcrição são proteínas que estão envolvidas em funções regulatórias. Eles regulam a taxa de transcrição de seus genes a jusante ligando-se ao DNA. Às vezes, a oligomerização ou formação de complexos de alta ordem por proteínas é um pré-requisito para a realização de suas funções2. Os fatores de transcrição da caixa MADS são genes homeóticos que regulam vários processos como transição floral, desenvolvimento de órgãos florais, fertilização, desenvolvimento de sementes, senescência e desenvolvimento vegetativo. Eles são conhecidos por formar complexos de alta ordem que se ligam ao DNA3,4. Estudar as redes de PPI entre fatores de transcrição e seus interagirres fornece insights sobre a complexidade subjacente à regulação transcricional.
A expressão proteica transitória em Nicotiana benthamiana tem sido uma abordagem popular para estudar a localização de proteínas ou interações proteína-proteína in vivo5. BiFC e FRET são métodos para estudar interações proteína-proteína in vivo usando sistemas de repórteres fluorescentes6. Uma combinação dessas duas técnicas tem sido demonstrada para revelar a interação entre três proteínas7. O FRET é medido usando a técnica de fotobleaching aceitador, emissão sensibilizada e Fluorescência Lifetime Imaging (FLIM). O ensaio FRET baseado em FLIM surgiu como uma ferramenta que fornece quantificação precisa e especificidade espacial para medições de transferência de energia entre duas moléculas com base em suas vidas de fluorescência8. FLIM mede o tempo em que um fluorohore permanece em estado animado antes de emitir um fóton e é melhor do que técnicas que usam medidas de intensidadesozinhas 9,10. Além de sistemas heterologos como nicotiana benthamiana e cascas epidérmicas de cebola, relatórios mais recentes demonstraram o uso de raízes arabidopsis e mudas de arroz jovens, etc., para análise in vivo de interações proteína-proteína em condições nativas11,12.
Além de um sistema de expressão adequado, a seleção de parceiros interajantes para ensaios BiFC e FRET também é crucial para o sucesso deste experimento. O PPI entre os parceiros utilizados na configuração biFC deve ser validado utilizando controles apropriados antes de seu uso como parceiro conjugado no experimento FRET13. O BiFC utiliza a complementação estrutural das partes terminais N e C da proteína fluorescente. Uma limitação comum na maioria, se não todas as proteínas fluorescentes utilizadas nos ensaios biFC tem sido o auto-montagem entre os dois fragmentos não fluorescentes derivados, contribuindo para a fluorescência falso-positiva e diminui a razão sinal-ruído (S/N)razão 14. Desenvolvimentos recentes, incluindo mutações pontuais ou posição de divisão da proteína fluorescente, deram origem a pares de BiFC com maior intensidade, maior especificidade, alta razão S/N15,16. Essas proteínas fluorescentes também podem ser usadas para a realização de BiFC, dependendo da adequação do experimento.
Tradicionalmente, CFP e YFP têm sido usados como o doador e o par de aceitadores em experimentos FRET17. No entanto, YFP ou m-Citrine foram encontrados como melhores doadores de FRET (quando usados com RFP como aceitador) devido ao alto rendimento quântico (QY) durante a expressão nativa de proteínas-alvo no sistema raiz arabidopsis. A seleção de promotores (constitutivo versus nativo/endógeno) e fluorógono também desempenham um papel crucial na concepção de um experimento bifc-fret-flim bem sucedido. É essencial notar que a eficiência dos doadores de FRET e a adequação dos pares FRET tendem a mudar com a mudança no promotor e no sistema biológico que está sendo utilizado para a expressão. O QY do fluoróforo, que se relaciona com seu brilho, depende do pH, temperatura e do sistema biológico em uso. Sugerimos que esses critérios sejam cuidadosamente considerados antes de escolher o par fluoróforo para o experimento FRET. O sistema biológico, os promotores e as proteínas utilizadas para este protocolo funcionaram bem com fluoroforos CFP-YFP para o experimento BiFC FRET-FLIM.
No presente estudo, incorporamos a característica da FLIM para visualizar a interação entre três moléculas de proteínas utilizando FRET baseada em BiFC. Nesta técnica, duas proteínas são marcadas com proteína YFP dividida e a terceira proteína com PCP. Como estávamos interessados em estudar a interação de um homodimer de proteína mads-box (M) com uma proteína sensor de cálcio (C), essas proteínas foram marcadas com proteínas fluorescentes nos vetores pSITE-1CA e pSITE-3CA18. Dois dos parceiros interativos, neste ensaio, foram marcados com partes terminais N e C do YFP nos vetores pSPYNE-35S e pSPYCE-35S19, e sua interação resulta na reconstituição do YFP funcional que atua como um aceitador de FRET para o terceiro parceiro interagente, que é marcado com CFP (atuando como doador de FRET) (Figura 1 ). Neste caso específico, o PPI entre dois monômeros M e entre M e C foi validado realizando BiFC em três sistemas diferentes, juntamente com o sistema levedura-dois-híbridos. Esses vetores foram mobilizados na cepa Agrobacterium tumifaciens GV3101 por eletroporação. A cepa GV3101 tem um pMP90 de Ti plasmídeo desarmado (pTiC58DT-DNA) com resistência à gentamicina20. Uma cepa p19 Agrobacterium foi adicionada juntamente com todas as infiltrações para evitar o silenciamento transgene21. Recomendamos que as três proteínas também sejam usadas em conformações opostas para validar as interações tripartites.
Nesta técnica, utilizamos flim, onde primeiro, a vida fluorescência do doador (vida de doador nãonchado) é medida na ausência de um aceitor. Depois disso, sua vida útil é medida na presença do aceitador (extinto de vida do doador). Essa diferença na vida útil da fluorescência de doadores é usada para calcular a eficiência do FRET, que depende do número de fótons que apresentam uma redução na vida útil da fluorescência. Mencionado abaixo é um protocolo detalhado para determinar a formação de um complexo ternário entre quaisquer três proteínas expressando transitoriamente as proteínas fluorescentes marcadas em Nicotiana benthamiana e avaliando sua interação pelo BiFC-FRET-FLIM.
Access restricted. Please log in or start a trial to view this content.
Protocolo
1. Clonagem de genes em vetores de entrada e destino (Figura2)
- Amplie a sequência de codificação (CDS) dos genes de interesse (genes M e C em nosso caso) por PCR e clone-os em vetores de entrada apropriados (por exemplo, vetor pENTR/D-TOPO; ver Tabela 1 para vetores usados neste experimento).
- Cresça os clones em placas contendo antibióticos. Validar clones selecionados nos antibióticos por restrição de digestão e sequenciamento de DNA22,23.
- Mobilize os CDS reconstituídos dos clones de entrada para os vetores de destino (pSPYNE-35S, pSPYCE-35S, pSITE-1CA e pSITE-3CA) e confirme a transferência de sequências da entrada para os vetores de destino por restrição de digestão enzimada.
NOTA: Todos os vetores utilizados neste experimento estão listados na Tabela 1. - Por fim, transforme as células Agrobacterium GV3101 (pMP90 (GentR))com os vetores de destino por eletroporação(Figura 3)24.
2. Condições de crescimento para as plantas nicotiana benthamiana
NOTA: Cultivar plantas de Nicotiana até 4-6 estágio de folha em condições de controle.
- Para cultivar as plantas de Nicotiana, prepare a mistura do solo misturando misturas de solo comercialmente disponíveis com cocopeat e adubo em uma proporção de 2:1:1.
- Espalhe uma camada de 1 polegada de espessura desta mistura de solo em uma bandeja de plástico para fazer o leito do solo e satura-lo com água deionizada. Polvilhe cerca de 200 sementes neste leito de solo.
- Transfira para uma bandeja maior contendo 1 cm de água parada. Cubra esta bandeja com plástico para criar uma câmara de umidade.
- Transfira esta configuração para uma câmara de crescimento fixada em 23 °C com luz de 16 h e ciclo escuro de 8h com intensidade de luz de 150-170 μmol/m2.
- Após duas semanas, transfira mudas jovens para pequenos potes de 3-4 polegadas contendo mistura de solo saturada de água.
- Coloque esses potes em bandejas de plástico e transfira-os para a câmara de crescimento por mais quatro semanas.
3. Prepare cepas bacterianas para a agro-infiltração
NOTA: Para a agro-infiltração, as cepas bacterianas precisam ser recém-subculturadas e misturadas juntamente com a cepa p19 de Agrobacterium em razões apropriadas.
- Prepare placas de ágar 2xYT contendo rifampicina (100 μg/mL), gentamicina (25 μg/mL) e kanamicina (50 μg/mL) para agrobacterium abrigando pSPYNE-35S e vetor pSPYCE-35S. Para o vetor pSITE contendo cepa, use rifampicina (100 μg/mL), gentamicina (25 μg/mL) e espectinomicina (50 μg/mL).
- Listrar as cepas de Agrobacterium contendo os plasmídeos nestas placas usando laços de inoculação estéreis em uma capa de fluxo laminar.
- Incubar-os a 28 °C por 48 h no escuro.
- Inicie este procedimento inoculando a cepa Agrobacterium GV3101 que abriga construções BiFC e FRET (preparada em pSPYNE-35S, pSPYCE-35S e vetores pSITE) de placas listradas em 10 mL de caldo 2xYT contendo antibióticos apropriados (Rifampicina (100 μg/mL), gentamicina (25 μg/mL), kanamycin (50 μg/mL) ou espectinomicina (50 μg/mL)).
- Além disso, inicie uma cultura de cepa p19 de Agrobacteria inoculando 10 mL de caldo 2xYT contendo rifampicina (100 μg/mL) e kanamycina (50 μg/mL).
NOTA: a cepa p19 é adicionada para evitar o silenciamento transgênico. - Cubra o frasco com uma folha de alumínio e mantenha-os no agitador da incubadora a 28 °C e 170 rpm por 16 h no escuro.
- Após o crescimento da noite para o dia, transfira 1 mL desta cultura para um cuvette descartável para medir a densidade óptica (O.D.) das culturas a 600 nm usando um espectrômetro.
- Misture as culturas do parceiro BiFC e FRET apropriados contendo cepas para que a O.D. final de cada cultura seja 0,5 e a de p19 seja de 0,3 em um volume total de 2 mL.
- Para alcançar essas proporções, use a fórmula mencionada abaixo:
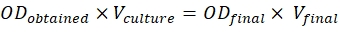
ODobtido = O.D. da cultura medida em 600 nm
Cultura V = Volume da cultura necessária
Final deOD = 0,5 para construções e 0,3 para p19
Vfinal = Volume final para a infiltração, que é de 2 mL
NOTA: As combinações de construção utilizadas neste estudo estão especificadas na Tabela 2. - Centrifugar as culturas de Agrobacterium mistas a 3.000 x g por 5 min à temperatura ambiente e descartar cuidadosamente o sobrenante. Resuspenque a pelota em 2 mL de tampão de infiltração recém-preparado (10 mM MES, 100 μM de Acetosyringona e 10 mM MgCl2). Use um misturador de vórtice para fazer uma suspensão celular homogênea.
- Incubar os tubos contendo células resuspended no escuro à temperatura ambiente por 3 h.
- Enquanto isso, rotule cada vaso de planta com a mistura de construção com a que ele vai ser infiltrado. Use duas plantas para cada mistura de infiltração.
- Encha uma seringa sem agulha de 1 mL com a mistura agrobacteriana. Pressione suavemente, mas firmemente, a seringa no lado abaxial da folha totalmente expandida enquanto apoia a folha do outro lado. Empurre suavemente o êmbolo até que as soluções se encham na área da folha equivalente a 2-3 vezes a ponta da seringa.
- Infiltrar-se em até quatro pontos em uma folha e 3-4 folhas por planta, como mostrado na Figura 4.
NOTA: Troque luvas ou limpe luvas com 70% de álcool entre as amostras para evitar contaminação cruzada. - Transfira todos os potes para uma bandeja e incubar em uma câmara de crescimento nas mesmas condições mencionadas na etapa 2.
- Verifique uma pequena parte da folha agroinfiltilhada em diferentes pontos de tempo usando um microscópio de fluorescência. Quando a fluorescência de YFP e CFP forem detectáveis em células, proceda ao microscópio confocal para o ensaio BiFC-FRET FLIM. Neste experimento, a análise foi realizada 3 dias após a agroindústria.
NOTA: Defina o período pós-agroinfiltração de incubação individualmente para cada promotor e combinação genética para evitar a superexpressão de proteínas quimricas utilizadas no ensaio BiFC-FRET FLIM. A superexpressão das proteínas parceiras pode levar a interações falso-positivas.
4. Prepare slides para visualização de fluorescência
- Quando as plantas estiverem prontas para visualização, corte amostras de folhas quadradas, 5-8 mm de distância da ferida de infiltração, e monte-as em água destilada em lâminas limpas.
NOTA: Para minimizar a fluorescência de fundo, limpe os slides com 80% de etanol seguido de água destilada 3-4 vezes, o ar seque-os e mantenha-os em uma folha absorvente. - Cubra a amostra da folha com uma mancha de cobertura limpa e vedação usando um esmalte de unha.
- Visualize essas amostras sob um microscópio de varredura a laser confocal.
5. Análise FRET-FLIM usando um microscópio de varredura a laser confocal
NOTA: Neste procedimento, a base de determinação e quantificação da interação entre duas proteínas é a redução da vida fluorescência do parceiro do doador de FRET após sua interação com o aceitador, que é usado para calcular a eficiência do FRET. A complexidade no caso da interação tripartite aumenta ainda mais porque o fret-acceptor, neste caso, não é uma única molécula, mas um par YFP-BiFC dividido, que deve primeiro ser reconstituído in vivo para se tornar um fluoróforo funcional que aceita FRET. Para realizar o FRET-FLIM, é preciso determinar a fluorescência da molécula do doador sozinho e, em seguida, na presença de um parceiro FRET.
- Abra o aplicativo FLIM no microscópio de varredura a laser confocal, inicie o console e use a contagem de fótons de reconhecimento de padrões para medir a vida útil da fluorescência. Selecione o modo de medição padrão 'Toda contagem de fótons'.
- Analisar amostras de dois tipos de plantas agroinduinadas: uma apenas com o doador (C-CFP) e outra com o doador e o aceitador (C-CFP, juntamente com M-YFP) ambas.
NOTA: Como a interação da proteína M já foi validada com a proteína C usando BiFC e Y2H, espera-se boa eficiência de FRET com este par de interação. - Em seguida, escaneie a folha agroindutorizada C-CFP e foque em uma célula que mostra boa fluorescência de CFP. Inicie o modo de varredura a laser e ajuste o sistema para visualização de CFP e medições FLIM (λex 440 nm laser de pulso, λem 480-520 nm por detectores híbridos, velocidade de varredura 512 x 512 pixels a 400 Hz).
- Ajuste o foco, o zoom e o ganho inteligente para focar na área que precisa ser capturada.
- Ilumine a amostra com potência laser suficiente para alcançar a captura de aproximadamente 0,1 fótons por pulso. Para amostras com intensidade variável de fluorescência, capture 50 quadros para coletar fótons adequados necessários para a medição vitalícia. O PCP exibe duas vidas de fluorescência devido à sua adaptação conformacional; portanto, encaixe os dados usando o modelo de reconvolução n-exponencial, mantendo o valor de n igual a 2.
- Nessas configurações, o PCP mostra duas vidas de 1,0 e 3,2 ns. Aqui o maior, 3,2 ns, a vida útil é utilizada para todos os cálculos subsequentes25,26.
- Para calcular a eficiência do FRET utilizando flim, que é a medida do grau de interação entre duas proteínas, pegue uma amostra de folha que foi co-infiltrada com C-CFP e M-YFP. Procure por uma célula que expresse tanto C-CFP quanto M-YFP e confirme seus respectivos padrões de emissão, excitando-os usando λex 440 nm laser de pulso, λem 480-520 nm e λex 514 nm laser de luz branca com λem 526-550 nm. Digitalizar e identificar sequencialmente uma célula que mostra fluorescência CFP e YFP.
- Depois de confirmar a fluorescência de ambas as proteínas, mude para o console FLIM para medir a vida útil do CFP usando as mesmas configurações que usamos anteriormente para medir a vida útil do C-CFP (etapa 5.5).
NOTA: Esta célula também está expressando M-YFP que pode potencialmente interagir com c-CFP e causar uma redução na vida útil do C-CFP. - Ajuste o gráfico obtido utilizando o modelo de reconvolução n-exponencial, com n = 2. Observou-se redução da vida útil do PCP de 3,2 para 2,6 ns, indicando a transferência de energia de ressonância förster entre CFP eYFP (Figura 5A).
- Agora inicie o console FRET no software e calcule a eficiência do FRET inserindo manualmente a vida útil do doador não saciado na equação fornecida no software. E a eficiência observada do FRET é de: 56%.
- Interação tripartite
- Finalmente, para visualizar as interações entre três parceiros, pegue a amostra de folha de uma planta que foi co-infiltrada com C-CFP, M-YFPn e M-YFPc.
- Escaneie a explanta da folha para uma célula que mostra tanto a fluorescência YFP reconstituída emanando da interação biFC entre duas proteínas M. Use os mesmos comprimentos de onda de laser e emissão usados anteriormente.
- Posteriormente, desligue o laser de 514 nm e mova-se para o console FLIM.
NOTA: Se o dimer M-YFP interagir com o C-CFP, deve-se ver uma redução na vida útil do C-CFP, como observado durante sua interação com o M-YFP. No entanto, se o C-CFP não interagir com o dimer M-YFP, sua vida útil de fluorescência deve permanecer em 3,2 ns. - Utilizando configurações semelhantes às mencionadas acima, meça a vida útil do CFP na presença de YFP reconstituído. Ajuste o gráfico obtido usando o modelo de reconvolução n-exponencial, com n = 2, e mova-se para o console FRET.
NOTA: Há um declínio na vida útil do CFP de 3,2 para 2,3 ns. Calcule a eficiência do FRET conforme descrito acima. A eficiência calculada do FRET é de 55%. A redução da vida útil do doador e a boa eficiência de FRET de 55% confirma a interação tripartite entre duas proteínas M e a proteína C in vivo (ver Figura 5B).
Access restricted. Please log in or start a trial to view this content.
Resultados
Este protocolo representa um método otimizado para estudar interações proteína-proteína in vivo tripartite nas plantas. O princípio básico do protocolo é combinar duas técnicas de interação proteica marcadas por fluorescência, ou seja, BiFC e FRET, para criar um ensaio para medir a formação do complexo ternário entre três parceiros proteicos. Aqui, utilizamos a FLIM para medir a vida fluorescência do parceiro doador DA FRET na presença e ausência do aceitador da FRET. Esperava-se uma redução...
Access restricted. Please log in or start a trial to view this content.
Discussão
O presente protocolo demonstra o uso de ensaios FRET-FLIM baseados em BiFC para verificar a formação de um complexo ternário entre dois monômeros de uma proteína de caixa MADS e uma proteína de sensor de cálcio. O protocolo é adaptado a partir de um relatório de Y. John Shyu et al. onde eles desenvolveram um método FRET baseado em BiFC para visualizar complexo ternário formado entre heterodimers Fos-Jun e NFAT ou p65 usando o método de emissão sensibilizado7. Anteriormente, um sistema...
Access restricted. Please log in or start a trial to view this content.
Divulgações
Os autores não declaram conflitos de interesse.
Agradecimentos
NB, GG, SB, KC agradecem sinceramente à Comissão de Subsídios Universitários (UGC), UGC-BSR, DBT-INSPIRE e Conselho de Pesquisa Científica e Industrial (CSIR) por suas bolsas de pesquisa. Agradecemos, felizmente, o Departamento de Biotecnologia (DBT), o Governo da Índia, o Departamento de Ciência e Tecnologia (DST-FIST), o Governo da Índia pelo apoio financeiro.
Access restricted. Please log in or start a trial to view this content.
Materiais
| Name | Company | Catalog Number | Comments |
| 1 ml Syringes without needles | Dispovan | - | |
| Acetosyringone | Sigma-Aldrich | D134406 | |
| Gateway LR Clonase II Enzyme mix | Thermo Fischer Scientific | 11791020 | The vectors used in the study are Gateway based |
| Gentamycin Sulphate | Himedia | CMS461 | |
| Kanamycin Sulphate | Himedia | MB105 | |
| MES hydrate | Sigma-Aldrich | M2933 | |
| MgCl2 | Sigma-Aldrich | M2670 | |
| pENTR/D-TOPO Cloning Kit | Thermo Fischer Scientific | K240020 | The vectors used in the study are Gateway based |
| Phusion high fidelity Taq DNA polymerase | Thermo Fischer Scientific | F530-S | Any High fidelity Polymerase can work |
| Rifampicin | Himedia | CMS1889 | |
| SP8 FALCON Confocal laser scanning microscope | Leica | SP8 FALCON | Any CLSM with FLIM capabilities can be used for this analysis |
| Spectinomycin dihydrochloride pentahydrate | Himedia | TC034 |
Referências
- Grove, C. A., Walhout, A. J. M. Transcription factor functionality and transcription regulatory networks. Molecular Biosystem. (4), 309-314 (2008).
- Amoutzias, G. D., Robertson, D. L., Van de Peer, Y., Oliver, S. G. Choose your partners: dimerization in eukaryotic transcription factors. Trends in Biochemical Sciences. 33 (5), 220-229 (2008).
- Arora, R., et al. MADS-box gene family in rice: genome-wide identification, organization and expression profiling during reproductive development and stress. BMC Genomics. 8 (242), (2007).
- Theißen, G., Gramzow, L. Structure and evolution of plant MADS domain transcription factors. Plant Transcription Factors: Evolutionary, Structural and Functional Aspects. , 127-138 (2016).
- Schweiger, R., Schwenkert, S. Protein-protein interactions visualized by bimolecular fluorescence complementation in tobacco protoplasts and leaves. Journal of Visualized Experiments: JoVE. (85), (2014).
- Bracha-Drori, K., et al. Detection of protein-protein interactions in plants using bimolecular fluorescence complementation. Plant Journal. 40 (3), 419-427 (2004).
- Shyu, Y. J., Suarez, C. D., Hu, C. Visualization of ternary complexes in living cells by using a BiFC-based FRET assay. Nature Protocols. 3 (11), 1693-1702 (2008).
- Kwaaitaal, M., Keinath, N. F., Pajonk, S., Biskup, C., Panstruga, R. Combined bimolecular fluorescence complementation and förster resonance energy transfer reveals ternary SNARE complex formation in living plant cells. Plant Physiology. 152 (3), 1135-1147 (2010).
- Margineanu, A., et al. Screening for protein-protein interactions using Förster resonance energy transfer (FRET) and fluorescence lifetime imaging microscopy (FLIM). Scientific Reports. 6, (2016).
- Datta, R., Heaster, T. M., Sharick, J. T., Gillette, A. A., Skala, M. C. Fluorescence lifetime imaging microscopy: fundamentals and advances in instrumentation, analysis, and applications. Journal of Biomedical Optics. 25 (07), 1(2020).
- Long, Y., et al. Optimizing FRET-FLIM labeling conditions to detect nuclear protein interactions at native expression levels in living Arabidopsis roots. Frontiers in Plant Science. 9, 1-13 (2018).
- Burman, N., Chandran, D., Khurana, J. P. A Rapid and highly efficient method for transient gene expression in rice plants. Frontiers in Plant Science. , 11(2020).
- Kudla, J., Bock, R. Lighting the way to protein-protein interactions: Recommendations on best practices for bimolecular fluorescence complementation analyses. Plant Cell. 28 (5), 1002-1008 (2016).
- Shyu, Y. J., Hu, C. D. Fluorescence complementation: an emerging tool for biological research. Trends in Biotechnology. 26 (11), 622-630 (2008).
- Kodama, Y., Hu, C. D. An improved bimolecular fluorescence complementation assay with a high signal-to-noise ratio. BioTechniques. 49 (5), 793-803 (2010).
- Kodama, Y., Hu, C. D. Bimolecular fluorescence complementation (BiFC): A 5-year update and future perspectives. BioTechniques. 53 (5), 285-298 (2012).
- Piston, D. W., Kremers, G. J. Fluorescent protein FRET: the good, the bad and the ugly. Trends in Biochemical Sciences. 32 (9), 407-414 (2007).
- Chakrabarty, R., et al. pSITE vectors for stable integration or transient expression of autofluorescent protein fusions in plants: Probing Nicotiana benthamiana- Virus Interactions. Molecular Plant-Microbe Interactions. 20 (7), 740-750 (2007).
- Walter, M., et al. Visualization of protein interactions in living plant cells using bimolecular fluorescence complementation. Plant Journal. 40 (3), 428-438 (2004).
- Hellens, R., Mullineaux, P., Klee, H. A guide to Agrobacterium binary Ti vectors. Trends in Plant Science. 5 (10), 446-451 (2000).
- Van Der Hoorn, R. A. L., Rivas, S., Wulff, B. B. H., Jones, J. D. G., Joosten, M. H. A. J. Rapid migration in gel filtration of the Cf-4 and Cf-9 resistance proteins is an intrinsic property of Cf proteins and not because of their association with high-molecular-weight proteins. Plant Journal. 35 (3), 305-315 (2003).
- Xie, X., et al. Engineering SARS-CoV-2 using a reverse genetic system. Nature Protocols. 16, (2021).
- Xu, J., et al. Optimized plasmid construction strategy for Cas9. Cellular Physiology and Biochemistry. 48, 131-137 (2018).
- Mattanovich, D., et al. Efficient transformation of Agrobacterium spp. by electroporation. Nucleic Acids Research. 17 (16), 6747(1989).
- Rizzo, M. A., Springer, G. H., Granada, B., Piston, D. W. An improved cyan fluorescent protein variant useful for FRET. Nature Biotechnology. 22 (4), 445-449 (2004).
- Tramier, M., et al. Picosecond-hetero-FRET microscopy to probe protein-protein interactions in live cells. Biophysical Journal. 83 (6), 3570-3577 (2002).
- Alvarez, L. A. J., et al. SP8 FALCON: a novel concept in fluorescence lifetime imaging enabling video-rate confocal FLIM. Nature Methods. 20, 2-4 (2019).
- Postma, M., Goedhart, J. Plotsofdata-a web app for visualizing data together with their summaries. PLoS Biology. 17 (3), 1-8 (2019).
- Galperin, E., Verkhusha, V. V., Sorkin, A. Three-chromophore fret microscopy to analyze multiprotein interactions in living cells. Nature Methods. 1 (3), 209-217 (2004).
- Waadt, R., Kudla, J. In plant visualization of protein interactions using bimolecular fluorescence complementation (BiFC). Cold Spring Harbor Protocols. 3 (4), (2008).
Access restricted. Please log in or start a trial to view this content.
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados