Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Determinación de la interacción tripartita entre dos monómeros de un factor de transcripción MADS-box y una proteína sensora de calcio mediante ensayo BiFC-FRET-FLIM
* Estos autores han contribuido por igual
En este artículo
Resumen
Aquí presentamos, un método para visualizar la formación de complejos ternarios entre tres socios de proteínas utilizando proteínas marcadas con fluorescentes mediante el ensayo FRET-FLIM basado en BiFC. Este método es valioso para estudiar los complejos de interacción proteína-proteína in vivo.
Resumen
Las interacciones proteína-proteína son una parte integral de todos los procesos biológicos en las células, ya que desempeñan un papel crucial en la regulación, el mantenimiento y la modificación de las funciones celulares. Estas interacciones están involucradas en una amplia gama de fenómenos como la transducción de señales, la respuesta a patógenos, las interacciones célula-célula, los procesos metabólicos y de desarrollo. En el caso de los factores de transcripción, estas interacciones pueden conducir a la oligomerización de las subunidades, secuestrando en contextos subcelulares específicos como el núcleo, el citoplasma, etc., lo que, a su vez, podría tener un efecto más profundo en la expresión de los genes aguas abajo. Aquí, demostramos una metodología para visualizar la interacción tripartita in vivo utilizando la Transferencia de Energía de Resonancia de Förster (FRET) basada en la Complementación de Fluorescencia Bimolecular (BiFC) que involucra Imágenes de Por Vida de Fluorescencia (FLIM). Dos de las proteínas seleccionadas para esta demostración interactúan como socios biFC, y su actividad de fluorescencia reconstituida se utiliza para ensayar FRET-FLIM con el tercer socio. Se han utilizado plantas de Nicotiana benthamiana cultivadas en cámaras de crecimiento de cuatro a cinco semanas de edad como el sistema de planta modelo para esta demostración.
Introducción
Las interacciones proteína-proteína (IBP) forman la base del buen funcionamiento de las células eucariotas mediante la regulación de diversos procesos metabólicos y de desarrollo. Algunos IBP son estables, mientras que otros son de naturaleza transitoria. Las interacciones se pueden clasificar en función del número y tipo de miembros en la interacción, como diméricos, triméricos, tetrámeros homoméricos y heteroméricos1. La identificación y caracterización de las interacciones proteicas puede conducir a una mejor comprensión de las funciones proteicas y las redes reguladoras.
Los factores de transcripción son proteínas que están involucradas en las funciones reguladoras. Regulan la velocidad de transcripción de sus genes aguas abajo uniéndose al ADN. A veces, la oligomerización o formación de complejos de orden superior por proteínas es un requisito previo para llevar a cabo sus funciones2. Los factores de transcripción MADS-box de plantas son genes homeóticos que regulan diversos procesos como la transición floral, el desarrollo de órganos florales, la fertilización, el desarrollo de semillas, la senescencia y el desarrollo vegetativo. Se sabe que forman complejos de orden superior que se unen al ADN3,4. El estudio de las redes PPI entre los factores de transcripción y sus interactores proporciona información sobre la complejidad subyacente a la regulación transcripcional.
La expresión transitoria de proteínas en Nicotiana benthamiana ha sido un enfoque popular para estudiar la localización de proteínas o las interacciones proteína-proteína in vivo5. BiFC y FRET son métodos para estudiar las interacciones proteína-proteína in vivo utilizando sistemas de reportero fluorescente6. Se ha demostrado que una combinación de estas dos técnicas revela la interacción entre tres proteínas7. FreT se mide mediante fotoblanqueo aceptor, emisión sensibilizada y técnica de imágenes de por vida de fluorescencia (FLIM). El ensayo FRET basado en FLIM ha surgido como una herramienta que proporciona cuantificación precisa y especificidad espaciotemporal a las mediciones de transferencia de energía entre dos moléculas en función de sus vidas de fluorescencia8. FLIM mide el tiempo que un fluoróforo permanece en un estado excitado antes de emitir un fotón y es mejor que las técnicas que utilizan medidas de intensidad solas9,10. Además de los sistemas heterólogos como Nicotiana benthamiana y las cáscaras epidérmicas de cebolla, informes más recientes han demostrado el uso de raíces de Arabidopsis y plántulas de arroz jóvenes, etc., para el análisis in vivo de las interacciones proteína-proteína en condiciones nativas11,12.
Además de un sistema de expresión adecuado, la selección de socios que interactúan para los ensayos BiFC y FRET también es crucial para el éxito de este experimento. El PPI entre los socios utilizados en la configuración de BiFC debe validarse utilizando los controles apropiados antes de su uso como socio conjugado en el experimento FRET13. BiFC utiliza la complementación estructural de las partes N- y C-terminal de la proteína fluorescente. Una limitación común en la mayoría, si no en todas, las proteínas fluorescentes utilizadas en los ensayos de BiFC ha sido el autoensamblaje entre los dos fragmentos derivados no fluorescentes, lo que contribuye a la fluorescencia falsa positiva y disminuye la relación señal-ruido (S / N)14. Los desarrollos recientes, incluidas las mutaciones puntuales o la posición de división de la proteína fluorescente, han dado lugar a pares biFC con mayor intensidad, mayor especificidad, alta relación S / N15,16. Estas proteínas fluorescentes también se pueden utilizar para llevar a cabo BiFC dependiendo de la idoneidad del experimento.
Tradicionalmente, CFP y YFP se han utilizado como el par donante y aceptor en experimentos FRET17. Sin embargo, se encontró que YFP o m-citrino son mejores donantes de FRET (cuando se usan con RFP como aceptor) debido al alto rendimiento cuántico (QY) durante la expresión nativa de proteínas objetivo en el sistema radicular de Arabidopsis. La selección de promotores (constitutivos versus nativos/endógenos) y fluoróforos también juegan un papel crucial en el diseño de un exitoso experimento BiFC-FRET-FLIM. Es esencial tener en cuenta que la eficiencia de los donantes fret y la idoneidad de los pares FRET tienden a cambiar con el cambio en el promotor y el sistema biológico que se utiliza para la expresión. El QY del fluoróforo, que se relaciona con su brillo, depende del pH, la temperatura y el sistema biológico en uso. Sugerimos que estos criterios se consideren a fondo antes de elegir el par de fluoróforos para el experimento FRET. El sistema biológico, los promotores y las proteínas utilizadas para este protocolo funcionaron bien con los fluoróforos CFP-YFP para el experimento BiFC FRET-FLIM.
En el presente estudio, incorporamos la característica de FLIM para visualizar la interacción entre tres moléculas de proteína utilizando FRET basado en BiFC. En esta técnica, dos proteínas se etiquetan con proteína YFP dividida y la tercera proteína con PPC. Dado que estábamos interesados en estudiar la interacción de un homodímero de proteína MADS-box (M) con una proteína sensora de calcio (C), estas proteínas fueron marcadas con proteínas fluorescentes en vectores pSITE-1CA y pSITE-3CA18. Dos de los socios que interactúan, en este ensayo, fueron etiquetados con partes N- y C-terminales del YFP en los vectores pSPYNE-35S y pSPYCE-35S19, y su interacción da como resultado la reconstitución del YFP funcional que actúa como aceptor fret para el tercer socio que interactúa, que está etiquetado con CFP (actuando como donante FRET) (Figura 1 ). En este caso particular, el PPI entre dos monómeros M y entre M y C ha sido validado mediante la realización de BiFC en tres sistemas diferentes junto con el sistema de levadura-dos-híbrido. Estos vectores fueron movilizados en la cepa Agrobacterium tumifaciens GV3101 por electroporación. La cepa GV3101 tiene un plásmido Ti desarmado pMP90 (pTiC58DT-DNA) con resistencia a la gentamicina20. Se añadió una cepa p19 Agrobacterium junto con todas las infiltraciones para evitar el silenciamiento detransgenes 21. Recomendamos que las tres proteínas también se utilicen en conformaciones opuestas para validar las interacciones tripartitas.
En esta técnica, hemos empleado FLIM, donde en primer lugar, la vida útil de fluorescencia del donante (vida útil del donante no exprimida) se mide en ausencia de un aceptor. Después de eso, su vida útil se mide en presencia del aceptor (vida del donante apagado). Esta diferencia en las vidas de fluorescencia de los donantes se utiliza para calcular la eficiencia de FRET, que depende del número de fotones que exhiben una reducción en la vida útil de la fluorescencia. A continuación se menciona un protocolo detallado para determinar la formación de un complejo ternario entre tres proteínas cualesquiera expresando transitoriamente las proteínas marcadas con fluorescentes en Nicotiana benthamiana y ensayando su interacción por BiFC-FRET-FLIM.
Access restricted. Please log in or start a trial to view this content.
Protocolo
1. Clonación de genes en vectores de entrada y destino (Figura2)
- Amplificar la secuencia codificante (CDS) de los genes de interés (genes M y C en nuestro caso) mediante PCR y clonarlos en vectores de entrada apropiados (por ejemplo, vector pENTR/D-TOPO; ver Tabla 1 para los vectores utilizados en este experimento).
- Cultive los clones en placas que contengan antibióticos. Validar clones que se seleccionan en los antibióticos por restricción de digestión y secuenciación de ADN22,23.
- Movilizar los CDS reconstituidos de los clones de entrada a los vectores de destino (pSPYNE-35S, pSPYCE-35S, pSITE-1CA y pSITE-3CA) y confirmar la transferencia de secuencias desde la entrada a los vectores de destino por digestión enzimática de restricción.
NOTA: Todos los vectores utilizados en este experimento se enumeran en la Tabla 1. - Finalmente, transformar las celdas de Agrobacterium GV3101 (pMP90 (GentR))con los vectores de destino por electroporación (Figura 3)24.
2. Condiciones de crecimiento para las plantas de Nicotiana benthamiana
NOTA: Cultive plantas de Nicotiana hasta la etapa de 4-6 hojas en condiciones de control.
- Para cultivar plantas de Nicotiana, prepare la mezcla de suelo mezclando mezclas de suelo disponibles comercialmente con cocopeat y compost en una proporción de 2: 1: 1.
- Extienda una capa de 1 pulgada de espesor de esta mezcla de suelo en una bandeja de plástico para hacer el lecho de suelo y saturarlo con agua desionizada. Espolvorea alrededor de 200 semillas en este lecho de suelo.
- Transfiéralo a una bandeja más grande que contenga 1 cm de agua estancada. Cubra esta bandeja con una envoltura de plástico para crear una cámara de humedad.
- Transfiera esta configuración a una cámara de crecimiento a 23 °C con 16 h de luz y 8 h de ciclo oscuro con 150-170 μmol/m2s de intensidad de luz.
- Después de dos semanas, transfiera las plántulas jóvenes a macetas pequeñas de 3-4 pulgadas que contengan una mezcla de suelo saturada de agua.
- Coloque estas macetas en bandejas de plástico y transfiéralas a la cámara de crecimiento durante cuatro semanas más.
3. Preparar cepas bacterianas para la agroinfiltración
NOTA: Para la agroinfiltración, las cepas bacterianas deben ser recién subcultivadas y mezcladas junto con la cepa p19 de Agrobacterium en proporciones apropiadas.
- Preparar placas de agar 2xYT que contengan rifampicina (100 μg/mL), gentamicina (25 μg/mL) y kanamicina (50 μg/mL) para Agrobacterium que albergue vector pSPYNE-35S y pSPYCE-35S. Para el vector pSITE que contiene cepa, use rifampicina (100 μg/ml), gentamicina (25 μg/ml) y espectinomicina (50 μg/ml).
- Rayar las cepas de Agrobacterium que contienen los plásmidos en estas placas utilizando bucles de inoculación estériles en una campana de flujo laminar.
- Incubarlos a 28 °C durante 48 h en la oscuridad.
- Comience este procedimiento inoculando la cepa Agrobacterium GV3101 que alberga construcciones BiFC y FRET (preparadas en vectores pSPYNE-35S, pSPYCE-35S y pSITE) a partir de placas rayadas en 10 mL de caldo 2xYT que contienen antibióticos apropiados (Rifampicina (100 μg / ml), gentamicina (25 μg / ml), kanamicina (50 μg / ml) o espectinomicina (50 μg / ml)).
- Además, iniciar un cultivo de cepa p19 de Agrobacteria inoculando 10 mL de caldo 2xYT que contenga rifampicina (100 μg/mL) y kanamicina (50 μg/mL).
NOTA: se añade la cepa p19 para evitar el silenciamiento de los transgénicos. - Cubra el matraz con un papel de aluminio y manténgalos en el agitador de la incubadora a 28 ° C y 170 rpm durante 16 h en la oscuridad.
- Después del crecimiento durante la noche, transfiera 1 ml de este cultivo a una cubeta desechable para medir la densidad óptica (O.D.) de los cultivos a 600 nm utilizando un espectrofotómetro.
- Mezclar los cultivos de las cepas apropiadas de BiFC y FRET partner que contengan cepas de modo que el O.D. final de cada cultivo sea de 0.5 y el de p19 sea de 0.3 en un volumen total de 2 mL.
- Para lograr estas proporciones, use la fórmula que se menciona a continuación:
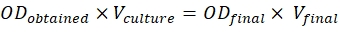
ODobtenida = O.D. del cultivo medido a 600 nm
Vcultura = Volumen de la cultura requerida
ODfinal = 0,5 para constructos y 0,3 para p19
Vfinal = Volumen final para la infiltración, que es de 2 mL
NOTA: Las combinaciones de constructo utilizadas en este estudio se especifican en la Tabla 2. - Centrifugar los cultivos mixtos de Agrobacterium a 3.000 x g durante 5 min a temperatura ambiente y desechar cuidadosamente el sobrenadante. Resuspender el pellet en 2 mL de tampón de infiltración recién preparado (10 mM MES, 100 μM de acetosiringona y 10 mM MgCl2). Use un mezclador de vórtice para hacer una suspensión celular homogénea.
- Incubar los tubos que contienen células resuspendidas en la oscuridad a temperatura ambiente durante 3 h.
- Mientras tanto, etiquete cada maceta con la mezcla de construcción con la que se va a infiltrar. Use dos plantas para cada mezcla de infiltración.
- Llene una jeringa sin aguja de 1 ml con la mezcla agrobacteriana. Presione suave pero firmemente la jeringa sobre el lado abaxial de la hoja completamente expandida mientras apoya la hoja desde el otro lado. Empuje suavemente el émbolo hasta que las soluciones se llenen en el área de la hoja equivalente a 2-3 veces la punta de la jeringa.
- Infiltrar hasta cuatro manchas en una hoja y 3-4 hojas por planta, como se muestra en la Figura 4.
NOTA: Cambie los guantes o limpie los guantes con alcohol al 70% entre las muestras para evitar la contaminación cruzada. - Transfiera todas las macetas a una bandeja e incube en una cámara de crecimiento en las mismas condiciones que se mencionan en el paso 2.
- Revise una pequeña parte de la hoja agroinfiltrada en diferentes puntos de tiempo utilizando un microscopio de fluorescencia. Cuando la fluorescencia de YFP y CFP sea detectable en las células, proceda al microscopio confocal para el ensayo BiFC-FRET FLIM. En este experimento, el análisis se llevó a cabo 3 días después de la agroinfiltración.
NOTA: Establezca el período de incubación posterior a la agroinfiltración individualmente para cada promotor y combinación de genes para evitar la sobreexpresión de proteínas quiméricas utilizadas en el ensayo BiFC-FRET FLIM. La sobreexpresión de las proteínas asociadas puede conducir a interacciones falsas positivas.
4. Prepare diapositivas para la visualización de fluorescencia
- Cuando las plantas estén listas para la visualización, corte muestras de hojas cuadradas, a 5-8 mm de distancia de la herida de infiltración, y móntelas en agua destilada en toboganes limpios.
NOTA: Para minimizar la fluorescencia de fondo, limpie las diapositivas con etanol al 80% seguido de agua destilada 3-4 veces, séquelas al aire y manténgalas en una lámina absorbente. - Cubra la muestra de la hoja con una funda limpia y selle con un esmalte de uñas.
- Visualice estas muestras bajo un microscopio de barrido láser confocal.
5. Análisis FRET-FLIM utilizando un microscopio de barrido láser confocal
NOTA: En este procedimiento, la base para determinar y cuantificar la interacción entre dos proteínas es la reducción de la vida útil de fluorescencia del socio donante de FRET sobre su interacción con el aceptor, que se utiliza para calcular la eficiencia de FRET. La complejidad en el caso de la interacción tripartita aumenta aún más porque el aceptor FRET, en este caso, no es una sola molécula sino un par YFP-BiFC dividido, que primero debe reconstituirse in vivo para convertirse en un fluoróforo aceptor FRET funcional. Para llevar a cabo FRET-FLIM, es necesario determinar la vida útil de fluorescencia de la molécula donante, primero solo y luego en presencia de un socio FRET.
- Abra la aplicación FLIM en el microscopio de barrido láser confocal, inicie la consola y utilice el conteo de fotones de reconocimiento de patrones para medir la vida útil de la fluorescencia. Seleccione el modo de medición estándar 'Conteo de todos los fotones'.
- Analizar muestras de dos tipos de plantas agroinfiltradas: una solo con el donante (C-CFP) y la otra con el donante y el aceptor (C-CFP, junto con M-YFP) ambos.
NOTA: Debido a que la interacción de la proteína M ya ha sido validada con la proteína C utilizando BiFC e Y2H, se espera una buena eficiencia FRET con este par que interactúa. - A continuación, escanee la hoja agroinfiltrada C-CFP y concéntrese en una célula que muestre buena fluorescencia CFP. Inicie el modo de escaneo láser y configure el sistema para la visualización CFP y las mediciones FLIM (láser de pulso λex 440 nm, λem 480-520 nm por detectores híbridos, velocidad de escaneo 512 x 512 píxeles a 400 Hz).
- Ajuste el enfoque, el zoom y la ganancia inteligente para enfocar el área que necesita ser capturada.
- Ilumine la muestra a suficiente potencia láser para lograr la captura de aproximadamente 0,1 fotones por pulso. Para muestras con intensidad de fluorescencia variable, capture 50 fotogramas para recolectar los fotones adecuados necesarios para la medición de la vida útil. La PPC exhibe dos vidas de fluorescencia debido a su adaptación conformacional; por lo tanto, ajuste los datos utilizando el modelo de reconvolución n-exponencial manteniendo el valor de n igual a 2.
- En estos ajustes, el CFP muestra dos vidas útiles de 1.0 y 3.2 ns. Aquí el más alto, 3.2 ns, la vida útil se utiliza para todos los cálculos posteriores25,26.
- Para calcular la eficiencia de FRET utilizando FLIM, que es la medida del grado de interacción entre dos proteínas, tome una muestra de hoja que ha sido coinfiltrada con C-CFP y M-YFP. Busque una célula que exprese tanto C-CFP como M-YFP y confirme sus respectivos patrones de emisión ex ex excitandolos usando láser de pulso λex 440 nm, λem 480-520 nm y láser de luz blanca λex 514 nm con λem 526-550 nm. Escanee e identifique secuencialmente una célula que muestre fluorescencia cfp y YFP.
- Después de confirmar la fluorescencia de ambas proteínas, cambie a la consola FLIM para medir la vida útil de CFP utilizando la misma configuración que usamos anteriormente para medir la vida útil de C-CFP (paso 5.5).
NOTA: Esta célula también está expresando M-YFP que potencialmente puede interactuar con C-CFP y causar una reducción en la vida útil de C-CFP. - Ajustar la gráfica obtenida utilizando el modelo de reconvolución n-exponencial, con n = 2. Se observó una disminución en la vida útil de la PPC de 3,2 a 2,6 ns, lo que indica la transferencia de energía de resonancia de Förster entre la PPC y la YFP(Figura 5A).
- Ahora inicie la consola FRET en el software y calcule la eficiencia FRET ingresando manualmente la vida útil del donante no exprimida en la ecuación proporcionada en el software. Y la eficiencia FRET observada es: 56%.
- Interacción tripartita
- Finalmente, para visualizar las interacciones entre tres socios, tome la muestra de hoja de una planta que fue coinfiltrada con C-CFP, M-YFPn y M-YFPc.
- Escanee el explante de la hoja en busca de una célula que muestre tanto CFP como fluorescencia YFP reconstituida que emana de la interacción BiFC entre dos proteínas M. Utilice el mismo láser y longitudes de onda de emisión que se usaron anteriormente.
- Posteriormente, apague el láser de 514 nm y muévase a la consola FLIM.
NOTA: Si el dímero M-YFP interactúa con C-CFP, se debe ver una reducción en la vida útil de C-CFP como se observa durante su interacción con M-YFP. Sin embargo, si el C-CFP no interactúa con el dímero M-YFP, su vida útil de fluorescencia debe permanecer en 3.2 ns. - Utilizando configuraciones similares a las mencionadas anteriormente, mida la vida útil de la PPC en presencia de YFP reconstituida. Ajuste el gráfico obtenido utilizando el modelo de reconvolución n-exponencial, con n = 2, y muévase a la consola FRET.
NOTA: Hay una disminución en la vida útil de la PPC de 3.2 a 2.3 ns. Calcule la eficiencia fret como se describió anteriormente. La eficiencia FRET calculada es del 55%. La reducción en la vida útil del donante y la buena eficiencia FRET del 55% confirma la interacción tripartita entre dos proteínas M y la proteína C in vivo (ver Figura 5B).
Access restricted. Please log in or start a trial to view this content.
Resultados
Este protocolo representa un método optimizado para estudiar in vivo las interacciones tripartitas proteína-proteína en plantas. El principio básico del protocolo es combinar dos técnicas de interacción de proteínas marcadas con fluorescencia, es decir, BiFC y FRET, para crear un ensayo para medir la formación de complejos ternarios entre tres socios de proteínas. Aquí, hemos utilizado FLIM para medir la vida útil de fluorescencia del socio donante fret en presencia y ausencia del aceptor FRET. Se esp...
Access restricted. Please log in or start a trial to view this content.
Discusión
El presente protocolo demuestra el uso del ensayo FRET-FLIM basado en BiFC para determinar la formación de un complejo ternario entre dos monómeros de una proteína MADS-box y una proteína sensora de calcio. El protocolo está adaptado de un informe de Y. John Shyu et al. donde han desarrollado un método FRET basado en BiFC para visualizar el complejo ternario formado entre heterodímeros de Fos-Jun y NFAT o p65 utilizando el método de emisión sensibilizada7. Anteriormente, galperin y sus co...
Access restricted. Please log in or start a trial to view this content.
Divulgaciones
Los autores declaran que no hay conflictos de intereses.
Agradecimientos
NB, GG, SB, KC agradecen sinceramente a la Comisión de Becas Universitarias (UGC), UGC-BSR, DBT-INSPIRE y el Consejo de Investigación Científica e Industrial (CSIR) por sus becas de investigación. Afortunadamente, agradecemos al Departamento de Biotecnología (DBT), al Gobierno de la India, al Departamento de Ciencia y Tecnología (DST-FIST), al Gobierno de la India por su apoyo financiero.
Access restricted. Please log in or start a trial to view this content.
Materiales
| Name | Company | Catalog Number | Comments |
| 1 ml Syringes without needles | Dispovan | - | |
| Acetosyringone | Sigma-Aldrich | D134406 | |
| Gateway LR Clonase II Enzyme mix | Thermo Fischer Scientific | 11791020 | The vectors used in the study are Gateway based |
| Gentamycin Sulphate | Himedia | CMS461 | |
| Kanamycin Sulphate | Himedia | MB105 | |
| MES hydrate | Sigma-Aldrich | M2933 | |
| MgCl2 | Sigma-Aldrich | M2670 | |
| pENTR/D-TOPO Cloning Kit | Thermo Fischer Scientific | K240020 | The vectors used in the study are Gateway based |
| Phusion high fidelity Taq DNA polymerase | Thermo Fischer Scientific | F530-S | Any High fidelity Polymerase can work |
| Rifampicin | Himedia | CMS1889 | |
| SP8 FALCON Confocal laser scanning microscope | Leica | SP8 FALCON | Any CLSM with FLIM capabilities can be used for this analysis |
| Spectinomycin dihydrochloride pentahydrate | Himedia | TC034 |
Referencias
- Grove, C. A., Walhout, A. J. M. Transcription factor functionality and transcription regulatory networks. Molecular Biosystem. (4), 309-314 (2008).
- Amoutzias, G. D., Robertson, D. L., Van de Peer, Y., Oliver, S. G. Choose your partners: dimerization in eukaryotic transcription factors. Trends in Biochemical Sciences. 33 (5), 220-229 (2008).
- Arora, R., et al. MADS-box gene family in rice: genome-wide identification, organization and expression profiling during reproductive development and stress. BMC Genomics. 8 (242), (2007).
- Theißen, G., Gramzow, L. Structure and evolution of plant MADS domain transcription factors. Plant Transcription Factors: Evolutionary, Structural and Functional Aspects. , 127-138 (2016).
- Schweiger, R., Schwenkert, S. Protein-protein interactions visualized by bimolecular fluorescence complementation in tobacco protoplasts and leaves. Journal of Visualized Experiments: JoVE. (85), (2014).
- Bracha-Drori, K., et al. Detection of protein-protein interactions in plants using bimolecular fluorescence complementation. Plant Journal. 40 (3), 419-427 (2004).
- Shyu, Y. J., Suarez, C. D., Hu, C. Visualization of ternary complexes in living cells by using a BiFC-based FRET assay. Nature Protocols. 3 (11), 1693-1702 (2008).
- Kwaaitaal, M., Keinath, N. F., Pajonk, S., Biskup, C., Panstruga, R. Combined bimolecular fluorescence complementation and förster resonance energy transfer reveals ternary SNARE complex formation in living plant cells. Plant Physiology. 152 (3), 1135-1147 (2010).
- Margineanu, A., et al. Screening for protein-protein interactions using Förster resonance energy transfer (FRET) and fluorescence lifetime imaging microscopy (FLIM). Scientific Reports. 6, (2016).
- Datta, R., Heaster, T. M., Sharick, J. T., Gillette, A. A., Skala, M. C. Fluorescence lifetime imaging microscopy: fundamentals and advances in instrumentation, analysis, and applications. Journal of Biomedical Optics. 25 (07), 1(2020).
- Long, Y., et al. Optimizing FRET-FLIM labeling conditions to detect nuclear protein interactions at native expression levels in living Arabidopsis roots. Frontiers in Plant Science. 9, 1-13 (2018).
- Burman, N., Chandran, D., Khurana, J. P. A Rapid and highly efficient method for transient gene expression in rice plants. Frontiers in Plant Science. , 11(2020).
- Kudla, J., Bock, R. Lighting the way to protein-protein interactions: Recommendations on best practices for bimolecular fluorescence complementation analyses. Plant Cell. 28 (5), 1002-1008 (2016).
- Shyu, Y. J., Hu, C. D. Fluorescence complementation: an emerging tool for biological research. Trends in Biotechnology. 26 (11), 622-630 (2008).
- Kodama, Y., Hu, C. D. An improved bimolecular fluorescence complementation assay with a high signal-to-noise ratio. BioTechniques. 49 (5), 793-803 (2010).
- Kodama, Y., Hu, C. D. Bimolecular fluorescence complementation (BiFC): A 5-year update and future perspectives. BioTechniques. 53 (5), 285-298 (2012).
- Piston, D. W., Kremers, G. J. Fluorescent protein FRET: the good, the bad and the ugly. Trends in Biochemical Sciences. 32 (9), 407-414 (2007).
- Chakrabarty, R., et al. pSITE vectors for stable integration or transient expression of autofluorescent protein fusions in plants: Probing Nicotiana benthamiana- Virus Interactions. Molecular Plant-Microbe Interactions. 20 (7), 740-750 (2007).
- Walter, M., et al. Visualization of protein interactions in living plant cells using bimolecular fluorescence complementation. Plant Journal. 40 (3), 428-438 (2004).
- Hellens, R., Mullineaux, P., Klee, H. A guide to Agrobacterium binary Ti vectors. Trends in Plant Science. 5 (10), 446-451 (2000).
- Van Der Hoorn, R. A. L., Rivas, S., Wulff, B. B. H., Jones, J. D. G., Joosten, M. H. A. J. Rapid migration in gel filtration of the Cf-4 and Cf-9 resistance proteins is an intrinsic property of Cf proteins and not because of their association with high-molecular-weight proteins. Plant Journal. 35 (3), 305-315 (2003).
- Xie, X., et al. Engineering SARS-CoV-2 using a reverse genetic system. Nature Protocols. 16, (2021).
- Xu, J., et al. Optimized plasmid construction strategy for Cas9. Cellular Physiology and Biochemistry. 48, 131-137 (2018).
- Mattanovich, D., et al. Efficient transformation of Agrobacterium spp. by electroporation. Nucleic Acids Research. 17 (16), 6747(1989).
- Rizzo, M. A., Springer, G. H., Granada, B., Piston, D. W. An improved cyan fluorescent protein variant useful for FRET. Nature Biotechnology. 22 (4), 445-449 (2004).
- Tramier, M., et al. Picosecond-hetero-FRET microscopy to probe protein-protein interactions in live cells. Biophysical Journal. 83 (6), 3570-3577 (2002).
- Alvarez, L. A. J., et al. SP8 FALCON: a novel concept in fluorescence lifetime imaging enabling video-rate confocal FLIM. Nature Methods. 20, 2-4 (2019).
- Postma, M., Goedhart, J. Plotsofdata-a web app for visualizing data together with their summaries. PLoS Biology. 17 (3), 1-8 (2019).
- Galperin, E., Verkhusha, V. V., Sorkin, A. Three-chromophore fret microscopy to analyze multiprotein interactions in living cells. Nature Methods. 1 (3), 209-217 (2004).
- Waadt, R., Kudla, J. In plant visualization of protein interactions using bimolecular fluorescence complementation (BiFC). Cold Spring Harbor Protocols. 3 (4), (2008).
Access restricted. Please log in or start a trial to view this content.
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados