È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Determinazione dell'interazione tripartita tra due monomeri di un fattore di trascrizione MADS-box e una proteina sensore di calcio mediante saggio BiFC-FRET-FLIM
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
Qui presentiamo un metodo per visualizzare la formazione di complessi ternari tra tre partner proteici utilizzando proteine fluorescenti marcate dal test FRET-FLIM basato su BiFC. Questo metodo è prezioso per studiare i complessi di interazione proteina-proteina in vivo.
Abstract
Le interazioni proteina-proteina sono parte integrante di tutti i processi biologici nelle cellule in quanto svolgono un ruolo cruciale nella regolazione, nel mantenimento e nella modifica delle funzioni cellulari. Queste interazioni sono coinvolte in una vasta gamma di fenomeni come la trasduzione del segnale, la risposta dei patogeni, le interazioni cellula-cellula, i processi metabolici e di sviluppo. Nel caso di fattori di trascrizione, queste interazioni possono portare all'oligomerizzazione di subunità, sequestrando in specifici contesti subcellulari come il nucleo, il citoplasma, ecc., Che, a sua volta, potrebbe avere un effetto più profondo sull'espressione dei geni a valle. Qui, dimostriamo una metodologia per visualizzare l'interazione tripartita in vivo utilizzando la complementazione di fluorescenza bimolecolare (BiFC) basata sul trasferimento di energia di risonanza di Förster (FRET) che coinvolge l'imaging a vita di fluorescenza (FLIM). Due delle proteine selezionate per questa dimostrazione interagiscono come partner BiFC e la loro attività di fluorescenza ricostituita viene utilizzata per analizzare FRET-FLIM con il terzo partner. Le piante di Nicotiana benthamiana coltivate a camera di crescita da quattro a cinque settimane sono state utilizzate come sistema vegetale modello per questa dimostrazione.
Introduzione
Le interazioni proteina-proteina (PPI) costituiscono la base del corretto funzionamento delle cellule eucariotiche regolando vari processi metabolici e di sviluppo. Alcuni IPP sono stabili, mentre altri sono di natura transitoria. Le interazioni possono essere classificate in base al numero e al tipo di membri nell'interazione come dimerico, trimerico, tetramerico omomerico ed eteromerico1. L'identificazione e la caratterizzazione delle interazioni proteiche può portare a una migliore comprensione delle funzioni proteiche e delle reti regolatorie.
I fattori di trascrizione sono proteine coinvolte nelle funzioni regolatorie. Regolano il tasso di trascrizione dei loro geni a valle legandosi al DNA. A volte l'oligomerizzazione o la formazione di complessi di ordine superiore da parte delle proteine è un prerequisito per svolgere le loro funzioni2. I fattori di trascrizione MADS-box delle piante sono geni omeotici che regolano vari processi come la transizione floreale, lo sviluppo di organi floreali, la fecondazione, lo sviluppo dei semi, la senescenza e lo sviluppo vegetativo. Sono noti per formare complessi di ordine superiore che si legano al DNA3,4. Lo studio delle reti PPI tra i fattori di trascrizione e i loro interattori fornisce informazioni sulla complessità alla base della regolazione trascrizionale.
L'espressione proteica transitoria in Nicotiana benthamiana è stata un approccio popolare per studiare la localizzazione proteica o le interazioni proteina-proteina in vivo5. BiFC e FRET sono metodi per studiare le interazioni proteina-proteina in vivo utilizzando sistemi reporter fluorescenti6. Una combinazione di queste due tecniche ha dimostrato di rivelare l'interazione tra tre proteine7. Il FRET viene misurato utilizzando la fotosbiancamento dell'accettore, l'emissione sensibilizzata e la tecnica FLIM (Fluorescence Lifetime Imaging). Il test FRET basato su FLIM è emerso come uno strumento che fornisce una quantificazione accurata e una specificità spaziotemporale alle misurazioni del trasferimento di energia tra due molecole in base alla loro durata di fluorescenza8. FLIM misura il tempo in cui un fluoroforo rimane in uno stato eccitato prima di emettere un fotone ed è migliore delle tecniche che utilizzano solo misurazioni di intensità9,10. Oltre a sistemi eterologhi come la Nicotiana benthamiana e le bucce epidermiche di cipolla, rapporti più recenti hanno dimostrato l'uso di radici di Arabidopsis e giovani piantine di riso, ecc., per l'analisi in vivo delle interazioni proteina-proteina in condizioni native11,12.
Oltre a un adeguato sistema di espressione, anche la selezione di partner interagenti per i test BiFC e FRET è cruciale per il successo di questo esperimento. Il PPI tra i partner utilizzati nella configurazione BiFC deve essere convalidato utilizzando controlli appropriati prima del loro utilizzo come partner coniugato nell'esperimento FRET13. BiFC utilizza la complementazione strutturale di parti N- e C-terminali della proteina fluorescente. Una limitazione comune nella maggior parte se non in tutte le proteine fluorescenti utilizzate nei saggi BiFC è stata l'autoassemblaggio tra i due frammenti non fluorescenti derivati, contribuendo alla fluorescenza falsamente positiva e diminuendo il rapporto segnale-rumore (S / N)14. Recenti sviluppi, tra cui mutazioni puntiformi o posizione di scissione della proteina fluorescente, hanno dato origine a coppie BiFC con maggiore intensità, maggiore specificità, alto rapporto S/N15,16. Queste proteine fluorescenti possono anche essere utilizzate per eseguire BiFC a seconda dell'idoneità dell'esperimento.
Tradizionalmente, CFP e YFP sono stati utilizzati come coppia donatore e accettore negli esperimenti FRET17. Tuttavia, YFP o m-Citrino sono risultati essere migliori donatori di FRET (se usati con RFP come accettore) a causa dell'elevata resa quantistica (QY) durante l'espressione nativa delle proteine bersaglio nel sistema radicale di Arabidopsis. Anche la selezione di promotori (costitutivi rispetto a quelli nativi/endogeni) e fluorofori svolgono un ruolo cruciale nella progettazione di un esperimento BiFC-FRET-FLIM di successo. È essenziale notare che l'efficienza dei donatori di FRET e l'idoneità delle coppie FRET tendono a cambiare con il cambiamento del promotore e del sistema biologico utilizzato per l'espressione. Il QY del fluoroforo, che si riferisce alla sua luminosità, dipende dal pH, dalla temperatura e dal sistema biologico in uso. Suggeriamo che questi criteri siano attentamente considerati prima di scegliere la coppia di fluorofori per l'esperimento FRET. Il sistema biologico, i promotori e le proteine utilizzate per questo protocollo hanno funzionato bene con i fluorofori CFP-YFP per l'esperimento BiFC FRET-FLIM.
Nel presente studio, incorporiamo la funzione di FLIM per visualizzare l'interazione tra tre molecole proteiche utilizzando FRET basato su BiFC. In questa tecnica, due proteine sono etichettate con la proteina YFP divisa e la terza proteina con CFP. Poiché eravamo interessati a studiare l'interazione di un omodimero MADS-box protein (M) con una proteina sensore di calcio (C), queste proteine sono state etichettate con proteine fluorescenti nei vettori pSITE-1CA e pSITE-3CA18. Due dei partner interagenti, in questo test, sono stati etichettati con parti N- e C-terminali della YFP nei vettori pSPYNE-35S e pSPYCE-35S19e la loro interazione si traduce nella ricostituzione della YFP funzionale che funge da accettore FRET per il terzo partner interagente, che è taggato con CFP (che agisce come donatore FRET) (Figura 1 ). In questo caso particolare, il PPI tra due monomeri M e tra M e C è stato convalidato eseguendo BiFC in tre diversi sistemi insieme al sistema lievito-due-ibrido. Questi vettori sono stati mobilitati nel ceppo Agrobacterium tumifaciens GV3101 mediante elettroporazione. Il ceppo GV3101 ha un plasmide Ti disarmato pMP90 (pTiC58DT-DNA) con resistenza alla gentamicina20. Un ceppo di Agrobacterium p19 è stato aggiunto insieme a tutte le infiltrazioni per prevenire il silenziamento transgenico21. Raccomandiamo che le tre proteine siano utilizzate anche in conformazioni opposte per convalidare le interazioni tripartite.
In questa tecnica, abbiamo impiegato FLIM, dove in primo luogo, la durata di fluorescenza del donatore (vita del donatore inestinguibile) viene misurata in assenza di un accettore. Successivamente, la sua durata viene misurata in presenza dell'accettore (vita del donatore estinto). Questa differenza nella durata di fluorescenza del donatore viene utilizzata per calcolare l'efficienza FRET, che dipende dal numero di fotoni che presentano una riduzione della durata di fluorescenza. Di seguito è menzionato un protocollo dettagliato per determinare la formazione di un complesso ternario tra tre proteine qualsiasi esprimendo transitoriamente le proteine marcate fluorescenti in Nicotiana benthamiana e analizzando la loro interazione con BiFC-FRET-FLIM.
Protocollo
1. Clonazione di geni in vettori di entrata e di destinazione (Figura 2)
- Amplificare la sequenza codificante (CDS) dei geni di interesse (geni M e C nel nostro caso) mediante PCR e clonarli in opportuni vettori di ingresso (ad esempio, vettore pENTR/D-TOPO; vedi Tabella 1 per i vettori utilizzati in questo esperimento).
- Coltiva i cloni su piastre contenenti antibiotici. Convalidare i cloni che vengono selezionati sugli antibiotici mediante digestione di restrizione e sequenziamento del DNA22,23.
- Mobilitare i CDS ricostituiti dai cloni di ingresso ai vettori di destinazione (pSPYNE-35S, pSPYCE-35S, pSITE-1CA e pSITE-3CA) e confermare il trasferimento di sequenze dai vettori di ingresso ai vettori di destinazione mediante digestione enzimatica di restrizione.
NOTA: tutti i vettori utilizzati in questo esperimento sono elencati nella Tabella 1. - Infine, trasformare le celle Agrobacterium GV3101 (pMP90 (GentR)) con i vettori di destinazione per elettroporazione (Figura 3)24.
2. Condizioni di crescita per le piante di Nicotiana benthamiana
NOTA: Coltivare piante di Nicotiana fino allo stadio di 4-6 foglie in condizioni di controllo.
- Per coltivare le piante di Nicotiana, preparare il mix di terreno mescolando miscele di terreno disponibili in commercio con cocopeat e compost in un rapporto di 2: 1: 1.
- Stendere uno strato di 1 pollice di spessore di questa miscela di terreno in un vassoio di plastica per rendere il letto del terreno e saturarlo con acqua deionizzata. Cospargere circa 200 semi in questo letto di terreno.
- Trasferirlo in un vassoio più grande contenente 1 cm di acqua stagnante. Coprire questo vassoio con un involucro di plastica per creare una camera di umidità.
- Trasferire questa configurazione in una camera di crescita impostata a 23 °C con 16 ore di luce e 8 ore di ciclo di buio con intensità luminosa di 150-170 μmol/m2s.
- Dopo due settimane, trasferire le giovani piantine in piccoli vasi da 3-4 pollici contenenti miscela di terreno saturo d'acqua.
- Metti questi vasi in vassoi di plastica e trasferiscili nella camera di crescita per altre quattro settimane.
3. Preparare ceppi batterici per l'agro-infiltrazione
NOTA: Per l'agro-infiltrazione, i ceppi batterici devono essere appena sottocoltati e miscelati insieme al ceppo p19 di Agrobacterium in rapporti appropriati.
- Preparare piastre di agar 2xYT contenenti rifampicina (100 μg/mL), gentamicina (25 μg/mL) e kanamicina (50 μg/mL) per Agrobacterium che ospita il vettore pSPYNE-35S e pSPYCE-35S. Per il vettore pSITE contenente ceppo, utilizzare rifampicina (100 μg/mL), gentamicina (25 μg/mL) e spectinomicina (50 μg/mL).
- Strisciare i ceppi di Agrobacterium contenenti i plasmidi su queste piastre utilizzando anelli di inoculazione sterili in una cappa a flusso laminare.
- Incubarli a 28 °C per 48 ore al buio.
- Iniziare questa procedura inoculando il ceppo Agrobacterium GV3101 che ospita costrutti BiFC e FRET (preparati in vettori pSPYNE-35S, pSPYCE-35S e pSITE) da piastre striate in 10 mL di brodo 2xYT contenente antibiotici appropriati (rifampicina (100 μg / mL), gentamicina (25 μg / mL), kanamicina (50 μg / mL) o spectinomicina (50 μg / mL)).
- Inoltre, iniziare una coltura del ceppo p19 di Agrobacteria inoculando 10 mL di brodo 2xYT contenente rifampicina (100 μg / mL) e kanamicina (50 μg / mL).
NOTA: il ceppo p19 viene aggiunto per evitare il silenziamento transgenico. - Coprire il matraccio con un foglio di alluminio e tenerli nello shaker dell'incubatrice a 28 °C e 170 giri/min per 16 ore al buio.
- Dopo la crescita durante la notte, trasferire 1 mL di questa coltura in una cuvetta usa e getta per misurare la densità ottica (O.D.) delle colture a 600 nm utilizzando uno spettrofotometro.
- Mescolare le colture di biFC appropriato e partner FRET contenenti ceppi in modo che l'O.D. finale di ciascuna coltura sia 0,5 e quello di p19 sia 0,3 in un volume totale di 2 ml.
- Per ottenere questi rapporti, utilizzare la formula indicata di seguito:
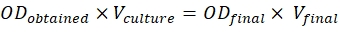
ODottenuto = O.D. della coltura misurata a 600 nm
Vcultura = Volume della lingua richiesta
ODfinale = 0,5 per i costrutti e 0,3 per p19
Vfinale = Volume finale per l'infiltrazione, che è di 2 ml
NOTA: le combinazioni di costrutti utilizzate in questo studio sono specificate nella Tabella 2. - Centrifugare le colture miste di Agrobacterium a 3.000 x g per 5 minuti a temperatura ambiente e scartare accuratamente il surnatante. Risospendare il pellet in 2 mL di tampone di infiltrazione appena preparato (10 mM MES, 100 μM di Acetosiringone e 10 mM MgCl2). Utilizzare un miscelatore a vortice per creare una sospensione cellulare omogenea.
- Incubare i tubi contenenti cellule risospese al buio a temperatura ambiente per 3 ore.
- Nel frattempo, etichetta ogni vaso con la miscela di costruzione con cui si infiltrerà. Utilizzare due piante per ogni miscela di infiltrazione.
- Riempire una siringa senza ago da 1 mL con la miscela agrobatterica. Premere delicatamente ma con fermezza la siringa sul lato abassiale della foglia completamente espansa mentre si sostiene la foglia dall'altro lato. Spingere delicatamente lo stantuffo fino a quando le soluzioni si riempiono nell'area fogliare equivalente a 2-3 volte la punta della siringa.
- Infiltrare fino a quattro punti su una foglia e 3-4 foglie per pianta, come mostrato nella Figura 4.
NOTA: Cambiare i guanti o pulire i guanti con il 70% di alcol tra i campioni per evitare contaminazioni incrociate. - Trasferire tutti i vasi in un vassoio e incubare in una camera di crescita nelle stesse condizioni menzionate nel passaggio 2.
- Controllare una piccola parte della foglia agroinfiltrata in diversi punti temporali utilizzando un microscopio a fluorescenza. Quando la fluorescenza di YFP e CFP è rilevabile nelle cellule, procedere al microscopio confocale per il test BiFC-FRET FLIM. In questo esperimento, l'analisi è stata effettuata 3 giorni dopo l'agro-infiltrazione.
NOTA: Impostare il periodo di incubazione post-agroinfiltrazione individualmente per ogni combinazione di promotori e geni per evitare la sovraespressione delle proteine chimeriche utilizzate nel test BiFC-FRET FLIM. La sovraespressione delle proteine partner può portare a interazioni false positive.
4. Preparare le diapositive per la visualizzazione a fluorescenza
- Quando le piante sono pronte per la visualizzazione, tagliare campioni di foglie quadrate, a 5-8 mm di distanza dalla ferita di infiltrazione, e montarli in acqua distillata su scivoli puliti.
NOTA: Per ridurre al minimo la fluorescenza di fondo, pulire i vetrini con etanolo all'80% seguito da acqua distillata 3-4 volte, asciugarli all'aria e tenerli su un foglio assorbente. - Coprire il campione di foglie con una copertina pulita e sigillare usando uno smalto per unghie.
- Visualizza questi campioni sotto un microscopio a scansione laser confocale.
5. Analisi FRET-FLIM con microscopio a scansione laser confocale
NOTA: In questa procedura, la base per determinare e quantificare l'interazione tra due proteine è la riduzione della durata di fluorescenza del partner donatore di FRET sulla sua interazione con l'accettore, che viene utilizzata per calcolare l'efficienza di FRET. La complessità nel caso dell'interazione tripartita aumenta ulteriormente perché l'accettore FRET, in questo caso, non è una singola molecola ma una coppia YFP-BiFC divisa, che dovrebbe prima essere ricostituita in vivo per diventare un fluoroforo fret-accettore funzionale. Per eseguire FRET-FLIM, è necessario determinare la durata di fluorescenza della molecola donatrice, prima da sola e poi in presenza di un partner FRET.
- Aprire l'applicazione FLIM nel microscopio a scansione laser confocale, avviare la console e utilizzare il conteggio dei fotoni di riconoscimento dei modelli per misurare la durata della fluorescenza. Selezionare la modalità di misurazione standard "Tutto il conteggio dei fotoni".
- Analizza campioni di due tipi di piante agro-infiltrate: una con il solo donatore (C-CFP) e l'altra con il donatore e l'accettore (C-CFP, insieme a M-YFP) entrambi.
NOTA: Poiché l'interazione della proteina M è già stata convalidata con la proteina C utilizzando BiFC e Y2H, ci si aspetta una buona efficienza FRET con questa coppia interagente. - Quindi, scansiona la foglia agro-infiltrata C-CFP e concentrati su una cellula che mostra una buona fluorescenza CFP. Avviare la modalità di scansione laser e impostare il sistema per la visualizzazione CFP e le misurazioni FLIM (λex laser a impulsi 440 nm, λem 480-520 nm da rilevatori ibridi, velocità di scansione 512 x 512 pixel a 400 Hz).
- Regola la messa a fuoco, lo zoom e il guadagno intelligente per mettere a fuoco l'area che deve essere acquisita.
- Illuminare il campione con una potenza laser sufficiente per ottenere la cattura di circa 0,1 fotoni per impulso. Per i campioni con intensità di fluorescenza variabile, acquisire 50 fotogrammi per raccogliere i fotoni adeguati necessari per la misurazione della durata. CFP presenta due durate di fluorescenza grazie al suo adattamento conformazionale; pertanto, adattare i dati utilizzando il modello di riconvoluzione n-esponenziale mantenendo il valore di n uguale a 2.
- A queste impostazioni, la STAMPANTE digitale mostra due vite di 1,0 e 3,2 ns. Qui la vita più alta, 3,2 ns, viene utilizzata per tutti i calcoli successivi25,26.
- Per calcolare l'efficienza FRET utilizzando FLIM, che è la misura del grado di interazione tra due proteine, prelevare un campione di foglie che è stato co-infiltrato con C-CFP e M-YFP. Cerca una cella che esprima sia C-CFP che M-YFP e conferma i rispettivi modelli di emissione eccitandoli usando λex 440 nm laser a impulsi, λem 480-520 nm e λex 514 nm laser a luce bianca con λem 526-550 nm. Scansiona e identifica in sequenza una cellula che mostra sia la fluorescenza CFP che YFP.
- Dopo aver confermato la fluorescenza di entrambe le proteine, passare alla console FLIM per misurare la durata della CFP utilizzando le stesse impostazioni utilizzate in precedenza per misurare la durata della C-CFP (passaggio 5.5).
NOTA: questa cella esprime anche M-YFP che può potenzialmente interagire con C-CFP e causare una riduzione della durata di C-CFP. - Adatta il grafico ottenuto usando il modello di riconvoluzione n-esponenziale, con n = 2. È stata osservata una diminuzione della durata della CFP da 3,2 a 2,6 ns, indicando il trasferimento di energia di risonanza di Förster tra CFP e YFP (Figura 5A).
- Ora avvia la console FRET nel software e calcola l'efficienza FRET inserendo manualmente la durata del donatore inestinguibile nell'equazione fornita nel software. E l'efficienza FRET osservata è: 56%.
- Interazione tripartita
- Infine, per visualizzare le interazioni tra tre partner, prelevare il campione di foglie da una pianta che è stata co-infiltrata con C-CFP, M-YFPn e M-YFPc.
- Scansiona l'espianto fogliare per una cellula che mostra sia CFP che fluorescenza YFP ricostituita che emana dall'interazione BiFC tra due proteine M. Utilizzare le stesse lunghezze d'onda laser ed di emissione utilizzate in precedenza.
- Successivamente, spegnere il laser a 514 nm e passare alla console FLIM.
NOTA: Se il dimero M-YFP interagisce con C-CFP, si dovrebbe vedere una riduzione della durata di C-CFP come osservato durante la sua interazione con M-YFP. Tuttavia, se la C-CFP non riesce a interagire con il dimero M-YFP, la sua durata di fluorescenza dovrebbe rimanere a 3,2 ns. - Utilizzando impostazioni simili a quelle sopra menzionate, misurare la durata della CFP in presenza di YFP ricostituita. Adatta il grafico ottenuto usando il modello di ricovoluzione n-esponenziale, con n = 2, e spostati sulla console FRET.
NOTA: c'è un calo della durata della PCP da 3,2 a 2,3 ns. Calcola l'efficienza FRET come descritto sopra. L'efficienza FRET calcolata è del 55%. La riduzione della durata di vita del donatore e una buona efficienza FRET del 55% confermano l'interazione tripartita tra due proteine M e la proteina C in vivo (vedi Figura 5B).
Risultati
Questo protocollo rappresenta un metodo ottimizzato per studiare in vivo le interazioni tripartite proteina-proteina nelle piante. Il principio di base del protocollo è quello di combinare due tecniche di interazione proteica marcate con fluorescenza, cioè BiFC e FRET, per creare un test per misurare la formazione di complessi ternari tra tre partner proteici. Qui, abbiamo usato FLIM per misurare la durata di fluorescenza del partner donatore FRET in presenza e assenza dell'accettore FRET. Ci si aspettava una ...
Discussione
Il presente protocollo dimostra l'uso del test FRET-FLIM basato su BiFC per accertare la formazione di un complesso ternario tra due monomeri di una proteina MADS-box e una proteina sensore di calcio. Il protocollo è adattato da un rapporto di Y. John Shyu et al. dove hanno sviluppato un metodo FRET basato su BiFC per visualizzare il complesso ternario formato tra eterodimeri Fos-Jun e NFAT o p65 utilizzando il metodo di emissione sensibilizzata7. In precedenza, un sistema FRET a tre fluorofori ?...
Divulgazioni
Gli autori non dichiarano conflitti di interesse.
Riconoscimenti
NB, GG, SB, KC ringraziano sinceramente la University Grants Commission (UGC), UGC-BSR, DBT-INSPIRE e il Council for Scientific and Industrial Research (CSIR) per le loro borse di ricerca. Per fortuna riconosciamo il Dipartimento di Biotecnologia (DBT), il Governo dell'India, il Dipartimento di Scienza e Tecnologia (DST-FIST), il Governo dell'India per il sostegno finanziario.
Materiali
| Name | Company | Catalog Number | Comments |
| 1 ml Syringes without needles | Dispovan | - | |
| Acetosyringone | Sigma-Aldrich | D134406 | |
| Gateway LR Clonase II Enzyme mix | Thermo Fischer Scientific | 11791020 | The vectors used in the study are Gateway based |
| Gentamycin Sulphate | Himedia | CMS461 | |
| Kanamycin Sulphate | Himedia | MB105 | |
| MES hydrate | Sigma-Aldrich | M2933 | |
| MgCl2 | Sigma-Aldrich | M2670 | |
| pENTR/D-TOPO Cloning Kit | Thermo Fischer Scientific | K240020 | The vectors used in the study are Gateway based |
| Phusion high fidelity Taq DNA polymerase | Thermo Fischer Scientific | F530-S | Any High fidelity Polymerase can work |
| Rifampicin | Himedia | CMS1889 | |
| SP8 FALCON Confocal laser scanning microscope | Leica | SP8 FALCON | Any CLSM with FLIM capabilities can be used for this analysis |
| Spectinomycin dihydrochloride pentahydrate | Himedia | TC034 |
Riferimenti
- Grove, C. A., Walhout, A. J. M. Transcription factor functionality and transcription regulatory networks. Molecular Biosystem. (4), 309-314 (2008).
- Amoutzias, G. D., Robertson, D. L., Van de Peer, Y., Oliver, S. G. Choose your partners: dimerization in eukaryotic transcription factors. Trends in Biochemical Sciences. 33 (5), 220-229 (2008).
- Arora, R., et al. MADS-box gene family in rice: genome-wide identification, organization and expression profiling during reproductive development and stress. BMC Genomics. 8 (242), (2007).
- Theißen, G., Gramzow, L. Structure and evolution of plant MADS domain transcription factors. Plant Transcription Factors: Evolutionary, Structural and Functional Aspects. , 127-138 (2016).
- Schweiger, R., Schwenkert, S. Protein-protein interactions visualized by bimolecular fluorescence complementation in tobacco protoplasts and leaves. Journal of Visualized Experiments: JoVE. (85), (2014).
- Bracha-Drori, K., et al. Detection of protein-protein interactions in plants using bimolecular fluorescence complementation. Plant Journal. 40 (3), 419-427 (2004).
- Shyu, Y. J., Suarez, C. D., Hu, C. Visualization of ternary complexes in living cells by using a BiFC-based FRET assay. Nature Protocols. 3 (11), 1693-1702 (2008).
- Kwaaitaal, M., Keinath, N. F., Pajonk, S., Biskup, C., Panstruga, R. Combined bimolecular fluorescence complementation and förster resonance energy transfer reveals ternary SNARE complex formation in living plant cells. Plant Physiology. 152 (3), 1135-1147 (2010).
- Margineanu, A., et al. Screening for protein-protein interactions using Förster resonance energy transfer (FRET) and fluorescence lifetime imaging microscopy (FLIM). Scientific Reports. 6, (2016).
- Datta, R., Heaster, T. M., Sharick, J. T., Gillette, A. A., Skala, M. C. Fluorescence lifetime imaging microscopy: fundamentals and advances in instrumentation, analysis, and applications. Journal of Biomedical Optics. 25 (07), 1 (2020).
- Long, Y., et al. Optimizing FRET-FLIM labeling conditions to detect nuclear protein interactions at native expression levels in living Arabidopsis roots. Frontiers in Plant Science. 9, 1-13 (2018).
- Burman, N., Chandran, D., Khurana, J. P. A Rapid and highly efficient method for transient gene expression in rice plants. Frontiers in Plant Science. , 11 (2020).
- Kudla, J., Bock, R. Lighting the way to protein-protein interactions: Recommendations on best practices for bimolecular fluorescence complementation analyses. Plant Cell. 28 (5), 1002-1008 (2016).
- Shyu, Y. J., Hu, C. D. Fluorescence complementation: an emerging tool for biological research. Trends in Biotechnology. 26 (11), 622-630 (2008).
- Kodama, Y., Hu, C. D. An improved bimolecular fluorescence complementation assay with a high signal-to-noise ratio. BioTechniques. 49 (5), 793-803 (2010).
- Kodama, Y., Hu, C. D. Bimolecular fluorescence complementation (BiFC): A 5-year update and future perspectives. BioTechniques. 53 (5), 285-298 (2012).
- Piston, D. W., Kremers, G. J. Fluorescent protein FRET: the good, the bad and the ugly. Trends in Biochemical Sciences. 32 (9), 407-414 (2007).
- Chakrabarty, R., et al. pSITE vectors for stable integration or transient expression of autofluorescent protein fusions in plants: Probing Nicotiana benthamiana- Virus Interactions. Molecular Plant-Microbe Interactions. 20 (7), 740-750 (2007).
- Walter, M., et al. Visualization of protein interactions in living plant cells using bimolecular fluorescence complementation. Plant Journal. 40 (3), 428-438 (2004).
- Hellens, R., Mullineaux, P., Klee, H. A guide to Agrobacterium binary Ti vectors. Trends in Plant Science. 5 (10), 446-451 (2000).
- Van Der Hoorn, R. A. L., Rivas, S., Wulff, B. B. H., Jones, J. D. G., Joosten, M. H. A. J. Rapid migration in gel filtration of the Cf-4 and Cf-9 resistance proteins is an intrinsic property of Cf proteins and not because of their association with high-molecular-weight proteins. Plant Journal. 35 (3), 305-315 (2003).
- Xie, X., et al. Engineering SARS-CoV-2 using a reverse genetic system. Nature Protocols. 16, (2021).
- Xu, J., et al. Optimized plasmid construction strategy for Cas9. Cellular Physiology and Biochemistry. 48, 131-137 (2018).
- Mattanovich, D., et al. Efficient transformation of Agrobacterium spp. by electroporation. Nucleic Acids Research. 17 (16), 6747 (1989).
- Rizzo, M. A., Springer, G. H., Granada, B., Piston, D. W. An improved cyan fluorescent protein variant useful for FRET. Nature Biotechnology. 22 (4), 445-449 (2004).
- Tramier, M., et al. Picosecond-hetero-FRET microscopy to probe protein-protein interactions in live cells. Biophysical Journal. 83 (6), 3570-3577 (2002).
- Alvarez, L. A. J., et al. SP8 FALCON: a novel concept in fluorescence lifetime imaging enabling video-rate confocal FLIM. Nature Methods. 20, 2-4 (2019).
- Postma, M., Goedhart, J. Plotsofdata-a web app for visualizing data together with their summaries. PLoS Biology. 17 (3), 1-8 (2019).
- Galperin, E., Verkhusha, V. V., Sorkin, A. Three-chromophore fret microscopy to analyze multiprotein interactions in living cells. Nature Methods. 1 (3), 209-217 (2004).
- Waadt, R., Kudla, J. In plant visualization of protein interactions using bimolecular fluorescence complementation (BiFC). Cold Spring Harbor Protocols. 3 (4), (2008).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon