A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
تعزيز الاسترخاء المغنطيسي لاكتشاف وتوصيف الارتباطات الذاتية للبروتينات المضطربة جوهريا
In This Article
Summary
يتم تقديم بروتوكول لتطبيق التحليل الطيفي بالرنين المغناطيسي النووي لتعزيز الاسترخاء المغنطيسي للكشف عن التفاعلات الضعيفة والعابرة بين الجزيئات وداخلها في البروتينات المضطربة جوهريا.
Abstract
تشكل البروتينات المضطربة جوهريا والمناطق المضطربة جوهريا داخل البروتينات جزءا كبيرا ومهما وظيفيا من البروتين البشري. تسمح الطبيعة المرنة للغاية لهذه التسلسلات بتكوين تفاعلات ضعيفة وطويلة المدى وعابرة مع شركاء جزيئيين حيويين متنوعين. تعمل التفاعلات المحددة ذات التقارب المنخفض على تعزيز الارتباط غير المشروع وتمكين جزء واحد مضطرب جوهريا من التفاعل مع العديد من المواقع المستهدفة. بسبب الطبيعة العابرة لهذه التفاعلات ، قد يكون من الصعب توصيفها بطرق البيولوجيا الهيكلية التي تعتمد على البروتينات لتشكيل شكل واحد سائد. يعد الرنين المغناطيسي النووي لتعزيز الاسترخاء المغنطيسي أداة مفيدة لتحديد وتعريف الأساس الهيكلي للتفاعلات الضعيفة والعابرة. تم وصف بروتوكول مفصل لاستخدام تعزيز الاسترخاء المغنطيسي لتوصيف مجمعات المواجهة منخفضة السكان التي تتشكل بين البروتينات المضطربة جوهريا وبروتينها أو حمضها النووي أو شركاء جزيئيين حيويين آخرين.
Introduction
يصف الاضطراب الداخلي (ID) البروتينات (IDPs) أو المناطق داخل البروتينات (IDRs) التي لا تطوى تلقائيا في هياكل ثانوية أو ثالثية مستقرة ولكنها نشطة بيولوجيا. بشكل عام ، تتمثل وظيفة IDP / IDRs في تسهيل تفاعلات محددة ولكن قابلة للعكس مع الجزيئات الحيوية في الظروف الفسيولوجية1. وهكذا، يشارك النازحون داخليا والنازحون داخليا في مجموعة من الوظائف الخلوية، بما في ذلك تجنيد وتنظيم وتثبيت المجمعات متعددة البروتينات، على سبيل المثال، تجميع ونشاط المركب2، وتجنيد وتنظيم المكونات في مواقع تلف الحمض النووي3، وتنظيم وتثبيت تجنيد معقدات النسخ4، أو معيد تشكيل الكروماتين BAF5. بالإضافة إلى ذلك، يوجد النازحون داخليا في روابط الإشارة حيث تمكنهم اختلاطهم مع شركاء ملزمين مختلفين من التوسط في نقل المعلومات من خلال شبكات البروتين الخلوي6. كما كشفت الأعمال الأخيرة عن ميل مناطق IDR إلى ربط المكثفات الجزيئية الحيوية ذاتية التكوين من خلال عملية فصل الطور السائلعن السائل 7. يعتقد الآن أن العديد من الوظائف المذكورة أعلاه التي تتضمن ID تتضمن بعض جوانب تكوين المكثفات8. على الرغم من أهمية تحديد الهوية للتجميع المعقد الجزيئي الحيوي ، والاستقرار ، والسقالات ، ونقل الإشارات ، يصعب تحديد التفاصيل الذرية لتفاعلاتها المحددة نظرا لأن النازحين داخليا و IDRs عادة ما يكونون غير قابلين للتحقيقات الهيكلية باستخدام علم البلورات بالأشعة السينية أو المجهر الإلكتروني المبرد.
الرنين المغناطيسي النووي (NMR) هو تقنية مثالية لفحص الهوية لأنه لا يعتمد على وجود مجموعات هيكلية صلبة أو متجانسة ولكنه يقدم تقارير عن البيئة المحلية المباشرة للنوى الفردية. يتأثر تردد الرنين ، أو التحول الكيميائي ، للنواة في جزيء معين بالمجالات المغناطيسية الضعيفة التي يسببها التوزيع الإلكتروني المحلي ، والذي يعتمد بدوره على أطوال الروابط والزوايا وقرب النوى الأخرى والتفاعلات مع شركاء الربط وعوامل أخرى9. وبالتالي ، تعمل كل نواة كمسبار هيكلي فريد خاص بالموقع حساس للتغيرات في بيئته الكيميائية المحلية. على الرغم من هذه المزايا ، فإن الرنين المغناطيسي النووي هو تقنية سائبة ، والتحول الكيميائي المرصود هو متوسط جميع البيئات التي تم أخذ عينات منها بواسطة نواة معينة. تم تطوير مجموعة من تقنيات الرنين المغناطيسي النووي ، والتي تم وصف العديد منها في هذا العدد ، لاستعادة المعلومات الهيكلية والديناميكية والحركية حول المطابقات الجزيئية الحيوية عالية الطاقة ومنخفضة السكان الموجودة في متوسط التحول الكيميائي10,11. على الرغم من أن هذه الحالات مكتظة بالسكان بشكل عابر ، إلا أن تحديد هذه الحالات وقياسها كميا مهمان لتحديد تفاصيل الآليات الوظيفية12. على سبيل المثال، في حالة النازحين داخليا والنازحين داخليا، قد تكون المجموعة التوافقية متحيزة لأخذ عينات تفضيلية من المطابقات المنتجة لتشكيل مجمعات المواجهة مع شركاء الربط الفسيولوجي. يعد الكشف عن هذه الحالات ، وكذلك تحديد التفاعلات والديناميات الجزيئية الخاصة بالبقايا وداخلها ، أمرا مهما لتحديد الآليات الهيكلية الأساسية لوظيفة البروتين والتكوين المعقد.
تم وصف بروتوكول لاستخدام الرنين المغناطيسي النووي (PRE) لتعزيز الاسترخاء المغنطيسي (PRE) للتحقيق في الحالات العابرة ذات الكثافة السكانية المنخفضة المهمة لتشكيل المعقدات الجزيئية الحيوية بوساطة IDP / IDR13. هذا النهج مفيد لدراسة تفاعلات البروتين والبروتين العابرة مثل تلك التي تعزز تجميع ألياف الأميلويد من α-synuclein 14,15 أو الارتباط الذاتي ل FUS16 ، وكذلك لتوصيف تفاعلات البروتين والبروتين المحددة مثل بين بروتينات الإشارة17. يتم تقديم مثال على IDP المرتبط ذاتيا ، حيث تؤدي التفاعلات المحددة بين الجزيئات وداخلها إلى حالات مضغوطة بشكل تفضيلي بالإضافة إلى تفاعلات خاصة بالموقع تدفع الارتباط الذاتي.
ينشأ PRE من التفاعل المغناطيسي ثنائي القطب للنواة إلى مركز مغناطيسي مع موتر g متناحي الخواص ، يتم توفيره عادة في شكل إلكترون غير مزاوج على مجموعة نيتروكسيد أو كذرة فلز بارامغناطيسية18 (الشكل 1). في حين أن الذرات ذات الموترات g-tensors متباينة الخواص تنتج أيضا تأثير PRE ، فإن تحليل هذه الأنظمة يكون أكثر صعوبة بسبب التأثيرات المربكة التي تساهم بها تحولات الاتصال الزائفة (PCS) أو الاقتران ثنائي القطب المتبقي (RDC)13,19. تعتمد قوة التفاعل بين النواة والمركز المغنطيسي على المسافة <r-6> بين الاثنين. ينتج عن هذا التفاعل زيادة في معدلات الاسترخاء النووي ، مما يؤدي إلى توسيع الخط القابل للكشف حتى بالنسبة للتفاعلات طويلة المدى (~ 10-35 Å) ، لأن اللحظة المغناطيسية للإلكترون غير المزاوج قوية جدا20,21. يمكن اكتشاف الحالات العابرة باستخدام PRE إذا تم استيفاء الشرطين التاليين ؛ (1) التفاعل العابر في تبادل سريع على المقياس الزمني للرنين المغناطيسي النووي (التحول الكيميائي المرصود هو متوسط مرجح للسكان لحالات التبادل) ؛ و (2) تكون المسافة بين النوى والمركز المغنطيسي أقصر في الحالة المأهولة بالسكان بشكل عابر منها في الحالة الرئيسية11. يشار إلى PRE المستعرض Γ2 ، ولأغراض عملية ، يتم حسابه من الفرق في معدلات الاسترخاء المستعرض 1H بين عينة تحتوي على مركز مغناطيسي وتحكم مغناطيسي. للحصول على معالجة متعمقة لنظرية PRE وتحولات الاتصال الزائف ذات الصلة في أنظمة التبادل السريع والبطيء ، تتم إحالة القارئ إلى المراجعات الشاملة التي أجراها كلور وزملاؤهفي العمل 13،22. هنا ، يتم النظر فقط في الحالة التي يكون فيها 1H N-Γ2 في نظام التبادل السريع ، حيث بسبب اعتماد r-6 على PRE، يرتبط معدل الاسترخاء الملحوظ بكل من المسافة التي يقترب منها المركز المغنطيسي من النواة وكذلك مقدار الوقت الذي يقضيه في هذا التشكل. لذلك ، فإن المطابقات العابرة التي لا تنطوي على نهج وثيق تنتج PRE صغيرا بينما التفاعلات الأوثق ، حتى لو كانت قصيرة الأجل ، ستنتج PRE.
بالنسبة للنازحين داخليا ، يتم استخدام PRE لقياس وتمييز التفاعلات التي تحدث داخل جزيء واحد (داخل الجزيئات) وبين جزيئات منفصلة (بين الجزيئات). من خلال ربط مركز مغناطيسي ببروتين الرنين المغناطيسي النووي المرئي (على سبيل المثال ، 15N-labeld) أو NMR غير المرئي (على سبيل المثال ، الوفرة الطبيعية 14N) ، يمكن تحديد المصدر (بين أو داخل الجزء) من PRE (الشكل 2). يعد الطفرات الموجهة للموقع والتي تقدم بقايا السيستين طريقة ملائمة لربط مركز مغناطيسي (ملصق مغزلي) ببروتين23. تم اقتراح عدة أنواع من الجزيئات لاستخدامها كعلامات مغزلية ، بما في ذلك مخلب المعادن (القائم على EDTA) والجذور الحرة (القائمة على النيتروكسيد)24. تم وصف العديد من ملصقات دوران النيتروكسيد وهي متوفرة مع كيمياء مختلفة تفاعلية للسيستين مثل ميثانثيوسلفونات وماليميد ويودواسيتاميد25,26 (الشكل 1). قد تكون المرونة المتأصلة في العلامة أو الرابط مشكلة بالنسبة لبعض التحليلات ، وفي هذه الحالات ، تم اقتراح استراتيجيات مختلفة للحد من حركة العلامة ، مثل إضافة مجموعات كيميائية ضخمة أو استخدام رابط ثان لتثبيت العلامة على البروتين (ملحق موقعين)27 ، 28. بالإضافة إلى ذلك ، قد تحتوي العلامات المتاحة تجاريا على بروتينات دياستيرومريك ولكن هذا بشكل عام لن يساهم في PRE29 المرصود. يوصف استخدام 3-Maleimido-PROXYL المرتبط بالسيستين الحر عبر كيمياء ماليميد لأنه متاح بسهولة وفعال من حيث التكلفة وغير قابل للعكس ، ويمكن الحفاظ على عامل الاختزال tris (2-carboxyethyl) phosphine (TCEP) في المحلول طوال تفاعل وضع العلامات. نظرا لأن 3-Maleimido-PROXYL يحتوي على موتر g متناحي الخواص ، فلا يتم تحفيز PCS أو RDCs ، ويمكن استخدام نفس تخصيصات التحول الكيميائي لكل من العينات المغناطيسية والمغناطيسية13.
يتم قياس 1HN-T 2 باستخدام استراتيجية نقطتين زمنيتين (T a ، Tb) التي ثبت سابقا أنها دقيقة مثل جمع سلسلة تطور كاملة تتكون من 8 إلى 12 نقطة زمنية30. يتم تعيين النقطة الزمنية الأولى (T a) بالقرب من الصفر قدر الإمكاني ، ويعتمد الطول الأمثل للنقطة الزمنية الثانية على حجم أكبر PRE متوقع لعينة معينة ويمكن تقديره من: Tb ~ 1.15 / (R 2، dia + Γ 2) حيث R 2 ، dia يمثل R 2 للعينة المغناطيسية13. إذا كان حجم أكبر PREs غير معروف ، فإن ضبط T b على ~ مرة واحدة في 1H T 2 للبروتين هو تقدير أولي جيد ويتم تحسينه بشكل أكبر عن طريق ضبط T2 لتحسين الإشارة إلى الضوضاء. تقلل استراتيجية القياس المكونة من نقطتين بشكل كبير من الوقت التجريبي اللازم لقياس PREs وتتيح الوقت لمزيد من متوسط الإشارة ، خاصة وأن العينات المخففة نسبيا تستخدم لتقليل آثار الاتصالات غير المحددة بين الجزيئات. يتم استخدام تسلسل النبض القائم على HSQC لقياس 1H N-T2 وقد تم وصفه بالتفصيل في مكان آخر30. لتحسين الحساسية ، يمكن استبدال النبضات الصلبة لعمليات نقل INEPT الأمامية والخلفية بنبضات على شكل ؛ بدلا من ذلك ، يتم تحويل التسلسل بسهولة إلى قراءاتتستند إلى TROSY-based 31. وبما أن النازحين داخليا عادة ما يكون لديهم معدلات استرخاء عرضية أطول بكثير مما يؤدي إلى عرض خط أضيق (بسبب الاضطراب المتأصل) من البروتينات الكروية ذات الحجم المماثل، يمكن استخدام أوقات اكتساب طويلة في البعد غير المباشر لتحسين الاستبانة الطيفية والتخفيف من قيود تشتت التحول الكيميائي المتأصلة في النازحين داخليا.
PRE هي أداة مفيدة لدراسة تفاعلات البروتين والبروتين والحمض النووي ، وخاصة التفاعلات العابرة أو منخفضة السكان. يتم توفير بروتوكول مفصل لإعداد عينة الرنين المغناطيسي النووي المناسبة لقياس PREs ، بما في ذلك خطوات تنقية البروتين ، ووضع العلامات الدورانية الموجهة للموقع ، وإعداد ومعايرة برنامج النبض ، ومعالجة وتفسير بيانات الرنين المغناطيسي النووي. لوحظت اعتبارات تجريبية مهمة في جميع أنحاء والتي قد تؤثر على جودة البيانات والنتائج التجريبية ، بما في ذلك تركيز العينة ، واختيار التسمية المغزلية ، وإزالة المكونات البارامغناطيسية.
Protocol
المتطلبات العامة للبروتوكول: مرافق تنقية البروتين ، مطياف الأشعة المرئية وفوق البنفسجية ، مطياف الرنين المغناطيسي النووي عالي المجال وبرمجيات التشغيل ، برامج تحليل ما بعد المعالجة بما في ذلك ؛ NMRPipe32 أو Sparky33 (أو تحليل CCPN 34 أو NMRViewJ35).
1. التعبير المؤتلف وتنقية البروتين لقياسات PRE
- صمم بنية تعبير للبروتين محل الاهتمام بحيث يكون هناك بقايا سيستين واحدة. ستكون هناك حاجة إلى طفرات متعددة لإدخال السيستين الحر في مواضع مختلفة في البروتين محل الاهتمام36.
- التعبير عن وتنقية وفرة طبيعية (14N) أو 15N المسمى عينة من البروتين محل الاهتمام باستخدام بروتوكول المعمول به37.
ملاحظة: توفر أنظمة التعبير عن الإشريكية القولونية طريقة فعالة من حيث التكلفة وقوية لتعبير البروتين المؤتلف لأن التخصيب النظيري ل 15نيوتن هو الحد الأدنى من المتطلبات للتحليل الطيفي للرنين المغناطيسي النووي غير المتجانس الجزيئي الحيوي. الخطوات النموذجية هي التعبير في الحد الأدنى من الوسائط ، والتنقية الكروماتوغرافية ، وإزالة علامة تنقية التقارب. يفترض هذا البروتوكول أنه تم إنشاء بروتوكول قوي للتعبير والتنقية يمكنه إنتاج بروتين كاف بجودة مناسبة لتحقيقات الرنين المغناطيسي النووي.- الحفاظ على عامل اختزال 1 مللي متر (DTT أو TCEP) في المخازن المؤقتة في جميع خطوات التنقية لمنع تفاعل السيستين الحر وتشكيل روابط ثاني كبريتيد بين الجزيئات للنازحين داخليا.
ملاحظة: قد تكون بعض الأنظمة أكثر تحملا وأقل عرضة للتجميع للظروف غير المختزلة اعتمادا على الخصائص المحددة للبروتين ، بالإضافة إلى درجة الحرارة ودرجة الحموضة ونظام المخزن المؤقت المختار للتنقية38. - قم بإزالة علامات التقارب المستخدمة للتنقية قبل المتابعة لأنها قد تتفاعل بشكل غير محدد مع البروتين بطرق غير متوقعة أو ربما تحتوي على بقايا السيستين التفاعلية التي يمكن أن تكون بمثابة موقع ارتباط غير مقصود.
- قم بإعداد عينة مرجعية موسومة 15N بدون طفرة (طفرات) السيستين ممزوجة بنسخة قابلة للذوبان من ملصق الدوران لتقييم مساهمة PREs المذيبات.
- الحفاظ على عامل اختزال 1 مللي متر (DTT أو TCEP) في المخازن المؤقتة في جميع خطوات التنقية لمنع تفاعل السيستين الحر وتشكيل روابط ثاني كبريتيد بين الجزيئات للنازحين داخليا.
2. تصريف ملصق 3-ماليميدو-بروكسيل نيتروكسيد
- تخزين أو تبادل البروتين المنقى في مخزن مؤقت منزوع الغازات يحتوي على 50 mM Tris pH 7 و 1 mM TCEP ؛ قد يحتوي المخزن المؤقت أيضا على ما يصل إلى 8 ملايين من اليوريا إذا لزم الأمر للمساعدة في ذوبان البروتين.
بدلا من ذلك ، قم بتخفيف محلول مخزون البروتين بسرعة إلى ما لا يقل عن 10 مكافئات حجم من 50 mM Tris pH 7 و 1 mM TCEP buffer. تأكد من أن تركيز البروتين قبل إضافة ملصق الدوران لا يقل عن 100 ميكرومتر. - أضف 3-ماليميدو بروكسيل من محلول مخزون إلى 20x مولار فائض من البروتين محل الاهتمام. حماية العينة من الضوء والأكسجين واحتضانها طوال الليل في درجة حرارة الغرفة أو 4 درجات مئوية ؛ قد يؤدي التأرجح اللطيف أو التعويذة إلى تحسين كفاءة وضع العلامات.
- تحضير محاليل المخزون من الملصق الدوار عن طريق إذابة مسحوق بروكسيل 3-Maleimido في 95٪ إيثانول. يمكن تخزين حصص المخزون عند -80 درجة مئوية لمدة تقل عن 6 أشهر.
- الخطوة الحاسمة: قم بإزالة ملصق الدوران الحر غير المتفاعل لمنع المذيبات غير المحددة. حقق ذلك عن طريق ترشيح الهلام أو (يفضل) غسيل الكلى الشامل لعينة البروتين. ستؤدي هذه الخطوة أيضا إلى إدخال البروتين في مخزن مؤقت مناسب للرنين المغناطيسي النووي.
ملاحظة: يجب تحضير عوامل الاختزال طازجة ، ويجب مراعاة التوافق بين مكونات المخزن المؤقت ؛ على سبيل المثال ، يتحلل TCEP بسرعة في المخازن المؤقتة القائمة على الفوسفات ، ويجب تجنب هذا المزيج39. - عالج جميع المخازن المؤقتة المستخدمة من هذه الخطوة إلى الأمام باستخدام راتنج مخلب انتقائي للمعادن ثنائية التكافؤ والانتقالية لإزالة الأيونات البارامغناطيسية أو أجهزة التبريد ذات الملصقات الدورانية. إذا تعذر تخزين البروتين في مخزن مؤقت للرنين المغناطيسي النووي ، فقم بتركيز البروتين لتخفيفه بسرعة في مخزن مؤقت مناسب للرنين المغناطيسي النووي.
- مراقبة كفاءة دمج الملصقات الدورانية.
- استخدم كاشف إيلمان (5،5-ديثيو-مكرر- (2-حمض النيتروبنزويك) لتحديد مجموعات سلفهيدريل الحرة في المحلول40.
ملاحظة: تتوفر بروتوكولات مفصلة من الشركة المصنعة. للأغراض هنا ، من المهم تحديد دمج الملصق المغزلي ، تتم مقارنة تركيز مجموعات سلفهيدريل الحرة مع تركيز البروتين الكلي. النسبة المئوية لمجموعات سلفهيدريل الحرة هي النسبة المئوية للجزيئات التي لا تحتوي على ملصق دوران نيتروكسيد مرفق. - راقب شدة الذروة المقابلة لبقايا السيستين الموسومة للحكم على دمج التسمية المغزلية في البروتين محل الاهتمام.
ملاحظة: هذا هو نهج سريع وفعال لتحديد درجة وضع العلامات على دوران البروتين. سيؤدي الدمج الكامل للتسمية المغزلية إلى اختفاء الذروة من الطيف. ومع خاصية التشتت الضعيفة للمشردين داخليا، قد لا يتم دائما تحديد الذروة المقابلة لبقايا السيستين الطافرة بسهولة، وبالتالي يوصى باستخدام كاشف إيلمان (الخطوة 2-6-1).
- استخدم كاشف إيلمان (5،5-ديثيو-مكرر- (2-حمض النيتروبنزويك) لتحديد مجموعات سلفهيدريل الحرة في المحلول40.
3. تحضير عينة الرنين المغناطيسي النووي لقياس PRE داخل أو بين الجزيئات
- تحضير عينة لقياس PRE داخل الجزيئات
- تحضير 15N بروتين مخصب نظائريا وموسوم بالدوران بتركيز لا يقل عن 100 ميكرومتر ولكن ليس أكثر من 300 ميكرومتر في مخزن مؤقت مناسب للرنين المغناطيسي النووي. إجمالي حجم العينة (بما في ذلك D2O) هو 500 - 550 ميكرولتر.
ملاحظة: تشمل مخازن الرنين المغناطيسي النووي الشائعة الفوسفات والأسيتات والكربونات (الثنائية) و TRIS. قد تكون المخازن المؤقتة الجيدة مثل MES و HEPES مناسبة أيضا. توخ الحذر عند اختيار المخازن المؤقتة لضمان عدم التفاعل المتبادل مع مكونات الحل الأخرى. - تأكد من أن الرقم الهيدروجيني ~ 7.2 أو أقل لتقليل آثار تبادل بروتون الأميد مع الماء. حافظ على تركيز الملح عند أدنى مستوى ممكن (عادة أقل من 150 مللي مول) ، على الرغم من أن الاعتبار الأساسي هو الحفاظ على استقرار البروتين.
ملاحظة: تم وصف نهج إجراء تجارب الرنين المغناطيسي النووي في ظروف عالية الملح في مكان آخر41.
- تحضير 15N بروتين مخصب نظائريا وموسوم بالدوران بتركيز لا يقل عن 100 ميكرومتر ولكن ليس أكثر من 300 ميكرومتر في مخزن مؤقت مناسب للرنين المغناطيسي النووي. إجمالي حجم العينة (بما في ذلك D2O) هو 500 - 550 ميكرولتر.
- تحضير عينة لقياس PRE بين الجزيئات
- اتبع هذه الخطوة أو الخطوة 3.1 ؛ لا يتم تنفيذها في وقت واحد. قم بإعداد 14N وفرة طبيعية ، بروتين يحمل علامة تدور في المخزن المؤقت NMR المختار.
- قم بإعداد عينة البروتين عن طريق خلط 15N بروتين مخصب نظائريا غير موسوم بالدوران مع 1٪ -50٪ 14N بروتين ذو وفرة طبيعية بحيث يكون التركيز النهائي مطابقا للعينة المحضرة في 3.1.1. إجمالي حجم العينة (بما في ذلك D2O) هو 500 - 550 ميكرولتر.
- تحسين نسبة البروتينات 15N و 14N تجريبيا لكل بروتين تمت دراسته. تعد نسب 1٪ و 5٪ و 20٪ من البروتين المسمى ب 14N نقاط انطلاق جيدة.
ملاحظة: يشير تراكم PRE كدالة للبروتين المضاف المسمى 14N-spin إلى تأثير محدد. PRE المرصود خاص بالعينة لأنه يعتمد على المسافة والسكان (كما تمت مناقشته أعلاه) ، وبالتالي ستكون هناك حاجة إلى نسب أعلى من البروتين المسمى ب 14N إذا كان التفاعل عابرا بشكل خاص17.
- انقل عينة الرنين المغناطيسي النووي (سواء داخل أو بين الجزيئيات) إلى أنبوب الرنين المغناطيسي النووي 5 مم مناسب للاستخدام في المغناطيسات عالية المجال باستخدام ماصة زجاجية طويلة الجذع (9 بوصات) أو ماصة ميكروماصة. تأكد من أن جميع عينات الرنين المغناطيسي النووي تحتوي على 5٪ -10٪ من D2O لتسهيل قفل الحقل.
ملاحظة: لا ينصح باستخدام أنابيب الرنين المغناطيسي النووي التي تستخدم سدادات البوليمر لتقليل حجم العينة الضروري لإجراء قياسات PRE بسبب الصعوبات المتعلقة بتلميع العينة الفعال.
4. إعداد مطياف الرنين المغناطيسي النووي وتجربة معلمات محددة
- توخ الحذر الشديد عند العمل حول مطياف الرنين المغناطيسي النووي فائق التوصيل وعالي المجال.
ملاحظة: تشمل المخاطر الإصابات الناجمة عن التسارع المفاجئ للأجسام المعدنية نحو المغناطيس ، والتداخل مع الأجهزة الطبية المزروعة ، والاختناق بسبب الإطلاق المفاجئ لغاز N 2 و He2 في حالة إخماد المغناطيس. تفترض الخطوات التالية أن القارئ قد خضع للتدريب المطلوب ، وأنه على دراية بهذه المخاطر وغيرها من المخاطر المحلية ، وحصل على موافقة من مدير المنشأة لتشغيل مطياف الرنين المغناطيسي النووي. عندما تكون في شك في خطوة أو تعليمات ، استشر مدير المنشأة أو المستخدم المتمرس لمنع الإصابة الشخصية المحتملة أو تلف مقياس الطيف. - تفترض الخطوات التالية مطياف NMR تجاريا يشغل نسخة حديثة من برنامج التحكم في الاحتياز. قم بتنزيل برنامج النبض وملفات المعلمات وضعها في الدلائل المناسبة.
ملاحظة: يتوفر برنامج نبض ومجموعة معلمات مناسبة للاستخدام مع مطياف Bruker و TopSpin (3.2 أو أحدث) عند الطلب من المؤلفين.- خطوة حاسمة: يفترض الإلمام بتثبيت برامج نبض الرنين المغناطيسي النووي غير الأصلية ؛ استشر مدير المنشأة أو مستخدم متمرس إذا لزم الأمر.
- ضع العينة في المغناطيس ، وقم بقفل إشارة 2H باستخدام أمر القفل ، وقم بضبط ومطابقة قناة 1H وفقا لبروتوكولات المنشأة (سيعتمد الإجراء الدقيق على ما إذا كان المسبار مزودا بوحدة ضبط ومطابقة عن بعد).
- اضبط الحشوات باستخدام الروتين الفرعي topshim لتحسين قمع إشارة المذيبات.
- قم بمعايرة النبضات 1H و 15N 90 ° باستخدام الطرق القياسية.
- قم بمعايرة نبضة 1H باستخدام برنامج popt (استخدم النبض أولا لتقدير طول النبضة).
- معايرة نبضة 15نيوتن مقابل عينة قياسية ؛ تأكد من معايرة هذه القيمة مؤخرا من خلال المناقشة مع مدير فني أو مستخدم متمرس.
- بدلا من ذلك ، قم بمعايرة نبضة 15N على العينة عن طريق تغيير إحدى نبضات 90 درجة لتجربة HMQC حتى يتم تحقيق إشارة فارغة.
- حدد التوهين الصحيح للنبضات المشكلة باستخدام الروتين الفرعي لأداة الشكل (stdisp).
- افتح ملف شكل النبض المناسب بالنقر فوق رمز المجلد. تم العثور على النبضات المشكلة في قسم معلمات النبض في ACQUPARS.
- قم بتحميل ملف تعريف النبض وانقر فوق تحليل الشكل الموجي > دمج الشكل. أدخل النبضة الصلبة المعايرة 1H 90 ° ، وطول النبضة على الشكل المطلوب ، والدوران (90 درجة أو 180 درجة).
- احسب مستوى طاقة النبضة المشكلة عن طريق إضافة تغيير مستوى الطاقة إلى التوهين للنبضة المعايرة بزاوية 90 درجة.
- سجل 1 Hقياسي ، 15N HSQC (hsqcetfpf3gpsi) لتحسين عرض المسح وتردد الموجة الحاملة والتحقق من قمع المياه25.
- اضبط عرض الاجتياح وعدد زيادات الأبعاد غير المباشرة باستخدام أمري sw وtd أو مباشرة في شاشات الاتصال المناسبة. عادة ، لجمع PREs ، يتم اختيار العروض الطيفية بحيث لا يتم طي الطيف.
5. قم بإعداد تجربة 1H N-T2
- معايرة النبضات المشكلة كما هو موضح أعلاه (4.4.5-4.5.7). ملفات معلمات النبض المشكلة لتجربة PRE هي Eburp2.1000 (نبضة 90 درجة) و Reburp.1000 و Iburp2.1000. أدخل أطوال النبضات المعايرة في قسم معلمات النبض في علامة التبويب ACQUPARS .
- تقيس هذه التجربة 1H N-T2 باستخدام نهج نقطتي تأخيرالوقت 30.
- اضبط التأخيرات الزمنية عن طريق تحرير ملف vdlist ، يتم تعيين التأخير الأول (Ta) على 0.01 مللي ثانية.
- اختر التأخير الثاني ، (T b) باستخدام العلاقة مع الحد الأقصى المتوقع PRE (T b ~ 1.15 / (R 2 ، dia + Γ 2) حيث يمثل R 2 ، dia R 2 للعينة المغناطيسية13. بدون معرفة مسبقة بحجم مساهمة PRE في الاسترخاء الملحوظ ، فإن نقطة البداية الجيدة هي ضبط Tb على ~ 1x 1H T 2.
- ثم حدد قيمة مناسبة من خلال مقارنة الزيادات الأولى (المعالجة باستخدام الأمر efp) لأطياف T a و T b وضبط T b بحيث تضمحل الإشارة إلى ما بين 40٪ -50٪ من قيمتها الأولية.
ملاحظة: يعمل هذا النهج على تحسين الإشارة الطيفية إلى الضوضاء، وهو اعتبار ضروري للعينات التي لا يمكن تركيزها بدرجة عالية (< 50 ميكرومتر). تعتمد القيم المناسبة ل Tb على العينة ولكنها تتراوح عادة من 8 إلى 40 مللي ثانية لبروتين متوسط الحجم.
- حدد عدد النقاط المعقدة المراد تسجيلها وعدد عمليات المسح للحصول على متوسط إشارة كاف. وبما أن المشردين داخليا لديهم 15 T2 أطول من البروتينات المطوية ذات الحجم المماثل، يمكن استخدام أوقات اكتساب أطول في البعد غير المباشر.
ملاحظة: تعتمد هذه القيمة على الخصائص المحددة للبروتين ولكن يمكن تقديرها تقريبا من 15 N T2 وتحسينها من خلال مراقبة اضمحلال الإشارة في FID. بالنسبة للبعد المباشر، تكفي 1024* نقطة معقدة (عرض مسح 13 جزء في المليون، ووقت اكتساب 112.6 مللي ثانية) لمعظم العينات. - استخدم الأمر expt لحساب وقت التجربة ثم ابدأ التجربة باستخدام الأمر zg.
6. قم بعمل عينة مغناطيسية عن طريق تقليل ملصق الدوران بحمض الأسكوربيك
- قم بإذابة أسكوربات الصوديوم في المخزن المؤقت للرنين المغناطيسي النووي واضبط الأس الهيدروجيني لمطابقة المخزن المؤقت الأصلي للرنين المغناطيسي النووي.
- احسب تركيز مخزون أسكوربات الصوديوم بحيث يمكن إضافة فائض 10x المولي من الأسكوربات على تركيز ملصق الدوران بأقل تغيير في حجم العينة. على سبيل المثال ، بالنسبة لعينة بروتين 100 ميكرومتر ، يكون مخزون 100 مللي متر من الأسكوربات مناسبا. سيتطلب تقليل ملصق الدوران إضافة 5.5 ميكرولتر من محلول مخزون حمض الأسكوربيك ، وهو ما يمثل 1٪ فقط من إجمالي حجم العينة.
- أضف الكمية المطلوبة من حمض الأسكوربيك إلى أنبوب الرنين المغناطيسي النووي عن طريق وضع قطرة أسفل حافة الأنبوب ، وقم بتغطية الأنبوب ، واقلب الأنبوب بعناية للخلط ، ثم قم بتدويره عند 200-400 × جم لمدة 10-20 ثانية في جهاز طرد مركزي يدوي لتسوية العينة في قاع الأنبوب.
- لف أنبوب الرنين المغناطيسي النووي بورق القصدير للحماية من الضوء والسماح للتفاعل بالاستمرار لمدة 3 ساعات على الأقل.
- سجل 1HN-T 2 على العينة المغناطيسية باستخدام نفس المعلمات المستخدمة للعينة المغناطيسية.
- أعد معايرة النبضات. ومع ذلك ، لا ينبغي أن تتغير من القياسات المغناطيسية ؛ إذا كانت مختلفة اختلافا كبيرا (فرق > 0.5 ميكروس) ، ففكر في جودة العينة (على سبيل المثال ، التدهور ، هطول الأمطار).
- تأكد من أن جميع معلمات الاستحواذ ، بما في ذلك تأخيرات الاسترخاء المحددة (vdlist) ، وعدد عمليات الفحص الوهمية ، وعدد عمليات الفحص التي تم جمعها ، وعدد النقاط المعقدة التي تم جمعها ، ووقت الاستحواذ ، وعرض المسح ، وترددات الموجة الحاملة تظل كما هي بالنسبة للعينات المغناطيسية والبارامغناطيسية.
7. عملية الأطياف المغناطيسية والمغناطيسية
- انسخ البيانات إلى الكمبيوتر المحلي أو محطة العمل التي تم تثبيت NMRPipe و Sparky وتكوينها. أنشئ مجلدا باسم proc في دليل بيانات التجربة الذي يحتوي على ملف ser.
- انسخ البرامج النصية NMRPipe fid.com و p3d.com و nmrproc.com إلى proc (تتوفر البرامج النصية للمعالجة عند الطلب من المؤلفين).
- استخدم البرنامج النصي fid.com لتحويل تنسيق بيانات Bruker (ser) إلى تنسيق NMRPipe.
- استخدم البرنامج النصي p3D.com لتقسيم طائرات pseudo3D إلى أطياف فردية.
- استخدم البرنامج النصي nmrproc.com لقراءة إخراج البرنامج النصي fid.com ، وتطبيق قمع المذيبات ، ووظيفة النافذة ، وإلحاق الأصفار بالبيانات الأولية (صفر تعبئة) ، وتطبيق تصحيح الطور ، وتنفيذ تحويل فورييه ، وتقليم البيانات للعرض وكتابة البيانات المعالجة على القرص. سيقوم البرنامج النصي بإخراج ملف واحد لكل تأخير استرخاء مسجل (T a و Tb).
ملاحظة: كل من هذه البرامج النصية قابلة للتخصيص لتحسين المعالجة للتفاصيل المحددة لكل تجربة. يتم تضمين البرامج التعليمية ومجموعات البيانات النموذجية في توزيع NMRPipe المتاح من موقع NMRPipe32. يمكن استخدام NMRDraw للعرض الطيفي أثناء المعالجة (على سبيل المثال ، تحديد زوايا الطور المناسبة وما إلى ذلك). يمكن عرض الخيارات المتاحة لأوامر NMRPipe باستخدام الأمر nmrPipe -help.
8. نقل تعيينات الرنين واستخراج ارتفاعات الذروة
- قم بتغيير معلومات رأس الملف لكل ملف طيف (T a ، Tb لكل من العينات المغناطيسية والمغناطيسية) باستخدام الأمر sethdr [اسم الملف] -ndim 2.
- استخدم Sparky لاستخراج ارتفاعاتالذروة 33 باتباع الخطوات 8.3-8.5. حزم البرامج الأخرى ، بما في ذلك NMRPipe (NMRDraw) 32 و CCPN Analysis 34 و NMRViewJ35 مناسبة أيضا.
- اقرأ الملفات الطيفية في سباركي. في هذه الخطوة ، ستتألف مجموعة البيانات من طيف واحد لكل أطياف نقطة زمنية (T a ، Tb) ، لكل من العينات البارامغناطيسية والمغناطيسية ، المقاسة لكل موضع من علامة الدوران في البروتين.
- استخدم Sparky لاختيار القمم (الأمر: F8، ثم انقر واسحب) ونقل المهام باستخدام أداة قائمة ذروة النقل من قائمة ذروة مرجعية.
ملاحظة: تخصيصات الرنين للبروتين محل الاهتمام ضرورية للتفسير الخاص بالتسلسل ل PREs36 المرصودة.- اضبط الخطوط في كل من الأطياف المغناطيسية والمغناطيسية إلى نفس المستوى. تأكد من ضبط الخطوط بحيث لا تستبعد الأطياف التي تم جمعها بعد التأخير الزمني القمم عن قصد ولكنها مرتفعة بما يكفي بحيث لا تكون أطياف T a صاخبة بشكل مفرط.
- احفظ قوائم الذروة الجديدة لكل طيف وقم بتضمين شدة الذروة المقاسة ونسبة الإشارة إلى الضوضاء المحسوبة من Sparky (الأمر: lt لفتح قائمة الذروة ، انقر فوق الخيارات لتضمين أعمدة الكثافة و SNR ، الأمر: حفظ).
9. استخراج 1HN-T 2 معدلات لكل بقايا وحساب PRE
- قم باستيراد قوائم الذروة إلى برنامج جداول البيانات أو لغة برمجة مفضلة مثل Python.
ملاحظة: لكل موضع تسمية مغزلية على البروتين ، ستتألف مجموعة البيانات من أربع قوائم ذروة مع شدة الذروة المرتبطة بها ، واحدة لكل من T a و Tb لكل من التجارب البارامغناطيسية والمغناطيسية. - احسب 1HN R2 لكل من العينات المغناطيسية والمغناطيسية باستخدام المعادلة:
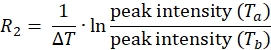
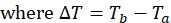
- استخدم المعادلة أعلاه لتحديد معدل الانحلال لكل بقايا للعينات البارامغناطيسية والمغناطيسية.
- حدد معدل 1HN-Γ 2 لكل بقايا باستخدام المعادلة:
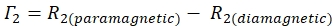
- استخدم نسبة الإشارة إلى الضوضاء المحسوبة من Sparky (SNR) لحساب عدم اليقين في ارتفاع الذروة لكل بقايا.
- انشر الخطأ باستخدام المعادلة:

- قطعة 1HN-Γ 2 كدالة لعدد المخلفات باستخدام مخطط مبعثر بما في ذلك الخطأ المحسوب في 9.6.
النتائج
تم تسجيل الجزيئات 1HN-Γ 2 PREs على جزء مرتبط ذاتيا ومضطرب جوهريا (بقايا 171-264) مشتق من مجال منخفض التعقيد لبروتين ربط الحمض النووي الريبي EWSR142 (الشكل 3). ومن المتوقع أن تتسع المخلفات المتتالية القريبة من نقطة التعلق بعلامة الدوران (مثل البقايا 178...
Discussion
تم تقديم طريقة لتوصيف التفاعلات العابرة الموجودة في مجموعات منخفضة بين البروتينات المضطربة جوهريا ومختلف شركاء الربط باستخدام PRE. في المثال الموضح، يرتبط البروتين ذاتيا، ومن ثم قد ينشأ PRE من مزيج من التفاعلات بين الجزيئات وداخلها. تمتد هذه الطريقة بسهولة إلى عينات غير متجانسة حيث يمكن تم?...
Disclosures
لقد قرأ جميع المؤلفين المخطوطة ووافقوا عليها. لم يتم الإعلان عن أي تضارب في المصالح.
Acknowledgements
نشكر الدكتورة جينفا ينغ وكريستين كانو على المناقشات المفيدة والمساعدة التقنية. DSL هو باحث في سانت بالدريك ويعترف بدعم مؤسسة سانت بالدريك (634706). تم دعم هذا العمل جزئيا من قبل مؤسسة ويلش (AQ-2001-20190330) إلى DSL ، وصندوق Max and Minnie Tomerlin Voelcker (منحة الباحث الشاب لمؤسسة Voelcker إلى DSL) ، وصناديق بدء UTHSA إلى DSL ، وزمالة Greehey Graduate في صحة الأطفال إلى CNJ. يعتمد هذا العمل على الأبحاث التي أجريت في المرافق الأساسية للبيولوجيا الهيكلية ، وهي جزء من مراكز الأبحاث المؤسسية في مركز العلوم الصحية بجامعة تكساس في سان أنطونيو بدعم من مكتب نائب الرئيس للأبحاث والموارد المشتركة لاكتشاف الأدوية والبيولوجيا الهيكلية في مركز ميس للسرطان (NIH P30 CA054174).
Materials
| Name | Company | Catalog Number | Comments |
| 0.45 µm and 0.22 µm syringe filters | Millipore Sigma | SLHVM33RS SLGVR33RS | Filter lysate before first purification step and before size exclusion chromatography. |
| 100 mm Petri Dish | Fisher | FB0875713 | Agar plates for bacterial transformation. |
| 14N Ammonium chloride | Sigma Aldrich | 576794 | Use of 15N in M9 medium will produce an NMR visible protein, 14N will produces an NMR invisible protein |
| 15N Ammonium chloride | Sigma Aldrich | 299251 | Use of 15N in M9 medium will produce an NMR visible protein, 14N will produces an NMR invisible protein |
| 3 L Fernbach baffled flask | Corning | 431523 | Bacterial expression culture |
| 3-Maleimido-Proxyl | Sigma Aldrich | 253375 | Nitroxide spin label |
| 50 mL conical centrifuge tubes | Thermo Fisher | 14-432-22 | Solution/protein storage |
| Amicon centrifugal filter | Millipore Sigma | UFC900308 | Protein concentration |
| Ampicillan | Sigma Aldrich | A5354 | Antibiotic for a selective marker, exact choice depends on the expression construct plasmid |
| Analytical balance | Oahus | 30061978 | Explorer Pro, for weighing reagents |
| Ascorbic acid | Sigma Aldrich | AX1775 | Reduces nitroxide spin label |
| Autoclave | Sterilize glassware and culture media | ||
| Calcium chloride | Sigma Aldrich | C4901 | M9 media component |
| Centrifuge bottles | Thermo Fisher | 010-1459 | Harvest E. coli cells after recombinant protein expression |
| Centrifuge, hand-crank | Thomas Scientific | 0241C68 | Boekel hand-driven, low-speed centrifuge with 15 mL buckets that can accommodate NMR tubes |
| Chelex 100 | Sigma Aldrich | C7901 | Remove contaminating paramagnetic compounds from buffer solutions |
| Computer workstation | Linux or Mac OS compatable with NMR data processing and analysis software packages such as NMRPipe and Sparky | ||
| Deuterium oxide | Sigma Aldrich | 151882 | Needed for NMR lock signal |
| Dextrose | Sigma Aldrich | D9434 | M9 media component |
| Dibasic Sodium Phosphate | Sigma Aldrich | S5136 | M9 media component |
| Ellman's reagent (5,5-dithio-bis-(2-nitrobenzoic acid) | Thermo Fisher | 22582 | Quantification of free cystiene residues |
| High speed centrifuge tubes | Thermo Fisher | 3114-0050 | Used to clear bacterial lysate. |
| High-field NMR instrument (600 - 800 MHz) | Bruker | Equiped with a multichannel cryogenic probe and temperature control | |
| IMAC column, HisTrap FF | Cytvia | 17528601 | Initial fractionation of crude bacterial lysate |
| Isopropyl B-D-thiogalactoside (IPTG) | Sigma Aldrich | I6758 | Induces protein expression for genes under control of lac operator |
| LB agar | Thermo Fisher | 22700025 | Items are used for transforming E. coli to express protein of interest, substitions for any of these items with like products is acceptable. |
| LB broth | Thermo Fisher | 12780052 | |
| Low-pressure chromatography system | Bio-Rad | 7318300 | BioRad BioLogic is used for low-pressure chomatograph such as running IMAC columns |
| Magnesium sulfate | Sigma Aldrich | M7506 | M9 media component |
| Medium pressure chromatography system | Bio-Rad | 7880007 | BioRad NGC equipped with a multi-wavelength detector, pH and conductivity monitors, and automatic fraction collector |
| MEM vitamin solution | Sigma Aldrich | M6895 | M9 media component |
| Microfluidizer | Avestin | EmulsiFlex-C3 | Provides rapid and efficient bacterial cell lysis |
| Micropipettes | Thermo Fisher | Calibrated set of micropippetters with properly fitting disposable tips (available from multiple manufacturers e.g. Eppendorf) | |
| Monobasic potassium phosphate | Sigma Aldrich | 1551139 | M9 media component |
| NMR pipettes | Sigma Aldrich | 255688 | To remove sample from NMR tube |
| NMR sample tube | NewEra | NE-SL5 | Suitable for high-field NMR spectrometers |
| Preparative Centrifuge | Beckman Coulter | Avanti J-HC | Harvest E. coli cells after recombinant protein expression |
| Round bottom polystyrene centrifuge tubes | Corning | 352057 | Clear bacterial lysate |
| Shaking incubator | Eppendorf | S44I200005 | Temperature controlled growth of E. coli starter and expression cultures |
| Sodium chloride | Sigma Aldrich | S5886 | M9 media component |
| Sonicating water bath and vacuum source | Thomas Scientific | Used to degas buffer solutions | |
| Sonicator | Thermo Fisher | FB505110 | Used for bacterial cell lysis or shearing bacterial DNA |
| Spectrophotometer | Implen | OD600 Diluphotometer | Monitor growth of E.coli protein expression cultures |
| Superdex 200 16/600 size exculsion colum | Cytvia | 28989333 | Final protein purification step |
| Topspin software, version 3.2 or later | Bruker | Operating software for the NMR instrument | |
| Transformation competent E. coli cells | Thermo Fisher | C600003 | One Shot BL21 Star (DE3) chemically competent E. coli, other strains may be compatable |
| Tris(2-carboxyethyl)phosphine (TCEP) | ThermoFisher | 20490 | Reducing agent compatable with some sulfhydryl-reactive conjugations |
| UV-Vis spectrophotometer | Implen | NP80 | Measure protein concentration. |
| Water bath, temperature controlled | ThermoFisher | FSGPD25 | For heat shock step of bacterial transformation |
| Yeast extract | Sigma Aldrich | Y1625 | For supplementing M9 media if required |
References
- Dyson, H. J., Wright, P. E. Intrinsically unstructured proteins and their functions. Nature Reviews: Molecular Cell Biology. 6 (3), 197-208 (2005).
- Korneta, I., Bujnicki, J. M. Intrinsic disorder in the human spliceosomal proteome. PLoS Computational Biology. 8 (8), 1002641 (2012).
- Frege, T., Uversky, V. N. Intrinsically disordered proteins in the nucleus of human cells. Biochemistry and Biophysics Reports. 1, 33-51 (2015).
- Liu, J., et al. Intrinsic disorder in transcription factors. Biochemistry. 45 (22), 6873-6888 (2006).
- El Hadidy, N., Uversky, V. N. Intrinsic disorder of the BAF complex: Roles in chromatin remodeling and disease development. International Journal of Molecular Sciences. 20 (21), (2019).
- Wright, P. E., Dyson, H. J. Intrinsically disordered proteins in cellular signalling and regulation. Nature Reviews: Molecular Cell Biology. 16 (1), 18-29 (2015).
- Brangwynne, C. P. Phase transitions and size scaling of membrane-less organelles. Journal of Cell Biology. 203 (6), 875-881 (2013).
- Shin, Y., Brangwynne, C. P. Liquid phase condensation in cell physiology and disease. Science. 357 (6357), (2017).
- Cavanagh, J. . Protein NMR spectroscopy : principles and practice. 1st edition. , (2018).
- Sekhar, A., Kay, L. E. NMR paves the way for atomic level descriptions of sparsely populated, transiently formed biomolecular conformers. Proceedings of the National Academy of Sciences of the United States of America. 110 (32), 12867-12874 (2013).
- Anthis, N. J., Clore, G. M. Visualizing transient dark states by NMR spectroscopy. Quarterly Reviews of Biophysics. 48 (1), 35-116 (2015).
- Alderson, T. R., Kay, L. E. NMR spectroscopy captures the essential role of dynamics in regulating biomolecular function. Cell. 184 (3), 577-595 (2021).
- Clore, G. M., Iwahara, J. Theory, practice, and applications of paramagnetic relaxation enhancement for the characterization of transient low-population states of biological macromolecules and their complexes. Chemical Reviews. 109 (9), 4108-4139 (2009).
- Wu, K. P., Baum, J. Detection of transient interchain interactions in the intrinsically disordered protein alpha-synuclein by NMR paramagnetic relaxation enhancement. Journal of the American Chemical Society. 132 (16), 5546-5547 (2010).
- Janowska, M. K., Wu, K. P., Baum, J. Unveiling transient protein-protein interactions that modulate inhibition of alpha-synuclein aggregation by beta-synuclein, a pre-synaptic protein that co-localizes with alpha-synuclein. Scientific Reports. 5, 15164 (2015).
- Murthy, A. C., et al. Molecular interactions underlying liquid-liquid phase separation of the FUS low-complexity domain. Nature Structural & Molecular Biology. 26 (7), 637-648 (2019).
- Fawzi, N. L., Doucleff, M., Suh, J. Y., Clore, G. M. Mechanistic details of a protein-protein association pathway revealed by paramagnetic relaxation enhancement titration measurements. Proceedings of the National Academy of Sciences of the United States of America. 107 (4), 1379-1384 (2010).
- Griffith, O. H., Waggoner, A. S. Nitroxide free radicals: spin labels for probing biomolecular structure. Accounts of Chemical Research. 2 (2), 17-24 (1969).
- Bertini, I., Luchinat, C., Parigi, G., Ravera, E. . NMR of Paramagnetic Macromolecules, Applications to Metallobiomolecules and Models. 2 edn. , (2016).
- Bloembergen, N., Purcell, E. M., Pound, R. V. Relaxation effects in nuclear magnetic resonance absorption. Physical Review. 73 (7), 679-712 (1948).
- Solomon, I. Relaxation processes in a system of two spins. Physical Review. 99 (2), 559 (1955).
- Clore, G. M. Practical aspects of paramagnetic relaxation enhancement in biological macromolecules. Methods in Enzymology. 564, 485-497 (2015).
- Klare, J. P. Site-directed spin labeling EPR spectroscopy in protein research. Biological Chemistry. 394 (10), 1281-1300 (2013).
- Clore, G. M., Tang, C., Iwahara, J. Elucidating transient macromolecular interactions using paramagnetic relaxation enhancement. Current Opinion in Structural Biology. 17 (5), 603-616 (2007).
- Melanson, M., Sood, A., Torok, F., Torok, M. Introduction to spin label electron paramagnetic resonance spectroscopy of proteins. Biochemistry and Molecular Biology Education. 41 (3), 156-162 (2013).
- Czogalla, A., Pieciul, A., Jezierski, A., Sikorski, A. F. Attaching a spin to a protein -- site-directed spin labeling in structural biology. Acta Biochimica Polonica. 54 (2), 235-244 (2007).
- Lindfors, H. E., de Koning, P. E., Drijfhout, J. W., Venezia, B., Ubbink, M. Mobility of TOAC spin-labelled peptides binding to the Src SH3 domain studied by paramagnetic NMR. Journal of Biomolecular NMR. 41 (3), 157-167 (2008).
- Fawzi, N. L., et al. A rigid disulfide-linked nitroxide side chain simplifies the quantitative analysis of PRE data. Journal of Biomolecular NMR. 51 (1-2), 105-114 (2011).
- Bleicken, S., et al. gem-Diethyl pyrroline nitroxide spin labels: Synthesis, EPR characterization, rotamer libraries and biocompatibility. ChemistryOpen. 8 (8), 1035 (2019).
- Iwahara, J., Tang, C., Clore, G. M. Practical aspects of 1H transverse paramagnetic relaxation enhancement measurements on macromolecules. Journal of Magnetic Resonance. 184, 185-195 (2007).
- Venditti, V., Fawzi, N. L. Probing the atomic structure of transient protein contacts by paramagnetic relaxation enhancement solution NMR. Methods in Molecular Biology. 1688, 243-255 (2018).
- Delaglio, F., et al. NMRPipe: a multidimensional spectral processing system based on UNIX pipes. Journal of Biomolecular NMR. 6 (3), 277-293 (1995).
- Lee, W., Tonelli, M., Markley, J. L. NMRFAM-SPARKY: enhanced software for biomolecular NMR spectroscopy. Bioinformatics. 31 (8), 1325-1327 (2015).
- Vranken, W. F., et al. The CCPN data model for NMR spectroscopy: development of a software pipeline. Proteins. 59 (4), 687-696 (2005).
- Johnson, B. A. Using NMRView to visualize and analyze the NMR spectra of macromolecules. Methods in Molecular Biology. 278, 313-352 (2004).
- Sjodt, M., Clubb, R. T. Nitroxide labeling of proteins and the determination of paramagnetic relaxation derived distance restraints for NMR studies. Bio-Protocol. 7 (7), (2017).
- Zhang, H., van Ingen, H. Isotope-labeling strategies for solution NMR studies of macromolecular assemblies. Current Opinion in Structural Biology. 38, 75-82 (2016).
- Rabdano, S. O., et al. Onset of disorder and protein aggregation due to oxidation-induced intermolecular disulfide bonds: case study of RRM2 domain from TDP-43. Scientific Reports. 7 (1), 11161 (2017).
- Burns, J. A., Butler, J. C., Moran, J., Whitesides, G. M. Selective reduction of disulfides by tris(2-carboxyethyl)phosphine. Journal of Organic Chemistry. 56 (8), 2648-2650 (1991).
- Ellman, G. L. Tissue sulfhydryl groups. Archives of Biochemistry and Biophysics. 82 (1), 70-77 (1959).
- Binbuga, B., Boroujerdi, A. F., Young, J. K. Structure in an extreme environment: NMR at high salt. Protein Science. 16 (8), 1783-1787 (2007).
- Schwartz, J. C., Cech, T. R., Parker, R. R. Biochemical properties and biological functions of FET proteins. Annual Review of Biochemistry. 84, 355-379 (2015).
- Nabuurs, S. M., de Kort, B. J., Westphal, A. H., van Mierlo, C. P. Non-native hydrophobic interactions detected in unfolded apoflavodoxin by paramagnetic relaxation enhancement. European Biophysics Journal. 39 (4), 689-698 (2010).
- Wiedemann, C., Kumar, A., Lang, A., Ohlenschlager, O. Cysteines and disulfide bonds as structure-forming units: Insights from different domains of life and the potential for characterization by NMR. Frontiers in Chemistry. 8, 280 (2020).
- Wommack, A. J., et al. NMR solution structure and condition-dependent oligomerization of the antimicrobial peptide human defensin 5. Biochemistry. 51 (48), 9624-9637 (2012).
- Taylor, A. M., et al. Detailed characterization of cysteine-less P-glycoprotein reveals subtle pharmacological differences in function from wild-type protein. British Journal of Pharmacology. 134 (8), 1609-1618 (2001).
- Hu, K., Doucleff, M., Clore, G. M. Using multiple quantum coherence to increase the 15N resolution in a three-dimensional TROSY HNCO experiment for accurate PRE and RDC measurements. Journal of Magnetic Resonance. 200 (2), 173-177 (2009).
- Anthis, N. J., Doucleff, M., Clore, G. M. Transient, sparsely populated compact states of apo and calcium-loaded calmodulin probed by paramagnetic relaxation enhancement: interplay of conformational selection and induced fit. Journal of the American Chemical Society. 133 (46), 18966-18974 (2011).
- Battiste, J. L., Wagner, G. Utilization of site-directed spin labeling and high-resolution heteronuclear nuclear magnetic resonance for global fold determination of large proteins with limited nuclear overhauser effect data. Biochemistry. 39 (18), 5355-5365 (2000).
- Donaldson, L. W., et al. Structural characterization of proteins with an attached ATCUN motif by paramagnetic relaxation enhancement NMR spectroscopy. Journal of the American Chemical Society. 123 (40), 9843-9847 (2001).
- Gaponenko, V., et al. Protein global fold determination using site-directed spin and isotope labeling. Protein Science. 9 (2), 302-309 (2000).
- Trindade, I. B., Invernici, M., Cantini, F., Louro, R. O., Piccioli, M. PRE-driven protein NMR structures: an alternative approach in highly paramagnetic systems. FEBS Journal. 288 (9), 3010-3023 (2021).
- Nitsche, C., Otting, G. Pseudocontact shifts in biomolecular NMR using paramagnetic metal tags. Progress in Nuclear Magnetic Resonance Spectroscopy. 98-99, 20-49 (2017).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved