需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
顺磁弛豫增强,用于检测和表征固有无序蛋白质的自缔合
摘要
提出了一种应用顺磁弛豫增强核磁共振波谱来检测固有无序蛋白质中弱和瞬态分子间和分子内相互作用的方案。
摘要
蛋白质中的内在无序蛋白质和内在无序区域构成了人类蛋白质组的重要组成部分。这些序列的高度灵活性使它们能够与不同的生物分子伙伴形成弱的、长距离的和短暂的相互作用。特异性但低亲和力的相互作用促进混杂结合,并使单个固有无序片段与多个靶位点相互作用。由于这些相互作用的瞬时性质,它们很难通过依赖蛋白质形成单一主要构象的结构生物学方法进行表征。顺磁弛豫增强核磁共振是识别和定义弱相互作用和瞬态相互作用的结构基础的有用工具。描述了使用顺磁弛豫增强来表征固有无序蛋白质与其蛋白质、核酸或其他生物分子伙伴之间形成的低人口遭遇复合物的详细协议。
引言
内在疾病 (ID) 描述了蛋白质 (IDP) 或蛋白质中的区域 (IDR),它们不会自发折叠成稳定的二级或三级结构,但具有生物活性。通常,IDP / IDR的功能是在生理条件下促进与生物分子的特定但可逆的相互作用1。因此,IDP 和 IDR 参与一系列细胞功能,包括多蛋白复合物的募集、组织和稳定,例如剪接体2 的组装和活性、DNA 损伤位点组件的募集和组织3、转录复合物4 或染色质重塑剂 BAF5 的募集的组织和稳定.此外,IDP存在于信号连接处,它们对不同结合伴侣的混杂性使它们能够通过细胞蛋白网络介导信息传递6。最近的工作还揭示了IDR区域通过液-液相分离过程自缔合形成生物分子冷凝物的倾向7。许多涉及ID的上述功能现在也被认为涉及冷凝水形成8的某些方面。尽管ID对于生物分子复合物组装,稳定,支架和信号转导很重要,但它们特定相互作用的原子细节很难识别,因为IDP和IDR通常不适合使用X射线晶体学或低温电子显微镜进行结构研究。
核磁共振(NMR)是研究ID的理想技术,因为它不依赖于刚性或均匀结构集合的存在,而是报告单个细胞核的直接局部环境。给定分子中原子核的共振频率或化学位移受到局部电子分布引起的弱磁场的影响,而局部电子分布又取决于键长、角度、其他原子核的接近程度、与结合伙伴的相互作用以及其他因素9.因此,每个原子核都充当独特的、特定于位点的结构探针,对其局部化学环境的变化敏感。尽管有这些优点,核磁共振是一种体型技术,观察到的化学位移是特定原子核采样的所有环境的平均值。已经开发了一系列核磁共振技术(本期中描述了其中许多技术)来恢复平均化学位移中包含的高能量、低人口生物分子构象的结构、动态和动力学信息10,11。虽然是短暂填充的,但这些状态的识别和量化对于确定功能机制的细节很重要12。例如,在IDP和IDR的情况下,构象系综可能偏向于优先采样构象,这些构象对于与生理结合伴侣形成相遇复合物是有效的。检测这些状态,以及鉴定残基特异性分子间和分子内相互作用和动力学,对于确定蛋白质功能和复合物形成的潜在结构机制非常重要。
描述了使用顺磁弛豫增强(PRE)NMR来研究对IDP / IDR介导的生物分子复合物的形成很重要的瞬态,人口稀少的状态的协议13。这种方法可用于研究瞬时蛋白质-蛋白质相互作用,例如促进α-突触核蛋白14,15 的淀粉样纤维组装或 FUS 16 的自缔合的相互作用,以及表征特定的蛋白质-蛋白质相互作用,例如信号蛋白之间的相互作用17。提出了一个自缔合IDP的例子,其中特定的分子间和分子内相互作用导致优先压缩状态以及驱动自缔合的位点特异性相互作用。
PRE起源于原子核与具有各向同性g张量的顺磁中心的磁偶极相互作用,通常以氮氧化物基团上的不成对电子的形式提供或作为顺磁性金属原子18(图1)。虽然具有各向异性g张量的原子也会产生PRE效应,但由于伪接触位移(PCS)或残余偶极耦合(RDC)产生的混杂效应,对这些系统的分析更加困难13,19。原子核和顺磁中心之间相互作用的强度取决于两者之间的>距离。这种相互作用导致核弛豫速率的增加,即使对于长程相互作用(~10-35 Å),也会导致可检测的谱线展宽,因为未成对电子的磁矩非常强20,21。如果满足以下两个条件,则可以使用 PRE 检测瞬态;(1)瞬态相互作用在核磁共振时间尺度上处于快速交换状态(观察到的化学位移是交换态的群体加权平均值);(2)瞬态下原子核到顺磁中心的距离比主态短11。横向 PRE 表示Γ2,出于实际目的,根据包含顺磁中心和抗磁性对照的样品之间 1H 横向弛豫速率的差异计算得出。为了深入探讨快速和慢速交换制度中的PRE理论和相关伪接触位移,读者可以参考Clore及其同事的综合评论13,22。在这里,只考虑1H N-Γ2处于快速交换状态的情况,其中由于PRE的r-6依赖性,观察到的弛豫速率与顺磁中心接近原子核的距离以及在该构象中花费的时间有关。因此,不涉及接近的瞬态构象会产生较小的PRE,而较近的相互作用,即使是短暂的,也会产生较大的PRE。
对于 IDP,PRE 用于测量和区分单个分子(分子内)和单独分子(分子间)之间发生的相互作用。通过将顺磁中心连接到NMR可见(例如,15N标记)或NMR不可见(例如,天然丰度14N)蛋白上,可以确定PRE的来源(分子间或分子内)(图2)。引入半胱氨酸残基的定点诱变是将顺磁中心(自旋标记)连接到蛋白质的便捷方法23。已经提出了几种类型的分子用作自旋标记,包括金属螯合(基于EDTA)和自由基(基于氮氧化物)24。已经描述了各种氮氧化物自旋标记物,并且具有不同的半胱氨酸反应性化学物质,例如硫代磺酸盐,马来酰亚胺和碘乙酰胺25,26(图1)。标签或接头的固有灵活性对于某些分析可能是有问题的,在这些情况下,已经提出了不同的策略来限制标签的运动,例如通过添加庞大的化学基团或使用第二个接头将标签锚定到蛋白质上(两个位点附着)27,28.此外,市售标签可能含有非对映异构体蛋白,但通常这不会有助于观察到的PRE29。描述了通过马来酰亚胺化学连接到游离半胱氨酸上的3-Maleimido-PROXY的使用,因为它易于获得,具有成本效益,不可逆,并且还原剂三(2-羧乙基)膦(TCEP)可以在整个标记反应中维持在溶液中。由于3-Maleimido-PROXYL具有各向同性的g-张量,因此不会诱导PCS或RDC,并且相同的化学位移分配可用于顺磁性和抗磁性样品13。
1HN-T 2 使用两个时间点策略(T a,T b)进行测量,该策略先前已被证明与收集由 8 到 12 个时间点30 组成的完整进化序列一样准确。第一个时间点(T a)设置为尽可能接近零,第二个时间点的最佳长度取决于给定样品的最大预期PRE的大小,可以由下式估计:Tb ~ 1.15/(R 2,dia + Γ 2),其中R 2,dia表示抗磁性样品13的R 2。如果最大 PREs 的大小未知,将 T b 设置为 ~ 蛋白质 1 H T 2 的 1倍是一个很好的初始估计,并通过调整 T2 来进一步优化以改善信噪比。这种两点测量策略显著减少了测量PRE所需的实验时间,并允许有更多的时间进行更多的信号平均,特别是因为使用相对稀释的样品来最小化分子之间非特异性接触的影响。基于HSQC的脉冲序列用于测量1H N-T2,并在其他地方进行了详细描述30。为了提高灵敏度,前向和后向INEPT转移的硬脉冲可以用整形脉冲代替;或者,该序列容易地转换为基于TROSY的读出31。由于IDP通常具有更长的横向弛豫率,导致线宽(由于固有的紊乱)比类似大小的球状蛋白更窄,因此间接维度的长采集时间可用于提高光谱分辨率并减轻IDP固有的化学位移色散限制。
PRE是研究蛋白质-蛋白质和蛋白质-核酸相互作用的有用工具,特别是瞬时或人口稀少的相互作用。提供了用于制备适用于测量PRE的NMR样品的详细方案,包括蛋白质纯化,定点自旋标记,设置和校准脉冲程序,处理和解释NMR数据的步骤。自始至终都注意到了可能影响数据质量和实验结果的重要实验考虑因素,包括样品浓度、自旋标记的选择和顺磁性成分的去除。
研究方案
协议的一般要求:蛋白质纯化设施、紫外-可见光谱仪、高场核磁共振波谱仪和操作软件、后处理分析软件包括;NMRPipe 32,Sparky33(或CCPN分析34,或NMRViewJ35)。
1. 用于 PRE 测量的蛋白质的重组表达和纯化
- 为目标蛋白设计表达构建体,以便存在单个半胱氨酸残基。需要多个突变才能在感兴趣的蛋白质的不同位置引入游离半胱氨酸36。
- 使用已建立的方案表达和纯化目标蛋白质的天然丰度(14N)或 15N标记样品37。
注意: 大肠杆菌 表达系统为重组蛋白表达提供了经济高效且稳健的方法,因为 15N的同位素富集是生物分子异核NMR波谱的最低要求。典型的步骤是在最小培养基中表达、色谱纯化和去除亲和纯化标签。该协议假设已经建立了稳健的表达和纯化方案,可以产生足够质量的蛋白质用于核磁共振研究。- 在所有纯化步骤中,在缓冲液中维持 1 mM 还原剂(DTT 或 TCEP),以防止游离半胱氨酸的反应和 IDP 分子间二硫键的形成。
注意:某些系统可能更耐受且不易聚集,容易出现非还原条件,具体取决于蛋白质的特定特性以及选择用于纯化的温度、pH 和缓冲液系统38。 - 在继续之前去除用于纯化的亲和标签,因为它们可能以不可预测的方式与蛋白质非特异性相互作用,或者可能含有反应性半胱氨酸残基,这些残基可能无意中充当意外的附着位点。
- 制备无半胱氨酸突变的 15N 标记参比样品,与可溶性版本的自旋标记物混合,以评估溶剂 PRE 的贡献。
- 在所有纯化步骤中,在缓冲液中维持 1 mM 还原剂(DTT 或 TCEP),以防止游离半胱氨酸的反应和 IDP 分子间二硫键的形成。
2. 共轭 3-马来酰亚胺基-丙氧基氮氧化物自旋标记
- 将纯化的蛋白质储存或交换到含有50 mM Tris pH 7和1 mM TCEP的脱气缓冲液中;如果需要,缓冲液还可以含有高达8 M尿素以帮助蛋白质溶解。
或者,将蛋白质储备溶液快速稀释到至少 10 体积当量的脱气 50 mM Tris pH 7 和 1 mM TCEP 缓冲液中。确保添加自旋标记之前的蛋白质浓度至少为100μM。 - 将储备溶液中的 3-Maleimido 丙烯添加到目标蛋白质的 20 倍摩尔过量中。保护样品免受光照和氧气的影响,并在室温或4°C下孵育过夜;轻柔摇晃或章动可以提高贴标效率。
- 通过将3-Maleimido丙基粉末溶解在95%乙醇中来制备自旋标记的储备溶液。原液的等分试样可以在-80°C下储存不到6个月。
- 关键步骤:去除未反应的游离自旋标签以防止非特异性溶剂PRE。 通过凝胶过滤或(优选)蛋白质样品的广泛透析来实现这一点。此步骤还将蛋白质引入适合NMR的缓冲液中。
注意:还原剂应新鲜制备,并应考虑缓冲液组分之间的相容性;例如,TCEP在磷酸盐缓冲液中快速降解,应避免这种组合39。 - 用选择性二价和过渡金属的螯合树脂处理此步骤中使用的所有缓冲液,以去除顺磁性离子或自旋标记淬灭剂。如果蛋白质不能储存在核磁共振缓冲液中,请将要快速稀释的蛋白质浓缩到适合核磁共振的缓冲液中。
- 监控纺丝标签合并的效率。
- 使用Ellman试剂(5,5-二硫代双(2-硝基苯甲酸)定量溶液40中的游离巯基。
注:详细协议可从制造商处获得。出于这里的目的,重要的是确定自旋标记的掺入,将游离巯基的浓度与总蛋白质浓度进行比较。游离巯基的百分比是没有附着氮氧化物自旋标记的分子的百分比。 - 监测对应于标记的半胱氨酸残基的峰的强度,以判断自旋标记掺入目标蛋白质中。
注意:这是确定蛋白质自旋标记程度的快速有效方法。完全掺入自旋标记将导致峰从光谱中消失。由于IDP的分散特性较差,与突变半胱氨酸残基对应的峰可能并不总是容易识别,因此建议使用Ellman试剂(步骤2.6.1)。
- 使用Ellman试剂(5,5-二硫代双(2-硝基苯甲酸)定量溶液40中的游离巯基。
3. 制备用于测量分子内或分子间 PRE 的 NMR 样品
- 制备用于测量分子内 PRE 的样品
- 在适合NMR的缓冲液中制备浓度至少为100μM但不大于300μM的 15N同位素富集的自旋标记蛋白。总样品体积(包括D2O)为500 - 550 μL。
注意:常见的NMR缓冲液包括磷酸盐、乙酸盐、(双)碳酸酯和TRIS。好的缓冲器如MES,HEPES也可能是合适的。选择缓冲液时要小心,以确保与其他溶液组分没有交叉反应。 - 确保pH值为~7.2或更低,以尽量减少酰胺质子与水交换的影响。保持盐的浓度尽可能低(通常低于150 mM),尽管主要考虑因素是保持蛋白质稳定性。
注:在高盐条件下进行核磁共振实验的方法已在别处41中描述。
- 在适合NMR的缓冲液中制备浓度至少为100μM但不大于300μM的 15N同位素富集的自旋标记蛋白。总样品体积(包括D2O)为500 - 550 μL。
- 准备用于测量分子间 PRE 的样品
- 按照此步骤或步骤 3.1;它们不是同时执行的。在所选的NMR缓冲液中制备 14N天然丰度的自旋标记蛋白。
- 通过将 15N同位素富集的非自旋标记蛋白与1%-50% 14N天然丰度自旋标记蛋白混合来制备蛋白质样品,以使最终浓度与3.1.1中制备的样品相同。总样品体积(包括D2O)为500 - 550 μL。
- 根据经验优化所研究每种蛋白质的 15N 和 14N 蛋白的比例。 14个N-自旋标记蛋白的1%、5%和20%的比例是很好的起点。
注意:作为添加的 14个 N-自旋标记蛋白的函数的 PRE 的积累表明特定效果;观察到的PRE是样品特异性的,因为它取决于距离和群体(如上所述),因此如果相互作用特别短暂,则需要 更高的14个N-自旋标记蛋白的比率17。
- 使用长柄(9")玻璃移液器或微量移液器将NMR(分子内或分子间)样品转移到适用于高场磁体的5 mm NMR管中。确保所有 NMR 样品包含 5%-10% 的 D2O,以便于磁场锁定。
注意:由于与有效样品垫片相关的困难,不建议使用聚合物塞来减少必要样品体积的NMR管进行PRE测量。
4. 设置核磁共振波谱仪并实验特定参数
- 在超导高场核磁共振波谱仪周围工作时要格外小心。
注意:危险包括由于金属物体突然向磁铁加速而导致的伤害、对植入式医疗设备的干扰,以及在磁铁淬火时突然释放 N 2 和He 2 气体而导致的窒息。以下步骤假设读者已经接受了所需的培训,意识到这些和其他当地危害,并已获得设施经理的批准来操作核磁共振波谱仪。当对步骤或说明有疑问时,请咨询设施经理或有经验的用户,以防止潜在的人身伤害或光谱仪损坏。 - 以下步骤假设商用 NMR 波谱仪运行现代版本的采集控制软件。下载脉冲程序和参数文件,并将它们放在适当的目录中。
注意:适合与布鲁克光谱仪和 TopSpin(3.2 或更高版本)一起使用的脉冲程序和参数集可向作者索取。- 关键步骤:假设熟悉安装非天然核磁共振脉冲程序;如有必要,请咨询设施经理或有经验的用户。
- 将样品放入磁体中,使用 Lock 命令锁定 2H 信号,根据设施协议调谐和匹配 1H 通道(确切的程序将取决于探头是否配备远程调谐和匹配模块)。
- 使用顶垫子程序调整垫片,以优化溶剂信号抑制。
- 使用标准方法校准 1H 和 15N 90° 脉冲。
- 使用弹出程序校准 1H 脉冲(首先使用脉冲来估计脉冲长度)。
- 针对标准样品校准 15N 脉冲;通过与技术总监或有经验的用户讨论,确保最近已校准此值。
- 或者,通过改变HMQC实验的90°脉冲之一来校准样品上的 15N脉冲,直到获得零信号。
- 使用形状工具 (stdisp) 子程序确定成形脉冲的正确衰减。
- 通过单击文件夹图标打开相应的脉冲形状文件。形状的脉冲可在 ACQUPARS的脉冲参数部分找到。
- 加载脉冲定义文件,然后单击分析 波形>积分形状。输入校准的 1H 90° 硬脉冲、所需的形状脉冲长度和旋转(90° 或 180°)。
- 通过将功率电平的变化与校准的 90° 脉冲的衰减相加来计算成形脉冲的功率电平。
- 记录标准 1H, 15N HSQC (hsqcetfpf3gpsi) 以优化扫描宽度、载波频率并检查抑水25.
- 使用 sw 和 td 命令或直接在相应的对话框中调整扫描宽度和间接尺寸增量的数量。通常,为了收集PRE,选择光谱宽度以使光谱不折叠。
5. 设置 1HN-T 2 实验
- 如上所述校准成形脉冲(4.4.5-4.5.7)。PRE实验的成形脉冲参数文件为Eburp2.1000(90°脉冲),Reburp.1000和Iburp2.1000。在 ACQUPARS 选项卡的脉冲参数部分中输入校准的脉冲长度。
- 本实验使用两个延时点方法30测量1 HN-T 2。
- 通过编辑 vdlist 文件设置时间延迟,第一个延迟 (Ta) 设置为 0.01 毫秒。
- 选择第二个延迟,(T b)使用与预期最大PRE(Tb ~ 1.15/(R 2,dia + Γ 2)的关系,其中R 2,dia表示抗磁性样品13的R 2。在没有事先了解PRE对观察到的弛豫的贡献大小的情况下,一个好的起点是将T b设置为~1x 1H T2。
- 然后通过比较 T a 和 T b 频谱的第一个增量(使用 efp 命令处理)并调整 T b 使信号衰减到其初始值的 40%-50% 之间来确定合适的值。
注意:这种方法优化了光谱信噪比,这是不能高度浓缩(<50μM)的样品的必要考虑因素。 Tb 的合适值取决于样品,但对于平均大小的蛋白质,通常范围为 8 - 40 ms。
- 确定要记录的复杂点的数量和扫描次数,以获得足够的信号平均。由于IDP比同等大小的折叠蛋白具有更长 的15N T2 ,因此可以使用更长的间接维度采集时间。
注意:该值取决于蛋白质的特定特性,但可以从 15N T2 中粗略估计,并通过监测FID中的信号衰减进行优化。对于直接尺寸,1024*复点(13 ppm扫描宽度,112.6 ms采集时间)对于大多数样品来说就足够了。 - 使用命令 expt 计算实验时间,然后使用命令 zg 开始实验。
6. 用抗坏血酸还原自旋标记制作抗磁性样品
- 将抗坏血酸钠溶解在NMR缓冲液中,并调节pH值以匹配原始NMR缓冲液。
- 计算抗坏血酸钠原液的浓度,以便以最小的样品体积变化添加超过自旋标记浓度的抗坏血酸摩尔数的10倍。例如,对于 100 μM 蛋白质样品,100 mM 抗坏血酸原液是合适的。减少自旋标记需要添加 5.5 μL 抗坏血酸储备溶液,仅占样品总量的 1%。
- 通过在管边缘下方放置液滴,将所需量的抗坏血酸添加到NMR管中,盖上管盖,小心地倒置管以混合,然后在手摇离心机中以200-400× g 旋转10-20秒,以将样品沉降在管底部。
- 用箔纸包裹NMR管以防止光线照射,并使反应进行至少3小时。
- 使用与顺磁性样品相同的参数在抗磁性样品上记录 1HN-T 2。
- 重新校准脉冲。但是,它们不应该从顺磁测量中改变;如果它们明显不同(> 0.5 μs 差异),请考虑样品质量(例如,降解、沉淀)。
- 确保抗磁性和顺磁性样品的所有采集参数(包括指定的松弛延迟 (vdlist)、虚拟扫描次数、收集的扫描次数、收集的复杂点数、采集时间、扫描宽度和载波频率保持不变。
7. 处理顺磁和抗磁光谱
- 将数据复制到安装和配置了NMRPipe和Sparky的本地计算机或工作站。在包含 ser 文件的试验数据目录中创建一个名为 proc 的文件夹。
- 将 NMRPipe 脚本 fid.com、 p3d.com 和 nmrproc.com 复制到 proc 中(可应作者要求提供处理脚本)。
- 使用 fid.com 脚本将布鲁克数据格式 (ser) 转换为 NMRPipe 格式。
- 使用 p3D.com 脚本将伪 3D 平面拆分为单个光谱。
- 使用 nmrproc.com 脚本读取 fid.com 脚本的输出、应用溶剂抑制、窗口函数、将零附加到原始数据(零填充)、应用相位校正、执行傅里叶变换、修剪数据以显示并将处理后的数据写入磁盘。该脚本将为记录的每个松弛延迟(T a 和 Tb)输出一个文件。
注意:这些脚本中的每一个都是可自定义的,以优化每个实验的特定细节的处理。教程和示例数据集包含在NMRPipe分布中,可从NMRPipe网站32获得。NMRDraw可用于加工过程中的光谱观察(例如,设置适当的相位角等)。NMRPipe 命令的可用选项可以使用命令 nmrPipe -help 进行查看。
8. 转移共振分配并提取峰高
- 使用命令 sethdr [文件名] -ndim 2 更改每个频谱文件的文件头信息(顺磁性和抗磁性样本为 Ta、Tb)。
- 按照步骤8.3-8.5使用Sparky提取峰高33。其他软件包,包括NMRPipe (NMRDraw)32,CCPN Analysis34和NMRViewJ 35也是合适的。
- 将光谱文件读取到Sparky中。在此步骤中,数据集将包括每个时间点光谱(Ta,T b)的一个光谱,用于顺磁性和抗磁性样品,针对蛋白质中自旋标记的每个位置进行测量。
- 使用 Sparky 选取峰(命令:F8,然后单击并拖动)并使用参考峰表中的转移峰列表工具转移分配。
注意:目标蛋白质的共振分配对于观察到的PREs36的序列特异性解释是必要的。- 将顺磁和抗磁光谱中的等值线设置为同一水平。确保设置轮廓,以便在时间延迟后收集的光谱不会故意排除峰值,而是足够高,以使 Ta 光谱不会过度嘈杂。
- 保存每个频谱的新峰值列表,并包括测量的峰值强度和Sparky计算的信噪比(命令:lt打开峰值列表,单击选项以包括强度和SNR列,命令:保存)。
9.提取每个残基的1HN-T 2速率并计算PRE
- 将峰值列表导入电子表格软件或首选编程语言(如 Python)。
注意:对于蛋白质上的每个自旋标记位置,数据集将包含四个具有相关峰强度的峰列表,顺磁性和抗磁性实验的T a和Tb各一个。 - 使用以下公式计算顺磁性和抗磁性样品的 1HN R2 :
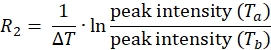
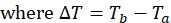
- 使用上述公式确定顺磁性和抗磁性样品中每个残基的弛豫率。
- 使用以下公式确定每个残基的 1HN-Γ 2 速率:
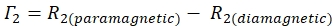
- 使用Sparky计算的信噪比(SNR)计算每个残基的峰高的不确定性。
- 使用以下公式传播误差:

- 使用散点图绘制 1HN-Γ 2 作为残差数的函数,包括 9.6 中计算的误差。
结果
分子内 1H N-Γ2 PRE 记录在源自 RNA 结合蛋白 EWSR142 的低复杂性结构域的自缔合、固有无序片段(残基 171-264)上(图 3)。与自旋标记附着点非常接近的残基(例如,图3中的残基178或260)预计将显着展宽,并且在光谱中无法检测到。从附着点依次间隔的残基显示出增强Γ 2在空间上接近自旋?...
讨论
已经提出了一种使用PRE表征固有无序蛋白质和各种结合伴侣之间在低群体中存在的瞬时相互作用的方法。在所示的示例中,蛋白质是自缔合的,因此PRE可能来自分子间和分子内相互作用的组合。该方法很容易扩展到异质样品,其中可以表征两种不同蛋白质之间的相互作用。通过将自旋标记放置在蛋白质内的不同位置,可以获得有关蛋白质不同区域如何相互作用的补充信息。此外,通过在NMR活性(<...
披露声明
所有作者均已阅读并批准该稿件。不声明任何利益冲突。
致谢
我们感谢应金发博士和克里斯汀·卡诺博士的有益讨论和技术援助。DSL是圣鲍德里克学者,并感谢圣鲍德里克基金会(634706)的支持。这项工作得到了韦尔奇基金会(AQ-2001-20190330)对DSL的部分支持,Max and Minnie Tomerlin Voelcker基金(Voelcker基金会青年研究员资助DSL),UTHSA启动基金DSL,以及Greehey儿童健康研究生奖学金CNJ。这项工作基于在结构生物学核心设施中进行的研究,这是德克萨斯大学圣安东尼奥健康科学中心机构研究核心的一部分,由研究副总统办公室和梅斯癌症中心药物发现和结构生物学共享资源(NIH P30 CA054174)支持。
材料
| Name | Company | Catalog Number | Comments |
| 0.45 µm and 0.22 µm syringe filters | Millipore Sigma | SLHVM33RS SLGVR33RS | Filter lysate before first purification step and before size exclusion chromatography. |
| 100 mm Petri Dish | Fisher | FB0875713 | Agar plates for bacterial transformation. |
| 14N Ammonium chloride | Sigma Aldrich | 576794 | Use of 15N in M9 medium will produce an NMR visible protein, 14N will produces an NMR invisible protein |
| 15N Ammonium chloride | Sigma Aldrich | 299251 | Use of 15N in M9 medium will produce an NMR visible protein, 14N will produces an NMR invisible protein |
| 3 L Fernbach baffled flask | Corning | 431523 | Bacterial expression culture |
| 3-Maleimido-Proxyl | Sigma Aldrich | 253375 | Nitroxide spin label |
| 50 mL conical centrifuge tubes | Thermo Fisher | 14-432-22 | Solution/protein storage |
| Amicon centrifugal filter | Millipore Sigma | UFC900308 | Protein concentration |
| Ampicillan | Sigma Aldrich | A5354 | Antibiotic for a selective marker, exact choice depends on the expression construct plasmid |
| Analytical balance | Oahus | 30061978 | Explorer Pro, for weighing reagents |
| Ascorbic acid | Sigma Aldrich | AX1775 | Reduces nitroxide spin label |
| Autoclave | Sterilize glassware and culture media | ||
| Calcium chloride | Sigma Aldrich | C4901 | M9 media component |
| Centrifuge bottles | Thermo Fisher | 010-1459 | Harvest E. coli cells after recombinant protein expression |
| Centrifuge, hand-crank | Thomas Scientific | 0241C68 | Boekel hand-driven, low-speed centrifuge with 15 mL buckets that can accommodate NMR tubes |
| Chelex 100 | Sigma Aldrich | C7901 | Remove contaminating paramagnetic compounds from buffer solutions |
| Computer workstation | Linux or Mac OS compatable with NMR data processing and analysis software packages such as NMRPipe and Sparky | ||
| Deuterium oxide | Sigma Aldrich | 151882 | Needed for NMR lock signal |
| Dextrose | Sigma Aldrich | D9434 | M9 media component |
| Dibasic Sodium Phosphate | Sigma Aldrich | S5136 | M9 media component |
| Ellman's reagent (5,5-dithio-bis-(2-nitrobenzoic acid) | Thermo Fisher | 22582 | Quantification of free cystiene residues |
| High speed centrifuge tubes | Thermo Fisher | 3114-0050 | Used to clear bacterial lysate. |
| High-field NMR instrument (600 - 800 MHz) | Bruker | Equiped with a multichannel cryogenic probe and temperature control | |
| IMAC column, HisTrap FF | Cytvia | 17528601 | Initial fractionation of crude bacterial lysate |
| Isopropyl B-D-thiogalactoside (IPTG) | Sigma Aldrich | I6758 | Induces protein expression for genes under control of lac operator |
| LB agar | Thermo Fisher | 22700025 | Items are used for transforming E. coli to express protein of interest, substitions for any of these items with like products is acceptable. |
| LB broth | Thermo Fisher | 12780052 | |
| Low-pressure chromatography system | Bio-Rad | 7318300 | BioRad BioLogic is used for low-pressure chomatograph such as running IMAC columns |
| Magnesium sulfate | Sigma Aldrich | M7506 | M9 media component |
| Medium pressure chromatography system | Bio-Rad | 7880007 | BioRad NGC equipped with a multi-wavelength detector, pH and conductivity monitors, and automatic fraction collector |
| MEM vitamin solution | Sigma Aldrich | M6895 | M9 media component |
| Microfluidizer | Avestin | EmulsiFlex-C3 | Provides rapid and efficient bacterial cell lysis |
| Micropipettes | Thermo Fisher | Calibrated set of micropippetters with properly fitting disposable tips (available from multiple manufacturers e.g. Eppendorf) | |
| Monobasic potassium phosphate | Sigma Aldrich | 1551139 | M9 media component |
| NMR pipettes | Sigma Aldrich | 255688 | To remove sample from NMR tube |
| NMR sample tube | NewEra | NE-SL5 | Suitable for high-field NMR spectrometers |
| Preparative Centrifuge | Beckman Coulter | Avanti J-HC | Harvest E. coli cells after recombinant protein expression |
| Round bottom polystyrene centrifuge tubes | Corning | 352057 | Clear bacterial lysate |
| Shaking incubator | Eppendorf | S44I200005 | Temperature controlled growth of E. coli starter and expression cultures |
| Sodium chloride | Sigma Aldrich | S5886 | M9 media component |
| Sonicating water bath and vacuum source | Thomas Scientific | Used to degas buffer solutions | |
| Sonicator | Thermo Fisher | FB505110 | Used for bacterial cell lysis or shearing bacterial DNA |
| Spectrophotometer | Implen | OD600 Diluphotometer | Monitor growth of E.coli protein expression cultures |
| Superdex 200 16/600 size exculsion colum | Cytvia | 28989333 | Final protein purification step |
| Topspin software, version 3.2 or later | Bruker | Operating software for the NMR instrument | |
| Transformation competent E. coli cells | Thermo Fisher | C600003 | One Shot BL21 Star (DE3) chemically competent E. coli, other strains may be compatable |
| Tris(2-carboxyethyl)phosphine (TCEP) | ThermoFisher | 20490 | Reducing agent compatable with some sulfhydryl-reactive conjugations |
| UV-Vis spectrophotometer | Implen | NP80 | Measure protein concentration. |
| Water bath, temperature controlled | ThermoFisher | FSGPD25 | For heat shock step of bacterial transformation |
| Yeast extract | Sigma Aldrich | Y1625 | For supplementing M9 media if required |
参考文献
- Dyson, H. J., Wright, P. E. Intrinsically unstructured proteins and their functions. Nature Reviews: Molecular Cell Biology. 6 (3), 197-208 (2005).
- Korneta, I., Bujnicki, J. M. Intrinsic disorder in the human spliceosomal proteome. PLoS Computational Biology. 8 (8), 1002641 (2012).
- Frege, T., Uversky, V. N. Intrinsically disordered proteins in the nucleus of human cells. Biochemistry and Biophysics Reports. 1, 33-51 (2015).
- Liu, J., et al. Intrinsic disorder in transcription factors. Biochemistry. 45 (22), 6873-6888 (2006).
- El Hadidy, N., Uversky, V. N. Intrinsic disorder of the BAF complex: Roles in chromatin remodeling and disease development. International Journal of Molecular Sciences. 20 (21), (2019).
- Wright, P. E., Dyson, H. J. Intrinsically disordered proteins in cellular signalling and regulation. Nature Reviews: Molecular Cell Biology. 16 (1), 18-29 (2015).
- Brangwynne, C. P. Phase transitions and size scaling of membrane-less organelles. Journal of Cell Biology. 203 (6), 875-881 (2013).
- Shin, Y., Brangwynne, C. P. Liquid phase condensation in cell physiology and disease. Science. 357 (6357), (2017).
- Cavanagh, J. . Protein NMR spectroscopy : principles and practice. 1st edition. , (2018).
- Sekhar, A., Kay, L. E. NMR paves the way for atomic level descriptions of sparsely populated, transiently formed biomolecular conformers. Proceedings of the National Academy of Sciences of the United States of America. 110 (32), 12867-12874 (2013).
- Anthis, N. J., Clore, G. M. Visualizing transient dark states by NMR spectroscopy. Quarterly Reviews of Biophysics. 48 (1), 35-116 (2015).
- Alderson, T. R., Kay, L. E. NMR spectroscopy captures the essential role of dynamics in regulating biomolecular function. Cell. 184 (3), 577-595 (2021).
- Clore, G. M., Iwahara, J. Theory, practice, and applications of paramagnetic relaxation enhancement for the characterization of transient low-population states of biological macromolecules and their complexes. Chemical Reviews. 109 (9), 4108-4139 (2009).
- Wu, K. P., Baum, J. Detection of transient interchain interactions in the intrinsically disordered protein alpha-synuclein by NMR paramagnetic relaxation enhancement. Journal of the American Chemical Society. 132 (16), 5546-5547 (2010).
- Janowska, M. K., Wu, K. P., Baum, J. Unveiling transient protein-protein interactions that modulate inhibition of alpha-synuclein aggregation by beta-synuclein, a pre-synaptic protein that co-localizes with alpha-synuclein. Scientific Reports. 5, 15164 (2015).
- Murthy, A. C., et al. Molecular interactions underlying liquid-liquid phase separation of the FUS low-complexity domain. Nature Structural & Molecular Biology. 26 (7), 637-648 (2019).
- Fawzi, N. L., Doucleff, M., Suh, J. Y., Clore, G. M. Mechanistic details of a protein-protein association pathway revealed by paramagnetic relaxation enhancement titration measurements. Proceedings of the National Academy of Sciences of the United States of America. 107 (4), 1379-1384 (2010).
- Griffith, O. H., Waggoner, A. S. Nitroxide free radicals: spin labels for probing biomolecular structure. Accounts of Chemical Research. 2 (2), 17-24 (1969).
- Bertini, I., Luchinat, C., Parigi, G., Ravera, E. . NMR of Paramagnetic Macromolecules, Applications to Metallobiomolecules and Models. 2 edn. , (2016).
- Bloembergen, N., Purcell, E. M., Pound, R. V. Relaxation effects in nuclear magnetic resonance absorption. Physical Review. 73 (7), 679-712 (1948).
- Solomon, I. Relaxation processes in a system of two spins. Physical Review. 99 (2), 559 (1955).
- Clore, G. M. Practical aspects of paramagnetic relaxation enhancement in biological macromolecules. Methods in Enzymology. 564, 485-497 (2015).
- Klare, J. P. Site-directed spin labeling EPR spectroscopy in protein research. Biological Chemistry. 394 (10), 1281-1300 (2013).
- Clore, G. M., Tang, C., Iwahara, J. Elucidating transient macromolecular interactions using paramagnetic relaxation enhancement. Current Opinion in Structural Biology. 17 (5), 603-616 (2007).
- Melanson, M., Sood, A., Torok, F., Torok, M. Introduction to spin label electron paramagnetic resonance spectroscopy of proteins. Biochemistry and Molecular Biology Education. 41 (3), 156-162 (2013).
- Czogalla, A., Pieciul, A., Jezierski, A., Sikorski, A. F. Attaching a spin to a protein -- site-directed spin labeling in structural biology. Acta Biochimica Polonica. 54 (2), 235-244 (2007).
- Lindfors, H. E., de Koning, P. E., Drijfhout, J. W., Venezia, B., Ubbink, M. Mobility of TOAC spin-labelled peptides binding to the Src SH3 domain studied by paramagnetic NMR. Journal of Biomolecular NMR. 41 (3), 157-167 (2008).
- Fawzi, N. L., et al. A rigid disulfide-linked nitroxide side chain simplifies the quantitative analysis of PRE data. Journal of Biomolecular NMR. 51 (1-2), 105-114 (2011).
- Bleicken, S., et al. gem-Diethyl pyrroline nitroxide spin labels: Synthesis, EPR characterization, rotamer libraries and biocompatibility. ChemistryOpen. 8 (8), 1035 (2019).
- Iwahara, J., Tang, C., Clore, G. M. Practical aspects of 1H transverse paramagnetic relaxation enhancement measurements on macromolecules. Journal of Magnetic Resonance. 184, 185-195 (2007).
- Venditti, V., Fawzi, N. L. Probing the atomic structure of transient protein contacts by paramagnetic relaxation enhancement solution NMR. Methods in Molecular Biology. 1688, 243-255 (2018).
- Delaglio, F., et al. NMRPipe: a multidimensional spectral processing system based on UNIX pipes. Journal of Biomolecular NMR. 6 (3), 277-293 (1995).
- Lee, W., Tonelli, M., Markley, J. L. NMRFAM-SPARKY: enhanced software for biomolecular NMR spectroscopy. Bioinformatics. 31 (8), 1325-1327 (2015).
- Vranken, W. F., et al. The CCPN data model for NMR spectroscopy: development of a software pipeline. Proteins. 59 (4), 687-696 (2005).
- Johnson, B. A. Using NMRView to visualize and analyze the NMR spectra of macromolecules. Methods in Molecular Biology. 278, 313-352 (2004).
- Sjodt, M., Clubb, R. T. Nitroxide labeling of proteins and the determination of paramagnetic relaxation derived distance restraints for NMR studies. Bio-Protocol. 7 (7), (2017).
- Zhang, H., van Ingen, H. Isotope-labeling strategies for solution NMR studies of macromolecular assemblies. Current Opinion in Structural Biology. 38, 75-82 (2016).
- Rabdano, S. O., et al. Onset of disorder and protein aggregation due to oxidation-induced intermolecular disulfide bonds: case study of RRM2 domain from TDP-43. Scientific Reports. 7 (1), 11161 (2017).
- Burns, J. A., Butler, J. C., Moran, J., Whitesides, G. M. Selective reduction of disulfides by tris(2-carboxyethyl)phosphine. Journal of Organic Chemistry. 56 (8), 2648-2650 (1991).
- Ellman, G. L. Tissue sulfhydryl groups. Archives of Biochemistry and Biophysics. 82 (1), 70-77 (1959).
- Binbuga, B., Boroujerdi, A. F., Young, J. K. Structure in an extreme environment: NMR at high salt. Protein Science. 16 (8), 1783-1787 (2007).
- Schwartz, J. C., Cech, T. R., Parker, R. R. Biochemical properties and biological functions of FET proteins. Annual Review of Biochemistry. 84, 355-379 (2015).
- Nabuurs, S. M., de Kort, B. J., Westphal, A. H., van Mierlo, C. P. Non-native hydrophobic interactions detected in unfolded apoflavodoxin by paramagnetic relaxation enhancement. European Biophysics Journal. 39 (4), 689-698 (2010).
- Wiedemann, C., Kumar, A., Lang, A., Ohlenschlager, O. Cysteines and disulfide bonds as structure-forming units: Insights from different domains of life and the potential for characterization by NMR. Frontiers in Chemistry. 8, 280 (2020).
- Wommack, A. J., et al. NMR solution structure and condition-dependent oligomerization of the antimicrobial peptide human defensin 5. Biochemistry. 51 (48), 9624-9637 (2012).
- Taylor, A. M., et al. Detailed characterization of cysteine-less P-glycoprotein reveals subtle pharmacological differences in function from wild-type protein. British Journal of Pharmacology. 134 (8), 1609-1618 (2001).
- Hu, K., Doucleff, M., Clore, G. M. Using multiple quantum coherence to increase the 15N resolution in a three-dimensional TROSY HNCO experiment for accurate PRE and RDC measurements. Journal of Magnetic Resonance. 200 (2), 173-177 (2009).
- Anthis, N. J., Doucleff, M., Clore, G. M. Transient, sparsely populated compact states of apo and calcium-loaded calmodulin probed by paramagnetic relaxation enhancement: interplay of conformational selection and induced fit. Journal of the American Chemical Society. 133 (46), 18966-18974 (2011).
- Battiste, J. L., Wagner, G. Utilization of site-directed spin labeling and high-resolution heteronuclear nuclear magnetic resonance for global fold determination of large proteins with limited nuclear overhauser effect data. Biochemistry. 39 (18), 5355-5365 (2000).
- Donaldson, L. W., et al. Structural characterization of proteins with an attached ATCUN motif by paramagnetic relaxation enhancement NMR spectroscopy. Journal of the American Chemical Society. 123 (40), 9843-9847 (2001).
- Gaponenko, V., et al. Protein global fold determination using site-directed spin and isotope labeling. Protein Science. 9 (2), 302-309 (2000).
- Trindade, I. B., Invernici, M., Cantini, F., Louro, R. O., Piccioli, M. PRE-driven protein NMR structures: an alternative approach in highly paramagnetic systems. FEBS Journal. 288 (9), 3010-3023 (2021).
- Nitsche, C., Otting, G. Pseudocontact shifts in biomolecular NMR using paramagnetic metal tags. Progress in Nuclear Magnetic Resonance Spectroscopy. 98-99, 20-49 (2017).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。