Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Paramagnetische Relaxationsverstärkung zur Detektion und Charakterisierung von Selbstassoziationen von intrinsisch ungeordneten Proteinen
In diesem Artikel
Zusammenfassung
Es wird ein Protokoll für die Anwendung der NMR-Spektroskopie zur Verstärkung der paramagnetischen Relaxation vorgestellt, um schwache und transiente inter- und intramolekulare Wechselwirkungen in intrinsisch ungeordneten Proteinen zu detektieren.
Zusammenfassung
Intrinsisch ungeordnete Proteine und intrinsisch ungeordnete Regionen innerhalb von Proteinen machen einen großen und funktionell bedeutsamen Teil des menschlichen Proteoms aus. Die hohe Flexibilität dieser Sequenzen ermöglicht es ihnen, schwache, langreichweitige und vorübergehende Wechselwirkungen mit verschiedenen biomolekularen Partnern einzugehen. Spezifische, aber wenig affine Interaktionen fördern die promiskuitive Bindung und ermöglichen es einem einzelnen, intrinsisch ungeordneten Segment, mit einer Vielzahl von Zielstellen zu interagieren. Aufgrund der vorübergehenden Natur dieser Wechselwirkungen kann es schwierig sein, sie mit strukturbiologischen Methoden zu charakterisieren, die auf Proteinen beruhen, um eine einzige, vorherrschende Konformation zu bilden. Die NMR zur Verbesserung der paramagnetischen Relaxation ist ein nützliches Werkzeug, um die strukturellen Grundlagen schwacher und transienter Wechselwirkungen zu identifizieren und zu definieren. Ein detailliertes Protokoll zur Verwendung der paramagnetischen Relaxationsverstärkung zur Charakterisierung der niedrig besiedelten Begegnungskomplexe, die sich zwischen intrinsisch ungeordneten Proteinen und ihren Proteinen, Nukleinsäuren oder anderen biomolekularen Partnern bilden, wird beschrieben.
Einleitung
Intrinsische Störung (ID) beschreibt Proteine (IDPs) oder Regionen innerhalb von Proteinen (IDRs), die sich nicht spontan zu stabilen Sekundär- oder Tertiärstrukturen falten, sondern biologisch aktiv sind. Im Allgemeinen besteht die Funktion von IDP/IDRs darin, spezifische, aber reversible Wechselwirkungen mit Biomolekülen unter physiologischen Bedingungen zu ermöglichen1. Somit sind IDPs und IDRs an einer Reihe von zellulären Funktionen beteiligt, einschließlich der Rekrutierung, Organisation und Stabilisierung von Multi-Protein-Komplexen, z. B. der Assemblierung und Aktivität des Spleißosoms2, der Rekrutierung und Organisation von Komponenten an den Stellen der DNA-Schädigung3, der Organisation und Stabilisierung der Rekrutierung von Transkriptionskomplexen4 oder des Chromatin-Remodelers BAF5. Darüber hinaus werden IDPs an Signalknoten gefunden, wo ihre Promiskuität für verschiedene Bindungspartner es ihnen ermöglicht, den Informationstransfer durch zelluläre Proteinnetzwerke zu vermitteln6. Neuere Arbeiten haben auch gezeigt, dass IDR-Regionen dazu neigen, sich selbst zu assoziieren und biomolekulare Kondensate durch den Prozess der Flüssig-Flüssig-Phasentrennung zu bilden7. Viele der oben erwähnten Funktionen, die ID betreffen, werden heute auch mit einem Aspekt der Kondensatbildung in Verbindung gebracht8. Trotz der Bedeutung von ID für die Assemblierung, Stabilisierung, Gerüstbildung und Signaltransduktion biomolekularer Komplexe sind die atomaren Details ihrer spezifischen Wechselwirkungen schwer zu identifizieren, da IDPs und IDRs in der Regel nicht für strukturelle Untersuchungen mit Röntgenkristallographie oder kryogener Elektronenmikroskopie geeignet sind.
Die Kernspinresonanz (NMR) ist eine ideale Technik zur Untersuchung der ID, da sie nicht vom Vorhandensein starrer oder homogener Strukturensembles abhängt, sondern über die unmittelbare lokale Umgebung einzelner Kerne berichtet. Die Resonanzfrequenz oder chemische Verschiebung eines Kerns in einem bestimmten Molekül wird durch schwache Magnetfelder beeinflusst, die durch die lokale elektronische Verteilung induziert werden, die wiederum von Bindungslängen, Winkeln, der Nähe anderer Kerne, Wechselwirkungen mit Bindungspartnern und anderen Faktoren abhängt9. So fungiert jeder Kern als einzigartige, ortsspezifische Struktursonde, die empfindlich auf Veränderungen in seiner lokalen chemischen Umgebung reagiert. Trotz dieser Vorteile ist die NMR eine Bulk-Technik, und die beobachtete chemische Verschiebung ist der Durchschnitt aller Umgebungen, die von einem bestimmten Kern beprobt wurden. Eine Reihe von NMR-Techniken, von denen viele in dieser Ausgabe beschrieben werden, wurden entwickelt, um strukturelle, dynamische und kinetische Informationen über hochenergetische, niedrig besiedelte biomolekulare Konformationen zu gewinnen, die in der gemittelten chemischen Verschiebung enthaltensind 10,11. Obwohl sie nur vorübergehend besiedelt sind, sind die Identifizierung und Quantifizierung dieser Zustände wichtig, um die Details der funktionellen Mechanismen zu bestimmen12. Zum Beispiel kann im Fall von IDPs und IDRs das Konformationsensemble verzerrt sein, um bevorzugt Konformationen zu probieren, die für die Bildung von Begegnungskomplexen mit physiologischen Bindungspartnern produktiv sind. Die Detektion dieser Zustände sowie die Identifizierung der restspezifischen inter- und intramolekularen Wechselwirkungen und Dynamiken sind wichtig, um die zugrundeliegenden strukturellen Mechanismen der Proteinfunktion und Komplexbildung zu bestimmen.
Es wird ein Protokoll zur Verwendung der paramagnetischen Relaxationsverstärkung (PRE) NMR zur Untersuchung transienter, niedrig besiedelter Zustände beschrieben, die für die Bildung von IDP/IDR-vermittelten biomolekularen Komplexen wichtig sind13. Dieser Ansatz ist nützlich für die Untersuchung der transienten Protein-Protein-Interaktionen, wie z. B. solche, die den Aufbau von Amyloidfibrillen aus α-Synuclein 14,15 oder die Selbstassoziation von FUS16 fördern, sowie für die Charakterisierung spezifischer Protein-Protein-Interaktionen, z. B. zwischen Signalproteinen17. Ein Beispiel für eine selbstassoziierende IDP wird vorgestellt, bei der spezifische inter- und intramolekulare Wechselwirkungen zu bevorzugt verdichteten Zuständen sowie zu ortsspezifischen Wechselwirkungen führen, die die Selbstassoziation vorantreiben.
Die PRE entsteht aus der magnetischen dipolaren Wechselwirkung eines Kerns mit einem paramagnetischen Zentrum mit einem isotropen g-Tensor, der üblicherweise in Form eines ungepaarten Elektrons auf einer Nitroxidgruppe oder als paramagnetisches Metallatom18 geliefert wird (Abbildung 1). Während Atome mit anisotropen g-Tensoren auch einen PRE-Effekt erzeugen, ist die Analyse dieser Systeme aufgrund von Störeffekten, die durch die Pseudo-Kontaktverschiebungen (PCS) oder die residuale dipolare Kopplung (RDC) verursacht werden, schwieriger13,19. Die Stärke der Wechselwirkung zwischen einem Kern und dem paramagnetischen Zentrum hängt vom Abstand <r-6> zwischen den beiden ab. Diese Wechselwirkung führt zu einem Anstieg der Kernrelaxationsraten, was zu einer nachweisbaren Linienverbreiterung auch für langreichweitige Wechselwirkungen (~10-35 Å) führt, da das magnetische Moment des ungepaarten Elektrons so stark ist20,21. Die Erkennung von transienten Zuständen mit dem PRE ist möglich, wenn die folgenden beiden Bedingungen erfüllt sind; (1) die transiente Wechselwirkung befindet sich in schnellem Austausch auf der NMR-Zeitskala (beobachtete chemische Verschiebung ist ein populationsgewichteter Durchschnitt der austauschenden Zustände); und (2) der Abstand zwischen Kernen und paramagnetischem Schwerpunkt ist im vorübergehend besiedelten Zustand kürzer als im Hauptzustand11. Die transversale PRE wird Γ2 bezeichnet und aus praktischen Gründen aus der Differenz der transversalenRelaxationsraten von 1 H zwischen einer Probe mit einem paramagnetischen Zentrum und einer diamagnetischen Steuerung berechnet. Für eine eingehende Behandlung der Theorie der PRE und der damit verbundenen Pseudokontaktverschiebungen in schnellen und langsamen Austauschregimen wird der Leser auf die umfassenden Übersichtsarbeiten von Clore und Mitarbeitern13,22 verwiesen. Hier wird nur die Situation betrachtet, in der sich 1HN-Γ 2 im schnellen Austauschregime befindet, wobei aufgrund der r-6-Abhängigkeit des PRE die beobachtete Relaxationsrate sowohl mit der Entfernung, in der sich das paramagnetische Zentrum dem Kern nähert, als auch mit der Zeit, die es in dieser Konformation verbringt, zusammenhängt. Daher erzeugen transiente Konformationen, die keine enge Annäherung beinhalten, eine kleine PRE, während nähere Wechselwirkungen, auch wenn sie nur von kurzer Dauer sind, eine größere PRE erzeugen.
Bei IDPs wird der PRE verwendet, um die Wechselwirkungen innerhalb eines einzelnen Moleküls (intramolekular) und zwischen einzelnen Molekülen (intermolekular) zu messen und zu differenzieren. Durch Anbringen eines paramagnetischen Zentrums an ein sichtbares (z. B. 15N-markiertes) oder NMR-unsichtbares (z. B. natürliches 14N) Protein kann die Quelle (inter- oder intramolekular) des PRE bestimmt werden (Abbildung 2). Die ortsgerichtete Mutagenese, die einen Cysteinrest einführt, ist ein bequemer Ansatz, um ein paramagnetisches Zentrum (Spin-Label) an ein Proteinanzuhängen 23. Es wurden verschiedene Arten von Molekülen für die Verwendung als Spin-Markierungen vorgeschlagen, darunter Metallchelat-Moleküle (EDTA-basiert) und freie Radikale (Nitroxid-basiert)24. Verschiedene Nitroxid-Spin-Markierungen wurden beschrieben und sind mit unterschiedlichen Cystein-reaktiven Chemikalien wie Methanethiosulfonat, Maleimid und Iodacetamid25,26 erhältlich (Abbildung 1). Die inhärente Flexibilität des Tags oder des Linkers kann für bestimmte Analysen problematisch sein, und in diesen Situationen wurden verschiedene Strategien vorgeschlagen, um die Bewegung des Tags zu begrenzen, z. B. durch Hinzufügen sperriger chemischer Gruppen oder die Verwendung eines zweiten Linkers, um den Tag am Protein zu verankern (Attachment an zwei Stellen)27, 28. S. Darüber hinaus können kommerziell erhältliche Tags diastereomere Proteine enthalten, was jedoch im Allgemeinen nicht zu der beobachteten PRE29 beiträgt. Die Verwendung des 3-Maleimido-PROXYLS, das über Maleimid-Chemie an ein freies Cystein gebunden ist, wird beschrieben, da es leicht verfügbar, kostengünstig und nicht reversibel ist und das Reduktionsmittel Tris(2-carboxyethyl)phosphin (TCEP) während der gesamten Markierungsreaktion in der Lösung beibehalten werden kann. Da 3-Maleimido-PROXYL einen isotropen g-Tensor aufweist, werden keine PCS oder RDCs induziert, und die gleichen chemischen Verschiebungszuweisungen können sowohl für die paramagnetischen als auch für die diamagnetischen Proben13 verwendet werden.
Die 1HN-T 2 wird unter Verwendung einer Zwei-Zeitpunkt-Strategie (T a, Tb) gemessen, die sich zuvor als so genau erwiesen hat wie das Sammeln einer vollständigen Evolutionsreihe, die aus 8 bis 12 Zeitpunkten besteht30. Der erste Zeitpunkt (T a) wird so nahe wie möglich an Null gesetzt, und die optimale Länge des zweiten Zeitpunkts hängt von der Größe der größten erwarteten PRE für eine gegebene Probe ab und kann geschätzt werden von: Tb ~ 1,15/(R2,dia + Γ2), wobei R2,dia die R2 der diamagnetischen Probe13 darstellt. Wenn die Größe der größten PREs unbekannt ist, ist die Einstellung von T b auf ~ das Einfache des 1H T2 des Proteins eine gute erste Schätzung und wird durch Anpassung von T2 weiter optimiert, um das Signal-Rausch-Verhältnis zu verbessern. Diese Zwei-Punkt-Messstrategie reduziert die experimentelle Zeit, die für die Messung von PREs erforderlich ist, erheblich und ermöglicht mehr Zeit für eine bessere Signalmittelung, zumal relativ verdünnte Proben verwendet werden, um die Auswirkungen unspezifischer Kontakte zwischen Molekülen zu minimieren. Eine HSQC-basierte Pulssequenz wird zur Messung von 1H, N-T2 verwendet und wurde an anderer Stelleausführlich beschrieben 30. Um die Empfindlichkeit zu verbessern, können die harten Impulse der Vorwärts- und Rückwärts-INEPT-Übertragungen durch geformte Impulse ersetzt werden. alternativ kann die Sequenz ohne weiteres in eine TROSY-basierte Auslesung31 umgewandelt werden. Da IDPs typischerweise viel längere transversale Relaxationsraten haben, was zu schmaleren Linienbreiten führt (aufgrund der inhärenten Unordnung) als ähnlich große globuläre Proteine, können lange Aufnahmezeiten in der indirekten Dimension genutzt werden, um die spektrale Auflösung zu verbessern und die für IDPs inhärente Begrenzung der chemischen Verschiebungsdispersion zu mildern.
PRE ist ein nützliches Werkzeug für die Untersuchung von Protein-Protein- und Protein-Nukleinsäure-Interaktionen, insbesondere von Interaktionen, die vorübergehend oder schwach besiedelt sind. Ein detailliertes Protokoll für die Vorbereitung einer NMR-Probe, die für die Messung von PREs geeignet ist, einschließlich der Schritte zur Proteinaufreinigung, der ortsgerichteten Spinmarkierung, der Einrichtung und Kalibrierung des Pulsprogramms, der Verarbeitung und Interpretation der NMR-Daten, wird bereitgestellt. Es werden wichtige experimentelle Überlegungen angestellt, die sich auf die Datenqualität und das experimentelle Ergebnis auswirken können, einschließlich der Probenkonzentration, der Auswahl der Spin-Markierung und der Entfernung paramagnetischer Komponenten.
Protokoll
Allgemeine Anforderungen an das Protokoll: Proteinreinigungseinrichtungen, UV-Vis-Spektrometer, Hochfeld-NMR-Spektrometer und Betriebssoftware, Post-Processing-Analysesoftware einschließlich; NMRPipe 32, Sparky33 (oder CCPN-Analyse34 oder NMRViewJ35).
1. Rekombinante Expression und Aufreinigung eines Proteins für PRE-Messungen
- Entwerfen Sie ein Expressionskonstrukt für das interessierende Protein, so dass ein einzelner Cysteinrest vorhanden ist. Mehrere Mutationen sind erforderlich, um ein freies Cystein an verschiedenen Stellen im interessierenden Protein einzuführen36.
- Exprimieren und reinigen Sie eine natürliche Häufigkeit (14N) oder 15N-markierte Probe des interessierenden Proteins unter Verwendung eines etablierten Protokolls37.
HINWEIS: E. coli-Expressionssysteme bieten eine kostengünstige und robuste Methode für die rekombinante Proteinexpression, da die Isotopenanreicherung von 15N eine minimale Voraussetzung für die biomolekulare heteronukleare NMR-Spektroskopie ist. Typische Schritte sind die Expression in minimalen Medien, die chromatographische Aufreinigung und das Entfernen der Affinitätsreinigungsmarkierung. Dieses Protokoll setzt voraus, dass ein robustes Expressions- und Reinigungsprotokoll etabliert wurde, das ausreichend Protein in geeigneter Qualität für NMR-Untersuchungen produzieren kann.- Halten Sie 1 mM Reduktionsmittel (DTT oder TCEP) in Puffern bei allen Reinigungsschritten aufrecht, um die Reaktion des freien Cysteins und die Bildung von intermolekularen Disulfidbrücken für IDPs zu verhindern.
ANMERKUNG: Einige Systeme können toleranter und weniger aggregationsanfällig für nicht reduzierende Bedingungen sein, abhängig von den spezifischen Eigenschaften des Proteins sowie der Temperatur, dem pH-Wert und dem Puffersystem, das für die Reinigung ausgewählt wurde38. - Entfernen Sie Affinitätsmarkierungen, die für die Reinigung verwendet werden, bevor Sie fortfahren, da sie auf unvorhersehbare Weise unspezifisch mit dem Protein interagieren oder möglicherweise reaktive Cysteinreste enthalten können, die versehentlich als unbeabsichtigte Bindungsstelle dienen könnten.
- Bereiten Sie eine 15N markierte Referenzprobe ohne Cysteinmutation(en) vor, die mit einer löslichen Version der Spin-Markierung gemischt ist, um den Beitrag von Lösungsmittel-PREs zu bewerten.
- Halten Sie 1 mM Reduktionsmittel (DTT oder TCEP) in Puffern bei allen Reinigungsschritten aufrecht, um die Reaktion des freien Cysteins und die Bildung von intermolekularen Disulfidbrücken für IDPs zu verhindern.
2. Konjugieren der 3-Maleimido-PROXYL-Nitroxid-Spin-Markierung
- Lagerung oder Austausch des gereinigten Proteins in einem entgasten Puffer, der 50 mM Tris pH 7 und 1 mM TCEP enthält; Der Puffer kann bei Bedarf auch bis zu 8 M Harnstoff enthalten, um die Proteinlöslichkeit zu unterstützen.
Alternativ kann eine Proteinstammlösung schnell in mindestens 10 Volumenäquivalente entgaster 50 mM Tris pH 7 und 1 mM TCEP-Puffer verdünnt werden. Stellen Sie sicher, dass die Proteinkonzentration vor dem Hinzufügen der Spin-Markierung mindestens 100 μM beträgt. - Fügen Sie 3-Maleimido-Proxyl aus einer Stammlösung zu einem 20-fachen molaren Überschuss des interessierenden Proteins hinzu. Die Probe vor Licht und Sauerstoff schützen und über Nacht bei Raumtemperatur oder 4 °C inkubieren; Sanftes Schaukeln oder kann die Etikettiereffizienz verbessern.
- Bereiten Sie Stammlösungen des Spin-Labels vor, indem Sie 3-Maleimido-Proxylpulver in 95%igem Ethanol auflösen. Aliquots der Brühe können bei -80 °C für weniger als 6 Monate gelagert werden.
- Kritischer Schritt: Entfernen Sie das nicht umgesetzte Freispin-Etikett, um unspezifische Lösungsmittel-PREs zu vermeiden. Dies wird durch Gelfiltration oder (vorzugsweise) umfangreiche Dialyse der Proteinprobe erreicht. In diesem Schritt wird das Protein auch in einen für NMR geeigneten Puffer eingebracht.
HINWEIS: Reduktionsmittel sollten frisch zubereitet werden, und die Kompatibilität zwischen den Pufferkomponenten sollte berücksichtigt werden. TCEP z. B. wird in Puffern auf Phosphatbasis schnell abgebaut, und diese Kombination sollte vermieden werden39. - Behandeln Sie alle Puffer, die ab diesem Schritt verwendet werden, mit einem chelatbildenden Harz, das selektiv für zweiwertige Metalle und Übergangsmetalle ist, um paramagnetische Ionen oder Spin-Label-Quencher zu entfernen. Wenn das Protein nicht in einem NMR-Puffer gespeichert werden kann, konzentrieren Sie das Protein, das schnell verdünnt werden soll, in einen für NMR geeigneten Puffer.
- Überwachung der Effizienz der Einarbeitung von Spin-Labels.
- Verwenden Sie das Ellman-Reagenz (5,5-Dithio-bis-(2-nitrobenzoesäure)) zur Quantifizierung freier Sulfhydrylgruppen in Lösung40.
HINWEIS: Detaillierte Protokolle sind vom Hersteller erhältlich. Für die Zwecke hier ist es wichtig, den Einbau der Spin-Markierung zu bestimmen, wobei die Konzentration der freien Sulfhydrylgruppen mit der Gesamtproteinkonzentration verglichen wird. Der Prozentsatz der freien Sulfhydrylgruppen ist der Prozentsatz der Moleküle, an denen keine Nitroxid-Spin-Markierung angebracht ist. - Überwachen Sie die Intensität des Peaks, der dem markierten Cysteinrest entspricht, um den Einbau der Spin-Markierung in das interessierende Protein zu beurteilen.
HINWEIS: Dies ist ein schneller und effektiver Ansatz, um den Grad der Spin-Markierung des Proteins zu bestimmen. Der vollständige Einbau der Spin-Markierung führt zum Verschwinden des Peaks aus dem Spektrum. Aufgrund der schlechten Dispersionscharakteristik von IDPs kann der Peak, der dem mutierten Cysteinrest entspricht, nicht immer leicht identifiziert werden, weshalb die Verwendung des Ellman-Reagenzes (Schritt 2.6.1) empfohlen wird.
- Verwenden Sie das Ellman-Reagenz (5,5-Dithio-bis-(2-nitrobenzoesäure)) zur Quantifizierung freier Sulfhydrylgruppen in Lösung40.
3. NMR-Probe für die Messung der intra- oder intermolekularen PRE vorbereiten
- Probenvorbereitung für die Messung der intramolekularen PRE
- 15N isotopenangereichertes, spinmarkiertes Protein wird in einem für NMR geeigneten Puffer auf eine Konzentration von mindestens 100 μM, aber nicht mehr als 300 μM hergestellt. Das Gesamtvolumen der Probe (einschließlichD2O) beträgt 500 - 550 μl.
HINWEIS: Zu den gängigen NMR-Puffern gehören Phosphat, Acetat, (Bi)carbonat und TRIS. Good's Puffer wie MES, HEPES können ebenfalls geeignet sein. Seien Sie vorsichtig bei der Auswahl von Puffern, um sicherzustellen, dass es nicht zu einer Kreuzreaktivität mit anderen Lösungskomponenten kommt. - Stellen Sie sicher, dass der pH-Wert ~7,2 oder niedriger ist, um die Auswirkungen des Amid-Protonenaustauschs mit Wasser zu minimieren. Halten Sie die Salzkonzentration so niedrig wie möglich (in der Regel weniger als 150 mM), obwohl die Hauptüberlegung darin besteht, die Proteinstabilität zu erhalten.
ANMERKUNG: Ansätze zur Durchführung von NMR-Experimenten unter salzreichen Bedingungen wurden an anderer Stelle beschrieben41.
- 15N isotopenangereichertes, spinmarkiertes Protein wird in einem für NMR geeigneten Puffer auf eine Konzentration von mindestens 100 μM, aber nicht mehr als 300 μM hergestellt. Das Gesamtvolumen der Probe (einschließlichD2O) beträgt 500 - 550 μl.
- Vorbereitung der Probe für die Messung der intermolekularen PRE
- Führen Sie diesen Schritt oder Schritt 3.1 aus. Sie werden nicht gleichzeitig durchgeführt. Bereiten Sie 14N natürliches, spinmarkiertes Protein im gewählten NMR-Puffer vor.
- Die Proteinprobe wird vorbereitet, indem 15N isotopenangereichertes, nicht spinmarkiertes Protein mit 1%-50% 14N natürlichem Spin-markiertem Protein gemischt wird, so dass die Endkonzentration mit der in 3.1.1 hergestellten Probe identisch ist. Das gesamte Probenvolumen (einschließlichD2O) beträgt 500 - 550 μl.
- Empirische Optimierung des Verhältnisses der 15N- und 14N-Proteine für jedes untersuchte Protein. Die Verhältnisse von 1 %, 5 % und 20 % des 14N-Spin-markierten Proteins sind gute Ausgangspunkte.
HINWEIS: Eine Anhäufung des PRE als Funktion des zugesetzten 14N-Spin-markierten Proteins deutet auf eine spezifische Wirkung hin; Die beobachtete PRE ist probenspezifisch, da sie von der Entfernung und der Population abhängt (wie oben diskutiert), und daher sind höhere Verhältnisse von 14N-Spin-markierten Proteinen erforderlich, wenn die Wechselwirkung besonders vorübergehend ist17.
- Übertragen Sie die NMR-Probe (entweder intra- oder intermolekular) mit einer Glaspipette oder Mikropipette mit langem Stiel (9") in ein 5-mm-NMR-Röhrchen, das für die Verwendung in Hochfeldmagneten geeignet ist. Stellen Sie sicher, dass alle NMR-Proben 5 % bis 10 % D2O enthalten, um die Feldsperre zu erleichtern.
HINWEIS: NMR-Röhrchen, die Polymerstopfen verwenden, um das erforderliche Probenvolumen zu reduzieren, werden aufgrund von Schwierigkeiten im Zusammenhang mit dem effektiven Proben-Shimming nicht für PRE-Messungen empfohlen.
4. NMR-Spektrometer einrichten und spezifische Parameter experimentieren
- Seien Sie äußerst vorsichtig, wenn Sie in der Nähe von supraleitenden Hochfeld-NMR-Spektrometern arbeiten.
HINWEIS: Zu den Gefahren gehören Verletzungen durch die plötzliche Beschleunigung metallischer Gegenstände in Richtung des Magneten, Interferenzen mit implantierten medizinischen Geräten und Ersticken durch eine plötzliche Freisetzung vonN2- und He2-Gas im Falle einer Magnetlöschung. Bei den folgenden Schritten wird davon ausgegangen, dass das Lesegerät die erforderliche Schulung durchlaufen hat, sich dieser und anderer lokaler Gefahren bewusst ist und vom Facility Manager die Genehmigung für den Betrieb des NMR-Spektrometers erhalten hat. Wenn Sie Zweifel an einem Schritt oder einer Anweisung haben, wenden Sie sich an den Einrichtungsleiter oder einen erfahrenen Benutzer, um mögliche Verletzungen oder Schäden am Spektrometer zu vermeiden. - Bei den folgenden Schritten wird von einem kommerziellen NMR-Spektrometer ausgegangen, auf dem eine moderne Version der Erfassungssteuerungssoftware ausgeführt wird. Laden Sie das Pulsprogramm und die Parameterdateien herunter und legen Sie sie in den entsprechenden Verzeichnissen ab.
HINWEIS: Ein Pulsprogramm und ein Parametersatz, die für die Verwendung mit einem Bruker-Spektrometer und TopSpin (3.2 oder höher) geeignet sind, sind auf Anfrage bei den Autoren erhältlich.- Kritischer Schritt: Vertrautheit mit der Installation nicht-nativer NMR-Pulsprogramme wird vorausgesetzt; Wenden Sie sich bei Bedarf an den Facility Manager oder einen erfahrenen Benutzer.
- Legen Sie die Probe in den Magneten, sperren Sie das 2-H-Signal mit dem Lock-Befehl, stimmen Sie den 1-H-Kanalgemäß den Einrichtungsprotokollen ab und passen Sie ihn an (das genaue Verfahren hängt davon ab, ob die Sonde mit einem Remote-Tune-and-Match-Modul ausgestattet ist).
- Passen Sie die Shims mit der Topshim-Unterroutine an, um die Unterdrückung des Lösungsmittelsignals zu optimieren.
- Kalibrieren Sie die 1-H- und 15-N-90°-Impulse mit Standardmethoden.
- Kalibrieren Sie den 1-H-Impuls mit dem Programm popt (verwenden Sie zuerst pulsecal zur Schätzung der Pulslänge).
- Kalibrieren Sie den 15-N-Impuls gegen eine Standardprobe. Stellen Sie sicher, dass dieser Wert kürzlich kalibriert wurde, indem Sie dies mit einem technischen Direktor oder einem erfahrenen Benutzer besprechen.
- Alternativ können Sie den 15-N-Impulsauf der Probe kalibrieren, indem Sie einen der 90°-Impulse eines HMQC-Experiments variieren, bis ein Nullsignal erreicht ist.
- Bestimmen Sie die richtige Dämpfung für geformte Impulse mit dem Unterprogramm shape tool (stdisp).
- Öffnen Sie die entsprechende Pulsformdatei, indem Sie auf das Ordnersymbol klicken. Die geformten Pulse befinden sich im Abschnitt Pulsparameter von ACQUPARS.
- Laden Sie die Impulsdefinitionsdatei und klicken Sie auf Wellenform analysieren > Form integrieren. Geben Sie den kalibrierten 1H 90° harten Impuls, die gewünschte Impulslänge und die Drehung (90° oder 180°) ein.
- Berechnen Sie den Leistungspegel des geformten Impulses, indem Sie die Änderung des Leistungspegels zur Dämpfung für den kalibrierten 90°-Impuls addieren.
- Zeichnen Sie einen Standard-HSQC von 1H, 15N (hsqcetfpf3gpsi) auf, um die Sweep-Breite und die Trägerfrequenz zu optimieren und die Wasserunterdrückungzu überprüfen 25.
- Passen Sie die Sweeping-Breite und die Anzahl der indirekten Bemaßungsinkremente mit den Befehlen sw und td oder direkt in den entsprechenden Dialogfeldern an. Typischerweise werden für die Erfassung von PREs spektrale Breiten gewählt, damit das Spektrum nicht gefaltet wird.
5. Aufbau des 1HN-T 2 Experiments
- Die geformten Impulse sind wie oben beschrieben (4.4.5-4.5.7) zu kalibrieren. Die geformten Pulsparameterdateien für das PRE-Experiment sind Eburp2.1000 (90°-Puls), Reburp.1000 und Iburp2.1000. Geben Sie die kalibrierten Pulslängen im Abschnitt Pulsparameter auf der Registerkarte ACQUPARS ein.
- In diesem Experiment wird der 1H N-T2unter Verwendung des Zwei-Zeit-Verzögerungspunkt-Ansatzes30 gemessen.
- Stellen Sie die Zeitverzögerungen ein, indem Sie die vdlist-Datei bearbeiten, die erste Verzögerung (Ta) wird auf 0,01 ms gesetzt.
- Wählen Sie die zweite Verzögerung (T b) unter Verwendung der Beziehung zum erwarteten maximalen PRE (T b ~ 1,15/(R 2,dia + Γ 2), wobei R 2,dia das R 2 der diamagnetischen Probe13 darstellt. Ohne Vorkenntnisse über die Größe des PRE-Beitrags zur beobachteten Relaxation ist es ein guter Ausgangspunkt, T b auf ~1x 1H T 2 zu setzen.
- Bestimmen Sie dann einen geeigneten Wert, indem Sie die ersten Inkremente (verarbeitet mit dem Befehl efp) der Ta und T b Spektren vergleichen und T b so einstellen, dass das Signal auf 40 % bis 50 % seines Anfangswerts abklingt.
HINWEIS: Dieser Ansatz optimiert das spektrale Signal-Rausch-Verhältnis, eine notwendige Überlegung für Proben, die nicht hochkonzentriert sein können (< 50 μM). Geeignete Werte von Tb sind probenabhängig, liegen aber typischerweise zwischen 8 und 40 ms für ein durchschnittlich großes Protein.
- Bestimmen Sie die Anzahl der aufzuzeichnenden komplexen Punkte und die Anzahl der Scans für eine ausreichende Signalmittelung. Da IDPs längere 15N T 2 aufweisen als gefaltete Proteine vergleichbarer Größe, können längere Aufnahmezeiten in der indirekten Dimension verwendet werden.
HINWEIS: Dieser Wert hängt von den spezifischen Eigenschaften des Proteins ab, kann aber grob aus dem 15N T2 geschätzt und durch die Überwachung des Signalabfalls im FID optimiert werden. Für die direkte Dimension sind 1024* komplexe Punkte (13 ppm Sweep-Breite, 112,6 ms Aufnahmezeit) für die meisten Proben ausreichend. - Berechnen Sie mit dem Befehl expt die Versuchszeit und starten Sie dann das Experiment mit dem Befehl zg.
6. Stellen Sie eine diamagnetische Probe her, indem Sie die Spin-Markierung mit Ascorbinsäure reduzieren
- Lösen Sie Natriumascorbat im NMR-Puffer auf und passen Sie den pH-Wert an den ursprünglichen NMR-Puffer an.
- Berechnen Sie die Konzentration des Natriumascorbat-Stocks so, dass ein 10-facher molarer Überschuss von Ascorbat über die Konzentration der Spin-Markierung mit der geringsten Änderung des Probenvolumens zugegeben werden kann. Zum Beispiel ist für eine Proteinprobe von 100 μM ein Vorrat von 100 mM Ascorbat angemessen. Um die Spin-Markierung zu reduzieren, müssen 5,5 μl Ascorbinsäure-Stammlösung zugegeben werden, was nur 1 % des gesamten Probenvolumens entspricht.
- Geben Sie die erforderliche Menge Ascorbinsäure in das NMR-Röhrchen, indem Sie einen Tropfen unter den Rand des Röhrchens geben, das Röhrchen verschließen, das Röhrchen vorsichtig umdrehen, um es zu mischen, und dann bei 200-400 x g für 10-20 s in einer handgekurbelten Zentrifuge schleudern, um die Probe am Boden des Röhrchens abzusetzen.
- Wickeln Sie das NMR-Röhrchen zum Schutz vor Licht in Folie ein und lassen Sie die Reaktion mindestens 3 Stunden lang ablaufen.
- 1HN-T 2 auf der diamagnetischen Probe mit den gleichen Parametern wie bei der paramagnetischen Probe aufzeichnen.
- Kalibrieren Sie die Impulse neu. Sie sollten sich jedoch gegenüber den paramagnetischen Messungen nicht verändert haben; Wenn sie sich signifikant unterscheiden (> 0,5 μs Unterschied), ist die Probenqualität zu berücksichtigen (z. B. Abbau, Ausfällung).
- Stellen Sie sicher, dass alle Erfassungsparameter, einschließlich der angegebenen Relaxationsverzögerungen (vdlist), der Anzahl der Dummy-Scans, der Anzahl der erfassten Scans, der Anzahl der erfassten komplexen Punkte, der Erfassungszeit, der Sweep-Breiten und der Trägerfrequenzen, für die diamagnetischen und paramagnetischen Proben gleich bleiben.
7. Paramagnetische und diamagnetische Spektren verarbeiten
- Kopieren Sie die Daten auf den lokalen Computer oder die Arbeitsstation, auf dem NMRPipe und Sparky installiert und konfiguriert sind. Erstellen Sie einen Ordner mit dem Namen proc im Experimentdatenverzeichnis, das die ser-Datei enthält.
- Kopieren Sie die NMRPipe-Skripte fid.com, p3d.com und nmrproc.com in proc (Verarbeitungsskripts sind auf Anfrage bei den Autoren erhältlich).
- Verwenden Sie das fid.com-Skript , um das Bruker-Datenformat (ser) in das NMRPipe-Format zu konvertieren.
- Verwenden Sie das Skript p3D.com , um die Pseudo3D-Ebenen in einzelne Spektren aufzuteilen.
- Verwenden Sie das nmrproc.com Skript, um die Ausgabe des fid.com Skripts zu lesen, Lösungsmittelunterdrückung und eine Fensterfunktion anzuwenden, Nullen an die Rohdaten anzuhängen (Nullfüllung), Phasenkorrektur anzuwenden, eine Fourier-Transformation auszuführen, die Daten für die Anzeige zu trimmen und die verarbeiteten Daten auf die Festplatte zu schreiben. Das Skript gibt für jede aufgezeichnete Relaxationsverzögerung (Ta und Tb) eine Datei aus.
HINWEIS: Jedes dieser Skripts kann angepasst werden, um die Verarbeitung für die spezifischen Details jedes Experiments zu optimieren. Tutorials und Beispieldatensätze sind in der NMRPipe-Distribution enthalten, die auf der NMRPipe-Websiteverfügbar ist 32. NMRDraw kann für die spektrale Betrachtung während der Verarbeitung verwendet werden (z. B. Einstellen geeigneter Phasenwinkel usw.). Die für NMRPipe-Befehle verfügbaren Optionen können mit dem Befehl nmrPipe -help angezeigt werden.
8. Resonanzzuweisungen übertragen und Spitzenhöhen extrahieren
- Ändern Sie die Datei-Header-Informationen für jede Spektrum-Datei (T a, Tb für paramagnetische und diamagnetische Proben) mit dem Befehl sethdr [Dateiname] -ndim 2.
- Verwenden Sie Sparky, um die Peakhöhen33 zu extrahieren, indem Sie die Schritte 8.3 bis 8.5 ausführen. Andere Softwarepakete, einschließlich NMRPipe (NMRDraw)32, CCPN Analysis34 und NMRViewJ35 , sind ebenfalls geeignet.
- Lesen Sie die Spektraldateien in Sparky ein. In diesem Schritt besteht der Datensatz aus einem Spektrum für jedes Zeitpunktspektren (Ta, Tb), sowohl für die paramagnetischen als auch für die diamagnetischen Proben, gemessen für jede Position der Spin-Markierung im Protein.
- Verwenden Sie Sparky, um Peaks auszuwählen (Befehl: F8, dann klicken und ziehen) und Zuweisungen mit dem Werkzeug "Peakliste übertragen" aus einer Referenz-Peakliste zu übertragen.
ANMERKUNG: Resonanzzuweisungen des interessierenden Proteins sind für die sequenzspezifische Interpretation der beobachteten PREs36 erforderlich.- Stellen Sie die Konturen sowohl in paramagnetischen als auch in diamagnetischen Spektren auf die gleiche Ebene ein. Stellen Sie sicher, dass die Konturen so eingestellt werden, dass die nach der Zeitverzögerung gesammelten Spektren nicht absichtlich Peaks ausschließen, sondern hoch genug sind, damit die T-a-Spektren nicht übermäßig verrauscht sind.
- Speichern Sie die neuen Peak-Listen für jedes Spektrum und fügen Sie die gemessene Peak-Intensität und das von Sparky berechnete Signal-Rausch-Verhältnis hinzu (Befehl: lt , um die Peakliste zu öffnen, klicken Sie auf Optionen, um die Spalten für Intensität und SNR einzubeziehen, Befehl: Speichern).
9. Extrahieren Sie 1HN-T 2-Raten für jeden Rückstand und berechnen Sie PRE
- Importieren Sie die Peak-Listen in eine Tabellenkalkulationssoftware oder eine bevorzugte Programmiersprache wie Python.
ANMERKUNG: Für jede Spin-Markierungsposition auf dem Protein besteht der Datensatz aus vier Peaklisten mit zugehörigen Peak-Intensitäten, jeweils eine von T a und Tb für das paramagnetische und diamagnetische Experiment. - Berechnen Sie 1HN R2 sowohl für die paramagnetischen als auch für die diamagnetischen Proben mit der Gleichung:
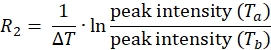
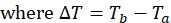
- Verwenden Sie die obige Gleichung, um die Relaxationsrate für jeden Rückstand für die paramagnetischen und diamagnetischen Proben zu bestimmen.
- Bestimmen Sie die 1-H-N-Γ-2-Rate für jeden Rückstand mit der folgenden Gleichung:
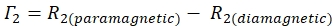
- Verwenden Sie das von Sparky berechnete Signal-Rausch-Verhältnis (SNR), um die Unsicherheit der Peakhöhe für jeden Rest zu berechnen.
- Propagieren Sie den Fehler mit der folgenden Gleichung:

- Plot 1HN-Γ 2 als Funktion der Rückstandszahl unter Verwendung eines Streudiagramms einschließlich des in 9.6 berechneten Fehlers.
Ergebnisse
Intramolekulare 1H N-Γ2 PREs wurden auf einem selbstassoziierenden, intrinsisch ungeordneten Fragment (Reste 171-264) detektiert, das von der Domäne mit geringer Komplexität des RNA-bindenden Proteins EWSR142 abgeleitet wurde (Abbildung 3). Es wird erwartet, dass Rückstände in unmittelbarer sequentieller Nähe des Spin-Markierungs-Bindungspunktes (z. B. Rückstand 178 oder 260 in Abbildung 3)...
Diskussion
Eine Methode zur Charakterisierung transienter Wechselwirkungen, die bei niedrigen Populationen zwischen intrinsisch ungeordneten Proteinen und verschiedenen Bindungspartnern bestehen, wurde mittels PRE vorgestellt. Im gezeigten Beispiel ist das Protein selbstassoziierend, so dass die PRE aus einer Kombination von inter- und intramolekularen Wechselwirkungen entstehen kann. Diese Methode lässt sich leicht auf heterogene Proben ausdehnen, bei denen die Wechselwirkungen zwischen zwei verschiedenen Proteinen charakterisier...
Offenlegungen
Alle Autoren haben das Manuskript gelesen und freigegeben. Es werden keine Interessenkonflikte deklariert.
Danksagungen
Wir danken Dr. Jinfa Ying und Dr. Kristin Cano für hilfreiche Gespräche und technische Unterstützung. DSL ist ein St. Baldrick's Scholar und bedankt sich für die Unterstützung der St. Baldrick's Foundation (634706). Diese Arbeit wurde teilweise von der Welch Foundation (AQ-2001-20190330) an DSL, dem Max and Minnie Tomerlin Voelcker Fund (Voelcker Foundation Young Investigator Grant an DSL), UTHSA Start-Up Funds an DSL und einem Greehey Graduate Fellowship in Children's Health an CNJ unterstützt. Diese Arbeit basiert auf Forschungsarbeiten, die in den Structural Biology Core Facilities durchgeführt werden, die Teil der institutionellen Forschungskerne am Health Science Center der University of Texas in San Antonio sind und vom Büro des Vizepräsidenten für Forschung und der Mays Cancer Center Drug Discovery and Structural Biology Shared Resource (NIH P30 CA054174) unterstützt werden.
Materialien
| Name | Company | Catalog Number | Comments |
| 0.45 µm and 0.22 µm syringe filters | Millipore Sigma | SLHVM33RS SLGVR33RS | Filter lysate before first purification step and before size exclusion chromatography. |
| 100 mm Petri Dish | Fisher | FB0875713 | Agar plates for bacterial transformation. |
| 14N Ammonium chloride | Sigma Aldrich | 576794 | Use of 15N in M9 medium will produce an NMR visible protein, 14N will produces an NMR invisible protein |
| 15N Ammonium chloride | Sigma Aldrich | 299251 | Use of 15N in M9 medium will produce an NMR visible protein, 14N will produces an NMR invisible protein |
| 3 L Fernbach baffled flask | Corning | 431523 | Bacterial expression culture |
| 3-Maleimido-Proxyl | Sigma Aldrich | 253375 | Nitroxide spin label |
| 50 mL conical centrifuge tubes | Thermo Fisher | 14-432-22 | Solution/protein storage |
| Amicon centrifugal filter | Millipore Sigma | UFC900308 | Protein concentration |
| Ampicillan | Sigma Aldrich | A5354 | Antibiotic for a selective marker, exact choice depends on the expression construct plasmid |
| Analytical balance | Oahus | 30061978 | Explorer Pro, for weighing reagents |
| Ascorbic acid | Sigma Aldrich | AX1775 | Reduces nitroxide spin label |
| Autoclave | Sterilize glassware and culture media | ||
| Calcium chloride | Sigma Aldrich | C4901 | M9 media component |
| Centrifuge bottles | Thermo Fisher | 010-1459 | Harvest E. coli cells after recombinant protein expression |
| Centrifuge, hand-crank | Thomas Scientific | 0241C68 | Boekel hand-driven, low-speed centrifuge with 15 mL buckets that can accommodate NMR tubes |
| Chelex 100 | Sigma Aldrich | C7901 | Remove contaminating paramagnetic compounds from buffer solutions |
| Computer workstation | Linux or Mac OS compatable with NMR data processing and analysis software packages such as NMRPipe and Sparky | ||
| Deuterium oxide | Sigma Aldrich | 151882 | Needed for NMR lock signal |
| Dextrose | Sigma Aldrich | D9434 | M9 media component |
| Dibasic Sodium Phosphate | Sigma Aldrich | S5136 | M9 media component |
| Ellman's reagent (5,5-dithio-bis-(2-nitrobenzoic acid) | Thermo Fisher | 22582 | Quantification of free cystiene residues |
| High speed centrifuge tubes | Thermo Fisher | 3114-0050 | Used to clear bacterial lysate. |
| High-field NMR instrument (600 - 800 MHz) | Bruker | Equiped with a multichannel cryogenic probe and temperature control | |
| IMAC column, HisTrap FF | Cytvia | 17528601 | Initial fractionation of crude bacterial lysate |
| Isopropyl B-D-thiogalactoside (IPTG) | Sigma Aldrich | I6758 | Induces protein expression for genes under control of lac operator |
| LB agar | Thermo Fisher | 22700025 | Items are used for transforming E. coli to express protein of interest, substitions for any of these items with like products is acceptable. |
| LB broth | Thermo Fisher | 12780052 | |
| Low-pressure chromatography system | Bio-Rad | 7318300 | BioRad BioLogic is used for low-pressure chomatograph such as running IMAC columns |
| Magnesium sulfate | Sigma Aldrich | M7506 | M9 media component |
| Medium pressure chromatography system | Bio-Rad | 7880007 | BioRad NGC equipped with a multi-wavelength detector, pH and conductivity monitors, and automatic fraction collector |
| MEM vitamin solution | Sigma Aldrich | M6895 | M9 media component |
| Microfluidizer | Avestin | EmulsiFlex-C3 | Provides rapid and efficient bacterial cell lysis |
| Micropipettes | Thermo Fisher | Calibrated set of micropippetters with properly fitting disposable tips (available from multiple manufacturers e.g. Eppendorf) | |
| Monobasic potassium phosphate | Sigma Aldrich | 1551139 | M9 media component |
| NMR pipettes | Sigma Aldrich | 255688 | To remove sample from NMR tube |
| NMR sample tube | NewEra | NE-SL5 | Suitable for high-field NMR spectrometers |
| Preparative Centrifuge | Beckman Coulter | Avanti J-HC | Harvest E. coli cells after recombinant protein expression |
| Round bottom polystyrene centrifuge tubes | Corning | 352057 | Clear bacterial lysate |
| Shaking incubator | Eppendorf | S44I200005 | Temperature controlled growth of E. coli starter and expression cultures |
| Sodium chloride | Sigma Aldrich | S5886 | M9 media component |
| Sonicating water bath and vacuum source | Thomas Scientific | Used to degas buffer solutions | |
| Sonicator | Thermo Fisher | FB505110 | Used for bacterial cell lysis or shearing bacterial DNA |
| Spectrophotometer | Implen | OD600 Diluphotometer | Monitor growth of E.coli protein expression cultures |
| Superdex 200 16/600 size exculsion colum | Cytvia | 28989333 | Final protein purification step |
| Topspin software, version 3.2 or later | Bruker | Operating software for the NMR instrument | |
| Transformation competent E. coli cells | Thermo Fisher | C600003 | One Shot BL21 Star (DE3) chemically competent E. coli, other strains may be compatable |
| Tris(2-carboxyethyl)phosphine (TCEP) | ThermoFisher | 20490 | Reducing agent compatable with some sulfhydryl-reactive conjugations |
| UV-Vis spectrophotometer | Implen | NP80 | Measure protein concentration. |
| Water bath, temperature controlled | ThermoFisher | FSGPD25 | For heat shock step of bacterial transformation |
| Yeast extract | Sigma Aldrich | Y1625 | For supplementing M9 media if required |
Referenzen
- Dyson, H. J., Wright, P. E. Intrinsically unstructured proteins and their functions. Nature Reviews: Molecular Cell Biology. 6 (3), 197-208 (2005).
- Korneta, I., Bujnicki, J. M. Intrinsic disorder in the human spliceosomal proteome. PLoS Computational Biology. 8 (8), 1002641 (2012).
- Frege, T., Uversky, V. N. Intrinsically disordered proteins in the nucleus of human cells. Biochemistry and Biophysics Reports. 1, 33-51 (2015).
- Liu, J., et al. Intrinsic disorder in transcription factors. Biochemistry. 45 (22), 6873-6888 (2006).
- El Hadidy, N., Uversky, V. N. Intrinsic disorder of the BAF complex: Roles in chromatin remodeling and disease development. International Journal of Molecular Sciences. 20 (21), (2019).
- Wright, P. E., Dyson, H. J. Intrinsically disordered proteins in cellular signalling and regulation. Nature Reviews: Molecular Cell Biology. 16 (1), 18-29 (2015).
- Brangwynne, C. P. Phase transitions and size scaling of membrane-less organelles. Journal of Cell Biology. 203 (6), 875-881 (2013).
- Shin, Y., Brangwynne, C. P. Liquid phase condensation in cell physiology and disease. Science. 357 (6357), (2017).
- Cavanagh, J. . Protein NMR spectroscopy : principles and practice. 1st edition. , (2018).
- Sekhar, A., Kay, L. E. NMR paves the way for atomic level descriptions of sparsely populated, transiently formed biomolecular conformers. Proceedings of the National Academy of Sciences of the United States of America. 110 (32), 12867-12874 (2013).
- Anthis, N. J., Clore, G. M. Visualizing transient dark states by NMR spectroscopy. Quarterly Reviews of Biophysics. 48 (1), 35-116 (2015).
- Alderson, T. R., Kay, L. E. NMR spectroscopy captures the essential role of dynamics in regulating biomolecular function. Cell. 184 (3), 577-595 (2021).
- Clore, G. M., Iwahara, J. Theory, practice, and applications of paramagnetic relaxation enhancement for the characterization of transient low-population states of biological macromolecules and their complexes. Chemical Reviews. 109 (9), 4108-4139 (2009).
- Wu, K. P., Baum, J. Detection of transient interchain interactions in the intrinsically disordered protein alpha-synuclein by NMR paramagnetic relaxation enhancement. Journal of the American Chemical Society. 132 (16), 5546-5547 (2010).
- Janowska, M. K., Wu, K. P., Baum, J. Unveiling transient protein-protein interactions that modulate inhibition of alpha-synuclein aggregation by beta-synuclein, a pre-synaptic protein that co-localizes with alpha-synuclein. Scientific Reports. 5, 15164 (2015).
- Murthy, A. C., et al. Molecular interactions underlying liquid-liquid phase separation of the FUS low-complexity domain. Nature Structural & Molecular Biology. 26 (7), 637-648 (2019).
- Fawzi, N. L., Doucleff, M., Suh, J. Y., Clore, G. M. Mechanistic details of a protein-protein association pathway revealed by paramagnetic relaxation enhancement titration measurements. Proceedings of the National Academy of Sciences of the United States of America. 107 (4), 1379-1384 (2010).
- Griffith, O. H., Waggoner, A. S. Nitroxide free radicals: spin labels for probing biomolecular structure. Accounts of Chemical Research. 2 (2), 17-24 (1969).
- Bertini, I., Luchinat, C., Parigi, G., Ravera, E. . NMR of Paramagnetic Macromolecules, Applications to Metallobiomolecules and Models. 2 edn. , (2016).
- Bloembergen, N., Purcell, E. M., Pound, R. V. Relaxation effects in nuclear magnetic resonance absorption. Physical Review. 73 (7), 679-712 (1948).
- Solomon, I. Relaxation processes in a system of two spins. Physical Review. 99 (2), 559 (1955).
- Clore, G. M. Practical aspects of paramagnetic relaxation enhancement in biological macromolecules. Methods in Enzymology. 564, 485-497 (2015).
- Klare, J. P. Site-directed spin labeling EPR spectroscopy in protein research. Biological Chemistry. 394 (10), 1281-1300 (2013).
- Clore, G. M., Tang, C., Iwahara, J. Elucidating transient macromolecular interactions using paramagnetic relaxation enhancement. Current Opinion in Structural Biology. 17 (5), 603-616 (2007).
- Melanson, M., Sood, A., Torok, F., Torok, M. Introduction to spin label electron paramagnetic resonance spectroscopy of proteins. Biochemistry and Molecular Biology Education. 41 (3), 156-162 (2013).
- Czogalla, A., Pieciul, A., Jezierski, A., Sikorski, A. F. Attaching a spin to a protein -- site-directed spin labeling in structural biology. Acta Biochimica Polonica. 54 (2), 235-244 (2007).
- Lindfors, H. E., de Koning, P. E., Drijfhout, J. W., Venezia, B., Ubbink, M. Mobility of TOAC spin-labelled peptides binding to the Src SH3 domain studied by paramagnetic NMR. Journal of Biomolecular NMR. 41 (3), 157-167 (2008).
- Fawzi, N. L., et al. A rigid disulfide-linked nitroxide side chain simplifies the quantitative analysis of PRE data. Journal of Biomolecular NMR. 51 (1-2), 105-114 (2011).
- Bleicken, S., et al. gem-Diethyl pyrroline nitroxide spin labels: Synthesis, EPR characterization, rotamer libraries and biocompatibility. ChemistryOpen. 8 (8), 1035 (2019).
- Iwahara, J., Tang, C., Clore, G. M. Practical aspects of 1H transverse paramagnetic relaxation enhancement measurements on macromolecules. Journal of Magnetic Resonance. 184, 185-195 (2007).
- Venditti, V., Fawzi, N. L. Probing the atomic structure of transient protein contacts by paramagnetic relaxation enhancement solution NMR. Methods in Molecular Biology. 1688, 243-255 (2018).
- Delaglio, F., et al. NMRPipe: a multidimensional spectral processing system based on UNIX pipes. Journal of Biomolecular NMR. 6 (3), 277-293 (1995).
- Lee, W., Tonelli, M., Markley, J. L. NMRFAM-SPARKY: enhanced software for biomolecular NMR spectroscopy. Bioinformatics. 31 (8), 1325-1327 (2015).
- Vranken, W. F., et al. The CCPN data model for NMR spectroscopy: development of a software pipeline. Proteins. 59 (4), 687-696 (2005).
- Johnson, B. A. Using NMRView to visualize and analyze the NMR spectra of macromolecules. Methods in Molecular Biology. 278, 313-352 (2004).
- Sjodt, M., Clubb, R. T. Nitroxide labeling of proteins and the determination of paramagnetic relaxation derived distance restraints for NMR studies. Bio-Protocol. 7 (7), (2017).
- Zhang, H., van Ingen, H. Isotope-labeling strategies for solution NMR studies of macromolecular assemblies. Current Opinion in Structural Biology. 38, 75-82 (2016).
- Rabdano, S. O., et al. Onset of disorder and protein aggregation due to oxidation-induced intermolecular disulfide bonds: case study of RRM2 domain from TDP-43. Scientific Reports. 7 (1), 11161 (2017).
- Burns, J. A., Butler, J. C., Moran, J., Whitesides, G. M. Selective reduction of disulfides by tris(2-carboxyethyl)phosphine. Journal of Organic Chemistry. 56 (8), 2648-2650 (1991).
- Ellman, G. L. Tissue sulfhydryl groups. Archives of Biochemistry and Biophysics. 82 (1), 70-77 (1959).
- Binbuga, B., Boroujerdi, A. F., Young, J. K. Structure in an extreme environment: NMR at high salt. Protein Science. 16 (8), 1783-1787 (2007).
- Schwartz, J. C., Cech, T. R., Parker, R. R. Biochemical properties and biological functions of FET proteins. Annual Review of Biochemistry. 84, 355-379 (2015).
- Nabuurs, S. M., de Kort, B. J., Westphal, A. H., van Mierlo, C. P. Non-native hydrophobic interactions detected in unfolded apoflavodoxin by paramagnetic relaxation enhancement. European Biophysics Journal. 39 (4), 689-698 (2010).
- Wiedemann, C., Kumar, A., Lang, A., Ohlenschlager, O. Cysteines and disulfide bonds as structure-forming units: Insights from different domains of life and the potential for characterization by NMR. Frontiers in Chemistry. 8, 280 (2020).
- Wommack, A. J., et al. NMR solution structure and condition-dependent oligomerization of the antimicrobial peptide human defensin 5. Biochemistry. 51 (48), 9624-9637 (2012).
- Taylor, A. M., et al. Detailed characterization of cysteine-less P-glycoprotein reveals subtle pharmacological differences in function from wild-type protein. British Journal of Pharmacology. 134 (8), 1609-1618 (2001).
- Hu, K., Doucleff, M., Clore, G. M. Using multiple quantum coherence to increase the 15N resolution in a three-dimensional TROSY HNCO experiment for accurate PRE and RDC measurements. Journal of Magnetic Resonance. 200 (2), 173-177 (2009).
- Anthis, N. J., Doucleff, M., Clore, G. M. Transient, sparsely populated compact states of apo and calcium-loaded calmodulin probed by paramagnetic relaxation enhancement: interplay of conformational selection and induced fit. Journal of the American Chemical Society. 133 (46), 18966-18974 (2011).
- Battiste, J. L., Wagner, G. Utilization of site-directed spin labeling and high-resolution heteronuclear nuclear magnetic resonance for global fold determination of large proteins with limited nuclear overhauser effect data. Biochemistry. 39 (18), 5355-5365 (2000).
- Donaldson, L. W., et al. Structural characterization of proteins with an attached ATCUN motif by paramagnetic relaxation enhancement NMR spectroscopy. Journal of the American Chemical Society. 123 (40), 9843-9847 (2001).
- Gaponenko, V., et al. Protein global fold determination using site-directed spin and isotope labeling. Protein Science. 9 (2), 302-309 (2000).
- Trindade, I. B., Invernici, M., Cantini, F., Louro, R. O., Piccioli, M. PRE-driven protein NMR structures: an alternative approach in highly paramagnetic systems. FEBS Journal. 288 (9), 3010-3023 (2021).
- Nitsche, C., Otting, G. Pseudocontact shifts in biomolecular NMR using paramagnetic metal tags. Progress in Nuclear Magnetic Resonance Spectroscopy. 98-99, 20-49 (2017).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten