Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Mejora de la relajación paramagnética para detectar y caracterizar las autoasociaciones de proteínas intrínsecamente desordenadas
En este artículo
Resumen
Se presenta un protocolo para la aplicación de espectroscopía de RMN de mejora de la relajación paramagnética para detectar interacciones inter e intramoleculares débiles y transitorias en proteínas intrínsecamente desordenadas.
Resumen
Las proteínas intrínsecamente desordenadas y las regiones intrínsecamente desordenadas dentro de las proteínas constituyen una parte grande y funcionalmente significativa del proteoma humano. La naturaleza altamente flexible de estas secuencias les permite formar interacciones débiles, de largo alcance y transitorias con diversos socios biomoleculares. Las interacciones específicas pero de baja afinidad promueven la unión promiscua y permiten que un solo segmento intrínsecamente desordenado interactúe con una multitud de sitios diana. Debido a la naturaleza transitoria de estas interacciones, pueden ser difíciles de caracterizar mediante métodos de biología estructural que se basan en proteínas para formar una conformación única y predominante. La RMN de mejora de la relajación paramagnética es una herramienta útil para identificar y definir la base estructural de las interacciones débiles y transitorias. Se describe un protocolo detallado para el uso de la mejora de la relajación paramagnética para caracterizar los complejos de encuentro poco poblados que se forman entre las proteínas intrínsecamente desordenadas y sus proteínas, ácidos nucleicos u otros socios biomoleculares.
Introducción
El trastorno intrínseco (DI) describe proteínas (IDP) o regiones dentro de las proteínas (IDR) que no se pliegan espontáneamente en estructuras secundarias o terciarias estables, sino que son biológicamente activas. En general, la función de los IDP/IDR es facilitar interacciones específicas pero reversibles con biomoléculas en condiciones fisiológicas1. Por lo tanto, las IDP y las IDR están involucradas en una variedad de funciones celulares, incluido el reclutamiento, la organización y la estabilización de complejos multiproteicos, por ejemplo, el ensamblaje y la actividad del espliceosoma2, el reclutamiento y la organización de componentes en los sitios de daño del ADN3, la organización y estabilización del reclutamiento de complejos de transcripción4 o del remodelador de cromatina BAF5. Además, los IDP se encuentran en nexos de señalización donde su promiscuidad para diferentes socios de unión les permite mediar en la transferencia de información a través de redes de proteínas celulares6. Trabajos recientes también han revelado una propensión de las regiones IDR a autoasociarse formando condensados biomoleculares a través del proceso de separación de fases líquido-líquido7. Muchas de las funciones mencionadas anteriormente que involucran el DI también se cree que involucran algún aspecto de la formación de condensado8. A pesar de la importancia de la identificación para el ensamblaje de complejos biomoleculares, la estabilización, el andamiaje y la transducción de señales, los detalles atómicos de sus interacciones específicas son difíciles de identificar, ya que las IDP y las IDR no suelen ser susceptibles de investigaciones estructurales mediante cristalografía de rayos X o microscopía electrónica criogénica.
La resonancia magnética nuclear (RMN) es una técnica ideal para investigar el DI, ya que no depende de la presencia de conjuntos estructurales rígidos u homogéneos, sino que informa sobre el entorno local inmediato de los núcleos individuales. La frecuencia de resonancia, o cambio químico, de un núcleo en una molécula dada está influenciada por campos magnéticos débiles inducidos por la distribución electrónica local, que a su vez depende de las longitudes de enlace, los ángulos, la proximidad de otros núcleos, las interacciones con los socios de unión yotros factores. Por lo tanto, cada núcleo actúa como una sonda estructural única y específica del sitio sensible a los cambios en su entorno químico local. A pesar de estas ventajas, la RMN es una técnica a granel, y el cambio químico observado es el promedio de todos los ambientes muestreados por un núcleo en particular. Se han desarrollado una serie de técnicas de RMN, muchas de las cuales se describen en este número, para recuperar información estructural, dinámica y cinética sobre conformaciones biomoleculares de alta energía y poco pobladas contenidas en el desplazamiento químico promedio10,11. Aunque transitoriamente poblados, la identificación y cuantificación de estos estados son importantes para determinar los detalles de los mecanismos funcionales12. Por ejemplo, en el caso de IDPs e IDRs, el conjunto conformacional puede estar sesgado para muestrear preferentemente conformaciones que son productivas para la formación de complejos de encuentro con compañeros de unión fisiológicos. La detección de estos estados, así como la identificación de las interacciones y dinámicas inter e intramoleculares específicas del residuo, son importantes para determinar los mecanismos estructurales subyacentes de la función de las proteínas y la formación de complejos.
Se describe un protocolo para el uso de la RMN de mejora de la relajación paramagnética (PRE) para investigar estados transitorios poco poblados importantes para la formación de complejos biomoleculares mediados por IDP/IDR13. Este enfoque es útil para estudiar las interacciones transitorias proteína-proteína, como las que promueven el ensamblaje de fibrillas amiloides a partir de la α-sinucleína 14,15 o la autoasociación de FUS16, así como para caracterizar interacciones proteína-proteínas específicas, como entre proteínas de señalización17. Se presenta un ejemplo de un IDP autoasociado, en el que las interacciones inter e intramoleculares específicas dan lugar a estados preferentemente compactados, así como a interacciones específicas del sitio que impulsan la autoasociación.
El PRE surge de la interacción dipolar magnética de un núcleo a un centro paramagnético con un tensor g isótropo, comúnmente suministrado en forma de un electrón desapareado en un grupo nitrógeno o como un átomo metálico paramagnético18 (Figura 1). Mientras que los átomos con tensores g anisotrópicos también producen un efecto PRE, el análisis de estos sistemas es más difícil debido a los efectos de confusión aportados por los desplazamientos de pseudocontacto (PCS) o el acoplamiento dipolar residual (RDC)13,19. La fuerza de la interacción entre un núcleo y el centro paramagnético depende de la distancia <r-6> entre los dos. Esta interacción da como resultado un aumento en las tasas de relajación nuclear, lo que provoca un ensanchamiento de la línea detectable incluso para interacciones de largo alcance (~10-35 Å), porque el momento magnético del electrón desapareado es muy fuerte20,21. La detección de estados transitorios con el PRE es posible si se cumplen las dos condiciones siguientes; (1) la interacción transitoria está en intercambio rápido en la escala de tiempo de RMN (el desplazamiento químico observado es un promedio ponderado por población de los estados de intercambio); y (2) la distancia entre los núcleos y el centro paramagnético es más corta en el estado transitoriamente poblado que en el estado principal11. El PRE transversal se denota Γ2 y, a efectos prácticos, se calcula a partir de la diferencia en las tasas de relajación transversal de 1H entre una muestra que contiene un centro paramagnético y un control diamagnético. Para un tratamiento en profundidad de la teoría del PRE y los cambios de pseudocontacto relacionados en regímenes de intercambio rápido y lento, se remite al lector a las revisiones exhaustivas de Clore y colaboradores13,22. Aquí, solo se considera la situación en la que 1H N-Γ 2 se encuentra en el régimen de intercambio rápido, donde debido a la dependencia r-6 del PRE, la tasa de relajación observada está relacionada tanto con la distancia a la que el centro paramagnético se acerca al núcleo como con la cantidad de tiempo que pasa en esa conformación. Por lo tanto, las conformaciones transitorias que no implican un acercamiento cercano producen un PRE pequeño, mientras que las interacciones más cercanas, incluso si son de corta duración, producirán un PRE más grande.
En el caso de los PDI, el PRE se utiliza para medir y diferenciar las interacciones que se producen dentro de una sola molécula (intramolecular) y entre moléculas separadas (intermolecular). Al unir un centro paramagnético a una proteína visible de RMN (p. ej., marcada con 15N) o invisible de RMN (p. ej., abundancia natural de 14N), se puede determinar la fuente (inter o intramolecular) del PRE (Figura 2). La mutagénesis dirigida al sitio que introduce un residuo de cisteína es un enfoque conveniente para unir un centro paramagnético (etiqueta de espín) a una proteína23. Se han propuesto varios tipos de moléculas para su uso como etiquetas de espín, incluyendo la quelación de metales (basada en EDTA) y la de radicales libres (basada en nitrógeno)24. Se han descrito varias etiquetas de centrifugado de nitrógeno y están disponibles con diferentes químicas reactivas a la cisteína, como metanetiosulfonato, maleimida y yodoacetamida25,26 (Figura 1). La flexibilidad inherente de la etiqueta o del enlazador puede ser problemática para ciertos análisis y, en estas situaciones, se han propuesto diferentes estrategias para limitar el movimiento de la marca, como la adición de grupos químicos voluminosos o el uso de un segundo enlazador para anclar la etiqueta a la proteína (unión de dos sitios)27, Artículo 28. Además, las etiquetas disponibles comercialmente pueden contener proteínas diastereoméricas, pero generalmente esto no contribuirá a la PRE29 observada. Se describe el uso del 3-Maleimido-PROXYL unido a una cisteína libre a través de la química de la maleimida, ya que está fácilmente disponible, es rentable, no reversible y el agente reductor tris(2-carboxietil)fosfina (TCEP) se puede mantener en la solución durante toda la reacción de marcado. Dado que el 3-maleimido-proxyl tiene un tensor g isotrópico, no se inducen PCS ni RDC, y se pueden utilizar las mismas asignaciones de desplazamiento químico tanto para las muestras paramagnéticas como para las diamagnéticas13.
El 1HN-T 2 se mide utilizando una estrategia de dos puntos de tiempo (T a, Tb) que previamente ha demostrado ser tan precisa como la recopilación de una serie de evolución completa que consta de 8 a 12 puntos de tiempo30. El primer punto de tiempo (T a) se establece lo más cerca posible de cero, y la longitud óptima del segundo punto de tiempo depende de la magnitud del mayor PRE esperado para una muestra dada y se puede estimar a partir de: Tb ~ 1,15/(R 2,dia + Γ 2) donde R 2,dia representa el R 2 de la muestra diamagnética13. Si se desconoce la magnitud de los PRE más grandes, establecer T b en ~ una vez 1 HT 2 de la proteína es una buena estimación inicial y optimizarse aún más ajustando T2 para mejorar la señal a ruido. Esta estrategia de medición de dos puntos reduce significativamente el tiempo experimental requerido para medir los PRE y permite tiempo para un mayor promedio de señales, particularmente porque se utilizan muestras relativamente diluidas para minimizar los efectos de los contactos no específicos entre moléculas. Para medir 1H,N-T, 2 se utiliza una secuencia de impulsos basada en HSQC y se ha descrito en detalle en otro lugar30. Para mejorar la sensibilidad, los pulsos duros de las transferencias INEPT hacia adelante y hacia atrás pueden reemplazarse con pulsos con forma; alternativamente, la secuencia se convierte fácilmente en una lectura31 basada en TROSY. Dado que las IDP suelen tener tasas de relajación transversal mucho más largas, lo que da lugar a anchos de línea más estrechos (debido al desorden inherente) que las proteínas globulares de tamaño similar, se pueden utilizar tiempos de adquisición largos en la dimensión indirecta para mejorar la resolución espectral y aliviar la limitación de dispersión por desplazamiento químico inherente a las IDP.
PRE es una herramienta útil para estudiar las interacciones proteína-proteína y proteína-ácido nucleico, en particular las interacciones que son transitorias o poco pobladas. Se proporciona un protocolo detallado para la preparación de una muestra de RMN adecuada para medir los PRE, incluidos los pasos para la purificación de proteínas, el etiquetado de espín dirigido al sitio, la configuración y calibración del programa de pulsos, el procesamiento y la interpretación de los datos de RMN. A lo largo de todo el estudio se señalan importantes consideraciones experimentales que pueden afectar a la calidad de los datos y al resultado experimental, como la concentración de la muestra, la selección de la etiqueta de espín y la eliminación de los componentes paramagnéticos.
Protocolo
Requisitos generales para el protocolo: instalaciones de purificación de proteínas, espectrómetro UV-Vis, espectrómetro de RMN de alto campo y software operativo, software de análisis de posprocesamiento que incluye; NMRPipe32, Sparky33, (o CCPN Analysis 34, o NMRViewJ35).
1. Expresión recombinante y purificación de una proteína para mediciones de PRE
- Diseñe una construcción de expresión para la proteína de interés de modo que haya un solo residuo de cisteína presente. Se requerirán múltiples mutaciones para introducir una cisteína libre en diferentes posiciones de la proteína de interés36.
- Extraer y purificar una muestra de abundancia natural (14N) o marcada con 15N de la proteína de interés utilizando un protocolo establecido37.
NOTA: Los sistemas de expresión de E. coli proporcionan un método rentable y robusto para la expresión de proteínas recombinantes, ya que el enriquecimiento isotópico de 15N es un requisito mínimo para la espectroscopia de RMN heteronuclear biomolecular. Los pasos típicos son la expresión en medios mínimos, la purificación cromatográfica y la eliminación de la etiqueta de purificación de afinidad. Este protocolo asume que se ha establecido un protocolo robusto de expresión y purificación que puede producir suficiente proteína de calidad adecuada para las investigaciones de RMN.- Mantener 1 mM de agente reductor (DTT o TCEP) en tampones en todos los pasos de purificación para evitar la reacción de la cisteína libre y la formación de enlaces disulfuro intermoleculares para los IDP.
NOTA: Algunos sistemas pueden ser más tolerantes y menos propensos a la agregación a condiciones no reductoras dependiendo de las características específicas de la proteína, así como de la temperatura, el pH y el sistema tampón elegido para la purificación38. - Retire las etiquetas de afinidad utilizadas para la purificación antes de continuar, ya que pueden interactuar de forma no específica con la proteína de forma impredecible o posiblemente contener residuos de cisteína reactiva que podrían servir inadvertidamente como un sitio de unión no intencionado.
- Preparar una muestra de referencia marcada con 15N sin mutación(es) de cisteína mezclada con una versión soluble de la etiqueta de centrifugado para evaluar la contribución de los PREs del disolvente.
- Mantener 1 mM de agente reductor (DTT o TCEP) en tampones en todos los pasos de purificación para evitar la reacción de la cisteína libre y la formación de enlaces disulfuro intermoleculares para los IDP.
2. Conjugación de la etiqueta de centrifugación de nitrógeno 3-Maleimido-PROXYL
- Almacenar o intercambiar la proteína purificada en un tampón desgasificado que contenga 50 mM de Tris pH 7 y 1 mM de TCEP; el tampón también puede contener hasta 8 M de urea si es necesario para ayudar a la solubilidad de las proteínas.
Alternativamente, diluya rápidamente una solución madre de proteína en al menos 10 equivalentes de volumen de tampón Tris pH 7 desgasificado de 50 mM y TCEP de 1 mM. Asegúrese de que la concentración de proteínas antes de añadir spin-label sea de al menos 100 μM. - Añadir 3-Maleimido proxyl de una solución madre a 20 veces el exceso molar de la proteína de interés. Proteger la muestra de la luz y del oxígeno e incubar durante la noche a temperatura ambiente o 4 °C; El balanceo suave o la nutación pueden mejorar la eficiencia del etiquetado.
- Prepare las soluciones madre de la etiqueta giratoria disolviendo el polvo de proxilo 3-Maleimido en etanol al 95%. Las alícuotas del stock pueden almacenarse a -80 °C durante menos de 6 meses.
- Paso crítico: Retire la etiqueta de giro libre sin reaccionar para evitar la presencia de PRE de disolventes no específicos. Consiga esto mediante filtración en gel o (preferiblemente) diálisis extensa de la muestra de proteína. Este paso también introducirá la proteína en un tampón adecuado para RMN.
NOTA: Los agentes reductores deben prepararse frescos y se debe considerar la compatibilidad entre los componentes tampón; por ejemplo, el TCEP se degrada rápidamente en tampones a base de fosfato, y esta combinación debe evitarse39. - Trate todos los tampones utilizados a partir de este paso con una resina quelante selectiva para metales divalentes y de transición para eliminar iones paramagnéticos o extintores de etiquetas de centrifugado. Si la proteína no se puede almacenar en un tampón de RMN, concéntrela para diluirla rápidamente en un tampón adecuado para RMN.
- Supervisión de la eficiencia de la incorporación de etiquetas giratorias.
- Utilice el reactivo de Ellman (ácido 5,5-ditio-bis-(2-nitrobenzoico) para cuantificar los grupos sulfhidrilo libres en la solución40.
NOTA: Los protocolos detallados están disponibles en el fabricante. Para los propósitos que aquí se presentan, es importante determinar la incorporación de la etiqueta de espín, la concentración de grupos sulfhidrilo libres se compara con la concentración total de proteína. El porcentaje de grupos sulfhidrilo libres es el porcentaje de moléculas que no tienen una etiqueta de espín de nitrógeno adherida. - Monitorizar la intensidad del pico correspondiente al residuo de cisteína marcado para juzgar la incorporación de la etiqueta de espín en la proteína de interés.
NOTA: Este es un enfoque rápido y efectivo para determinar el grado de marcaje de espín de la proteína. La incorporación completa de la etiqueta de espín dará como resultado la desaparición del pico del espectro. Dada la escasa dispersión característica de los IDP, el pico correspondiente al residuo de cisteína mutante no siempre puede identificarse fácilmente, por lo que se recomienda el uso del reactivo de Ellman (paso 2.6.1).
- Utilice el reactivo de Ellman (ácido 5,5-ditio-bis-(2-nitrobenzoico) para cuantificar los grupos sulfhidrilo libres en la solución40.
3. Preparar la muestra de RMN para medir el PRE intra o intermolecular
- Preparación de la muestra para la medición de PRE intramolecular
- Preparar 15proteínas enriquecidas isotópicamente y marcadas con espín hasta una concentración de al menos 100 μM pero no más de 300 μM en un tampón adecuado para RMN. El volumen total de la muestra (incluyendoD2O) es de 500 - 550 μL.
NOTA: Los tampones comunes de RMN incluyen fosfato, acetato, (bi)carbonato y TRIS. Los búferes de Good, como MES, HEPES, también pueden ser apropiados. Tenga cuidado al seleccionar búferes para asegurarse de que no haya reactividad cruzada con otros componentes de la solución. - Asegúrese de que el pH sea ~7.2 o inferior para minimizar los efectos del intercambio de protones amida con el agua. Mantenga la concentración de sal lo más baja posible (normalmente menos de 150 mM), aunque la consideración principal es mantener la estabilidad de la proteína.
NOTA: Los enfoques para llevar a cabo experimentos de RMN en condiciones de alta sal se han descrito en otro lugar41.
- Preparar 15proteínas enriquecidas isotópicamente y marcadas con espín hasta una concentración de al menos 100 μM pero no más de 300 μM en un tampón adecuado para RMN. El volumen total de la muestra (incluyendoD2O) es de 500 - 550 μL.
- Preparar la muestra para la medición de PRE intermolecular
- Siga este paso o el paso 3.1; no se realizan simultáneamente. Prepare 14N de proteína marcada con espín de abundancia natural en el tampón de RMN elegido.
- Preparar la muestra de proteína mezclando 15N de proteína enriquecida isotópicamente sin espín con 1%-50% de proteína marcada con espín de 14N de abundancia natural, de modo que la concentración final sea idéntica a la muestra preparada en el punto 3.1.1. El volumen total de la muestra (incluyendoD2O) es de 500 - 550 μL.
- Optimizar empíricamente la proporción de las proteínas 15N y 14N para cada proteína estudiada. Las proporciones del 1%, 5% y 20% de proteína marcada con 14N-spin son buenos puntos de partida.
NOTA: Una acumulación de PRE en función de la adición de proteína marcada con 14N-spin indica un efecto específico; el PRE observado es específico de la muestra, ya que depende de la distancia y la población (como se discutió anteriormente) y, por lo tanto, se requerirán proporciones más altas de proteína marcada con 14N-spin si la interacción es particularmente transitoria17.
- Transfiera la muestra de RMN (ya sea intra o intermolecular) a un tubo de RMN de 5 mm que sea apropiado para su uso en imanes de alto campo utilizando una pipeta de vidrio o micropipeta de vástago largo (9"). Asegúrese de que todas las muestras de RMN incluyan entre el 5 % y el 10 % de D2O para facilitar el bloqueo de campo.
NOTA: Los tubos de RMN que utilizan tapones de polímero para reducir el volumen de muestra necesario no se recomiendan para las mediciones de PRE debido a las dificultades relacionadas con la calzada efectiva de la muestra.
4. Configure el espectrómetro de RMN y experimente los parámetros específicos
- Tenga mucho cuidado cuando trabaje cerca de espectrómetros de RMN superconductores de alto campo.
NOTA: Los peligros incluyen lesiones debidas a la aceleración repentina de objetos metálicos hacia el imán, interferencia con dispositivos médicos implantados y asfixia debido a una liberación repentina de gas N 2 y He2 en caso de enfriamiento del imán. Los siguientes pasos asumen que el lector ha recibido la capacitación requerida, es consciente de estos y otros peligros locales y ha recibido la aprobación del gerente de la instalación para operar el espectrómetro de RMN. En caso de duda sobre un paso o instrucción, consulte con el administrador de la instalación o el usuario experimentado para evitar posibles lesiones personales o daños al espectrómetro. - En los siguientes pasos se supone un espectrómetro de RMN comercial que ejecuta una versión moderna del software de control de adquisición. Descargue el programa de pulso y los archivos de parámetros y colóquelos en los directorios apropiados.
NOTA: Un programa de pulsos y un conjunto de parámetros adecuados para su uso con un espectrómetro Bruker y TopSpin (3.2 o posterior) están disponibles a petición de los autores.- Paso crítico: Se asume que está familiarizado con la instalación de programas de impulsos de RMN no nativos; Consulte con el gerente de la instalación o con un usuario experimentado si es necesario.
- Coloque la muestra en el imán, bloquee la señal de 2H con el comando Bloquear, sintonice y haga coincidir el canal de 1 H de acuerdo con los protocolos de la instalación (el procedimiento exacto dependerá de si la sonda está equipada con un módulo desintonización y coincidencia remota).
- Ajuste las cuñas utilizando la subrutina topshim para optimizar la supresión de la señal de disolvente.
- Calibrar los pulsos de 1H y 15N a 90° utilizando métodos estándar.
- Calibrar el pulso de 1H usando el programa popt (use pulsecal primero para estimar la longitud del pulso).
- Calibrar el pulso de 15N contra una muestra estándar; Asegúrese de que este valor se haya calibrado recientemente consultándolo con un director técnico o un usuario experimentado.
- Alternativamente, calibre el pulso de 15N en la muestra variando uno de los pulsos de 90° de un experimento HMQC hasta que se logre una señal nula.
- Determine la atenuación correcta para pulsos con forma utilizando la subrutina de la herramienta de forma (stdisp).
- Abra el archivo de forma de pulso apropiado haciendo clic en el icono de la carpeta. Los pulsos con forma se encuentran en la sección de parámetros de pulso de ACQUPARS.
- Cargue el archivo de definición de pulso y haga clic en Analizar forma de onda > Integrar forma. Introduzca el pulso duro calibrado de 1H 90°, la longitud del pulso con la forma deseada y la rotación (90° o 180°).
- Calcule el nivel de potencia del pulso con forma sumando el cambio de nivel de potencia a la atenuación del pulso calibrado de 90°.
- Registre un HSQC estándar de 1H, 15N (hsqcetfpf3gpsi) para optimizar el ancho de barrido, la frecuencia portadora y verificar la supresión del agua25.
- Ajuste la anchura de barrido y el número de incrementos de cota indirectos mediante los comandos sw y td o directamente en los cuadros de diálogo correspondientes. Normalmente, para recoger los PRE, se eligen anchos espectrales de modo que el espectro no se pliegue.
5. Configurar el experimento 1H N-T2
- Calibrar los impulsos conformados como se ha descrito anteriormente (4.4.5-4.5.7). Los archivos de parámetros de pulso con forma para el experimento PRE son Eburp2.1000 (pulso de 90°), Reburp.1000 e Iburp2.1000. Introduzca las longitudes de pulso calibradas en la sección de parámetros de pulso de la pestaña ACQUPARS .
- Este experimento mide el 1H N-T2 utilizando el enfoque de dos puntos de retardo de tiempo30.
- Establezca los retardos de tiempo editando el archivo vdlist, el primer retardo (Ta) se establece en 0,01 ms.
- Elija el segundo retardo, (T b) utilizando la relación con el máximo esperado PRE (T b ~ 1.15/(R 2,dia + Γ 2) donde R 2,dia representa el R 2 de la muestra diamagnética13. Sin conocimiento previo de la magnitud de la contribución del PRE a la relajación observada, un buen punto de partida es establecer T b en ~1x 1H T2.
- A continuación, determine un valor adecuado comparando los primeros incrementos (procesados con el comando efp) de los espectros T a y T b y ajustando T b de modo que la señal decaiga entre el 40% y el 50% de su valor inicial.
NOTA: Este enfoque optimiza la relación señal/ruido espectral, una consideración necesaria para las muestras que no pueden estar altamente concentradas (< 50 μM). Los valores adecuados de Tb dependen de la muestra, pero suelen oscilar entre 8 y 40 ms para una proteína de tamaño medio.
- Determine el número de puntos complejos que se van a registrar y el número de escaneos para obtener un promedio de señal suficiente. Dado que las IDP tienen 15N T2 más largas que las proteínas plegadas de tamaño comparable, se pueden utilizar tiempos de adquisición más largos en la dimensión indirecta.
NOTA: Este valor depende de las características específicas de la proteína, pero puede estimarse aproximadamente a partir de la 15 N T2 y optimizarse mediante el monitoreo de la decaimiento de la señal en el FID. Para la dimensión directa, 1024* puntos complejos (13 ppm de ancho de barrido, 112,6 ms de tiempo de adquisición) es suficiente para la mayoría de las muestras. - Utilice el comando expt para calcular el tiempo del experimento y, a continuación, inicie el experimento con el comando zg.
6. Hacer una muestra diamagnética reduciendo la etiqueta giratoria con ácido ascórbico
- Disuelva el ascorbato de sodio en el tampón de RMN y ajuste el pH para que coincida con el tampón de RMN original.
- Calcule la concentración de stock de ascorbato de sodio de modo que se pueda agregar un exceso molar de 10 veces el de ascorbato sobre la concentración de la etiqueta giratoria con el menor cambio en el volumen de la muestra. Por ejemplo, para una muestra de proteína de 100 μM, es apropiado un stock de 100 mM de ascorbato. La reducción de la etiqueta de centrifugado requerirá la adición de 5,5 μl de solución madre de ácido ascórbico, que es solo el 1 % del volumen total de la muestra.
- Agregue la cantidad requerida de ácido ascórbico al tubo de RMN colocando una gota debajo del borde del tubo, tape el tubo, invierta con cuidado el tubo para mezclar y luego gire a 200-400 x g durante 10-20 s en una centrífuga manual para asentar la muestra en el fondo del tubo.
- Envuelva el tubo de RMN en papel de aluminio para protegerlo de la luz y deje que la reacción continúe durante al menos 3 h.
- Registre 1HN-T 2 en la muestra diamagnética utilizando los mismos parámetros utilizados para la muestra paramagnética.
- Recalibrar los pulsos. Sin embargo, no deberían haber cambiado con respecto a las mediciones paramagnéticas; Si son significativamente diferentes (> diferencia de 0,5 μs), considere la calidad de la muestra (p. ej., degradación, precipitación).
- Asegúrese de que todos los parámetros de adquisición, incluidos los retardos de relajación especificados (vdlist), el número de escaneos ficticios, el número de escaneos recopilados, el número de puntos complejos recopilados, el tiempo de adquisición, los anchos de barrido y las frecuencias portadoras permanezcan iguales para las muestras diamagnéticas y paramagnéticas.
7. Procesar espectros paramagnéticos y diamagnéticos
- Copie los datos en el equipo local o en la estación de trabajo que tenga instalados y configurados NMRPipe y Sparky. Cree una carpeta denominada proc en el directorio de datos del experimento que contenga el archivo ser.
- Copie los scripts NMRPipe fid.com, p3d.com y nmrproc.com en proc (los scripts de procesamiento están disponibles a petición de los autores).
- Utilice el script fid.com para convertir el formato de datos Bruker (ser) en formato NMRPipe.
- Utilice el script p3D.com para dividir los planos pseudo3D en espectros individuales.
- Utilice el script nmrproc.com para leer la salida del script fid.com, aplicar supresión de disolvente, una función de ventana, añadir ceros a los datos sin procesar (relleno de cero), aplicar corrección de fase, ejecutar una transformación de Fourier, recortar los datos para su visualización y escribir los datos procesados en el disco. El script generará un archivo por cada retardo de relajación grabado (T a y Tb).
NOTA: Cada uno de estos scripts se puede personalizar para optimizar el procesamiento de los detalles específicos de cada experimento. Se incluyen tutoriales y conjuntos de datos de ejemplo en la distribución NMRPipe disponible en el sitio web de NMRPipe32. NMRDraw se puede utilizar para la visualización espectral durante el procesamiento (por ejemplo, establecer ángulos de fase adecuados, etc.). Las opciones disponibles para los comandos NMRPipe se pueden ver mediante el comando nmrPipe -help.
8. Transfiera asignaciones de resonancia y extraiga alturas de pico
- Cambie la información del encabezado del archivo para cada archivo de espectro (T a, Tb para muestras paramagnéticas y diamagnéticas) usando el comando sethdr [nombre de archivo] -ndim 2.
- Utilice Sparky para extraer alturas de pico33 siguiendo los pasos 8.3-8.5. Otros paquetes de software, incluidos NMRPipe (NMRDraw)32, CCPN Analysis34 y NMRViewJ35 también son apropiados.
- Lee los archivos espectrales en Sparky. En este paso, el conjunto de datos consistirá en un espectro para cada espectro de punto de tiempo (T a, Tb), tanto para las muestras paramagnéticas como diamagnéticas, medidas para cada posición de la etiqueta de espín en la proteína.
- Utilice Sparky para seleccionar picos (comando: F8 y, a continuación, haga clic y arrastre) y transfiera asignaciones con la herramienta de transferencia de lista de picos desde una lista de picos de referencia.
NOTA: Las asignaciones de resonancia de la proteína de interés son necesarias para la interpretación específica de la secuencia de los PRE observados36.- Establezca los contornos de los espectros paramagnéticos y diamagnéticos al mismo nivel. Asegúrese de establecer los contornos de modo que los espectros recogidos después del retardo de tiempo no excluyan deliberadamente los picos, sino que sean lo suficientemente altos como para que los espectros Ta no sean demasiado ruidosos.
- Guarde las nuevas listas de picos para cada espectro e incluya la intensidad máxima medida y la relación señal/ruido calculada por Sparky (comando: lt para abrir la lista de picos, haga clic en las opciones para incluir las columnas de intensidad y SNR, comando: guardar).
9. Extraiga 1H N-T2 dosis para cada residuo y calcule PRE
- Importe las listas de picos en un software de hoja de cálculo o en un lenguaje de programación preferido, como Python.
NOTA: Para cada posición de la etiqueta de espín en la proteína, el conjunto de datos consistirá en cuatro listas de picos con intensidades de pico asociadas, una de Ta y otra de Tb para los experimentos paramagnéticos y diamagnéticos. - Calcule 1HN R2 para las muestras paramagnéticas y diamagnéticas utilizando la ecuación:
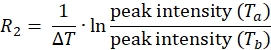
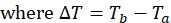
- Utilice la ecuación anterior para determinar la tasa de relajación de cada residuo para las muestras paramagnéticas y diamagnéticas.
- Determine la tasa 1H N-Γ2 para cada residuo utilizando la ecuación:
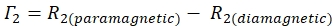
- Utilice la relación señal/ruido (SNR) calculada por Sparky para calcular la incertidumbre de la altura máxima de cada residuo.
- Propague el error usando la ecuación:

- Gráfica 1HN-Γ 2 en función del número de residuos utilizando un diagrama de dispersión que incluya el error calculado en 9.6.
Resultados
Se registraron PREs intramoleculares 1HN-Γ 2 en un fragmento autoasociado, intrínsecamente desordenado (residuos 171-264) derivado del dominio de baja complejidad de la proteína de unión al ARN EWSR142 (Figura 3). Se espera que los residuos en estrecha proximidad secuencial al punto de fijación de la etiqueta giratoria (por ejemplo, el residuo 178 o 260 en la Figura 3) se amplíen significati...
Discusión
Se ha presentado un método para caracterizar las interacciones transitorias que existen en poblaciones bajas entre proteínas intrínsecamente desordenadas y varios socios de unión utilizando PRE. En el ejemplo mostrado, la proteína se asocia a sí misma y, por lo tanto, el PRE puede surgir de una combinación de interacciones intermoleculares e intramoleculares. Este método se extiende fácilmente a muestras heterogéneas en las que se pueden caracterizar las interacciones entre dos proteínas diferentes. La informa...
Divulgaciones
Todos los autores han leído y aprobado el manuscrito. No se declaran conflictos de intereses.
Agradecimientos
Agradecemos a las Dras. Jinfa Ying y Kristin Cano por sus útiles discusiones y asistencia técnica. DSL es un St. Baldrick's Scholar y agradece el apoyo de la Fundación St. Baldrick's (634706). Este trabajo fue apoyado en parte por la Fundación Welch (AQ-2001-20190330) para DSL, el Fondo Max y Minnie Tomerlin Voelcker (Beca para Jóvenes Investigadores de la Fundación Voelcker para DSL), los Fondos de Puesta en Marcha de UTHSA para DSL y una Beca de Posgrado Greehey en Salud Infantil para CNJ. Este trabajo se basa en la investigación realizada en las Instalaciones Centrales de Biología Estructural, parte de los Núcleos de Investigación Institucional en el Centro de Ciencias de la Salud de la Universidad de Texas en San Antonio, con el apoyo de la Oficina del Vicepresidente de Investigación y el Recurso Compartido de Descubrimiento de Fármacos y Biología Estructural del Centro Oncológico Mays (NIH P30 CA054174).
Materiales
| Name | Company | Catalog Number | Comments |
| 0.45 µm and 0.22 µm syringe filters | Millipore Sigma | SLHVM33RS SLGVR33RS | Filter lysate before first purification step and before size exclusion chromatography. |
| 100 mm Petri Dish | Fisher | FB0875713 | Agar plates for bacterial transformation. |
| 14N Ammonium chloride | Sigma Aldrich | 576794 | Use of 15N in M9 medium will produce an NMR visible protein, 14N will produces an NMR invisible protein |
| 15N Ammonium chloride | Sigma Aldrich | 299251 | Use of 15N in M9 medium will produce an NMR visible protein, 14N will produces an NMR invisible protein |
| 3 L Fernbach baffled flask | Corning | 431523 | Bacterial expression culture |
| 3-Maleimido-Proxyl | Sigma Aldrich | 253375 | Nitroxide spin label |
| 50 mL conical centrifuge tubes | Thermo Fisher | 14-432-22 | Solution/protein storage |
| Amicon centrifugal filter | Millipore Sigma | UFC900308 | Protein concentration |
| Ampicillan | Sigma Aldrich | A5354 | Antibiotic for a selective marker, exact choice depends on the expression construct plasmid |
| Analytical balance | Oahus | 30061978 | Explorer Pro, for weighing reagents |
| Ascorbic acid | Sigma Aldrich | AX1775 | Reduces nitroxide spin label |
| Autoclave | Sterilize glassware and culture media | ||
| Calcium chloride | Sigma Aldrich | C4901 | M9 media component |
| Centrifuge bottles | Thermo Fisher | 010-1459 | Harvest E. coli cells after recombinant protein expression |
| Centrifuge, hand-crank | Thomas Scientific | 0241C68 | Boekel hand-driven, low-speed centrifuge with 15 mL buckets that can accommodate NMR tubes |
| Chelex 100 | Sigma Aldrich | C7901 | Remove contaminating paramagnetic compounds from buffer solutions |
| Computer workstation | Linux or Mac OS compatable with NMR data processing and analysis software packages such as NMRPipe and Sparky | ||
| Deuterium oxide | Sigma Aldrich | 151882 | Needed for NMR lock signal |
| Dextrose | Sigma Aldrich | D9434 | M9 media component |
| Dibasic Sodium Phosphate | Sigma Aldrich | S5136 | M9 media component |
| Ellman's reagent (5,5-dithio-bis-(2-nitrobenzoic acid) | Thermo Fisher | 22582 | Quantification of free cystiene residues |
| High speed centrifuge tubes | Thermo Fisher | 3114-0050 | Used to clear bacterial lysate. |
| High-field NMR instrument (600 - 800 MHz) | Bruker | Equiped with a multichannel cryogenic probe and temperature control | |
| IMAC column, HisTrap FF | Cytvia | 17528601 | Initial fractionation of crude bacterial lysate |
| Isopropyl B-D-thiogalactoside (IPTG) | Sigma Aldrich | I6758 | Induces protein expression for genes under control of lac operator |
| LB agar | Thermo Fisher | 22700025 | Items are used for transforming E. coli to express protein of interest, substitions for any of these items with like products is acceptable. |
| LB broth | Thermo Fisher | 12780052 | |
| Low-pressure chromatography system | Bio-Rad | 7318300 | BioRad BioLogic is used for low-pressure chomatograph such as running IMAC columns |
| Magnesium sulfate | Sigma Aldrich | M7506 | M9 media component |
| Medium pressure chromatography system | Bio-Rad | 7880007 | BioRad NGC equipped with a multi-wavelength detector, pH and conductivity monitors, and automatic fraction collector |
| MEM vitamin solution | Sigma Aldrich | M6895 | M9 media component |
| Microfluidizer | Avestin | EmulsiFlex-C3 | Provides rapid and efficient bacterial cell lysis |
| Micropipettes | Thermo Fisher | Calibrated set of micropippetters with properly fitting disposable tips (available from multiple manufacturers e.g. Eppendorf) | |
| Monobasic potassium phosphate | Sigma Aldrich | 1551139 | M9 media component |
| NMR pipettes | Sigma Aldrich | 255688 | To remove sample from NMR tube |
| NMR sample tube | NewEra | NE-SL5 | Suitable for high-field NMR spectrometers |
| Preparative Centrifuge | Beckman Coulter | Avanti J-HC | Harvest E. coli cells after recombinant protein expression |
| Round bottom polystyrene centrifuge tubes | Corning | 352057 | Clear bacterial lysate |
| Shaking incubator | Eppendorf | S44I200005 | Temperature controlled growth of E. coli starter and expression cultures |
| Sodium chloride | Sigma Aldrich | S5886 | M9 media component |
| Sonicating water bath and vacuum source | Thomas Scientific | Used to degas buffer solutions | |
| Sonicator | Thermo Fisher | FB505110 | Used for bacterial cell lysis or shearing bacterial DNA |
| Spectrophotometer | Implen | OD600 Diluphotometer | Monitor growth of E.coli protein expression cultures |
| Superdex 200 16/600 size exculsion colum | Cytvia | 28989333 | Final protein purification step |
| Topspin software, version 3.2 or later | Bruker | Operating software for the NMR instrument | |
| Transformation competent E. coli cells | Thermo Fisher | C600003 | One Shot BL21 Star (DE3) chemically competent E. coli, other strains may be compatable |
| Tris(2-carboxyethyl)phosphine (TCEP) | ThermoFisher | 20490 | Reducing agent compatable with some sulfhydryl-reactive conjugations |
| UV-Vis spectrophotometer | Implen | NP80 | Measure protein concentration. |
| Water bath, temperature controlled | ThermoFisher | FSGPD25 | For heat shock step of bacterial transformation |
| Yeast extract | Sigma Aldrich | Y1625 | For supplementing M9 media if required |
Referencias
- Dyson, H. J., Wright, P. E. Intrinsically unstructured proteins and their functions. Nature Reviews: Molecular Cell Biology. 6 (3), 197-208 (2005).
- Korneta, I., Bujnicki, J. M. Intrinsic disorder in the human spliceosomal proteome. PLoS Computational Biology. 8 (8), 1002641 (2012).
- Frege, T., Uversky, V. N. Intrinsically disordered proteins in the nucleus of human cells. Biochemistry and Biophysics Reports. 1, 33-51 (2015).
- Liu, J., et al. Intrinsic disorder in transcription factors. Biochemistry. 45 (22), 6873-6888 (2006).
- El Hadidy, N., Uversky, V. N. Intrinsic disorder of the BAF complex: Roles in chromatin remodeling and disease development. International Journal of Molecular Sciences. 20 (21), (2019).
- Wright, P. E., Dyson, H. J. Intrinsically disordered proteins in cellular signalling and regulation. Nature Reviews: Molecular Cell Biology. 16 (1), 18-29 (2015).
- Brangwynne, C. P. Phase transitions and size scaling of membrane-less organelles. Journal of Cell Biology. 203 (6), 875-881 (2013).
- Shin, Y., Brangwynne, C. P. Liquid phase condensation in cell physiology and disease. Science. 357 (6357), (2017).
- Cavanagh, J. . Protein NMR spectroscopy : principles and practice. 1st edition. , (2018).
- Sekhar, A., Kay, L. E. NMR paves the way for atomic level descriptions of sparsely populated, transiently formed biomolecular conformers. Proceedings of the National Academy of Sciences of the United States of America. 110 (32), 12867-12874 (2013).
- Anthis, N. J., Clore, G. M. Visualizing transient dark states by NMR spectroscopy. Quarterly Reviews of Biophysics. 48 (1), 35-116 (2015).
- Alderson, T. R., Kay, L. E. NMR spectroscopy captures the essential role of dynamics in regulating biomolecular function. Cell. 184 (3), 577-595 (2021).
- Clore, G. M., Iwahara, J. Theory, practice, and applications of paramagnetic relaxation enhancement for the characterization of transient low-population states of biological macromolecules and their complexes. Chemical Reviews. 109 (9), 4108-4139 (2009).
- Wu, K. P., Baum, J. Detection of transient interchain interactions in the intrinsically disordered protein alpha-synuclein by NMR paramagnetic relaxation enhancement. Journal of the American Chemical Society. 132 (16), 5546-5547 (2010).
- Janowska, M. K., Wu, K. P., Baum, J. Unveiling transient protein-protein interactions that modulate inhibition of alpha-synuclein aggregation by beta-synuclein, a pre-synaptic protein that co-localizes with alpha-synuclein. Scientific Reports. 5, 15164 (2015).
- Murthy, A. C., et al. Molecular interactions underlying liquid-liquid phase separation of the FUS low-complexity domain. Nature Structural & Molecular Biology. 26 (7), 637-648 (2019).
- Fawzi, N. L., Doucleff, M., Suh, J. Y., Clore, G. M. Mechanistic details of a protein-protein association pathway revealed by paramagnetic relaxation enhancement titration measurements. Proceedings of the National Academy of Sciences of the United States of America. 107 (4), 1379-1384 (2010).
- Griffith, O. H., Waggoner, A. S. Nitroxide free radicals: spin labels for probing biomolecular structure. Accounts of Chemical Research. 2 (2), 17-24 (1969).
- Bertini, I., Luchinat, C., Parigi, G., Ravera, E. . NMR of Paramagnetic Macromolecules, Applications to Metallobiomolecules and Models. 2 edn. , (2016).
- Bloembergen, N., Purcell, E. M., Pound, R. V. Relaxation effects in nuclear magnetic resonance absorption. Physical Review. 73 (7), 679-712 (1948).
- Solomon, I. Relaxation processes in a system of two spins. Physical Review. 99 (2), 559 (1955).
- Clore, G. M. Practical aspects of paramagnetic relaxation enhancement in biological macromolecules. Methods in Enzymology. 564, 485-497 (2015).
- Klare, J. P. Site-directed spin labeling EPR spectroscopy in protein research. Biological Chemistry. 394 (10), 1281-1300 (2013).
- Clore, G. M., Tang, C., Iwahara, J. Elucidating transient macromolecular interactions using paramagnetic relaxation enhancement. Current Opinion in Structural Biology. 17 (5), 603-616 (2007).
- Melanson, M., Sood, A., Torok, F., Torok, M. Introduction to spin label electron paramagnetic resonance spectroscopy of proteins. Biochemistry and Molecular Biology Education. 41 (3), 156-162 (2013).
- Czogalla, A., Pieciul, A., Jezierski, A., Sikorski, A. F. Attaching a spin to a protein -- site-directed spin labeling in structural biology. Acta Biochimica Polonica. 54 (2), 235-244 (2007).
- Lindfors, H. E., de Koning, P. E., Drijfhout, J. W., Venezia, B., Ubbink, M. Mobility of TOAC spin-labelled peptides binding to the Src SH3 domain studied by paramagnetic NMR. Journal of Biomolecular NMR. 41 (3), 157-167 (2008).
- Fawzi, N. L., et al. A rigid disulfide-linked nitroxide side chain simplifies the quantitative analysis of PRE data. Journal of Biomolecular NMR. 51 (1-2), 105-114 (2011).
- Bleicken, S., et al. gem-Diethyl pyrroline nitroxide spin labels: Synthesis, EPR characterization, rotamer libraries and biocompatibility. ChemistryOpen. 8 (8), 1035 (2019).
- Iwahara, J., Tang, C., Clore, G. M. Practical aspects of 1H transverse paramagnetic relaxation enhancement measurements on macromolecules. Journal of Magnetic Resonance. 184, 185-195 (2007).
- Venditti, V., Fawzi, N. L. Probing the atomic structure of transient protein contacts by paramagnetic relaxation enhancement solution NMR. Methods in Molecular Biology. 1688, 243-255 (2018).
- Delaglio, F., et al. NMRPipe: a multidimensional spectral processing system based on UNIX pipes. Journal of Biomolecular NMR. 6 (3), 277-293 (1995).
- Lee, W., Tonelli, M., Markley, J. L. NMRFAM-SPARKY: enhanced software for biomolecular NMR spectroscopy. Bioinformatics. 31 (8), 1325-1327 (2015).
- Vranken, W. F., et al. The CCPN data model for NMR spectroscopy: development of a software pipeline. Proteins. 59 (4), 687-696 (2005).
- Johnson, B. A. Using NMRView to visualize and analyze the NMR spectra of macromolecules. Methods in Molecular Biology. 278, 313-352 (2004).
- Sjodt, M., Clubb, R. T. Nitroxide labeling of proteins and the determination of paramagnetic relaxation derived distance restraints for NMR studies. Bio-Protocol. 7 (7), (2017).
- Zhang, H., van Ingen, H. Isotope-labeling strategies for solution NMR studies of macromolecular assemblies. Current Opinion in Structural Biology. 38, 75-82 (2016).
- Rabdano, S. O., et al. Onset of disorder and protein aggregation due to oxidation-induced intermolecular disulfide bonds: case study of RRM2 domain from TDP-43. Scientific Reports. 7 (1), 11161 (2017).
- Burns, J. A., Butler, J. C., Moran, J., Whitesides, G. M. Selective reduction of disulfides by tris(2-carboxyethyl)phosphine. Journal of Organic Chemistry. 56 (8), 2648-2650 (1991).
- Ellman, G. L. Tissue sulfhydryl groups. Archives of Biochemistry and Biophysics. 82 (1), 70-77 (1959).
- Binbuga, B., Boroujerdi, A. F., Young, J. K. Structure in an extreme environment: NMR at high salt. Protein Science. 16 (8), 1783-1787 (2007).
- Schwartz, J. C., Cech, T. R., Parker, R. R. Biochemical properties and biological functions of FET proteins. Annual Review of Biochemistry. 84, 355-379 (2015).
- Nabuurs, S. M., de Kort, B. J., Westphal, A. H., van Mierlo, C. P. Non-native hydrophobic interactions detected in unfolded apoflavodoxin by paramagnetic relaxation enhancement. European Biophysics Journal. 39 (4), 689-698 (2010).
- Wiedemann, C., Kumar, A., Lang, A., Ohlenschlager, O. Cysteines and disulfide bonds as structure-forming units: Insights from different domains of life and the potential for characterization by NMR. Frontiers in Chemistry. 8, 280 (2020).
- Wommack, A. J., et al. NMR solution structure and condition-dependent oligomerization of the antimicrobial peptide human defensin 5. Biochemistry. 51 (48), 9624-9637 (2012).
- Taylor, A. M., et al. Detailed characterization of cysteine-less P-glycoprotein reveals subtle pharmacological differences in function from wild-type protein. British Journal of Pharmacology. 134 (8), 1609-1618 (2001).
- Hu, K., Doucleff, M., Clore, G. M. Using multiple quantum coherence to increase the 15N resolution in a three-dimensional TROSY HNCO experiment for accurate PRE and RDC measurements. Journal of Magnetic Resonance. 200 (2), 173-177 (2009).
- Anthis, N. J., Doucleff, M., Clore, G. M. Transient, sparsely populated compact states of apo and calcium-loaded calmodulin probed by paramagnetic relaxation enhancement: interplay of conformational selection and induced fit. Journal of the American Chemical Society. 133 (46), 18966-18974 (2011).
- Battiste, J. L., Wagner, G. Utilization of site-directed spin labeling and high-resolution heteronuclear nuclear magnetic resonance for global fold determination of large proteins with limited nuclear overhauser effect data. Biochemistry. 39 (18), 5355-5365 (2000).
- Donaldson, L. W., et al. Structural characterization of proteins with an attached ATCUN motif by paramagnetic relaxation enhancement NMR spectroscopy. Journal of the American Chemical Society. 123 (40), 9843-9847 (2001).
- Gaponenko, V., et al. Protein global fold determination using site-directed spin and isotope labeling. Protein Science. 9 (2), 302-309 (2000).
- Trindade, I. B., Invernici, M., Cantini, F., Louro, R. O., Piccioli, M. PRE-driven protein NMR structures: an alternative approach in highly paramagnetic systems. FEBS Journal. 288 (9), 3010-3023 (2021).
- Nitsche, C., Otting, G. Pseudocontact shifts in biomolecular NMR using paramagnetic metal tags. Progress in Nuclear Magnetic Resonance Spectroscopy. 98-99, 20-49 (2017).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados