JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
Paramagnetic Relaxation Enhancement for Detecting and Characterizing Self-Associations of Intrinsically Disordered Proteins(본질적으로 무질서한 단백질의 자기 연관성을 검출하고 특성화하기 위한 상자성 이완 향상)
요약
본질적으로 무질서한 단백질에서 약하고 일시적인 분자 간 및 분자 내 상호 작용을 감지하기 위한 상자성 이완 강화 NMR 분광법의 적용을 위한 프로토콜이 제시됩니다.
초록
본질적으로 무질서한 단백질과 단백질 내의 본질적으로 무질서한 영역은 인간 프로테옴의 크고 기능적으로 중요한 부분을 구성합니다. 이러한 서열의 매우 유연한 특성으로 인해 다양한 생체 분자 파트너와 약하고 장거리이며 일시적인 상호 작용을 형성할 수 있습니다. 구체적이지만 친화도가 낮은 상호 작용은 무차별 결합을 촉진하고 본질적으로 무질서한 단일 세그먼트가 다수의 표적 부위와 상호 작용할 수 있도록 합니다. 이러한 상호 작용의 일시적인 특성으로 인해 단백질에 의존하여 하나의 우세한 형태를 형성하는 구조 생물학 방법으로 특성화하기 어려울 수 있습니다. 상자성 이완 강화 NMR은 약하고 일시적인 상호 작용의 구조적 토대를 식별하고 정의하는 데 유용한 도구입니다. 본질적으로 무질서한 단백질과 단백질, 핵산 또는 기타 생체 분자 파트너 사이에 형성되는 저인구 만남 복합체를 특성화하기 위해 상자성 이완 향상을 사용하기 위한 자세한 프로토콜이 설명됩니다.
서문
내인성 장애(ID)는 안정적인 2차 또는 3차 구조로 자발적으로 접히지 않고 생물학적으로 활성인 단백질(IDP) 또는 단백질 내 영역(IDR)을 설명합니다. 일반적으로 IDP/IDR의 기능은 생리학적 조건에서 생체 분자와의 특이적이면서도 가역적인 상호작용을 촉진하는 것이다1. 따라서, 국내실향민 및 IDR은 다중단백질 복합체의 모집, 조직 및 안정화, 예를 들어, 스플라이소좀2의 조립 및 활성, DNA 손상부위에서의 성분의 모집 및 조직화3, 전사 복합체4 또는 염색질 리모델러BAF5의 모집의 조직 및 안정화를 포함하는 다양한 세포 기능에 관여한다. 또한 IDP는 서로 다른 결합 파트너에 대한 난잡함으로 인해 세포 단백질 네트워크를 통한 정보 전달을 매개할 수 있는 신호 연결 고리에서 발견됩니다6. 최근 연구에서는 IDR 영역이 액체-액체 상 분리 과정을 통해 생체 분자 응축수를 자가 결합하는 경향이 있음이 밝혀졌다7. ID를 포함하는 전술한 기능들 중 많은 것들은 또한 응축수 형성(condensate formation)8의 어떤 측면을 포함하는 것으로 생각된다. 생체 분자 복합체 조립, 안정화, 스캐폴딩 및 신호 전달에 대한 ID의 중요성에도 불구하고, IDP 및 IDR은 일반적으로 X선 결정학 또는 극저온 전자 현미경을 사용한 구조 조사에 적합하지 않기 때문에 특정 상호 작용의 원자 세부 사항을 식별하기 어렵습니다.
핵 자기 공명(NMR)은 단단하거나 균질한 구조 앙상블의 존재에 의존하지 않고 개별 핵의 즉각적인 국소 환경에 대해 보고하기 때문에 ID를 조사하는 데 이상적인 기술입니다. 주어진 분자 내 핵의 공명 주파수(resonant frequency) 또는 화학적 이동(chemical shift)은 국소 전자 분포에 의해 유도된 약한 자기장의 영향을 받으며, 이는 다시 결합 길이, 각도, 다른 핵의 근접성, 결합 파트너와의 상호작용 및 기타 요인에 따라 달라진다9. 따라서 각 핵은 지역 화학 환경의 변화에 민감한 고유한 부위별 구조 프로브 역할을 합니다. 이러한 장점에도 불구하고 NMR은 벌크 기술이며 관찰된 화학적 이동은 특정 핵에 의해 샘플링된 모든 환경의 평균입니다. 이 문제에 설명된 다양한 NMR 기술은 평균 화학적 이동10,11에 포함된 고에너지, 저인구 생체 분자 형태에 대한 구조적, 동적 및 운동학적 정보를 복구하기 위해 개발되었습니다. 비록 일시적인 인구가 채워져 있기는 하지만, 이들 상태의 식별과 정량화는 기능적 메커니즘의 세부사항을 결정하는 데 중요하다12. 예를 들어, IDP 및 IDR의 경우, 구조적 앙상블은 생리학적 결합 파트너와의 만남 복합체의 형성을 위해 생산적인 샘플 형태를 우선적으로 샘플링하도록 편향될 수 있습니다. 이러한 상태의 검출과 잔류물 특이적 분자 간 및 분자 내 상호 작용 및 역학의 식별은 단백질 기능 및 복합체 형성의 기본 구조적 메커니즘을 결정하는 데 중요합니다.
IDP/IDR 매개 생체 분자 복합체의 형성에 중요한 일시적이고 인구가 적은 상태를 조사하기 위해 상자성 이완 향상(PRE) NMR을 사용하기 위한 프로토콜이 설명되어 있습니다13. 이 접근법은 α-시누클레인 14,15 또는 FUS 16의 자가 결합으로부터 아밀로이드 원섬유의 조립을 촉진하는 것과 같은 일시적인 단백질-단백질 상호작용을 연구하고 신호 전달 단백질17 사이의 특정 단백질-단백질 상호작용을 특성화하는 데 유용합니다. 자기 연관 IDP의 예가 제시되며, 특정 분자 간 및 분자 내 상호 작용이 우선적으로 압축된 상태와 자기 결합을 유도하는 부위별 상호 작용을 초래합니다.
PRE는 등방성 g-텐서가 있는 상자성 중심에 대한 핵의 자기 쌍극자 상호 작용에서 발생하며, 일반적으로 질산화물 그룹 또는 상자성 금속 원자18에 짝을 이루지 않은 전자의 형태로 공급됩니다(그림 1). 이방성 g-텐서를 가진 원자도 PRE 효과를 생성하지만, 이러한 시스템의 분석은 의사 접촉 이동(PCS) 또는 잔류 쌍극자 결합(RDC)에 의해 기여되는 교란 효과로 인해 더 어렵습니다13,19. 핵과 상자성 중심 사이의 상호 작용 강도는 둘 사이의 <r-6> 거리에 따라 달라집니다. 이 상호 작용은 핵 이완 속도를 증가시켜 장거리 상호 작용 (~ 10-35 Å)에서도 검출 가능한 선이 넓어지게하는데, 이는 짝을 이루지 않은 전자의 자기 모멘트가 매우 강하기 때문입니다20,21. PRE로 과도 상태를 감지하는 것은 다음 두 가지 조건이 충족되는 경우 가능합니다. (1) 일시적인 상호 작용은 NMR 시간 척도에서 빠른 교환에 있습니다(관찰된 화학적 이동은 교환 상태의 모집단 가중 평균임). (2) 상자성 중심 거리까지의 핵은 주요 상태(11)에서보다 일시적으로 채워진 상태에서 더 짧다. 횡방향 PRE는 Γ2로 표시되며, 실용적인 목적을 위해 상자성 중심과 반자성 대조군을 포함하는 샘플 사이의 1H횡방향 이완 속도의 차이로부터 계산됩니다. 빠르고 느린 교환 체제에서 PRE 이론 및 관련 의사 접촉 교대에 대한 심층적 인 치료를 위해 독자는 Clore와 동료13,22의 포괄적 인 검토를 참조합니다. 여기서, 1HN-Γ 2 가 빠른 교환 체제에있는 상황 만 고려되며, 여기서 PRE의 r-6- 의존성으로 인해 관찰 된 이완 속도는 상자성 중심이 핵에 접근하는 거리와 그 형태에서 소비하는 시간과 관련이 있습니다. 따라서 근접 접근을 포함하지 않는 과도 형태는 작은 PRE를 생성하는 반면, 근접 상호 작용은 수명이 짧더라도 더 큰 PRE를 생성합니다.
IDP의 경우 PRE는 단일 분자 내(분자 내)와 개별 분자 간(분자 간)에서 발생하는 상호 작용을 측정하고 구별하는 데 사용됩니다. 가시적(예: 15N-표지) 또는 가시적(예: 14N-자연적 존재비) NMR 단백질에 상자성 중심을 부착하여 PRE의 소스(분자간 또는 분자내)를 결정할 수 있습니다(그림 2). 시스테인 잔기를 도입하는 부위-지시적 돌연변이유발은 단백질23에 상자성 중심(스핀-라벨)을 부착하는 편리한 접근법이다. 금속 킬레이트화(EDTA 기반) 및 자유 라디칼(니트록시드 기반)을 포함하여 스핀 라벨로 사용하기 위해 여러 유형의 분자가 제안되었습니다.24. 다양한 산화질소 스핀 표지가 설명되었으며 메탄티오술포네이트, 말레이미드 및 요오도아세트아미드25,26과 같은 다양한 시스테인 반응성 화학 물질과 함께 사용할 수 있습니다(그림 1). 태그 또는 링커의 고유한 유연성은 특정 분석에 문제가 될 수 있으며, 이러한 상황에서는 부피가 큰 화학 그룹을 추가하거나 태그를 단백질에 고정하기 위해 두 번째 링커를 사용하는 것과 같이 태그의 움직임을 제한하기 위한 다양한 전략이 제안되었습니다(27). 28. 추가로, 상업적으로 이용가능한 태그는 부분입체이성질체 단백질을 함유할 수 있지만, 일반적으로 이것은 관찰된 PRE29에 기여하지 않을 것이다. 말레이미드 화학을 통해 유리 시스테인에 부착된 3-말레이미도-PROXYL의 사용은 쉽게 구할 수 있고 비용 효율적이며 비가역적이며 환원제인 트리스(2-카르복시에틸)포스핀(TCEP)이 라벨링 반응 전반에 걸쳐 용액에서 유지될 수 있기 때문에 설명됩니다. 3-Maleimido-PROXYL은 등방성 g-텐서를 갖기 때문에, PCS 또는 RDC가 유도되지 않으며, 상자성 및 반자성 샘플(13) 모두에 대해 동일한 화학적 이동 할당이 사용될 수 있다.
상기 1HNT2는 8 내지 12개의 시점으로 구성된 완전한 진화 시리즈를 수집하는 것만큼 정확한 것으로 이전에 밝혀진 2개의 시점 전략(Ta, Tb)을 사용하여 측정된다(30). 제 1 시점 (Ta)은 실제적으로 0에 가깝게 설정되고, 제 2 시점의 최적 길이는 주어진 샘플에 대해 예상되는 가장 큰 PRE의 크기에 의존하며, 다음으로부터 추정될 수 있다: Tb~1.15/(R2,dia+Γ2) 여기서 R2,dia는 반자성 샘플(13)의 R2를 나타낸다. 가장 큰 PRE의 크기가 알려지지 않은 경우, Tb를 단백질의 1H T2의 ~1배로 설정하는 것은 양호한 초기 추정치이며, 신호 대 잡음을 개선하기 위해 T2를 조정함으로써 추가로 최적화된다. 이 2점 측정 전략은 PRE를 측정하는 데 필요한 실험 시간을 크게 단축하고, 특히 분자 간의 비특이적 접촉의 영향을 최소화하기 위해 상대적으로 희석된 샘플이 사용되기 때문에 더 많은 신호 평균화를 위한 시간을 허용합니다. HSQC 기반 펄스 시퀀스는 1H, N-T2를 측정하는 데 사용되며 다른 곳에서도30에 자세히 설명되어 있습니다. 감도 향상을 위해 순방향 및 후방 INEPT 전송의 하드 펄스를 성형 펄스로 대체할 수 있습니다. 대안적으로, 시퀀스는 TROSY-기반 판독(31)으로 쉽게 변환된다. IDP는 일반적으로 유사한 크기의 구형 단백질보다 훨씬 더 긴 횡방향 이완 속도를 가지므로 (내재적 장애로 인해) 더 좁은 선폭을 초래하기 때문에 간접 차원에서 긴 획득 시간을 사용하여 스펙트럼 분해능을 개선하고 IDP에 내재된 화학적 이동 분산 제한을 완화할 수 있습니다.
PRE는 단백질-단백질 및 단백질-핵산 상호작용, 특히 일시적이거나 인구가 적은 상호작용을 연구하는 데 유용한 도구입니다. 단백질 정제, 부위 지정 스핀 라벨링, 펄스 프로그램 설정 및 보정, NMR 데이터 처리 및 해석을 위한 단계를 포함하여 PRE 측정에 적합한 NMR 샘플을 준비하기 위한 자세한 프로토콜이 제공됩니다. 데이터 품질 및 실험 결과에 영향을 미칠 수 있는 중요한 실험 고려 사항(샘플 농도, 스핀 라벨 선택, 상자성 성분 제거)이 전체적으로 언급됩니다.
프로토콜
프로토콜에 대한 일반 요구 사항: 단백질 정제 시설, UV-Vis 분광계, 고자기장 NMR 분광계 및 운영 소프트웨어, 다음을 포함한 후처리 분석 소프트웨어; NMRPipe32, Sparky 33 (또는 CCPN 분석 34 또는 NMRViewJ35).
1. PRE 측정을 위한 단백질의 재조합 발현 및 정제
- 단일 시스테인 잔기가 존재하도록 관심 단백질에 대한 발현 구조체를 설계합니다. 관심 단백질의 상이한 위치에 유리 시스테인을 도입하기 위해 다중 돌연변이가 필요할 것이다36.
- 확립된 프로토콜37을 사용하여 관심 단백질의 천연 풍부도(14N) 또는 15N-표지된 샘플을 발현하고 정제합니다.
참고: 대장균 발현 시스템은 15N의 동위원소 농축이 생체 분자 이종 핵 NMR 분광법에 대한 최소 요구 사항이기 때문에 재조합 단백질 발현을 위한 비용 효율적이고 강력한 방법을 제공합니다. 일반적인 단계는 최소 배지에서의 발현, 크로마토그래피 정제 및 친화성 정제 태그 제거입니다. 이 프로토콜은 NMR 조사에 적합한 품질의 충분한 단백질을 생산할 수 있는 강력한 발현 및 정제 프로토콜이 확립되었다고 가정합니다.- 모든 정제 단계에서 완충액에 1mM 환원제(DTT 또는 TCEP)를 유지하여 유리 시스테인의 반응과 IDP에 대한 분자간 이황화 결합 형성을 방지합니다.
참고: 일부 시스템은 단백질의 특정 특성뿐만 아니라 정제를 위해 선택된 온도, pH 및 완충 시스템에 따라 비환원 조건에 대해 더 관대하고 응집되기 쉬운 경향이 있을 수 있다38. - 예측할 수 없는 방식으로 단백질과 비특이적으로 상호 작용하거나 의도하지 않은 부착 부위로 작용할 수 있는 반응성 시스테인 잔기를 포함할 수 있으므로 진행하기 전에 정제에 사용되는 친화성 태그를 제거하십시오.
- 용매 PRE의 기여도를 평가하기 위해 스핀 라벨의 가용성 버전과 혼합된 시스테인 돌연변이가 없는 15N표지 기준 샘플을 준비합니다.
- 모든 정제 단계에서 완충액에 1mM 환원제(DTT 또는 TCEP)를 유지하여 유리 시스테인의 반응과 IDP에 대한 분자간 이황화 결합 형성을 방지합니다.
2. 3-Maleimido-PROXYL nitroxide 스핀 라벨 접합
- 정제된 단백질을 50 mM 트리스 pH 7 및 1 mM TCEP를 함유하는 탈기된 완충액으로 저장 또는 교환하고; 완충액은 단백질 용해도를 돕기 위해 필요한 경우 최대 8M 요소를 포함할 수도 있습니다.
대안적으로, 단백질 원액을 탈기된 50 mM 트리스 pH 7 및 1 mM TCEP 완충액의 적어도 10 부피 당량으로 급속히 희석한다. 스핀 라벨을 추가하기 전에 단백질 농도가 100μM 이상인지 확인하십시오. - 원액의 3-Maleimido proxyl을 관심 단백질의 20배 몰 초과량에 추가합니다. 샘플을 빛과 산소로부터 보호하고 실온 또는 4°C에서 밤새 배양합니다. 부드럽게 흔들거나 너트를 사용하면 라벨링 효율성이 향상될 수 있습니다.
- 3-Maleimido proxyl 분말을 95% 에탄올에 용해시켜 스핀 라벨의 원액을 준비합니다. 주식의 분취량은 6 달 미만을 위한 -80 °C에 저장될 수 있습니다.
- 중요 단계: 비특이적 용매 PRE를 방지하기 위해 미반응 유리 스핀 라벨을 제거합니다. 겔 여과 또는 (바람직하게는) 단백질 샘플의 광범위한 투석에 의해 이를 달성합니다. 이 단계는 또한 NMR에 적합한 완충액에 단백질을 도입합니다.
알림: 환원제는 신선하게 준비해야 하며 완충액 성분 간의 호환성을 고려해야 합니다. 예를 들어, TCEP는 인산염 기반 완충액에서 빠르게 분해되며,이 조합은 피해야한다39. - 이 단계부터 사용되는 모든 완충액을 2가 및 전이 금속에 선택적인 킬레이트 수지로 처리하여 상자성 이온 또는 스핀 라벨 소광제를 제거합니다. 단백질을 NMR 완충액에 저장할 수 없는 경우 NMR에 적합한 완충액으로 빠르게 희석할 단백질을 농축합니다.
- 스핀 라벨 통합의 효율성 모니터링.
- 용액40에서 유리 설프하이드릴기를 정량하기 위해 Ellman's 시약(5,5-디티오비스-(2-니트로벤조산))을 사용합니다.
참고: 자세한 프로토콜은 제조업체에서 구할 수 있습니다. 여기서의 목적을 위해, 스핀 표지의 혼입을 결정하는 것이 중요하며, 유리 설프하이드릴기의 농도는 전체 단백질 농도와 비교된다. 유리 설프하이드릴기의 백분율은 니트록사이드 스핀 라벨이 부착되지 않은 분자의 백분율입니다. - 태그된 시스테인 잔기에 해당하는 피크의 강도를 모니터링하여 관심 단백질로의 스핀 라벨 결합을 판단합니다.
참고: 이것은 단백질의 스핀 라벨링 정도를 결정하기 위한 빠르고 효과적인 접근 방식입니다. 스핀 라벨의 완전한 통합은 스펙트럼에서 피크의 소멸을 초래할 것이다. IDP의 불량한 분산 특성으로 인해 돌연변이 시스테인 잔기에 해당하는 피크가 항상 쉽게 식별되는 것은 아니므로 Ellman의 시약(단계 2.6.1)을 사용하는 것이 좋습니다.
- 용액40에서 유리 설프하이드릴기를 정량하기 위해 Ellman's 시약(5,5-디티오비스-(2-니트로벤조산))을 사용합니다.
3. 분자간 또는 분자간 PRE 측정을 위한 NMR 샘플 준비
- 분자 내 PRE 측정을 위한 시료 준비
- NMR에 적합한 완충액에서 15N 동위원소가 풍부한 스핀 표지된 단백질을 최소 100 μM 이상 300 μM 이하의 농도로 준비합니다. 총 시료 부피(D2O포함)는 500 - 550 μL이다.
참고: 일반적인 NMR 완충액에는 인산염, 아세테이트, (이)탄산염 및 TRIS가 포함됩니다. MES, HEPES와 같은 Good's 버퍼도 적절할 수 있습니다. 버퍼를 선택할 때 다른 용액 구성 요소와의 교차 반응이 없도록 주의하십시오. - pH가 ~7.2 이하인지 확인하여 물과 아미드 양성자 교환의 영향을 최소화합니다. 염의 농도를 가능한 한 낮게 유지(일반적으로 150mM 미만)하지만 주요 고려 사항은 단백질 안정성을 유지하는 것입니다.
참고: 고염분 조건에서 NMR 실험을 수행하기 위한 접근 방식은 다른 곳에서 설명되었습니다41.
- NMR에 적합한 완충액에서 15N 동위원소가 풍부한 스핀 표지된 단백질을 최소 100 μM 이상 300 μM 이하의 농도로 준비합니다. 총 시료 부피(D2O포함)는 500 - 550 μL이다.
- 분자간 PRE 측정을 위한 시료 준비
- 이 단계 또는 3.1 단계를 따르십시오. 동시에 수행되지 않습니다. 선택한 NMR 완충액에서 14N천연 풍부함, 스핀 표지 단백질을 준비합니다.
- 최종농도가 3.1.1에서 제조된 샘플과 동일하도록 15N 동위원소가 풍부한 비스핀 표지 단백질과 1%-50% 14N천연 풍부 스핀 표지 단백질을 혼합하여 단백질 샘플을 준비합니다. 총 시료 부피(D2O포함)는 500 - 550 μL입니다.
- 연구된 각 단백질에 대한 15N및 14N단백질의 비율을 경험적으로 최적화합니다. 14개의 N-스핀 표지 단백질의 1%, 5% 및 20%의 비율이 좋은 출발점입니다.
참고: 추가된 14개의 N-스핀 표지 단백질의 함수로서 PRE의 축적은 특정 효과를 나타냅니다. 관찰된 PRE는 거리와 개체군에 의존하기 때문에 샘플에 특이적이며(위에서 논의한 바와 같이), 따라서 상호 작용이 특히 일시적인 경우 14N-스핀 표지 단백질의 더 높은 비율이 필요할 것이다17.
- NMR(분자 내 또는 분자 간) 샘플을 긴 줄기(5") 유리 피펫 또는 마이크로피펫을 사용하여 고자기장 자석에 사용하기에 적합한 9mm NMR 튜브로 옮깁니다. 필드 잠금을 용이하게 하기 위해 모든 NMR 샘플에D2O의 5%-10%가 포함되어 있는지 확인하십시오.
참고: 필요한 샘플을 줄이기 위해 폴리머 플러그를 사용하는 NMR 튜브ample 부피는 효과적인 샘플 shimming과 관련된 어려움으로 인해 PRE 측정에 권장되지 않습니다.
4. NMR 분광계 설정 및 특정 매개변수 실험
- 초전도 고자기장 NMR 분광계 주변에서 작업할 때는 각별한 주의를 기울이십시오.
알림: 자석을 향한 금속 물체의 갑작스러운 가속으로 인한 부상, 이식된 의료 기기와의 간섭, 자석 소멸 시 N 2 및 He2 가스의 갑작스러운 방출로 인한 질식 등의 위험이 있습니다. 다음 단계에서는 판독기가 필요한 교육을 받았고 이러한 위험 및 기타 지역 위험을 알고 있으며 NMR 분광계를 작동할 수 있도록 시설 관리자로부터 승인을 받았다고 가정합니다. 단계나 지침이 의심되는 경우 시설 관리자 또는 숙련된 사용자와 상의하여 잠재적인 부상이나 분광계 손상을 방지하십시오. - 다음 단계에서는 최신 버전의 획득 제어 소프트웨어를 실행하는 상용 NMR 분광계를 가정합니다. 펄스 프로그램 및 매개변수 파일을 다운로드하여 적절한 디렉토리에 배치합니다.
참고: Bruker 분광계 및 TopSpin(3.2 이상)과 함께 사용하기에 적합한 펄스 프로그램 및 매개변수 세트는 저자의 요청에 따라 제공됩니다.- 중요 단계: 네이티브가 아닌 NMR 펄스 프로그램 설치에 익숙하다고 가정합니다. 필요한 경우 시설 관리자 또는 숙련된 사용자와 상담하십시오.
- 샘플을 자석에 넣고, Lock 명령을 사용하여 2H신호를 잠그고, 시설 프로토콜에 따라 1H채널을 튜닝 및 매칭합니다(정확한 절차는 프로브에 원격 튜닝 및 매치 모듈이 장착되어 있는지 여부에 따라 다름).
- topshim 서브루틴을 사용하여 심을 조정하여 용매 신호 억제를 최적화합니다.
- 표준 방법을 사용하여 1H 및 15N 90° 펄스를 교정합니다.
- popt 프로그램을 사용하여 1H펄스를 보정합니다(펄스 길이를 추정하려면 먼저 펄스 사용을 사용).
- 표준 샘플에 대해 15N펄스를 교정합니다. 기술 책임자 또는 숙련된 사용자와 논의하여 이 값이 최근에 보정되었는지 확인하십시오.
- 또는 null 신호가 달성될 때까지 HMQC 실험의 90° 펄스 중 하나를 변경하여 샘플의 15N펄스를 보정합니다.
- shape tool(stdisp) 서브루틴을 사용하여 형태화된 펄스에 대한 올바른 감쇠를 판별하십시오.
- 폴더 아이콘을 클릭하여 적절한 펄스 모양 파일을 엽니다. 성형된 펄스는 ACQUPARS의 펄스 매개변수 섹션에서 찾을 수 있습니다.
- 펄스 정의 파일을 로드하고 Analyze Waveform > Integrate Shape를 클릭합니다. 보정된 1H 90° 하드 펄스, 원하는 모양의 펄스 길이 및 회전(90° 또는 180°)을 입력합니다.
- 보정된 90° 펄스에 대한 감쇠에 전력 레벨 변화를 추가하여 성형된 펄스의 전력 레벨을 계산합니다.
- 표준 1H, 15N HSQC(hsqcetfpf3gpsi)를 기록하여 스윕 폭, 캐리어 주파수를 최적화하고 물 억제를 확인합니다25.
- sw 및 td 명령을 사용하거나 해당 대화상자에서 직접 스윕 폭과 간접 치수 증분 수를 조정합니다. 일반적으로 PRE를 수집하기 위해 스펙트럼이 접히지 않도록 스펙트럼 폭이 선택됩니다.
5. 1HN-T 2 실험 설정
- 위에서 설명한 대로 모양의 펄스를 보정합니다(4.4.5-4.5.7). PRE 실험을 위한 성형 펄스 매개변수 파일은 Eburp2.1000(90° 펄스), Reburp.1000 및 Iburp2.1000입니다. ACQUPARS 탭의 펄스 매개변수 섹션에 보정된 펄스 길이를 입력합니다.
- 본 실험은 2개의 시간지연 시점 접근법(30)을 이용하여1HN-T2를 측정한다.
- vdlist 파일을 편집하여 지연 시간을 설정하고, 제1 지연 시간(Ta)은 0.01 ms로 설정한다.
- 예상 최대 PRE(Tb~1.15/(R2,dia+Γ 2)와의 관계를 사용하여 제2 지연(Tb)을 선택하고, 여기서 R2,dia는 반자성 샘플13의 R2를 나타낸다. 관찰된 이완에 대한 PRE 기여의 크기에 대한 사전 지식 없이, 좋은 출발점은 Tb를 ~1x 1H T2로 설정하는 것입니다.
- 그런 다음 T a 및 Tb 스펙트럼의 첫 번째 증분(efp 명령으로 처리됨)을 비교하고 신호가 초기 값의 40%-50% 사이로 감쇠하도록 Tb를 조정하여 적절한 값을 결정합니다.
참고: 이 접근 방식은 스펙트럼 신호 대 잡음비를 최적화하며, 이는 고농축(< 50μM)이 불가능한 샘플에 필요한 고려 사항입니다. Tb의 적합한 값은 샘플에 따라 다르지만, 전형적으로 평균 크기의 단백질에 대해 8 - 40 ms의 범위이다.
- 충분한 신호 평균화를 위해 기록할 복소점의 수와 스캔 횟수를 결정합니다. IDP는 비슷한 크기의 접힌 단백질보다 더 긴 15NT2를 갖기 때문에, 간접 차원에서 더 긴 획득 시간이 사용될 수 있다.
참고: 이 값은 단백질의 특정 특성에 따라 달라지지만, 15NT2로부터 대략적으로 추정될 수 있고, FID에서 신호 붕괴를 모니터링함으로써 최적화될 수 있다. 직접 치수의 경우 대부분의 샘플에 대해 1024*개의 복소점(13ppm 스윕 폭, 112.6ms 획득 시간)이면 충분합니다. - 명령 expt를 사용하여 실험 시간을 계산한 다음 명령 zg로 실험을 시작합니다.
6. 아스코르브산으로 스핀 라벨을 줄여 반자성 시료 만들기
- 아스코르브산나트륨을 NMR 완충액에 녹이고 원래의 NMR 완충액과 일치하도록 pH를 조정합니다.
- 소듐 아스코르브산염 스톡의 농도를 계산하여 스핀 라벨의 농도에 비해 10배 몰 초과량의 아스코르브산염을 시료 부피의 변화를 최소화하면서 첨가할 수 있습니다. 예를 들어, 100μM 단백질 샘플의 경우 100mM 스코르브산염 스톡이 적합합니다. 스핀 라벨을 줄이려면 총 시료 부피의 1%에 불과한 5.5μL의 아스코르브산 원액을 추가해야 합니다.
- 튜브의 가장자리 아래에 물방울을 놓고 튜브를 덮고 튜브를 조심스럽게 뒤집어 혼합한 다음 200-400 x g 에서 10-20초 동안 회전하여 NMR 튜브에 필요한 양의 아스코르브산을 추가합니다.
- NMR 튜브를 호일로 감싸 빛으로부터 보호하고 반응이 최소 3시간 동안 진행되도록 합니다.
- 1 HN-T 2 반자성 샘플에 대해 상자성 샘플에 사용된 것과 동일한 파라미터를 사용합니다.
- 펄스를 재보정합니다. 그러나 상자성 측정에서 변경되어서는 안됩니다. 두 값이 크게 다른 경우(> 0.5μs 차이) 샘플 품질(예: 분해, 침전)을 고려하십시오.
- 지정된 완화 지연(vdlist), 더미 스캔 수, 수집된 스캔 수, 수집된 복소점 수, 획득 시간, 스윕 폭 및 반송파 주파수를 포함한 모든 획득 파라미터가 반자성 및 상자성 샘플에 대해 동일하게 유지되는지 확인합니다.
7. 상자성 및 반자성 스펙트럼 처리
- NMRPipe 및 Sparky가 설치 및 구성된 로컬 컴퓨터 또는 워크스테이션에 데이터를 복사합니다. ser 파일이 포함된 실험 데이터 디렉터리에 proc라는 폴더를 만듭니다.
- NMRPipe 스크립트 fid.com, p3d.com 및 nmrproc.com 를 proc에 복사합니다(처리 스크립트는 작성자의 요청에 따라 사용할 수 있음).
- fid.com 스크립트를 사용하여 Bruker 데이터 형식(ser)을 NMRPipe 형식으로 변환합니다.
- p3D.com 스크립트를 사용하여 pseudo3D 평면을 개별 스펙트럼으로 분할합니다.
- nmrproc.com 스크립트를 사용하여 fid.com 스크립트의 출력값을 읽고, 용매 억제 및 창 함수를 적용하고, 원시 데이터에 0을 추가(0 채우기)하고, 위상 보정을 적용하고, 푸리에 변환을 실행하고, 표시할 데이터를 트리밍하고, 처리된 데이터를 디스크에 씁니다. 스크립트는 기록된 각 완화 지연(T a 및 Tb)에 대해 하나의 파일을 출력합니다.
참고: 이러한 각 스크립트는 각 실험의 특정 세부 사항에 대한 처리를 최적화하도록 사용자 지정할 수 있습니다. 튜토리얼 및 예시 데이터 세트는 NMRPipe 웹 사이트(32)로부터 이용가능한 NMRPipe 배포판에 포함된다. NMRDraw는 처리 중 스펙트럼 보기(예: 적절한 위상각 설정 등)에 사용할 수 있습니다. NMRPipe 명령에 사용할 수 있는 옵션은 nmrPipe -help 명령을 사용하여 볼 수 있습니다.
8. 공명 할당 전송 및 피크 높이 추출
- sethdr [filename] -ndim 2 명령을 사용하여 각 스펙트럼 파일(상자성 및 반자성 샘플 모두에 대한 Ta, Tb)에 대한 파일 헤더 정보를 변경합니다.
- Sparky를 사용하여 8.3-8.5단계에 따라 피크 높이33을 추출합니다. NMRPipe (NMRDraw) 32, CCPN Analysis 34 및 NMRViewJ35를 포함한 다른 소프트웨어 패키지도 적합합니다.
- 스펙트럼 파일을 Sparky로 읽어 들입니다. 이 단계에서 데이터 세트는 단백질에서 스핀-라벨의 각 위치에 대해 측정된 상자성 및 반자성 샘플 모두에 대해 각 시점 스펙트럼(Ta, Tb)에 대해 하나의 스펙트럼으로 구성됩니다.
- Sparky를 사용하여 피크를 선택하고( 명령: F8 키를 누른 다음 클릭하고 드래그) 참조 피크 목록에서 피크 목록 전송 도구를 사용하여 할당을 전송합니다.
참고: 관심 단백질의 공명 할당은 관찰된 PREs36의 서열 특이적 해석에 필요합니다.- 상자성 스펙트럼과 반자성 스펙트럼의 윤곽을 동일한 수준으로 설정합니다. 시간 지연 후에 수집된 스펙트럼이 의도적으로 피크를 제외하지 않고 Ta 스펙트럼이 지나치게 노이즈가 발생하지 않도록 충분히 높도록 윤곽을 설정해야 합니다.
- 각 스펙트럼에 대한 새 피크 목록을 저장하고 측정된 피크 강도와 계산된 Sparky 신호 대 잡음비를 포함합니다(명령: 피크 목록을 열려면 lt, 강도 및 SNR 열을 포함하려면 옵션 클릭, 명령: 저장).
9. 각 잔류물에 대해 1H, N-T2 비율을 추출하고 PRE를 계산합니다.
- 피크 목록을 스프레드시트 소프트웨어 또는 Python과 같은 기본 프로그래밍 언어로 가져옵니다.
참고: 단백질의 각 스핀 라벨 위치에 대해 데이터 세트는 관련 피크 강도가 있는 4개의 피크 목록으로 구성되며, 상자성 및 반자성 실험 모두에 대해 각각 Ta 및 Tb 가 하나씩 구성됩니다. - 다음 방정식을 사용하여 상자성 및 반자성 샘플 모두에 대해 1HN R2를 계산합니다.
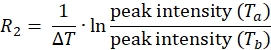
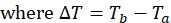
- 위의 방정식을 사용하여 상자성 및 반자성 샘플의 각 잔류물에 대한 이완 속도를 결정합니다.
- 다음 방정식을 사용하여 각 잔류물에 대한 1HN-Γ 2 비율을 결정합니다.
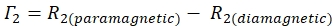
- Sparky에서 계산된 신호 대 잡음비(SNR)를 사용하여 각 잔류물에 대한 피크 높이의 불확도를 계산합니다.
- 다음 방정식을 사용하여 오류를 전파합니다.

- 플롯 1H,N-Γ 2는 9.6에서 계산된 오차를 포함하는 산점도를 사용하여 잔차 수의 함수입니다.
결과
분자 내 1HN-Γ 2 PRE는 RNA 결합 단백질 EWSR142의 저복잡성 도메인에서 유래된 자가 회합, 본질적으로 무질서한 단편(잔기 171-264)에 기록되었습니다(그림 3). 스핀-라벨 부착 지점에 근접한 순차적 근접 잔기(예를 들어, 도 3의 잔기 178 또는 260)는 상당히 넓어질 것으로 예상되며, 스펙트럼에서 검출가능하지 ?...
토론
PRE를 사용하여 본질적으로 무질서한 단백질과 다양한 결합 파트너 사이에 낮은 집단에 존재하는 일시적인 상호 작용을 특성화하는 방법이 제시되었습니다. 제시된 예에서, 단백질은 자가 회합이며, 따라서 PRE는 분자간 및 분자내 상호작용의 조합으로부터 발생할 수 있다. 이 방법은 두 개의 서로 다른 단백질 간의 상호 작용을 특성화할 수 있는 이종 샘플로 쉽게 확장됩니다. 단백질의 상이한 영?...
공개
모든 저자는 원고를 읽고 승인했습니다. 이해 상충은 선언되지 않습니다.
감사의 말
유용한 토론과 기술 지원을 해주신 Jinfa Ying 박사와 Kristin Cano에게 감사드립니다. DSL은 St. Baldrick's Scholar이며 St. Baldrick's Foundation(634706)의 지원을 인정합니다. 이 작업은 Welch Foundation(AQ-2001-20190330)에서 DSL로, Max and Minnie Tomerlin Voelcker Fund(DSL에 대한 Voelcker Foundation Young Investigator Grant), DSL에 대한 UTHSA 스타트업 펀드, CNJ에 대한 Greehey Graduate Fellowship in Children's Health에서 부분적으로 지원되었습니다. 이 작업은 샌안토니오에 있는 텍사스 대학교 보건 과학 센터의 기관 연구 핵심의 일부인 구조 생물학 핵심 시설에서 수행된 연구를 기반으로 하며, 연구 담당 부사장실과 Mays Cancer Center 약물 발견 및 구조 생물학 공유 리소스(NIH P30 CA054174).
자료
| Name | Company | Catalog Number | Comments |
| 0.45 µm and 0.22 µm syringe filters | Millipore Sigma | SLHVM33RS SLGVR33RS | Filter lysate before first purification step and before size exclusion chromatography. |
| 100 mm Petri Dish | Fisher | FB0875713 | Agar plates for bacterial transformation. |
| 14N Ammonium chloride | Sigma Aldrich | 576794 | Use of 15N in M9 medium will produce an NMR visible protein, 14N will produces an NMR invisible protein |
| 15N Ammonium chloride | Sigma Aldrich | 299251 | Use of 15N in M9 medium will produce an NMR visible protein, 14N will produces an NMR invisible protein |
| 3 L Fernbach baffled flask | Corning | 431523 | Bacterial expression culture |
| 3-Maleimido-Proxyl | Sigma Aldrich | 253375 | Nitroxide spin label |
| 50 mL conical centrifuge tubes | Thermo Fisher | 14-432-22 | Solution/protein storage |
| Amicon centrifugal filter | Millipore Sigma | UFC900308 | Protein concentration |
| Ampicillan | Sigma Aldrich | A5354 | Antibiotic for a selective marker, exact choice depends on the expression construct plasmid |
| Analytical balance | Oahus | 30061978 | Explorer Pro, for weighing reagents |
| Ascorbic acid | Sigma Aldrich | AX1775 | Reduces nitroxide spin label |
| Autoclave | Sterilize glassware and culture media | ||
| Calcium chloride | Sigma Aldrich | C4901 | M9 media component |
| Centrifuge bottles | Thermo Fisher | 010-1459 | Harvest E. coli cells after recombinant protein expression |
| Centrifuge, hand-crank | Thomas Scientific | 0241C68 | Boekel hand-driven, low-speed centrifuge with 15 mL buckets that can accommodate NMR tubes |
| Chelex 100 | Sigma Aldrich | C7901 | Remove contaminating paramagnetic compounds from buffer solutions |
| Computer workstation | Linux or Mac OS compatable with NMR data processing and analysis software packages such as NMRPipe and Sparky | ||
| Deuterium oxide | Sigma Aldrich | 151882 | Needed for NMR lock signal |
| Dextrose | Sigma Aldrich | D9434 | M9 media component |
| Dibasic Sodium Phosphate | Sigma Aldrich | S5136 | M9 media component |
| Ellman's reagent (5,5-dithio-bis-(2-nitrobenzoic acid) | Thermo Fisher | 22582 | Quantification of free cystiene residues |
| High speed centrifuge tubes | Thermo Fisher | 3114-0050 | Used to clear bacterial lysate. |
| High-field NMR instrument (600 - 800 MHz) | Bruker | Equiped with a multichannel cryogenic probe and temperature control | |
| IMAC column, HisTrap FF | Cytvia | 17528601 | Initial fractionation of crude bacterial lysate |
| Isopropyl B-D-thiogalactoside (IPTG) | Sigma Aldrich | I6758 | Induces protein expression for genes under control of lac operator |
| LB agar | Thermo Fisher | 22700025 | Items are used for transforming E. coli to express protein of interest, substitions for any of these items with like products is acceptable. |
| LB broth | Thermo Fisher | 12780052 | |
| Low-pressure chromatography system | Bio-Rad | 7318300 | BioRad BioLogic is used for low-pressure chomatograph such as running IMAC columns |
| Magnesium sulfate | Sigma Aldrich | M7506 | M9 media component |
| Medium pressure chromatography system | Bio-Rad | 7880007 | BioRad NGC equipped with a multi-wavelength detector, pH and conductivity monitors, and automatic fraction collector |
| MEM vitamin solution | Sigma Aldrich | M6895 | M9 media component |
| Microfluidizer | Avestin | EmulsiFlex-C3 | Provides rapid and efficient bacterial cell lysis |
| Micropipettes | Thermo Fisher | Calibrated set of micropippetters with properly fitting disposable tips (available from multiple manufacturers e.g. Eppendorf) | |
| Monobasic potassium phosphate | Sigma Aldrich | 1551139 | M9 media component |
| NMR pipettes | Sigma Aldrich | 255688 | To remove sample from NMR tube |
| NMR sample tube | NewEra | NE-SL5 | Suitable for high-field NMR spectrometers |
| Preparative Centrifuge | Beckman Coulter | Avanti J-HC | Harvest E. coli cells after recombinant protein expression |
| Round bottom polystyrene centrifuge tubes | Corning | 352057 | Clear bacterial lysate |
| Shaking incubator | Eppendorf | S44I200005 | Temperature controlled growth of E. coli starter and expression cultures |
| Sodium chloride | Sigma Aldrich | S5886 | M9 media component |
| Sonicating water bath and vacuum source | Thomas Scientific | Used to degas buffer solutions | |
| Sonicator | Thermo Fisher | FB505110 | Used for bacterial cell lysis or shearing bacterial DNA |
| Spectrophotometer | Implen | OD600 Diluphotometer | Monitor growth of E.coli protein expression cultures |
| Superdex 200 16/600 size exculsion colum | Cytvia | 28989333 | Final protein purification step |
| Topspin software, version 3.2 or later | Bruker | Operating software for the NMR instrument | |
| Transformation competent E. coli cells | Thermo Fisher | C600003 | One Shot BL21 Star (DE3) chemically competent E. coli, other strains may be compatable |
| Tris(2-carboxyethyl)phosphine (TCEP) | ThermoFisher | 20490 | Reducing agent compatable with some sulfhydryl-reactive conjugations |
| UV-Vis spectrophotometer | Implen | NP80 | Measure protein concentration. |
| Water bath, temperature controlled | ThermoFisher | FSGPD25 | For heat shock step of bacterial transformation |
| Yeast extract | Sigma Aldrich | Y1625 | For supplementing M9 media if required |
참고문헌
- Dyson, H. J., Wright, P. E. Intrinsically unstructured proteins and their functions. Nature Reviews: Molecular Cell Biology. 6 (3), 197-208 (2005).
- Korneta, I., Bujnicki, J. M. Intrinsic disorder in the human spliceosomal proteome. PLoS Computational Biology. 8 (8), 1002641 (2012).
- Frege, T., Uversky, V. N. Intrinsically disordered proteins in the nucleus of human cells. Biochemistry and Biophysics Reports. 1, 33-51 (2015).
- Liu, J., et al. Intrinsic disorder in transcription factors. Biochemistry. 45 (22), 6873-6888 (2006).
- El Hadidy, N., Uversky, V. N. Intrinsic disorder of the BAF complex: Roles in chromatin remodeling and disease development. International Journal of Molecular Sciences. 20 (21), (2019).
- Wright, P. E., Dyson, H. J. Intrinsically disordered proteins in cellular signalling and regulation. Nature Reviews: Molecular Cell Biology. 16 (1), 18-29 (2015).
- Brangwynne, C. P. Phase transitions and size scaling of membrane-less organelles. Journal of Cell Biology. 203 (6), 875-881 (2013).
- Shin, Y., Brangwynne, C. P. Liquid phase condensation in cell physiology and disease. Science. 357 (6357), (2017).
- Cavanagh, J. . Protein NMR spectroscopy : principles and practice. 1st edition. , (2018).
- Sekhar, A., Kay, L. E. NMR paves the way for atomic level descriptions of sparsely populated, transiently formed biomolecular conformers. Proceedings of the National Academy of Sciences of the United States of America. 110 (32), 12867-12874 (2013).
- Anthis, N. J., Clore, G. M. Visualizing transient dark states by NMR spectroscopy. Quarterly Reviews of Biophysics. 48 (1), 35-116 (2015).
- Alderson, T. R., Kay, L. E. NMR spectroscopy captures the essential role of dynamics in regulating biomolecular function. Cell. 184 (3), 577-595 (2021).
- Clore, G. M., Iwahara, J. Theory, practice, and applications of paramagnetic relaxation enhancement for the characterization of transient low-population states of biological macromolecules and their complexes. Chemical Reviews. 109 (9), 4108-4139 (2009).
- Wu, K. P., Baum, J. Detection of transient interchain interactions in the intrinsically disordered protein alpha-synuclein by NMR paramagnetic relaxation enhancement. Journal of the American Chemical Society. 132 (16), 5546-5547 (2010).
- Janowska, M. K., Wu, K. P., Baum, J. Unveiling transient protein-protein interactions that modulate inhibition of alpha-synuclein aggregation by beta-synuclein, a pre-synaptic protein that co-localizes with alpha-synuclein. Scientific Reports. 5, 15164 (2015).
- Murthy, A. C., et al. Molecular interactions underlying liquid-liquid phase separation of the FUS low-complexity domain. Nature Structural & Molecular Biology. 26 (7), 637-648 (2019).
- Fawzi, N. L., Doucleff, M., Suh, J. Y., Clore, G. M. Mechanistic details of a protein-protein association pathway revealed by paramagnetic relaxation enhancement titration measurements. Proceedings of the National Academy of Sciences of the United States of America. 107 (4), 1379-1384 (2010).
- Griffith, O. H., Waggoner, A. S. Nitroxide free radicals: spin labels for probing biomolecular structure. Accounts of Chemical Research. 2 (2), 17-24 (1969).
- Bertini, I., Luchinat, C., Parigi, G., Ravera, E. . NMR of Paramagnetic Macromolecules, Applications to Metallobiomolecules and Models. 2 edn. , (2016).
- Bloembergen, N., Purcell, E. M., Pound, R. V. Relaxation effects in nuclear magnetic resonance absorption. Physical Review. 73 (7), 679-712 (1948).
- Solomon, I. Relaxation processes in a system of two spins. Physical Review. 99 (2), 559 (1955).
- Clore, G. M. Practical aspects of paramagnetic relaxation enhancement in biological macromolecules. Methods in Enzymology. 564, 485-497 (2015).
- Klare, J. P. Site-directed spin labeling EPR spectroscopy in protein research. Biological Chemistry. 394 (10), 1281-1300 (2013).
- Clore, G. M., Tang, C., Iwahara, J. Elucidating transient macromolecular interactions using paramagnetic relaxation enhancement. Current Opinion in Structural Biology. 17 (5), 603-616 (2007).
- Melanson, M., Sood, A., Torok, F., Torok, M. Introduction to spin label electron paramagnetic resonance spectroscopy of proteins. Biochemistry and Molecular Biology Education. 41 (3), 156-162 (2013).
- Czogalla, A., Pieciul, A., Jezierski, A., Sikorski, A. F. Attaching a spin to a protein -- site-directed spin labeling in structural biology. Acta Biochimica Polonica. 54 (2), 235-244 (2007).
- Lindfors, H. E., de Koning, P. E., Drijfhout, J. W., Venezia, B., Ubbink, M. Mobility of TOAC spin-labelled peptides binding to the Src SH3 domain studied by paramagnetic NMR. Journal of Biomolecular NMR. 41 (3), 157-167 (2008).
- Fawzi, N. L., et al. A rigid disulfide-linked nitroxide side chain simplifies the quantitative analysis of PRE data. Journal of Biomolecular NMR. 51 (1-2), 105-114 (2011).
- Bleicken, S., et al. gem-Diethyl pyrroline nitroxide spin labels: Synthesis, EPR characterization, rotamer libraries and biocompatibility. ChemistryOpen. 8 (8), 1035 (2019).
- Iwahara, J., Tang, C., Clore, G. M. Practical aspects of 1H transverse paramagnetic relaxation enhancement measurements on macromolecules. Journal of Magnetic Resonance. 184, 185-195 (2007).
- Venditti, V., Fawzi, N. L. Probing the atomic structure of transient protein contacts by paramagnetic relaxation enhancement solution NMR. Methods in Molecular Biology. 1688, 243-255 (2018).
- Delaglio, F., et al. NMRPipe: a multidimensional spectral processing system based on UNIX pipes. Journal of Biomolecular NMR. 6 (3), 277-293 (1995).
- Lee, W., Tonelli, M., Markley, J. L. NMRFAM-SPARKY: enhanced software for biomolecular NMR spectroscopy. Bioinformatics. 31 (8), 1325-1327 (2015).
- Vranken, W. F., et al. The CCPN data model for NMR spectroscopy: development of a software pipeline. Proteins. 59 (4), 687-696 (2005).
- Johnson, B. A. Using NMRView to visualize and analyze the NMR spectra of macromolecules. Methods in Molecular Biology. 278, 313-352 (2004).
- Sjodt, M., Clubb, R. T. Nitroxide labeling of proteins and the determination of paramagnetic relaxation derived distance restraints for NMR studies. Bio-Protocol. 7 (7), (2017).
- Zhang, H., van Ingen, H. Isotope-labeling strategies for solution NMR studies of macromolecular assemblies. Current Opinion in Structural Biology. 38, 75-82 (2016).
- Rabdano, S. O., et al. Onset of disorder and protein aggregation due to oxidation-induced intermolecular disulfide bonds: case study of RRM2 domain from TDP-43. Scientific Reports. 7 (1), 11161 (2017).
- Burns, J. A., Butler, J. C., Moran, J., Whitesides, G. M. Selective reduction of disulfides by tris(2-carboxyethyl)phosphine. Journal of Organic Chemistry. 56 (8), 2648-2650 (1991).
- Ellman, G. L. Tissue sulfhydryl groups. Archives of Biochemistry and Biophysics. 82 (1), 70-77 (1959).
- Binbuga, B., Boroujerdi, A. F., Young, J. K. Structure in an extreme environment: NMR at high salt. Protein Science. 16 (8), 1783-1787 (2007).
- Schwartz, J. C., Cech, T. R., Parker, R. R. Biochemical properties and biological functions of FET proteins. Annual Review of Biochemistry. 84, 355-379 (2015).
- Nabuurs, S. M., de Kort, B. J., Westphal, A. H., van Mierlo, C. P. Non-native hydrophobic interactions detected in unfolded apoflavodoxin by paramagnetic relaxation enhancement. European Biophysics Journal. 39 (4), 689-698 (2010).
- Wiedemann, C., Kumar, A., Lang, A., Ohlenschlager, O. Cysteines and disulfide bonds as structure-forming units: Insights from different domains of life and the potential for characterization by NMR. Frontiers in Chemistry. 8, 280 (2020).
- Wommack, A. J., et al. NMR solution structure and condition-dependent oligomerization of the antimicrobial peptide human defensin 5. Biochemistry. 51 (48), 9624-9637 (2012).
- Taylor, A. M., et al. Detailed characterization of cysteine-less P-glycoprotein reveals subtle pharmacological differences in function from wild-type protein. British Journal of Pharmacology. 134 (8), 1609-1618 (2001).
- Hu, K., Doucleff, M., Clore, G. M. Using multiple quantum coherence to increase the 15N resolution in a three-dimensional TROSY HNCO experiment for accurate PRE and RDC measurements. Journal of Magnetic Resonance. 200 (2), 173-177 (2009).
- Anthis, N. J., Doucleff, M., Clore, G. M. Transient, sparsely populated compact states of apo and calcium-loaded calmodulin probed by paramagnetic relaxation enhancement: interplay of conformational selection and induced fit. Journal of the American Chemical Society. 133 (46), 18966-18974 (2011).
- Battiste, J. L., Wagner, G. Utilization of site-directed spin labeling and high-resolution heteronuclear nuclear magnetic resonance for global fold determination of large proteins with limited nuclear overhauser effect data. Biochemistry. 39 (18), 5355-5365 (2000).
- Donaldson, L. W., et al. Structural characterization of proteins with an attached ATCUN motif by paramagnetic relaxation enhancement NMR spectroscopy. Journal of the American Chemical Society. 123 (40), 9843-9847 (2001).
- Gaponenko, V., et al. Protein global fold determination using site-directed spin and isotope labeling. Protein Science. 9 (2), 302-309 (2000).
- Trindade, I. B., Invernici, M., Cantini, F., Louro, R. O., Piccioli, M. PRE-driven protein NMR structures: an alternative approach in highly paramagnetic systems. FEBS Journal. 288 (9), 3010-3023 (2021).
- Nitsche, C., Otting, G. Pseudocontact shifts in biomolecular NMR using paramagnetic metal tags. Progress in Nuclear Magnetic Resonance Spectroscopy. 98-99, 20-49 (2017).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유