Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Amélioration de la relaxation paramagnétique pour détecter et caractériser des auto-associations de protéines intrinsèquement désordonnées
Dans cet article
Résumé
Un protocole pour l’application de la spectroscopie RMN d’amélioration de la relaxation paramagnétique pour détecter les interactions inter- et intramoléculaires faibles et transitoires dans les protéines intrinsèquement désordonnées est présenté.
Résumé
Les protéines intrinsèquement désordonnées et les régions intrinsèquement désordonnées dans les protéines constituent une partie importante et fonctionnellement significative du protéome humain. La nature très flexible de ces séquences leur permet de former des interactions faibles, à longue portée et transitoires avec divers partenaires biomoléculaires. Des interactions spécifiques mais de faible affinité favorisent la liaison de promiscuité et permettent à un seul segment intrinsèquement désordonné d’interagir avec une multitude de sites cibles. En raison de la nature transitoire de ces interactions, elles peuvent être difficiles à caractériser par des méthodes de biologie structurale qui reposent sur des protéines pour former une seule conformation prédominante. La RMN d’amélioration de la relaxation paramagnétique est un outil utile pour identifier et définir le fondement structurel des interactions faibles et transitoires. Un protocole détaillé pour l’utilisation de l’amélioration de la relaxation paramagnétique pour caractériser les complexes de rencontre faiblement peuplés qui se forment entre les protéines intrinsèquement désordonnées et leurs protéines, acides nucléiques ou autres partenaires biomoléculaires est décrit.
Introduction
Le trouble intrinsèque (DI) décrit des protéines (IDP) ou des régions à l’intérieur des protéines (IDR) qui ne se replient pas spontanément en structures secondaires ou tertiaires stables, mais qui sont biologiquement actives. Généralement, la fonction des IDP/IDR est de faciliter des interactions spécifiques mais réversibles avec des biomolécules dans des conditions physiologiques1. Ainsi, les IDP et les IDR sont impliqués dans une gamme de fonctions cellulaires, y compris le recrutement, l’organisation et la stabilisation de complexes multiprotéiques, par exemple, l’assemblage et l’activité du splicéosome2, le recrutement et l’organisation des composants sur les sites de dommages à l’ADN3, l’organisation et la stabilisation du recrutement des complexes de transcription4, ou du remodeleur de chromatine BAF5. En outre, les PDI se trouvent à des carrefours de signalisation où leur promiscuité pour différents partenaires de liaison leur permet de servir de médiateur dans le transfert d’informations via des réseaux de protéines cellulaires6. Des travaux récents ont également révélé une propension des régions IDR à s’associer en formant des condensats biomoléculaires par le processus de séparation en phase liquide-liquide7. Bon nombre des fonctions susmentionnées impliquant l’ID sont également considérées comme impliquant un aspect de la formation de condensats8. Malgré l’importance de l’ID pour l’assemblage de complexes biomoléculaires, la stabilisation, l’échafaudage et la transduction du signal, les détails atomiques de leurs interactions spécifiques sont difficiles à identifier car les IDP et les IDR ne se prêtent généralement pas à des études structurelles utilisant la cristallographie aux rayons X ou la microscopie électronique cryogénique.
La résonance magnétique nucléaire (RMN) est une technique idéale pour étudier l’ID car elle ne dépend pas de la présence d’ensembles structurels rigides ou homogènes, mais rend compte de l’environnement local immédiat des noyaux individuels. La fréquence de résonance, ou décalage chimique, d’un noyau dans une molécule donnée est influencée par les faibles champs magnétiques induits par la distribution électronique locale, qui à son tour dépend de la longueur des liaisons, des angles, de la proximité d’autres noyaux, des interactions avec les partenaires de liaison et d’autres facteurs9. Ainsi, chaque noyau agit comme une sonde structurelle unique, spécifique au site, sensible aux changements de son environnement chimique local. Malgré ces avantages, la RMN est une technique de masse, et le décalage chimique observé est la moyenne de tous les environnements échantillonnés par un noyau particulier. Une gamme de techniques de RMN, dont beaucoup sont décrites dans ce numéro, ont été développées pour récupérer des informations structurelles, dynamiques et cinétiques sur les conformations biomoléculaires à haute énergie et faiblement peuplées contenues dans le décalage chimique moyen10,11. Bien que transitoirement peuplés, l’identification et la quantification de ces états sont importantes pour déterminer les détails des mécanismes fonctionnels12. Par exemple, dans le cas des PDI et des IDR, l’ensemble conformationnel peut être biaisé pour échantillonner préférentiellement les conformations productives pour la formation de complexes de rencontre avec des partenaires de liaison physiologiques. La détection de ces états, ainsi que l’identification des interactions et de la dynamique inter- et intramoléculaires spécifiques aux résidus, sont importantes pour déterminer les mécanismes structurels sous-jacents de la fonction protéique et de la formation complexe.
Un protocole d’utilisation de la RMN d’amélioration de la relaxation paramagnétique (PRE) pour étudier les états transitoires et peu peuplés importants pour la formation de complexes biomoléculaires médiés par IDP/IDR est décrit13. Cette approche est utile pour étudier les interactions transitoires protéine-protéine telles que celles qui favorisent l’assemblage des fibrilles amyloïdes à partir de la α-synucléine 14,15 ou l’auto-association de FUS16, ainsi que pour caractériser des interactions protéine-protéine spécifiques telles que entre protéines de signalisation17. Un exemple d’IDP auto-associatif est présenté, où des interactions inter- et intramoléculaires spécifiques entraînent des états préférentiellement compactés ainsi que des interactions spécifiques au site qui conduisent à l’auto-association.
Le PRE résulte de l’interaction dipolaire magnétique d’un noyau à un centre paramagnétique avec un tenseur g isotrope, généralement fourni sous la forme d’un électron non apparié sur un groupe nitroxide ou sous forme d’atome métallique paramagnétique18 (Figure 1). Alors que les atomes avec des tenseurs g anisotropes produisent également un effet PRE, l’analyse de ces systèmes est plus difficile en raison des effets de confusion apportés par les décalages de pseudo-contact (PCS) ou le couplage dipolaire résiduel (RDC)13,19. La force de l’interaction entre un noyau et le centre paramagnétique dépend de la distance <r-6> entre les deux. Cette interaction entraîne une augmentation des taux de relaxation nucléaire, ce qui provoque un élargissement détectable de la raie même pour les interactions à longue portée (~10-35 Å), car le moment magnétique de l’électron non apparié est si fort20,21. La détection d’états transitoires avec le PRE est possible si les deux conditions suivantes sont remplies; (1) l’interaction transitoire est en échange rapide sur l’échelle de temps RMN (le déplacement chimique observé est une moyenne pondérée en fonction de la population des états échangeurs); et (2) la distance entre les noyaux et le centre paramagnétique est plus courte à l’état de population transitoire qu’à l’état principal11. Le PRE transversal est noté Γ2 et, pour des raisons pratiques, est calculé à partir de la différence de vitesse de relaxation transversale 1H entre un échantillon contenant un centre paramagnétique et un contrôle diamagnétique. Pour un traitement approfondi de la théorie du PRE et des changements de pseudo-contact connexes dans les régimes d’échange rapide et lent, le lecteur est renvoyé aux examens complets de Clore et de ses collègues13,22. Ici, seule la situation où 1H N-Γ2 est dans le régime d’échange rapide est considérée, où, en raison de la dépendance r-6 du PRE, le taux de relaxation observé est lié à la fois à la distance à laquelle le centre paramagnétique s’approche du noyau ainsi qu’au temps qu’il passe dans cette conformation. Par conséquent, les conformations transitoires qui n’impliquent pas une approche rapprochée produisent un petit PRE tandis que des interactions plus étroites, même si elles sont de courte durée, produiront un PRE plus grand.
Pour les PDI, le PRE est utilisé pour mesurer et différencier les interactions qui se produisent au sein d’une seule molécule (intramoléculaire) et entre des molécules distinctes (intermoléculaires). En attachant un centre paramagnétique à une protéine RMN visible (p. ex., 15N-marquée) ou RMN invisible (p. ex., abondance naturelle 14N), la source (inter- ou intra-moléculaire) de l’ERP peut être déterminée (Figure 2). La mutagénèse dirigée qui introduit un résidu de cystéine est une approche pratique pour attacher un centre paramagnétique (spin-label) à une protéine23. Plusieurs types de molécules ont été proposés pour être utilisés comme marqueurs de spin, y compris la chélation des métaux (à base d’EDTA) et les radicaux libres (à base de nitroxide)24. Diverses étiquettes de spin de nitroxide ont été décrites et sont disponibles avec différentes chimies réactives à la cystéine telles que le méthanethiosulfonate, le maléimide et l’iodoacétamide25,26 (Figure 1). La flexibilité inhérente de l’étiquette ou de l’agent de liaison peut être problématique pour certaines analyses, et dans ces situations, différentes stratégies ont été proposées pour limiter le mouvement de l’étiquette, par exemple en ajoutant des groupes chimiques volumineux ou l’utilisation d’un deuxième agent de liaison pour ancrer l’étiquette à la protéine (fixation à deux sites)27, 28. De plus, les étiquettes disponibles dans le commerce peuvent contenir des protéines diastéréomères, mais cela ne contribuera généralement pas à l’ERP29 observée. L’utilisation du 3-Maleimido-PROXYL attaché à une cystéine libre via la chimie maléimide est décrite car il est facilement disponible, rentable, non réversible et l’agent réducteur tris(2-carboxyéthyl)phosphine (TCEP) peut être maintenu dans la solution tout au long de la réaction de marquage. Étant donné que le 3-maléimido-PROXYL possède un tenseur g isotrope, aucun PCS ou RDC n’est induit, et les mêmes assignations de décalage chimique peuvent être utilisées pour les échantillons paramagnétiques et diamagnétiques13.
Le 1HN-T 2 est mesuré à l’aide d’une stratégie à deux points temporels (T a, Tb) qui s’est déjà révélée aussi précise que la collecte d’une série d’évolution complète composée de 8 à 12 points temporels30. Le premier point temporel (T a) est fixé aussi près de zéro que possible, et la longueur optimale du deuxième point de temps dépend de l’amplitude du plus grand PRE attendu pour un échantillon donné et peut être estimée à partir de: Tb ~ 1,15 / (R 2,dia + Γ 2) où R 2,dia représente le R2 de l’échantillon diamagnétique13. Si la magnitude des plus grands PRE est inconnue, définir T b à ~ une fois le 1H T 2 de la protéine est une bonne estimation initiale et optimisée en ajustant T2 pour améliorer le signal au bruit. Cette stratégie de mesure en deux points réduit considérablement le temps expérimental requis pour mesurer les PRE et laisse le temps de faire plus de moyenne des signaux, d’autant plus que des échantillons relativement dilués sont utilisés pour minimiser les effets des contacts non spécifiques entre les molécules. Une séquence d’impulsions basée sur HSQC est utilisée pour mesurer 1HN-T 2 et a été décrite en détail ailleurs30. Pour améliorer la sensibilité, les impulsions dures des transferts INEPT avant et arrière peuvent être remplacées par des impulsions façonnées; alternativement, la séquence est facilement convertie en une lecture basée sur TROSY31. Étant donné que les PDI ont généralement des taux de relaxation transversale beaucoup plus longs, ce qui entraîne des largeurs de raies plus étroites (en raison du désordre inhérent) que les protéines globulaires de taille similaire, de longs temps d’acquisition dans la dimension indirecte peuvent être utilisés pour améliorer la résolution spectrale et atténuer la limitation de la dispersion par décalage chimique inhérente aux PDI.
Le PRE est un outil utile pour étudier les interactions protéine-protéine et protéine-acide nucléique, en particulier les interactions transitoires ou peu peuplées. Un protocole détaillé pour la préparation d’un échantillon RMN adapté à la mesure des PRE, y compris les étapes de purification des protéines, le marquage par centrifugation dirigé sur site, la mise en place et l’étalonnage du programme d’impulsions, le traitement et l’interprétation des données RMN, est fourni. D’importantes considérations expérimentales sont notées tout au long de la période qui peuvent avoir une incidence sur la qualité des données et les résultats expérimentaux, y compris la concentration de l’échantillon, la sélection de l’étiquette de spin et l’élimination des composants paramagnétiques.
Protocole
Exigences générales pour le protocole: installations de purification des protéines, spectromètre UV-Vis, spectromètre RMN à haut champ et logiciel d’exploitation, logiciel d’analyse post-traitement, y compris; NMRPipe32, Sparky 33, (ou CCPN Analysis 34, ou NMRViewJ35).
1. Expression recombinante et purification d’une protéine pour les mesures PRE
- Concevoir une construction d’expression pour la protéine d’intérêt afin qu’il y ait un seul résidu de cystéine présent. De multiples mutations seront nécessaires pour introduire une cystéine libre à différentes positions dans la protéine d’intérêt36.
- Exprimer et purifier un échantillon d’abondance naturelle (14N) ou 15N-marqué de la protéine d’intérêt en utilisant un protocole établi37.
REMARQUE: Les systèmes d’expression d’E. coli fournissent une méthode rentable et robuste pour l’expression de protéines recombinantes, car l’enrichissement isotopique de 15N est une exigence minimale pour la spectroscopie RMN hétéronucléaire biomoléculaire. Les étapes typiques sont l’expression dans un milieu minimal, la purification chromatographique et le retrait de l’étiquette de purification d’affinité. Ce protocole suppose qu’un protocole d’expression et de purification robuste a été établi qui peut produire suffisamment de protéines de qualité appropriée pour les investigations RMN.- Maintenir l’agent réducteur de 1 mM (DTT ou TCEP) dans les tampons à toutes les étapes de purification pour empêcher la réaction de la cystéine libre et la formation de liaisons disulfures intermoléculaires pour les PDI.
NOTE: Certains systèmes peuvent être plus tolérants et moins sujets à l’agrégation dans des conditions non réductrices en fonction des caractéristiques spécifiques de la protéine, ainsi que de la température, du pH et du système tampon choisi pour la purification38. - Retirez les étiquettes d’affinité utilisées pour la purification avant de continuer, car elles peuvent interagir de manière imprévisible avec la protéine ou éventuellement contenir des résidus de cystéine réactifs qui pourraient servir par inadvertance de site de fixation involontaire.
- Préparer un échantillon de référence marqué à 15N sans mutation(s) de cystéine mélangé à une version soluble de l’étiquette de spin pour évaluer la contribution des PRE solvants.
- Maintenir l’agent réducteur de 1 mM (DTT ou TCEP) dans les tampons à toutes les étapes de purification pour empêcher la réaction de la cystéine libre et la formation de liaisons disulfures intermoléculaires pour les PDI.
2. Conjuguer l’étiquette de spin de nitroxide 3-Maleimido-PROXYL
- Stocker ou échanger la protéine purifiée dans un tampon dégazé contenant 50 mM Tris pH 7 et 1 mM de PTCE ; le tampon peut également contenir jusqu’à 8 M d’urée si nécessaire pour faciliter la solubilité des protéines.
Sinon, diluer rapidement une solution mère de protéines dans au moins 10 équivalents volumiques de tampon de PTCE Tris pH 7 dégazé de 50 mM et 1 mM. Assurez-vous que la concentration de protéines avant d’ajouter le spin-label est d’au moins 100 μM. - Ajouter 3-Maleimido proxyl d’une solution mère à 20x l’excès molaire de la protéine d’intérêt. Protéger l’échantillon de la lumière et de l’oxygène et incuber pendant la nuit à température ambiante ou à 4 °C; Un balancement doux ou une nutation peut améliorer l’efficacité de l’étiquetage.
- Préparer les solutions mères de l’étiquette de spin en dissolvant la poudre de 3-Maleimido proxyl dans de l’éthanol à 95%. Les aliquotes du stock peuvent être conservées à -80 °C pendant moins de 6 mois.
- Étape critique : Retirer l’étiquette de spin libre n’ayant pas réagi pour éviter les PRE de solvants non spécifiques. Pour ce faire, vous pouvez le faire par filtration sur gel ou (de préférence) dialyse extensive de l’échantillon de protéines. Cette étape introduira également la protéine dans un tampon adapté à la RMN.
NOTA : Les agents réducteurs doivent être préparés frais et la compatibilité entre les composants tampons doit être prise en compte; par exemple, le PTCE se dégrade rapidement dans les tampons à base de phosphate, et cette combinaison doit être évitée39. - Traiter tous les tampons utilisés à partir de cette étape avec une résine chélatante sélective pour les métaux divalents et de transition afin d’éliminer les ions paramagnétiques ou les quenchers de spin-label. Si la protéine ne peut pas être stockée dans un tampon RMN, concentrez-la pour qu’elle soit rapidement diluée dans un tampon adapté à la RMN.
- Surveillance de l’efficacité de l’incorporation spin-label.
- Utiliser le réactif d’Ellman (acide 5,5-dithio-bis-(2-nitrobenzoïque)) pour quantifier les groupes sulfhydryles libres dans la solution40.
REMARQUE: Des protocoles détaillés sont disponibles auprès du fabricant. Aux fins de la présente étude, il est important de déterminer l’incorporation du spin-label, la concentration des groupes sulfhydryle libres est comparée à la concentration totale en protéines. Le pourcentage de groupes sulfhydryles libres est le pourcentage de molécules qui n’ont pas d’étiquette de spin de nitroxide attachée. - Surveillez l’intensité du pic correspondant au résidu de cystéine marqué pour juger de l’incorporation du spin-label dans la protéine d’intérêt.
REMARQUE: Il s’agit d’une approche rapide et efficace pour déterminer le degré de marquage de spin de la protéine. L’incorporation complète de l’étiquette de spin entraînera la disparition du pic du spectre. Compte tenu de la faible dispersion caractéristique des PDI, le pic correspondant au résidu de cystéine mutante peut ne pas toujours être facilement identifié, et l’utilisation du réactif d’Ellman (étape 2.6.1) est donc recommandée.
- Utiliser le réactif d’Ellman (acide 5,5-dithio-bis-(2-nitrobenzoïque)) pour quantifier les groupes sulfhydryles libres dans la solution40.
3. Préparer l’échantillon RMN pour mesurer l’ERP intra ou intermoléculaire
- Préparer l’échantillon pour la mesure de l’ERP intramoléculaire
- Préparer 15 protéines marquées par un spin enrichies en isotopie de 15N à une concentration d’au moins 100 μM mais pas plus de 300 μM dans un tampon adapté à la RMN. Le volume total de l’échantillon (y compris D2O) est compris entre 500 et 550 μL.
REMARQUE: Les tampons RMN courants comprennent le phosphate, l’acétate, le (bi)carbonate et le TRIS. Les tampons de Good tels que MES, HEPES peuvent également être appropriés. Faites preuve de prudence lors de la sélection des tampons pour éviter toute réactivité croisée avec d’autres composants de la solution. - Assurez-vous que le pH est ~7,2 ou inférieur pour minimiser les effets de l’échange amide de protons avec l’eau. Maintenez la concentration de sel aussi faible que possible (généralement inférieure à 150 mM), bien que la principale considération soit de maintenir la stabilité des protéines.
NOTA : Des approches pour mener des expériences de RMN dans des conditions à haute teneur en sel ont été décrites ailleurs41.
- Préparer 15 protéines marquées par un spin enrichies en isotopie de 15N à une concentration d’au moins 100 μM mais pas plus de 300 μM dans un tampon adapté à la RMN. Le volume total de l’échantillon (y compris D2O) est compris entre 500 et 550 μL.
- Préparer l’échantillon pour la mesure de l’ERP intermoléculaire
- Suivez cette étape ou l’étape 3.1; ils ne sont pas effectués simultanément. Préparer une protéine d’abondance naturelle de 14N, marquée par spin, dans le tampon RMN choisi.
- Préparer l’échantillon de protéines en mélangeant 15 N de protéines non marquées par spin enrichies isotopiquement avec des protéines marquées par spin d’abondance naturelle de 15à 50 % à 14N, de sorte que la concentration finale soit identique à celle de l’échantillon préparé en 3.1.1. Le volume total de l’échantillon (y compris D2O) est compris entre 500 et 550 μL.
- Optimiser empiriquement le rapport des protéines 15N et 14N pour chaque protéine étudiée. Les ratios de 1%, 5% et 20% de 14protéines marquées par N-spin sont de bons points de départ.
REMARQUE: Une accumulation du PRE en fonction de l’ajout de 14protéines marquées par le spin N indique un effet spécifique; l’EPR observée est spécifique à l’échantillon puisqu’elle dépend de la distance et de la population (comme discuté ci-dessus), et par conséquent, des ratios plus élevés de 14protéines marquées par un spin N seront nécessaires si l’interaction est particulièrement transitoire17.
- Transférer l’échantillon RMN (intra- ou intermoléculaire) dans un tube RMN de 5 mm qui convient à une utilisation dans des aimants à champ élevé à l’aide d’une pipette ou d’une micropipette en verre à longue tige (9 pouces). S’assurer que tous les échantillons RMN contiennent de 5 % à 10 % deD2Opour faciliter le verrouillage sur le terrain.
REMARQUE: Les tubes RMN qui utilisent des bouchons en polymère pour réduire le volume d’échantillon nécessaire ne sont pas recommandés pour les mesures PRE en raison de difficultés liées au calage efficace de l’échantillon.
4. Configurer le spectromètre RMN et expérimenter les paramètres spécifiques
- Faites preuve d’une extrême prudence lorsque vous travaillez autour de spectromètres RMN supraconducteurs à haut champ.
NOTE: Les dangers comprennent les blessures dues à l’accélération soudaine d’objets métalliques vers l’aimant, l’interférence avec les dispositifs médicaux implantés et l’asphyxie due à une libération soudaine de gaz N 2 et He2 en cas d’extinction de l’aimant. Les étapes suivantes supposent que le lecteur a suivi la formation requise, qu’il est conscient de ces dangers et d’autres dangers locaux et qu’il a reçu l’approbation du gestionnaire de l’installation pour utiliser le spectromètre RMN. En cas de doute sur une étape ou une instruction, consultez le gestionnaire de l’installation ou l’utilisateur expérimenté pour éviter d’éventuelles blessures ou dommages au spectromètre. - Les étapes suivantes supposent un spectromètre RMN commercial exécutant une version moderne du logiciel de contrôle d’acquisition. Téléchargez le programme pulse et les fichiers de paramètres et placez-les dans les répertoires appropriés.
REMARQUE: Un programme d’impulsions et un ensemble de paramètres adaptés à une utilisation avec un spectromètre Bruker et TopSpin (3.2 ou ultérieur) sont disponibles sur demande auprès des auteurs.- Étape critique : On suppose qu’il est familier avec l’installation de programmes d’impulsions RMN non natifs; Consultez le gestionnaire de l’installation ou un utilisateur expérimenté si nécessaire.
- Placez l’échantillon dans l’aimant, verrouillez le signal 2H à l’aide de la commande Verrouiller, réglez et faites correspondre le canal 1H selon les protocoles de l’installation (la procédure exacte dépendra si la sonde est équipée d’un module de réglage et de correspondance à distance).
- Ajustez les cales à l’aide du sous-programme topshim pour optimiser la suppression du signal de solvant.
- Calibrer les impulsions de 1H et 15N 90° à l’aide de méthodes standard.
- Calibrez l’impulsion de 1H à l’aide du programme popt (utilisez d’abord pulsecal pour estimer la longueur de l’impulsion).
- Calibrer l’impulsion de 15N par rapport à un échantillon étalon; Assurez-vous que cette valeur a été calibrée récemment en discutant avec un directeur technique ou un utilisateur expérimenté.
- Vous pouvez également calibrer l’impulsion de 15N sur l’échantillon en faisant varier l’une des impulsions à 90° d’une expérience HMQC jusqu’à ce qu’un signal nul soit atteint.
- Déterminez l’atténuation correcte des impulsions façonnées à l’aide de la sous-routine de l’outil de forme (stdisp).
- Ouvrez le fichier de forme d’impulsion approprié en cliquant sur l’icône du dossier. Les impulsions façonnées se trouvent dans la section des paramètres d’impulsion d’ACQUPARS.
- Chargez le fichier de définition d’impulsion et cliquez sur Analyser la forme d’onde > Intégrer la forme. Entrez l’impulsion dure calibrée de 1H 90°, la longueur d’impulsion de forme souhaitée et la rotation (90° ou 180°).
- Calculez le niveau de puissance de l’impulsion façonnée en ajoutant le changement de niveau de puissance à l’atténuation pour l’impulsion calibrée à 90°.
- Enregistrer une HSQC standard 1H, 15N (hsqcetfpf3gpsi) pour optimiser la largeur de balayage, la fréquence de la porteuse et vérifier la suppression de l’eau25.
- Ajustez la largeur de balayage et le nombre d’incréments de cotes indirectes à l’aide des commandes sw et td ou directement dans les boîtes de dialogue appropriées. En règle générale, pour la collecte des PRE, les largeurs spectrales sont choisies de manière à ce que le spectre ne soit pas replié.
5. Configurer l’expérience 1HN-T 2
- Calibrer les impulsions façonnées comme décrit ci-dessus (4.4.5-4.5.7). Les fichiers de paramètres d’impulsion façonnée pour l’expérience PRE sont Eburp2.1000 (impulsion de 90°), Reburp.1000 et Iburp2.1000. Entrez les longueurs d’impulsion calibrées dans la section des paramètres d’impulsion de l’onglet ACQUPARS .
- Cette expérience mesure le 1H N-T2 en utilisant l’approche à deux points de retard30.
- Définissez les délais en éditant le fichier vdlist, le premier délai (Ta) est défini sur 0,01 ms.
- Choisir le second délai, (T b) en utilisant la relation avec le PRE maximum attendu (T b ~ 1,15/(R2,dia + Γ2) où R2,dia représente le R2 de l’échantillon diamagnétique13. Sans connaissance préalable de l’ampleur de la contribution du PRE à la relaxation observée, un bon point de départ est de fixer T b à ~1x 1H T2.
- Ensuite, déterminez une valeur appropriée en comparant les premiers incréments (traités avec la commande efp) des spectres T a et T b et en ajustant T b de telle sorte que le signal diminue entre 40% et 50% de sa valeur initiale.
REMARQUE : Cette approche optimise le rapport signal/bruit spectral, une considération nécessaire pour les échantillons qui ne peuvent pas être fortement concentrés (< 50 μM). Les valeurs appropriées de Tb dépendent de l’échantillon, mais varient généralement de 8 à 40 ms pour une protéine de taille moyenne.
- Déterminez le nombre de points complexes à enregistrer et le nombre de balayages pour une moyenne de signal suffisante. Étant donné que les PDI ont 15 N T2 plus longs que les protéines repliées de taille comparable, des temps d’acquisition plus longs dans la dimension indirecte peuvent être utilisés.
NOTE: Cette valeur dépend des caractéristiques spécifiques de la protéine mais peut être approximativement estimée à partir du 15 N T2 et optimisée en surveillant la désintégration du signal dans le FID. Pour la dimension directe, 1024* points complexes (largeur de balayage de 13 ppm, temps d’acquisition de 112,6 ms) est suffisant pour la plupart des échantillons. - Utilisez la commande expt pour calculer la durée de l’expérience, puis démarrez l’expérience avec la commande zg.
6. Faire un échantillon diamagnétique en réduisant l’étiquette de spin avec de l’acide ascorbique
- Dissoudre l’ascorbate de sodium dans le tampon RMN et ajuster le pH pour qu’il corresponde au tampon RMN d’origine.
- Calculer la concentration du stock d’ascorbate de sodium de manière à ajouter un excès molaire d’ascorbate de 10 fois par rapport à la concentration de l’étiquette de spin avec le moins de changement possible du volume de l’échantillon. Par exemple, pour un échantillon de protéines de 100 μM, un stock d’ascorbate de 100 mM est approprié. Pour réduire l’étiquette d’essorage, il faudra ajouter 5,5 μL de solution mère d’acide ascorbique, ce qui ne représente que 1 % du volume total de l’échantillon.
- Ajouter la quantité requise d’acide ascorbique au tube RMN en plaçant une gouttelette sous le bord du tube, boucher le tube, retourner soigneusement le tube pour mélanger, puis tourner à 200-400 x g pendant 10-20 s dans une centrifugeuse à manivelle pour déposer l’échantillon au fond du tube.
- Enveloppez le tube RMN dans une feuille d’aluminium pour le protéger de la lumière et laisser la réaction se dérouler pendant au moins 3 heures.
- Noter 1H N-T2 sur l’échantillon diamagnétique en utilisant les mêmes paramètres que ceux utilisés pour l’échantillon paramagnétique.
- Recalibrez les impulsions. Cependant, ils n’auraient pas dû changer par rapport aux mesures paramagnétiques; S’ils sont significativement différents (> différence de 0,5 μs), tenir compte de la qualité de l’échantillon (p. ex. dégradation, précipitation).
- Assurez-vous que tous les paramètres d’acquisition, y compris les délais de relaxation spécifiés (vdlist), le nombre de scans factices, le nombre de scans collectés, le nombre de points complexes collectés, le temps d’acquisition, les largeurs de balayage et les fréquences porteuses restent les mêmes pour les échantillons diamagnétiques et paramagnétiques.
7. Traiter les spectres paramagnétiques et diamagnétiques
- Copiez les données sur l’ordinateur local ou le poste de travail sur lequel NMRPipe et Sparky sont installés et configurés. Créez un dossier nommé proc dans le répertoire de données d’expérience qui contient le fichier ser.
- Copiez les scripts NMRPipe fid.com, p3d.com et nmrproc.com dans le processus (les scripts de traitement sont disponibles sur demande auprès des auteurs).
- Utilisez le script fid.com pour convertir le format de données Bruker (ser) au format NMRPipe.
- Utilisez le script p3D.com pour diviser les plans pseudo3D en spectres individuels.
- Utilisez le script nmrproc.com pour lire la sortie du script fid.com, appliquer une fonction de suppression de solvant, une fonction de fenêtre, ajouter des zéros aux données brutes (remplissage zéro), appliquer une correction de phase, exécuter une transformation de Fourier, rogner les données pour les afficher et écrire les données traitées sur le disque. Le script produira un fichier pour chaque délai de relaxation enregistré (T a et Tb).
Remarque : Chacun de ces scripts est personnalisable pour optimiser le traitement pour les détails spécifiques de chaque expérience. Des tutoriels et des exemples d’ensembles de données sont inclus dans la distribution NMRPipe disponible sur le site Web NMRPipe32. NMRDraw peut être utilisé pour la visualisation spectrale pendant le traitement (par exemple, réglage des angles de phase appropriés, etc.). Les options disponibles pour les commandes NMRPipe peuvent être visualisées à l’aide de la commande nmrPipe -help.
8. Transférer les assignations de résonance et extraire les hauteurs de pointe
- Modifiez les informations d’en-tête de fichier pour chaque fichier de spectre (T a, Tb pour les échantillons paramagnétiques et diamagnétiques) à l’aide de la commande sethdr [nom du fichier] -ndim 2.
- Utilisez Sparky pour extraire les hauteursde pic 33 en suivant les étapes 8.3-8.5. D’autres logiciels, y compris NMRPipe (NMRDraw)32, CCPN Analysis34 et NMRViewJ35 sont également appropriés.
- Lisez les fichiers spectraux dans Sparky. À cette étape, l’ensemble de données consistera en un spectre pour chaque spectre de point temporel (T a, Tb), pour les échantillons paramagnétiques et diamagnétiques, mesuré pour chaque position du spin-marqueur dans la protéine.
- Utilisez Sparky pour sélectionner des pics (commande : F8, puis cliquer et glisser) et transférer des affectations à l’aide de l’outil de liste de pics de transfert à partir d’une liste de pics de référence.
NOTE: Les assignations par résonance de la protéine d’intérêt sont nécessaires pour l’interprétation spécifique à la séquence des PREobservés 36.- Définissez les contours des spectres paramagnétique et diamagnétique au même niveau. Assurez-vous de définir les contours de manière à ce que les spectres collectés après le délai n’excluent pas délibérément les pics, mais soient suffisamment élevés pour que les spectres Ta ne soient pas trop bruyants.
- Enregistrez les nouvelles listes de pics pour chaque spectre et incluez l’intensité de crête mesurée et le rapport signal sur bruit calculé par Sparky (commande: lt pour ouvrir la liste des pics, cliquez sur les options pour inclure les colonnes intensité et SNR, commande: enregistrer).
9. Extraire 1HN-T 2 taux pour chaque résidu et calculer PRE
- Importez les listes de pics dans un tableur ou un langage de programmation préféré tel que Python.
REMARQUE : Pour chaque position de spin-étiquette sur la protéine, l’ensemble de données sera composé de quatre listes de pics avec les intensités de pic associées, une de Ta et une de Tb pour les expériences paramagnétiques et diamagnétiques. - Calculer 1HN R2 pour les échantillons paramagnétiques et diamagnétiques à l’aide de l’équation:
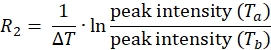
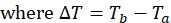
- Utilisez l’équation ci-dessus pour déterminer le taux de relaxation de chaque résidu pour les échantillons paramagnétiques et diamagnétiques.
- Déterminer le taux de 1H N-Γ2 pour chaque résidu à l’aide de l’équation suivante :
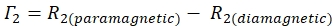
- Utilisez le rapport signal sur bruit (SNR) calculé par Sparky pour calculer l’incertitude de la hauteur maximale pour chaque résidu.
- Propagez l’erreur à l’aide de l’équation :

- Graphique 1HN-Γ 2 en fonction du nombre de résidus à l’aide d’un nuage de points comprenant l’erreur calculée au point 9.6.
Résultats
Des PRE intramoléculaires 1H N-Γ 2 ont été enregistrées sur un fragment auto-associatif, intrinsèquement désordonné (résidus 171-264) dérivé du domaine de faible complexité de la protéine de liaison à l’ARN EWSR142 (Figure 3). Les résidus à proximité séquentielle étroite du point de fixation de l’étiquette de spin (p. ex. résidus 178 ou 260 à la figure 3) devraient être ...
Discussion
Une méthode pour caractériser les interactions transitoires qui existent à de faibles populations entre des protéines intrinsèquement désordonnées et divers partenaires de liaison utilisant PRE a été présentée. Dans l’exemple montré, la protéine s’auto-associa, et donc le PRE peut provenir d’une combinaison d’interactions inter et intramoléculaires. Cette méthode est facilement étendue à des échantillons hétérogènes où les interactions entre deux protéines différentes peuvent être caract?...
Déclarations de divulgation
Tous les auteurs ont lu et approuvé le manuscrit. Aucun conflit d’intérêts n’est déclaré.
Remerciements
Nous remercions les Dres Jinfa Ying et Kristin Cano pour leurs discussions utiles et leur assistance technique. DSL est un boursier St. Baldrick’s et reconnaît le soutien de la St. Baldrick’s Foundation (634706). Ce travail a été soutenu en partie par la Welch Foundation (AQ-2001-20190330) à DSL, le Max and Minnie Tomerlin Voelcker Fund (Voelcker Foundation Young Investigator Grant à DSL), UTHSA Start-Up Funds to DSL et une bourse d’études supérieures Greehey en santé des enfants au CNJ. Ce travail est basé sur des recherches menées dans les installations de base de biologie structurale, qui font partie des noyaux de recherche institutionnelle du Centre des sciences de la santé de l’Université du Texas à San Antonio, avec le soutien du Bureau du vice-président pour la recherche et la ressource partagée de découverte de médicaments et de biologie structurale du Mays Cancer Center (NIH P30 CA054174).
matériels
| Name | Company | Catalog Number | Comments |
| 0.45 µm and 0.22 µm syringe filters | Millipore Sigma | SLHVM33RS SLGVR33RS | Filter lysate before first purification step and before size exclusion chromatography. |
| 100 mm Petri Dish | Fisher | FB0875713 | Agar plates for bacterial transformation. |
| 14N Ammonium chloride | Sigma Aldrich | 576794 | Use of 15N in M9 medium will produce an NMR visible protein, 14N will produces an NMR invisible protein |
| 15N Ammonium chloride | Sigma Aldrich | 299251 | Use of 15N in M9 medium will produce an NMR visible protein, 14N will produces an NMR invisible protein |
| 3 L Fernbach baffled flask | Corning | 431523 | Bacterial expression culture |
| 3-Maleimido-Proxyl | Sigma Aldrich | 253375 | Nitroxide spin label |
| 50 mL conical centrifuge tubes | Thermo Fisher | 14-432-22 | Solution/protein storage |
| Amicon centrifugal filter | Millipore Sigma | UFC900308 | Protein concentration |
| Ampicillan | Sigma Aldrich | A5354 | Antibiotic for a selective marker, exact choice depends on the expression construct plasmid |
| Analytical balance | Oahus | 30061978 | Explorer Pro, for weighing reagents |
| Ascorbic acid | Sigma Aldrich | AX1775 | Reduces nitroxide spin label |
| Autoclave | Sterilize glassware and culture media | ||
| Calcium chloride | Sigma Aldrich | C4901 | M9 media component |
| Centrifuge bottles | Thermo Fisher | 010-1459 | Harvest E. coli cells after recombinant protein expression |
| Centrifuge, hand-crank | Thomas Scientific | 0241C68 | Boekel hand-driven, low-speed centrifuge with 15 mL buckets that can accommodate NMR tubes |
| Chelex 100 | Sigma Aldrich | C7901 | Remove contaminating paramagnetic compounds from buffer solutions |
| Computer workstation | Linux or Mac OS compatable with NMR data processing and analysis software packages such as NMRPipe and Sparky | ||
| Deuterium oxide | Sigma Aldrich | 151882 | Needed for NMR lock signal |
| Dextrose | Sigma Aldrich | D9434 | M9 media component |
| Dibasic Sodium Phosphate | Sigma Aldrich | S5136 | M9 media component |
| Ellman's reagent (5,5-dithio-bis-(2-nitrobenzoic acid) | Thermo Fisher | 22582 | Quantification of free cystiene residues |
| High speed centrifuge tubes | Thermo Fisher | 3114-0050 | Used to clear bacterial lysate. |
| High-field NMR instrument (600 - 800 MHz) | Bruker | Equiped with a multichannel cryogenic probe and temperature control | |
| IMAC column, HisTrap FF | Cytvia | 17528601 | Initial fractionation of crude bacterial lysate |
| Isopropyl B-D-thiogalactoside (IPTG) | Sigma Aldrich | I6758 | Induces protein expression for genes under control of lac operator |
| LB agar | Thermo Fisher | 22700025 | Items are used for transforming E. coli to express protein of interest, substitions for any of these items with like products is acceptable. |
| LB broth | Thermo Fisher | 12780052 | |
| Low-pressure chromatography system | Bio-Rad | 7318300 | BioRad BioLogic is used for low-pressure chomatograph such as running IMAC columns |
| Magnesium sulfate | Sigma Aldrich | M7506 | M9 media component |
| Medium pressure chromatography system | Bio-Rad | 7880007 | BioRad NGC equipped with a multi-wavelength detector, pH and conductivity monitors, and automatic fraction collector |
| MEM vitamin solution | Sigma Aldrich | M6895 | M9 media component |
| Microfluidizer | Avestin | EmulsiFlex-C3 | Provides rapid and efficient bacterial cell lysis |
| Micropipettes | Thermo Fisher | Calibrated set of micropippetters with properly fitting disposable tips (available from multiple manufacturers e.g. Eppendorf) | |
| Monobasic potassium phosphate | Sigma Aldrich | 1551139 | M9 media component |
| NMR pipettes | Sigma Aldrich | 255688 | To remove sample from NMR tube |
| NMR sample tube | NewEra | NE-SL5 | Suitable for high-field NMR spectrometers |
| Preparative Centrifuge | Beckman Coulter | Avanti J-HC | Harvest E. coli cells after recombinant protein expression |
| Round bottom polystyrene centrifuge tubes | Corning | 352057 | Clear bacterial lysate |
| Shaking incubator | Eppendorf | S44I200005 | Temperature controlled growth of E. coli starter and expression cultures |
| Sodium chloride | Sigma Aldrich | S5886 | M9 media component |
| Sonicating water bath and vacuum source | Thomas Scientific | Used to degas buffer solutions | |
| Sonicator | Thermo Fisher | FB505110 | Used for bacterial cell lysis or shearing bacterial DNA |
| Spectrophotometer | Implen | OD600 Diluphotometer | Monitor growth of E.coli protein expression cultures |
| Superdex 200 16/600 size exculsion colum | Cytvia | 28989333 | Final protein purification step |
| Topspin software, version 3.2 or later | Bruker | Operating software for the NMR instrument | |
| Transformation competent E. coli cells | Thermo Fisher | C600003 | One Shot BL21 Star (DE3) chemically competent E. coli, other strains may be compatable |
| Tris(2-carboxyethyl)phosphine (TCEP) | ThermoFisher | 20490 | Reducing agent compatable with some sulfhydryl-reactive conjugations |
| UV-Vis spectrophotometer | Implen | NP80 | Measure protein concentration. |
| Water bath, temperature controlled | ThermoFisher | FSGPD25 | For heat shock step of bacterial transformation |
| Yeast extract | Sigma Aldrich | Y1625 | For supplementing M9 media if required |
Références
- Dyson, H. J., Wright, P. E. Intrinsically unstructured proteins and their functions. Nature Reviews: Molecular Cell Biology. 6 (3), 197-208 (2005).
- Korneta, I., Bujnicki, J. M. Intrinsic disorder in the human spliceosomal proteome. PLoS Computational Biology. 8 (8), 1002641 (2012).
- Frege, T., Uversky, V. N. Intrinsically disordered proteins in the nucleus of human cells. Biochemistry and Biophysics Reports. 1, 33-51 (2015).
- Liu, J., et al. Intrinsic disorder in transcription factors. Biochemistry. 45 (22), 6873-6888 (2006).
- El Hadidy, N., Uversky, V. N. Intrinsic disorder of the BAF complex: Roles in chromatin remodeling and disease development. International Journal of Molecular Sciences. 20 (21), (2019).
- Wright, P. E., Dyson, H. J. Intrinsically disordered proteins in cellular signalling and regulation. Nature Reviews: Molecular Cell Biology. 16 (1), 18-29 (2015).
- Brangwynne, C. P. Phase transitions and size scaling of membrane-less organelles. Journal of Cell Biology. 203 (6), 875-881 (2013).
- Shin, Y., Brangwynne, C. P. Liquid phase condensation in cell physiology and disease. Science. 357 (6357), (2017).
- Cavanagh, J. . Protein NMR spectroscopy : principles and practice. 1st edition. , (2018).
- Sekhar, A., Kay, L. E. NMR paves the way for atomic level descriptions of sparsely populated, transiently formed biomolecular conformers. Proceedings of the National Academy of Sciences of the United States of America. 110 (32), 12867-12874 (2013).
- Anthis, N. J., Clore, G. M. Visualizing transient dark states by NMR spectroscopy. Quarterly Reviews of Biophysics. 48 (1), 35-116 (2015).
- Alderson, T. R., Kay, L. E. NMR spectroscopy captures the essential role of dynamics in regulating biomolecular function. Cell. 184 (3), 577-595 (2021).
- Clore, G. M., Iwahara, J. Theory, practice, and applications of paramagnetic relaxation enhancement for the characterization of transient low-population states of biological macromolecules and their complexes. Chemical Reviews. 109 (9), 4108-4139 (2009).
- Wu, K. P., Baum, J. Detection of transient interchain interactions in the intrinsically disordered protein alpha-synuclein by NMR paramagnetic relaxation enhancement. Journal of the American Chemical Society. 132 (16), 5546-5547 (2010).
- Janowska, M. K., Wu, K. P., Baum, J. Unveiling transient protein-protein interactions that modulate inhibition of alpha-synuclein aggregation by beta-synuclein, a pre-synaptic protein that co-localizes with alpha-synuclein. Scientific Reports. 5, 15164 (2015).
- Murthy, A. C., et al. Molecular interactions underlying liquid-liquid phase separation of the FUS low-complexity domain. Nature Structural & Molecular Biology. 26 (7), 637-648 (2019).
- Fawzi, N. L., Doucleff, M., Suh, J. Y., Clore, G. M. Mechanistic details of a protein-protein association pathway revealed by paramagnetic relaxation enhancement titration measurements. Proceedings of the National Academy of Sciences of the United States of America. 107 (4), 1379-1384 (2010).
- Griffith, O. H., Waggoner, A. S. Nitroxide free radicals: spin labels for probing biomolecular structure. Accounts of Chemical Research. 2 (2), 17-24 (1969).
- Bertini, I., Luchinat, C., Parigi, G., Ravera, E. . NMR of Paramagnetic Macromolecules, Applications to Metallobiomolecules and Models. 2 edn. , (2016).
- Bloembergen, N., Purcell, E. M., Pound, R. V. Relaxation effects in nuclear magnetic resonance absorption. Physical Review. 73 (7), 679-712 (1948).
- Solomon, I. Relaxation processes in a system of two spins. Physical Review. 99 (2), 559 (1955).
- Clore, G. M. Practical aspects of paramagnetic relaxation enhancement in biological macromolecules. Methods in Enzymology. 564, 485-497 (2015).
- Klare, J. P. Site-directed spin labeling EPR spectroscopy in protein research. Biological Chemistry. 394 (10), 1281-1300 (2013).
- Clore, G. M., Tang, C., Iwahara, J. Elucidating transient macromolecular interactions using paramagnetic relaxation enhancement. Current Opinion in Structural Biology. 17 (5), 603-616 (2007).
- Melanson, M., Sood, A., Torok, F., Torok, M. Introduction to spin label electron paramagnetic resonance spectroscopy of proteins. Biochemistry and Molecular Biology Education. 41 (3), 156-162 (2013).
- Czogalla, A., Pieciul, A., Jezierski, A., Sikorski, A. F. Attaching a spin to a protein -- site-directed spin labeling in structural biology. Acta Biochimica Polonica. 54 (2), 235-244 (2007).
- Lindfors, H. E., de Koning, P. E., Drijfhout, J. W., Venezia, B., Ubbink, M. Mobility of TOAC spin-labelled peptides binding to the Src SH3 domain studied by paramagnetic NMR. Journal of Biomolecular NMR. 41 (3), 157-167 (2008).
- Fawzi, N. L., et al. A rigid disulfide-linked nitroxide side chain simplifies the quantitative analysis of PRE data. Journal of Biomolecular NMR. 51 (1-2), 105-114 (2011).
- Bleicken, S., et al. gem-Diethyl pyrroline nitroxide spin labels: Synthesis, EPR characterization, rotamer libraries and biocompatibility. ChemistryOpen. 8 (8), 1035 (2019).
- Iwahara, J., Tang, C., Clore, G. M. Practical aspects of 1H transverse paramagnetic relaxation enhancement measurements on macromolecules. Journal of Magnetic Resonance. 184, 185-195 (2007).
- Venditti, V., Fawzi, N. L. Probing the atomic structure of transient protein contacts by paramagnetic relaxation enhancement solution NMR. Methods in Molecular Biology. 1688, 243-255 (2018).
- Delaglio, F., et al. NMRPipe: a multidimensional spectral processing system based on UNIX pipes. Journal of Biomolecular NMR. 6 (3), 277-293 (1995).
- Lee, W., Tonelli, M., Markley, J. L. NMRFAM-SPARKY: enhanced software for biomolecular NMR spectroscopy. Bioinformatics. 31 (8), 1325-1327 (2015).
- Vranken, W. F., et al. The CCPN data model for NMR spectroscopy: development of a software pipeline. Proteins. 59 (4), 687-696 (2005).
- Johnson, B. A. Using NMRView to visualize and analyze the NMR spectra of macromolecules. Methods in Molecular Biology. 278, 313-352 (2004).
- Sjodt, M., Clubb, R. T. Nitroxide labeling of proteins and the determination of paramagnetic relaxation derived distance restraints for NMR studies. Bio-Protocol. 7 (7), (2017).
- Zhang, H., van Ingen, H. Isotope-labeling strategies for solution NMR studies of macromolecular assemblies. Current Opinion in Structural Biology. 38, 75-82 (2016).
- Rabdano, S. O., et al. Onset of disorder and protein aggregation due to oxidation-induced intermolecular disulfide bonds: case study of RRM2 domain from TDP-43. Scientific Reports. 7 (1), 11161 (2017).
- Burns, J. A., Butler, J. C., Moran, J., Whitesides, G. M. Selective reduction of disulfides by tris(2-carboxyethyl)phosphine. Journal of Organic Chemistry. 56 (8), 2648-2650 (1991).
- Ellman, G. L. Tissue sulfhydryl groups. Archives of Biochemistry and Biophysics. 82 (1), 70-77 (1959).
- Binbuga, B., Boroujerdi, A. F., Young, J. K. Structure in an extreme environment: NMR at high salt. Protein Science. 16 (8), 1783-1787 (2007).
- Schwartz, J. C., Cech, T. R., Parker, R. R. Biochemical properties and biological functions of FET proteins. Annual Review of Biochemistry. 84, 355-379 (2015).
- Nabuurs, S. M., de Kort, B. J., Westphal, A. H., van Mierlo, C. P. Non-native hydrophobic interactions detected in unfolded apoflavodoxin by paramagnetic relaxation enhancement. European Biophysics Journal. 39 (4), 689-698 (2010).
- Wiedemann, C., Kumar, A., Lang, A., Ohlenschlager, O. Cysteines and disulfide bonds as structure-forming units: Insights from different domains of life and the potential for characterization by NMR. Frontiers in Chemistry. 8, 280 (2020).
- Wommack, A. J., et al. NMR solution structure and condition-dependent oligomerization of the antimicrobial peptide human defensin 5. Biochemistry. 51 (48), 9624-9637 (2012).
- Taylor, A. M., et al. Detailed characterization of cysteine-less P-glycoprotein reveals subtle pharmacological differences in function from wild-type protein. British Journal of Pharmacology. 134 (8), 1609-1618 (2001).
- Hu, K., Doucleff, M., Clore, G. M. Using multiple quantum coherence to increase the 15N resolution in a three-dimensional TROSY HNCO experiment for accurate PRE and RDC measurements. Journal of Magnetic Resonance. 200 (2), 173-177 (2009).
- Anthis, N. J., Doucleff, M., Clore, G. M. Transient, sparsely populated compact states of apo and calcium-loaded calmodulin probed by paramagnetic relaxation enhancement: interplay of conformational selection and induced fit. Journal of the American Chemical Society. 133 (46), 18966-18974 (2011).
- Battiste, J. L., Wagner, G. Utilization of site-directed spin labeling and high-resolution heteronuclear nuclear magnetic resonance for global fold determination of large proteins with limited nuclear overhauser effect data. Biochemistry. 39 (18), 5355-5365 (2000).
- Donaldson, L. W., et al. Structural characterization of proteins with an attached ATCUN motif by paramagnetic relaxation enhancement NMR spectroscopy. Journal of the American Chemical Society. 123 (40), 9843-9847 (2001).
- Gaponenko, V., et al. Protein global fold determination using site-directed spin and isotope labeling. Protein Science. 9 (2), 302-309 (2000).
- Trindade, I. B., Invernici, M., Cantini, F., Louro, R. O., Piccioli, M. PRE-driven protein NMR structures: an alternative approach in highly paramagnetic systems. FEBS Journal. 288 (9), 3010-3023 (2021).
- Nitsche, C., Otting, G. Pseudocontact shifts in biomolecular NMR using paramagnetic metal tags. Progress in Nuclear Magnetic Resonance Spectroscopy. 98-99, 20-49 (2017).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon