Method Article
التشريح المجهري بالليزر الصناعي الموجه بالذكاء الاصطناعي للتحليل البروتيني المجهري للبيئة الدقيقة للورم
* These authors contributed equally
In This Article
Summary
يصف هذا البروتوكول سير عمل عالي الإنتاجية للتجزئة القائمة على الذكاء الاصطناعي للمناطق ذات الأهمية المؤكدة من قبل علم الأمراض من صور قسم الأنسجة الرقيقة الملطخة لإثراء مجموعات الخلايا التي تم حلها بواسطة علم الأنسجة باستخدام التشريح المجهري بالليزر. تتضمن هذه الاستراتيجية خوارزمية جديدة تمكن من نقل الحدود التي تشير إلى مجموعات الخلايا ذات الأهمية المباشرة إلى المجاهر الليزرية.
Abstract
تمثل البيئة الدقيقة للورم (TME) نظاما بيئيا معقدا يتكون من عشرات أنواع الخلايا المتميزة ، بما في ذلك الورم والسدى والخلايا المناعية. لتوصيف التباين على مستوى البروتيوم وعدم تجانس الورم على نطاق واسع ، هناك حاجة إلى طرق عالية الإنتاجية لعزل المجموعات الخلوية المنفصلة بشكل انتقائي في الأورام الخبيثة الصلبة. يصف هذا البروتوكول سير عمل عالي الإنتاجية ، يتم تمكينه بواسطة الذكاء الاصطناعي (الذكاء الاصطناعي) ، والذي يقسم صور أقسام الأنسجة الرقيقة الملطخة بالهيماتوكسيلين والإيوسين (H & E) إلى مناطق ذات أهمية مؤكدة من قبل علم الأمراض للحصاد الانتقائي لمجموعات الخلايا التي تم حلها بواسطة علم الأنسجة باستخدام التشريح المجهري بالليزر (LMD). تتضمن هذه الاستراتيجية خوارزمية جديدة تمكن من نقل المناطق التي تشير إلى مجموعات الخلايا ذات الأهمية ، والتي تم شرحها باستخدام برنامج الصور الرقمية ، مباشرة إلى مجاهر الليزر ، وبالتالي تمكين مجموعات أكثر سهولة. تم تنفيذ سير العمل هذا بنجاح ، مما يدل على فائدة هذه الطريقة المنسقة لحصاد مجموعات الخلايا السرطانية بشكل انتقائي من TME للتحليل البروتيني الكمي متعدد الإرسال بواسطة مطياف الكتلة عالي الدقة. تتكامل هذه الاستراتيجية تماما مع المراجعة الروتينية لعلم الأنسجة المرضي ، والاستفادة من تحليل الصور الرقمية لدعم إثراء المجموعات الخلوية ذات الأهمية وهي قابلة للتعميم بالكامل ، مما يتيح حصادا منسقا لمجموعات الخلايا من TME للتحليلات المتعددة.
Introduction
يمثل TME نظاما بيئيا معقدا تسكنه مجموعة متنوعة للغاية من أنواع الخلايا ، مثل الخلايا السرطانية والخلايا اللحمية والخلايا المناعية والخلايا البطانية وأنواع الخلايا الوسيطة الأخرى والخلايا الشحمية ، إلى جانب مصفوفة معقدة خارج الخلية1. يختلف هذا النظام البيئي الخلوي داخل وعبر مواقع أعضاء المرض المختلفة ، مما يؤدي إلى عدم تجانس الورم المعقد 2,3. أظهرت الدراسات الحديثة أن الأورام غير المتجانسة والأورام ذات الخلايا السرطانية المنخفضة (النقاء المنخفض) غالبا ما ترتبط بسوء تشخيص المرض 2,3.
لفهم التفاعل الجزيئي بين مجموعات الخلايا السرطانية وغير السرطانية داخل TME على نطاق واسع ، تعد الاستراتيجيات الموحدة وعالية الإنتاجية ضرورية لحصاد مجموعات خلوية متميزة ذات أهمية انتقائية للتحليل متعدد الخلايا في المراحل النهائية. تمثل البروتينات الكمية تقنية سريعة التطور ومتزايدة الأهمية لتعزيز فهم بيولوجيا السرطان. حتى الآن ، فإن غالبية الدراسات التي تستخدم البروتيوميات قد فعلت ذلك مع البروتينات المستخرجة من مستحضرات أنسجة الورم الكاملة (على سبيل المثال ، المسحوقة بالتبريد) ، مما أدى إلى ندرة في فهم عدم التجانس على مستوى البروتيوم في TME 4,5,6.
إن تطوير استراتيجيات جمع العينات التي تتكامل بسلاسة مع المعلومات المستمدة من سير عمل علم الأمراض السريري وتسخرها سيمكن جيلا جديدا من البروتينات التي تم حلها في علم الأنسجة والتي تعد مكملة للغاية لسير عمل علم الأمراض التشخيصي ذي المعيار الذهبي. يتيح LMD الجمع المباشر والانتقائي للسكان الفرعيين الخلويين أو المناطق ذات الأهمية (ROIs) من خلال الفحص المجهري للأقسام الرقيقة من الأنسجة الملطخة نسيجيا7. أظهرت التطورات الرئيسية الحديثة في علم الأمراض الرقمي والتحليل المدعوم الذكاء الاصطناعي القدرة على تحديد الميزات التركيبية الفريدة وعائد الاستثمار داخل TME بطريقة آلية ، يرتبط الكثير منها بالتغيرات الجزيئية وميزات الأمراض السريرية ، مثل مقاومة العلاج وتشخيص المرض8.
يستفيد سير العمل الموصوف في البروتوكول المعروض هنا من حلول البرمجيات التجارية لتعليق عائد الاستثمار على الورم بشكل انتقائي داخل صور علم الأنسجة الرقمية ، ويستخدم أدوات برمجية تم تطويرها داخليا لنقل عائد الاستثمار على الورم إلى مجاهر الليزر للجمع الآلي للمجموعات الخلوية المنفصلة ذات الأهمية التي تتكامل بسلاسة مع سير عمل التحليل متعدد الأورام في المراحل النهائية. تقلل هذه الاستراتيجية المتكاملة بشكل كبير من وقت مشغل LMD وتقلل من المدة التي يلزم فيها أن تكون الأنسجة في درجة الحرارة المحيطة. يتم إثبات تكامل الاختيار الآلي للميزات وحصاد LMD مع البروتينات الكمية عالية الإنتاجية من خلال تحليل تفاضلي ل TME من نوعين فرعيين نسيجيين تمثيليين لسرطان المبيض الظهاري ، سرطان المبيض المصلي عالي الجودة (HGSOC) وسرطان الخلايا المبيض الشفاف (OCCC).
Protocol
تمت الموافقة على جميع بروتوكولات الدراسة للاستخدام بموجب بروتوكول غربي معتمد من IRB "تحليل جزيئي متكامل لسرطان بطانة الرحم والمبيض لتحديد والتحقق من صحة المؤشرات الحيوية الإعلامية سريريا" التي تعتبر معفاة بموجب اللائحة الفيدرالية الأمريكية 45 CFR 46.102 (f). كانت جميع البروتوكولات التجريبية التي تتضمن بيانات بشرية في هذه الدراسة متوافقة مع إعلان هلسنكي. وتم الحصول على الموافقة المستنيرة من جميع الأشخاص المشاركين في الدراسة.
تحذير: الكواشف التالية المستخدمة في جميع أنحاء البروتوكول هي مواد مسرطنة معروفة أو مشتبه بها و / أو تحتوي على مواد خطرة: الإيثانول ، ماء DEPC ، محلول الهيماتوكسيلين من ماير ، محلول Eosin Y ، الميثانول ، الأسيتونيتريل ، وحمض الفورميك. ويعد التعامل السليم مع الآخرين، كما هو موضح في صحائف بيانات السلامة ذات الصلة، واستخدام معدات الحماية الشخصية المناسبة أمرا إلزاميا.
1. إنشاء ملف بيانات قائمة الأشكال الافتراضية (.sld) الذي يحتوي على بيانات معايرة
ملاحظة: خطوات البروتوكول الموضحة في هذا القسم محددة للاستخدام مع مجهر ليزر مقلوب والبرامج المرتبطة به (انظر جدول المواد). إنشاء ملف .sld افتراضي ضروري مرة واحدة فقط لكل مجهر ليزر. يمكن استخدام الملف الناتج لقطع fiducials في جميع شرائح PEN المستخدمة بعد ذلك. الوقت التقريبي: 5 دقائق (مرة واحدة فقط).
- افتح برنامج LMD وقم بتحميل غشاء البولي إيثيلين النفثالات (PEN) على مرحلة LMD متجها لأسفل ، مع الملصق الأقرب إلى المستخدم. قم بإلغاء تحديد المربع إغلاق الخط (الأسطر) على الجانب الأيسر من نافذة البرنامج.
- استخدم الدالة PtoP (من نقطة إلى نقطة) تحت التكبير العالي (63x) لرسم ثلاثة أسهم "V" لتكون بمثابة ائتمانات معايرة. بدءا من نقطة خارجية واحدة على V ، ارسم خطا إلى نقطة منتصف V ونقرة واحدة. ثم ارسم خطا ثانيا من النقطة المركزية V إلى نهاية النقطة V الخارجية الثانية، وانقر نقرا مزدوجا لإنشاء شكل V واحد غير مغلق من الخطين.
ملاحظة: يجب وضع هذه المعايرة الائتمانية في ثلاث زوايا من الشريحة: اليمين الأمامي واليمين الخلفي واليسار الخلفي. - حدد خيار التركيز البؤري التلقائي (AF) قبل القص . قم بقص الشريحة في الموضع 1، وحركها في كل موضع من مواضع الشرائح المتبقية، وتتبع بدقة عمليات المعايرة.
- احفظ ملف .sld وحدد خيار حفظ بدون معايرة من مربع الحوار المنبثق لتجنب قطع ائتمانات المعايرة في الغشاء.
ملاحظة: يتم توفير ملف .sld تمثيلي يحتوي على اعتمادات معايرة قياسية لأربعة مواضع شرائح في الملف التكميلي 1.
2. إعداد شريحة (شرائح) LMD
ملاحظة: خطوات البروتوكول الموضحة في هذا القسم محددة للاستخدام مع مجهر ليزر مقلوب والبرامج المرتبطة به (انظر جدول المواد). الوقت التقريبي: 5 دقائق.
- تأكد من أن الشريحة جافة تماما قبل قطع المعايرة المرجعية. افتح برنامج LMD وافتح ملف .sld الافتراضي للمعايرة ضمن الخيار استيراد الأشكال .
- حدد خيار التركيز البؤري التلقائي (AF) قبل القص . قم بتحميل الشريحة (الشريحات) مع توجيه الأنسجة لأسفل وانزلاق الملصق بالقرب من المشغل إلى حامل الشريحة على مرحلة LMD.
- باستخدام مجهر الليزر وملف .sld للمعايرة الافتراضية ، قم بقطع ائتمانات المعايرة في غشاء PEN.
- اختياري: قم بقص المقاطع الائتمانية للمعايرة في غشاء القلم إما قبل أو بعد وضع قسم (أقسام) الأنسجة على الشريحة. إذا تم قطع المعايرة الائتمانية قبل وضع الأنسجة، فتأكد من عدم تداخل الأنسجة و/أو المثبت مع المعايرات عند وضع النسيج على الشريحة في الخطوة 2.5. إذا تم قطع معايرة fiducials بعد وضع الأنسجة، فتوقف بعد الانتهاء من الخطوة 2.4 وانتقل إلى القسم 3.
- راجع جميع أجهزة المعايرة بشكل فردي للتأكد من أن كل عملية قص كاملة ومرئية.
ملاحظة: استخدم ميزة " تحريك وقطع" لتوجيه الليزر يدويا فوق أي اعتمادات معايرة لم تقطع غشاء القلم بالكامل. - ضع قسم الأنسجة المجمدة أو المثبتة بالفورمالين والمدمجة في البارافين (FFPE) على الشريحة التي تحتوي على بيانات المعايرة.
3. تلطيخ الأنسجة
ملاحظة: الوقت التقريبي: 30 دقيقة.
- إصلاح شرائح الأنسجة LMD المجمدة في 70٪ الإيثانول (EtOH) التي تحتوي على كواشف كوكتيل مثبطات الفوسفاتيز لمدة 5 دقائق.
- اغسل الشرائح في ماء ثنائي إيثيل بيروكاربومات (DEPC) الذي يحتوي على كواشف كوكتيل مثبطات الفوسفاتيز لمدة 1 دقيقة.
- اغسل الشرائح في ماء DEPC لمدة 1 دقيقة.
- احتضان الشرائح في محلول الهيماتوكسيلين من ماير لمدة 3 دقائق.
- اشطف الشرائح في ماء DEPC لمدة 3 دقائق.
- شطف الشرائح في تبادل جديد من الماء DEPC لمدة 1 دقيقة.
- احتضان الشرائح في محلول Eosin Y المائي لمدة 1 ثانية.
- شطف الشرائح 2 × 5 ثانية في 95٪ EtOH.
- شطف الشرائح 3 × 10 ثانية في 100٪ EtOH.
- امسح EtOH الزائد من الجزء الخلفي من الشرائح واترك الشرائح لتجف في الهواء.
- قم بتخزين الشرائح عند -80 درجة مئوية إذا لم يتم تنفيذ LMD على الفور.
4. تصوير الشرائح
ملاحظة: خطوات البروتوكول الموضحة في هذا القسم خاصة بالشرائح التي تم مسحها ضوئيا (راجع جدول المواد) والصور الناتجة المحفوظة كملفات .svs. استخدم أي ماسح ضوئي والبرامج المرتبطة به التي تنشئ ملفات صور بتنسيق يمكن لبرنامج تحليل الصور (انظر جدول المواد) فتحه. تتضمن أنواع الملفات التي تستخدم المقاطع الهرمية المضغوطة المدعومة JPG ، TIF ، MRXS ، QPTIFF ، المكون TIFF ، SVS، AFI، SCN، LIF، DCM، OME. TIFF و ND2 و VSI و NDPI و NDPIS و CVI و BIF و KFB و ISYNTAX. الوقت التقريبي: 5 دقائق.
- قم بتشغيل الماسحة الضوئية وافتح برنامج الماسح الضوئي للشرائح. قم بتحميل الشريحة بحيث تكون الأنسجة متجهة لأعلى إلى مرحلة الشريحة الواحدة في الماسحة الضوئية. تأكد من أن الشريحة جافة تماما ووضع غطاء بلطف على المنديل. لا تستخدم الإيثانول أو زيت الغمر تحت الغطاء.
- التقط صورة المجهر باستخدام الإعدادات التي تمت معايرتها لضبط غشاء PEN بدلا من الخلفية الزجاجية وتجاهل تلطيخ غشاء الخلفية ، وفقا لتعليمات الشركة المصنعة.
- اضبط منطقة التصوير عن طريق سحب المحيط الأخضر الداخلي وتغيير حجمه لالتقاط منطقة غشاء القلم بالكامل، حسب الحاجة. أضف أربع نقاط تركيز بؤري على الأنسجة بالنقر المزدوج على صورة نظرة عامة على اللقطة، وثلاث نقاط تركيز على الغشاء بالقرب من ائتمانات المعايرة (نقطة تركيز بؤري واحدة لكل من مؤشرات المعايرة الثلاثة).
ملاحظة: يمكن وضع نقاط التركيز البؤري الأربع في أي مكان تقريبا على قسم الأنسجة، على الرغم من أن وضعها على أنسجة ملطخة بلون داكن جدا وتبدو سوداء قد يتسبب في فشل الفحص. - ضمن القائمة عرض ، حدد شاشة الفيديو. اضبط التركيز البؤري يدويا باستخدام شريط تمرير التركيز البؤري الدقيق و/أو الماكرو، حسب الحاجة، لكل نقطة حول نسيج LMD. التقط المسح الضوئي للصورة تحت تكبير عال (20x). تأكد من أن جميع اعتمادات المعايرة مرئية وواضحة في الصورة المحفوظة.
5. اختيار الميزات الآلي باستخدام برنامج تحليل الصور
- لمجموعات الورم بأكملها (الوقت التقريبي: 5 دقائق ؛ حسب الحالة):
- افتح برنامج تحليل الصور (انظر جدول المواد). حدد فتح الصور ومن النافذة المنبثقة، حدد ملف صورة .svs الذي تم إنشاؤه من مسح الشريحة ضوئيا على الماسح الضوئي AT2.
ملاحظة: يتم توفير ملف صورة .svs تمثيلي في الملف التكميلي 2. - انتقل إلى علامة التبويب التعليقات التوضيحية . حدد أداة القلم على شريط أدوات التعليق التوضيحي وارسم شكلا حول الأنسجة.
- حدد الشكل وانقر بزر الماوس الأيمن على الصورة. من القائمة المنسدلة خيارات متقدمة، حدد التقسيم (البلاط)". اضبط حجم البلاط والمسافة بين 500 و40 على التوالي، وحدد موافق لإنشاء التجانبات. حدد وحذف شكل المحيط المستخدم لإنشاء التجانبات في الخطوة 5.1.2.
- حدد القائمة المنسدلة إجراءات الطبقة | تصدير لحفظ التعليقات التوضيحية المتجانبة كملف .annotation.
ملاحظة: يتم توفير ملف .annotation تمثيلي لمجموعة أنسجة الورم بأكملها في الملف التكميلي 3. - قم بإنشاء مجلد لجلسة العمل أو المشروع، واحفظ ملف .annotation داخل مجلد فرعي يحمل المعرف الفريد للشريحة.
- انتقل إلى علامة التبويب التعليقات التوضيحية . حدد القائمة المنسدلة إجراءات الطبقة | احذف كل الطبقات لإزالة كل التعليقات التوضيحية من الصورة. حدد أداة القلم وارسم خطا قصيرا من الطرف الداخلي لرأس السهم لكل عملية معايرة ائتمانية. ارسم الخطوط من العلامات بالترتيب التالي: أعلى اليسار ، أعلى اليمين ، أسفل اليمين.
- حدد القائمة المنسدلة إجراءات الطبقة | تصدير لحفظ التعليقات التوضيحية للأسطر كملف .annotation. أضف _calib إلى اسم الملف وضع الملف في المجلد الفرعي الذي يحتوي على إحداثيات الأشكال المتجانبة.
ملاحظة: يتم توفير ملف _calib.annotation تمثيلي في الملف التكميلي 4. - انسخ عنوان المشروع الرئيسي أو مجلد جلسة العمل. افتح البرنامج النصي لتوليد استيراد XML، "Malleator" (متوفر عبر https://github.com/GYNCOE/Mitchell.et.al.2022)، باستخدام بيئة التطوير المتكاملة IDLE والصق عنوان مجلد المشروع بين علامات الاقتباس في أسفل البرنامج النصي.
- حدد القائمة المنسدلة تشغيل | قم بتشغيل الوحدة النمطية لتنفيذ البرنامج النصي.
ملاحظة: سيتم إنشاء ملف استيراد LMD .xml داخل المجلد الفرعي الذي تم إنشاؤه للصورة/الشريحة. يتم توفير ملف .xml تمثيلي في الملف التكميلي 5.
- افتح برنامج تحليل الصور (انظر جدول المواد). حدد فتح الصور ومن النافذة المنبثقة، حدد ملف صورة .svs الذي تم إنشاؤه من مسح الشريحة ضوئيا على الماسح الضوئي AT2.
- بالنسبة للمجموعات الغنية ب LMD فقط (الوقت التقريبي: 15 دقيقة؛ حسب الحالة):
- افتح برنامج تحليل الصور (انظر جدول المواد). حدد فتح الصور ومن النافذة المنبثقة، حدد ملف صورة .svs الذي تم إنشاؤه من مسح الشريحة.
- انتقل إلى علامة التبويب التعليقات التوضيحية . حدد أداة التعليق التوضيحي المستطيل واستخدمها لرسم مربع حول الأنسجة.
- حدد التعليق التوضيحي للمربع وانقر بزر الماوس الأيمن على الصورة. حدد القائمة المنسدلة خيارات متقدمة | خيار التقسيم (البلاط). اضبط حجم البلاط والمسافة بين 500 و40 على التوالي، وحدد موافق لإنشاء التجانبات. حدد التعليق التوضيحي لمربع المحيط المستخدم لإنشاء التجانبات في الخطوة 5.2.2 واحذفه.
- حدد القائمة المنسدلة إجراءات الطبقة | تصدير لحفظ التعليقات التوضيحية المتجانبة كملف .annotation.
- ضع نسخة محفوظة من خوارزمية Python "Dapọ" (المتوفرة عبر https://github.com/GYNCOE/Mitchell.et.al.2022) ، التي تم تطويرها لدمج طبقات التعليقات التوضيحية المصنفة الذكاء الاصطناعي ، في نفس المجلد مثل ملف التعليقات التوضيحية المتجانبة.
- انسخ اسم ملف التعليقات التوضيحية المتجانبة. افتح برنامج Python باستخدام بيئة التطوير المتكاملة IDLE والصق اسم ملف التعليقات التوضيحية المتجانبة بين علامات الاقتباس الموجودة أسفل البرنامج.
- حدد القائمة المنسدلة تشغيل | تشغيل الوحدة النمطية. انتظر حتى يتم إنشاء ملف جديد يحتوي على جميع التعليقات التوضيحية المتجانبة المدمجة تحت طبقة واحدة.
- افتح برنامج تحليل الصور وانتقل إلى علامة التبويب التعليقات التوضيحية . حدد القائمة المنسدلة إجراءات الطبقة | احذف كل الطبقات لإزالة كل التعليقات التوضيحية من الصورة.
- حدد القائمة المنسدلة إجراءات الطبقة | استيراد ملف التعليق التوضيحي المحلي. في النافذة المنبثقة، حدد ملف .annotation المدمج الذي تم إنشاؤه بواسطة البرنامج النصي. تأكد من أن كل التجانبات المستوردة تقع تحت نفس طبقة التعليقات التوضيحية.
- انتقل إلى علامة التبويب المصنف واتبع إرشادات الشركة المصنعة لإنشاء أشكال لعائد الاستثمار. قبل تشغيل المصنف، حدد طبقة (طبقات) التعليقات التوضيحية المطلوبة (أي طبقة الورم أو السدى) عن طريق تحديد مربع عائد الاستثمار أو المربعات في علامة التبويب التعليقات التوضيحية ، ضمن خيارات المصنف المتقدم. استخدم خيار طبقة التعليقات التوضيحية من قائمة إجراءات المصنف لتشغيل المصنف.
- بمجرد اكتمال تحليل المصنف، انتقل إلى علامة التبويب التعليقات التوضيحية وحدد طبقة التعليقات التوضيحية التي تم إنشاؤها من التحليل. حدد القائمة المنسدلة إجراءات الطبقة | حذف كل الطبقات ولكن الحالية لإزالة كل طبقات التعليقات التوضيحية الأخرى من الصورة.
- حدد القائمة المنسدلة إجراءات الطبقة | تصدير لحفظ التعليقات التوضيحية كملف .annotation. قم بإنشاء مجلد لجلسة العمل أو المشروع واحفظ ملف .annotation داخل مجلد فرعي يحمل المعرف الفريد للشريحة.
ملاحظة: يتم توفير ملف .annotation تمثيلي لمجموعة أنسجة LMD مخصبة مصنفة في الملف التكميلي 6. - انتقل إلى علامة التبويب التعليقات التوضيحية ، وحدد القائمة المنسدلة إجراءات الطبقة | احذف كل الطبقات لإزالة كل التعليقات التوضيحية من الصورة. حدد أداة القلم وارسم خطا قصيرا من كل عملية معايرة ائتمانية. ارسم خطوطا من العلامات بالترتيب التالي: أعلى اليسار وأعلى اليمين وأسفل اليمين.
- حدد القائمة المنسدلة إجراءات الطبقة | تصدير لحفظ التعليقات التوضيحية للأسطر كملف .annotation. أضف _calib إلى اسم الملف وضع الملف في المجلد الفرعي الذي يحتوي على إحداثيات الأشكال المتجانبة.
- انسخ عنوان المشروع الرئيسي أو مجلد جلسة العمل. افتح البرنامج النصي لإنشاء استيراد XML، "Malleator" (متوفر عبر https://github.com/GYNCOE/Mitchell.et.al.2022)، باستخدام بيئة التطوير المتكاملة IDLE، ثم الصق عنوان مجلد المشروع بين علامات الاقتباس الموجودة أسفل البرنامج النصي.
- حدد القائمة المنسدلة تشغيل | قم بتشغيل الوحدة النمطية لتنفيذ البرنامج النصي.
ملاحظة: سيتم إنشاء ملف استيراد LMD .xml داخل المجلد الفرعي الذي تم إنشاؤه للصورة/الشريحة.
6. التشريح المجهري بالليزر
ملاحظة: خطوات البروتوكول الموضحة في هذا القسم محددة للاستخدام مع مجهر ليزر مقلوب والبرامج المرتبطة به (انظر جدول المواد). الوقت التقريبي: 2 ساعة; حالة الاعتماد.
- قم بتحميل شريحة الغشاء المحددة (التي تحتوي على ائتمانات المعايرة) بحيث يكون النسيج متجها لأسفل وجانب الملصق الأقرب إلى المشغل في حامل الشريحة على مرحلة المجهر الليزر.
- حدد استيراد الأشكال من القائمة المنسدلة ملف. حدد ملف استيراد LMD .xml الذي تم إنشاؤه للشريحة. حدد لا في النافذة المنبثقة لتجنب تحميل النقاط المرجعية من الملف و لا في النافذة المنبثقة الثانية لتجنب استخدام أي نقاط مرجعية مخزنة مسبقا للمعايرة.
- اتبع المطالبات من تطبيق LMD وقم بمحاذاة تقاطع المعايرة مع كل من مراجع المعايرة الثلاثة على الشريحة. ابحث عن اعتمادات المعايرة التي تظهر في أعلى اليسار وأعلى اليمين وأسفل يمين صورة الشريحة في برنامج تحليل الصور التي تتوافق مع النقاط المرجعية في الزوايا الأمامية اليمنى والخلفية اليمنى والخلفية اليسرى ، على التوالي ، من شريحة LMD المقلوبة على مرحلة المجهر. قم بالتبديل بين استخدام العدسة الموضوعية 5x لتحديد الموقع والهدف 63x لمحاذاة كل ائتمان معايرة. حدد لا في النافذة المنبثقة لتجنب حفظ النقاط المرجعية في الملف وموافق في النافذة المنبثقة الثانية للتأكد من إدراج الشريحة.
- حرك عدسة الهدف 5x إلى موضعها وحدد نعم في النافذة المنبثقة لاستخدام التكبير الفعلي. بمجرد ظهور الأشكال المستوردة، ركز الكاميرا على الأنسجة.
- قم بتمييز وتحديد كافة الأشكال في نافذة قائمة الأشكال ، واسحبها إلى مكانها باستخدام تعليق توضيحي واحد أو اثنين داخل مجال الرؤية كمراجع، وقم بمحاذاة المحور z الرأسي للقطع باستخدام الليزر.
- راجع الأشكال المستوردة وقم بتعيينها إلى موضع الأنبوب المناسب للتجميع. اضغط على Start Cut لبدء تشغيل الليزر.
ملاحظة: سيتم تعيين الأشكال المستوردة في ملف .xml تلقائيا إلى الموضع "بدون غطاء" في نافذة قائمة الأشكال . لحصاد الأنسجة ، يجب إعادة تعيين الأشكال المستوردة إلى موضع يحتوي على أنبوب محمل.
7. هضم البروتين بواسطة تكنولوجيا دورة الضغط (PCT)
ملاحظة: الوقت التقريبي: 4 ساعات (3 ساعات بدون وقت تجفيف أجهزة الطرد المركزي الفراغية).
- ضع أنابيب 0.5 مل تحتوي على الأنابيب الدقيقة المغطاة بموجب معاهدة التعاون بشأن البراءات التي تحتوي على أنسجة LMD المحصودة في 20 ميكرولتر من 100 mM TEAB / 10٪ acetonitrile في جهاز تدوير حراري والحرارة عند 99 درجة مئوية لمدة 30 دقيقة ، ثم تبرد إلى 50 درجة مئوية لمدة 10 دقائق.
- قم بتدوير الأنابيب لمدة 30 ثانية عند 4000 × جم ثم قم بإزالة MicroTubes من أنابيب 0.5 مل. باستخدام أداة MicroCap ، قم بإزالة MicroCaps والتخلص منها من MicroTubes لمعاهدة التعاون بشأن البراءات. أضف التربسين (انظر جدول المواد) بنسبة 1 ميكروغرام لكل 30 مم2 من الأنسجة وأدخل MicroPestle في MicroTube باستخدام أداة MicroCap.
- انقل MicroTubes إلى خرطوشة barocycler وقم بتجميع الخرطوشة بالكامل. ضع الخرطوشة في غرفة الضغط الباروسيكلر وقم بتأمين الغطاء. الباروسايكل عند 45000 رطل لكل بوصة مربعة لمدة 50 ثانية والضغط الجوي لمدة 10 ثوان عند 50 درجة مئوية لمدة 60 دورة.
- بمجرد اكتمال التدوير الباروكي ، انقل الأنابيب الدقيقة إلى أنبوب طرد مركزي دقيق 0.5 مل وأجهزة طرد مركزي لمدة 2 دقيقة عند 4000 × جم.
- قم بإزالة MicroTube من أنبوب الطرد المركزي الدقيق 0.5 مل باستخدام أداة الغطاء. قم بإزالة المدقة بعناية باستخدام أداة الغطاء وشطف النصف السفلي من المدقة ب 20 ميكرولتر من الماء السائل من درجة الكروماتوغرافيا - قياس الطيف الكتلي (LC-MS) وجمع الغسيل في أنبوب طرد مركزي دقيق نظيف 0.5 مل.
- اضغط برفق على MicroTube على سطح المقعد لنقل السائل إلى الأسفل ونقل كل المحلول من MicroTube إلى أنبوب الطرد المركزي الدقيق 0.5 مل.
- أضف 20 ميكرولتر من الماء من فئة LC-MS إلى MicroTube واضغط عليه بلطف على سطح المقعد. انقل محلول الغسيل إلى أنبوب 0.5 مل وكرر خطوة الغسيل هذه مرة أخرى.
- جهاز طرد مركزي فراغي لتجفيف العينات إلى ~ 2 ميكرولتر وإضافة 100 ميكرولتر من 100 mM TEAB ، درجة الحموضة 8.0.
- حدد تركيز الببتيد باستخدام مقايسة لونية (مقايسة حمض البيتشينكونينيك (BCA) ؛ انظر جدول المواد) وفقا لبروتوكول الشركة المصنعة.
8. وضع العلامات جنبا إلى جنب (TMT) ووضع العلامات وتنظيف EasyPep
ملاحظة: الوقت التقريبي: 7 ساعات و 20 دقيقة (2 ساعة و 20 دقيقة بدون وقت تجفيف أجهزة الطرد المركزي الفراغية).
- أحضر كواشف وضع العلامات TMT متساوي الضغط إلى درجة الحرارة المحيطة قبل الفتح. أضف 500 ميكرولتر من الأسيتونيتريل 100٪ إلى كل قارورة TMT (5 ملغ). احتضان لمدة 10 دقائق مع دوامة عرضية.
- قم بإذابة 5 ميكروغرام من عينة الببتيد في 100 ميكرولتر من 100 mM TEAB ، ودرجة الحموضة 8.0 ، وإضافة 10 ميكرولتر من كاشف TMT معين. بناء وتضمين تجمعات مرجعية تمثل كل عينة فردية في التجربة في كل مجموعة من العينات متعددة الإرسال TMT لتسهيل تحديد كمية العينات عبر مضاعفات TMT متعددة9. احتضان التفاعلات لمدة 1 ساعة في درجة الحرارة المحيطة مع اهتزاز / نقر عرضي.
- قم بإخماد تفاعل وضع العلامات TMT عن طريق إضافة 10 ميكرولتر من 5٪ هيدروكسيلامين وحضانت لمدة 30 دقيقة في درجة الحرارة المحيطة مع التنصت العرضي. بعد التبريد ، ادمج العينات التي تحمل علامة TMT في أنبوب واحد وجففها إلى حوالي 200 ميكرولتر.
- أضف 1800 ميكرولتر من 0.1٪ حمض الفورميك. تحقق من الرقم الهيدروجيني مع ورقة الرقم الهيدروجيني: إذا كان الرقم الهيدروجيني ~ 3 ، أضف 1 مل من حمض الفورميك 0.1 ٪ ؛ إذا كان الرقم الهيدروجيني >3 ، أضف 10-20 ميكرولتر من حمض الفورميك بنسبة 5٪ حتى درجة الحموضة ~ 3. أضف 0.1٪ من حمض الفورميك للوصول إلى الحجم النهائي من 3 مل.
- قم بإزالة علامة التبويب الموجودة أسفل عمود تنظيف الببتيد، وقم بإزالة الغطاء، وضعه في أنبوب مخروطي بسعة 15 مل. انقل العينة التي تحمل علامة TMT إلى العمود وتابع التنظيف وفقا لبروتوكول الشركة المصنعة.
- جهاز طرد مركزي فراغي لتجفيف الببتيدات المطفأة إلى ~ 20 ميكرولتر ، ونقلها إلى قارورة LC باستخدام 25 mM بيكربونات الأمونيوم لحجم نهائي من 80 ميكرولتر ، والمضي قدما في تجزئة غير متصل بالإنترنت.
9. TMT تجزئة العينة متعددة الإرسال وتجميعها
ملاحظة: الوقت التقريبي: 3 ساعات و 30 دقيقة (1 ساعة و 30 دقيقة بدون وقت تجفيف أجهزة الطرد المركزي الفراغية).
- قم بتجزئة مضاعفات الببتيد المسماة TMT بواسطة كروماتوغرافيا الطور المعكوس الأساسية إلى 96 كسرا عن طريق تطوير تدرج خطي متزايد (0.69٪ min-1) من المرحلة المتنقلة B (acetonitrile) إلى المرحلة المتنقلة A (10 mM NH4HCO3 ، الرقم الهيدروجيني 8.0).
- توليد 36 كسرا متسلسلة عن طريق تجميع آبار العينة. جهاز طرد مركزي فراغي لتجفيف الكسور إلى ~ 2 ميكرولتر وإعادة تعليقه في 25 mM بيكربونات الأمونيوم (التركيز النهائي 1.5 ميكروغرام / 10 ميكرولتر) ، وأجهزة الطرد المركزي في 15000 × غرام لمدة 10-15 دقيقة ، ونقلها إلى قوارير LC لتحليل MS.
10. قياس الطيف الكتلي الترادف بالكروماتوغرافيا السائلة (LC-MS/MS)
ملاحظة: الوقت التقريبي: تعتمد طريقة الأداة والتصميم التجريبي.
- معايرة مطياف الكتلة وفقا لتعليمات/بروتوكولات الشركة المصنعة.
- إعداد مراحل ومعايير متنقلة جديدة وإجراء الاستعدادات المناسبة لما قبل تشغيل LC (بما في ذلك على سبيل المثال لا الحصر مذيبات التطهير والهواء المتدفق ونصوص اختبار التسرب للأداة المشار إليها [انظر جدول المواد]). موازنة الأعمدة المسبقة والتحليلية وحلقة العينة قبل بدء التحليلات.
- قبل التحليلات التسلسلية متعددة الإرسال TMT وفيما بينها، تحقق من أن نظام LC-MS يفي بمقاييس الأداء التي تم قياسها مسبقا باستخدام ملخصات الببتيد الموسومة بعلامة TMT لضمان الجودة / مراقبة الجودة (QA / QC) TMT و (على سبيل المثال ، MSPE (انظر جدول المواد) ، HeLa).
- قم بتحميل قوارير أخذ العينات التلقائية في المواضع المناسبة في جهاز أخذ العينات التلقائي LC. تحليل الكسور الفردية باستخدام طريقة التدرج / MS المناسبة. تتخلل عملية "غسل" مع معيار الببتيد (على سبيل المثال، معايرة وقت الاحتفاظ بالببتيد [PRTC]) مرة واحدة يوميا تقريبا لتقييم الأداء الكروماتوغرافي والطيفي الكتلي. بعد تحليل كل سلسلة كسور عينة TMT متعددة الإرسال ، قم بتشغيل معايير QA / QC TMT المرجعية لتقييم أداء النظام.
- قم بتشغيل إجراءات تقييم مقياس الطيف الكتلي بعد المعايير المرجعية QA/QC TMT لتقييم أداء ما بعد العينة ثم معايرة النظام كما في الخطوة 10.1 لمجموعة العينات التالية.
11. تحليل البيانات المعلوماتية الحيوية
ملاحظة: الوقت التقريبي: يعتمد التصميم التجريبي.
- انقل جميع عينات البيانات (على سبيل المثال، ملفات .raw) إلى محرك أقراص تخزين / كمبيوتر مناسب للشبكة.
- ابحث في جميع الكسور معا باستخدام تطبيق تحليل البيانات المطلوب (على سبيل المثال ، Proteome Discover ، Mascot) باستخدام المعلمات المناسبة9 مقابل قاعدة بيانات مرجعية للبروتين الخاص بالأنواع لتوليد تطابقات طيفية الببتيد (PSMs) واستخراج كثافة إشارة أيون مراسل TMT. قم بتصفية PSMs استنادا إلى مقاييس مراقبة الجودة المناسبة ، وإجمالي التدفقات الأيونية لمراسل TMT المحولة2 إلى وفرة عالمية لمستوى البروتين ، كما هو موضح سابقا 3,9.
- قارن تغيرات البروتين في ظروف الاهتمام باستخدام برنامج التحليل التفاضلي المطلوب.
النتائج
تم تحليل المقاطع الرقيقة للأنسجة المجمدة الطازجة من اثنين من HGSOC واثنين من مرضى OCCC باستخدام هذا العمل المتكامل القائم على الذكاء الاصطناعي لتحديد عائد الاستثمار في الأنسجة ، والتجزئة ، و LMD ، وسير عمل التحليل البروتيني الكمي (الشكل 1). تمت مراجعة أقسام الأنسجة الملطخة H & E التمثيلية لكل ورم من قبل أخصائي علم الأمراض المعتمد من مجلس الإدارة. تراوحت خلية الورم من 70٪ إلى 99٪. تم تقسيم الأنسجة إلى شرائح غشاء PEN (الملف التكميلي 2) وقطعها مسبقا باستخدام fiducials المعايرة (الملف التكميلي 1) ، مما مكن من دمج بيانات الاتجاه الموضعي من التعليقات التوضيحية التي تم إنشاؤها في برنامج تحليل الصور (انظر جدول المواد) مع توجيه الإحداثيات الديكارتية في برنامج LMD. بعد تلطيخ H & E ، تم التقاط صور عالية الدقة (20x) لشرائح PEN التي تحتوي على الأنسجة بالإضافة إلى أجهزة المعايرة.
تم تقسيم مجموعات الخلايا السرطانية واللحمية في المجهر باستخدام برنامج تحليل الصور (انظر جدول المواد) للحصاد الانتقائي بواسطة LMD ، إلى جانب الحصاد الذي يمثل القسم الرقيق بأكمله من الأنسجة (على سبيل المثال ، نسيج الورم الكامل) (الشكل 1). تم إنشاء تعليقات توضيحية غير تمييزية لمجموعات أنسجة الورم بأكملها عن طريق تقسيم قسم الأنسجة بأكمله ببلاط 500 ميكرومتر2 ، تاركا فجوة 40 ميكرومتر بين البلاط للحفاظ على سلامة غشاء القلم ومنع الغشاء من التجعيد أثناء LMD. على الشرائح الخاصة بإثراء LMD الذي تم حله بواسطة علم الأنسجة ، تم تدريب المصنف الذكاء الاصطناعي في برنامج تحليل الصور (انظر جدول المواد) على التمييز بين الخلايا السرطانية واللحمية ، إلى جانب خلفية الشريحة الزجاجية الفارغة. تم تمييز مناطق الورم التمثيلي والسدى والزجاج الفارغ يدويا ، وتم استخدام أداة المصنف لتقسيم عائد الاستثمار هذا في جميع أنحاء قسم الأنسجة بأكمله. تم حفظ الطبقات المجزأة التي تمثل الأنسجة الكاملة وظهارة الورم والسدى بشكل منفصل كملفات .annotation فردية (الملف التكميلي 3 والملف التكميلي 6). في نسخة منفصلة من ملف الصورة (بدون التعليقات التوضيحية لعائد الاستثمار المقسمة)، تم إضافة سطر قصير من الطرف الأوسط لكل من المعايرات الائتمانية الثلاثة وحفظه كملف .annotation باستخدام نفس اسم الملف مثل كل ملف من ملفات طبقة التعليقات التوضيحية LMD ولكن تم إلحاقه باللاحقة "_calib" (الملف التكميلي 4). تم استخدام هذه الخطوط للمشاركة في تسجيل موضع معايرات غشاء القلم مع بيانات قائمة شكل التعليق التوضيحي المرسومة في برنامج تحليل الصور.
توفر هذه الدراسة خوارزميتين ، "Malleator" و "Dapọ" في Python لدعم سير عمل LMD المدفوع الذكاء الاصطناعي ، والذي يتوفر في https://github.com/GYNCOE/Mitchell.et.al.2022. تستخرج خوارزمية Malleator الإحداثيات الديكارتية المحددة لجميع التعليقات التوضيحية الفردية (عائد استثمار الأنسجة والمعايرة) من ملفات .annotation المقترنة وتدمجها في ملف استيراد لغة ترميز واحدة قابلة للتوسيع (XML) (ملف تكميلي 5). على وجه التحديد، تستخدم خوارزمية Malleator اسم الدليل من مجلد أصل كإدخال للبحث في كافة مجلدات الدليل الفرعي وإنشاء ملفات .xml لأي مجلدات فرعية لا تحتوي بالفعل على ملف مدمج .xml. تقوم خوارزمية Malleator بدمج جميع طبقات التعليقات التوضيحية في برنامج تحليل الصور (انظر جدول المواد) في طبقة واحدة وتحويل بيانات قائمة الأشكال التي تم إنشاؤها بواسطة الذكاء الاصطناعي ، والتي يتم حفظها كنوع ملف التعليق التوضيحي الخاص ، إلى تنسيق .xml متوافق مع برنامج LMD. بعد دمج ملفات التعليقات التوضيحية والمعايرة، يتم حفظ ملف .xml الذي تم إنشاؤه بواسطة الخوارزمية واستيراده إلى برنامج LMD. تعد التعديلات الطفيفة ضرورية لضبط محاذاة التعليقات التوضيحية يدويا ، والتي تعمل أيضا على تسجيل الموضع الرأسي (z-plane) لمرحلة الشريحة على مجهر الليزر. تستخدم خوارزمية Dapọ خصيصا للمجموعات الغنية ب LMD. يتم تعيين البلاط المقسم تلقائيا إلى طبقات التعليقات التوضيحية الفردية بواسطة برنامج تحليل الصور. تقوم خوارزمية Dapọ بدمج جميع التجانبات المقسمة في طبقة تعليق توضيحي واحدة قبل استخدام أداة المصنف ، وبالتالي تقليل وقت تشغيل تحليل المصنف للمجموعات المخصبة LMD.
تم هضم عينات الورم والأنسجة المخصب ب LMD بأكملها ، وتصنيفها باستخدام كواشف TMT ، ومتعددة الإرسال ، ومجزأة في وضع عدم الاتصال ، وتحليلها عبر البروتيوميات الكمية القائمة على MS كما هو موضح سابقا9. وكان متوسط محصول الببتيد (43-60 ميكروغرام) والاسترداد (0.46-0.59 ميكروغرام/مم2) للعينات التي تم حصادها باستخدام سير العمل المدفوع الذكاء الاصطناعي هذا قابلا للمقارنة مع التقارير السابقة 9,10. تم تحديد ما مجموعه 5,971 بروتينا بشكل مشترك عبر جميع العينات (الجدول التكميلي S1). أدى التجميع الهرمي غير الخاضع للإشراف باستخدام أكثر 100 بروتين متغير إلى فصل الأنماط النسيجية HGSOC و OCCC عن عينات الورم الكامل المخصب ب LMD (الشكل 2A) ، على غرار تلك الموصوفة سابقا11. وعلى النقيض من ذلك، تجمعت عينات السدى المخصب ب LMD من كل من HGSOC و OCCC معا وبشكل مستقل عن الورم المخصب LMD وعينات الورم بأكملها. من بين 5,971 بروتينا كميا ، تم تغيير 215 بشكل كبير (LIMMA adj. p < 0.05) بين مجموعات الورم الكاملة من عينات HGSOC و OCCC (الجدول التكميلي S2). تمت مقارنة هذه البروتينات المعدلة مع تلك التي تم تحديدها للتمييز بين أنسجة ورم HGSOC و OCCC بواسطة Hughes et al.11. ومن بين 76 بروتينا مميزا حددها هيوز وآخرون، تم قياس 57 بروتينا كميا مشتركا في مجموعة البيانات هذه وكانت مترابطة إلى حد كبير (سبيرمان رو = 0.644، ص < 0.001) (الشكل 2 ب).
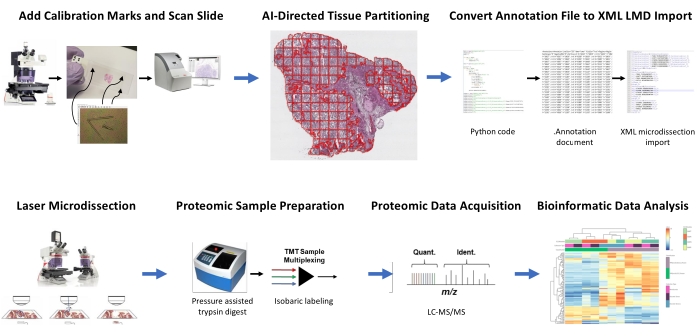
الشكل 1: ملخص سير العمل المتكامل لاختيار منطقة الأنسجة الآلية ذات الأهمية للتشريح المجهري بالليزر للبروتينات الكمية النهائية. يتم قطع ائتمانات المعايرة على شرائح غشاء القلم للمشاركة في تسجيل بيانات الاتجاه الموضعي من الأجزاء المشتقة من الذكاء الاصطناعي من عائد الاستثمار على الأنسجة في برنامج تحليل الصور ، HALO ، مع تحديد المواقع الأفقي على المجهر LMD. تستخدم خوارزمية Malleator لدمج بيانات التقسيم المشروحة عبر جميع طبقات التعليقات التوضيحية لشريحة مع الملف المرجعي _calib، وتحويلها إلى ملف .xml متوافق مع برنامج LMD. يتم هضم وتحليل الأنسجة التي يتم حصادها من LMD للتحليل البروتيني بواسطة البروتينات الكمية عالية الإنتاجية كما هو موضح سابقا9. الاختصارات: LMD = التشريح المجهري بالليزر; عائد الاستثمار = منطقة الاهتمام؛ TMT = علامة كتلة جنبا إلى جنب; الكمية. = القياس الكمي. الهوية. = تحديد الهوية؛ LC-MS/MS = كروماتوغرافيا سائلة-قياس الطيف الكتلي جنبا إلى جنب. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
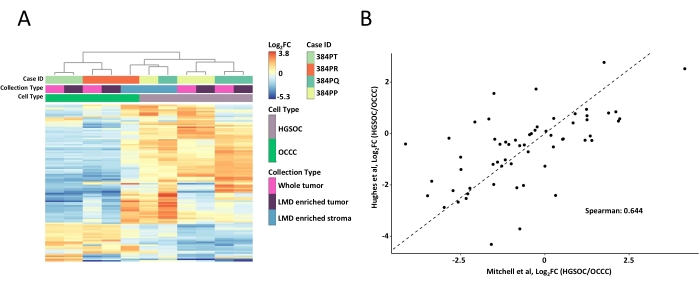
الشكل 2: تحليل البروتينات في عينات الورم الكامل المخصب ب LMD. (أ) التحليل العنقودي الهرمي غير الخاضع للإشراف ل 100 بروتين أكثر وفرة بشكل متغير في عينات الورم المخصب والكامل HGSOC و OCCC LMD. (ب) العلاقة بين وفرة البروتين ذي التغير الطوي2 بين حصاد الورم الكامل HGSOC و OCCC في هذه الدراسة (Mitchell et al.، x-axis) ودراسة مماثلة أجراها هيوز وآخرون (y-axis)11. الاختصارات: LMD = التشريح المجهري بالليزر; HGSOC = سرطان المبيض المصلي عالي الجودة. OCCC = سرطان الخلايا المبيض الواضح; log 2 FC = log2-transform-proteomicabunover. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
الجدول التكميلي S1: وفرة من 5,971 بروتينا تم تحديدها كميا بشكل مشترك عبر جميع عينات الورم المخصب والكامل LMD من عينات أنسجة HGSOC و OCCC. الاختصارات: LMD = التشريح المجهري بالليزر; HGSOC = سرطان المبيض المصلي عالي الجودة. OCCC = سرطان الخلايا المبيض الصافي. يرجى النقر هنا لتنزيل هذا الجدول.
الجدول التكميلي S2: البروتينات المعبر عنها بشكل تفاضلي (215) في مجموعات الورم بأكملها من HGSOC مقابل OCCC (LIMMA adj. p < 0.05). الاختصارات: HGSOC = سرطان المبيض المصلي عالي الجودة. OCCC = سرطان الخلايا المبيض الصافي. يرجى النقر هنا لتنزيل هذا الجدول.
الملف التكميلي 1: ملف بيانات قائمة الأشكال التمثيلية (.sld) الذي يحتوي على اعتمادات معايرة قياسية لأربعة مواضع للشرائح. يمكن استيراد الملف إلى برنامج LMD. يرجى النقر هنا لتنزيل هذا الملف.
الملف التكميلي 2: ملف صورة تمثيلي .svs لقسم أنسجة عالي الدقة (20x) ملطخ ب H&E. يمكن فتح الملف وعرضه باستخدام برنامج تحليل الصور أو برنامج LMD. اختصار: H & E = الهيماتوكسيلين والإيوزين. LMD = التشريح المجهري بالليزر. يرجى النقر هنا لتنزيل هذا الملف.
الملف التكميلي 3: ملف .annotation التمثيلي لشرائح الورم الكاملة المقسمة. يمكن استيراد الملف إلى برنامج تحليل الصور. يرجى النقر هنا لتنزيل هذا الملف.
الملف التكميلي 4: ملف التعليق التمثيلي _calib.annotation للمقاطع الائتمانية للمعايرة. تمثل معلومات الإحداثيات تحديد المواقع الشرقية لخطوط المعايرة القصيرة المستمدة من كل رأس سهم ائتماني. يمكن استيراد الملف إلى برنامج تحليل الصور. يرجى النقر هنا لتنزيل هذا الملف.
الملف التكميلي 5: ملف لغة الترميز التمثيلية القابلة للتوسيع (.xml) الذي تم إنشاؤه بواسطة خوارزمية Malleator. يمكن استيراد الملف إلى برنامج التشريح المجهري بالليزر. يرجى النقر هنا لتنزيل هذا الملف.
الملف التكميلي 6: ملف التعليق التوضيحي التمثيلي للشرائح المصنفة الذكاء الاصطناعي المجزأة للمجموعات المخصبة ب LMD. يمكن استيراد الملف إلى برنامج تحليل الصور. الاختصارات: الذكاء الاصطناعي = الذكاء الاصطناعي; LMD = التشريح المجهري بالليزر. يرجى النقر هنا لتنزيل هذا الملف.
Discussion
في حين كانت هناك سوابق دراسة متعددة تهدف إلى تطوير و / أو تحسين سير العمل لإثراء المجموعات الفرعية الخلوية المستهدفة من FFPE و / أو الأنسجة المجمدة الطازجة ومنهجيات للحفاظ على جودة العينات أثناء المعالجة9،12،13،14،15 ، هناك حاجة كبيرة لتطوير استراتيجيات آلية لإعداد عينات الأنسجة السريرية للتحليلات الجزيئية لتقليل التباين و زيادة قابلية التكرار. يصف سير العمل هذا بروتوكولا موحدا شبه آلي يدمج أدوات برامج تحليل الصور الحالية (انظر جدول المواد) للحصاد الذي تم حله بواسطة الأنسجة لمجموعات الخلايا المنفصلة بواسطة LMD من عينات الأنسجة السريرية.
يمثل إثراء LMD الذي تم حله مكانيا لعائد الاستثمار الذي يلتقط مجموعات الخلايا المنفصلة خطوة معالجة الأنسجة من الجيل التالي قبل التحليلات متعددة الأزمنة لتحسين التوصيف الجزيئي وتحديد الهوية وتسهيل اكتشاف المؤشرات الحيوية الانتقائية للخلايا. يحسن هذا البروتوكول من المنهجيات الحالية عن طريق تقليل التعرض الطويل في كثير من الأحيان لأقسام الأنسجة للبيئة المحيطة المرتبطة بالتقسيم اليدوي لعائد الاستثمار من قبل أخصائي الأنسجة (والذي يمكن أن يستغرق >1-2 ساعة قبل جمع LMD). يسمح سير العمل هذا بدلا من ذلك بتحديد عائد الاستثمار مسبقا من خلال التصنيف والتقسيم الموجه الذكاء الاصطناعي. إن الحد من وقت بقاء الأنسجة سيقلل من الاختلافات الزائفة في تقييمات الأهداف الجزيئية عالية القابلية للاختبار ، مثل الببتيدات الفوسفاتية و mRNA ، أو للتقنيات التحليلية القائمة على الأجسام المضادة التي تعتمد على البروتين المستهدف في تشكيله الأصلي للكشف.
يعد قطع اعتمادات المعايرة الأنيقة على شريحة غشاء PEN المرئية بوضوح في صورة الشريحة الممسوحة ضوئيا أحد المكونات الرئيسية التي تمكن من دمج برنامج تحليل الصور (انظر جدول المواد) مع سير عمل LMD. إن التأكد من أن المعايرات تحتوي على نقطة دقيقة ("نظيفة") في أسفل الشكل "V" يسمح باختيار نقطة دقيقة في برنامج تحليل الصور لخطوط المعايرة التي سيتم استخلاصها منها، كما هو موضح في الخطوتين 5.1.6 و 5.2.13. تعد محاذاة هذه النقاط أثناء الاستيراد إلى برنامج LMD أمرا بالغ الأهمية لتراكب التعليقات التوضيحية بشكل صحيح (يتم تسهيلها من خلال إنشاء ملف .xml متوافق باستخدام خوارزميات "Malleator" و / أو "Dapọ") على عائد الاستثمار على الأنسجة ذات الصلة على شريحة LMD المادية. من الضروري تسليط الضوء على جميع الأشكال و "السحب والإفلات" بشكل جماعي في مكانها حتى عندما تكون المحاذاة دقيقة عند الاستيراد إلى برنامج LMD لتسجيل الموضع الرأسي (z-plane) لمرحلة الشريحة على المجهر الليزر. يمكن أيضا إجراء تعديلات طفيفة على موضع التعليقات التوضيحية على عائد الاستثمار على الأنسجة خلال هذه الخطوة ، إذا لزم الأمر.
أحد قيود الإصدار الحالي من خوارزمية Malleator هو أنه غير متوافق مع أدوات شكل التعليق التوضيحي المحددة مسبقا التي يوفرها برنامج تحليل الصور (انظر جدول المواد) ، على الرغم من أن التحديثات / الإصدارات المستقبلية من الخوارزمية ستهدف إلى تحسين هذا التوافق. يحتوي ملف .annotation للأشكال المرسومة باستخدام هذه الأدوات على مجموعتين فقط من إحداثيات x وy المقترنة لكل تعليق توضيحي، بدون الاتجاه المكاني الكامل حول تلك النقاط. يؤدي الاستخدام الحالي لهذه الأدوات إلى تحويل التعليقات التوضيحية إلى خطوط مستقيمة محددة بنقطتين فقط أثناء عملية الاستيراد. مطلوب تعريف يدوي لشرائح عائد الاستثمار على الأنسجة للتحويل الناجح إلى تنسيق XML واستيراد LMD. يمكن القيام بذلك إما عن طريق تعريف كل عائد استثمار يدويا باستخدام تعليقات توضيحية فردية متعددة الأضلاع حرة خاصة بالمنطقة المستهدفة أو عن طريق تطبيق تعليق توضيحي دائري أو مستطيل تقريبي عبر جميع شرائح عائد الاستثمار في الأنسجة ، إذا رغبت في ذلك ، وسيكون متوافقا مع سير العمل هذا.
في حين تم إثبات سير العمل المعروض هنا للتحليل البروتيني لعينات أنسجة السرطان البشرية المجمدة حديثا ، يمكن استخدام سير عمل LMD المدفوع الذكاء الاصطناعي بشكل متساو مع أنسجة FFPE وأنواع الأنسجة غير السرطانية وتلك من مصادر غير بشرية. ويمكنه أيضا دعم سير عمل التنميط الجزيئي المصب الآخر ، بما في ذلك التحليلات النسخية أو الجينومية أو الفوسفوبروتيومية. يمكن لسير العمل هذا أيضا الاستفادة من الاستخدامات الأخرى لبرنامج تحليل الصور (انظر جدول المواد) ، بما في ذلك القدرات المرتبطة بحساب الخلايا أو الوحدات التحليلية الأخرى ، بما في ذلك وحدة "Multiplex IHC" أو "الوظيفة الإضافية Tissue Microarray (TMA)". قد تستفيد التطبيقات المستقبلية لسير العمل هذا أيضا من التحديد المسبق لعدد الخلايا لكل جزء من عائد الاستثمار ، وبالتالي ضمان مدخلات خلوية مكافئة عبر مجموعات متعددة ، أو باستخدام طرق بديلة لتحديد عائد الاستثمار الخلوي ذي الأهمية ، مثل الكيمياء النسيجية المناعية أو علم اجتماع الخلية.
Disclosures
T.P.C. هي عضو في ThermoFisher Scientific ، Inc SAB وتتلقى تمويلا بحثيا من AbbVie.
Acknowledgements
تم توفير التمويل لهذا المشروع جزئيا من قبل برنامج صحة الدفاع (HU0001-16-2-0006 و HU0001-16-2-00014) إلى جامعة الخدمات النظامية لمركز التميز لسرطان أمراض النساء. ولم يكن للرعاة أي دور في تصميم الدراسة أو تنفيذها أو تفسيرها أو كتابتها. اخلاء المسؤوليه: الآراء المعرب عنها هنا هي آراء المؤلفين ولا تعكس السياسة الرسمية لوزارة الجيش / البحرية / القوات الجوية أو وزارة الدفاع أو حكومة الولايات المتحدة.
Materials
| Name | Company | Catalog Number | Comments |
| 1260 Infinity II System | Agilent Technologies Inc | Offline LC system | |
| 96 MicroCaps (150uL) in bulk | Pressure Biosciences Inc | MC150-96 | |
| 96 MicroPestles in bulk | Pressure Biosciences Inc | MP-96 | |
| 96 MicroTubes in bulk (no caps) | Pressure Biosciences Inc | MT-96 | |
| 9mm MS Certified Clear Screw Thread Kits | Fisher Scientific | 03-060-058 | Sample vial for offline LC frationation and mass spectrometry |
| Acetonitrile, Optima LC/MS Grade | Fisher Chemical | A995-4 | Mobile phase solvent |
| Aperio AT2 | Leica Microsystems | 23AT2100 | Slide scanner |
| Axygen PCR Tubes with 0.5 mL Flat Cap | Fisher Scientific | 14-222-292 | Sample tubes; size fits PCT tubes and thermocycler |
| Barocycler 2320EXT | Pressure Biosciences Inc | 2320-EXT | Barocycler |
| BCA Protein Assay Kit | Fisher Scientific | P123225 | |
| cOmplete, Mini, EDTA-free Protease Inhibitor Cocktail | Roche | 11836170001 | |
| Easy-nLC 1200 | Thermo Fisher Scientific | Liquid Chromatography | |
| EasyPep Maxi Sample Prep Kit | Thermo Fisher Scientific | NCI5734 | Post-label sample clean up column |
| EASY-SPRAY C18 2UM 50CM X 75 | Fisher Scientific | ES903 | Analytical column |
| Eosin Y Solution Aqueous | Sigma Aldrich | HT110216 | |
| Formic Acid, 99+ % | Thermo Fisher Scientific | 28905 | Mobile phase additive |
| ggplot2 version 3.3.5 | CRAN | https://cran.r-project.org/web/packages/ggplot2/ | |
| HALO | Indica Labs | Image analysis software | |
| IDLE (Integrated Development and Learning Environment) | Python Software Foundation | ||
| iheatmapr version 0.5.1 | CRAN | https://cran.r-project.org/web/packages/iheatmapr/ | |
| iRT Kit | Biognosys | Ki-3002-1 | LC-MS QAQC Standard |
| limma version 3.42.2 | Bioconductor | https://bioconductor.org/packages/release/bioc/html/limma.html | |
| LMD Scanning stage Ultra LMT350 | Leica Microsystems | 11888453 | LMD stage model outfitted with PCT tube holder |
| LMD7 (software version 8.2.3.7603) | Leica Microsystems | LMD apparatus (microscope, laser, camera, PC, tablet) | |
| Mascot Server | Matrix Science | Data analysis software | |
| Mass Spec-Compatible Human Protein Extract, Digest | Promega | V6951 | LC-MS QAQC Standard |
| Mayer’s Hematoxylin Solution | Sigma Aldrich | MHS32 | |
| PEN Membrane Glass Slides | Leica Microsystems | 11532918 | |
| Peptide Retention Time Calibration Mixture | Thermo Fisher Scientific | 88321 | LC-MS QAQC Standard |
| Phosphatase Inhibitor Cocktail 2 | Sigma Aldrich | P5726 | |
| Phosphatase Inhibitor Cocktail 3 | Sigma Aldrich | P0044 | |
| Pierce LTQ Velos ESI Positive Ion Calibration Solution | Thermo Fisher Scientific | 88323 | Instrument calibration solution |
| PM100 C18 3UM 75UMX20MM NV 2PK | Fisher Scientific | 164535 | Pre-column |
| Proteome Discoverer | Thermo Fisher Scientific | OPTON-31040 | Data analysis software |
| Python | Python Software Foundation | ||
| Q Exactive HF-X | Thermo Fisher Scientific | Mass spectrometer | |
| R version 3.6.0 | CRAN | https://cran-archive.r-project.org/bin/windows/base/old/2.6.2/ | |
| RColorBrewer version 1.1-2 | CRAN | https://cran.r-project.org/web/packages/RColorBrewer/ | |
| Soluble Smart Digest Kit | Thermo Fisher Scientific | 3251711 | Digestion reagent |
| TMTpro 16plex Label Reagent Set | Thermo Fisher Scientific | A44520 | isobaric TMT labeling reagents |
| Veriti 60 well thermal cycler | Applied Biosystems | 4384638 | Thermocycler |
| Water, Optima LC/MS Grade | Fisher Chemical | W6-4 | Mobile phase solvent |
| ZORBAX Extend 300 C18, 2.1 x 12.5 mm, 5 µm, guard cartridge (ZGC) | Agilent Technologies Inc | 821125-932 | Offline LC trap column |
| ZORBAX Extend 300 C18, 2.1 x 150 mm, 3.5 µm | Agilent Technologies Inc | 763750-902 | Offline LC analytical column |
References
- Motohara, T., et al. An evolving story of the metastatic voyage of ovarian cancer cells: cellular and molecular orchestration of the adipose-rich metastatic microenvironment. Oncogene. 38 (16), 2885-2898 (2019).
- Aran, D., Sirota, M., Butte, A. J. Systematic pan-cancer analysis of tumour purity. Nature Communications. 6, 8971 (2015).
- Hunt, A. L., et al. Extensive three-dimensional intratumor proteomic heterogeneity revealed by multiregion sampling in high-grade serous ovarian tumor specimens. iScience. 24 (7), 102757 (2021).
- Dou, Y., et al. Proteogenomic characterization of endometrial carcinoma. Cell. 180 (4), 729-748 (2020).
- Zhang, H., et al. Integrated proteogenomic characterization of human high-grade serous ovarian cancer. Cell. 166 (3), 755-765 (2016).
- Gillette, M. A., et al. Proteogenomic characterization reveals therapeutic vulnerabilities in lung adenocarcinoma. Cell. 182 (1), 200-225 (2020).
- Silvestri, A., et al. Protein pathway biomarker analysis of human cancer reveals requirement for upfront cellular-enrichment processing. Laboratory Investigation. 90 (5), 787-796 (2010).
- Echle, A., et al. Deep learning in cancer pathology: a new generation of clinical biomarkers. British Journal of Cancer. 124 (4), 686-696 (2021).
- Lee, S., et al. Molecular analysis of clinically defined subsets of high-grade serous ovarian cancer. Cell Reports. 31 (2), 107502 (2020).
- Xuan, Y., et al. Standardization and harmonization of distributed multi-center proteotype analysis supporting precision medicine studies. Nature Communications. 11 (1), 5248 (2020).
- Hughes, C. S., et al. Quantitative profiling of single formalin fixed tumour sections: proteomics for translational research. Scientific Reports. 6 (1), 34949 (2016).
- Espina, V., et al. A portrait of tissue phosphoprotein stability in the clinical tissue procurement process. Molecular & Cellular Proteomics. 7 (10), 1998-2018 (2008).
- Espina, V., Heiby, M., Pierobon, M., Liotta, L. A. Laser capture microdissection technology. Expert Review of Molecular Diagnostics. 7 (5), 647-657 (2007).
- Havnar, C. A., et al. Automated dissection protocol for tumor enrichment in low tumor content tissues. Journal of Visualized Experiments. (169), e62394 (2021).
- Mueller, C., et al. One-step preservation of phosphoproteins and tissue morphology at room temperature for diagnostic and research specimens. PLoS One. 6 (8), (2011).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved