Method Article
工业化、人工智能引导的激光显微切割,用于肿瘤微环境的微尺度蛋白质组学分析
* 这些作者具有相同的贡献
摘要
该协议描述了一种高通量工作流程,用于人工智能驱动的从染色的薄组织切片图像中分割病理确认感兴趣的区域,以使用激光显微切割富集组织学分辨细胞群。该策略包括一种新颖的算法,能够将表示感兴趣细胞群的分界线直接转移到激光显微镜上。
摘要
肿瘤微环境(TME)代表了一个由数十种不同细胞类型组成的复杂生态系统,包括肿瘤,基质和免疫细胞群。为了大规模表征蛋白质组水平的变化和肿瘤异质性,需要高通量方法来选择性地分离实体瘤恶性肿瘤中的离散细胞群。该协议描述了一种由人工智能(AI)实现的高通量工作流程,该工作流程将苏木精和曙红(H&E)染色的薄组织切片的图像分割成病理学确认的感兴趣区域,以便使用激光显微切割(LMD)选择性收获组织学分辨细胞群。该策略包括一种新颖的算法,能够将表示感兴趣细胞群的区域直接转移到激光显微镜上,从而能够更轻松地收集,使用数字图像软件进行注释。成功实施了该工作流程,证明了这种协调方法的实用性,该方法可通过高分辨率质谱选择性地从TME中收获肿瘤细胞群,以进行定量,多路复用蛋白质组学分析。该策略与常规组织病理学检查完全集成,利用数字图像分析来支持感兴趣的细胞群的富集,并且是完全可推广的,能够从TME中协调收获细胞群以进行多组分析。
引言
TME代表了一个复杂的生态系统,由高度多样化的细胞类型阵列组成,例如肿瘤细胞,基质细胞,免疫细胞,内皮细胞,其他间充质细胞类型和脂肪细胞,以及复杂的细胞外基质1。这种细胞生态系统在不同疾病器官部位内和之间变化,导致复杂的肿瘤异质性2,3。最近的研究表明,异质性肿瘤和肿瘤细胞性低(低纯度)的肿瘤通常与不良的疾病预后相关2,3。
为了大规模了解TME内肿瘤和非肿瘤细胞群之间的分子相互作用,需要标准化和高通量策略来选择性地收获感兴趣的不同细胞群以进行下游多组分析。定量蛋白质组学代表了一种快速发展和日益重要的技术,以进一步了解癌症生物学。迄今为止,采用蛋白质组学的大多数研究已经使用从整个肿瘤组织制剂中提取的蛋白质(例如,冷冻粉碎)来做到这一点,导致对TME4,5,6中蛋白质组水平异质性的理解不足。
与临床病理学工作流程无缝集成和利用信息的样本收集策略的开发将使新一代组织学解决的蛋白质组学成为可能,这些蛋白质组学与金标准,诊断病理学工作流程高度互补。LMD能够通过对组织学染色的组织薄切片进行显微镜检查,直接和选择性地收集细胞亚群或感兴趣区域(ROI)7。数字病理学和人工智能分析的最新重大进展已经证明能够以自动化方式识别TME内独特的组成特征和ROI,其中许多与分子改变和临床疾病特征相关,例如对治疗的抵抗力和疾病预后8。
本文介绍的方案中描述的工作流程利用商业软件解决方案在数字组织病理学图像中选择性地注释肿瘤ROI,并利用内部开发的软件工具将这些肿瘤ROI传输到激光显微镜,以自动收集感兴趣的离散细胞群,与下游多组分析工作流程无缝集成。这种集成策略显著缩短了LMD操作员的时间,并最大限度地缩短了组织在环境温度下所需的时间。通过对两种代表性上皮性卵巢癌组织学亚型(高级别浆液性卵巢癌(HGSOC)和卵巢透明细胞癌(OCCC))的TME进行差异分析,证明了自动特征选择和LMD收获与高通量定量蛋白质组学的整合。
研究方案
所有研究方案均被批准用于西方IRB批准的方案"子宫内膜癌和卵巢癌的综合分子分析,以识别和验证临床信息生物标志物",根据美国联邦法规45 CFR 46.102(f)被视为豁免。本研究中涉及人类数据的所有实验方案均符合《赫尔辛基宣言》。获得了参与研究的所有受试者的知情同意。
注意:整个方案中使用的以下试剂是已知或可疑的致癌物质和/或含有有害物质:乙醇,DEPC水,迈耶苏木精溶液,曙红Y溶液,甲醇,乙腈和甲酸。必须按照相应的安全数据表(SDS)所述正确处理,并使用适当的个人防护装备(PPE)。
1. 生成包含校准器基准的默认形状列表数据 (.sld) 文件
注意:本节中描述的实验方案步骤专门用于倒置激光显微镜和相关软件(参见 材料表)。每个激光显微镜只需要创建一次默认的.sld文件。生成的文件可用于将基准切割成此后使用的所有 PEN 幻灯片。大约时间:5分钟(仅限一次)。
- 打开LMD软件,将聚萘二甲酸乙二醇酯(PEN)膜载玻片正面朝下装到LMD载物台上,标签离用户最近。取消选择程序窗口右侧 的"关闭行"框。
- 在高放大倍率(63x)下使用 PtoP (点对点)函数绘制三个"V"箭头作为校准基准。从 V 上的一个外部点开始,绘制一条线到 V 的中点,然后单击一下。然后,从 V 的中心点到第二个外部 V 点的末端绘制第二条线,然后双击以从两条线创建单个未闭合的 V 形。
注:这些校准基准应放置在滑块的三个角:右前角、右后角、左后方。 - 选择 "剪切前自动对焦" 选项。将载玻片切入位置 1,将其移动到每个剩余的载玻片位置,并在校准切口上精确跟踪。
- 保存 .sld 文件,然后从弹出对话框中选择" 保存而不校准 "选项,以避免将校准基准切割到膜中。
注: 补充文件 1 中提供了一个具有代表性的 .sld 文件,其中包含四个载玻片位置的标准校准器基准。
2. 改性活体压载玻片的制备
注意:本节中描述的实验方案步骤专门用于倒置激光显微镜和相关软件(参见 材料表)。大约时间:5分钟。
- 在切割参考校准基准之前,请确保载玻片完全干燥。打开 LMD 软件,然后在 "导入形状 "选项下打开默认校准 .sld 文件。
- 选择 "剪切前自动对焦" 选项。将纸巾朝下,将标签载玻片装入靠近操作员的位置,放入LMD载物台上的载玻片架中。
- 使用激光显微镜和默认校准.sld文件,将校准基准切割到PEN膜中。
- 可选:在将组织切片放置在载玻片上之前或之后,将校准基准切入PEN膜中。如果在组织放置之前切割校准基准,请确保在步骤2.5中将组织放置在载玻片上时组织和/或固定剂不与校准器重叠。如果在组织放置后切割校准基准,请在步骤2.4完成后停止并继续第3节。
- 单独检查所有校准器,以确保每个切割都是完整且可见的。
注:使用 "移动和剪切" 功能可手动将激光引导到任何未完全穿过 PEN 膜的校准基准上。 - 将冷冻或福尔马林固定的石蜡包埋(FFPE)组织切片放在含有校准基准的载玻片上。
3. 组织染色
注:大约时间:30分钟。
- 将冷冻的LMD组织载玻片固定在含有磷酸酶抑制剂鸡尾酒试剂的70%乙醇(EtOH)中5分钟。
- 在含有磷酸酶抑制剂鸡尾酒试剂的焦氢化物二乙酯(DEPC)水中洗涤载玻片1分钟。
- 在DEPC水中洗涤载玻片1分钟。
- 将载玻片在迈耶的苏木精溶液中孵育3分钟。
- 在DEPC水中冲洗载玻片3分钟。
- 在新鲜的DEPC水中冲洗载玻片1分钟。
- 将载玻片在Eosin Y水溶液中孵育1秒。
- 在 95% 环氧乙烷中冲洗载玻片 2 x 5 秒。
- 在 100% 环氧乙烷中冲洗载玻片 3 x 10 秒。
- 擦拭载玻片背面多余的EtOH,让载玻片风干。
- 如果不立即进行LMD,则将载玻片储存在-80°C。
4. 玻片成像
注意:本节中描述的实验方案步骤特定于扫描的幻灯片(请参阅 材料表)和另存为 .svs 文件的结果图像。使用任何扫描仪及其相关软件,以图像分析软件(参见 材料表)可以打开的格式生成图像文件。支持使用金字塔形短帧的文件类型包括 JPG、TIF、MRXS、QPTIFF、组件式 TIFF、超高频、AFI、超前置式、超音速、直流、OME。多伦多国际电影节、第二届、越南国家统计局、国家警察局、国家警察局、智利、印度边境论坛、科索沃联邦共和国和独立电视台。大约时间:5分钟。
- 打开扫描仪并打开幻灯片扫描仪软件。将载玻片装入纸巾朝上,装入扫描仪中的单载玻片载物台。确保载玻片完全干燥,并将盖玻片轻轻地放在纸巾上。不要在盖玻片下使用乙醇或浸油。
- 根据制造商的说明,使用经过校准的设置捕获显微照片图像,以调整 PEN 膜而不是玻璃背景,并忽略背景膜染色。
- 根据需要,通过拖动绿色内周界并调整其大小来调整成像区域,以捕获整个 PEN 膜区域。通过双击快照概览图像,将四个对焦点添加到组织上,并在校准基准点附近的膜上添加三个对焦点(三个校准基准中每个一个对焦点)。
注意:四个对焦点几乎可以放置在组织切片的任何地方,尽管放置在染色太深且呈黑色的纸上可能会导致扫描失败。 - 在" 视图 "菜单下,选择" 视频监视器"。根据需要,使用精细和/或宏焦滑块手动调整LMD组织周围每个点的焦点。在高放大倍率(20x)下捕获图像扫描。确认所有校准基准在保存的图像中都可见且清晰。
5. 使用图像分析软件自动选择特征
- 对于整个肿瘤集合(大约时间:5分钟,视病例而定):
- 打开图像分析软件(参见 材料表)。选择" 打开图像" ,然后从弹出窗口中选择在 AT2 扫描仪上扫描幻灯片时生成的 .svs 图像文件。
注意: 补充文件 2 中提供了具有代表性的 .svs 映像文件。 - 导航到" 批注 "选项卡。选择"批注"工具栏上的" 钢笔 "工具,然后在组织周围绘制一个形状。
- 选择形状,然后右键单击图像。从"高级"下拉菜单中,选择"分区(平铺)"。将"磁贴大小"和"间距介于两者之间"分别设置为 500 和 40,然后选择"确定"以生成磁贴。在步骤 5.1.2 中选择并删除用于生成切片的周长形状。
- 选择 图层操作 下拉菜单 |导出 以将平铺批注另存为 .annotation 文件。
注意: 补充文件 3 中提供了整个肿瘤组织集合的代表性 .annotation 文件。 - 为会话或项目创建一个文件夹,并将 .annotation 文件保存在标有幻灯片唯一标识符的子文件夹中。
- 导航到" 批注 "选项卡。选择 图层操作 下拉列表 |删除所有图层可 从图像中删除所有批注。选择 钢笔 工具,并从箭头的内尖为每个校准基准绘制一条短线。按以下顺序从标记中绘制线条:左上角、右上角、右下角。
- 选择 图层操作 下拉菜单 |导出 以将线条批注另存为 .annotation 文件。 将_calib 添加到文件名中,并将文件放在包含平铺形状坐标的子文件夹中。
注意: 补充文件 4 中提供了具有代表性的 _calib.annotation 文件。 - 复制主项目或会话文件夹的地址。使用 IDLE 集成开发环境打开 XML 导入生成脚本"Malleator"(可通过 https://github.com/GYNCOE/Mitchell.et.al.2022 获得),并将项目文件夹地址粘贴到脚本底部的引号之间。
- 选择 "运行 "下拉菜单 |运行模块 以执行脚本。
注意:.xml LMD 导入文件将在为图像/幻灯片创建的子文件夹中生成。具有代表性.xml文件在 补充文件5中提供。
- 打开图像分析软件(参见 材料表)。选择" 打开图像" ,然后从弹出窗口中选择在 AT2 扫描仪上扫描幻灯片时生成的 .svs 图像文件。
- 仅适用于富含 LMD 的馆藏(大约时间:15 分钟,视情况而定):
- 打开图像分析软件(参见 材料表)。选择" 打开图像" ,然后从弹出窗口中选择扫描幻灯片时生成的 .svs 图像文件。
- 导航到" 批注 "选项卡。选择并使用 矩形 批注工具在组织周围绘制一个框。
- 选择框注释,然后右键单击图像。选择"高级"下拉菜单|分区(平铺)选项。将"磁贴大小"和"间距介于两者之间"分别设置为 500 和 40,然后选择"确定"以生成磁贴。在步骤 5.2.2 中选择并删除用于生成切片的周长框批注。
- 选择 图层操作 下拉菜单 |导出 以将平铺批注另存为 .annotation 文件。
- 将 Python"Dapọ"( 可通过 https://github.com/GYNCOE/Mitchell.et.al.2022 获得)算法的已保存副本(开发用于合并 AI 分类的注释图层)放入与平铺注释文件相同的文件夹中。
- 复制平铺批注文件的名称。使用 IDLE 集成开发环境打开 Python 程序,并将平铺注释文件的名称粘贴到程序底部的引号之间。
- 选择 "运行 "下拉菜单 |运行模块。等待生成一个新文件,该文件将所有平铺批注合并到单个图层下。
- 打开影像分析软件并导航到 "注释 "选项卡。选择 图层操作 下拉菜单 |删除所有图层可 从图像中删除所有批注。
- 选择 图层操作 下拉菜单 |导入本地注释文件。在弹出窗口中,选择由脚本生成的合并的 .annotation 文件。确保所有导入的切片都位于同一注记图层下。
- 导航到"分类器"选项卡,然后按照制造商的说明为 ROI 生成形状。在运行分类器之前,通过选中"高级分类器选项"下"注释"选项卡上的"ROI"框或框,选择所需的注释层(即肿瘤或基质层)。使用分类器操作菜单中的注释图层选项运行分类器。
- 分类器分析完成后,导航到 注记 选项卡,然后选择从分析生成的注记图层。选择 图层操作 下拉菜单 |删除除当前图层之外的所有图层 可从图像中移除所有其他注记图层。
- 选择 图层操作 下拉菜单 |导出 以将批注另存为 .批注文件。为会话或项目创建一个文件夹,并将 .annotation 文件保存在标有幻灯片唯一标识符的子文件夹中。
注: 补充文件 6 中提供了分类 LMD 富集组织集合的代表性 .annotation 文件。 - 导航到 注记 选项卡,选择 图层操作 下拉列表 |删除所有图层可 从图像中删除所有批注。选择 钢笔 工具,然后从每个校准基准绘制一条短线。按以下顺序从标记中绘制线条:左上角、右上角、右下角。
- 选择 图层操作 下拉菜单 |导出 以将线条批注另存为 .annotation 文件。 将_calib 添加到文件名中,并将该文件放在包含平铺形状坐标的子文件夹中。
- 复制主项目或会话文件夹的地址。使用 IDLE 集成开发环境打开 XML 导入生成脚本"Malleator"( 可通过 https://github.com/GYNCOE/Mitchell.et.al.2022 获得),然后将项目文件夹地址粘贴到脚本底部的引号之间。
- 选择 "运行 "下拉菜单 |运行模块 以执行脚本。
注意:.xml LMD 导入文件将在为图像/幻灯片创建的子文件夹中生成。
6. 激光显微切割
注意:本节中描述的实验方案步骤专门用于倒置激光显微镜和相关软件(参见 材料表)。大约时间:2小时;视情况而定。
- 将标记的膜载玻片(包含校准基准)装载,组织朝下,标签侧更靠近操作员,装入激光显微镜载物台上的载玻片支架。
- 从文件下拉菜单中选择导入形状。选择为幻灯片生成的.xml LMD 导入文件。在弹出窗口中选择否以避免从文件加载参考点,在第二个弹出窗口中选择否以避免使用任何以前存储的参考点进行校准。
- 按照LMD应用程序的提示进行操作,并将校准十字与载玻片上的三个校准基准中的每一个对齐。在图像分析软件中查找出现在载玻片图像的左上角、右上角和右下角的校准基准,这些基准点将分别对应于显微镜载物台上倒置LMD载玻片的右前角、右后角和左后角的参考点。在使用5x物镜定位和使用63x物镜对齐每个校准基准之间进行切换。在弹出窗口中选择" 否 "以避免将参考点保存到文件,在第二个弹出窗口中选择 "确定" 以确认插入幻灯片。
- 将5倍物镜移动到适当的位置,然后在弹出窗口中选择" 是 "以使用实际放大倍率。导入的形状出现后,将相机对焦于纸巾上。
- 突出显示并选择" 形状列表 "窗口中的所有形状,使用视野中的一个或两个注释作为参照将它们拖动到位,然后对齐垂直 z 轴以使用激光进行切割。
- 查看导入的形状,并将它们指定到适当的管位置以进行收集。按 开始剪切 以启动激光。
注意:.xml文件中导入的形状将自动分配到" 形状列表 "窗口中的"无大写"位置。要收获组织,必须将导入的形状重新分配到包含加载管的位置。
7. 通过压力循环技术(PCT)进行蛋白质消化
注意:大约时间:4小时(3小时没有真空离心机干燥时间)。
- 将含有含有LMD收获组织的封盖PCT微管的0.5mL管放入20μL100mM TEAB / 10%乙腈中,放入热循环仪中,在99°C下加热30分钟,然后冷却至50°C10分钟。
- 以4,000×g旋转试管30秒,然后从0.5 mL试管中取出微量管。使用微电容工具,从PCT微管中移除并丢弃微电容。以每30mm2组织1μg的比例加入胰蛋白酶(参见材料表),并使用MicroCap工具将微胃插入微管中。
- 将微管转移到气压柱式墨盒中,并组装完整的墨盒。将墨盒放入气压罐中并盖上盖子。巴罗周期在 45,000 psi 下持续 50 秒,大气压下 10 秒,在 50 °C 下持续 60 次循环。
- 气压环切完成后,将微量管转移到0.5mL微量离心管中,并在4,000× g下离心2分钟。
- 使用盖子工具从0.5 mL微量离心管中取出微量离心管。使用盖子工具小心地取出杵,并用20μL液相色谱 - 质谱(LC-MS)级水冲洗杵的下半部分,并将洗涤液收集到干净的0.5mL微量离心管中。
- 轻轻敲击台面上的微量管,将液体移至底部,并将微量管中的所有溶液转移到0.5 mL微量离心管中。
- 向微量管中加入 20 μL LC-MS 级水,轻轻敲击台面。将洗涤液转移到0.5 mL管中,并再次重复该洗涤步骤。
- 真空离心机将样品干燥至~2 μL,并加入100 μL 100 mM TEAB,pH 8.0。
- 根据制造商的方案,使用比色测定法(双新胆酸(BCA)测定,参见 材料表)测定肽浓度。
8. 串联质量标签 (TMT) 标签和易闪清理
注意:大约时间:7小时20分钟(2小时20分钟,没有真空离心机干燥时间)。
- 打开前将等压TMT标记试剂置于环境温度。向每个TMT小瓶(5mg)中加入500μL100%乙腈。孵育10分钟,偶尔涡旋。
- 将5μg肽样品溶解在100μL100mM TEAB,pH 8.0中,并加入10μL给定的TMT试剂。在每个TMT多路复用样品集中构建并包括代表实验中每个单独样品的参考池,以促进对多个TMT多路复用器中的样品进行定量9。在环境温度下孵育反应1小时,偶尔摇动/敲击。
- 通过加入10μL5%羟胺来淬灭TMT标记反应,并在环境温度下孵育30分钟,偶尔敲击。淬灭后,将TMT标记的样品混合到一个管中并干燥至约200μL。
- 加入1,800μL 0.1%甲酸。用pH纸检查pH值:如果pH~3,则加入1mL 0.1%甲酸;如果pH>3,加入10-20μL5%甲酸,直到pH〜3。加入0.1%甲酸,使最终体积达到3 mL。
- 取下肽纯化柱底部的标签,取下盖子,然后将其置于15mL锥形管中。将TMT标记的样品转移到色谱柱中,然后根据制造商的协议进行清理。
- 真空离心机将洗脱的肽干燥至〜20μL,使用25mM碳酸氢铵转移到LC小瓶中,最终体积为80μL,然后进行离线分馏。
9. TMT多路复用样品分馏和池化
注意:大约时间:3小时30分钟(1小时30分钟,没有真空离心机干燥时间)。
- 通过将流动相B(乙腈)的线性梯度递增(0.69% min-1)发展为流动相A(10 mM NH4HCO3,pH 8.0),通过碱性反相色谱将TMT标记的肽多重分馏成96个级分。
- 通过池化样品孔生成 36 个串联的馏分。真空离心至干燥级分至~2μL,重悬于25mM碳酸氢铵(终浓度1.5μg/ 10μL),在15,000× g 离心10-15分钟,然后转移到LC小瓶中进行MS分析。
10. 液相色谱串联质谱 (LC-MS/MS)
注:大致时间:取决于仪器方法和实验设计。
- 根据制造商的说明/方案校准质谱仪。
- 制备新鲜的流动相和标准品,并进行适当的LC预运行准备(包括但不限于吹扫溶剂,冲洗空气和参考仪器的泄漏测试脚本[参见 材料表])。在开始分析之前平衡预分析柱和分析柱以及样品循环。
- 在连续TMT多重分析之前和之间,使用质量保证/质量控制(QA / QC)TMT标记肽消化物和(例如MSPE(参见 材料表),HeLa)验证LC-MS系统是否符合先前的基准性能指标。
- 将自动进样器小瓶加载到 LC 自动进样器的适当位置。使用适当的梯度/MS方法分析单个分数。将肽标准品(例如,肽保留时间校准 [PRTC])穿插一次"洗涤"运行,大约每天一次,以评估色谱和质谱性能。在分析每个TMT多重样品分数系列后,运行QA/QC TMT基准标准以评估系统性能。
- 在QA / QC TMT基准标准后运行质谱仪评估程序,以评估样品后性能,然后按照步骤10.1中的步骤校准系统以进行下一个样品集。
11. 生物信息学数据分析
注:大致时间:取决于实验设计。
- 将所有示例数据(例如,.raw文件)传输到适当的网络存储/计算机驱动器。
- 使用所需的数据分析应用程序(例如,蛋白质组发现,Mascot)使用适当的参数9与物种特异性蛋白质参考数据库一起搜索所有级分,以生成肽光谱匹配(PSM)并提取TMT报告离子信号强度。如前所述,根据适当的质量控制指标过滤PSM,并将归一化的中位数对数2转换TMT报告离子比丰度聚合为全局蛋白质水平丰度。
- 使用所需的差分分析软件比较感兴趣条件下的蛋白质变化。
结果
使用这种集成的AI驱动的组织ROI识别,分割,LMD和定量蛋白质组学分析工作流程分析来自两名HGSOC和两名OCCC患者的新鲜冷冻组织薄片(图1)。每个肿瘤的代表性H&E染色组织切片由董事会认证的病理学家进行审查;肿瘤细胞率从70%到99%不等。将组织薄片切到PEN膜载玻片上(补充文件2),并使用校准器基准(补充文件1)进行预切割,从而能够将图像分析软件(参见 材料表)中生成的注释中的位置方向数据与LMD软件中的笛卡尔坐标方向集成在一起。H&E染色后,捕获了包含组织和校准器的PEN载玻片的高分辨率图像(20x)。
使用图像分析软件(参见 材料表)对显微照片中的肿瘤和基质细胞群进行分割,以便通过LMD进行选择性收获,以及代表整个组织薄片(例如整个肿瘤组织)的收获(图1)。通过用500μm2的瓷砖分割整个组织切片来生成整个肿瘤组织集合的无区别注释,在瓷砖之间留出40μm的间隙以保持PEN膜的完整性并防止膜在LMD期间卷曲。在用于组织学分辨LMD富集的载玻片上,图像分析软件中的AI分类器(参见 材料表)被训练以区分肿瘤和基质细胞,以及空白载玻片背景。手动突出显示具有代表性的肿瘤,基质和空白玻璃区域,并使用分类器工具将这些ROI分割到整个组织切片中。代表整个组织,肿瘤上皮和基质的分割层分别保存为单独的.annotation文件(补充文件3 和 补充文件6)。在图像文件的单独副本(没有分区的 ROI 注释)中,对三个基准校准器中最中心的尖端处的一条短线进行注释,并使用与每个 LMD 注释图层文件相同的文件名保存为 .annotation 文件,但附加后缀为"_calib"(补充文件 4)。这些线用于将PEN膜校准器的位置与图像分析软件中绘制的注释形状列表数据共同注册。
本研究提供了两种算法,即Python中的"马利特"和"Dapọ",以支持这种AI驱动的LMD工作流程,这些工作流程可在 https://github.com/GYNCOE/Mitchell.et.al.2022 获得。Malleator 算法从配对的 .annotation 文件中提取所有单个注释(组织 ROI 和校准器)的特定笛卡尔坐标,并将其合并到单个可扩展标记语言 (XML) 导入文件(补充文件 5)中。具体而言,Malleator 算法使用父文件夹中的目录名称作为输入来搜索所有子目录文件夹,并为尚未具有.xml合并文件的任何子文件夹生成.xml文件。Malleator 算法将图像分析软件(参见 材料表)中的所有注释图层合并为单个图层,并将 AI 生成的形状列表数据(另存为专有 .annotation 文件类型)转换为与 LMD 软件兼容.xml格式。合并注释和校准器文件后,算法生成的.xml文件将被保存并导入到LMD软件中。手动调整注释的对齐方式需要稍作调整,这也可用于在激光显微镜上记录载玻片载物台的垂直(z平面)位置。Dapọ 算法专门用于 LMD 丰富的集合。分割切片由影像分析软件自动分配给各个注记图层。在使用分类器工具之前,Dapọ 算法会将所有分区切片合并到单个注记图层中,从而减少 LMD 丰富集合的分类器分析运行时间。
消化整个肿瘤和富含LMD的组织样品,用TMT试剂标记,多重检测,离线分馏, 并通过 基于MS的定量蛋白质组学进行分析,如前所述9。使用这种AI驱动的工作流程收获的样品的平均肽产量(43-60μg)和回收率(0.46-0.59μg/ mm2)与以前的报告9,10相当。在所有样品中共对5,971种蛋白质进行了共定量(补充表S1)。使用100种最可变的蛋白质的无监督分层聚类导致HGSOC和OCCC组织型从LMD富集和整个肿瘤样品中分离(图2A),类似于前面描述的11。相比之下,来自HGSOC和OCCC的LMD富集基质样品聚集在一起,独立于富含LMD的肿瘤和整个肿瘤样品。在5,971种定量蛋白质中,HGSOC和OCCC标本的整个肿瘤集合之间有215种显着改变(LIMMA调整后<0.05)(补充表S2)。将这些改变的蛋白质与Hughes等人鉴定的用于区分HGSOC和OCCC肿瘤组织的蛋白质进行比较。在Hughes等人定量的76种特征蛋白中,有57种在该数据集中共定量并且高度相关(Spearman Rho = 0.644,p< 0.001)(图2B)。
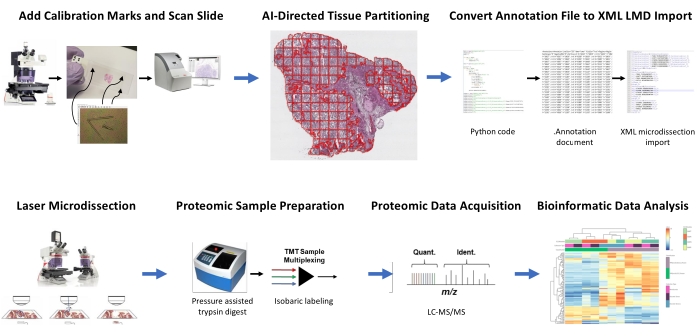
图 1:用于下游定量蛋白质组学的激光显微切割的自动组织感兴趣区域选择集成工作流程摘要。 校准基准被切割到PEN膜载玻片上,以在LMD显微镜上水平定位,在图像分析软件HALO中共同记录来自AI衍生的组织ROI片段的位置方向数据。Malleator 算法用于将幻灯片的所有注释图层的带注释的分割数据与_calib参考文件合并,并将其转换为与 LMD 软件兼容的.xml文件。用于蛋白质组学分析的LMD收获的组织通过高通量蛋白质组学消化和分析,如前所述9。缩写: LMD = 激光显微切割;投资回报率 = 感兴趣的区域;TMT = 串联质量标签;定量 = 量化;标识。= 识别;LC-MS/MS = 液相色谱-串联质谱。 请点击此处查看此图的大图。
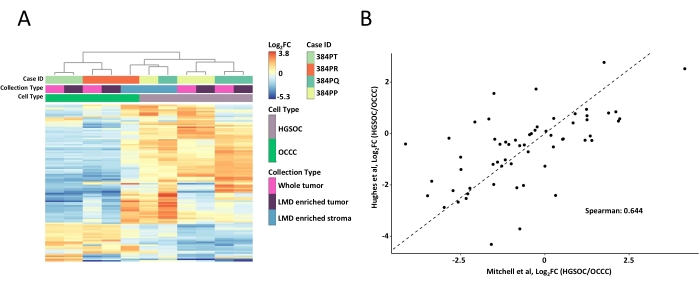
图2:LMD富集和整个肿瘤样品中蛋白质的分析(A)对HGSOC和OCCC LMD富集和整个肿瘤样品中100种最可变丰富的蛋白质进行无监督分层聚类分析。(B)本研究中HGSOC和OCCC全肿瘤收获量(米切尔等人,x轴)与休斯等人(y轴)的类似研究11的对数2倍变化蛋白质丰度的相关性。缩写: LMD = 激光显微切割;HGSOC = 高级别浆液性卵巢癌;OCCC = 卵巢透明细胞癌;对数2FC = 对数2 转化蛋白质组丰度。请点击此处查看此图的大图。
补充表S1:来自HGSOC和OCCC组织标本的所有LMD富集和整个肿瘤样品中共定量的5,971种蛋白质的丰度。 缩写: LMD = 激光显微切割;HGSOC = 高级别浆液性卵巢癌;OCCC = 卵巢透明细胞癌。 请按此下载此表格。
补充表S2:来自HGSOC与OCCC的整个肿瘤集合中差异表达的蛋白质(215)(LIMMA调整后第<0.05页)。 简称:HGSOC =高级别浆液性卵巢癌;OCCC = 卵巢透明细胞癌。 请按此下载此表格。
补充文件 1:包含四个载玻片位置的标准校准器基准的代表性形状列表数据 (.sld) 文件。 该文件可以导入到 LMD 软件中。 请按此下载此档案。
补充文件 2:H&E 染色的高分辨率 (20x) 组织切片的代表性 .svs 图像文件。 可以使用图像分析软件或LMD软件打开和查看该文件。缩写:H&E = 苏木精和曙红;LMD = 激光显微切割。 请按此下载此档案。
补充文件3:分区的整个肿瘤片段的代表性.注解文件。 该文件可以导入到图像分析软件中。 请按此下载此档案。
补充文件 4:校准器基准段的代表性_calib注释文件。 坐标信息表示从每个箭头基准点绘制的短校准器线的东方定位。该文件可以导入到图像分析软件中。 请按此下载此档案。
补充文件 5:由 Malleator 算法生成的具有代表性的可扩展标记语言 (.xml) 文件。 该文件可以导入到激光显微切割软件中。 请按此下载此档案。
补充文件 6:用于 LMD 丰富集合的分区 AI 分类段的代表性 .annotation 文件。 该文件可以导入到图像分析软件中。缩写:AI = 人工智能;LMD = 激光显微切割。 请按此下载此档案。
讨论
虽然已有多个研究先例旨在开发和/或改进工作流程,以富集FFPE和/或新鲜冷冻组织中的目标细胞亚群以及在处理过程中保持样品质量的方法9,12,13,14,15,但仍迫切需要开发自动化策略来制备用于分子分析的临床组织标本,以减少变异性和 提高再现性。该工作流程描述了一种标准化的半自动方案,该方案集成了现有的图像分析软件工具(参见材料表),可通过LMD从临床组织标本中以组织学方式分离收获离散细胞群。
捕获离散细胞群的ROI的空间分辨LMD富集代表了多组学分析之前的下一代组织处理步骤,以改善分子表征和鉴定并促进细胞选择性生物标志物的发现。该协议通过减少组织切片经常长时间暴露于周围环境(与组织学家手动分割ROI相关的环境)(在LMD收集之前可能需要>1-2小时)来改进现有方法。相反,此工作流允许通过 AI 引导的分类和细分来预先确定 ROI。限制组织停留时间将减少高度不稳定的分子靶标(如磷酸肽和mRNA)评估中的虚假变化,或用于依赖于靶蛋白在其天然构象中的基于抗体的分析技术进行检测。
在扫描的载玻片图像中清晰可见的 PEN 膜载玻片上切割整齐的校准器基准是实现图像分析软件(参见 材料表)与 LMD 工作流程集成的关键组件之一。确保校准器在"V"形底部有一个精确("干净")的点,可以在图像分析软件中选择一个精确的点,以便从中绘制校准器线,如步骤5.1.6和5.2.13所述。在导入到LMD软件的过程中,这些点的对齐对于将注释(通过使用"Malleator"和/或"Dapọ"算法生成兼容的.xml文件)正确叠加到物理LMD载玻片上的相关组织ROI上至关重要。有必要突出显示所有形状,并集体"拖放"到位,即使导入LMD软件时对齐是精确的,以在激光显微镜上记录载玻片载物台的垂直(z平面)位置。如果需要,也可以在此步骤中对组织ROI上的注释位置进行微小调整。
当前版本的 Malleator 算法的局限性在于它与图像分析软件提供的预定义注释形状工具不兼容(参见 材料表),尽管该算法的未来更新/版本将旨在提高这种兼容性。使用这些工具绘制的形状的 .annotation 文件对于每个注记仅包含两组成对的 x 和 y 坐标,而没有围绕这些点的完整空间方向。目前使用这些工具会导致注释在导入过程中转换为仅由两个点定义的直线。要成功转换为 XML 格式和 LMD 导入,需要手动定义组织 ROI 段。这可以通过手动定义每个ROI来执行,方法是使用特定于目标区域的单个徒手多边形注释,或者如果需要,可以在所有组织ROI段上应用近似的圆形或矩形注释,并且与此工作流程兼容。
虽然这里介绍的工作流程被证明用于新鲜冷冻的人类癌症组织标本的蛋白质组学分析,但这种AI驱动的LMD工作流程可以等效地用于FFPE组织,非癌组织类型以及来自非人类来源的组织类型。它还可以支持其他下游分子分析工作流程,包括转录组学、基因组学或磷酸化蛋白质组学分析。该工作流程还可以利用图像分析软件的其他用途(参见 材料表),包括与细胞计数或其他分析模块相关的功能,包括"多重IHC"模块或"组织微阵列(TMA)附加组件"。该工作流程的未来应用也可能受益于预定义每个ROI片段的细胞数量,从而确保多个集合之间的等效细胞输入,或者通过使用替代方法来定义感兴趣的细胞ROI,例如免疫组化或细胞社会学。
披露声明
T.P.C.是赛默飞世尔科技公司SAB成员,并获得艾伯维的研究资助。
致谢
该项目的资金部分由国防健康计划(HU0001-16-2-0006和HU0001-16-2-00014)提供给统一服务大学妇科癌症卓越中心。申办方在研究的设计、执行、解释或撰写中没有任何作用。 免責聲明: 本文表达的观点是作者的观点,并不反映陆军/海军/空军部,国防部或美国政府的官方政策。
材料
| Name | Company | Catalog Number | Comments |
| 1260 Infinity II System | Agilent Technologies Inc | Offline LC system | |
| 96 MicroCaps (150uL) in bulk | Pressure Biosciences Inc | MC150-96 | |
| 96 MicroPestles in bulk | Pressure Biosciences Inc | MP-96 | |
| 96 MicroTubes in bulk (no caps) | Pressure Biosciences Inc | MT-96 | |
| 9mm MS Certified Clear Screw Thread Kits | Fisher Scientific | 03-060-058 | Sample vial for offline LC frationation and mass spectrometry |
| Acetonitrile, Optima LC/MS Grade | Fisher Chemical | A995-4 | Mobile phase solvent |
| Aperio AT2 | Leica Microsystems | 23AT2100 | Slide scanner |
| Axygen PCR Tubes with 0.5 mL Flat Cap | Fisher Scientific | 14-222-292 | Sample tubes; size fits PCT tubes and thermocycler |
| Barocycler 2320EXT | Pressure Biosciences Inc | 2320-EXT | Barocycler |
| BCA Protein Assay Kit | Fisher Scientific | P123225 | |
| cOmplete, Mini, EDTA-free Protease Inhibitor Cocktail | Roche | 11836170001 | |
| Easy-nLC 1200 | Thermo Fisher Scientific | Liquid Chromatography | |
| EasyPep Maxi Sample Prep Kit | Thermo Fisher Scientific | NCI5734 | Post-label sample clean up column |
| EASY-SPRAY C18 2UM 50CM X 75 | Fisher Scientific | ES903 | Analytical column |
| Eosin Y Solution Aqueous | Sigma Aldrich | HT110216 | |
| Formic Acid, 99+ % | Thermo Fisher Scientific | 28905 | Mobile phase additive |
| ggplot2 version 3.3.5 | CRAN | https://cran.r-project.org/web/packages/ggplot2/ | |
| HALO | Indica Labs | Image analysis software | |
| IDLE (Integrated Development and Learning Environment) | Python Software Foundation | ||
| iheatmapr version 0.5.1 | CRAN | https://cran.r-project.org/web/packages/iheatmapr/ | |
| iRT Kit | Biognosys | Ki-3002-1 | LC-MS QAQC Standard |
| limma version 3.42.2 | Bioconductor | https://bioconductor.org/packages/release/bioc/html/limma.html | |
| LMD Scanning stage Ultra LMT350 | Leica Microsystems | 11888453 | LMD stage model outfitted with PCT tube holder |
| LMD7 (software version 8.2.3.7603) | Leica Microsystems | LMD apparatus (microscope, laser, camera, PC, tablet) | |
| Mascot Server | Matrix Science | Data analysis software | |
| Mass Spec-Compatible Human Protein Extract, Digest | Promega | V6951 | LC-MS QAQC Standard |
| Mayer’s Hematoxylin Solution | Sigma Aldrich | MHS32 | |
| PEN Membrane Glass Slides | Leica Microsystems | 11532918 | |
| Peptide Retention Time Calibration Mixture | Thermo Fisher Scientific | 88321 | LC-MS QAQC Standard |
| Phosphatase Inhibitor Cocktail 2 | Sigma Aldrich | P5726 | |
| Phosphatase Inhibitor Cocktail 3 | Sigma Aldrich | P0044 | |
| Pierce LTQ Velos ESI Positive Ion Calibration Solution | Thermo Fisher Scientific | 88323 | Instrument calibration solution |
| PM100 C18 3UM 75UMX20MM NV 2PK | Fisher Scientific | 164535 | Pre-column |
| Proteome Discoverer | Thermo Fisher Scientific | OPTON-31040 | Data analysis software |
| Python | Python Software Foundation | ||
| Q Exactive HF-X | Thermo Fisher Scientific | Mass spectrometer | |
| R version 3.6.0 | CRAN | https://cran-archive.r-project.org/bin/windows/base/old/2.6.2/ | |
| RColorBrewer version 1.1-2 | CRAN | https://cran.r-project.org/web/packages/RColorBrewer/ | |
| Soluble Smart Digest Kit | Thermo Fisher Scientific | 3251711 | Digestion reagent |
| TMTpro 16plex Label Reagent Set | Thermo Fisher Scientific | A44520 | isobaric TMT labeling reagents |
| Veriti 60 well thermal cycler | Applied Biosystems | 4384638 | Thermocycler |
| Water, Optima LC/MS Grade | Fisher Chemical | W6-4 | Mobile phase solvent |
| ZORBAX Extend 300 C18, 2.1 x 12.5 mm, 5 µm, guard cartridge (ZGC) | Agilent Technologies Inc | 821125-932 | Offline LC trap column |
| ZORBAX Extend 300 C18, 2.1 x 150 mm, 3.5 µm | Agilent Technologies Inc | 763750-902 | Offline LC analytical column |
参考文献
- Motohara, T., et al. An evolving story of the metastatic voyage of ovarian cancer cells: cellular and molecular orchestration of the adipose-rich metastatic microenvironment. Oncogene. 38 (16), 2885-2898 (2019).
- Aran, D., Sirota, M., Butte, A. J. Systematic pan-cancer analysis of tumour purity. Nature Communications. 6, 8971 (2015).
- Hunt, A. L., et al. Extensive three-dimensional intratumor proteomic heterogeneity revealed by multiregion sampling in high-grade serous ovarian tumor specimens. iScience. 24 (7), 102757 (2021).
- Dou, Y., et al. Proteogenomic characterization of endometrial carcinoma. Cell. 180 (4), 729-748 (2020).
- Zhang, H., et al. Integrated proteogenomic characterization of human high-grade serous ovarian cancer. Cell. 166 (3), 755-765 (2016).
- Gillette, M. A., et al. Proteogenomic characterization reveals therapeutic vulnerabilities in lung adenocarcinoma. Cell. 182 (1), 200-225 (2020).
- Silvestri, A., et al. Protein pathway biomarker analysis of human cancer reveals requirement for upfront cellular-enrichment processing. Laboratory Investigation. 90 (5), 787-796 (2010).
- Echle, A., et al. Deep learning in cancer pathology: a new generation of clinical biomarkers. British Journal of Cancer. 124 (4), 686-696 (2021).
- Lee, S., et al. Molecular analysis of clinically defined subsets of high-grade serous ovarian cancer. Cell Reports. 31 (2), 107502 (2020).
- Xuan, Y., et al. Standardization and harmonization of distributed multi-center proteotype analysis supporting precision medicine studies. Nature Communications. 11 (1), 5248 (2020).
- Hughes, C. S., et al. Quantitative profiling of single formalin fixed tumour sections: proteomics for translational research. Scientific Reports. 6 (1), 34949 (2016).
- Espina, V., et al. A portrait of tissue phosphoprotein stability in the clinical tissue procurement process. Molecular & Cellular Proteomics. 7 (10), 1998-2018 (2008).
- Espina, V., Heiby, M., Pierobon, M., Liotta, L. A. Laser capture microdissection technology. Expert Review of Molecular Diagnostics. 7 (5), 647-657 (2007).
- Havnar, C. A., et al. Automated dissection protocol for tumor enrichment in low tumor content tissues. Journal of Visualized Experiments. (169), e62394 (2021).
- Mueller, C., et al. One-step preservation of phosphoproteins and tissue morphology at room temperature for diagnostic and research specimens. PLoS One. 6 (8), (2011).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。