Method Article
מיקרו-דיסקציה מתועשת, מונחית בינה מלאכותית בלייזר לניתוח פרוטאומי בקנה מידה זעיר של המיקרו-סביבה של הגידול
* These authors contributed equally
In This Article
Summary
פרוטוקול זה מתאר זרימת עבודה בתפוקה גבוהה לסגמנטציה מונחית בינה מלאכותית של אזורי עניין שאושרו על-ידי פתולוגיה מתמונות מוכתמות ודקות של מקטעי רקמה להעשרת אוכלוסיות תאים שנפתרו על-ידי היסטולוגיה באמצעות מיקרו-דיסקציה של לייזר. אסטרטגיה זו כוללת אלגוריתם חדשני המאפשר העברת תיחום המציין אוכלוסיות תאים בעלי עניין ישירות למיקרוסקופי לייזר.
Abstract
המיקרו-סביבה של הגידול (TME) מייצגת מערכת אקולוגית מורכבת המורכבת מעשרות סוגי תאים מובחנים, כולל אוכלוסיות גידולים, סטרומה ותאי חיסון. כדי לאפיין את השונות ברמת הפרוטאום ואת ההטרוגניות של הגידול בקנה מידה גדול, יש צורך בשיטות בעלות תפוקה גבוהה כדי לבודד באופן סלקטיבי אוכלוסיות תאיות בדידות בממאירות של גידולים מוצקים. פרוטוקול זה מתאר זרימת עבודה בתפוקה גבוהה, המתאפשרת על ידי בינה מלאכותית (AI), המפלחת תמונות של חלקי רקמות דקים ומוכתמים בהמטוקסילין (H&E) ומוכתמים ב-Eosin (H&E) לאזורי עניין שאושרו על-ידי פתולוגיה לצורך קצירה סלקטיבית של אוכלוסיות תאים שנפתרו על-ידי היסטולוגיה באמצעות מיקרו-דיסקציה של לייזר (LMD). אסטרטגיה זו כוללת אלגוריתם חדשני המאפשר העברה של אזורים המציינים אוכלוסיות תאים בעלי עניין, מבוארים באמצעות תוכנת תמונה דיגיטלית, ישירות למיקרוסקופי לייזר, ובכך מאפשרים אוספים קלים יותר. יישום מוצלח של זרימת עבודה זו בוצע, והדגים את התועלת של שיטה הרמונית זו לקציר סלקטיבי של אוכלוסיות תאי גידול מה- TME לצורך ניתוח פרוטאומי כמותי, מרובב על ידי ספקטרומטריית מסה ברזולוציה גבוהה. אסטרטגיה זו משתלבת באופן מלא עם סקירה היסטופתולוגית שגרתית, תוך מינוף ניתוח תמונות דיגיטליות כדי לתמוך בהעשרה של אוכלוסיות תאיות בעלות עניין והיא ניתנת להכללה מלאה, ומאפשרת קציר הרמוני של אוכלוסיות תאים מה-TME לצורך ניתוחים רב-אומיים.
Introduction
ה-TME מייצג מערכת אקולוגית מורכבת המאוכלסת על ידי מערך מגוון מאוד של סוגי תאים, כגון תאי גידול, תאים סטרומליים, תאים סטרומליים, תאי מערכת החיסון, תאי אנדותל, סוגי תאים מזנכימליים אחרים ואדיפוציטים, יחד עם מטריצה חוץ-תאית מורכבת1. מערכת אקולוגית תאית זו משתנה בתוך אתרי איברי המחלה השונים ועל פניהם, וכתוצאה מכך ההטרוגניות המורכבת של הגידול 2,3. מחקרים אחרונים הראו כי גידולים הטרוגניים וגידולים עם תאי גידול נמוכים (טוהר נמוך) מתואמים לעתים קרובות עם פרוגנוזהשל מחלה לקויה 2,3.
כדי להבין את יחסי הגומלין המולקולריים בין אוכלוסיות של תאים סרטניים ולא סרטניים בתוך ה-TME בקנה מידה גדול, יש צורך באסטרטגיות סטנדרטיות ותפוקה גבוהה כדי לקצור באופן סלקטיבי אוכלוסיות תאיות שונות בעלות עניין לצורך ניתוח מולטיומי במורד הזרם. פרוטאומיקה כמותית מייצגת טכניקה מתפתחת במהירות וחשובה יותר ויותר לקידום ההבנה של הביולוגיה של הסרטן. עד כה, הנטייה המוקדמת של מחקרים שהשתמשו בפרוטאומיקה עשתה זאת עם חלבונים המופקים מהכנת רקמות גידול שלמות (למשל, בהקפאה), מה שהוביל לירידה בהבנת ההטרוגניות ברמת הפרוטאום ב-TME 4,5,6.
פיתוח אסטרטגיות איסוף דגימות המשתלבות בצורה חלקה עם זרימות עבודה של פתולוגיה קלינית ורותמות אותו, יאפשר דור חדש של פרוטאומיקה הנפתרת על-ידי היסטולוגיה, המשלימים מאוד את תהליכי העבודה של פתולוגיה אבחנתית בתקן זהב. LMD מאפשר איסוף ישיר וסלקטיבי של תת-אוכלוסיות תאיות או אזורי עניין (ROIs) באמצעות בדיקה מיקרוסקופית של מקטעים דקים של רקמות מוכתמות היסטולוגית7. ההתקדמות הגדולה האחרונה בפתולוגיה דיגיטלית ובניתוח מבוסס בינה מלאכותית הדגימו את היכולת לזהות תכונות הרכב ייחודיות ו- ROIs בתוך ה- TME באופן אוטומטי, שרבים מהם מתואמים עם שינויים מולקולריים ותכונות מחלה קליניות, כגון עמידות לטיפול ופרוגנוזה של מחלות8.
זרימת העבודה המתוארת בפרוטוקול המוצג כאן ממנפת פתרונות תוכנה מסחריים כדי לבאר באופן סלקטיבי ROIs של גידולים בתוך תמונות היסטופתולוגיה דיגיטליות, ומשתמשת בכלי תוכנה שפותחו בתוך החברה כדי להעביר את ה- ROIs של הגידולים הללו למיקרוסקופי לייזר לצורך איסוף אוטומטי של אוכלוסיות תאיות נפרדות בעלות עניין המשתלב בצורה חלקה עם זרימות עבודה של ניתוח רב-אומי במורד הזרם. אסטרטגיה משולבת זו מפחיתה באופן משמעותי את זמן מפעיל ה- LMD וממזערת את משך הזמן שעבורו רקמות נדרשות להיות בטמפרטורת הסביבה. השילוב של בחירת תכונות אוטומטית וקציר LMD עם פרוטאומיקה כמותית בתפוקה גבוהה מודגם באמצעות ניתוח דיפרנציאלי של ה-TME משני תת-סוגים היסטולוגיים מייצגים של סרטן השחלות האפיתליאלי, סרטן שחלות סרוס בדרגה גבוהה (HGSOC) וקרצינומה של תאים צלולים בשחלות (OCCC).
Protocol
כל פרוטוקולי המחקר אושרו לשימוש תחת פרוטוקול מערבי שאושר על ידי IRB "ניתוח מולקולרי משולב של סרטן רירית הרחם והשחלות כדי לזהות ולאמת סמנים ביולוגיים אינפורמטיביים מבחינה קלינית" הנחשבים פטורים על פי התקנה הפדרלית של ארה"ב 45 CFR 46.102(f). כל הפרוטוקולים הניסוייים שכללו נתונים אנושיים במחקר זה היו בהתאם להצהרת הלסינקי. הסכמה מדעת התקבלה מכל הנבדקים שהשתתפו במחקר.
אזהרה: הריאגנטים הבאים המשמשים לאורך הפרוטוקול ידועים או חשודים כמסרטן ו/או מכילים חומרים מסוכנים: אתנול, מי DEPC, תמיסת המטוקסילין של מאייר, תמיסת Eosin Y, מתנול, אצטוניטריל וחומצה פורמית. טיפול נכון, כמתואר בגיליונות נתוני הבטיחות המתאימים (SDS), ושימוש בציוד מגן אישי מתאים (PPE) הוא חובה.
1. יצירת קובץ נתוני רשימת הצורות המוגדר כברירת מחדל (.sld) המכיל פידוקיאלים של כיול
הערה: שלבי הפרוטוקול המתוארים בסעיף זה הם ספציפיים לשימוש עם מיקרוסקופ לייזר הפוך והתוכנה המשויכת (ראה טבלת החומרים). יצירת קובץ .sld המוגדר כברירת מחדל נחוצה רק פעם אחת לכל מיקרוסקופ לייזר. ניתן להשתמש בקובץ המתקבל לחיתוך fiducials לתוך כל שקופיות PEN המשמשות לאחר מכן. זמן משוער: 5 דקות (פעם אחת בלבד).
- פתח את תוכנת ה- LMD וטען את ממברנת הפוליאתילן נפטלט (PEN) החלקה על במת ה- LMD עם הפנים כלפי מטה, כאשר התווית קרובה ביותר למשתמש. בטל את הבחירה בתיבה 'סגור קווים' בצד ימין של חלון התוכנית.
- השתמש בפונקציית PtoP (נקודה לנקודה) תחת הגדלה גבוהה (63x) כדי לצייר שלושה חצים "V" שישמשו כפידוקיאלים לכיול. החל מנקודה חיצונית אחת ב- V, צייר קו לנקודת האמצע של ה- V ולחיצה בודדת. לאחר מכן, צייר קו שני מהנקודה המרכזית של ה-V לסוף נקודת ה-V החיצונית השנייה, ולחץ פעמיים כדי ליצור צורת V אחת לא סגורה משני הקווים.
הערה: יש למקם את הפידוקיאלים האלה בשלוש פינות של המגלשה: ימין קדמי, ימין אחורי, שמאל אחורי. - בחר את המיקוד האוטומטי (מיקוד אוטומטי) לפני אפשרות החיתוך . חתכו את השקופית במצב 1, העבירו אותה לכל אחד ממיקומי השקופיות הנותרים ועקבו במדויק אחר חיתוכי הכיול.
- שמור את קובץ ה- .sld ובחר באפשרות שמור ללא כיול מתיבת הדו-שיח המוקפצת כדי להימנע מחיתוך הפידוקיאלים של הכיול לממברנה.
הערה: קובץ .sld מייצג המכיל פידוקיאלים כיול סטנדרטיים לארבעה מיקומי שקופיות מסופק בקובץ משלים 1.
2. הכנת שקופיות LMD
הערה: שלבי הפרוטוקול המתוארים בסעיף זה הם ספציפיים לשימוש עם מיקרוסקופ לייזר הפוך והתוכנה המשויכת (ראה טבלת החומרים). זמן משוער: 5 דקות.
- ודא שהמגלשה יבשה לחלוטין לפני חיתוך פידוקיאלים של כיול הייחוס. פתח את תוכנת LMD ופתח את קובץ ה- .sld של כיול ברירת המחדל תחת האפשרות ייבוא צורות .
- בחר את המיקוד האוטומטי (מיקוד אוטומטי) לפני אפשרות החיתוך . טען את השקופיות כאשר הרקמה פונה כלפי מטה והתווית מחליקה קרוב יותר למפעיל למחזיק השקופיות בשלב ה- LMD.
- באמצעות מיקרוסקופ הלייזר וקובץ .sld כיול ברירת המחדל, חתכו את הפידוקיאלים של הכיול לממברנת PEN.
- אופציונלי: חתכו את הפידוקיאלים של הכיול לתוך קרום ה-PEN לפני או אחרי שמקטעי הרקמה ממוקמים/ממוקמים על המגלשה. אם חוטי הכיול נחתכים לפני מיקום הרקמה, ודא שהרקמה ו/או הקיבוע אינם חופפים לכיולים כאשר הרקמה ממוקמת על המגלשה בשלב 2.5. אם פיסוציאלי הכיול נחתכים לאחר מיקום הרקמה, יש להפסיק לאחר השלמת שלב 2.4 ולהמשיך לסעיף 3.
- סקור את כל הכיולים בנפרד כדי לוודא שכל חתך שלם וגלוי.
הערה: השתמש בתכונה 'הזזה וחיתוך' כדי לכוון את הלייזר באופן ידני מעל כל פידוקיאלים של כיול שלא חתכו באופן מלא את קרום ה-PEN. - מקם את מקטע הרקמה הקפואה או הקבועה בפורמלין, המוטבעת בפרפין (FFPE) על השקופית המכילה את הפידוקיאלים של הכיול.
3. צביעת רקמות
הערה: זמן משוער: 30 דקות.
- תקן את רקמת ה-LMD הקפואה ב-70% אתנול (EtOH) המכיל ריאגנטים של קוקטייל מעכבי פוספטאז למשך 5 דקות.
- שטפו את המגלשות במי דיאתיל פירוקרבומט (DEPC) המכילים ריאגנטים של קוקטייל מעכבי פוספטאז למשך דקה אחת.
- שטפו את המגלשות במי DEPC למשך דקה אחת.
- הדגירו את השקופיות בתמיסת ההמטוקסילין של מאייר למשך 3 דקות.
- יש לשטוף את המגלשות במי DEPC למשך 3 דקות.
- יש לשטוף את המגלשות בהחלפה טרייה של מי DEPC למשך דקה אחת.
- דגירה של השקופיות בתמיסת Eosin Y מימית למשך 1 שניות.
- יש לשטוף את השקופיות ב-2 x 5 שניות ב-95% EtOH.
- יש לשטוף את השקופיות 3 x 10 שניות ב-100% EtOH.
- נגבו את ה-EtOH העודף מהחלק האחורי של המגלשות ואפשרו למגלשות להתייבש באוויר.
- אחסן את השקופיות בטמפרטורה של -80 °C אם אין לבצע LMD באופן מיידי.
4. הדמיית שקופיות
הערה: שלבי הפרוטוקול המתוארים בסעיף זה הם ספציפיים לשקופיות שנסרקו (ראה טבלת החומרים) ולתמונות המתקבלות שנשמרו כקבצי .svs. השתמש בכל סורק ובתוכנות המשויכות אליו היוצרות קבצי תמונה בתבנית שתוכנת ניתוח התמונות (ראה טבלת החומרים) יכולה לפתוח. סוגי קבצים המשתמשים ב- tiffs פירמידאליים הנתמכים כוללים JPG, TIF, MRXS, QPTIFF, רכיב TIFF, SVS, AFI, SCN, LIF, DCM, OME. TIFF, ND2, VSI, NDPI, NDPIS, CZI, BIF, KFB ו- ISYNTAX. זמן משוער: 5 דקות.
- הפעל את הסורק ופתח את תוכנת סורק השקופיות. טען את המגלשה כשהרקמה פונה כלפי מעלה אל שלב ההחלקה הבודדת בסורק. ודאו שהמגלשה יבשה לחלוטין והניחו בעדינות כיסוי מעל הרקמה. אין להשתמש באתנול או בשמן טבילה מתחת לכיסוי.
- צלם את תמונת המיקרוגרף באמצעות הגדרות מכוילות כדי להתאים את קרום PEN במקום את רקע הזכוכית ולהתעלם מכתמי קרום הרקע, בהתאם להוראות היצרן.
- התאם את אזור ההדמיה על-ידי גרירה ושינוי גודל של ההיקף הירוק הפנימי כדי ללכוד את כל אזור הממברנה של PEN, לפי הצורך. הוסיפו ארבע נקודות מיקוד על הרקמה על ידי לחיצה כפולה על תמונת הסקירה של התמונה, ושלוש נקודות מיקוד על הממברנה ליד הפידוקיאלים של הכיול (נקודת מיקוד אחת לכל אחד משלושת הפידוקיאלים של הכיול).
הערה: ניתן למקם את ארבע נקודות המיקוד כמעט בכל מקום בחלק הרקמה, אם כי הצבתן על רקמה מוכתמת באופן כהה מדי ונראית שחורה עלולה לגרום לכשל בסריקה. - תחת תפריט תצוגה , בחר צג וידאו. כוונו את המיקוד באופן ידני באמצעות מחוון המיקוד העדין ו/או המאקרו, לפי הצורך, עבור כל נקודה סביב רקמת ה-LMD. צלם את סריקת התמונה בהגדלה גבוהה (20x). אשר כי כל fiducials כיול גלויים וברורים בתמונה שנשמרה.
5. בחירת תכונות אוטומטית באמצעות תוכנת ניתוח תמונות
- עבור אוספי גידולים שלמים (זמן משוער: 5 דקות; תלוי מקרה):
- פתח את תוכנת ניתוח התמונות (ראה טבלת החומרים). בחר פתח תמונות ומהחלון המוקפץ, בחר את קובץ התמונה .svs שנוצר מסריקת השקופית בסורק AT2.
הערה: קובץ תמונה מייצג של .svs מסופק בקובץ משלים 2. - נווט אל הכרטיסיה ביאורים . בחר את הכלי עט בסרגל הכלים של ביאור וצייר צורה סביב הרקמה.
- בחר את הצורה ולחץ לחיצה ימנית על התמונה. מהתפריט הנפתח מתקדם , בחר מחיצות (אריחים) הגדר את גודל האריחים ואת הרווח בין ל - 500 ו - 40, בהתאמה, ובחר אישור כדי ליצור את האריחים. בחר ומחק את הצורה ההיקפית המשמשת ליצירת האריחים בשלב 5.1.2.
- בחר בתפריט הנפתח 'פעולות שכבה' | ייצוא כדי לשמור את הביאורים המרוצפים באריחים כקובץ .annotation.
הערה: קובץ ביאור מייצג עבור אוסף רקמות גידול שלם מובא בקובץ משלים 3. - צור תיקיה עבור ההפעלה או הפרוייקט ושמור את קובץ ההערה .annotation בתוך תיקיית משנה המסומנת במזהה הייחודי של השקופית.
- נווט אל הכרטיסיה ביאורים . בחר בתפריט הנפתח פעולות שכבה | מחק את כל השכבות כדי להסיר את כל הביאורים מהתמונה. בחר את כלי העט וצייר קו קצר מהקצה הפנימי של ראש החץ עבור כל פידוקיאל כיול. צייר את הקווים מהסימנים בסדר הבא: למעלה משמאל, מימין למעלה, מימין למטה, מימין למטה.
- בחר בתפריט הנפתח 'פעולות שכבה ' | ייצא כדי לשמור את ביאורי השורה כקובץ .annotation. הוסף _calib לשם הקובץ והצב את הקובץ בתיקיית המשנה המכילה את הקואורדינטות עבור הצורות המרוצפות באריחים.
הערה: קובץ _calib.ביאור מייצגים מופיע בקובץ משלים 4. - העתק את הכתובת עבור הפרוייקט הראשי או תיקיית ההפעלה. פתח את קובץ ה- Script ליצירת ייבוא XML, "Malleator" (זמין באמצעות https://github.com/GYNCOE/Mitchell.et.al.2022), באמצעות סביבת הפיתוח המשולבת IDLE והדבק את כתובת תיקיית הפרוייקט בין המרכאות בתחתית קובץ ה- Script.
- בחר בתפריט הנפתח הפעל | הפעל מודול כדי לבצע את הסקריפט.
הערה: קובץ הייבוא .xml LMD ייווצר בתוך תיקיית המשנה שנוצרה עבור התמונה/השקופית. קובץ .xml מייצגים מופיע בקובץ משלים 5.
- פתח את תוכנת ניתוח התמונות (ראה טבלת החומרים). בחר פתח תמונות ומהחלון המוקפץ, בחר את קובץ התמונה .svs שנוצר מסריקת השקופית בסורק AT2.
- עבור אוספים מועשרים ב-LMD בלבד (זמן משוער: 15 דקות; תלויי רישיות):
- פתח את תוכנת ניתוח התמונות (ראה טבלת החומרים). בחר פתח תמונות ומהחלון המוקפץ בחר את קובץ התמונה .svs שנוצר מסריקת השקופית.
- נווט אל הכרטיסיה ביאורים . בחר והשתמש בכלי ביאור מלבן כדי לצייר תיבה סביב הרקמה.
- בחר את ביאור התיבה ולחץ באמצעות לחצן העכבר הימני על התמונה. בחר בתפריט הנפתח מתקדם | אפשרות מחיצות (אריחים). הגדר את גודל האריחים ואת הרווח בין ל- 500 ו- 40, בהתאמה, ובחר אישור כדי ליצור את האריחים. בחר ומחק את ביאור התיבה ההיקפית המשמש ליצירת האריחים בשלב 5.2.2.
- בחר בתפריט הנפתח 'פעולות שכבה ' | ייצוא כדי לשמור את הביאורים המרוצפים באריחים כקובץ .annotation.
- מקם עותק שמור של אלגוריתם Python "Dapọ" (זמין באמצעות https://github.com/GYNCOE/Mitchell.et.al.2022), שפותח כדי למזג את שכבות הביאור המסווגות על ידי AI, לתוך אותה תיקייה כמו קובץ הביאורים המרוצפת.
- העתק את שם קובץ הביאור המרוצפת. פתח את תוכנית Python באמצעות סביבת הפיתוח המשולבת IDLE והדבק את שם קובץ ההערה המרוצפת בין המרכאות בתחתית התוכנית.
- בחר בתפריט הנפתח הפעל | הפעל מודול. המתן עד שייווצר קובץ חדש שיכלול את כל הביאורים המרוצפים באריחים ממוזגים בשכבה אחת.
- פתח את תוכנת ניתוח התמונות ונווט אל הכרטיסיה ביאורים . בחר בתפריט הנפתח 'פעולות שכבה ' | מחק את כל השכבות כדי להסיר את כל הביאורים מהתמונה.
- בחר בתפריט הנפתח 'פעולות שכבה ' | ייבוא קובץ ביאור מקומי. בחלון המוקפץ, בחר את קובץ ה- .annotation הממוזג שנוצר על-ידי קובץ ה- Script. ודא שכל האריחים המיובאים נמצאים תחת אותה שכבת ביאור.
- נווט אל הכרטיסיה מסווג ופעל לפי הוראות היצרן כדי ליצור צורות עבור ה- ROIs. לפני הפעלת המסווג, בחר את שכבות הביאור הרצויות (כלומר, שכבת הגידול או השכבה סטרומה) על-ידי סימון תיבת ROI או תיבות בכרטיסיה ביאורים , תחת אפשרויות המסווג המתקדמות. השתמש באפשרות 'שכבת ביאור' מתפריט 'פעולות מסווגות' כדי להפעיל את המסווג.
- לאחר השלמת ניתוח המסווג, נווט אל הכרטיסיה ביאורים ובחר את שכבת הביאור שנוצרה מהניתוח. בחר בתפריט הנפתח 'פעולות שכבה ' | מחק את כל השכבות אך נוכחיות כדי להסיר את כל שכבות ההערה האחרות מהתמונה.
- בחר בתפריט הנפתח 'פעולות שכבה ' | ייצוא לשמירת הביאורים כקובץ .annotation. צור תיקיה עבור ההפעלה או הפרוייקט ושמור את קובץ .annotation בתוך תיקיית משנה המסומנת במזהה הייחודי של השקופית.
הערה: קובץ .annotation מייצג עבור אוסף רקמות מועשרות ב- LMD מסווג מסופק בקובץ משלים 6. - נווט אל הכרטיסיה ביאורים, בחר | הנפתח פעולות שכבה | מחק את כל השכבות כדי להסיר את כל הביאורים מהתמונה. בחר את כלי העט וצייר קו קצר מכל פידוקיאל כיול. ציירו קווים מהסימנים בסדר הבא: למעלה משמאל, מימין למעלה, מימין למטה.
- בחר בתפריט הנפתח 'פעולות שכבה ' | ייצא כדי לשמור את ביאורי השורה כקובץ .annotation. הוסף _calib לשם הקובץ והצב את הקובץ בתיקיית המשנה המכילה את הקואורדינטות עבור הצורות המרוצפות באריחים.
- העתק את הכתובת עבור הפרוייקט הראשי או תיקיית ההפעלה. פתח את סקריפט יצירת ייבוא XML, "Malleator" (זמין באמצעות https://github.com/GYNCOE/Mitchell.et.al.2022), באמצעות סביבת הפיתוח המשולבת IDLE, ולאחר מכן הדבק את כתובת תיקיית הפרוייקט בין המרכאות בתחתית קובץ ה- Script.
- בחר בתפריט הנפתח הפעל | הפעל מודול כדי לבצע את הסקריפט.
הערה: קובץ הייבוא .xml LMD ייווצר בתוך תיקיית המשנה שנוצרה עבור התמונה/השקופית.
6. מיקרו-דיסקציה של לייזר
הערה: שלבי הפרוטוקול המתוארים בסעיף זה הם ספציפיים לשימוש עם מיקרוסקופ לייזר הפוך והתוכנה המשויכת (ראה טבלת החומרים). זמן משוער: 2 שעות; תלוי מקרה.
- טען את שקופית הממברנה המסומנת (המכילה פידוקיאלים כיולים) כאשר הרקמה פונה כלפי מטה וצד התווית קרוב יותר למפעיל למחזיק המגלשה בשלב המיקרוסקופ של הלייזר.
- בחר ייבוא צורות מהתפריט הנפתח קובץ . בחר את קובץ הייבוא .xml LMD שנוצר עבור השקופית. בחר לא בחלון המוקפץ כדי להימנע מטעינת נקודות ייחוס מהקובץ ולא בחלון המוקפץ השני כדי להימנע משימוש בנקודות ייחוס שאוחסנו בעבר לצורך כיול.
- עקוב אחר ההנחיות מיישום LMD ויישר את צלב הכיול לכל אחד משלושת הפידוקיאלים של הכיול בשקופית. חפשו פידוקיאלים כיולים המופיעים בפינה השמאלית העליונה, הימנית העליונה והימנית התחתונה של תמונת השקופית בתוכנת ניתוח התמונה, אשר יתאימו לנקודות ייחוס בפינות הימניות הקדמיות, הימניות האחוריות והשמאליות האחוריות, בהתאמה, של שקופית ה-LMD ההפוכה על במת המיקרוסקופ. עברו בין שימוש בעדשת המטרה 5x לאיתור לבין המטרה של 63x כדי ליישר כל פידוקציה של כיול. בחר לא בחלון המוקפץ כדי להימנע משמירה על נקודות ההפניה בקובץ ובאשר בחלון המוקפץ השני כדי לאשר שהשקופית נוספה.
- הזז את העדשה האובייקטיבית של 5x למקומה ובחר כן בחלון הקופץ כדי להשתמש בהגדלה בפועל. לאחר הופעת הצורות המיובאות, מקד את המצלמה ברקמה.
- סמן ובחר את כל הצורות בחלון רשימת הצורות , גרור אותן למקומם באמצעות ביאור אחד או שניים בתוך שדה הראייה כהפניות, ויישר את ציר z האנכי לחיתוך עם הלייזר.
- סקור את הצורות המיובאות והקצה אותן למיקום הצינור המתאים לאיסוף. לחץ על התחל לחתוך כדי להפעיל את הלייזר.
הערה: צורות מיובאות בקובץ .xml יוקצו באופן אוטומטי למיקום "ללא מכסה" בחלון רשימת הצורות . כדי לקצור רקמות, יש להקצות מחדש את הצורות המיובאות למצב המכיל צינור טעון.
7. עיכול חלבונים על ידי טכנולוגיית מחזור לחץ (PCT)
הערה: זמן משוער: 4 שעות (3 שעות ללא זמן ייבוש צנטריפוגות ואקום).
- הניחו צינורות בגודל 0.5 מ"ל המכילים את ה-PCT MicroTubes המכוסים המכילים רקמה שנקטפה ב-LMD ב-20 μL של 100 mM TEAB/10% אצטוניטריל לתוך תרמוציפלר וחממו ב-99 מעלות צלזיוס למשך 30 דקות, ואז התקררו ל-50 מעלות צלזיוס למשך 10 דקות.
- סובב את הצינורות במשך 30 שניות ב-4,000 × גרם ולאחר מכן הסר את המיקרו-טיובים מצינורות ה-0.5 מ"ל. באמצעות הכלי MicroCap, הסר והשלך את ה- MicroCaps מה- MICROTubes של PCT. הוסף טריפסין (ראה טבלת החומרים) ביחס של 1 מיקרוגרם לכל רקמת 30 מ"מ2 והכנס MicroPestle לתוך MicroTube באמצעות הכלי MicroCap.
- העבר את המיקרו-טיובים למחסנית ברוציקלר והרכיב את המחסנית השלמה. מניחים את המחסנית בתא הלחץ של הברוציקלר ומהדקים את המכסה. Barocycle ב 45,000 psi עבור 50 s ולחץ אטמוספרי עבור 10 s ב 50 °C ב 50 °C עבור 60 מחזורים.
- לאחר השלמת הברוציקלינג, העבירו את המיקרו-צינוריות לצינור מיקרו-צנטריפוגה של 0.5 מ"ל וצנטריפוגה למשך 2 דקות ב-4,000 × גרם.
- הסר את ה- MicroTube מצינור microcentrifuge בגודל 0.5 מ"ל באמצעות כלי המכסה. הסירו בזהירות את המזיק באמצעות כלי הפקק ושטפו את החצי התחתון של המזיק ב-20 μL של מים ברמת ספקטרומטריית מסה של כרומטוגרפיה נוזלית (LC-MS) ואספו את הכביסה לצינור מיקרוצנטריפוג' נקי של 0.5 מ"ל.
- הקש בעדינות על ה- MicroTube בחלק העליון של הספסל כדי להעביר את הנוזל לתחתית ולהעביר את כל הפתרון מה- MicroTube לצינור microcentrifuge של 0.5 מ"ל.
- הוסיפו 20 μL של מים ברמת LC-MS ל-MicroTube והקשו עליהם בעדינות על החלק העליון של הספסל. העבירו את תמיסת הכביסה לצינור ה-0.5 מ"ל וחזרו על שלב הכביסה הזה פעם נוספת.
- צנטריפוגת ואקום לייבוש הדגימות ל~2 μL והוספת 100 μL של 100 mM TEAB, pH 8.0.
- קבע את ריכוז הפפטידים באמצעות בדיקה קולורימטרית (בדיקת חומצה ביסינצ'ונינית (BCA); ראו טבלת החומרים) בהתאם לפרוטוקול היצרן.
8. תיוג תג מסה טנדם (TMT) וניקוי EasyPep
הערה: זמן משוער: 7 שעות 20 דקות (2 שעות 20 דקות ללא זמן ייבוש צנטריפוגת ואקום).
- הביאו את ריאגנטים איזובריים לסימון TMT לטמפרטורת הסביבה לפני הפתיחה. הוסיפו 500 μL של 100% אצטוניטריל לכל בקבוקון TMT (5 מ"ג). דגירה במשך 10 דקות עם מערבולות מזדמנות.
- ממיסים 5 מיקרוגרם של דגימת הפפטיד ב-100 μL של 100 mM TEAB, pH 8.0, ומוסיפים 10 μL של מגיב TMT נתון. לבנות ולכלול מאגרי ייחוס המייצגים כל דגימה בודדת בניסוי בכל קבוצת דגימות מולטיפלקס TMT כדי להקל על כימות דגימות על פני מספר מולטיפלקסים TMT9. דגירה של תגובות למשך שעה אחת בטמפרטורת הסביבה עם רעידות/הקשות מזדמנות.
- מרווים את תגובת הסימון של TMT על ידי הוספת 10 μL של 5% הידרוקסילאמין ודגירה למשך 30 דקות בטמפרטורת הסביבה עם הקשה מזדמנת. לאחר המרווה, שלבו את הדגימות המסומנות ב-TMT בצינור אחד ויבשו לכ-200 מיקרול'.
- הוסף 1,800 μL של 0.1% חומצה פורמית. בדוק את ה- pH עם נייר pH: אם pH ~ 3, הוסף 1 מ"ל של 0.1% חומצה פורמית; אם pH >3, הוסף 10-20 μL של 5% חומצה פורמית עד pH ~ 3. הוסיפו 0.1% חומצה פורמית כדי להביא לנפח סופי של 3 מ"ל.
- הסר את הכרטיסייה בתחתית עמודת הניקוי של הפפטיד, הסר את המכסה והנח אותה בצינור חרוטי של 15 מ"ל. העבר את הדגימה המסומנת ב- TMT לעמודה והמשך בניקוי בהתאם לפרוטוקול היצרן.
- צנטריפוגת ואקום לייבוש הפפטידים הבוקעים לכ-20 μL, העברה לבקבוקון LC באמצעות אמוניום ביקרבונט של 25 mM לנפח סופי של 80 μL, והמשך לפיצול לא מקוון.
9. פיצול דגימת מולטיפלקס TMT ואיגום
הערה: זמן משוער: 3 שעות 30 דקות (1 שעה 30 דקות ללא זמן ייבוש צנטריפוגת ואקום).
- שברו את המולטיפלקסים הפפטידיים המסומנים ב-TMT על ידי כרומטוגרפיה בסיסית של פאזה הפוכה ל-96 שברים על-ידי פיתוח שיפוע ליניארי הולך וגדל (0.69% דקה-1) של פאזה B ניידת (אצטוניטריל) לפאזה ניידת A (10 mM NH4HCO3, pH 8.0).
- צור 36 שברים משורשרים על ידי איגום בארות לדוגמה. צנטריפוגת ואקום לשברים יבשים ל~2 μL והחזרה ב-25 mM אמוניום ביקרבונט (ריכוז סופי של 1.5 מיקרוגרם/10 μL), צנטריפוגה ב-15,000 × גרם למשך 10-15 דקות, והעברה לבקבוקוני LC לצורך ניתוח טרשת נפוצה.
10. ספקטרומטריית מסות טנדם כרומטוגרפית נוזלית (LC-MS/MS)
הערה: זמן משוער: שיטת מכשיר ותלוי תכנון ניסיוני.
- כיול ספקטרומטר המסה בהתאם להוראות/פרוטוקולים של היצרן.
- הכינו פאזות ותקנים ניידים טריים ובצעו הכנות מתאימות ל-LC לפני הריצה (כולל, בין היתר, ממסים לטיהור, אוויר שוטף וסקריפטים לבדיקת דליפות עבור המכשיר המוזכר [ראו טבלת החומרים]). שיווי משקל בין עמודות טרום ואנליזה ולולאת דגימה לפני תחילת הניתוחים.
- לפני ובין ניתוחי מולטיפלקס TMT טוריים, ודא שמערכת LC-MS עומדת במדדי ביצועים שנבדקו בעבר באמצעות עיכולי פפטידים עם תווית TMT (QA/QC) של אבטחת איכות/בקרת איכות (QA/QC) ו-(לדוגמה, MSPE (ראה טבלת החומרים), HeLa).
- טען בקבוקונים של autosampler במיקומים המתאימים ב- autosampler LC. נתח שברים בודדים בשיטת מעבר צבע/MS מתאימה. שוזרים ריצת "שטיפה" עם תקן פפטיד (לדוגמה, כיול זמן שימור פפטידים [PRTC]) בערך פעם ביום כדי להעריך את הביצועים הכרומטוגרפיים והספקטרליים של המסה. לאחר הניתוח של כל סדרת שברים לדוגמה של מולטיפלקס TMT, הפעל תקני אמת מידה של QA/QC TMT כדי להעריך את ביצועי המערכת.
- הפעל שגרות הערכה של ספקטרומטר מסה לאחר תקני השוואת ביצועים של QA/QC TMT כדי להעריך את הביצועים לאחר הדגימה ולאחר מכן לכייל את המערכת כמו בשלב 10.1 עבור ערכת הדגימות הבאה.
11. ניתוח נתונים ביואינפורמטיים
הערה: זמן משוער: תלוי תכנון ניסיוני.
- העבר את כל הנתונים לדוגמה (לדוגמה, קבצי .raw) לאחסון רשת/כונן מחשב מתאים.
- חפש את כל השברים יחד באמצעות יישום ניתוח הנתונים הרצוי (לדוגמה, Proteome Discover, Mascot) תוך שימוש בפרמטרים מתאימים9 כנגד מסד נתונים של ייחוס חלבונים ספציפיים למין כדי ליצור התאמות ספקטרליות פפטידיות (PSMs) ולחלץ עוצמות אות יונים של כתב TMT. סנן PSM המבוססים על מדדי בקרת איכות מתאימים, ושפע של יחס יונים של TMT המנורמל, החציונישל 2 לוחות השידורים, לשפע של רמת החלבון העולמית, כפי שתואר קודם לכן 3,9.
- השווה שינויים בחלבון בתנאי העניין באמצעות תוכנת ניתוח דיפרנציאלית רצויה.
תוצאות
מקטעים דקים של רקמות קפואות טריות משני חולי HGSOC ושני מטופלי OCCC נותחו באמצעות זיהוי משולב זה של החזר על רקמות מונחה בינה מלאכותית, סגמנטציה, LMD וזרימת עבודה של ניתוח פרוטאומי כמותי (איור 1). מקטעי רקמות מוכתמים ב-H&E מייצגים עבור כל גידול נבדקו על ידי פתולוג מוסמך; תאי הגידול נעו בין 70% ל-99%. הרקמות נחתכו דק על גבי שקופיות ממברנה PEN (קובץ משלים 2) וחתכו מראש עם פידוקיאלים של כיול (קובץ משלים 1), מה שאפשר שילוב של נתוני כיוון מיקום מתוך ביאורים שנוצרו בתוכנת ניתוח התמונה (ראו טבלת החומרים) עם אוריינטציה של קואורדינטות קרטזיות בתוכנת LMD. לאחר צביעת H&E, צולמו תמונות ברזולוציה גבוהה (פי 20) של שקופיות PEN המכילות את הרקמה בתוספת כיולים.
אוכלוסיות גידולים ותאים סטרומליים במיקרוגרפים חולקו באמצעות תוכנת ניתוח תמונות (ראו טבלת החומרים) לקציר סלקטיבי על ידי LMD, יחד עם יבולים המייצגים את כל החלק הדק של הרקמה (למשל, רקמת גידול שלמה) (איור 1). ביאורים לא מפלים לאיסוף רקמות גידול שלמות נוצרו על ידי חלוקת כל מקטע הרקמה באריחים של 500 מיקרומטר2, והותירו מרווח של 40 מיקרומטר בין אריחים כדי לשמור על שלמות ממברנת PEN ולמנוע מהממברנה להסתלסל במהלך LMD. בשקופיות להעשרת LMD שנפתרה על-ידי היסטולוגיה, מסווג הבינה המלאכותית בתוכנת ניתוח התמונות (ראו טבלת החומרים) אומן להבחין בין תאים סרטניים וסטרומליים, יחד עם רקע שקופית הזכוכית הריקה. גידולים מייצגים, סטרומה ואזורי זכוכית ריקה הודגשו באופן ידני, וכלי המסווג שימש לסגמנט ה-ROIs האלה לאורך כל מקטע הרקמה. השכבות המפולגות המייצגות רקמה שלמה, אפיתל גידול וסטרומה נשמרו בנפרד כקבצי ביאור בודדים (קובץ משלים 3 וקובץ משלים 6). בעותק נפרד של קובץ התמונה (ללא ביאורי ROI המחולקים), שורה קצרה מהקצה המרכזי ביותר של כל אחד משלושת הכיולים הפידוקיאליים הוערה ונשמרה כקובץ .annotation תוך שימוש באותו שם קובץ כמו כל אחד מקבצי שכבת הביאור של LMD, אך צורפה עם הסיומת "_calib" (קובץ משלים 4). קווים אלה שימשו לרישום משותף של מיקום כיול הממברנות PEN עם נתוני רשימת צורות ההערה שצוירו בתוכנת ניתוח התמונה.
המחקר הנוכחי מספק שני אלגוריתמים, "Malleator" ו-"Dapọ" ב-Python כדי לתמוך בזרימת עבודה זו של LMD מונחה בינה מלאכותית, הזמינים https://github.com/GYNCOE/Mitchell.et.al.2022. אלגוריתם Malleator מחלץ את הקואורדינטות הקרטזיות הספציפיות עבור כל הביאורים הבודדים (ROI של רקמות וכיולים) מקבצי ה-.annotation הזוגיים וממזג אותם לקובץ ייבוא יחיד של שפת סימון מורחבת (XML) (קובץ משלים 5). באופן ספציפי, אלגוריתם Malleator משתמש בשם הספרייה מתיקיית אב כקלט כדי לחפש בכל תיקיות ספריות המשנה ומייצר קבצי .xml עבור כל תיקיות משנה שאין להן עדיין קובץ ממוזג .xml. אלגוריתם Malleator ממזג את כל שכבות הביאור בתוכנת ניתוח התמונות (ראה טבלת החומרים) לשכבה אחת וממיר את נתוני רשימת הצורות שנוצרו על-ידי בינה מלאכותית, שנשמרים כסוג קובץ .annotation קנייני, לתבנית .xml התואמת לתוכנת LMD. לאחר מיזוג קבצי הביאור והכיול, קובץ .xml שנוצר על ידי האלגוריתם נשמר ומיובא לתוכנת LMD. התאמות קלות נחוצות כדי לכוונן באופן ידני את יישור הביאורים, המשמש גם לרישום המיקום האנכי (מישור z) של שלב ההחלקה במיקרוסקופ הלייזר. אלגוריתם Dapọ משמש במיוחד עבור אוספים מועשרים ב-LMD. אריחים מחולקים למחיצות מוקצים באופן אוטומטי לשכבות ביאור בודדות על-ידי תוכנת ניתוח התמונות. אלגוריתם Dapọ ממזג את כל האריחים המחולקים לשכבת ביאור אחת לפני השימוש בכלי המסווג, ובכך מצמצם את זמן הריצה של ניתוח המסווג עבור אוספים מועשרים ב-LMD.
כל דגימות הגידול והרקמות מועשרות ב-LMD עוכלו, סומנו בריאגנטים של TMT, הוכפלו, חולקו במצב לא מקוון ונותחו באמצעות פרוטאומיקה כמותית מבוססת טרשת נפוצה כפי שתואר קודם לכן9. תפוקת הפפטידים הממוצעת (43-60 מיקרוגרם) וההתאוששות (0.46-0.59 מיקרוגרם/מ"מ2) עבור דגימות שנקטפו באמצעות זרימת עבודה מונחית בינה מלאכותית זו היו דומות לדוחות קודמים 9,10. בסך הכל 5,971 חלבונים כומתו במשותף בכל הדגימות (טבלה משלימה S1). אשכולות היררכיים לא מפוקחים באמצעות 100 החלבונים המשתנים ביותר הביאו להפרדה של היסטוטיפים HGSOC ו-OCCC מדגימות הגידולים העשירים והשלם של LMD (איור 2A), בדומה לזה שתואר קודם לכן11. לעומת זאת, דגימות הסטרומה מועשרות ב-LMD הן מ-HGSOC והן מ-OCCC התקבצו יחד ובאופן בלתי תלוי בגידול מועשר ב-LMD ומדגימות גידול שלמות. מתוך 5,971 חלבונים מכמתים, 215 שונו באופן משמעותי (LIMMA adj. p < 0.05) בין אוספי גידולים שלמים מדגימות HGSOC ו-OCCC (טבלה משלימה S2). חלבונים שהשתנו הושוו לאלה שזוהו כמבדילים בין HGSOC לרקמת הגידול של OCCC על ידי Hughes et al.11. מתוך 76 חלבוני החתימה שכומתו על ידי Hughes et al., 57 כומתו במשותף במערך נתונים זה והיו מתואמים מאוד (Spearman Rho = 0.644, p < 0.001) (איור 2B).
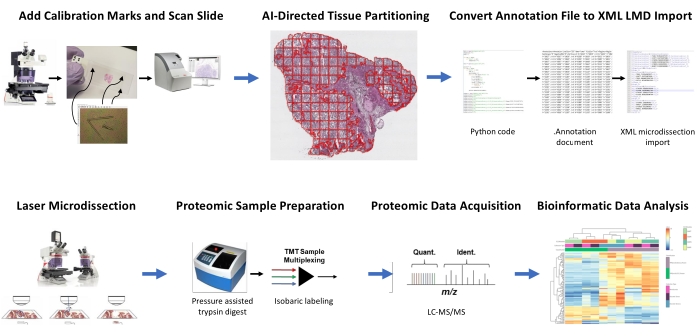
איור 1: סיכום של זרימת העבודה המשולבת עבור אזור רקמות אוטומטי של בחירת עניין עבור מיקרו-דיסקציה של לייזר עבור פרוטאומיקה כמותית במורד הזרם. פידוקיאלים של כיול נחתכים על שקופיות ממברנת PEN כדי לרשום במשותף נתוני כיוון מיקום מקטעים שמקורם בבינה מלאכותית של ROI של רקמות בתוכנת ניתוח התמונה, HALO, עם מיקום אופקי במיקרוסקופ LMD. אלגוריתם Malleator משמש למיזוג נתוני הסגמנטציה המבוארים בכל שכבות הביאור עבור שקופית עם קובץ הייחוס _calib, ולהמרתו לקובץ .xml התואם לתוכנת LMD. רקמה שנקטפה על ידי LMD לניתוח פרוטאומי מתעכלת ומנותחת על ידי פרוטאומיקה כמותית בתפוקה גבוהה כפי שתואר קודם לכן9. קיצורים: LMD = מיקרו-דיסקציה של לייזר; ROI = אזור עניין; TMT = תג מסה טנדם; כימות = כימות; מזהה. = זיהוי; LC-MS/MS = כרומטוגרפיה נוזלית-ספקטרומטריית מסה טנדם. אנא לחץ כאן כדי להציג גרסה גדולה יותר של נתון זה.
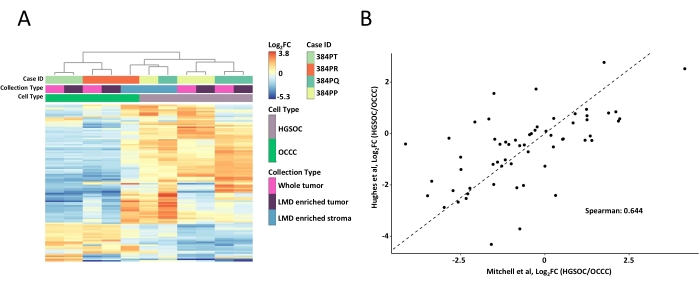
איור 2: ניתוח החלבונים בדגימות גידול מועשרות ב-LMD ובדגימות גידול שלמות מועשרות ב-LMD. (A) ניתוח אשכולות היררכי לא מפוקח של 100 החלבונים הנפוצים ביותר בדגימות HGSOC ו-OCCC LMD מועשרות ודגימות גידול שלמות. (B) קורלציה של שפע חלבונים שללוג 2 המשתנה בקיפול בין HGSOC לבין קציר גידולים שלמים של OCCC במחקר הנוכחי (Mitchell et al., x-axis) ובמחקר דומה של יוז ואחרים (ציר y)11. קיצורים: LMD = מיקרו-דיסקציה של לייזר; HGSOC = סרטן שחלות סרוס בדרגה גבוהה; OCCC = קרצינומה של תאים שקופים בשחלות; log2FC = לוג2-טרנספורמציה שפע פרוטאומי. אנא לחץ כאן כדי להציג גרסה גדולה יותר של נתון זה.
טבלה משלימה S1: שפע של 5,971 חלבונים כומתו במשותף בכל דגימות הגידול המועשרות והמלאות של LMD מדגימות רקמות HGSOC ו-OCCC. קיצורים: LMD = מיקרו-דיסקציה של לייזר; HGSOC = סרטן שחלות סרוס בדרגה גבוהה; OCCC = קרצינומה של תאים שקופים בשחלות. אנא לחץ כאן כדי להוריד טבלה זו.
טבלה משלימה S2: חלבונים המבוטאים באופן דיפרנציאלי (215) באוספי גידולים שלמים מ-HGSOC לעומת OCCC (LIMMA adj. p < 0.05). קיצורים: HGSOC = סרטן שחלות סרוסי בדרגה גבוהה; OCCC = קרצינומה של תאים שקופים בשחלות. אנא לחץ כאן כדי להוריד טבלה זו.
קובץ משלים 1: קובץ נתוני רשימת צורות מייצגות ( .sld) המכיל פידוקיאלים סטנדרטיים של כיול לארבעה מיקומי שקופיות. ניתן לייבא את הקובץ לתוכנת LMD. אנא לחץ כאן כדי להוריד קובץ זה.
קובץ משלים 2: קובץ תמונה מייצג של .svs של מקטע רקמה מוכתם H&E ברזולוציה גבוהה (20x). ניתן לפתוח ולהציג את הקובץ באמצעות תוכנת ניתוח תמונות או תוכנת LMD. קיצור: H&E = המטוקסילין ואוזין; LMD = מיקרו-דיסקציה של לייזר. אנא לחץ כאן כדי להוריד קובץ זה.
קובץ משלים 3: קובץ ביאור מייצג של מקטעי גידול שלמים מחולקים. ניתן לייבא את הקובץ לתוכנת ניתוח תמונות. אנא לחץ כאן כדי להוריד קובץ זה.
קובץ משלים 4: קובץ _calib.ביאור מייצג של מקטעי כיול פידוקיאליים. מידע קואורדינטות מייצג מיקום מזרחי של קווי הכיול הקצרים המצוירים מכל פידוקיאלי של ראש חץ. ניתן לייבא את הקובץ לתוכנת ניתוח תמונות. אנא לחץ כאן כדי להוריד קובץ זה.
קובץ משלים 5: קובץ שפת סימון ניתנת להרחבה מייצגת (.xml) שנוצר על ידי אלגוריתם Malleator. ניתן לייבא את הקובץ לתוכנת המיקרו-דיסקציה של הלייזר. אנא לחץ כאן כדי להוריד קובץ זה.
קובץ משלים 6: קובץ ביאור מייצג של מקטעים המסווגים ב- AI במחולקים עבור אוספים מועשרים ב- LMD. ניתן לייבא את הקובץ לתוכנת ניתוח תמונות. קיצורים: AI = בינה מלאכותית; LMD = מיקרו-דיסקציה של לייזר. אנא לחץ כאן כדי להוריד קובץ זה.
Discussion
אמנם היו תקדימים מחקריים רבים שמטרתם לפתח ו/או לשפר זרימות עבודה להעשרת תת-אוכלוסיות של תאי מטרה מ-FFPE ו/או רקמות קפואות טריות ומתודולוגיות לשמירה על איכות הדגימה במהלך עיבוד 9,12,13,14,15, אך קיים צורך משמעותי בפיתוח אסטרטגיות אוטומטיות להכנת דגימות רקמות קליניות לניתוחים מולקולריים כדי להפחית את השונות ו להגדיל את יכולת השכפול. זרימת עבודה זו מתארת פרוטוקול מתוקנן וחצוי למחצה המשלב כלי תוכנה קיימים לניתוח תמונות (ראו טבלת החומרים) לצורך קצירה פתורה היסטולוגית של אוכלוסיות תאים בדידים על ידי LMD מדגימות רקמות קליניות.
העשרת LMD בפתרון מרחבי של ROIs הלוכדים אוכלוסיות תאים בדידים מייצגת שלב עיבוד רקמות מהדור הבא לפני ניתוחים מולטיומיים כדי לשפר את האפיון והזיהוי המולקולריים ולהקל על גילוי סמנים ביולוגיים סלקטיביים של תאים. פרוטוקול זה משפר את המתודולוגיות הקיימות על ידי הפחתת החשיפה הארוכה לעתים קרובות של מקטעי רקמות לסביבה הסביבתית הקשורה לסגמנטציה ידנית של ROI על ידי היסטולוג (שיכולה להימשך >1-2 שעות לפני איסוף ה- LMD). במקום זאת, זרימת עבודה זו מאפשרת ל-ROI להיות מזוהה מראש על-ידי סיווג ופילוח מונחי בינה מלאכותית. הגבלת זמן השהייה של רקמות תפחית את השונות המופרכת בהערכות של מטרות מולקולריות מבוישות מאוד, כגון פוספופפטידים ו-mRNA, או עבור טכניקות אנליטיות מבוססות נוגדנים המסתמכות על כך שחלבון מטרה נמצא בקונפורמציה הטבעית שלו לזיהוי.
חיתוך פידוקיאלים כיולים מסודרים על שקופית הממברנה PEN הנראים בבירור בתמונת השקופית הסרוקה הוא אחד המרכיבים המרכזיים המאפשרים שילוב של תוכנת ניתוח התמונות (ראה טבלת החומרים) עם זרימת העבודה של LMD. ההקפדה על כך שלכיולים תהיה נקודה מדויקת ("נקייה") בתחתית צורת ה-"V" מאפשרת בחירה של נקודה מדויקת בתוכנת ניתוח התמונה שממנה יש לצייר את קווי הכיול, כמתואר בשלבים 5.1.6 ו-5.2.13. יישור של נקודות אלה במהלך הייבוא לתוכנת LMD הוא קריטי לכיסוי נכון של הביאורים (המתאפשרים באמצעות יצירת קובץ .xml תואם באמצעות האלגוריתמים "Malleator" ו/או "Dapọ") לתוך החזר ההשקעה הרלוונטי של הרקמה בשקופית LMD הפיזית. יש צורך להדגיש את כל הצורות ובאופן קולקטיבי "לגרור ולשחרר" למקומם גם כאשר היישור מדויק עם הייבוא לתוכנת LMD כדי לרשום את המיקום האנכי (z-plane) של שלב השקופית במיקרוסקופ הלייזר. התאמות קלות למיקום הביאורים על פני החזר ההשקעה על הרקמה יכולות גם הן להתבצע במהלך שלב זה, במידת הצורך.
מגבלה של הגרסה הנוכחית של אלגוריתם Malleator היא שהיא אינה תואמת את כלי צורת הביאור המוגדרים מראש המסופקים על ידי תוכנת ניתוח התמונה (ראה טבלת החומרים), אם כי עדכונים /גרסאות עתידיות של האלגוריתם ישאפו לשפר תאימות זו. קובץ .annotation עבור צורות שצוירו באמצעות כלים אלה מכיל רק שתי קבוצות של קואורדינטות x ו- y מזווגות עבור כל ביאור, ללא הכיוון המרחבי המלא סביב נקודות אלה. השימוש הנוכחי בכלים אלה גורם לכך שהביאורים מומרים לקווים ישרים המוגדרים על-ידי שתי נקודות בלבד במהלך תהליך הייבוא. הגדרה ידנית של מקטעי ROI של רקמות נדרשת להמרה מוצלחת לתבנית XML וייבוא LMD. ניתן לבצע זאת על ידי הגדרה ידנית של כל ROI עם ביאורים מצולעים נפרדים ביד חופשית הספציפיים לאזור היעד או על ידי החלת ביאור עגול או מלבני משוער על פני כל מקטעי ההחזר על ההשקעה ברקמות, אם תרצה בכך, ויהיה תואם לזרימת עבודה זו.
בעוד שזרימת העבודה שהוצגה כאן הודגמה לצורך ניתוח פרוטאומי של דגימות רקמות סרטן אנושיות טריות קפואות, ניתן להשתמש באופן שווה ערך בזרימת עבודה זו של LMD המונעת על ידי בינה מלאכותית עם רקמות FFPE, סוגי רקמות שאינן סרטניות ואלה ממקורות שאינם אנושיים. הוא יכול גם לתמוך בתהליכי עבודה אחרים של יצירת פרופיל מולקולרי במורד הזרם, כולל ניתוחים תעתיקיים, גנומיים או פוספופרוטאומיים. זרימת עבודה זו יכולה גם למנף שימושים אחרים בתוכנת ניתוח התמונה (ראה טבלת החומרים), כולל עם יכולות הקשורות לספירת תאים או למודולים אנליטיים אחרים, כולל מודול "Multiplex IHC" או תוסף "רקמות מיקרו-מערך (TMA)". יישומים עתידיים של זרימת עבודה זו עשויים גם להפיק תועלת מהגדרתם מראש של מספר התאים לכל מקטע ROI, ובכך להבטיח כניסות תאיות שוות ערך על פני אוספים מרובים, או על ידי שימוש בשיטות חלופיות להגדרת ROIs תאיים בעלי עניין, כגון על ידי אימונוהיסטוכימיה או סוציולוגיה של התא.
Disclosures
T.P.C. היא חברת ThermoFisher Scientific, Inc SAB ומקבלת מימון מחקר מ-AbbVie.
Acknowledgements
המימון לפרויקט זה ניתן בחלקו על ידי תוכנית הבריאות הביטחונית (HU0001-16-2-0006 ו- HU0001-16-2-00014) לאוניברסיטת השירותים במדים למרכז המצוינות לסרטן גינקולוגי. לנותני החסות לא היה כל תפקיד בתכנון, בביצוע, בפרשנות או בכתיבה של המחקר. כתב ויתור: הדעות המובעות כאן הן של המחברים ואינן משקפות את המדיניות הרשמית של מחלקת הצבא/הצי/חיל האוויר, משרד ההגנה או ממשלת ארצות הברית.
Materials
| Name | Company | Catalog Number | Comments |
| 1260 Infinity II System | Agilent Technologies Inc | Offline LC system | |
| 96 MicroCaps (150uL) in bulk | Pressure Biosciences Inc | MC150-96 | |
| 96 MicroPestles in bulk | Pressure Biosciences Inc | MP-96 | |
| 96 MicroTubes in bulk (no caps) | Pressure Biosciences Inc | MT-96 | |
| 9mm MS Certified Clear Screw Thread Kits | Fisher Scientific | 03-060-058 | Sample vial for offline LC frationation and mass spectrometry |
| Acetonitrile, Optima LC/MS Grade | Fisher Chemical | A995-4 | Mobile phase solvent |
| Aperio AT2 | Leica Microsystems | 23AT2100 | Slide scanner |
| Axygen PCR Tubes with 0.5 mL Flat Cap | Fisher Scientific | 14-222-292 | Sample tubes; size fits PCT tubes and thermocycler |
| Barocycler 2320EXT | Pressure Biosciences Inc | 2320-EXT | Barocycler |
| BCA Protein Assay Kit | Fisher Scientific | P123225 | |
| cOmplete, Mini, EDTA-free Protease Inhibitor Cocktail | Roche | 11836170001 | |
| Easy-nLC 1200 | Thermo Fisher Scientific | Liquid Chromatography | |
| EasyPep Maxi Sample Prep Kit | Thermo Fisher Scientific | NCI5734 | Post-label sample clean up column |
| EASY-SPRAY C18 2UM 50CM X 75 | Fisher Scientific | ES903 | Analytical column |
| Eosin Y Solution Aqueous | Sigma Aldrich | HT110216 | |
| Formic Acid, 99+ % | Thermo Fisher Scientific | 28905 | Mobile phase additive |
| ggplot2 version 3.3.5 | CRAN | https://cran.r-project.org/web/packages/ggplot2/ | |
| HALO | Indica Labs | Image analysis software | |
| IDLE (Integrated Development and Learning Environment) | Python Software Foundation | ||
| iheatmapr version 0.5.1 | CRAN | https://cran.r-project.org/web/packages/iheatmapr/ | |
| iRT Kit | Biognosys | Ki-3002-1 | LC-MS QAQC Standard |
| limma version 3.42.2 | Bioconductor | https://bioconductor.org/packages/release/bioc/html/limma.html | |
| LMD Scanning stage Ultra LMT350 | Leica Microsystems | 11888453 | LMD stage model outfitted with PCT tube holder |
| LMD7 (software version 8.2.3.7603) | Leica Microsystems | LMD apparatus (microscope, laser, camera, PC, tablet) | |
| Mascot Server | Matrix Science | Data analysis software | |
| Mass Spec-Compatible Human Protein Extract, Digest | Promega | V6951 | LC-MS QAQC Standard |
| Mayer’s Hematoxylin Solution | Sigma Aldrich | MHS32 | |
| PEN Membrane Glass Slides | Leica Microsystems | 11532918 | |
| Peptide Retention Time Calibration Mixture | Thermo Fisher Scientific | 88321 | LC-MS QAQC Standard |
| Phosphatase Inhibitor Cocktail 2 | Sigma Aldrich | P5726 | |
| Phosphatase Inhibitor Cocktail 3 | Sigma Aldrich | P0044 | |
| Pierce LTQ Velos ESI Positive Ion Calibration Solution | Thermo Fisher Scientific | 88323 | Instrument calibration solution |
| PM100 C18 3UM 75UMX20MM NV 2PK | Fisher Scientific | 164535 | Pre-column |
| Proteome Discoverer | Thermo Fisher Scientific | OPTON-31040 | Data analysis software |
| Python | Python Software Foundation | ||
| Q Exactive HF-X | Thermo Fisher Scientific | Mass spectrometer | |
| R version 3.6.0 | CRAN | https://cran-archive.r-project.org/bin/windows/base/old/2.6.2/ | |
| RColorBrewer version 1.1-2 | CRAN | https://cran.r-project.org/web/packages/RColorBrewer/ | |
| Soluble Smart Digest Kit | Thermo Fisher Scientific | 3251711 | Digestion reagent |
| TMTpro 16plex Label Reagent Set | Thermo Fisher Scientific | A44520 | isobaric TMT labeling reagents |
| Veriti 60 well thermal cycler | Applied Biosystems | 4384638 | Thermocycler |
| Water, Optima LC/MS Grade | Fisher Chemical | W6-4 | Mobile phase solvent |
| ZORBAX Extend 300 C18, 2.1 x 12.5 mm, 5 µm, guard cartridge (ZGC) | Agilent Technologies Inc | 821125-932 | Offline LC trap column |
| ZORBAX Extend 300 C18, 2.1 x 150 mm, 3.5 µm | Agilent Technologies Inc | 763750-902 | Offline LC analytical column |
References
- Motohara, T., et al. An evolving story of the metastatic voyage of ovarian cancer cells: cellular and molecular orchestration of the adipose-rich metastatic microenvironment. Oncogene. 38 (16), 2885-2898 (2019).
- Aran, D., Sirota, M., Butte, A. J. Systematic pan-cancer analysis of tumour purity. Nature Communications. 6, 8971 (2015).
- Hunt, A. L., et al. Extensive three-dimensional intratumor proteomic heterogeneity revealed by multiregion sampling in high-grade serous ovarian tumor specimens. iScience. 24 (7), 102757 (2021).
- Dou, Y., et al. Proteogenomic characterization of endometrial carcinoma. Cell. 180 (4), 729-748 (2020).
- Zhang, H., et al. Integrated proteogenomic characterization of human high-grade serous ovarian cancer. Cell. 166 (3), 755-765 (2016).
- Gillette, M. A., et al. Proteogenomic characterization reveals therapeutic vulnerabilities in lung adenocarcinoma. Cell. 182 (1), 200-225 (2020).
- Silvestri, A., et al. Protein pathway biomarker analysis of human cancer reveals requirement for upfront cellular-enrichment processing. Laboratory Investigation. 90 (5), 787-796 (2010).
- Echle, A., et al. Deep learning in cancer pathology: a new generation of clinical biomarkers. British Journal of Cancer. 124 (4), 686-696 (2021).
- Lee, S., et al. Molecular analysis of clinically defined subsets of high-grade serous ovarian cancer. Cell Reports. 31 (2), 107502 (2020).
- Xuan, Y., et al. Standardization and harmonization of distributed multi-center proteotype analysis supporting precision medicine studies. Nature Communications. 11 (1), 5248 (2020).
- Hughes, C. S., et al. Quantitative profiling of single formalin fixed tumour sections: proteomics for translational research. Scientific Reports. 6 (1), 34949 (2016).
- Espina, V., et al. A portrait of tissue phosphoprotein stability in the clinical tissue procurement process. Molecular & Cellular Proteomics. 7 (10), 1998-2018 (2008).
- Espina, V., Heiby, M., Pierobon, M., Liotta, L. A. Laser capture microdissection technology. Expert Review of Molecular Diagnostics. 7 (5), 647-657 (2007).
- Havnar, C. A., et al. Automated dissection protocol for tumor enrichment in low tumor content tissues. Journal of Visualized Experiments. (169), e62394 (2021).
- Mueller, C., et al. One-step preservation of phosphoproteins and tissue morphology at room temperature for diagnostic and research specimens. PLoS One. 6 (8), (2011).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved