Method Article
Tümör Mikroçevresinin Mikro Ölçekli Proteomik Analizi için Endüstriyelleşmiş, Yapay Zeka Rehberliğinde Lazer Mikrodiseksiyonu
* Bu yazarlar eşit katkıda bulunmuştur
Bu Makalede
Özet
Bu protokol, lazer mikrodiseksiyonu kullanarak histoloji ile çözülmüş hücre popülasyonlarının zenginleştirilmesi için lekeli, ince doku kesiti görüntülerinden patoloji onaylı ilgi alanlarının yapay zeka odaklı segmentasyonu için yüksek verimli bir iş akışını açıklamaktadır. Bu strateji, ilgilenilen hücre popülasyonlarını gösteren sınırların doğrudan lazer mikroskoplara aktarılmasını sağlayan yeni bir algoritma içerir.
Özet
Tümör mikroçevresi (TME), tümör, stroma ve bağışıklık hücresi popülasyonları dahil olmak üzere düzinelerce farklı hücre tipinden oluşan karmaşık bir ekosistemi temsil eder. Proteom düzeyindeki varyasyonu ve tümör heterojenitesini ölçekte karakterize etmek için, solid tümör malignitelerinde ayrık hücresel popülasyonları seçici olarak izole etmek için yüksek verimli yöntemlere ihtiyaç vardır. Bu protokol, lazer mikrodiseksiyonu (LMD) kullanarak histoloji ile çözülmüş hücre popülasyonlarının seçici hasadı için hematoksilin ve eozin (H & E) lekeli, ince doku bölümlerinin görüntülerini patoloji tarafından doğrulanmış ilgi alanlarına ayıran yapay zeka (AI) tarafından sağlanan yüksek verimli bir iş akışını tanımlar. Bu strateji, dijital görüntü yazılımı kullanılarak ek açıklamalı, ilgilenilen hücre popülasyonlarını gösteren bölgelerin doğrudan lazer mikroskoplara aktarılmasını sağlayan ve böylece daha kolay koleksiyonlara olanak tanıyan yeni bir algoritma içerir. Bu iş akışının başarılı bir şekilde uygulanması, bu uyumlaştırılmış yöntemin, yüksek çözünürlüklü kütle spektrometrisi ile kantitatif, çoklanmış proteomik analiz için TME'den tümör hücresi popülasyonlarını seçici olarak toplamak için faydasını gösteren bir uygulama gerçekleştirildi. Bu strateji, rutin histopatoloji incelemesi ile tamamen bütünleşir, ilgilenilen hücresel popülasyonların zenginleştirilmesini desteklemek için dijital görüntü analizinden yararlanır ve multiomik analizler için TME'den hücre popülasyonlarının uyumlu bir şekilde toplanmasını sağlayarak tamamen genelleştirilebilir.
Giriş
TME, tümör hücreleri, stromal hücreler, bağışıklık hücreleri, endotel hücreleri, diğer mezenkimal hücre tipleri ve adipositler gibi çok çeşitli hücre tipleri ile birlikte karmaşık bir hücre dışı matris1 ile doldurulan karmaşık bir ekosistemi temsil eder. Bu hücresel ekosistem, farklı hastalık organ bölgeleri içinde ve arasında değişir ve karmaşık tümör heterojenitesine neden olur 2,3. Son zamanlarda yapılan çalışmalar, heterojen tümörlerin ve düşük tümör hücreselliğine (düşük saflık) sahip tümörlerin sıklıkla kötü hastalık prognozu ile ilişkili olduğunu göstermiştir 2,3.
TME içindeki tümör ve tümör dışı hücre popülasyonları arasındaki moleküler etkileşimi ölçekli olarak anlamak için, aşağı akış multiomik analiz için ilgi çekici farklı hücresel popülasyonları seçici olarak toplamak için standartlaştırılmış ve yüksek verimli stratejiler gereklidir. Kantitatif proteomik, kanser biyolojisinin anlaşılmasını ilerletmek için hızla gelişen ve giderek daha önemli bir tekniği temsil eder. Bugüne kadar, proteomik kullanan çalışmaların üstünlüğü, tüm tümör dokusu preparatlarından (örneğin, kriyopulverize) ekstrakte edilen proteinlerle bunu yapmış ve TME 4,5,6'da proteom seviyesi heterojenitesinin anlaşılmasında bir kıtlığa yol açmıştır.
Klinik patoloji iş akışlarıyla sorunsuz bir şekilde entegre olan ve bunlardan yararlanan numune toplama stratejilerinin geliştirilmesi, altın standart, tanısal patoloji iş akışlarını son derece tamamlayıcı nitelikte olan yeni nesil histoloji çözümlü proteomiklere olanak sağlayacaktır. LMD, histolojik olarak boyanmış doku ince kesitlerinin mikroskobik incelemesi yoluyla hücresel alt popülasyonların veya ilgili bölgelerin (ROI'ler) doğrudan ve seçici bir şekilde toplanmasını sağlar7. Dijital patoloji ve AI özellikli analizdeki son büyük gelişmeler, TME içindeki benzersiz bileşimsel özellikleri ve ROI'leri, birçoğu moleküler değişiklikler ve tedaviye direnç ve hastalık prognozu gibi klinik hastalık özellikleri ile ilişkili olan otomatik bir şekilde tanımlama yeteneğini göstermiştir8.
Burada sunulan protokolde açıklanan iş akışı, dijital histopatoloji görüntülerindeki tümör YG'lerine seçici bir şekilde açıklama eklemek için ticari yazılım çözümlerinden yararlanır ve bu tümör yatırım getirilerini lazer mikroskoplara aktarmak için şirket içinde geliştirilen yazılım araçlarından yararlanır ve böylece aşağı akış multiomik analiz iş akışlarıyla sorunsuz bir şekilde bütünleşen ayrı hücresel popülasyonların otomatik olarak toplanması sağlanır. Bu entegre strateji, LMD operatör süresini önemli ölçüde azaltır ve dokuların ortam sıcaklığında olması gereken süreyi en aza indirir. Otomatik özellik seçimi ve LMD hasadının yüksek verimli kantitatif proteomiklerle entegrasyonu, TME'nin iki temsili epitelyal over kanseri histolojik alt tipinden, yüksek dereceli seröz over kanseri (HGSOC) ve yumurtalık berrak hücreli karsinomundan (OCCC) diferansiyel analizi ile gösterilmiştir.
Protokol
Tüm çalışma protokolleri, ABD Federal yönetmeliği 45 CFR 46.102 (f) kapsamında muaf tutulan Batı IRB onaylı bir protokol olan "Klinik Olarak Bilgilendirici Biyobelirteçleri Tanımlamak ve Doğrulamak için Endometriyal ve Yumurtalık Kanserinin Entegre Moleküler Analizi" kapsamında kullanılmak üzere onaylanmıştır. Bu çalışmada insan verilerini içeren tüm deneysel protokoller Helsinki Deklarasyonu'na uygundur. Çalışmaya dahil olan tüm deneklerden bilgilendirilmiş onam alınmıştır.
DİKKAT: Protokol boyunca kullanılan aşağıdaki reaktifler bilinen veya şüphelenilen kanserojenlerdir ve / veya tehlikeli maddeler içerir: etanol, DEPC suyu, Mayer'in Hematoksilin çözeltisi, Eozin Y çözeltisi, metanol, asetonitril ve formik asit. İlgili güvenlik bilgi formlarında (SDS) açıklandığı gibi uygun şekilde kullanım ve uygun kişisel koruyucu ekipmanların (KKD) kullanılması zorunludur.
1. Kalibratör referanslarını içeren varsayılan şekil listesi veri (.sld) dosyasını oluşturma
NOT: Bu bölümde açıklanan protokol adımları, ters lazer mikroskop ve ilgili yazılımla birlikte kullanıma özeldir (bkz. Varsayılan bir .sld dosyasının oluşturulması, lazer mikroskop başına yalnızca bir kez gereklidir. Elde edilen dosya, daha sonra kullanılan tüm PEN slaytlarına referansları kesmek için kullanılabilir. Yaklaşık süre: 5 dk (yalnızca bir kez).
- LMD yazılımını açın ve polietilen naftalat (PEN) membran kızağını LMD aşamasına kullanıcıya en yakın etiketle birlikte yüzü aşağı bakacak şekilde yükleyin. Program penceresinin sağ tarafındaki Satırları kapat kutusunun seçimini kaldırın.
- Kalibrasyon referansları olarak kullanılmak üzere üç "V" ok çizmek için yüksek büyütme (63x) altında PtoP (noktadan noktaya) işlevini kullanın. V üzerindeki bir dış noktadan başlayarak, V'nin orta noktasına bir çizgi çizin ve tek bir tıklama yapın. Ardından, V'nin merkezi noktasından ikinci harici V noktasının sonuna kadar ikinci bir çizgi çizin ve iki çizgiden tek, kapatılmamış bir V şekli oluşturmak için çift tıklatın.
NOT: Bu kalibrasyon referansları kızağın üç köşesine yerleştirilmelidir: ön sağ, arka sağ, arka sol. - Kesmeden önce AF (otomatik netleme) seçeneğini belirleyin. Slaytı 1 konumunda kesin, kalan slayt konumlarının her birine taşıyın ve kalibrasyon kesimleri üzerinde hassas bir şekilde izleyin.
- .sld dosyasını kaydedin ve kalibrasyon referanslarının membrana kesilmesini önlemek için açılır iletişim kutusundan Kalibrasyon olmadan kaydet seçeneğini belirleyin.
NOT: Dört slayt konumu için standart kalibratör referanslarını içeren temsili bir .sld dosyası Ek Dosya 1'de sağlanmıştır.
2. LMD slayt(lar)ının hazırlanması
NOT: Bu bölümde açıklanan protokol adımları, ters lazer mikroskop ve ilgili yazılımla birlikte kullanıma özeldir (bkz. Yaklaşık süre: 5 dk.
- Referans kalibrasyon referanslarını kesmeden önce kızağın tamamen kuru olduğundan emin olun. LMD yazılımını açın ve Şekilleri içe aktar seçeneğinin altındaki varsayılan kalibrasyon .sld dosyasını açın.
- Kesmeden önce AF (otomatik netleme) seçeneğini belirleyin. Kızakları, doku aşağı bakacak şekilde ve etiket operatörün yakınına kaydırarak LMD aşamasındaki slayt tutucuya yükleyin.
- Lazer mikroskobu ve varsayılan kalibrasyon .sld dosyasını kullanarak, kalibrasyon referanslarını PEN membranına kesin.
- İSTEĞE BAĞLI: Kalibrasyon referanslarını, doku kesitleri slayta yerleştirilmeden önce veya sonra PEN membranına kesin. Kalibrasyon referansları doku yerleştirilmeden önce kesilirse, doku Adım 2.5'te slayta yerleştirildiğinde doku ve / veya fiksatifin kalibratörlerle çakışmadığından emin olun. Kalibrasyon referansları doku yerleştirildikten sonra kesilirse, Adım 2.4'ün tamamlanmasından sonra durun ve bölüm 3'e geçin.
- Her kesimin eksiksiz ve görünür olduğundan emin olmak için tüm kalibratörleri ayrı ayrı gözden geçirin.
NOT: Lazeri, PEN membranını tam olarak kesmeyen kalibrasyon referansları üzerinde manuel olarak yönlendirmek için Taşı ve Kes özelliğini kullanın. - Dondurulmuş veya formalin sabit, parafin gömülü (FFPE) doku bölümünü, kalibrasyon referanslarını içeren slayda yerleştirin.
3. Doku boyama
NOT: Yaklaşık süre: 30 dk.
- Dondurulmuş LMD doku slaytlarını fosfataz inhibitörü kokteyl reaktifleri içeren %70 etanol (EtOH) içinde 5 dakika boyunca sabitleyin.
- Slaytları fosfataz inhibitörü kokteyl reaktifleri içeren dietil pirokarbomat (DEPC) suda 1 dakika boyunca yıkayın.
- Slaytları DEPC suyunda 1 dakika yıkayın.
- Mayer'in Hematoksilin çözeltisindeki slaytları 3 dakika boyunca inkübe edin.
- Kızakları DEPC suyunda 3 dakika durulayın.
- Slaytları 1 dakika boyunca taze DEPC suyu değişiminde durulayın.
- Slaytları 1 s için sulu Eosin Y çözeltisinde inkübe edin.
- Slaytları 2 x 5 sn %95 EtOH ile durulayın.
- Slaytları 3 x 10 sn hızda% 100 EtOH'de durulayın.
- Fazla EtOH'yi slaytların arkasından silin ve slaytların hava ile kurumasını bekleyin.
- LMD hemen yapılmayacaksa slaytları -80 °C'de saklayın.
4. Slayt görüntüleme
NOT: Bu bölümde açıklanan protokol adımları, taranan slaytlara ( Malzeme Tablosu'na bakın) ve .svs dosyaları olarak kaydedilen sonuç görüntülerine özgüdür. Görüntü analiz yazılımının (bkz. Malzeme Tablosu) açabileceği bir biçimde görüntü dosyaları oluşturan herhangi bir tarayıcı ve ilişkili yazılımı kullanın. Desteklenen piramidal tiff'leri kullanan dosya türleri arasında JPG, TIF, MRXS, QPTIFF, TIFF bileşeni, SVS, AFI, SCN, LIF, DCM, OME bulunur. TIFF, ND2, VSI, NDPI, NDPIS, CZI, BIF, KFB ve ISYNTAX. Yaklaşık süre: 5 dk.
- Tarayıcıyı açın ve slayt tarayıcı yazılımını açın. Slaytı, doku yukarı bakacak şekilde tarayıcıdaki tek slayt aşamasına yükleyin. Slaytın tamamen kuru olduğundan emin olun ve dokunun üzerine yavaşça bir örtü parçası yerleştirin. Kapak kapağının altına etanol veya daldırma yağı kullanmayın.
- Üreticinin talimatlarına göre, cam arka plan yerine PEN membranı ayarlamak ve arka plan membran boyamasını yoksaymak için kalibre edilmiş ayarları kullanarak mikrograf görüntüsünü yakalayın.
- Gerektiğinde tüm PEN membran alanını yakalamak için iç yeşil çevreyi sürükleyip yeniden boyutlandırarak görüntüleme alanını ayarlayın. Anlık görüntüye genel bakış görüntüsüne çift tıklayarak dokuya dört netleme noktası ve membran üzerinde kalibrasyon referanslarının yakınında üç netleme noktası (üç kalibrasyon referansının her biri için bir netleme noktası) ekleyin.
NOT: Dört netleme noktası doku kesitinin hemen hemen her yerine yerleştirilebilir, ancak çok koyu lekeli ve siyah görünen dokuya yerleştirmek taramanın başarısız olmasına neden olabilir. - Görünüm menüsü altında Video Monitörü'nü seçin. LMD dokusunun etrafındaki her nokta için ince ve/veya makro netleme kaydırıcısını kullanarak gerektiğinde odağı manuel olarak ayarlayın. Görüntü taramasını yüksek büyütme (20x) altında yakalayın. Kaydedilen görüntüde tüm kalibrasyon referanslarının görünür ve net olduğunu onaylayın.
5. Görüntü analiz yazılımı kullanarak otomatik özellik seçimi
- Tüm tümör koleksiyonları için (yaklaşık süre: 5 dakika; vakaya bağlı):
- Görüntü analiz yazılımını açın ( bkz. Malzeme Tablosu). Görüntüleri Aç'ı seçin ve açılır pencereden, AT2 tarayıcısında slaytın taranmasından oluşturulan .svs görüntü dosyasını seçin.
NOT: Ek Dosya 2'de temsili bir .svs görüntü dosyası verilmiştir. - Ek Açıklamalar sekmesine gidin. Ek Açıklama araç çubuğunda Kalem aracını seçin ve dokunun etrafına bir şekil çizin.
- Şekli seçin ve resme sağ tıklayın. Gelişmiş açılır menüsünden Bölümleme (Döşemeli)'yi seçin. Kutucuk Boyutu ve Boşluk Aralığı'nı sırasıyla 500 ile 40 arasında ayarlayın ve kutucukları oluşturmak için Tamam'ı seçin. Adım 5.1.2'de kutucukları oluşturmak için kullanılan çevre şeklini seçin ve silin.
- Katman Eylemleri açılır menüsünü seçin | Döşenmiş ek açıklamaları .annotation dosyası olarak kaydetmek için dışa aktarın.
NOT: Tüm tümör dokusu koleksiyonu için temsili bir .ek açıklama dosyası Ek Dosya 3'te verilmiştir. - Oturum veya proje için bir klasör oluşturun ve .annotation dosyasını, slaydın benzersiz tanımlayıcısıyla etiketlenmiş bir alt klasörün içine kaydedin.
- Ek Açıklamalar sekmesine gidin. Katman Eylemleri açılır | Görüntüdeki tüm ek açıklamaları kaldırmak için Tüm Katmanları Sil. Kalem aracını seçin ve her kalibrasyon referansı için ok ucunun iç ucundan kısa bir çizgi çizin. İşaretlerden çizgileri aşağıdaki sırayla çizin: sol üst, sağ üst, sağ alt.
- Katman Eylemleri açılır menüsünü seçin | Satır detaylandırmalarını .annotation dosyası olarak kaydetmek için dışa aktarın. Dosya adına _calib ekleyin ve dosyayı döşenmiş şekillerin koordinatlarını içeren alt klasöre yerleştirin.
NOT: Ek Dosya 4'te temsili bir _calib.ek açıklama dosyası sağlanmıştır. - Ana projenin veya oturum klasörünün adresini kopyalayın. IDLE tümleşik geliştirme ortamını kullanarak XML içe aktarma oluşturma komut dosyası "Malleator" (https://github.com/GYNCOE/Mitchell.et.al.2022 aracılığıyla kullanılabilir) açın ve proje klasörü adresini komut dosyasının altındaki tırnak işaretleri arasına yapıştırın.
- Çalıştır açılır menüsünü seçin | Komut dosyasını yürütmek için Modül'ü çalıştırın.
NOT: .xml LMD içe aktarma dosyası, görüntü/slayt için oluşturulan alt klasörün içinde oluşturulur. Ek Dosya 5'te temsili bir .xml dosyası verilmiştir.
- Görüntü analiz yazılımını açın ( bkz. Malzeme Tablosu). Görüntüleri Aç'ı seçin ve açılır pencereden, AT2 tarayıcısında slaytın taranmasından oluşturulan .svs görüntü dosyasını seçin.
- Yalnızca LMD ile zenginleştirilmiş koleksiyonlar için (yaklaşık süre: 15 dakika; büyük/küçük harfe bağlı):
- Görüntü analiz yazılımını açın ( bkz. Malzeme Tablosu). Görüntüleri Aç'ı seçin ve açılır pencereden slaytın taranmasıyla oluşturulan .svs görüntü dosyasını seçin.
- Ek Açıklamalar sekmesine gidin. Dokunun etrafına bir kutu çizmek için Dikdörtgen ek açıklama aracını seçin ve kullanın.
- Kutu ek açıklamasını seçin ve resme sağ tıklayın. Gelişmiş açılır menü | Bölümleme (Döşenmiş) seçeneği. Kutucuk Boyutu ve Boşluk Aralığı'nı sırasıyla 500 ile 40 arasında ayarlayın ve kutucukları oluşturmak için Tamam'ı seçin. Adım 5.2.2'de kutucukları oluşturmak için kullanılan çevre kutusu ek açıklamasını seçin ve silin.
- Katman Eylemleri açılır menüsünü seçin | Döşenmiş ek açıklamaları .annotation dosyası olarak kaydetmek için dışa aktarın.
- AI tarafından sınıflandırılmış ek açıklama katmanlarını birleştirmek için geliştirilen Python "Dapọ" (https://github.com/GYNCOE/Mitchell.et.al.2022 aracılığıyla kullanılabilir) algoritmasının kaydedilmiş bir kopyasını, döşenmiş ek açıklamalar dosyasıyla aynı klasöre yerleştirin.
- Döşenmiş ek açıklama dosyasının adını kopyalayın. IDLE tümleşik geliştirme ortamını kullanarak Python programını açın ve döşenmiş ek açıklama dosyasının adını programın altındaki alıntılar arasına yapıştırın.
- Çalıştır açılır menüsünü seçin | Modülü çalıştırın. Tüm döşenmiş ek açıklamaların tek bir katman altında birleştirileceği yeni bir dosyanın oluşturulmasını bekleyin.
- Görüntü analizi yazılımını açın ve Ek Açıklamalar sekmesine gidin. Katman Eylemleri açılır menüsünü seçin | Görüntüdeki tüm ek açıklamaları kaldırmak için Tüm Katmanları Sil.
- Katman Eylemleri açılır menüsünü seçin | Yerel Ek Açıklama Dosyasını İçe Aktarın. Açılır pencerede, komut dosyası tarafından oluşturulan birleştirilmiş .annotation dosyasını seçin. İçe aktarılan tüm kutucukların aynı ek açıklama katmanı altında olduğundan emin olun.
- Sınıflandırıcı sekmesine gidin ve YG'ler için şekiller oluşturmak üzere üreticinin yönergelerini izleyin. Sınıflandırıcıyı çalıştırmadan önce, Gelişmiş Sınıflandırıcı Seçenekleri altındaki Ek Açıklamalar sekmesindeki YG kutusunu veya kutularını işaretleyerek istediğiniz ek açıklama katmanlarını (yani tümör veya stroma katmanını) seçin. Sınıflandırıcıyı çalıştırmak için Sınıflandırıcı Eylemleri menüsünden Ek Açıklama Katmanı seçeneğini kullanın.
- Sınıflandırıcı analizi tamamlandıktan sonra, Ek Açıklamalar sekmesine gidin ve analizden oluşturulan detaylandırma katmanını seçin. Katman Eylemleri açılır menüsünü seçin | Diğer tüm ek açıklama katmanlarını görüntüden kaldırmak için Tüm Katmanları Sil Ancak Geçerli Olan.
- Katman Eylemleri açılır menüsünü seçin | Ek açıklamaları .annotation dosyası olarak kaydetmek için dışa aktarın. Oturum veya proje için bir klasör oluşturun ve .annotation dosyasını, slaydın benzersiz tanımlayıcısıyla etiketlenmiş bir alt klasörün içine kaydedin.
NOT: Sınıflandırılmış LMD ile zenginleştirilmiş doku koleksiyonu için temsili bir .ek açıklama dosyası Ek Dosya 6'da verilmiştir. - Ek Açıklamalar sekmesine gidin, Katman Eylemleri açılır menüsünü seçin | Görüntüdeki tüm ek açıklamaları kaldırmak için Tüm Katmanları Sil. Kalem aletini seçin ve her kalibrasyon referansından kısa bir çizgi çizin. İşaretlerden çizgileri aşağıdaki sırayla çizin: sol üst, sağ üst, sağ alt.
- Katman Eylemleri açılır menüsünü seçin | Satır detaylandırmalarını .annotation dosyası olarak kaydetmek için dışa aktarın. Dosya adına _calib ekleyin ve dosyayı döşenmiş şekillerin koordinatlarını içeren alt klasöre yerleştirin.
- Ana projenin veya oturum klasörünün adresini kopyalayın. IDLE tümleşik geliştirme ortamını kullanarak XML içe aktarma oluşturma komut dosyası "Malleator" (https://github.com/GYNCOE/Mitchell.et.al.2022 aracılığıyla kullanılabilir) açın, ardından proje klasörü adresini komut dosyasının altındaki tırnak işaretleri arasına yapıştırın.
- Çalıştır açılır menüsünü seçin | Komut dosyasını yürütmek için Modül'ü çalıştırın.
NOT: .xml LMD içe aktarma dosyası, görüntü/slayt için oluşturulan alt klasörün içinde oluşturulur.
6. Lazer mikrodiseksiyonu
NOT: Bu bölümde açıklanan protokol adımları, ters lazer mikroskop ve ilgili yazılımla birlikte kullanıma özeldir (bkz. Yaklaşık süre: 2 saat; duruma bağlı.
- İşaretli membran kızağını (kalibrasyon referansları içeren) doku aşağı bakacak şekilde ve etiket tarafı operatöre daha yakın olacak şekilde lazer mikroskop aşamasındaki slayt tutucuya yükleyin.
- Dosya açılır menüsünden Şekilleri İçe Aktar'ı seçin. Slayt için oluşturulan .xml LMD içe aktarma dosyasını seçin. Dosyadan referans noktalarının yüklenmesini önlemek için açılır pencerede Hayır'ı ve kalibrasyon için önceden depolanmış referans noktalarını kullanmaktan kaçınmak için ikinci açılır pencerede Hayır'ı seçin.
- LMD uygulamasındaki istemleri izleyin ve kalibrasyon çarpı işaretini slayttaki üç kalibrasyon referansının her birine hizalayın. Görüntü analiz yazılımında, mikroskop aşamasında ters çevrilmiş LMD slaytının sırasıyla ön sağ, arka sağ ve arka sol köşelerindeki referans noktalarına karşılık gelecek olan slayt görüntüsünün sol üst, sağ üst ve sağ alt kısımlarında görünen kalibrasyon referanslarını arayın. Konumlandırmak için 5x objektif lensi ve her kalibrasyon referansını hizalamak için 63x objektifi kullanma arasında geçiş yapın. Referans noktalarının dosyaya kaydedilmesini önlemek için açılır pencerede Hayır'ı ve slaytın eklendiğini onaylamak için ikinci açılır pencerede Tamam'ı seçin.
- 5x objektif lensi konumuna getirin ve gerçek büyütmeyi kullanmak için açılır pencerede Evet'i seçin. İçe aktarılan şekiller göründüğünde, kamerayı dokuya odaklayın.
- Şekil Listesi penceresindeki tüm şekilleri vurgulayın ve seçin, referans olarak görüş alanındaki bir veya iki ek açıklamayı kullanarak bunları yerlerine sürükleyin ve lazerle kesmek için dikey z eksenini hizalayın.
- İçe aktarılan şekilleri gözden geçirin ve toplama için uygun tüp konumuna atayın. Lazeri başlatmak için Start Cut (Kesimi Başlat) düğmesine basın.
NOT: .xml dosyasındaki içe aktarılan şekiller, Şekil Listesi penceresindeki "kapaksız" konuma otomatik olarak atanır. Doku toplamak için, içe aktarılan şekiller yüklü bir tüp içeren bir konuma yeniden atanmalıdır.
7. Basınç döngüsü teknolojisi (PCT) ile protein sindirimi
NOT: Yaklaşık süre: 4 saat (vakumlu santrifüj kuruma süresi olmadan 3 saat).
- LMD hasat edilmiş dokuyu içeren kapaklı PCT Mikrotüplerini içeren 0,5 mL tüpleri 20 μL 100 mM TEAB/%10 asetonitril içinde bir termosikler içine yerleştirin ve 30 dakika boyunca 99 °C'de ısıtın, ardından 10 dakika boyunca 50 °C'ye soğutun.
- Tüpleri 4.000 × g'da 30 s döndürün ve ardından Mikrotüpleri 0,5 mL tüplerden çıkarın. MicroCap aracını kullanarak, MicroCap'leri PCT MicroTubes'tan çıkarın ve atın. 30mm2 doku başına 1 μg oranında tripsin ekleyin (Malzeme Tablosuna bakın) ve MicroCap aracını kullanarak MicroTube'a bir MicroPestle yerleştirin.
- MicroTubes'u bir barosikler kartuşuna aktarın ve kartuşun tamamını monte edin. Kartuşu barosikler basınç odasına yerleştirin ve kapağı sabitleyin. 50 s için 45.000 psi'de barocycle ve 60 döngü için 50 °C'de 10 s atmosferik basınç.
- Barocycling tamamlandıktan sonra, mikrotüpleri 0,5 mL'lik bir mikrosantrifüj tüpüne aktarın ve 4.000 × g'da 2 dakika boyunca santrifüj yapın.
- Kapak aletini kullanarak MicroTube'u 0,5 mL mikrosantrifüj tüpünden çıkarın. Kapak aletini kullanarak havaneyi dikkatlice çıkarın ve havanenin alt yarısını 20 μL sıvı kromatografi-kütle spektrometresi (LC-MS) sınıfı su ile durulayın ve yıkamayı temiz bir 0,5 mL mikrosantrifüj tüpünde toplayın.
- Sıvıyı aşağıya taşımak ve tüm çözeltiyi MicroTube'dan 0,5 mL mikrosantrifüj tüpüne aktarmak için tezgahın üstündeki MicroTube'a hafifçe dokunun.
- MicroTube'a 20 μL LC-MS sınıfı su ekleyin ve tezgahın üstüne hafifçe dokunun. Yıkama solüsyonunu 0,5 mL tüpe aktarın ve bu yıkama adımını bir kez daha tekrarlayın.
- Numuneleri ~2 μL'ye kurutmak için vakumlu santrifüj yapın ve 100 μL 100 mM TEAB, pH 8.0 ekleyin.
- Üreticinin protokolüne göre kolorimetrik bir tahlil (biskinkoninik asit (BCA) testi; Malzeme Tablosuna bakınız) kullanarak peptid konsantrasyonunu belirleyin.
8. Tandem-kütle etiketi (TMT) etiketleme ve EasyPep temizleme
NOT: Yaklaşık süre: 7 sa 20 dk (vakumlu santrifüj kuruma süresi olmadan 2 sa 20 dk).
- İzobarik TMT etiketleme reaktiflerini açmadan önce ortam sıcaklığına getirin. Her TMT şişesine (5 mg) 500 μL% 100 asetonitril ekleyin. Ara sıra vorteks ile 10 dakika boyunca kuluçkaya yatın.
- Peptit numunesinin 5 μg'ını 100 mM TEAB, pH 8.0'ın 100 μL'sinde çözün ve belirli bir TMT reaktifinin 10 μL'sini ekleyin. Birden fazla TMT multipleks arasında numunelerin nicelleştirilmesini kolaylaştırmak için her bir TMT multipleks numune setinde deneydeki her bir numuneyi temsil eden referans havuzları oluşturun ve dahil edin9. Ara sıra sallama/dokunma ile ortam sıcaklığında 1 saat boyunca reaksiyonları inkübe edin.
- TMT etiketleme reaksiyonunu 10 μL %5 hidroksilamin ekleyerek söndürün ve ara sıra dokunarak ortam sıcaklığında 30 dakika inkübe edin. Söndürdükten sonra, TMT etiketli numuneleri tek bir tüpte birleştirin ve yaklaşık 200 μL'ye kadar kurutun.
- 1.800 μL% 0.1 formik asit ekleyin. pH kağıdı ile pH'ı kontrol edin: pH ~ 3 ise, 1 mL% 0.1 formik asit ekleyin; pH >3 ise, pH ~ 3'e kadar 10-20 μL% 5 formik asit ekleyin. 3 mL'lik bir son hacme getirmek için% 0.1 formik asit ekleyin.
- Peptit Temizleme Sütununun altındaki tırnağı çıkarın, kapağı çıkarın ve 15 mL'lik konik bir tüpe yerleştirin. TMT etiketli numuneyi sütuna aktarın ve üreticinin protokolüne göre temizlemeye devam edin.
- Salınımlı peptitleri ~ 20 μL'ye kurutmak için vakumlu santrifüj, 80 μL'lik son hacim için 25 mM amonyum bikarbonat kullanarak bir LC şişesine aktarın ve çevrimdışı fraksiyonasyona geçin.
9. TMT multipleks numune fraksiyonasyonu ve havuzlaması
NOT: Yaklaşık süre: 3 sa 30 dk (vakumlu santrifüj kuruma süresi olmadan 1 sa 30 dk).
- TMT etiketli peptid multiplekslerini, mobil faz A'ya (10 mM NH4HCO3, pH 8.0) artan bir doğrusal gradyan (% 0.69 min-1) mobil faz B (asetonitril) geliştirerek 96 fraksiyona bölün.
- Örnek kuyucukları bir araya getirerek 36 birleştirilmiş fraksiyon oluşturun. Kuru fraksiyonları ~2 μL'ye kadar vakumlayın ve 25 mM amonyum bikarbonatta (son konsantrasyon 1.5 μg / 10 μL) yeniden askıya alın, 10-15 dakika boyunca 15.000 × g'da santrifüj yapın ve MS analizi için LC şişelerine aktarın.
10. Sıvı kromatografisi tandem kütle spektrometresi (LC-MS / MS)
NOT: Yaklaşık süre: Alet yöntemine ve deneysel tasarıma bağlıdır.
- Kütle spektrometresini üreticinin talimatlarına/protokollerine göre kalibre edin.
- Yeni mobil aşamalar ve standartlar hazırlayın ve uygun LC çalıştırma öncesi hazırlıkları gerçekleştirin (solventleri temizleme, yıkama havası ve referans verilen cihaz için sızıntı testi komut dosyaları dahil ancak bunlarla sınırlı olmamak üzere [ Malzeme Tablosuna bakınız]). Analizlere başlamadan önce ön ve analitik sütunları ve numune döngüsünü dengeleyin.
- Seri TMT multipleks analizlerinden önce ve bu analizler arasında, LC-MS sisteminin kalite güvencesi/kalite kontrol (QA/QC) TMT etiketli peptid sindirimlerini ve (örneğin, MSPE ( Malzeme Tablosuna bakınız), HeLa) kullanarak daha önce karşılaştırılmış performans metriklerini karşıladığını doğrulayın.
- Otomatik numune alma şişelerini LC otomatik numune alma cihazında uygun konumlara yükleyin. Tek tek kesirleri uygun bir gradyan/MS yöntemiyle analiz edin. Kromatografik ve kütle spektral performansını değerlendirmek için günde yaklaşık bir kez peptid standardı (örneğin, peptid tutma süresi kalibrasyonu [PRTC]) ile bir "yıkama" çalışmasını serpiştirin. Her TMT multipleks numune fraksiyon serisinin analizini takiben, sistem performansını değerlendirmek için QA/QC TMT kıyaslama standartlarını çalıştırın.
- Numune sonrası performansı değerlendirmek için QA/QC TMT kıyaslama standartlarından sonra kütle spektrometresi değerlendirme rutinlerini çalıştırın ve ardından bir sonraki numune seti için adım 10.1'deki gibi sistemi kalibre edin.
11. Biyoinformatik veri analizi
NOT: Yaklaşık süre: Deneysel tasarıma bağlıdır.
- Tüm örnek verileri (ör. .raw dosyaları) uygun bir ağ depolama alanına/bilgisayar sürücüsüne aktarın.
- Peptit spektral eşleşmeleri (PSM'ler) oluşturmak ve TMT raporlayıcı iyon sinyali yoğunluklarını çıkarmak için türe özgü bir protein referans veritabanına karşı uygun parametreler9'u kullanarak istenen veri analizi uygulamasını (örneğin, Proteome Discover, Mascot) kullanarak tüm fraksiyonları birlikte arayın. PSM'leri uygun kalite kontrol metriklerine göre filtreleyin ve daha önce açıklandığı gibi normalleştirilmiş, medyan log2 dönüştürülmüş TMT raporlayıcı iyon oranı bolluklarını küresel protein seviyesi bolluklarına toplayın 3,9.
- İstenilen diferansiyel analiz yazılımını kullanarak ilgili koşullardaki protein değişikliklerini karşılaştırın.
Sonuçlar
İki HGSOC ve iki OCCC hastasından elde edilen taze dondurulmuş doku ince kesitleri, bu entegre AI güdümlü doku ROI tanımlama, segmentasyon, LMD ve kantitatif proteomik analiz iş akışı kullanılarak analiz edildi (Şekil 1). Her tümör için temsili H&E boyalı doku kesitleri, kurul onaylı bir patolog tarafından gözden geçirildi; tümör hücreselliği% 70 ila% 99 arasında değişmiştir. Dokular PEN membran slaytları üzerine ince kesitli (Ek Dosya 2) ve kalibratör referansları (Ek Dosya 1) ile önceden kesilerek görüntü analiz yazılımında oluşturulan ek açıklamalardan ( Malzeme Tablosuna bakınız) konumsal oryantasyon verilerinin LMD yazılımındaki Kartezyen koordinat oryantasyonu ile entegrasyonunu sağlamıştır. H&E boyamasını takiben, doku ve kalibratörleri içeren PEN slaytlarının yüksek çözünürlüklü görüntüleri (20x) yakalandı.
Mikrograflardaki tümör ve stromal hücre popülasyonları, LMD tarafından seçici hasat için görüntü analiz yazılımı (Malzeme Tablosuna bakınız) ve tüm doku ince kesitini (örneğin, tüm tümör dokusu) temsil eden hasatlar kullanılarak bölümlere ayrılmıştır (Şekil 1). Tüm tümör dokusu koleksiyonları için ayrımcı olmayan ek açıklamalar, tüm doku bölümünün 500μm2'lik fayanslarla bölünmesiyle, PEN membran bütünlüğünü korumak ve LMD sırasında membranın kıvrılmasını önlemek için fayanslar arasında 40 μm'lik bir boşluk bırakılarak oluşturulmuştur. Histoloji tarafından çözümlenmiş LMD zenginleştirme slaytlarında, görüntü analiz yazılımındaki AI sınıflandırıcısı (Malzeme Tablosuna bakınız), boş cam slayt arka planı ile birlikte tümör ve stromal hücreler arasında ayrım yapmak üzere eğitildi. Temsili tümör, stroma ve boş cam bölgeleri manuel olarak vurgulandı ve sınıflandırıcı araç, bu ROI'leri tüm doku bölümü boyunca segmentlere ayırmak için kullanıldı. Tüm doku, tümör epiteli ve stromayı temsil eden parçalanmış katmanlar, ayrı ayrı .ek açıklama dosyaları olarak ayrı ayrı kaydedildi (Ek Dosya 3 ve Ek Dosya 6). Görüntü dosyasının ayrı bir kopyasında (bölümlenmiş ROI ek açıklamaları olmadan), üç referans kalibratörünün her birinin en orta ucundan kısa bir satır açıklama eklenmiş ve LMD ek açıklama katmanı dosyalarının her biriyle aynı dosya adı kullanılarak bir .annotasyon dosyası olarak kaydedilmiş, ancak sonuna "_calib" soneki eklenmiştir (Ek Dosya 4). Bu çizgiler, PEN membran kalibratörlerinin konumunu, görüntü analiz yazılımında çizilen ek açıklama şekli listesi verileriyle birlikte kaydetmek için kullanılmıştır.
Bu çalışma, https://github.com/GYNCOE/Mitchell.et.al.2022 mevcut olan bu AI odaklı LMD iş akışını desteklemek için Python'da "Malleator" ve "Dapọ" olmak üzere iki algoritma sunmaktadır. Malleator algoritması, eşleştirilmiş .annotasyon dosyalarından tüm bireysel ek açıklamalar (doku ROI ve kalibratörler) için belirli Kartezyen koordinatları ayıklar ve bunları tek bir Genişletilebilir Biçimlendirme Dili (XML) içe aktarma dosyasında birleştirir (Ek Dosya 5). Özellikle, Malleator algoritması tüm alt dizin klasörlerini aramak için bir üst klasördeki dizin adını giriş olarak kullanır ve .xml birleştirilmiş bir dosyaya sahip olmayan alt klasörler için .xml dosyaları oluşturur. Malleator algoritması, görüntü analiz yazılımındaki tüm ek açıklama katmanlarını ( Malzeme Tablosuna bakın) tek bir katmanda birleştirir ve tescilli .annotation dosya türü olarak kaydedilen AI tarafından oluşturulan şekil listesi verilerini LMD yazılımıyla uyumlu .xml biçime dönüştürür. Ek açıklama ve kalibratör dosyaları birleştirildikten sonra, algoritma tarafından oluşturulan .xml dosyası kaydedilir ve LMD yazılımına aktarılır. Ek açıklamaların hizalamasını manuel olarak ayarlamak için hafif ayarlamalar gereklidir, bu da lazer mikroskop üzerindeki slayt aşamasının dikey (z-düzlemi) konumunu kaydetmeye yarar. Dapọ algoritması özellikle LMD ile zenginleştirilmiş koleksiyonlar için kullanılır. Bölümlenmiş kutucuklar, görüntü analiz yazılımı tarafından tek tek ek açıklama katmanlarına otomatik olarak atanır. Dapọ algoritması, Sınıflandırıcı aracını kullanmadan önce tüm bölümlenmiş kutucukları tek bir ek açıklama katmanında birleştirir, böylece LMD ile zenginleştirilmiş koleksiyonlar için Sınıflandırıcı analizi çalışma süresini azaltır.
Tüm tümör ve LMD ile zenginleştirilmiş doku örnekleri sindirildi, TMT reaktifleri ile etiketlendi, multiplekleştirildi, çevrimdışı fraksiyone edildi ve daha önce tarif edildiği gibi kantitatif MS tabanlı proteomikler aracılığıyla analiz edildi9. Bu AI güdümlü iş akışı kullanılarak hasat edilen numuneler için ortalama peptid verimi (43-60 μg) ve geri kazanım (0.46-0.59 μg /mm2), önceki raporlar 9,10 ile karşılaştırılabilirdi. Tüm örneklerde toplam 5.971 protein birlikte ölçülmüştür (Ek Tablo S1). En değişken 100 proteini kullanan denetimsiz hiyerarşik kümeleme, HGSOC ve OCCC histotiplerinin LMD ile zenginleştirilmiş ve tüm tümör örneklerinden ayrılmasıyla sonuçlandı (Şekil 2A), daha önce tarif edilene benzer11. Buna karşılık, hem HGSOC hem de OCCC'den LMD ile zenginleştirilmiş stroma örnekleri, LMD ile zenginleştirilmiş tümör ve tüm tümör örneklerinden bağımsız olarak birlikte ve bağımsız olarak kümelenmiştir. 5.971 niceliklendirilmiş protein arasında 215'i, HGSOC ve OCCC örneklerinden (Ek Tablo S2) tüm tümör koleksiyonları arasında anlamlı olarak değişmiştir (LIMMA adj. p < 0.05). Bu değiştirilmiş proteinler, Hughes ve ark.11 tarafından HGSOC ve OCCC tümör dokusunu ayırt etmek için tanımlananlarla karşılaştırılmıştır. Hughes ve ark. tarafından nicelleştirilen 76 imza proteininden 57'si bu veri kümesinde birlikte nicelleştirildi ve yüksek oranda korelasyona sahipti (Spearman Rho = 0.644, p < 0.001) (Şekil 2B).
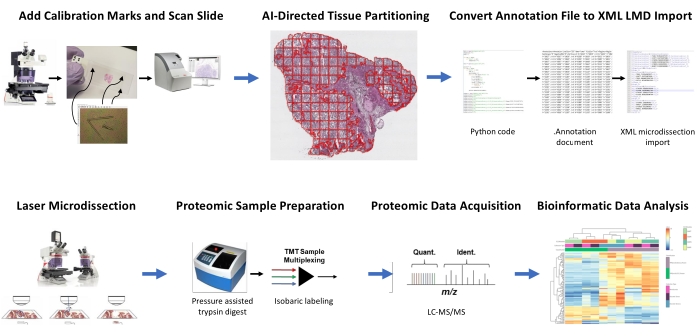
Şekil 1: Aşağı akış kantitatif proteomikleri için lazer mikrodiseksiyonu için otomatik doku bölgesi seçimi için entegre iş akışının özeti. Kalibrasyon referansları, görüntü analiz yazılımı HALO'da yapay zeka kaynaklı doku yatırım getirisi segmentlerinden gelen konumsal oryantasyon verilerini LMD mikroskobunda yatay konumlandırma ile birlikte kaydetmek için PEN membran kızaklarına kesilir. Malleator algoritması, bir slayt için tüm ek açıklama katmanlarındaki ek açıklamalı segmentasyon verilerini _calib referans dosyasıyla birleştirmek ve LMD yazılımıyla uyumlu bir .xml dosyasına dönüştürmek için kullanılır. Proteomik analiz için LMD ile hasat edilen doku, daha önce tarif edildiği gibi yüksek verimli kantitatif proteomikler tarafından sindirilir ve analizedilir 9. Kısaltmalar: LMD = lazer mikrodiseksiyon; ROI = ilgilenilen bölge; TMT = tandem kütle etiketi; Nicelik. = nicelik; Ident. = tanımlama; LC-MS/MS = sıvı kromatografisi-tandem kütle spektrometrisi. Bu şeklin daha büyük bir versiyonunu görüntülemek için lütfen buraya tıklayın.
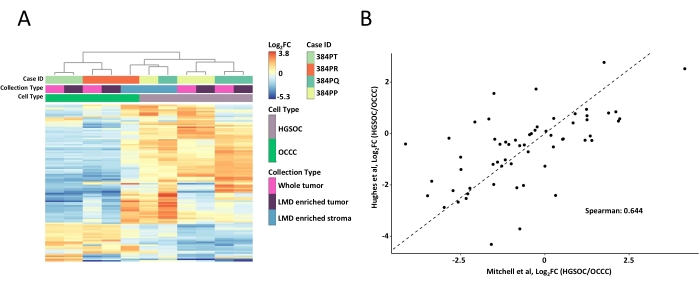
Şekil 2: LMD ile zenginleştirilmiş ve tüm tümör örneklerinde proteinlerin analizi. (A) HGSOC ve OCCC LMD ile zenginleştirilmiş ve tüm tümör örneklerinde en değişken olarak bol bulunan 100 proteinin denetimsiz hiyerarşik küme analizi. (B) Bu çalışmada HGSOC ve OCCC tüm tümör hasatları arasındaki log2 kat değişimli protein bolluklarının korelasyonu (Mitchell ve ark., x ekseni) ve Hughes ve ark. (y ekseni) 11 tarafından yapılan benzer bir çalışma. Kısaltmalar: LMD = lazer mikrodiseksiyon; HGSOC = yüksek dereceli seröz yumurtalık kanseri; OCCC = yumurtalık berrak hücreli karsinom; log 2 FC = log2-dönüştürülmüşproteomik bolluk. Bu şeklin daha büyük bir versiyonunu görüntülemek için lütfen buraya tıklayın.
Ek Tablo S1: HGSOC ve OCCC doku örneklerinden elde edilen tüm LMD ile zenginleştirilmiş ve tüm tümör örneklerinde birlikte nicelleştirilmiş 5.971 proteinin bolluğu. Kısaltmalar: LMD = lazer mikrodiseksiyon; HGSOC = yüksek dereceli seröz yumurtalık kanseri; OCCC = yumurtalık berrak hücreli karsinom. Bu tabloyu indirmek için lütfen tıklayınız.
Ek Tablo S2: HGSOC vs OCCC'den (LIMMA adj. p < 0.05) tüm tümör koleksiyonlarında diferansiyel olarak eksprese edilen proteinler (215). Kısaltmalar: HGSOC = yüksek dereceli seröz yumurtalık kanseri; OCCC = yumurtalık berrak hücreli karsinom. Bu tabloyu indirmek için lütfen tıklayınız.
Ek Dosya 1: Dört slayt konumu için standart kalibratör referanslarını içeren temsili şekil listesi verileri (.sld) dosyası. Dosya LMD yazılımına aktarılabilir. Bu Dosyayı indirmek için lütfen tıklayınız.
Ek Dosya 2: H&E boyalı yüksek çözünürlüklü (20x) doku bölümünün temsili .svs görüntü dosyası. Dosya, görüntü analiz yazılımı veya LMD yazılımı kullanılarak açılabilir ve görüntülenebilir. Kısaltma: H & E = hematoksilin ve eozin; LMD = lazer mikrodiseksiyonu. Bu Dosyayı indirmek için lütfen tıklayınız.
Ek Dosya 3: Bölümlenmiş tüm tümör segmentlerinin temsili .ek açıklama dosyası. Dosya görüntü analiz yazılımına aktarılabilir. Bu Dosyayı indirmek için lütfen tıklayınız.
Ek Dosya 4: Kalibratör referans segmentlerinin temsili _calib.annotasyon dosyası. Koordinat bilgileri, her bir ok ucu referansından çizilen kısa kalibratör çizgilerinin oryantal konumlandırılmasını temsil eder. Dosya görüntü analiz yazılımına aktarılabilir. Bu Dosyayı indirmek için lütfen tıklayınız.
Ek Dosya 5: Malleator algoritması tarafından oluşturulan temsili genişletilebilir biçimlendirme dili (.xml) dosyası. Dosya lazer mikrodiseksiyon yazılımına aktarılabilir. Bu Dosyayı indirmek için lütfen tıklayınız.
Ek Dosya 6: LMD ile zenginleştirilmiş koleksiyonlar için bölümlenmiş AI sınıfı segmentlerin temsili .annotasyon dosyası. Dosya görüntü analiz yazılımına aktarılabilir. Kısaltmalar: AI = yapay zeka; LMD = lazer mikrodiseksiyonu. Bu Dosyayı indirmek için lütfen tıklayınız.
Tartışmalar
9,12,13,14,15işleme sırasında numune kalitesini korumak için FFPE ve/veya taze dondurulmuş dokulardan hedef hücresel alt popülasyonların zenginleştirilmesi için iş akışları ve metodolojileri geliştirmeyi ve/veya iyileştirmeyi amaçlayan çok sayıda çalışma emsali bulunmakla birlikte, değişkenliği azaltmak için moleküler analizler için klinik doku örnekleri hazırlamak üzere otomatik stratejiler geliştirmek için önemli bir ihtiyaç vardır. tekrarlanabilirliği artırmak. Bu iş akışı, klinik doku örneklerinden LMD tarafından ayrık hücre popülasyonlarının histoloji tarafından çözümlenmiş hasadı için mevcut görüntü analizi yazılım araçlarını (Malzeme Tablosuna bakınız) entegre eden standartlaştırılmış, yarı otomatik bir protokolü tanımlar.
Ayrık hücre popülasyonlarını yakalayan ROI'ların mekansal olarak çözümlenmiş LMD zenginleştirmesi, moleküler karakterizasyonu ve tanımlamayı iyileştirmek ve hücre seçici biyobelirteç keşfini kolaylaştırmak için multiomik analizlerden önce yeni nesil bir doku işleme adımını temsil eder. Bu protokol, doku kesitlerinin bir histolog tarafından ROI'nin manuel olarak bölümlendirilmesiyle ilişkili ortam ortamına sıklıkla uzun süre maruz kalmasını azaltarak mevcut metodolojileri geliştirir (LMD toplanmasından önce >1-2 saat sürebilir). Bunun yerine bu iş akışı, YG'nin yapay zeka destekli sınıflandırma ve segmentasyon ile önceden tanımlanmasına olanak tanır. Doku bekleme süresinin sınırlandırılması, fosfopeptitler ve mRNA gibi son derece kararsız moleküler hedeflerin değerlendirilmesinde veya bir hedef proteinin tespit için doğal konformasyonunda olmasına dayanan antikor tabanlı analitik tekniklerdeki sahte varyasyonları azaltacaktır.
Taranan slayt görüntüsünde açıkça görülebilen PEN membran slaydı üzerine düzgün kalibratör referanslarının kesilmesi, görüntü analiz yazılımının ( Malzeme Tablosuna bakın) LMD iş akışıyla entegrasyonunu sağlayan temel bileşenlerden biridir. Kalibratörlerin "V" şeklinin altında hassas ("temiz") bir noktaya sahip olmasını sağlamak, 5.1.6 ve 5.2.13 numaralı adımlarda açıklandığı gibi, çizilecek kalibratör hatları için görüntü analiz yazılımında hassas bir noktanın seçilmesine olanak tanır. LMD yazılımına içe aktarma sırasında bu noktaların hizalanması, ek açıklamaların ("Malleator" ve/veya "Dapọ" algoritmaları kullanılarak uyumlu bir .xml dosyasının oluşturulmasıyla kolaylaştırılır) fiziksel LMD slaytındaki ilgili doku yatırım getirisine düzgün bir şekilde bindirilmesi için kritik öneme sahiptir. Lazer mikroskopta slayt aşamasının dikey (z-düzlemi) konumunu kaydetmek için LMD yazılımına içe aktarma sırasında hizalama hassas olsa bile tüm şekilleri vurgulamak ve toplu olarak "sürükleyip bırakmak" gerekir. Ek açıklamaların doku yatırım getirisi üzerindeki konumlandırılmasında küçük ayarlamalar da gerekirse bu adımda yapılabilir.
Malleator algoritmasının mevcut sürümünün bir sınırlaması, görüntü analiz yazılımı tarafından sağlanan önceden tanımlanmış ek açıklama şekli araçlarıyla uyumlu olmamasıdır ( bkz. Malzeme Tablosu), ancak algoritmanın gelecekteki güncellemeleri / sürümleri bu uyumluluğu geliştirmeyi hedefleyecektir. Bu araçlar kullanılarak çizilen şekiller için .annotation dosyası, bu noktaların etrafında tam uzamsal yönlendirme olmadan, her bir detaylandırma için yalnızca iki eşleştirilmiş x ve y koordinat kümesi içerir. Bu araçların geçerli kullanımı, ek açıklamaların içe aktarma işlemi sırasında yalnızca iki nokta tarafından tanımlanan düz çizgilere dönüştürülmesine neden olur. XML formatına başarılı bir şekilde dönüştürme ve LMD içe aktarma için doku yatırım getirisi segmentlerinin manuel olarak tanımlanması gerekir. Bu, hedef alana özgü bağımsız serbest el poligonal ek açıklamalarıyla her bir yatırım getirisini manuel olarak tanımlayarak veya istenirse tüm doku yatırım getirisi segmentlerine yaklaşık dairesel veya dikdörtgen bir ek açıklama uygulayarak gerçekleştirilebilir ve bu iş akışıyla uyumlu olacaktır.
Burada sunulan iş akışı, taze dondurulmuş insan kanser dokusu örneklerinin proteomik analizi için gösterilmiş olsa da, bu AI güdümlü LMD iş akışı, FFPE dokuları, kanserli olmayan doku tipleri ve insan dışı kaynaklardan gelenlerle eşdeğer olarak kullanılabilir. Ayrıca, transkriptomik, genomik veya fosfoproteomik analizler dahil olmak üzere diğer aşağı akış moleküler profil oluşturma iş akışlarını da destekleyebilir. Bu iş akışı ayrıca, hücre sayımı veya "Multiplex IHC" modülü veya "Doku Mikrodizisi (TMA) Eklentisi" dahil olmak üzere diğer analitik modüllerle ilişkili yetenekler de dahil olmak üzere görüntü analiz yazılımının diğer kullanımlarından da yararlanabilir ( Malzeme Tablosuna bakınız). Bu iş akışının gelecekteki uygulamaları, ROI segmenti başına hücre sayısını önceden tanımlamaktan ve böylece birden fazla koleksiyonda eşdeğer hücresel girdiler sağlamaktan veya immünohistokimya veya hücre sosyolojisi gibi ilgilenilen hücresel YG'leri tanımlamak için alternatif yöntemler kullanmaktan da yararlanabilir.
Açıklamalar
T.P.C. ThermoFisher Scientific, Inc SAB üyesidir ve AbbVie'den araştırma fonu almaktadır.
Teşekkürler
Bu projenin finansmanı kısmen Savunma Sağlık Programı (HU0001-16-2-0006 ve HU0001-16-2-00014) tarafından Jinekolojik Kanser Mükemmeliyet Merkezi için Üniformalı Hizmetler Üniversitesi'ne sağlanmıştır. Sponsorların çalışmanın tasarımı, yürütülmesi, yorumlanması veya yazılmasında hiçbir rolü yoktu. Feragatname: Burada ifade edilen görüşler yazarların görüşleridir ve Ordu / Deniz / Hava Kuvvetleri Bakanlığı, Savunma Bakanlığı veya ABD Hükümeti'nin resmi politikasını yansıtmamaktadır.
Malzemeler
| Name | Company | Catalog Number | Comments |
| 1260 Infinity II System | Agilent Technologies Inc | Offline LC system | |
| 96 MicroCaps (150uL) in bulk | Pressure Biosciences Inc | MC150-96 | |
| 96 MicroPestles in bulk | Pressure Biosciences Inc | MP-96 | |
| 96 MicroTubes in bulk (no caps) | Pressure Biosciences Inc | MT-96 | |
| 9mm MS Certified Clear Screw Thread Kits | Fisher Scientific | 03-060-058 | Sample vial for offline LC frationation and mass spectrometry |
| Acetonitrile, Optima LC/MS Grade | Fisher Chemical | A995-4 | Mobile phase solvent |
| Aperio AT2 | Leica Microsystems | 23AT2100 | Slide scanner |
| Axygen PCR Tubes with 0.5 mL Flat Cap | Fisher Scientific | 14-222-292 | Sample tubes; size fits PCT tubes and thermocycler |
| Barocycler 2320EXT | Pressure Biosciences Inc | 2320-EXT | Barocycler |
| BCA Protein Assay Kit | Fisher Scientific | P123225 | |
| cOmplete, Mini, EDTA-free Protease Inhibitor Cocktail | Roche | 11836170001 | |
| Easy-nLC 1200 | Thermo Fisher Scientific | Liquid Chromatography | |
| EasyPep Maxi Sample Prep Kit | Thermo Fisher Scientific | NCI5734 | Post-label sample clean up column |
| EASY-SPRAY C18 2UM 50CM X 75 | Fisher Scientific | ES903 | Analytical column |
| Eosin Y Solution Aqueous | Sigma Aldrich | HT110216 | |
| Formic Acid, 99+ % | Thermo Fisher Scientific | 28905 | Mobile phase additive |
| ggplot2 version 3.3.5 | CRAN | https://cran.r-project.org/web/packages/ggplot2/ | |
| HALO | Indica Labs | Image analysis software | |
| IDLE (Integrated Development and Learning Environment) | Python Software Foundation | ||
| iheatmapr version 0.5.1 | CRAN | https://cran.r-project.org/web/packages/iheatmapr/ | |
| iRT Kit | Biognosys | Ki-3002-1 | LC-MS QAQC Standard |
| limma version 3.42.2 | Bioconductor | https://bioconductor.org/packages/release/bioc/html/limma.html | |
| LMD Scanning stage Ultra LMT350 | Leica Microsystems | 11888453 | LMD stage model outfitted with PCT tube holder |
| LMD7 (software version 8.2.3.7603) | Leica Microsystems | LMD apparatus (microscope, laser, camera, PC, tablet) | |
| Mascot Server | Matrix Science | Data analysis software | |
| Mass Spec-Compatible Human Protein Extract, Digest | Promega | V6951 | LC-MS QAQC Standard |
| Mayer’s Hematoxylin Solution | Sigma Aldrich | MHS32 | |
| PEN Membrane Glass Slides | Leica Microsystems | 11532918 | |
| Peptide Retention Time Calibration Mixture | Thermo Fisher Scientific | 88321 | LC-MS QAQC Standard |
| Phosphatase Inhibitor Cocktail 2 | Sigma Aldrich | P5726 | |
| Phosphatase Inhibitor Cocktail 3 | Sigma Aldrich | P0044 | |
| Pierce LTQ Velos ESI Positive Ion Calibration Solution | Thermo Fisher Scientific | 88323 | Instrument calibration solution |
| PM100 C18 3UM 75UMX20MM NV 2PK | Fisher Scientific | 164535 | Pre-column |
| Proteome Discoverer | Thermo Fisher Scientific | OPTON-31040 | Data analysis software |
| Python | Python Software Foundation | ||
| Q Exactive HF-X | Thermo Fisher Scientific | Mass spectrometer | |
| R version 3.6.0 | CRAN | https://cran-archive.r-project.org/bin/windows/base/old/2.6.2/ | |
| RColorBrewer version 1.1-2 | CRAN | https://cran.r-project.org/web/packages/RColorBrewer/ | |
| Soluble Smart Digest Kit | Thermo Fisher Scientific | 3251711 | Digestion reagent |
| TMTpro 16plex Label Reagent Set | Thermo Fisher Scientific | A44520 | isobaric TMT labeling reagents |
| Veriti 60 well thermal cycler | Applied Biosystems | 4384638 | Thermocycler |
| Water, Optima LC/MS Grade | Fisher Chemical | W6-4 | Mobile phase solvent |
| ZORBAX Extend 300 C18, 2.1 x 12.5 mm, 5 µm, guard cartridge (ZGC) | Agilent Technologies Inc | 821125-932 | Offline LC trap column |
| ZORBAX Extend 300 C18, 2.1 x 150 mm, 3.5 µm | Agilent Technologies Inc | 763750-902 | Offline LC analytical column |
Referanslar
- Motohara, T., et al. An evolving story of the metastatic voyage of ovarian cancer cells: cellular and molecular orchestration of the adipose-rich metastatic microenvironment. Oncogene. 38 (16), 2885-2898 (2019).
- Aran, D., Sirota, M., Butte, A. J. Systematic pan-cancer analysis of tumour purity. Nature Communications. 6, 8971 (2015).
- Hunt, A. L., et al. Extensive three-dimensional intratumor proteomic heterogeneity revealed by multiregion sampling in high-grade serous ovarian tumor specimens. iScience. 24 (7), 102757 (2021).
- Dou, Y., et al. Proteogenomic characterization of endometrial carcinoma. Cell. 180 (4), 729-748 (2020).
- Zhang, H., et al. Integrated proteogenomic characterization of human high-grade serous ovarian cancer. Cell. 166 (3), 755-765 (2016).
- Gillette, M. A., et al. Proteogenomic characterization reveals therapeutic vulnerabilities in lung adenocarcinoma. Cell. 182 (1), 200-225 (2020).
- Silvestri, A., et al. Protein pathway biomarker analysis of human cancer reveals requirement for upfront cellular-enrichment processing. Laboratory Investigation. 90 (5), 787-796 (2010).
- Echle, A., et al. Deep learning in cancer pathology: a new generation of clinical biomarkers. British Journal of Cancer. 124 (4), 686-696 (2021).
- Lee, S., et al. Molecular analysis of clinically defined subsets of high-grade serous ovarian cancer. Cell Reports. 31 (2), 107502 (2020).
- Xuan, Y., et al. Standardization and harmonization of distributed multi-center proteotype analysis supporting precision medicine studies. Nature Communications. 11 (1), 5248 (2020).
- Hughes, C. S., et al. Quantitative profiling of single formalin fixed tumour sections: proteomics for translational research. Scientific Reports. 6 (1), 34949 (2016).
- Espina, V., et al. A portrait of tissue phosphoprotein stability in the clinical tissue procurement process. Molecular & Cellular Proteomics. 7 (10), 1998-2018 (2008).
- Espina, V., Heiby, M., Pierobon, M., Liotta, L. A. Laser capture microdissection technology. Expert Review of Molecular Diagnostics. 7 (5), 647-657 (2007).
- Havnar, C. A., et al. Automated dissection protocol for tumor enrichment in low tumor content tissues. Journal of Visualized Experiments. (169), e62394 (2021).
- Mueller, C., et al. One-step preservation of phosphoproteins and tissue morphology at room temperature for diagnostic and research specimens. PLoS One. 6 (8), (2011).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır