A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
أتمتة مقايسة النواة الدقيقة باستخدام قياس التدفق الخلوي للتصوير والذكاء الاصطناعي
In This Article
Summary
اختبار النواة الدقيقة (MN) هو اختبار راسخ لقياس تلف الحمض النووي. ومع ذلك ، فإن تسجيل الفحص باستخدام التقنيات التقليدية مثل الفحص المجهري اليدوي أو تحليل الصور المستند إلى الميزات أمر شاق وصعب. تصف هذه الورقة منهجية تطوير نموذج ذكاء اصطناعي لتسجيل مقايسة MN باستخدام بيانات قياس التدفق الخلوي للتصوير.
Abstract
يتم استخدام اختبار النواة الدقيقة (MN) في جميع أنحاء العالم من قبل الهيئات التنظيمية لتقييم المواد الكيميائية للسمية الجينية. يمكن إجراء الفحص بطريقتين: عن طريق تسجيل MN في الخلايا ثنائية النواة المقسمة مرة واحدة أو المسدودة بالسيتوكينات أو الخلايا أحادية النوى المقسمة بالكامل. تاريخيا ، كان الفحص المجهري الضوئي هو الطريقة القياسية الذهبية لتسجيل الفحص ، لكنه شاق وغير موضوعي. تم استخدام قياس التدفق الخلوي في السنوات الأخيرة لتسجيل الفحص ، ولكنه محدود بسبب عدم القدرة على تأكيد الجوانب الرئيسية للصور الخلوية بصريا. يجمع قياس التدفق الخلوي التصويري (IFC) بين التقاط الصور عالية الإنتاجية وتحليل الصور الآلي ، وقد تم تطبيقه بنجاح للحصول بسرعة على صور لجميع الأحداث الرئيسية وتسجيلها في اختبار MN. في الآونة الأخيرة ، ثبت أنه يمكن استخدام طرق الذكاء الاصطناعي (الذكاء الاصطناعي) القائمة على الشبكات العصبية التلافيفية لتسجيل بيانات مقايسة MN التي حصلت عليها مؤسسة التمويل الدولية. تصف هذه الورقة جميع خطوات استخدام الذكاء الاصطناعي البرنامج لإنشاء نموذج تعلم عميق لتسجيل جميع الأحداث الرئيسية وتطبيق هذا النموذج لتسجيل بيانات إضافية تلقائيا. تقارن نتائج نموذج التعلم العميق الذكاء الاصطناعي بشكل جيد بالفحص المجهري اليدوي ، وبالتالي تمكين التسجيل الآلي بالكامل لمقايسة MN من خلال الجمع بين مؤسسة التمويل الدولية و الذكاء الاصطناعي.
Introduction
يعد فحص النواة الدقيقة (MN) أمرا أساسيا في علم السموم الوراثية لتقييم تلف الحمض النووي في تطوير مستحضرات التجميل والمستحضرات الصيدلانية والمواد الكيميائية للاستخدام البشري1،2،3،4. تتكون النوى الدقيقة من كروموسومات كاملة أو شظايا كروموسوم لا تندمج في النواة بعد الانقسام وتتكثف في أجسام دائرية صغيرة منفصلة عن النواة. وبالتالي ، يمكن استخدام MN كنقطة نهاية لتحديد تلف الحمض النووي في اختبار السمية الجينية1.
الطريقة المفضلة لقياس MN هي داخل الخلايا ثنائية النواة المقسمة مرة واحدة (BNCs) عن طريق منع الانقسام باستخدام Cytochalasin-B (Cyt-B). في هذا الإصدار من الفحص ، يتم تقييم السمية الخلوية أيضا عن طريق تسجيل الخلايا أحادية النواة (MONO) ومتعددة النوى (POLY). يمكن أيضا إجراء الفحص عن طريق تسجيل MN في خلايا أحادية اللون غير محظورة ، وهو أسرع وأسهل في التسجيل ، مع تقييم السمية الخلوية باستخدام عدد الخلايا قبل وبعد التعرض لتقييم الانتشار 5,6.
تم إجراء التسجيل المادي للفحص تاريخيا من خلال الفحص المجهري اليدوي ، لأن هذا يسمح بتأكيد مرئي لجميع الأحداث الرئيسية. ومع ذلك ، فإن الفحص المجهري اليدوي يمثل تحديا وشخصيا1. وهكذا ، تم تطوير تقنيات آلية ، بما في ذلك مسح الشرائح المجهرية وقياس التدفق الخلوي ، ولكل منها مزاياها وقيودها. بينما تسمح طرق مسح الشرائح بتصور الأحداث الرئيسية ، يجب إنشاء الشرائح بكثافة الخلية المثلى ، والتي قد يكون من الصعب تحقيقها. بالإضافة إلى ذلك ، غالبا ما تفتقر هذه التقنية إلى التصور السيتوبلازمي ، والذي يمكن أن يضر بتسجيل الخلايا أحادية اللونو POLY 7,8. بينما يوفر قياس التدفق الخلوي التقاط بيانات عالية الإنتاجية ، يجب تحليل الخلايا ، وبالتالي عدم السماح باستخدام شكل Cyt-B للفحص. بالإضافة إلى ذلك ، كتقنية غير تصويرية ، لا يوفر قياس التدفق الخلوي التقليدي التحقق البصري من الأحداث الرئيسية 9,10.
لذلك ، تم التحقيق في قياس التدفق الخلوي التصويري (IFC) لإجراء فحص MN. تجمع ImageStreamX Mk II بين السرعة والمتانة الإحصائية لقياس التدفق الخلوي التقليدي مع إمكانات التصوير عالية الدقة للفحص المجهري في نظام واحد11. لقد ثبت أنه باستخدام مؤسسة التمويل الدولية ، يمكن التقاط صور عالية الدقة لجميع الأحداث الرئيسية وتسجيلها تلقائيا باستخدام تقنيات 12,13 أو الذكاء الاصطناعي (الذكاء الاصطناعي)القائمة على الميزات 14,15. باستخدام IFC لإجراء مقايسة MN ، يمكن تحقيق التسجيل التلقائي للعديد من الخلايا مقارنة بالفحص المجهري في فترة زمنية أقصر.
ينحرف هذا العمل عن سير عمل تحليل الصور الموصوف سابقا16 ويناقش جميع الخطوات المطلوبة لتطوير وتدريب نموذج غابة عشوائية (RF) و / أو شبكة عصبية تلافيفية (CNN) باستخدام برنامج Amnis الذكاء الاصطناعي (يشار إليه من الآن فصاعدا باسم "برنامج الذكاء الاصطناعي"). يتم وصف جميع الخطوات اللازمة ، بما في ذلك ملء بيانات الحقيقة الأرضية باستخدام أدوات وضع العلامات بمساعدة الذكاء الاصطناعي ، وتفسير نتائج تدريب النموذج ، وتطبيق النموذج لتصنيف البيانات الإضافية ، مما يسمح بحساب السمية الجينية والسمية الخلوية15.
Protocol
1. الحصول على البيانات باستخدام قياس التدفق الخلوي للتصوير
ملاحظة: يرجى الرجوع إلى Rodrigues et al.16 مع التعديلات التالية، مع ملاحظة أن مناطق الاقتناء التي تستخدم مؤسسة التمويل الدولية قد تحتاج إلى تعديل من أجل التقاط الصور على النحو الأمثل:
- بالنسبة للطريقة غير Cyt-B ، قم بإجراء عدد الخلايا باستخدام عداد خلايا متاح تجاريا باتباع تعليمات الشركة المصنعة (انظر جدول المواد) على كل مزرعة مباشرة قبل الاستزراع وبعد فترة الاسترداد مباشرة.
- في حالة تشغيل عينات على مقياس تدفق تدفق التصوير بكاميرا واحدة ، ضع Brightfield (BF) في القناة 4. يستعاض عن M01 ب M04 و M07 ب M01.
ملاحظة: يشير الحرف "M" إلى قناة الكاميرا على مؤسسة التمويل الدولية. - استخدم التكبير 40x أثناء الاستحواذ.
- في مخطط نسبة العرض إلى الارتفاع لمنطقة BF مقابل BF أثناء الاستحواذ ، استخدم إحداثيات المنطقة التالية:
إحداثيات X: 100 و 900 ؛ إحداثيات Y: 0.7 و 1 (طريقة Cyt-B)
إحداثيات X: 100 و 600 ؛ إحداثيات Y: 0.7 و 1 - في مخطط شدة Hoechst ، استخدم إحداثيات المنطقة التالية:
إحداثيات X: 55 و 75 ؛ إحداثيات Y: 9.5 و 15 (طريقة Cyt-B)
إحداثيات X: 55 و 75 ؛ إحداثيات Y: 13 و 21 (طريقة غير Cyt-B) - لإزالة صور الكائنات المبرمج والميتة من البيانات ، قم بتشغيل حزمة برامج IDEAS 6.3 (يشار إليها من الآن فصاعدا باسم "برنامج تحليل الصور" ؛ انظر جدول المواد).
ملاحظة: تم تصميم برنامج الذكاء الاصطناعي للعمل مع ملفات .daf التي تمت معالجتها باستخدام أحدث إصدار من برنامج تحليل الصور. تأكد من تحديث برنامج تحليل الصور. - احفظ هذا العمل كملف قالب (.ast).
2. إنشاء ملفات .daf لجميع ملفات .rif
- يسمح برنامج الذكاء الاصطناعي فقط باستيراد ملفات .daf. قم بإنشاء ملفات .daf لجميع ملفات .rif في التجربة من خلال المعالجة المجمعة.
- ضمن قائمة الأدوات ، انقر فوق ملفات البيانات المجمعة ثم انقر فوق إضافة دفعة.
- في النافذة الجديدة ، حدد إضافة ملفات وحدد ملفات .rif المراد إضافتها إلى الدفعة. ضمن الخيار تحديد قالب أو ملف تحليل بيانات (.ast، .daf)، حدد ملف .ast الذي تم إنشاؤه مسبقا.
- قم بتعيين اسم دفعة إذا لزم الأمر وانقر فوق "موافق " لإنشاء ملفات .daf لجميع ملفات .rif المحملة.
3. إنشاء تجربة في برنامج الذكاء الاصطناعي
- ارجع إلى المخطط الانسيابي في الشكل 1 الذي يصف عملية إنشاء نموذج التعلم العميق باستخدام برنامج الذكاء الاصطناعي.
- قم بتشغيل برنامج الذكاء الاصطناعي وتأكد من تثبيت أحدث إصدار بالنقر فوق حول في الركن الأيسر السفلي من النافذة. إذا لم يتم تثبيت أحدث إصدار، فاتصل support@luminexcorp.com للحصول عليه.
- الشاشة الافتراضية في البرنامج هي شاشة التجربة الجديدة . استخدم رمز المجلد لاختيار مكان حفظ التجربة، واكتب اسما للتجربة (على سبيل المثال، "نموذج MN").
- ضمن نوع التجربة، انقر فوق زر الاختيار الموجود بجانب تدريب لبدء تجربة تدريبية لبدء إنشاء نموذج CNN. انقر فوق التالي.
- اختياري: إذا تم تدريب نموذج مسبقا، فيمكن استخدامه كقالب لنموذج الذكاء الاصطناعي جديد، ويمكن تحديده كقالب لإنشاء نموذج جديد من شاشة تحديد نموذج قالب . في حالة عدم وجود نموذج قالب ، ما عليك سوى تخطي هذه الخطوة بالنقر فوق التالي.
- الشاشة التالية هي شاشة تحديد نموذج جديد . ضمن النموذج، سيتم ملء الاسم الذي تم إعطاؤه للنموذج في الخطوة 3.3 تلقائيا.
- ضمن الوصف، اكتب وصفا للنموذج (اختياري) واترك الحد الأقصى لحجم الصورة عند 150 بكسل.
- ضمن القنوات ، انقر فوق إضافة BF لإضافة قناة برايتفيلد إلى القائمة. ضمن الاسم ، انقر نقرا مزدوجا فوق Brightfield وأعد تسمية هذه القناة إلى BF. انقر فوق إضافة FL لإضافة قناة الفلورسنت إلى القائمة. ضمن الاسم ، انقر نقرا مزدوجا فوق الفلورسنت وأعد تسمية هذه القناة إلى DNA.
- ضمن أسماء الفئات ، انقر فوق إضافة. في النافذة المنبثقة ، اكتب أحادي النواة وانقر فوق موافق. يؤدي هذا إلى إضافة الفئة أحادية النواة إلى قائمة أسماء الفئات. كرر هذه العملية للتأكد من تعريف الفئات الست التالية في القائمة:
أحادي النواة
أحادي النواة مع MN
ثنائي النواة
ثنائي النواة مع MN
متعدد النوى
مورفولوجيا غير منتظمة
انقر فوق التالي.
ملاحظة: ستمثل فئات نماذج الحقيقة الأرضية الستة هذه الأحداث الرئيسية التي سيتم تسجيلها ، بالإضافة إلى الصور ذات التشكل التي تختلف عن معايير التسجيلالمقبولة 5.- اختياري: إذا رغبت في ذلك، يمكن تضمين قالب التحليل من 1.7 لاستخدام ميزات من برنامج تحليل الصور. إذا كنت ترغب في تضمين هذه الميزات في نموذج الذكاء الاصطناعي ، فاستعرض للوصول إلى ملف .ast ، ثم من القوائم المنسدلة الخاصة بالقناة ، اختر المجموعات الفرعية للميزات التي ترغب في تضمينها.
- ضمن تحديد الملفات ، انقر فوق إضافة ملفات واستعرض بحثا عن الملفات المطلوبة لإضافتها إلى برنامج الذكاء الاصطناعي لإنشاء بيانات الحقيقة الأساسية. انقر فوق التالي.
ملاحظة: من المهم إضافة ملفات بيانات متعددة (على سبيل المثال ، بيانات التحكم الإيجابية والسلبية) تحتوي على عدد كاف من جميع الأحداث الرئيسية.
- بعد ذلك، في شاشة تحديد مجموعات سكانية أساسية ، حدد موقع المحتوى غير المبرمج من التسلسل الهرمي للسكان. انقر بزر الماوس الأيمن فوق السكان غير المبرمجين وحدد تحديد جميع المجموعات السكانية المطابقة. انقر فوق التالي.
ملاحظة: من المهم استبعاد أي مجموعات سكانية لا ينبغي تصنيفها (على سبيل المثال ، الخرز ، الحطام ، الزوجي ، إلخ.) - هذه الشاشة هي شاشة تحديد مجموعات الحقيقة .
- إذا لم يتم إنشاء مجموعات الحقيقة الموسومة للأحداث الرئيسية في برنامج تحليل الصور ، فانقر فوق التالي.
- إذا تم إنشاء مجموعات الحقيقة الموسومة في برنامج تحليل الصور ، فقم بتعيينها إلى فئة النموذج المناسبة.
- لتعيين مجموعة حقيقة موسومة للخلايا أحادية اللون مع MN ، انقر فوق فئة Mononucleated with MN ضمن فئات النموذج على اليسار. ثم انقر فوق مجموعة الحقيقة المناسبة الموسومة على اليمين الذي يحتوي على هذه الأحداث.
- إذا تم إنشاء مجموعات معلومات الحقيقة ذات العلامات في أكثر من ملف بيانات واحد، فانقر بزر الماوس الأيمن فوق إحدى مجموعات معلومات الحقيقة وحدد تحديد كافة مجموعات التحقق المطابقة لإضافة مجموعات سكانية ذات علامات تمييز من ملفات متعددة إلى الفئة المناسبة.
- بمجرد تعيين جميع مجموعات الحقيقة المناسبة ، انقر فوق التالي.
- في شاشة تحديد القنوات ، اختر القنوات المناسبة للتجربة. هنا ، اضبط BF على القناة 1 و Hoechst على القناة 7. انقر بزر الماوس الأيمن فوق قناة وحدد تطبيق على الكل. انقر فوق التالي.
- أخيرا ، في شاشة التأكيد ، انقر فوق إنشاء تجربة.
- يقوم برنامج الذكاء الاصطناعي بتحميل الصور من ملفات البيانات وإنشاء فئات النموذج المحددة في الخطوة 3.5.3 مع صور الحقيقة الأرضية التي تم تعيينها في الخطوة 3.7. انقر فوق إنهاء.
- بمجرد إنشاء التجربة ، يتم تقديم خمسة خيارات:
التجربة: توفر تفاصيل التجربة، بما في ذلك ملفات البيانات التي تم تحميلها، والقنوات المختارة، وفئات نموذج الحقيقة الأساسية المحددة.
وضع العلامات: تشغيل أداة وضع العلامات التي يمكن للمستخدمين من خلالها ملء بيانات الحقيقة الأساسية.
التدريب: يدرب نموذجا يعتمد على بيانات الحقيقة على أرض الواقع.
تصنيف: يستخدم نماذج مدربة لتصنيف البيانات.
النتائج: توفر نتائج من كل من تجربة التدريب وتجربة التصنيف.
4. ملء بيانات الحقيقة الأساسية باستخدام أدوات وضع العلامات بمساعدة الذكاء الاصطناعي
- انقر فوق وضع العلامات لتشغيل واجهة أداة وضع العلامات.
- انقر فوق أدوات التكبير / التصغير (أيقونات العدسة المكبرة) لاقتصاص الصور لتسهيل عرضها.
- انقر فوق شريط التمرير لضبط حجم الصورة لتغيير عدد الصور المعروضة في المعرض.
- انقر فوق خيار إعداد العرض ، واختر Min-Max ، والذي يوفر أفضل صورة تباين لتحديد جميع الأحداث الرئيسية.
- انقر فوق إعداد عرض المعرض لتغيير لون صورة الحمض النووي إلى الأصفر أو الأبيض ، مما سيحسن تصور الأشياء الصغيرة (على سبيل المثال ، MN).
- انقر فوق Cluster لتشغيل الخوارزمية لتجميع الكائنات ذات التشكل المماثل معا. بمجرد اكتمال التجميع، يتم عرض المجموعات الفردية التي تحتوي على عدد الكائنات لكل مجموعة في قائمة ضمن مجموعات سكانية غير معروفة. حدد المجموعات الفردية لعرض الكائنات داخل الكتلة وقم بتعيين هذه الكائنات إلى فئات النموذج المناسبة لها.
- بعد تعيين ما لا يقل عن 25 كائنا لكل فئة نموذج، تصبح خوارزمية التنبؤ متاحة. انقر فوق توقع.
ملاحظة: تظل الكائنات التي لا تتناسب بشكل جيد مع أي مجموعة مصنفة على أنها غير معروفة. مع إضافة المزيد من الكائنات إلى مجموعات الحقيقة ، تتحسن دقة التنبؤ. - استمر في ملء فئات نموذج الحقيقة الأرضية بالصور المناسبة حتى يتم الوصول إلى عدد كاف من الكائنات في كل فئة.
- بمجرد تعيين ما لا يقل عن 100 كائن لكل فئة طراز ، انقر فوق علامة التبويب التدريب في الجزء العلوي من الشاشة. انقر فوق الزر Train لإنشاء نموذج باستخدام خوارزميات Random Forest و CNN.
ملاحظة: يقوم برنامج الذكاء الاصطناعي بإنشاء نماذج باستخدام كل من خوارزميات Random Forest و CNN ، وتسمح مربعات الاختيار بإنشاء نماذج باستخدام خوارزميات Random Forest of CNN فقط.
5. تقييم دقة النموذج
- بمجرد اكتمال تدريب النموذج ، انقر فوق عرض النتائج.
- استخدم شاشة النتائج لتقييم دقة النموذج. استخدم القائمة المنسدلة للتبديل بين Random Forest و CNN.
ملاحظة: يمكن تحديث مجموعات الحقيقة ، ويمكن إعادة تدريب النموذج أو استخدامه كما هو لتصنيف البيانات الإضافية.- لتحديث مجموعات الحقيقة ، انقر فوق وضع العلامات في الأعلى واتبع القسم 4.
6. تصنيف البيانات باستخدام النموذج
- قم بتشغيل برنامج الذكاء الاصطناعي. الشاشة الافتراضية هي شاشة تجربة جديدة . استخدم أيقونة المجلد لاختيار مكان حفظ التجربة واكتب اسما للتجربة.
- ضمن نوع التجربة، انقر فوق زر الاختيار بجانب تصنيف لبدء تجربة تصنيف . انقر فوق التالي.
- انقر فوق النموذج المراد استخدامه للتصنيف ، ثم انقر فوق التالي.
- في شاشة تحديد الملفات ، انقر فوق إضافة ملفات واستعرض الملفات المراد تصنيفها حسب نموذج CNN. انقر فوق التالي.
- بعد ذلك ، في شاشة تحديد السكان الأساسيين ، انقر فوق مربع الاختيار بجوار السكان غير المبرمجين في أحد الملفات المحملة. انقر بزر الماوس الأيمن فوق السكان غير المبرمجين وانقر فوق تحديد كافة المجموعات السكانية المطابقة لتحديد هذه المجموعة من جميع الملفات المحملة. انقر فوق التالي.
- اختياري: إذا كانت البيانات المراد تصنيفها تحتوي على مجموعات الحقيقة، فيمكن تعيينها إلى فئات النموذج المناسبة على شاشة تحديد مجموعات الحقيقة. وإلا، فانقر فوق التالي لتخطي هذه الخطوة.
- في شاشة تحديد القنوات ، اختر القناة 1 ل brightfield والقناة 7 لصبغة الحمض النووي. انقر بزر الماوس الأيمن فوق قناة وانقر فوق تطبيق على الكل. ثم انقر فوق التالي.
- أخيرا ، في شاشة التأكيد ، انقر فوق إنشاء تجربة. يقوم برنامج الذكاء الاصطناعي بتحميل النموذج المحدد وجميع الصور من ملفات البيانات المختارة. انقر فوق إنهاء.
- انقر فوق تصنيف لتشغيل شاشة التصنيف. انقر فوق الزر تصنيف . يبدأ هذا عملية استخدام نموذج RF و CNN لتصنيف البيانات الإضافية وتحديد جميع الكائنات التي تنتمي إلى فئات النموذج المحددة.
ملاحظة: يمكن استخدام خانات الاختيار لتحديد طراز RF و / أو طراز CNN. - بمجرد اكتمال التصنيف ، انقر فوق عرض النتائج.
- انقر فوق الزر تحديث DAFs لإظهار نافذة تحديث DAFs مع نتائج التصنيف. انقر فوق "موافق" لتحديث ملفات .daf.
7. إنشاء تقرير بنتائج التصنيف
- في شاشة النتائج ، انقر فوق إنشاء تقرير. حدد خانة الاختيار الموجودة بجانب إنشاء تقرير لكل DAF إدخال إذا كان هناك حاجة إلى تقرير فردي لكل DAF إدخال . انقر فوق موافق.
- بمجرد الانتهاء ، افتح المجلد حيث تم حفظ ملفات التقرير. داخل المجلد، يوجد تقرير .pdf التجربة ومجلد الموارد .
- افتح .pdf لعرض التقرير. يحتوي التقرير على معلومات النموذج والتجربة ، وقائمة ملفات الإدخال .daf ، وعدد الفئات والنسب المئوية للفئة بتنسيق جدولي ومدرج تكراري ، ومصفوفة ارتباك تلخص متوسط احتمال التنبؤ عبر جميع ملفات الإدخال .daf.
- افتح مجلد الموارد ثم مجلد CNN . يوجد داخل هذا المجلد ملفات .png لعدد الفئات والرسوم البيانية الشريطية للنسبة المئوية ، بالإضافة إلى مصفوفة الارتباك. بالإضافة إلى ذلك ، هناك ملفات .csv تحتوي على أعداد الفئات والنسب المئوية لكل ملف إدخال.
8. تحديد تردد MN والسمية الخلوية
- حساب تردد MN
- طريقة غير Cyt-B: لتحديد تردد MN افتح ملف class_count.csv من الخطوة 7.4. لكل ملف إدخال ، قسم الأعداد في مجتمع "أحادي النواة مع MN" على الأعداد في المحتوى "أحادي النوى" واضربه في 100:

- طريقة Cyt-B: لتحديد تردد MN افتح ملف class_count.csv من الخطوة 7.4. لكل ملف إدخال ، قسم الأعداد في مجتمع "ثنائي النواة مع MN" على الأعداد في السكان "ثنائي النواة" واضربه في 100:
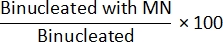
- طريقة غير Cyt-B: لتحديد تردد MN افتح ملف class_count.csv من الخطوة 7.4. لكل ملف إدخال ، قسم الأعداد في مجتمع "أحادي النواة مع MN" على الأعداد في المحتوى "أحادي النوى" واضربه في 100:
- حساب السمية الخلوية
- طريقة غير الخلايا البائية:
- باستخدام عدد الخلايا الأولي وعدد الخلايا بعد المعالجة ، احسب أولا مضاعفة عدد السكان (PD) لكل عينة2:
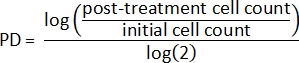
- بعد ذلك ، احسب تضاعف عدد السكان النسبي2:

- أخيرا ، احسب السمية الخلوية2 لكل عينة:
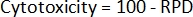
- باستخدام عدد الخلايا الأولي وعدد الخلايا بعد المعالجة ، احسب أولا مضاعفة عدد السكان (PD) لكل عينة2:
- طريقة الخلية البائية:
- لحساب مؤشر انتشار كتلة التحريك الخلوي (CBPI)2، استخدم الأعداد في الفئات أحادية النواة وثنائية النواة ومتعددة النوى لكل عينة من الملف class_count.csv :
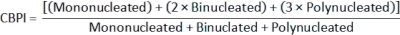
- لحساب السمية الخلوية2 ، استخدم CBPI من ثقافات التحكم (C) والثقافات المكشوفة (T):
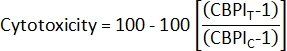
- لحساب مؤشر انتشار كتلة التحريك الخلوي (CBPI)2، استخدم الأعداد في الفئات أحادية النواة وثنائية النواة ومتعددة النوى لكل عينة من الملف class_count.csv :
- طريقة غير الخلايا البائية:
النتائج
يوضح الشكل 1 سير العمل لاستخدام برنامج الذكاء الاصطناعي لإنشاء نموذج لمقايسة MN. يقوم المستخدم بتحميل ملفات .daf المطلوبة في برنامج الذكاء الاصطناعي ، ثم يعين الكائنات إلى فئات نموذج الحقيقة الأرضية باستخدام المجموعة بمساعدة الذكاء الاصطناعي (الشكل 2) والتنب...
Discussion
يصف العمل المقدم هنا استخدام خوارزميات التعلم العميق لأتمتة تسجيل مقايسة MN. أظهرت العديد من المنشورات الحديثة أن الأدوات البديهية والتفاعلية تسمح بإنشاء نماذج التعلم العميق لتحليل بيانات الصور دون الحاجة إلى معرفة حسابية متعمقة18,19. تم تصميم البروتوكول ال...
Disclosures
يتم توظيف المؤلفين من قبل شركة Luminex Corporation ، وهي شركة DiaSorin ، الشركة المصنعة لمقياس التدفق الخلوي للتصوير ImageStream وبرنامج Amnis الذكاء الاصطناعي المستخدم في هذا العمل.
Acknowledgements
اي.
Materials
| Name | Company | Catalog Number | Comments |
| 15 mL centrifuge tube | Falcon | 352096 | |
| Cleanser - Coulter Clenz | Beckman Coulter | 8546931 | Fill container with 200 mL of Cleanser. https://www.beckmancoulter.com/wsrportal/page/itemDetails?itemNumber=8546931#2/10//0/25/ 1/0/asc/2/8546931///0/1//0/ |
| Colchicine | MilliporeSigma | 64-86-8 | |
| Corning bottle-top vacuum filter | MilliporeSigma | CLS430769 | 0.22 µm filter, 500 mL bottle |
| Cytochalasin B | MilliporeSigma | 14930-96-2 | 5 mg bottle |
| Debubbler - 70% Isopropanol | MilliporeSigma | 1.3704 | Fill container with 200 mL of Debubbler. http://www.emdmillipore.com/US/en/product/2-Propanol-70%25-%28V%2FV%29-0.1-%C2%B5m-filtred,MDA_CHEM-137040?ReferrerURL=https%3A%2F%2Fwww.google.com%2F |
| Dimethyl Sulfoxide (DMSO) | MilliporeSigma | 67-68-5 | |
| Dulbecco's Phosphate Buffered Saline 1X | EMD Millipore | BSS-1006-B | PBS Ca++MG++ Free |
| Fetal Bovine Serum | HyClone | SH30071.03 | |
| Formaldehyde, 10%, methanol free, Ultra Pure | Polysciences, Inc. | 04018 | This is what is used for the 4% and 1% Formalin. CAUTION: Formalin/Formaldehyde toxic by inhalation and if swallowed. Irritating to the eyes, respiratory systems and skin. May cause sensitization by inhalation or skin contact. Risk of serious damage to eyes. Potential cancer hazard. http://www.polysciences.com/default/catalog-products/life-sciences/histology-microscopy/fixatives/formaldehydes/formaldehyde-10-methanol-free-pure/ |
| Guava Muse Cell Analyzer | Luminex | 0500-3115 | A standard configuration Guava Muse Cell Analyzer was used. |
| Hoechst 33342 | Thermo Fisher | H3570 | 10 mg/mL solution |
| Mannitol | MilliporeSigma | 69-65-8 | |
| MEM Non-Essential Amino Acids 100X | HyClone | SH30238.01 | |
| MIFC - ImageStreamX Mark II | Luminex, a DiaSorin company | 100220 | A 2 camera ImageStreamX Mark II eqiped with the 405 nm, 488 nm, and 642 nm lasers was used. |
| MIFC analysis software - IDEAS | Luminex, a DiaSorin company | 100220 | "Image analysis sofware" The companion software to the MIFC (ImageStreamX MKII) |
| MIFC software - INSPIRE | Luminex, a DiaSorin company | 100220 | "Image acquisition software" This is the software that runs the MIFC (ImageStreamX MKII) |
| Amnis AI software | Luminex, a DiaSorin company | 100221 | "AI software" This is the software that permits the creation of artificial intelligence models to analyze data |
| Mitomycin C | MilliporeSigma | 50-07-7 | |
| NEAA Mixture 100x | Lonza BioWhittaker | 13-114E | |
| Penicllin/Streptomycin/Glutamine solution 100X | Gibco | 15070063 | |
| Potassium Chloride (KCl) | MilliporeSigma | P9541 | |
| Rinse - Ultrapure water or deionized water | NA | NA | Use any ultrapure water or deionized water. Fill container with 900 mL of Rinse. |
| RNase | MilliporeSigma | 9001-99-4 | |
| RPMI-1640 Medium 1x | HyClone | SH30027.01 | |
| Sheath - PBS | MilliporeSigma | BSS-1006-B | This is the same as Dulbecco's Phosphate Buffered Saline 1x Ca++MG++ free. Fill container with 900 mL of Sheath. |
| Sterile water | HyClone | SH30529.01 | |
| Sterilizer - 0.4%–0.7% Hypochlorite | VWR | JT9416-1 | This is assentually 10% Clorox bleach that can be made by deluting Clorox bleach with water. Fill container with 200 mL of Sterilzer. |
| T25 flask | Falcon | 353109 | |
| T75 flask | Falcon | 353136 | |
| TK6 cells | MilliporeSigma | 95111735 |
References
- Fenech, M., et al. HUMN project initiative and review of validation, quality control and prospects for further development of automated micronucleus assays using image cytometry systems. International Journal of Hygiene and Environmental Health. 216 (5), 541-552 (2013).
- OECD. Test No. 487: In Vitro Mammalian Cell Micronucleus Test. Section 4. OECD Guidelines for the Testing of Chemicals. , (2016).
- Fenech, M. The in vitro micronucleus technique. Mutation Research/Fundamental and Molecular Mechanisms of Mutagenesis. 455 (1), 81-95 (2000).
- Bonassi, S., et al. An increased micronucleus frequency in peripheral blood lymphocytes predicts the risk of cancer in humans. Carcinogenesis. 28 (3), 625-631 (2007).
- Fenech, M. Cytokinesis-block micronucleus cytome assay. Nature Protocols. 2 (5), 1084-1104 (2007).
- Fenech, M. Commentary on the SFTG international collaborative study on the in vitro micronucleus test: To Cyt-B or not to Cyt-B. Mutation Research/Fundamental and Molecular Mechanisms of Mutagenesis. 607 (1), 9-12 (2006).
- Seager, A. L., et al. Recommendations, evaluation and validation of a semi-automated, fluorescent-based scoring protocol for micronucleus testing in human cells. Mutagenesis. 29 (3), 155-164 (2014).
- Rossnerova, A., Spatova, M., Schunck, C., Sram, R. J. Automated scoring of lymphocyte micronuclei by the MetaSystems Metafer image cytometry system and its application in studies of human mutagen sensitivity and biodosimetry of genotoxin exposure. Mutagenesis. 26 (1), 169-175 (2011).
- Bryce, S. M., Bemis, J. C., Avlasevich, S. L., Dertinger, S. D. In vitro micronucleus assay scored by flow cytometry provides a comprehensive evaluation of cytogenetic damage and cytotoxicity. Mutation Research/Genetic Toxicology and Environmental Mutagenesis. 630 (1), 78-91 (2007).
- Avlasevich, S. L., Bryce, S. M., Cairns, S. E., Dertinger, S. D. In vitro micronucleus scoring by flow cytometry: Differential staining of micronuclei versus apoptotic and necrotic chromatin enhances assay reliability. Environmental and Molecular Mutagenesis. 47 (1), 56-66 (2006).
- Basiji, D. A. Principles of Amnis imaging flow cytometry. Methods in Molecular Biology. 1389, 13-21 (2016).
- Rodrigues, M. A. Automation of the in vitro micronucleus assay using the Imagestream® imaging flow cytometer. Cytometry Part A. 93 (7), 706-726 (2018).
- Verma, J. R., et al. Investigating FlowSight® imaging flow cytometry as a platform to assess chemically induced micronuclei using human lymphoblastoid cells in vitro. Mutagenesis. 33 (4), 283-289 (2018).
- Wills, J. W., et al. Inter-laboratory automation of the in vitro micronucleus assay using imaging flow cytometry and deep learning. Archives of Toxicology. 95 (9), 3101-3115 (2021).
- Rodrigues, M. A., et al. The in vitro micronucleus assay using imaging flow cytometry and deep learning. Npj Systems Biology and Applications. 7 (1), 20 (2021).
- Rodrigues, M. A. An automated method to perform the in vitro micronucleus assay using multispectral imaging flow cytometry. Journal of Visualized Experiments. (147), e59324 (2019).
- Lovell, D. P., et al. Analysis of negative historical control group data from the in vitro micronucleus assay using TK6 cells. Mutation Research/Genetic Toxicology and Environmental Mutagenesis. 825, 40-50 (2018).
- Berg, S., et al. ilastik: interactive machine learning for (bio)image analysis. Nature Methods. 16 (12), 1226-1232 (2019).
- Hennig, H., et al. An open-source solution for advanced imaging flow cytometry data analysis using machine learning. Methods. 112, 201-210 (2017).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved