JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
이미징 유세포 분석 및 인공 지능을 사용한 소핵 분석의 자동화
요약
소핵(MN) 분석은 DNA 손상을 정량화하기 위한 잘 확립된 검사입니다. 그러나 수동 현미경 또는 특징 기반 이미지 분석과 같은 기존 기술을 사용하여 분석에 점수를 매기는 것은 힘들고 어렵습니다. 이 논문은 이미징 유세포 분석 데이터를 사용하여 MN 분석의 점수를 매기는 인공 지능 모델을 개발하는 방법론을 설명합니다.
초록
소핵(MN) 분석은 유전 독성에 대한 화학 물질을 평가하기 위해 전 세계적으로 규제 기관에서 사용됩니다. 분석은 두 가지 방법으로 수행할 수 있습니다: 한 번 분할된 사이토카인시스 차단 이핵세포 또는 완전히 분할된 단핵구 세포에서 MN을 채점합니다. 역사적으로 광학 현미경은 분석에 점수를 매기는 황금 표준 방법이었지만 힘들고 주관적입니다. 유세포 분석은 최근 몇 년 동안 분석의 점수를 매기기 위해 사용되었지만 세포 이미지의 주요 측면을 시각적으로 확인할 수 없기 때문에 제한적입니다. 이미징 유세포 분석(IFC)은 고처리량 이미지 캡처와 자동화된 이미지 분석을 결합하며, MN 분석에서 모든 주요 이벤트의 이미지를 빠르게 획득하고 점수를 매기는 데 성공적으로 적용되었습니다. 최근에는 컨볼루션 신경망을 기반으로 하는 인공 지능(AI) 방법을 사용하여 IFC에서 획득한 MN 분석 데이터의 점수를 매길 수 있음이 입증되었습니다. 이 백서에서는 AI 소프트웨어를 사용하여 딥 러닝 모델을 만들어 모든 주요 이벤트에 점수를 매기고 이 모델을 적용하여 추가 데이터에 자동으로 점수를 매기는 모든 단계를 설명합니다. AI 딥 러닝 모델의 결과는 수동 현미경과 잘 비교되므로 IFC와 AI를 결합하여 MN 분석의 완전 자동 스코어링이 가능합니다.
서문
소핵(micronucleus, MN) 분석은 인체용 화장품, 의약품 및 화학물질 개발에서 DNA 손상을 평가하기 위한 유전독성학의 기본이다 1,2,3,4. 소핵은 분열 후 핵에 통합되지 않고 핵과 분리된 작은 원형체로 응축되는 전체 염색체 또는 염색체 조각으로 형성됩니다. 따라서, MN은 유전독성 시험1에서 DNA 손상을 정량화하기 위한 종말점으로 사용될 수 있다.
MN을 정량화하는 데 선호되는 방법은 Cyt-B(Cyt-B)를 사용하여 분열을 차단하여 일회성 분열 이핵 세포(BNC) 내에서 하는 것입니다. 이 버전의 분석에서 세포 독성은 단핵구(MONO) 및 다핵구(POLY) 세포의 점수를 매겨 평가됩니다. 이 분석은 또한 증식을 평가하기 위해 노출 전 및 노출 후 세포 수를 사용하여 세포 독성을 평가하여 더 빠르고 쉽게 점수를 매길 수 있는 차단되지 않은 MONO 세포에서 MN을 채점하여 수행할 수 있습니다 5,6.
분석의 물리적 채점은 역사적으로 수동 현미경을 통해 수행되어 왔으며, 이를 통해 모든 주요 이벤트를 시각적으로 확인할 수 있습니다. 그러나 수동 현미경 검사는 어렵고 주관적입니다1. 따라서 현미경 슬라이드 스캐닝 및 유세포 분석을 포함한 자동화 기술이 개발되었으며 각각 고유한 장점과 한계가 있습니다. 슬라이드 스캐닝 방법을 사용하면 주요 이벤트를 시각화할 수 있지만 슬라이드는 최적의 세포 밀도로 생성되어야 하므로 달성하기 어려울 수 있습니다. 또한, 이 기술은 종종 세포질 시각화가 부족하여 MONO 및 POLY 세포의 스코어링을 손상시킬 수 있습니다 7,8. 유세포 분석은 고처리량 데이터 캡처를 제공하지만 세포를 용해해야 하므로 Cyt-B 형태의 분석을 사용할 수 없습니다. 추가적으로, 비이미징 기술로서, 종래의 유세포 분석은 주요 이벤트(9,10)의 시각적 검증을 제공하지 않는다.
따라서, MN 분석을 수행하기 위해 이미징 유세포 분석법(IFC)이 조사되었다. ImageStreamX Mk II는 기존 유세포 분석의 속도 및 통계적 견고성과 현미경의 고분해능 이미징 기능을 단일 시스템11에 결합합니다. IFC를 사용함으로써, 모든 주요 이벤트들의 고해상도 이미지가 캡처될 수 있고, 특징-기반(12,13) 또는 인공지능(AI) 기술들(14,15)을 사용하여 자동으로 스코어링될 수 있다는 것이 보여졌다. IFC를 사용하여 MN 분석을 수행함으로써 더 짧은 시간 내에 현미경에 비해 더 많은 세포의 자동 스코어링을 달성할 수 있습니다.
이 작업은 이전에 설명된 이미지 분석 워크플로우(16 )에서 벗어나 Amnis AI 소프트웨어(이하 "AI 소프트웨어"로 지칭됨)를 사용하여 랜덤 포레스트(RF) 및/또는 컨볼루션 신경망(CNN) 모델을 개발하고 훈련하는 데 필요한 모든 단계를 논의합니다. AI 지원 태깅 도구를 사용하여 실측 데이터를 채우고, 모델 학습 결과를 해석하고, 추가 데이터를 분류하기 위한 모델 적용, 유전독성 및 세포독성계산 허용 등 필요한 모든 단계를 설명합니다 15.
프로토콜
1. 이미징 유세포 분석을 이용한 데이터 수집
참고: 최적의 이미지 캡처를 위해 IFC를 사용하는 획득 영역을 수정해야 할 수도 있다는 점에 유의하여 다음과 같이 수정한 Rodrigues et al.16 을 참조하십시오.
- non-Cyt-B 방법의 경우, 배양 직전 및 회수 기간 직후의 각 배양물에 대해 제조업체의 지침( 재료 표 참조)에 따라 시판되는 세포 계수기를 사용하여 세포 계수를 수행합니다.
- 단일 카메라 이미징 유세포분석기에서 샘플을 실행하는 경우 명시야(BF)를 채널 4에 배치합니다. M01을 M04로, M07을 M01로 바꿉니다.
알림: "M"은 IFC의 카메라 채널을 나타냅니다. - 획득 중에 40x 배율을 사용합니다.
- 획득 중 BF 영역 대 BF 종횡비 플롯에서 다음 영역 좌표를 사용합니다.
X 좌표 : 100 및 900; Y 좌표: 0.7 및 1(Cyt-B 방법)
X 좌표: 100 및 600; Y 좌표: 0.7 및 1 - Hoechst 강도 그림에서 다음 영역 좌표를 사용합니다.
X 좌표 : 55 및 75; Y 좌표: 9.5 및 15(Cyt-B 방법)
X 좌표 : 55 및 75; Y 좌표: 13 및 21(비 Cyt-B 방법) - 데이터에서 세포자멸사 및 괴사 물체의 이미지를 제거하려면 IDEAS 6.3 소프트웨어 패키지(이하 "이미지 분석 소프트웨어"라고 함, 재료 표 참조)를 시작합니다.
참고: AI 소프트웨어는 최신 버전의 이미지 분석 소프트웨어를 사용하여 처리된 .daf 파일과 함께 작동하도록 설계되었습니다. 이미지 분석 소프트웨어가 최신 버전인지 확인합니다. - 이 작업을 서식 파일(.ast)로 저장합니다.
2. 모든 .rif 파일에 대한 .daf 파일 만들기
- AI 소프트웨어는 .daf 파일 가져오기만 허용합니다. 일괄 처리를 통해 실험의 모든 .rif 파일에 대한 .daf 파일을 만듭니다.
- Tools( 도구 ) 메뉴에서 Batch Data Files(배치 데이터 파일 )를 클릭한 다음 Add Batch(배치 추가)를 클릭합니다.
- 새 창에서 파일 추가를 선택하고 배치에 추가할 .rif 파일을 선택합니다. 템플릿 또는 데이터 분석 파일(.ast, .daf) 선택 옵션에서 이전에 만든 .ast 파일을 선택합니다.
- 필요한 경우 배치 이름을 지정하고 확인을 클릭하여 로드된 모든 .rif 파일에 대해 .daf 파일을 만듭니다.
3. AI 소프트웨어에서 실험 만들기
- AI 소프트웨어를 사용하여 딥러닝 모델을 생성하는 과정을 설명하는 그림 1 의 순서도를 참조하십시오.
- AI 소프트웨어를 실행하고 창의 왼쪽 하단 모서리에 있는 정보를 클릭하여 최신 버전이 설치되어 있는지 확인합니다. 최신 버전이 설치되어 있지 않으면 support@luminexcorp.com 문의하여 구하십시오.
- 소프트웨어의 기본 화면은 새 실험 화면입니다. 폴더 아이콘을 사용하여 실험을 저장할 위치를 선택하고 실험 이름(예: "MN 모델")을 입력합니다.
- 실험 유형에서 학습 실험 옆에 있는 라디오 단추를 클릭하여 CNN 모델 빌드를 시작합니다. 다음을 클릭합니다.
- 선택 사항: 모델이 이전에 학습된 경우 새 AI 모델의 템플릿으로 사용할 수 있으며 템플릿 모델 선택 화면에서 새 모델을 만들기 위한 템플릿으로 선택할 수 있습니다. 템플릿 모델이 없으면 다음을 클릭하여 이 단계를 건너뜁니다.
- 다음 화면은 새 모델 정의(Define New Model ) 화면입니다. 모델 아래에는 3.3단계에서 모델에 지정된 이름이 자동으로 채워집니다.
- 설명에서 모델에 대한 설명(선택 사항)을 입력하고 최대 이미지 크기를 150픽셀로 유지합니다.
- 채널에서 BF 추가를 클릭하여 명시야 채널을 목록에 추가합니다. 이름에서 Brightfield를 두 번 클릭하고 이 채널의 이름을 BF로 바꿉니다. Add FL(FL 추가)을 클릭하여 목록에 형광 채널을 추가합니다. 이름에서 형광등을 두 번 클릭하고 이 채널의 이름을 DNA로 바꿉니다.
- Class Names(클래스 이름)에서 Add(추가)를 클릭합니다. 팝업 창에서 Mononucleated를 입력하고 확인을 클릭합니다. 이렇게 하면 단핵화된 클래스가 클래스 이름 목록에 추가됩니다. 이 프로세스를 반복하여 다음 6개의 클래스가 목록에 정의되어 있는지 확인합니다.
단핵성
MN으로 단핵화
이핵화
MN으로 이중화
다핵(Polynucleated)
불규칙한 형태
다음을 클릭합니다.
참고: 이 6개의 실측 모델 클래스는 채점할 주요 이벤트와 허용된 채점 기준5와 다른 형태의 이미지를 나타냅니다.- 선택 사항: 원하는 경우 1.7의 분석 템플릿을 포함하여 이미지 분석 소프트웨어의 기능을 사용할 수 있습니다. AI 모델에 이러한 기능을 포함하려면 .ast 파일을 찾은 다음 채널별 드롭다운에서 포함할 기능 하위 집합을 선택합니다.
- 파일 선택에서 파일 추가를 클릭하고 AI 소프트웨어에 추가할 원하는 파일을 찾아 실측 데이터를 구축합니다. 다음을 클릭합니다.
참고: 충분한 수의 모든 주요 이벤트를 포함하는 여러 데이터 파일(예: 양성 및 음성 대조군 데이터)을 추가하는 것이 중요합니다.
- 다음으로, Select Base Populations(기본 모집단 선택) 화면에서 모집단 계층에서 Non-Apoptotic 모집단을 찾습니다. Non-Apoptotic 모집단을 마우스 오른쪽 버튼으로 클릭하고 Select All Matching Populations를 선택합니다. 다음을 클릭합니다.
참고: 분류해서는 안 되는 개체군(예: 구슬, 파편, 이중선 등)을 제외하는 것이 중요합니다. - 이 화면은 진실 모집단 선택 화면입니다.
- 이미지 분석 소프트웨어에서 주요 이벤트의 태그가 지정된 진실 모집단이 생성되지 않은 경우 Next(다음)를 클릭합니다.
- 이미지 분석 소프트웨어에서 태그가 지정된 진리 모집단이 생성된 경우 적절한 모델 클래스에 할당합니다.
- MN을 사용하여 MONO 세포의 태그가 지정된 진리 집단을 할당하려면 왼쪽의 Model Classes(모델 클래스)에서 Mononucleated with MN(MN으로 단핵화됨) 클래스를 클릭합니다. 그런 다음 이러한 이벤트가 포함된 오른쪽의 적절한 태그가 지정된 진실 모집단을 클릭합니다.
- 태그가 지정된 진리 집단이 둘 이상의 데이터 파일에서 생성된 경우 진리 집단 중 하나를 마우스 오른쪽 버튼으로 클릭하고 일치하는 모든 모집단 선택을 선택하여 여러 파일의 태그가 지정된 모집단을 적절한 클래스에 추가합니다.
- 모든 적절한 진리 집단이 할당되면 다음을 클릭합니다.
- 채널 선택 화면에서 실험에 적합한 채널을 선택합니다. 여기에서 BF를 채널 1로, Hoechst를 채널 7로 설정합니다. 채널을 마우스 오른쪽 버튼으로 클릭하고 Apply to All(모두 적용)을 선택합니다. 다음을 클릭합니다.
- 마지막으로 확인 화면에서 실험 만들기를 클릭합니다.
- AI 소프트웨어는 데이터 파일에서 이미지를 로드하고 3.7단계에서 할당된 실측 영상을 사용하여 3.5.3단계에서 정의된 모델 클래스를 생성합니다. Finish( 마침)를 클릭합니다.
- 실험이 만들어지면 다음과 같은 5가지 옵션이 제공됩니다.
실험: 로드된 데이터 파일, 선택한 채널, 정의된 실측 자료 모델 클래스를 포함하여 실험의 세부 정보를 제공합니다.
태깅: 사용자가 실측 데이터를 채울 수 있는 태깅 도구를 시작합니다.
학습: 실측 데이터를 기반으로 모델을 학습합니다.
분류: 학습된 모델을 사용하여 데이터를 분류합니다.
결과: 학습 실험과 분류 실험의 결과를 모두 제공합니다.
4. AI 지원 태깅 도구를 사용하여 실측 자료 채우기
- Tagging (태깅)을 클릭하여 태깅 도구 인터페이스를 시작합니다.
- 확대/축소 도구(돋보기 아이콘)를 클릭하여 더 쉽게 볼 수 있도록 이미지를 자릅니다.
- 슬라이더 막대를 클릭하여 이미지 크기를 조정하여 갤러리에 표시되는 이미지 수를 변경합니다.
- 디스플레이 설정 옵션을 클릭하고 모든 주요 이벤트를 식별하는 데 가장 적합한 대비 이미지를 제공하는 Min-Max를 선택합니다.
- 갤러리 디스플레이 설정을 클릭하여 DNA 이미지의 색상을 노란색 또는 흰색으로 변경하면 작은 물체(예: MN)의 시각화가 향상됩니다.
- Cluster( 클러스터 )를 클릭하여 유사한 형태의 객체를 함께 그룹화하는 알고리즘을 실행합니다. 클러스터링이 완료되면 클러스터당 개체 수가 있는 개별 클러스터가 알 수 없는 모집단(Unknown Populations) 아래의 목록에 표시됩니다. 개별 군집을 선택하여 군집 내의 객체를 보고 이러한 객체를 적절한 모델 클래스에 지정합니다.
- 각 모델 클래스에 최소 25개의 개체가 할당되면 예측 알고리즘을 사용할 수 있게 됩니다. 예측을 클릭합니다.
참고: 어떤 모집단에도 잘 맞지 않는 개체는 알 수 없음으로 분류됩니다. 더 많은 객체가 진리 모집단에 추가될수록 예측 정확도가 향상됩니다. - 각 클래스의 충분한 개체 수에 도달할 때까지 Ground Truth Model 클래스를 적절한 이미지로 계속 채웁니다.
- 각 모델 클래스에 최소 100개의 개체가 할당되면 화면 상단의 학습 탭을 클릭합니다. 학습(Train ) 버튼을 클릭하여 랜덤 포레스트(Random Forest) 및 CNN 알고리즘을 사용하여 모델을 생성합니다.
참고: AI 소프트웨어는 랜덤 포레스트와 CNN 알고리즘을 모두 사용하여 모델을 생성하며, 확인란을 사용하면 CNN 알고리즘의 랜덤 포레스트만 사용하여 모델을 생성할 수 있습니다.
5. 모델 정확도 평가
- 모델 학습이 완료되면 결과 보기를 클릭합니다.
- 결과 화면을 사용하여 모델 정확도를 평가합니다. 풀다운 메뉴를 사용하여 랜덤 포레스트와 CNN 사이를 전환합니다.
참고: 진리 모집단을 업데이트할 수 있으며, 모델을 다시 훈련하거나 있는 그대로 사용하여 추가 데이터를 분류할 수 있습니다.- 진실 모집단을 업데이트하려면 상단의 태그 지정 을 클릭하고 섹션 4를 따르십시오.
6. 모델을 이용한 데이터 분류
- AI 소프트웨어를 실행합니다. 기본 화면은 새 실험 화면입니다. 폴더 아이콘을 사용하여 실험을 저장할 위치를 선택하고 실험의 이름을 입력합니다.
- 실험 유형에서 분류 옆에 있는 라디오 버튼을 클릭하여 분류 실험을 시작합니다. 다음을 클릭합니다.
- 분류에 사용할 모델을 클릭한 후 Next(다음)를 클릭합니다.
- 파일 선택 화면에서 파일 추가를 클릭하고 CNN 모델로 분류할 파일을 찾습니다. 다음을 클릭합니다.
- 그런 다음 Select Base Populations 화면에서 로드된 파일 중 하나에서 Non-Apoptotic 모집단 옆에 있는 확인란을 클릭합니다. Non-Apoptotic 모집단을 마우스 오른쪽 버튼으로 클릭하고 Select All Matching Populations 를 클릭하여 로드된 모든 파일에서 이 모집단을 선택합니다. 다음을 클릭합니다.
- 선택 사항: 분류할 데이터에 진리 집단이 포함된 경우 진리 모집단 선택 화면에서 적절한 모델 클래스에 지정할 수 있습니다. 그렇지 않으면 다음을 클릭하여 이 단계를 건너뜁니다.
- 채널 선택 화면에서 명시야의 경우 채널 1을 선택하고 DNA 염색의 경우 채널 7을 선택합니다. 채널을 마우스 오른쪽 버튼으로 클릭하고 Apply to All(모두 적용)을 클릭합니다. 그런 다음 다음을 클릭합니다.
- 마지막으로 확인 화면에서 실험 만들기를 클릭합니다. AI 소프트웨어는 선택한 데이터 파일에서 선택한 모델과 모든 이미지를 로드합니다. Finish( 마침)를 클릭합니다.
- Classify를 클릭하여 분류 화면을 시작합니다. 분류 버튼을 클릭합니다. 이렇게 하면 RF 및 CNN 모델을 사용하여 추가 데이터를 분류하고 지정된 모델 클래스에 속하는 모든 객체를 식별하는 프로세스가 시작됩니다.
알림: 확인란을 사용하여 RF 모델 및/또는 CNN 모델을 선택할 수 있습니다. - 분류가 완료되면 결과 보기를 클릭합니다.
- Update DAFs 버튼을 클릭하여 Update DAFs with Classification Results(분류 결과로 DAF 업데이트 ) 창을 표시합니다. 확인을 클릭하여 .daf 파일을 업데이트합니다.
7. 분류 결과 보고서 생성
- Results( 결과 ) 화면에서 Generate Report(보고서 생성)를 클릭합니다. 각 입력 DAF에 대한 개별 보고서가 필요한 경우 각 입력 DAF에 대한 보고서 만들기 옆의 확인란을 선택합니다. 확인을 클릭합니다.
- 완료되면 보고서 파일이 저장된 폴더를 엽니다. 폴더 내에는 실험 .pdf 보고서와 리소스 폴더가 있습니다.
- .pdf 열어 보고서를 봅니다. 이 보고서에는 모형 및 실험 정보, 입력 .daf 파일 목록, 표 형식 및 히스토그램 형식의 클래스 개수 및 클래스 백분율, 모든 입력 .daf 파일의 예측 확률 중앙값을 요약하는 혼동행렬이 포함되어 있습니다.
- Resources 폴더를 연 다음 CNN 폴더를 엽니다. 이 폴더 안에는 클래스 개수 및 백분율 막대 그래프의 .png 파일과 혼동 행렬이 있습니다. 또한 각 입력 파일에 대한 클래스 개수 및 백분율을 포함하는 .csv 파일이 있습니다.
8. MN 빈도 및 세포 독성 결정
- MN 주파수 계산
- Non Cyt-B 방법: MN 빈도를 확인하려면 7.4단계의 class_count.csv 파일을 엽니다. 각 입력 파일에 대해 "MN으로 단핵화된" 모집단의 카운트를 "모노핵화된" 모집단의 카운트로 나누고 100을 곱합니다.

- Cyt-B 방법: MN 주파수를 확인하려면 7.4단계의 class_count.csv 파일을 엽니다. 각 입력 파일에 대해 "MN으로 이중화된" 모집단의 개수를 "이분자형" 모집단의 개수로 나누고 100을 곱합니다.
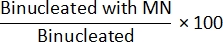
- Non Cyt-B 방법: MN 빈도를 확인하려면 7.4단계의 class_count.csv 파일을 엽니다. 각 입력 파일에 대해 "MN으로 단핵화된" 모집단의 카운트를 "모노핵화된" 모집단의 카운트로 나누고 100을 곱합니다.
- 세포 독성 계산
- 비 Cyt-B 방법:
- 초기 세포 계수 및 처리 후 세포 계수를 사용하여 먼저 각 샘플 2에 대한 모집단 배가(PD)를 계산합니다.
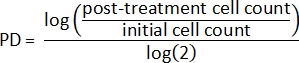
- 다음으로, 상대 모집단을 두 배로 늘리는2를 계산합니다.

- 마지막으로, 각 샘플에 대한 세포독성2 을 계산한다.
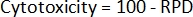
- 초기 세포 계수 및 처리 후 세포 계수를 사용하여 먼저 각 샘플 2에 대한 모집단 배가(PD)를 계산합니다.
- Cyt-B 방법:
- CBPI(Cytokinesis-Block Proliferation Index)2를 계산하려면 class_count.csv 파일의 각 샘플에 대해 단핵, 이핵화 및 다핵화 클래스의 카운트를 사용합니다.
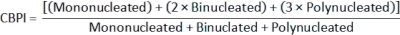
- 세포독성2를 계산하려면 대조군 배양(C)과 노출된 배양(T)의 CBPI를 사용합니다.
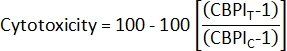
- CBPI(Cytokinesis-Block Proliferation Index)2를 계산하려면 class_count.csv 파일의 각 샘플에 대해 단핵, 이핵화 및 다핵화 클래스의 카운트를 사용합니다.
- 비 Cyt-B 방법:
결과
그림 1은 AI 소프트웨어를 사용하여 MN 분석을 위한 모델을 생성하는 워크플로우를 보여줍니다. 사용자는 원하는 .daf 파일을 AI 소프트웨어에 로드한 다음 AI 지원 클러스터(그림 2) 및 예측(그림 3) 태깅 알고리즘을 사용하여 실측 자료 모델 클래스에 객체를 할당합니다. 모든 실측 모델 클래스가 충분한 객체로 채워지면 RF 또는 CNN ?...
토론
여기에 제시된 작업은 딥 러닝 알고리즘을 사용하여 MN 분석의 스코어링을 자동화하는 방법을 설명합니다. 몇몇 최근의 간행물들은 직관적이고, 상호작용적인 도구들이 심층적인 계산 지식(18,19)에 대한 필요 없이 이미지 데이터를 분석하기 위한 딥 러닝 모델들의 생성들을 허용한다는 것을 보여주었다. 사용자 인터페이스 기반 소프트웨어 패키지를 사...
공개
저자는 DiaSorin Company인 Luminex Corporation, ImageStream 이미징 유세포 분석기 및 이 작업에 사용된 Amnis AI 소프트웨어 제조업체에 고용되어 있습니다.
감사의 말
없음.
자료
| Name | Company | Catalog Number | Comments |
| 15 mL centrifuge tube | Falcon | 352096 | |
| Cleanser - Coulter Clenz | Beckman Coulter | 8546931 | Fill container with 200 mL of Cleanser. https://www.beckmancoulter.com/wsrportal/page/itemDetails?itemNumber=8546931#2/10//0/25/ 1/0/asc/2/8546931///0/1//0/ |
| Colchicine | MilliporeSigma | 64-86-8 | |
| Corning bottle-top vacuum filter | MilliporeSigma | CLS430769 | 0.22 µm filter, 500 mL bottle |
| Cytochalasin B | MilliporeSigma | 14930-96-2 | 5 mg bottle |
| Debubbler - 70% Isopropanol | MilliporeSigma | 1.3704 | Fill container with 200 mL of Debubbler. http://www.emdmillipore.com/US/en/product/2-Propanol-70%25-%28V%2FV%29-0.1-%C2%B5m-filtred,MDA_CHEM-137040?ReferrerURL=https%3A%2F%2Fwww.google.com%2F |
| Dimethyl Sulfoxide (DMSO) | MilliporeSigma | 67-68-5 | |
| Dulbecco's Phosphate Buffered Saline 1X | EMD Millipore | BSS-1006-B | PBS Ca++MG++ Free |
| Fetal Bovine Serum | HyClone | SH30071.03 | |
| Formaldehyde, 10%, methanol free, Ultra Pure | Polysciences, Inc. | 04018 | This is what is used for the 4% and 1% Formalin. CAUTION: Formalin/Formaldehyde toxic by inhalation and if swallowed. Irritating to the eyes, respiratory systems and skin. May cause sensitization by inhalation or skin contact. Risk of serious damage to eyes. Potential cancer hazard. http://www.polysciences.com/default/catalog-products/life-sciences/histology-microscopy/fixatives/formaldehydes/formaldehyde-10-methanol-free-pure/ |
| Guava Muse Cell Analyzer | Luminex | 0500-3115 | A standard configuration Guava Muse Cell Analyzer was used. |
| Hoechst 33342 | Thermo Fisher | H3570 | 10 mg/mL solution |
| Mannitol | MilliporeSigma | 69-65-8 | |
| MEM Non-Essential Amino Acids 100X | HyClone | SH30238.01 | |
| MIFC - ImageStreamX Mark II | Luminex, a DiaSorin company | 100220 | A 2 camera ImageStreamX Mark II eqiped with the 405 nm, 488 nm, and 642 nm lasers was used. |
| MIFC analysis software - IDEAS | Luminex, a DiaSorin company | 100220 | "Image analysis sofware" The companion software to the MIFC (ImageStreamX MKII) |
| MIFC software - INSPIRE | Luminex, a DiaSorin company | 100220 | "Image acquisition software" This is the software that runs the MIFC (ImageStreamX MKII) |
| Amnis AI software | Luminex, a DiaSorin company | 100221 | "AI software" This is the software that permits the creation of artificial intelligence models to analyze data |
| Mitomycin C | MilliporeSigma | 50-07-7 | |
| NEAA Mixture 100x | Lonza BioWhittaker | 13-114E | |
| Penicllin/Streptomycin/Glutamine solution 100X | Gibco | 15070063 | |
| Potassium Chloride (KCl) | MilliporeSigma | P9541 | |
| Rinse - Ultrapure water or deionized water | NA | NA | Use any ultrapure water or deionized water. Fill container with 900 mL of Rinse. |
| RNase | MilliporeSigma | 9001-99-4 | |
| RPMI-1640 Medium 1x | HyClone | SH30027.01 | |
| Sheath - PBS | MilliporeSigma | BSS-1006-B | This is the same as Dulbecco's Phosphate Buffered Saline 1x Ca++MG++ free. Fill container with 900 mL of Sheath. |
| Sterile water | HyClone | SH30529.01 | |
| Sterilizer - 0.4%–0.7% Hypochlorite | VWR | JT9416-1 | This is assentually 10% Clorox bleach that can be made by deluting Clorox bleach with water. Fill container with 200 mL of Sterilzer. |
| T25 flask | Falcon | 353109 | |
| T75 flask | Falcon | 353136 | |
| TK6 cells | MilliporeSigma | 95111735 |
참고문헌
- Fenech, M., et al. HUMN project initiative and review of validation, quality control and prospects for further development of automated micronucleus assays using image cytometry systems. International Journal of Hygiene and Environmental Health. 216 (5), 541-552 (2013).
- OECD. Test No. 487: In Vitro Mammalian Cell Micronucleus Test. Section 4. OECD Guidelines for the Testing of Chemicals. , (2016).
- Fenech, M. The in vitro micronucleus technique. Mutation Research/Fundamental and Molecular Mechanisms of Mutagenesis. 455 (1), 81-95 (2000).
- Bonassi, S., et al. An increased micronucleus frequency in peripheral blood lymphocytes predicts the risk of cancer in humans. Carcinogenesis. 28 (3), 625-631 (2007).
- Fenech, M. Cytokinesis-block micronucleus cytome assay. Nature Protocols. 2 (5), 1084-1104 (2007).
- Fenech, M. Commentary on the SFTG international collaborative study on the in vitro micronucleus test: To Cyt-B or not to Cyt-B. Mutation Research/Fundamental and Molecular Mechanisms of Mutagenesis. 607 (1), 9-12 (2006).
- Seager, A. L., et al. Recommendations, evaluation and validation of a semi-automated, fluorescent-based scoring protocol for micronucleus testing in human cells. Mutagenesis. 29 (3), 155-164 (2014).
- Rossnerova, A., Spatova, M., Schunck, C., Sram, R. J. Automated scoring of lymphocyte micronuclei by the MetaSystems Metafer image cytometry system and its application in studies of human mutagen sensitivity and biodosimetry of genotoxin exposure. Mutagenesis. 26 (1), 169-175 (2011).
- Bryce, S. M., Bemis, J. C., Avlasevich, S. L., Dertinger, S. D. In vitro micronucleus assay scored by flow cytometry provides a comprehensive evaluation of cytogenetic damage and cytotoxicity. Mutation Research/Genetic Toxicology and Environmental Mutagenesis. 630 (1), 78-91 (2007).
- Avlasevich, S. L., Bryce, S. M., Cairns, S. E., Dertinger, S. D. In vitro micronucleus scoring by flow cytometry: Differential staining of micronuclei versus apoptotic and necrotic chromatin enhances assay reliability. Environmental and Molecular Mutagenesis. 47 (1), 56-66 (2006).
- Basiji, D. A. Principles of Amnis imaging flow cytometry. Methods in Molecular Biology. 1389, 13-21 (2016).
- Rodrigues, M. A. Automation of the in vitro micronucleus assay using the Imagestream® imaging flow cytometer. Cytometry Part A. 93 (7), 706-726 (2018).
- Verma, J. R., et al. Investigating FlowSight® imaging flow cytometry as a platform to assess chemically induced micronuclei using human lymphoblastoid cells in vitro. Mutagenesis. 33 (4), 283-289 (2018).
- Wills, J. W., et al. Inter-laboratory automation of the in vitro micronucleus assay using imaging flow cytometry and deep learning. Archives of Toxicology. 95 (9), 3101-3115 (2021).
- Rodrigues, M. A., et al. The in vitro micronucleus assay using imaging flow cytometry and deep learning. Npj Systems Biology and Applications. 7 (1), 20 (2021).
- Rodrigues, M. A. An automated method to perform the in vitro micronucleus assay using multispectral imaging flow cytometry. Journal of Visualized Experiments. (147), e59324 (2019).
- Lovell, D. P., et al. Analysis of negative historical control group data from the in vitro micronucleus assay using TK6 cells. Mutation Research/Genetic Toxicology and Environmental Mutagenesis. 825, 40-50 (2018).
- Berg, S., et al. ilastik: interactive machine learning for (bio)image analysis. Nature Methods. 16 (12), 1226-1232 (2019).
- Hennig, H., et al. An open-source solution for advanced imaging flow cytometry data analysis using machine learning. Methods. 112, 201-210 (2017).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유